È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Pull-down di Calmodulina proteine leganti
In questo articolo
Riepilogo
Calmodulina (CaM) pull-down saggio è un modo efficace per studiare l'interazione del CAM con varie proteine. Questo metodo utilizza CaM-sefarosio perle per l'analisi efficiente e specifica di CaM proteine leganti. Questo fornisce un importante strumento per esplorare segnalazione CaM in funzione cellulare.
Abstract
Calcio (Ca 2 +) è uno ione fondamentale nella regolazione della funzione cellulare attraverso una varietà di meccanismi. Gran parte del Ca 2 + segnalazione è mediato attraverso la proteina legante il calcio conosciuto come calmodulina (CaM) 1,2. CaM è coinvolta a più livelli in quasi tutti i processi cellulari, tra cui l'apoptosi, il metabolismo, la contrazione della muscolatura liscia, la plasticità sinaptica, la crescita dei nervi, l'infiammazione e la risposta immunitaria. Un certo numero di proteine aiutano a regolare queste vie attraverso la loro interazione con la CaM. Molte di queste interazioni dipende dalla conformazione della CAM, che è nettamente diverso quando si lega al Ca 2 + (Ca 2 +-Cam), in opposizione alla sua Ca 2 + libero stato (ApoCaM) 3.
Mentre la maggior parte delle proteine bersaglio legano Ca 2 +-CAM, solo alcune proteine si legano ai ApoCaM. Alcuni CaM si legano attraverso il loro IQ-dominio, tra cui neuromodulin 4, neurogranin (Ng) 5, e alcuni miosine 7, funzione post-sinaptici 8, e la contrazione muscolare 9, rispettivamente. La loro capacità di legare e rilasciare CaM in assenza o in presenza di Ca 2 + è fondamentale nella loro funzione. Al contrario, molte proteine si legano solo Ca 2 +-CAM e necessitano di questo legame per la loro attivazione. Gli esempi includono miosina chinasi della catena leggera 10, Ca 2 + / CaM chinasi-dipendenti (CaMKs) 11 e fosfatasi (ad esempio calcineurina) 12, e spettrina chinasi 13, che hanno una varietà di effetti diretti ed a valle 14.
Gli effetti di queste proteine sulla funzione cellulare dipendono spesso dalla loro capacità di legarsi al CaM in Ca 2 +-dipendente. Per esempio, abbiamo testato la rilevanza di Ng-CAM obbligatorio in funzione sinaptica e di come diverse mutazioni influenzano questa associazione. Abbiamo generato un GFP-tagged con Ngstruct con mutazioni specifiche nel IQ-dominio che avrebbe cambiato la capacità di Ng di legare CaM in Ca 2 +-dipendente. Lo studio di queste mutazioni diverse ci ha dato grande intuizione in importanti processi coinvolti nella funzione sinaptica 8,15. Tuttavia, in tali studi, è essenziale per dimostrare che le proteine mutate hanno atteso il legame alterato per CAM.
Qui vi presentiamo un metodo per testare la capacità delle proteine di legarsi al CaM in presenza o in assenza di Ca 2 +, con CaMKII e Ng come esempi. Questo metodo è una forma di cromatografia di affinità indicato come un pull-down assay CaM. Utilizza CaM-Sepharose perline per testare le proteine che si legano CaM e l'influenza di Ca 2 + su questa associazione. E 'molto più tempo efficiente e richiede meno proteine rispetto alla cromatografia su colonna, ed altri test. Complessivamente, questo fornisce un valido strumento per esplorare Ca 2 + / CaM e proteine di segnalazione che interact con CaM.
Protocollo
Fare riferimento alla Figura 1 per uno schema di base della procedura a partire con l'omogeneizzato. Tempo stimato dalla preparazione di estratti cellulari a eluizione di Cam-bound proteine è di circa 6-7 ore.
1. Preparazione del tessuto
- Iniettare organotipica fettine di ippocampo con un virus che contiene un plasmide che esprime la proteina ricombinante di interesse (in questo esempio, proteina fluorescente verde (GFP)-tagged Ng) e permettono di esprimere la proteina dei tessuti durante la notte.
- Circa 12 a 18 ore dopo l'iniezione virale (a seconda del tempo di espressione virale), si preparano a raccogliere i tessuti. Aggiungere 1 ml di buffer dissezione (glucosio 10mM, KCl 4 mm, 26mm NaHCO3, saccarosio 233mM, 5mm MgCl 2, 1 mM CaCl 2, e 0,1% di fenolo-rosso, gasati con il 5% di CO 2 95% O 2) a una piastra di Petri. Trasferimento dei tessuti in coltura / inserto a Petri e aggiungere 2 ml di buffer dissezione, per l'inserto di immergere il tessuto.
- Raccoltat organotipica tessuto dell'ippocampo (tra 5 e 10 fette) con delicatezza raschiando il tessuto libero della membrana inserire utilizzando un bisturi. In particolare la rimozione di una particolare regione di interesse (ad esempio CA1) è anche un'opzione. Trasferire la sospensione di tessuto in una provetta da microcentrifuga 1,5 ml utilizzando una pipetta Pasteur invertita.
- Centrifugare i campioni a 1.500 rcf per 1 min per separare i tessuti dal buffer dissezione. Rimuovere con attenzione il surnatante tramite aspirazione. Assicurarsi di non disturbare il pellet.
- Per ogni fetta utilizzato, aggiungere 30-60 ml di buffer di omogeneizzazione (150mm NaCl, 20 mM Tris pH 7.5, 1mM DTT, 1μg/mL leupeptina, 1μg/mL chemostatin, 1μg/mL antidolore, pepstatina 1μg/mL, e l'1% Triton X -100) al tessuto e omogeneizzare accuratamente con pestello.
- Al fine di rimuovere i detriti cellulari, centrifugare la omogenato restante a 1.100 rcf per 10 minuti e rimuovere con attenzione il surnatante tramite aspirazione evitando la contaminazione da pellet.
- Prendere il 10% del supernatante come un campione di input (esempio 1). Conservare il surnatante rimanente sul ghiaccio durante la preparazione del CAM-sefarosio perle per l'utilizzo al punto 3.
Nota: Il tessuto usato qui sono organotipica fettine di ippocampo. Tuttavia, si potrebbero usare i neuroni dissociati o qualsiasi altro sistema di coltura cellulare. In tal caso, iniziare a passo 1,4 dopo aver raccolto il fazzoletto di carta nel modo appropriato.
2. Preparazione di perline per pull-down
Nel maneggiare le perline, è importante per salvare le perline e massimizzare l'efficienza delle reazioni, impedendo le perline si secchi ai lati del tubo. Per fare questo, è meglio ruotare il tubo dalla loro parte, permettendo la soluzione per bagnare le perline sulle pareti del tubo, immediatamente prima della centrifugazione.
- Per ogni pull-down, pipetta 400 ml di sospensione Calmodulina-Sepharose perle in una provetta 2 mL con un relativamente flat-basso per massimizzare la superficie e l'interazione delle soluzioni con le perline durante il vostro incubazione.
- Centrifugare perline a 21.000 rcf per 30 secondi e rimuovere con attenzione il surnatante tramite aspirazione. Assicurarsi di non disturbare le perline.
- Per lavare le perline, aggiungere 100 ml di buffer di omogeneizzazione rispettivi contenente 2 mM CaCl 2 a quelli utilizzati per pull-down Ca 2 +-CaM proteine di legame e 2 mM EDTA (un chelante del noto Ca 2 +) per perline abbattimento proteine ApoCaM vincolanti. Battere delicatamente la provetta per risospendere perline e centrifugare a 1500 rcf per 1 min. Rimuovere con attenzione il surnatante tramite aspirazione, facendo attenzione a non disturbare le perline.
Nota: per tutte le fasi di aspirazione, si raccomanda di usare un puntale che ha una apertura multa (ad esempio punte di carico gel) per permettere la rimozione della soluzione senza rimuovere perline.
3. CaM-sefarosio legame delle proteine
- Dividere il surnatante in due condizioni che contiene un uguale volume. A seconda del vostro stato, aggiungere la giusta quantità di CaCl 2 o EDTA al surnatante fino a 2 mm per ogni concentrazione.
- Aggiungi surnatante dal passo 1,7 a perline lavati in buffer di omogeneizzazione corrispondente. Battere delicatamente il tubo per mescolare.
- Incubare i campioni a 4 ° C per 3 ore su un agitatore. Risospendere perline ogni 30 minuti o giù di lì per aumentare l'efficienza di legame.
- Provetta da centrifuga contenente campioni e perline a 1500 rcf per 3 min.
- Prendere 50μL di surnatante come un campione di proteina non legata (campione 2) e rimuovere con attenzione il surnatante per aspirazione e scartare.
- Lavare perle tre volte come descritto al punto 2.3 Uso 100μL di buffer di omogeneizzazione rispettivi proprietari.
4. Eluizione
- Aggiungere 50μL di tampone di eluizione (50mM Tris-HCl pH 7,5 e 150mm NaCl) contenente la condizione opposta (10mM Cacl 2 o 10mM EDTA) ai talloni. Per esempio, i campioni che sono stati omogeneizzati e legato in tampone contenente Ca 2 + vengono eluiti secondo tampone di eluizione contenente EDTA e viceversa.
Opzionale: Il riscaldamento del tampone di eluizione di 37 ° C prima di aggiungere al perline può aumentare il rendimento.
- Incubare soluzione con perline a temperatura ambiente per 30 minuti su un agitatore. Mescolare le perle picchiettando delicatamente il tubo circa ogni 5 min.
- Centrifugare perline a 1.500 rcf per 3 minuti e rimuovere con attenzione il 50μL di supernatante per il legato alle proteine (campione 3) per aspirazione. Assicurarsi di non disturbare le perline.
- Aggiungi il caricamento del buffer proteine a tutti i campioni (ad esempio proteine omogenato, liberi e legati così come quelle ancora legato ai talloni).
Nota: Per massimizzare eluizione (specialmente nel caso di eluizione inefficienti), aggiungere 50μL del buffer di eluizione corrispondente (ad esempio l'aggiunta di buf contenente EDTAfer di perle legate in CaCl 2) per le perle di riscaldamento prima di campioni per eluire le proteine rimanenti legato e ripetere i passaggi descritti in 4,3 per rimuovere le proteine legate rimanenti.
5. SDS-PAGE e Western Blot
Condotta SDS-PAGE e analizzare usando western blot sondando per la proteina di interesse e di sonda per una proteina nota per legare CaM nella condizione opposta come controllo positivo.
6. Rappresentante Risultati
Figura 2B mostra un esempio di Cam-pull-down test di prova il legame di CaM GFP-tagged Ng rispetto al Ng endogena. Per farlo, GFP-Ng è stato sovra-espresso nei nostri organotipica fettine ippocampali durante la notte e il tessuto è stato omogeneizzato. L'omogenato è stato incubato con CaM-sefarosio perline in presenza di entrambi i Ca 2 + o EDTA. Ingresso omogenato mostra che la GFP-Ng è stato espresso oltre che endogene Ng e Ca 2 + / CaM chinasi-dipendenti II(CaMKII). Come previsto in base al legame noto di Ng endogeni (illustrato nella fig. 2A), GFP-tagged Ng è stato eluito in assenza di Ca 2 + (proteina legata EDTA) e non legato alla presenza di Ca 2 + (Fig. 2B) . Al contrario, il controllo, CaMKII, è stato eluito solo in presenza di Ca 2 + (legato alle proteine) ed è stato legato in sua assenza (EDTA). Questo dimostra che le perline CaM erano funziona bene e il elutions erano efficienti. Ancora più importante, questo dimostra che GFP-Ng si lega a ApoCaM in modo simile alla forma endogena, suggerendo che il tag GFP non ha alterato la funzione della nostra proteina ricombinante.
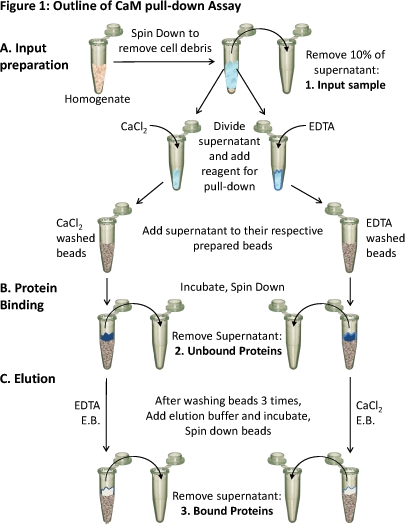
Figura 1. Schema di CaM pull-down assay
(A) omogenato del tessuto è filato giù per rimuovere i detriti cellulari. Circa il 10% del surnatante è preso come un esempio di ingresso (1). Il surnatante rimanente viene diviso in parti uguali per i diversicondizioni ed i reagenti appropriati (CaCl 2 o EDTA) vengono aggiunti alla prova vincolante in quelle condizioni. Ogni surnatante (contenente CaCl 2 o EDTA) viene caricato sulla rispettivamente preparato CaM-sefarosio perline e (B) incubato per consentire vincolanti. Proteine non legate vengono rimossi (2) e (C) le proteine legate (3) vengono eluiti fuori delle perle utilizzando tampone di eluizione (EB) contenente la condizione opposta, come l'associazione. La composizione proteica di questi tre campioni di proteine possono essere analizzati mediante SDS-PAGE e analisi Western Blot.
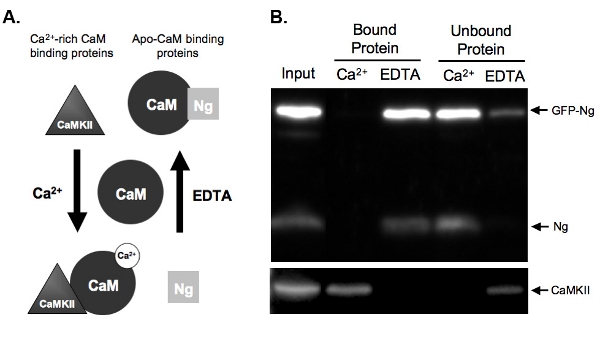
Figura 2. A.) Schema di Ca 2 +-dipendente vincolante CaM e eluizione di pull-down Esempi dosaggio sono dati di due tipi di proteine che legano CaM in Ca 2 +-dipendente. Neurogranin (Ng) rappresenta proteine che si legano apo-CAM e CaMKII rappresenta proteine che si legano a Ca 2 + ricchiCaM. CaM è mostrato nel suo stato dissociato prima dell'incubazione con le proteine omogenato. Una volta incubate in condizioni di alte concentrazioni di Ca 2 + (2 mm) o in presenza di un chelante del Ca 2 +, EDTA (2 mm), le proteine si legano a CaM di conseguenza. Ng lega CaM nella condizione EDTA come non vi è poca o nessuna Ca 2 + presenti, e sarebbe eluiti fuori dal Cam-sefarosio perline in presenza di Ca 2 +. CaMKII, tuttavia, si legano ai CaM in presenza di elevate quantità di Ca 2 + e si dissociano una volta che il Ca 2 + è stato chelati.
B) I risultati di CaM esempio di pull-down test. Questo dato dimostra il risultato finale atteso di un Cam-sefarosio tirare giù con i campioni sondato per Ng e CaMKII. Sia il endogene Ng e GFP-Ng sono presenti nelle corsie di proteine legate a CaM in presenza di EDTA. Ng non è legato quando i campioni sono incubati con CaM in presenza di Ca 2 +, a dimostrazione che Ng lega solo apo-CAM. Il nostro controllo positivo, CaMKII, invece, si lega alla CaM solo in presenza di Ca 2 +.
Discussione
Il protocollo fornito utilizza CaM-sefarosio perline per indagare il Ca 2 +-dipendenza di CaM proteine leganti. Molte proteine si legano CaM in Ca 2 +-dipendente. Queste interazioni sono di grande importanza dato il numero di proteine che legano CaM e il loro ruolo fondamentale in molte vie di segnalazione. In questo protocollo, Cam-sefarosio perline vengono utilizzate per separare CaM-binding proteins da tessuto omogenato in presenza o in assenza di Ca 2 +. I risultati...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Gli autori desiderano ringraziare Tiffany Cherry nel suo aiuto per ottimizzare questo protocollo. Questo lavoro è stato finanziato dal National Institute of Aging (AG032320) così come Avanzando un più sano Wisconsin.
Materiali
| Prodotto | Azienda | Numero di catalogo | Note |
| Calmodulina-Sepharose perline | GE Healthcare | 17-0529-01 | |
| Anti-alfa CaMKII | Sigma-Aldrich | C6974 | |
| Anti-neurogranin | Millipore | 07-425 | |
| Gel Loading Pipettare Consigli | Pescatore | 02-707-138 | L'uso per l'aspirazione di surnatanti |
| Microprovette (2,0 ml) | Pescatore | 05-408-146 | Utilizzare per tutte le fasi che coinvolgono calmodulina-sefarosio perline |
Riferimenti
- Vincenzi, F. F. Calmodulin in the regulation of intracellular calcium. Proc. West Pharmacol Soc. 22, 289-294 (1979).
- Cheung, W. Y. Calmodulin plays a pivotal role in cellular regulation. Science. 207, 19-27 (1980).
- Zhang, M., Tanaka, T., Ikura, M. Calcium-induced conformational transition revealed by the solution structure of apo calmodulin. Nat. Struct. Biol. 2, 758-767 (1995).
- Alexander, K. A., Wakim, B. T., Doyle, G. S., Walsh, K. A., Storm, D. R. Identification and characterization of the calmodulin-binding domain of neuromodulin, a neurospecific calmodulin-binding protein. J. Biol. Chem. 263, 7544-7549 (1988).
- Huang, K. P., Huang, F. L., Chen, H. C. Characterization of a 7.5-kDa protein kinase C substrate (RC3 protein, neurogranin) from rat brain. Arch. Biochem. Biophys. 305, 570-580 (1993).
- Bahler, M., Rhoads, A. Calmodulin signaling via the IQ motif. FEBS Lett. 513, 107-113 (2002).
- Routtenberg, A. Protein kinase C activation leading to protein F1 phosphorylation may regulate synaptic plasticity by presynaptic terminal growth. Behav. Neural. Biol. 44, 186-200 (1985).
- Zhong, L., Cherry, T., Bies, C. E., Florence, M. A., Gerges, N. Z. Neurogranin enhances synaptic strength through its interaction with calmodulin. EMBO J. 28, 3027-3039 (2009).
- Needham, D. M. Myosin and adenosinetriphosphate in relation to muscle contraction. Biochim. Biophys. Acta. 4, 42-49 (1950).
- Hathaway, D. R., Adelstein, R. S. Human platelet myosin light chain kinase requires the calcium-binding protein calmodulin for activity. Proc. Natl. Acad. Sci. U.S.A. 76, 1653-1657 (1979).
- Fukunaga, K., Yamamoto, H., Matsui, K., Higashi, K., Miyamoto, E. Purification and characterization of a Ca2+- and calmodulin-dependent protein kinase from rat brain. J. Neurochem. 39, 1607-1617 (1982).
- Yang, S. D., Tallant, E. A., Cheung, W. Y. Calcineurin is a calmodulin-dependent protein phosphatase. Biochem. Biophys. Res. Commun. 106, 1419-1425 (1982).
- Huestis, W. H., Nelson, M. J., Ferrell, J. E. J. Calmodulin-dependent spectrin kinase activity in human erythrocytes. Prog. Clin. Biol. Res. 56, 137-155 (1981).
- Yamniuk, A. P., Vogel, H. J. Calmodulin's flexibility allows for promiscuity in its interactions with target proteins and peptides. Mol. Biotechnol. 27, 33-57 (2004).
- Zhong, L., Kaleka, K. S., Gerges, N. Z. Neurogranin phosphorylation fine-tunes long-term potentiation. Eur. J. Neurosci. 33, 244-250 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon