È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
GC-based Detection di acetato derivatizzata Glucosamina e muramico Acid Aldononitrile per la determinazione dei residui microbica nel terreno
In questo articolo
Riepilogo
Noi descriviamo un protocollo di metodo per la GC-analisi, basata su strumenti derivati di acetato di aldonitrile di glucosamina e acido muramico estratti dal suolo. Per chiarire il meccanismo chimico, si presentano anche una strategia per confermare la struttura del derivato ed i frammenti di ioni formate sulla ionizzazione elettronica.
Abstract
Quantitavo dei microrganismi che caratterizzano sono essenziali per una comprensione più ampia dello stato microbica e la funzione all'interno degli ecosistemi. Attuali strategie per l'analisi microbiologica di laboratorio tradizionali comprendono sia la cultura-dipendenti tecniche e quelle basate su estrazione diretta e la determinazione di alcuni biomarcatori 1, 2. Pochi tra la diversità delle specie microbiche abitano suolo possono essere coltivate, in modo da cultura-dipendenti metodi introdurre distorsioni significative, una limitazione assente nelle analisi di biomarker.
L'acido glucosamina, mannosammina, galattosamina e muramico sono stati ben servito come misure sia i vivi ei morti di massa microbica, di questi l'acido della glucosamina (più abbondante) e muramico (unicamente dalla cellula batterica) sono costituenti più importanti nei sistemi suolo 3 , 4. Tuttavia, la mancanza di conoscenza sulle analisi limita la divulgazione a livello scientifico tra pari. Tra tutti existing metodi analitici, derivatizzazione di aldononitrile acetati seguita da GC-based analisi è emerso come una buona opzione rispetto a bilanciare in modo ottimale precisione, sensibilità, semplicità, buona separazione cromatografica, e la stabilità durante lo stoccaggio del campione 5.
Qui vi presentiamo un protocollo dettagliato per un'analisi affidabile e relativamente semplice di glucosamina e acido muramico dal terreno dopo la loro conversione al acetati aldononitrile. Il protocollo comprende principalmente quattro fasi: digestione acida, purificazione del campione, derivatizzazione e determinazione GC. Il passo-passo procedura viene modificato secondo ex pubblicazioni 6, 7. Inoltre, presentiamo una strategia per convalidare strutturalmente lo ione molecolare del derivato e suoi frammenti ionici formato sulla ionizzazione elettronica. Abbiamo applicato GC-EI-MS-SIM, LC-ESI-TOF-MS e reagenti marcati con isotopi per determinare il peso molecolare di acetato glucosamina aldononitrile derivatizzata e murL'acido Amic, abbiamo usato lo spostamento di massa di isotopi marcati derivati nello spettro di ioni di indagare frammenti di ioni di ogni derivati 8. Oltre alla spiegazione teorica, la convalida di ione molecolare del derivato e suoi frammenti ionici sarà utile per ricercatori utilizzando δ 13 C o frammenti ionici di questi marcatori in studi biogeochimici 9, 10.
Protocollo
1. Preparazione del campione e l'estrazione degli acidi
- Liofilizzare campioni di terreno dopo la raccolta campo.
- Macinare e omogeneizzare campioni di suolo mediante un mulino a sfere, smerigliatrice suolo, o un mortaio e pestello.
- Pesare campioni di suolo (contenente> 0,3 mg N) in un pallone da 25 mL idrolisi.
- Aggiungere 10 ml di HCl 6M in ogni pallone idrolisi, compilare N 2 gas nei fiaschi, e tappo.
- Idrolizzano a 105 ° C in un incubatore per 8 ore utilizzando un timer automatico.
2. Purificazione del campione
- Rimuovere i palloni dall 'incubatrice; raffreddare a temperatura ambiente.
- Aggiungere 100 ul di standard interno myo-inositolo (1 mg mL -1 in acqua) in ciascuno dei palloni, mescolare agitando.
- Impostare imbuti di plastica drenanti in 200 ml flaconi a forma di pera con ST / NS 24/40 articolazioni, situato in bicchieri di plastica per la stabilità.
- Fold Whatman # 2 Circles qualitativi (11 cm di diametro) in quarti e set in imbuti.
- Swirl ogni pallone idrolisi, e versare il liquame nel imbuto per filtrare (si può ulteriormente lavare ogni pallone con ~ 3 ml di acqua).
- Asciugare il filtrato con un evaporatore a ~ 45 ° C, sotto vuoto applicando.
- Risospendere il residuo secco da ciascuna beuta pera con 3 ~ 5 ml di acqua (usare bagno a ultrasuoni se lo si desidera), e versare in una provetta da 40 ml in teflon, lavare il pallone con una seconda aliquota di acqua.
- Regolare il pH a 6,6-6,8 usando 1M soluzione di KOH a ioni metallici precipitare e altre molecole organiche.
- Rimuovere il precipitati tramite centrifugazione a 2000 rcf per 10 minuti.
- Versare il surnatante in un tubo di 40 ml di vetro, coprire l'apertura del tubo con parafilm, congelare a -20 ° C, poi colpire buchi nel parafilm.
- Liofilizzare il surnatante congelata per rimuovere tutto il liquido.
- Sciogliere il residuo con 3 ml di metanolo a secco, vortex accuratamente (usare bagno a ultrasuoni se lo si desidera), tappare la provetta, quindi si centrifuga a 2000 rcf per 10 minuti per risolverei sali.
- Trasferire il surnatante in una fiala da 3 ml di reazione, porta a secco con macchine RapidVap a 45 ° C (o sotto un leggero flusso di gas di azoto secco se lo si desidera).
- Per ogni flacone, aggiungere 100 microlitri di recupero standard N-metilglucammina (1 mg mL -1 in acqua) e 1 mL H 2 O, coprire le bocche flacone con parafilm, congelare a -20 ° C, perforare la parafilm, e poi congelare a secco .
- Fare standard: aggiungere 100 microlitri di acido muramico (0,5 mg mL -1 in metanolo), 100 pl glucosamina (1 mg mL -1 in acqua), 100 pl myo-inositolo (1 mg mL -1 in acqua), 100 N-pl metilglucamina (1 mg mL -1 in acqua), e 1 ml di H 2 O, parafilm ogni flaconcino, congelare a -20 ° C, perforare la parafilm, poi congelare-dry.
3. Derivatizzazione
- Preparare derivatizzazione reagente contenente 32 mg mL -1 idrossilammina cloridrato e 40 mg mL -1 4-dimetilammino-PYridine in piridina-metanolo (4:1 v / v).
- Aggiungere 300 pl di reagente derivatizzazione a ciascuno dei reactivials, tappo a, e miscelare accuratamente.
- Mettere le provette in bagnomaria 75-80 ° C per 35 minuti (ben sigillate le fiale per evitare che l'acqua per inserire i flaconi).
- Rimuovere le fiale dal bagnomaria, e lasciare raffreddare a temperatura ambiente.
- Aggiungere 1 ml di anidride acetica a ciascuno dei tre reactivials mL, tappo ben, e vortex accuratamente, poi riscaldare 75-80 ° C in bagnomaria per 25 minuti.
- Rimuovere le fiale dal bagnomaria, e lasciare raffreddare a temperatura ambiente.
4. Separazione e Misura
- Aggiungere 1,5 mL di diclorometano ad ogni tappo del flaconcino, strettamente, e miscelare accuratamente.
- Aggiungere 1 ml di HCl 1M per ogni tappo del flaconcino, con forza e vortice a fondo per permettere alle soluzioni per riposare fino a quando le due frasi separate, aspirare ed eliminare la parte superiore (acquosa) fase con 1000 pipettatore ul.
- Nella stessa fashion, estrarre la fase organica 3 volte, ma con 1 mL H 2 O (con l'ultimo stadio di lavaggio, prestare particolare attenzione per garantire che tutta la fase acquosa superiore è stato rimosso).
- Essiccare la soluzione finale utilizzando RapidVap a 45 ° C (o secco utilizzando gas azoto se desiderato).
- Sciogliere in 300 ul di acetato di etile-esano (1:1 v / v) e trasferimento a 2 ml ambra con tappo a vite fiale con inserto in un piccolo volume e tappo.
- Per la quantificazione, analizzare mediante GC-FID utilizzando un fuso colonna capillare di silice polare: 30 m di lunghezza, 0,25 mm, spessore 0,25 um; fase stazionaria fenil-5%, 95% di metil-polisilossano (DB-5 o equivalente) con idrogeno o elio come gas di trasporto, a 0,5 mL min -1 flusso costante. La cromatografia è migliore su premium "inerti" fasi e carrier gas idrogeno, ma è accettabile sulle varietà più comuni e con elio. Le impostazioni del rivelatore sono 300 ° C, 400 mL min -1 aria e 30 mL min -1 sia per l'azoto etrucco gas idrogeno. L'iniezione e parametri forno sono i seguenti: 1 pl iniezione a separazione (30:1) con l'entrata GC impostata a 250 ° C, temperatura del forno iniziale, 120 ° C; premuto 1 min; aumentare la temperatura del forno a 10 ° C min. -1 a 250 ° C; tenere 2,5 min;. rampa a 270 ° C a 20 ° C min -1; tenere 2 minuti; rampa a 290 ° C a 20 ° C min -1, tenere premuto 5 min. Regolare il flusso del gas vettore in modo che la eluiscono inositolo, glucosammina e derivati dell'acido muramico a 250 ° C, e le N-metilglucamina eluisce a 270 ° C.
5. Derivata di convalida
- Utilizzare ionizzazione morbida LC-ESI-TOF-MS per identificare lo ione molecolare e determinare il peso molecolare del derivato.
- Utilizzare GC-EI-MS-SIM o GC-EI-MSMS per il miglioramento della sensibilità per indagare ioni mirati del derivato.
- Utilizzare più reagenti marcati con isotopi per la preparazione dei derivati, e quindi utilizzare lo spostamento di massadi questi derivati marcati con isotopi a MS per indagare ione molecolare e frammenti di ioni.
6. Risultati rappresentativi
Il protocollo del metodo comprende essenzialmente quattro fasi: digestione acido, purificazione del campione, derivatizzazione e determinazione GC (Figura 1). Un esempio di analisi per glucosammina e acido muramico dalle scorte standard e da un campione di terreno viene mostrato nella Figura 2. Oltre glucosammina e acido muramico, mannosammina e galattosamina (due isomeri di glucosammina) possono anche essere determinati simultaneamente utilizzando il metodo. Sulla base di fattori di risposta standard rispetto alla standard interno myo-inositolo, si può quantificare questi marcatori in campioni di terreno. Lo standard di recupero è stato usato per monitorare qualitativamente il processo di derivatizzazione. I regimi per la formazione del aldononitrile derivatizzata glucosammina e acido muramico acetato sono mostrati in figura 3 .
Le strutture proposte dei derivati sono stati determinati mediante GC-EIMS-SIM per miglioramento della sensibilità, o ionizzazione morbida LC-ESI-TOF-MS 8; le strutture proposte dei frammenti ionici formate sulla ionizzazione elettronica stati studiati confrontando gli spettri ione del I campioni preparati con isotopi vari incorporazioni 11. La figura 4 mostra lo spostamento della massa dominante di ioni m / z 187 di acetato glucosamina aldononitrile derivatizzato in tre scenari, preparati con agenti non etichettati, D-acetico anidrite, e U -13 C-glucosamina. Altre informazioni dettagliate e chiarimenti è possibile fare riferimento alla nostra recente pubblicazioni 8, 11. Questa strategia potrebbe servire da modello per studiare la chimica derivata.
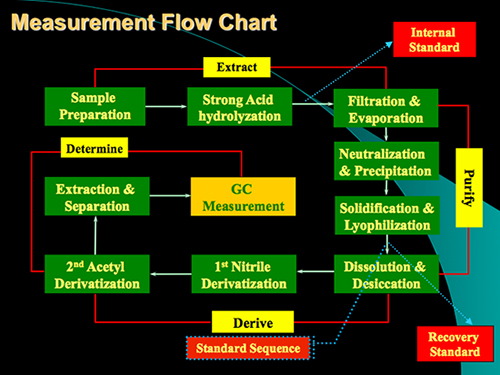
Figura 1. Flusso tabella di misura del protocollo per l'analisi di glucosammina e acido muramico nel suolocampioni. Il protocollo contiene principalmente 4 fasi: digestione acida, purificazione, derivatizzazione e determinazione GC.

Figura 2. GC-FID cromatogrammi di acetati aldononitrile di inositolo, glucosamina, muramico acido, mannosammina, galattosamina e metilglucamina per le norme e del suolo.
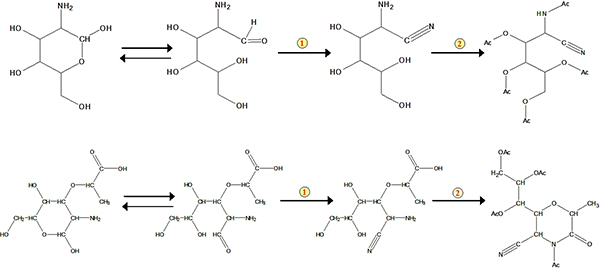
Figura 3. Schemi per la formazione del aldononitrile derivatizzata muramico glucosamina e acido acetico. Il numero 1 rappresenta la reazione nitrile. Il numero 2 rappresenta l'acetilazione.

Figura 4. Spettri di massa di acetato di glucosamina aldononitrile derivatizzata associato vede una struttura ionica dominantees in modalità EI preparato con (A) Agenti non-etichettati, (B), D-acetico anidrite, (C) U -13 C-glucosamina. Stella rappresenta isotopo pesante atomo o gruppo di isotopo.
Discussione
Il presentato GC-based metodo per l'analisi di acetato derivatizzata glucosammina e acido muramico aldononitrile fornisce un metodo relativamente rapido per quantificare questi amminoacidi, zuccheri estratti dal suolo. I derivati sono chimicamente stabili, e può essere determinata in analisi. Il metodo non è limitato a campioni di terreno, e può essere semplificata per campioni di suolo non-matrice.
La pompa a vuoto utilizzata in questo metodo è costruito per essere resistenti ...
Divulgazioni
Non abbiamo nulla da rivelare.
Riconoscimenti
Questo lavoro è stato supportato anche da finanziamenti DOE Bioenergy Great Lakes Research Center (DOE BER Office of Science DE-FC02-07ER64494). Siamo grati al Dr. Zhang Xudong membri del gruppo e dei suoi utili per le discussioni tecniche e commenti preziosi sulla messa a punto del protocollo.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | |
| L'acido muramico | Sigma-Aldrich | M2503-25mg | |
| D-(+)-glucosammina cloridrato | Sigma-Aldrich | G1514-100G | |
| N-metil-D-glucammina | Sigma-Aldrich | M2004-500G | |
| Myo-inositolo | Fisher Scientific | A307003G025 | |
| Metanolo (secco) | Acros organici | AC326950010 | |
| 4-dimetilammino-piridina | Acros organici | AC148270050 | |
| Acetato di etile | VWR International | BJGC100-4 | |
| Hydroxlamine cloridrato | Fisher Scientific | H330-100 | |
| Pyridine | Fisher Scientific | P368-500 | |
| Anidride acetica | Fisher Scientific | A10-100 | |
| Diclorometano (cloruro di metilene) | Fisher Scientific | D37-500 | |
| Esano | Fisher Scientific | H303-4 | |
| Acido cloridrico (6M) | Fisher Scientific | S456-4 | |
| Idrossilammina cloridrato -15 N | Icona servizi | IN5280 | |
| Anidride acetica -2 H (D 6 C 4 O 3) | Acros organici | AC174670050 | |
| D-glucosio-U -13 C | Cambridge isotopo lab | CLM-1396-1 | |
| Ammonio sufate -15 N | Cambridge isotopo lab | NLM-713-1 | |
| Nome dell'apparecchiatura | Azienda | Tipo | |
| Rapid-Vap | Labconco | 790002 | |
| Vacum pompa | KNF Neuberger | D-79.112 | |
| Idrolisi pallone | Fisher Scientific | 06 423A | |
| Derivatizzazione microprovetta | Fisher Scientific | 06-100E | |
| GC | Hewlett-Packard | 6890 | |
| MS | Hewlett-Packard | 5972 | |
| LC-ESI-TOF-MS | Agilent | Un Agilent 1200 Series HPLC accoppiato ad un LC Agilent / MSD-TOF |
Riferimenti
- Zelles, L. Fatty acid patterns of phospholipids and lipopolysaccharides in the characterisation of microbial communities in soil: a review. Biology and Fertility of Soils. 29, 111-129 (1999).
- Kirk, J. L. Methods of studying soil microbial diversity. Journal of Microbiological Methods. 58, 169-188 (2004).
- Joergensen, R. G., Emmerling, C. Methods for evaluating human impact on soil microorganisms based on their activity, biomass, and diversity in agricultural soils. Journal of Plant Nutrition and Soil Science. 169, 295-309 (2006).
- Guggenberger, G., Frey, S. D., Six, J., Paustian, K., Elliott, E. T. Bacterial and fungal cell-wall residues in conventional and no-tillage agroecosystems. Soil Science Society of America Journal. 63, 1188-1198 (1999).
- Amelung, W., Lal, R., Kimble, J. M., Follett, R. F., Stewart, B. A. . Assessment Methods for Soil Carbon. , 233-270 (2001).
- Guerrant, G. O., Moss, C. W. Determination of monosaccharides as aldononitrile, O-methyloxime, alditol, and cyclitol acetate derivatives by gas-chromatography. Analytical Chemistry. 56, 633-638 (1984).
- Zhang, X., Amelung, W. Gas chromatographic determination of muramic acid, glucosamine, mannosamine, and galactosamine in soils. Soil Biology and Biochemistry. 28, 1201-1206 (1996).
- Liang, C. Investigation of the molecular ion structure for aldononitrile acetate derivatized muramic acid. Journal of Microbiological Methods. 86, 224-230 (2011).
- He, H., Xie, H., Zhang, X. A novel GC/MS technique to assess 15N and 13C incorporation into soil amino sugars. Soil Biology and Biochemistry. 38, 1083-1091 (2006).
- Glaser, B., Gross, S. Compound-specific delta 13C analysis of individual amino sugars - a tool to quantify timing and amount of soil microbial residue stabilization. Rapid Communications in Mass Spectrometry. 19, 1409-1416 (2005).
- Liang, C., Balser, T. C. Mass spectrometric characterization of amino sugar aldononitrile acetate derivatives used for isotope enrichment assessment of microbial residues. Soil Biology and Biochemistry. 42, 904-909 (2010).
- Liang, C., Pedersen, J. A., Balser, T. C. Aminoglycoside antibiotics may interfere with microbial amino sugar analysis. Journal of Chromatography A. 1216, 5296-5301 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon