È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Purificazione delle proteine superficiali delle cellule biotinilate da
In questo articolo
Riepilogo
Una metodologia basata sulla gradiente di centrifugazione a densità modificata è stata utilizzata per isolare le cellule epiteliali del tessuto intestinale di Rhipicephalus microplus . Le proteine legate alle superfici sono state biotinilate e purificate mediante perline magnetiche di streptavidina per l'utilizzo nelle applicazioni a valle.
Abstract
Rhipicephalus microplus - il bestiame bestiame - è l'ectoparasite più significativo in termini di impatto economico sul bestiame come vettore di diversi agenti patogeni. Gli sforzi sono stati dedicati al controllo del bestiame da bere per diminuire i suoi effetti deleteri, con particolare attenzione alla scoperta di candidati vaccinali, come BM86, localizzati sulla superficie delle cellule epiteliali delle ghiandole. La ricerca attuale si concentra sull'utilizzo di cDNA e librerie genomiche, per la ricerca di altri candidati vaccinali. L'isolamento delle cellule gutricole costituisce un importante vantaggio nell'individuare la composizione delle proteine superficiali sulla membrana delle cellule gutiche. Questa carta costituisce un metodo nuovo e fattibile per l'isolamento delle cellule epiteliali, dal contenuto di gocce del polmone R. microplus semiaffuso. Questo protocollo utilizza TCEP e EDTA per rilasciare le cellule epiteliali dai tessuti di supporto subepitheliali e una gradi di centrifugazione a densità discontinuaNt per separare le cellule epiteliali da altri tipi di cellule. Le proteine superficiali delle cellule sono state biotinilate e isolate dalle cellule epiteliali delle ghiandole, usando branelli magnetici legati alla streptavidina, che consentono applicazioni a valle in analisi FACS o LC-MS / MS.
Introduzione
Rhipicephalus microplus , il segno del bestiame, è l'ectoparasite più significativo in termini di impatto economico sull'industria del bestiame delle regioni tropicali e subtropicali in quanto vectors bovine tick-tick (babesiosis), anaplasmosis e piroplasmosis equestre 1 , 2 , 3 , 4 . Gli sforzi sono stati dedicati al controllo dei bovini da bestiame, per diminuire l'effetto deleterio, tuttavia i metodi convenzionali come l'uso di acaricidi chimici hanno inconvenienti impliciti, come la presenza di residui chimici nel latte e la carne e l'aumento della prevalenza di zecche chimicamente resistenti 5 , 6 , 7 . Di conseguenza, è stato studiato lo sviluppo di metodi alternativi di controllo del bersaglio, come l'uso di bovini di resistenza naturale, il controllo biologico (biopesticidi) e il vaccinoInes 4 , 5 , 6 , 7 , 8 , 9 .
Nel perseguimento di proteine in grado di essere utilizzate come candidati per il vaccino, la ricerca attuale è incentrata sul guscio del timo. La parete midgut è costruita da un unico strato di cellule epiteliali che poggiano su una sottile lamina basale, con l'esterno della lamina basale che forma una rete muscolare. Le osservazioni del microscopio della luce e dell'elettronica indicano che il midgut è costituito da tre tipi di cellule: riserva (indifferenziata), secrezione e digestione. Il numero di tipi di cellule varia notevolmente a seconda della fase fisiologica. Le cellule di secretory e digestive sono entrambe originate dalle cellule di riserva 18 , 19 , 20 .
La costruzione di librerie cDNAPer esaminare la composizione dell'intestino del guscio ha portato all'identificazione di proteine antigeniche, come Bm86, come candidati potenziali per la vaccinazione 2 , 3 , 4 . La glicoproteina Bm86 è localizzata sulla superficie delle cellule gutiche del tick e induce una risposta immunitaria protettiva contro il bestiame bovino ( R. microplus ) nei bovini vaccinati. Le IgG anti-Bm86 prodotte dall'host immunizzato vengono ingerite dal ticchettio, riconoscono questo antigene sulla superficie delle cellule intestinali, e quindi disturbano la funzionalità e l'integrità del tessuto intestinale. I vaccini basati sugli antigeni Bm86 hanno mostrato un controllo efficace di R. microplus e Rhipicephalus annulatus, riducendo il numero, il peso e la capacità riproduttiva delle femmine incurvando, con conseguente riduzione delle infestazioni di larvali nelle successive generazioni di ticchetti 4 . Tuttavia, i vaccini basati su Bm86 non sono efficaci contro tutte le fasi del tick e hannoHa dimostrato un'efficacia insoddisfacente nei confronti di alcuni ceppi geografici di R. microplus , di conseguenza le industrie bovine e lattiero-caseari hanno adottato male questi vaccini 2 , 4 .
La capacità di isolare le cellule epiteliali dall'intestino è una innovazione significativa che consentirebbe la progressione della ricerca a determinare la composizione della membrana proteica, compresa la morfologia e la fisiologia in condizioni ambientali differenti. Il metodo qui descritto utilizza l'agente chelante acido etilendiamintetraoacetico (EDTA) e l'agente riducente tris (2-carbossiletil) fosfina (TCEP) per rilasciare l'epitelio dai suoi tessuti di supporto sub-epiteliali 10 . L'epitelio viene recuperato a seguito di scosse meccaniche dei tessuti agitando, seguita da centrifugazione a gradiente discontinua in Percoll. Questo articolo descrive una tecnica fattibile e novella per l'isolamento di epi di zuccheroCellule teliali. Le proteine superficiali delle cellule biotinilate, isolate dalla superficie di queste cellule epiteliali, possono successivamente essere analizzate in applicazioni a valle come FACS e / o analisi LC-MS / MS.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Dissezione dell'epitelio di Gut da R. microplus
- Raccogli le zecche semi-inguine dal bestiame nel giorno dell'esperimento. Disseccare le zecche entro 24 ore dopo la rimozione dall'ospite.
- Aderire una striscia di nastro adesivo alla parte inferiore del piatto di Petri da 92 mm x 16 mm. Aggiungere una goccia di colla super al nastro. Posizionare il ticchettio, lato ventrale verso il basso sulla colla super, lasciare asciugare per 2 minuti.
- Versare 100 ml di soluzione salina fosfata (PBS) nel piatto di Petri, o fino a quando la zecca è completamente sommersa.
- Utilizzando uno scalpello di taglia 11, tagliato dalla parte superiore degli occhi ai festoni inferiori, su entrambi i lati del ticchettio.
- Utilizzando pinze sterili, rimuovere completamente i scutum e alloscutum, per esporre gli organi interni.
- Rimuovere gli organi filettati bianchi fine (trachea) e altre membrane per impedire la contaminazione.
- Rimuovere l'intestino con le pinze, pizzicando la regione superiore e tirandola dallacarcassa. Rimuovere eventuali tessuti intestinali rimanenti, assicurando che nessun altro tessuto venga disciolto.
- Conservare l'intestino in soluzione salina equilibrata di Hank (HBSS) ghiacciata senza cloruro di calcio e solfato di magnesio con il cocktail inibitore della proteina (PIC). Snap congelare il budello in ghiaccio secco e conservare a -20 ° C.
Nota: per assistere con la dissezione delle intestine e dettagli sugli organi interni del ticco, fare riferimento al capitolo 3.1 di D. Sonenshine, "Biologia delle zecche" 19 .
2. Dissociazione delle cellule epiteliali
- Versare l'intestino sezionato su un filtro cellulare da 70 μm all'interno di un tubo da 50 ml.
- Lavare il tessuto intestinale con 50 ml di ghiaccio freddo HBSS con PIC fino a quando la soluzione scorre chiara e il budello assume un aspetto bianco / chiaro.
- Riassemblare il budello in 30 mL di ghiaccio freddo HBSS con PIC, mescolare delicatamente e centrifugare a 500 xg a 4 ° C per 10 minuti per far pelliccare l'intestino. Rimuovere il surnatante e ripetere il processo di lavaggio tre volte.
- Per dispiegare le cellule dell'intestino epiteliale, riposizionare l'intestino in 10 ml di terreno di coltura cellulare Dulbecco's medium modificato (DMEM), 2% di siero fetale (FCS), 0,5 mM di etilendiammina tetraacetico (EDTA), 1 mM Tris (TCEP), PIC e incubare per 60 minuti a 37 ° C in rotazione lenta utilizzando un rullo a 6 giri / min.
- Filtrare la sospensione attraverso un filtro di cellule da 250 μm, vortexare il flusso e filtrare attraverso un filtro di cellule da 70 μm raccogliendo il restante flusso.
- Centrifugare la sospensione a 500 xg a 4 ° C per 20 minuti per far pelliccare le singole cellule.
3. Isolamento di singole cellule epiteliali utilizzando un gradiente di centrifugazione di densità
- Preparare il gradiente di centrifugazione di densità ( ad esempio Percoll) filtrando attraverso una carta AP15 prefiltrata. Preparare il 40% e un 20% di Percoll in mqH 2 0 (v / v) e raffreddare a 4 ° C per 1 ora prima di stratificare il gradiente.
- Utilizzando un peristalticoLa pompa è impostata alla velocità più bassa, con 3 ml di gradiente di centrifugazione di densità del 40% in un tubo di ultracentrifuga da 16 ml, lasciandolo saldare a ghiaccio per 15 minuti. La velocità della pompa deve portare ad una velocità di flusso di <1 mL per min.
- Inclinando il tubo ad un angolo di 45 °, utilizzare la pompa peristaltica per aumentare il gradiente di centrifugazione di densità del 20% sulla parte superiore del livello del 40%. La velocità della pompa deve portare a <1 mL per min. Lasciare che i livelli si stabiliscano in ghiaccio per 15 minuti.
- Utilizzare la pompa peristaltica a una velocità di flusso minima di 1 mL per 3 ml di substrato DMEM contenente le cellule gutriche sopra il gradiente di centrifugazione di densità del 20-40%.
- Programmare la centrifuga per l'accelerazione massima e la decelerazione minima. Centrifugare a 600 xg per 10 min. Raccogli le interfacce tra il gradiente di centrifugazione DMEM: 20% di densità e le gradienti di centrifugazione di densità del 20%: 40% per isolare singole cellule epiteliali. Conservare le interfase raccolte a 4 ° C per le successive analisi.
4. Valutazione dell'isolamento cellulare
- emocitometro
- Pulire la diapositiva di hemacytometer con alcool.
- Sospendere le cellule pipettando delicatamente le cellule su e giù. Pipettare 100 μL della sospensione cellulare e collocare in un tubo di microcentrifuga da 1,5 ml.
- Aggiungere 400 μl di 0.4% Trypan Blue. Mescolare delicatamente spostando il tubo.
- Pipettare 100 μL della sospensione cellulare della Trypan blu per riempire lentamente entrambe le camere dell'emocitometro.
- Posizionare l'emocitometro sotto un microscopio leggero, concentrandosi sulle linee di griglia dell'emocitometro con un obiettivo di 10x.
- Utilizzando un contatore di contatti tenuto in mano, conteggi le cellule non sporche dal vivo all'interno di un insieme di 16 quadrati. All'interno della stessa piazza, contano le cellule morte blu. Continuare a contare finché non si contano quattro set di 16 quadrati.
- Calcolare le cellule totali per ml utilizzando la formula:
47eq1.jpg "/> - Calcola la vitalità cellulare percentuale utilizzando la formula:
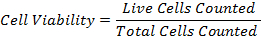
- Visualizzazione delle isole della cellula
- Diluire 1 μL delle cellule isolate in 9 μL di HBSS in un tubo di microcentrifuga da 1,5 ml. Spostare delicatamente il tubo di microcentrifuga per mescolare.
- Pipettare 5 μL di cellule isolate in HBSS nel mezzo di una vetrata. Applicare tre gocce di supporto di montaggio con 4 ', 6-diammino-2-fenilindolo (DAPI).
- Incubare la diapositiva a temperatura ambiente per 5 min. Posizionare con cura un coperchio di copertura sulla preparazione evitando le bolle d'aria.
- Visualizzare le cellule colorate con DAPI all'eccitazione a 360 nm e emettere a 460 nm sotto un microscopio fluorescente.
5. Biotinilazione della proteina della superficie cellulare
- Biotinilate 100 μL di cellule epiteliali a cellule singole usando la coniugazione di Biotina (tipo A)Kit, a un rapporto molare di proteine superficiali da 1: 1 a coniugare, secondo le istruzioni del produttore
- Per la lisi delle cellule aggiungere 100 μL di PBS, 1% Triton X-100, 10% glicerolo, 100 μM glutatione ossidato e PIC alle cellule biotinilate. Incubare su ghiaccio per 1 h con mescolanza dolce ogni 10 min.
- Centrifugare le cellule biotinilate a 20.000 xg a 4 ° C per 20 minuti per far pelliccare il materiale insolubile. Raccogliere il surnatante contenente proteine citoplasmatiche e biotinilate.
- Determinare la concentrazione proteica usando il saggio di Bradford.
6. Isolamento delle proteine superficiali biotinilate
- Aggiungere 50 μL di perline magnetiche di Streptavidin in un tubo di microcentrifuga da 1,5 ml.
- Mettere il tubo in un supporto magnetico, raccogliendo le perle contro il lato del tubo. Rimuovere e scartare il surnatante.
- Aggiungere 1000 μL di TBS, 0,1% Tween-20 al tubo. Mescolare delicatamente e raccogliere le perle con l'appartamento magneticod. Rimuovere e scartare il surnatante.
- Combinare 40 μg di proteine superficiali delle cellule biotinilate, diluite a 300 μL in 1x PBS con perline magnetiche lavate. Incubare per 2 ore a temperatura ambiente con agitazione.
- Raccogliere le perle con un supporto magnetico, rimuovere e scartare il surnatante.
- Aggiungere 300 μl di TBS, 0.1% Tween-20 al tubo, mescolando delicatamente per ri-sospendere le perle. Raccogli le perline, rimuove e scarta il supernatante. Ripetere due volte questo passaggio di lavaggio.
- Aggiungere 100 μL di 0,1 M glicina pH 2,0 alle perline magnetiche e incubare a temperatura ambiente per 5 min. Raccogliere le perle e rimuovere il surnatante contenente proteine superficiali biotinilate eluate.
- Visualizzare proteine superficiali isolate su un gel di Tris-MOPS SDS-PAGE 4-20%.
7. Valutazione della proteina superficiale biotinilata
- Dot Blot
- Tagliare una striscia di nitrocellulosa da 7 cm x 3 cm.
- Applicare 10 μg di zucchero totaleT (da 1.8 sopra) e 10 μg di proteine superficiali biotinilate alla membrana. Lasciare asciugare per 15 minuti a temperatura ambiente.
- Trasferire in un contenitore e immergerlo in 100 mL di tampone di blocco. Incubare a temperatura ambiente con agitazione per un'ora. Eliminare il buffer di blocco.
- Lavare la membrana di nitrocellulosa in 100 μl di PBS, 0,05% di Tween-20 per 5 minuti con agitazione. Scartare il tampone di lavaggio e ripetere le lavaggi per tre volte.
- Incubare in 1/5000 Streptavidin-perossidasi di rafano (HRP) in 100 μl di PBS, 0,05% di Tween-20 per 2 ore con agitazione a temperatura ambiente e scartare ogni residua soluzione.
- Lavare la membrana di nitrocellulosa in 100 μl di PBS, 0,05% Tween-20 per 5 minuti con agitazione. Scartare il tampone di lavaggio e ripetere il lavaggio tre volte.
- Per il rilevamento, sciogliere 1 compressa di 4-cloro-1-naftolo in 10 ml di metanolo a ghiaccio freddo. Aggiungere 4 ml di materiale di metanolo a 20 ml di TBS. Aggiungere 10 μl di fresco perossido di idrogeno al 30% e immediatamenteSi applicano alla membrana di nitrocellulosa.
- Incubare con agitazione a temperatura ambiente finché il substrato non produce un prodotto finale blu insolubile. Questo può richiedere tra 2-15 min.
- Eliminare la soluzione di rilevazione e lavare la membrana tre volte in 100 μL mqH 2 O.
- ELISA Assay
- Diluire 200 ng di ciascun campione con 400 μL di tampone di rivestimento in carbonato da 100 mM e coprire 2 corsie della prima fila (A #) della piastra ELISA (pozzetti piani)
- Aggiungere 100 μL di buffer di rivestimento carbonato da 100 mM a tutti gli altri pozzetti corrispondenti in righe (BH).
- Preparare le diluizioni seriali di ciascun campione pipettando 100 μl da ogni pozzetto nella riga A e trasferendolo nella riga B. Mescolare delicatamente pipettando ed evitare di produrre bolle. Ripetere le diluizioni in giù per ogni riga, scartando le ultime 100 μL da ogni pozzetto nella riga H.
- Coprire la lastra con il parafilm e incubare a 4 ° C per una notte.
- Lavare la piastraE con 200 μl PBS, 0,05% Tween-20 per pozzetto tre volte.
- Aggiungere 200 μl di tampone di bloccaggio ai pozzetti ricoperti, coprire con Parafilm e incubare a 4 ° C per una notte.
- Diluire 1 / 15.000 Streptavidin-HRP nel buffer di blocco e aggiungere 100 μL ad ogni pozzetto della piastra. Coprire con il parafilm e incubare a 4 ° C per una notte.
- Lavare la piastra in 200 μl di PBS, 0,05% Tween-20 per pozzetto cinque volte.
- Per rilevare, aggiungere 100 μL di reagente TMB per pozzetto. Dopo un sufficiente sviluppo di colore, di solito fra 10-15 min, aggiungere 100 μL di acido fosforico da 1 M per fermare la reazione.
- Leggere l'assorbanza di ogni pozzetto a λ = 450nm.
Access restricted. Please log in or start a trial to view this content.
Risultati
Le cellule epiteliali sono state isolate dai tessuti intestinali di R. microplus secondo lo schema presentato in Figura 1 . Le rappresentazioni rappresentative di microscopia a fluorescenza delle cellule epiteliali delle ghiandole del guscio preparate usando questo protocollo sono mostrate nella Figura 2A E 2B. Poiché l'isolamento delle cellu...
Access restricted. Please log in or start a trial to view this content.
Discussione
Le infestazioni di zootecnia costituiscono un grave problema per l'industria del bestiame nelle regioni tropicali e subtropicali del mondo, con il metodo più comune di controllo dipendente dall'uso degli acaricidi 1 , 4 . Bm86 è stato precedentemente individuato all'interno della superficie epiteliale della ghiera di zecca come antigene protettivo contro l'infestazione di R. microplus 10 , con un successo limita...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno niente da rivelare.
Riconoscimenti
Gli autori vogliono ringraziare la Biosecurity Tick Colony (Queensland Department of Agriculture & Fisheries, Australia) per la fornitura di Rhipicephalus microplus ticks utilizzati per questo studio e Lucas Karbanowicz per l'assistenza con la ripresa video.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.4% Trypan Blue | ThermoFisher Scientific | 15250061 | |
| 1.5 mL microcentrifuge tube | Eppendorf | 3322 | |
| 100mM Carbonate Buffer | 3.03 g Na2CO3, 6.0 g NaHCO3 1000 ml distilled water pH 9.6 | ||
| 16 mL centrifuge tubes with sealing cap | Thermo Scientific | 3138-0016 | Cool in ice prior to gradient |
| 250 µM cell strainer | Thermo Fisher | 87791 | |
| 3,3′,5,5′-Tetramethylbenzidine (TMB) Liquid Substrate System for ELISA | Sigma | T0440 | Stored at 4C |
| 30% Hydrogen Peroxide | Labscene | BSPA5.500 | |
| 4-20% Tris-MOPS Gel | Gen Script | M42015 | |
| 4-Chloro-1-naphthol tablet | Sigma-Aldrich | C6788 | |
| 50 mL Falcon Tube | Corning Blue | 30 x 115mm style. Polyproplyene conical tube. | |
| 70 µM cell strainer | BD Falcon | 352350 | |
| AP15 filter paper | Millipore | AO1504200 | |
| Biotin (Type A) Conjugation Kit | Abcam | Ab102865 | |
| Dissection microscope | Olympus | SZX7 | |
| DP Manager | Olympus | 2.2.1.195 | Cell imagery software |
| Duct Tape | Home Handyman | 48mm x 25mm Duct Tape | |
| Dulbecco’s Modified Eagle Medium | Gibco | 11995-065 | DMEM - ice cold for protocol |
| EDTA | Amresco | 0105-500G | |
| F96 Maxisorp Immuno Plate | Nunc | 439454 | |
| Fetal Bovine Serum | Sigma-Aldrich | 12003C | FCS |
| Fluorescence microscope | Olympus | BX51 | |
| Fluoroshield with DAPI | Sigma-Aldrich | F6057-20ML | DAPI |
| Forceps | Dumont | #9 Dumont - Switerzland | |
| Glycerol | Sigma-Aldrich | G5516 | Glycerol for molecular biology >99% |
| Glycine | Sigma-Aldrich | 410225 | |
| Hand-Held Counter | Officeworks | JA0376230 | |
| Hank’s Balanced Salt Solution | Sigma Life Sciences | H9394 | HBSS – ice cold for protocol |
| Hemacytometer | Optik Lakor | - | - |
| L-Glutathione oxidized | Sigma-Aldrich | G4376 | |
| Magnetic Separation Stand | Novagen | - | 4-Tube Magnetic Separation Rack |
| Methanol | Sigma-Aldrich | 179337 | |
| Milli-Q Water | Millipore | ZRXQ003WW | Integral Water Purification System for Ultrapure Water |
| Nitrocellulose Membrane | Life Sciences | 66485 | 30cm x 3M pure nitrocellulose membrane |
| PageRuler Prestained protein Ladder | Thermo-Fisher | SM0671 | |
| PBS | 1.16 g Na2HPO4, 0.1 g KCl, 0.1 g K3PO4, 4.0 g NaCl (500 ml distilled water) pH 7.4 | ||
| Percoll | Sigma-Aldrich | P1644-500ML | |
| Peristaltic Pump | Masterflex | 7518-10 | |
| Phosphoric Acid | Sigma-Aldrich | P6560 | |
| Pierce Protein-Free T20 PBS Blocking Buffer | Thermo-Scientific | 37573 | Stored at 4C. Blocking Buffer |
| Protease Inhibitor Cocktail | Sigma-Aldrich | P8215-5ML | PIC – stored at -20 °C |
| Quick Start Bradford Dye Reagent 1x | Biorad | 500-0205 | For Bradford Assay |
| Quick Start BSA Standards | Biorad | 500-0207 | BSA standards for Bradford Assay |
| Scalpel | Lab. Co | Size 11 Scalpel | |
| SilverQuest TM Staining Kit | Invitrogen | LC6070 | |
| Simply Blue TM Safe Stain | Invitrogen | LC6060 | |
| Sorvall C6+ Ultracentrifuge | Thermo Scientific | 46910 | |
| Streptavidin (HRP) | Abcam | AB7403 | |
| Streptavidin Magnetic Beads | New England Biolabs | S1420S | |
| Super Glue - Ultra Fast Mini | UHU | UHU Super Glue 1mg. Ultra Fast mini | |
| Table-top Centrifuge | Eppendorf | 22331 | |
| TCEP | Thermo Fisher | 20490 | |
| Triton X-100 | Biorad | 161-0407 | |
| Tween-20 | Sigma | P2287-500ML | |
| Vortex Mixer | Ratek | VM1 | |
| Water Bath | Grant | GD100 |
Riferimenti
- Rodriguez-Valle, M., et al. Efficacy of Rhipicephalus (Boophilus) microplus Bm86 against Hyalomma dromedarii and Amblyomma cajennense tick infestations in camels and cattle. Vaccine. 30, 3453-3458 (2012).
- De Rose, R., et al. Bm86 antigen induces a protective immune-response against Boophilus microplus following DNA and protein vaccination in sheep. Vet. Immunol. Immunopathol. 71, 151-160 (1999).
- García-García, J. C., et al. Sequence variations in the Boophilus microplus Bm86 locus and implications for immunoprotection in cattle vaccinated with this antigen. Exp. Appl. Acarol. 23, 883-895 (1999).
- Abbas, R. Z., Zaman, M. A., Colwell, D. D., Gilleard, J., Iqbal, Z. Acaricide resistance in cattle ticks and approaches to its management: The state of play. Vet. Parasitol. 203, 6-20 (2014).
- Kearney, S. Acaricide (chemical) resistance in cattle ticks. , AgNote No. K58. (2013).
- Foil, L. D., et al. Factors that influence the prevalence of acaricide resistance and tick-borne diseases. Vet. Parasitol. 125, 163-181 (2004).
- Rodriguez, M., et al. High level expression of the B. microplus Bm86 antigen in the yeast Pichia pastoris forming highly immunogenic particles for cattle. J Biotechnol. 33, 135-146 (1994).
- Rodriguez, M., et al. Effect of vaccination with a recombinant Bm86 antigen preparation on natural infestations of Boophilus microplus in grazing dairy and beef pure and cross-bred cattle in Brazil. Vaccine. 13 (18), 1804-1808 (1995).
- Lew-Tabor, A. E., Rodriguez Valle, M. A review of reverse vaccinology approaches for the development of vaccines against ticks and tick borne diseases. Ticks Tick Borne Dis. 7, 573-585 (2016).
- Capella, A. N., Terra, W. R., Ribeiro, A. F., Ferreira, C. Cytoskeleton removal and characterization of the microvillar membranes isolated from two midgut regions of Spodoptera frugiperda (Lepidoptera). Insect Biochem. Mol. Biol. 27, 793-801 (1997).
- Cioffi, M., Wolfersberger, M. G. Isolation of separate apical, lateral and basal plasma membrane from cells of an insect epithelium. A procedure based on tissue organization and ultrastructure. Tissue Cell. 15, 781-803 (1983).
- Koefoed, B. M. A simple mechanical method to isolate the basal lamina of insect midgut epithelial cells. Tissue Cell. 17, 763-768 (1985).
- Roche, J. K. Isolation of a purified epithelial cell population from human colon. Methods Mol. Med. 50, 15-20 (2001).
- Terra, W. R., Costa, R. H., Ferreira, C. Plasma membranes from insect midgut cells. An. Acad. Bras. Ciênc. 78, 255-269 (2006).
- Vargas, A. E., Markoski, M. M., Cañedo, A. D., Helena, F., Nardi, N. B. Identification, isolation and culture of intestinal epithelial stem cells from murine intestine. Stem Cells. 879, 479-490 (2012).
- Autengruber, A., Gereke, M., Hansen, G., Hennig, C., Bruder, D. Impact of enzymatic tissue disintegration on the level of surface molecule expression and immune cell function. Eur. J. Microbiol. Immunol. 2, 112-120 (2012).
- Karhemo, P. R., et al. An optimized isolation of biotinylated cell surface proteins reveals novel players in cancer metastasis. J. Proteomics. 77, 87-100 (2012).
- Obenchain, F. R., Galun, R. Physiology of Ticks. Current Themes in Tropical Science Volume 1. , Pergamon Press. 201-205 (1982).
- Sonenshine, D., Roe, R. Chapter 3.1. "Biology of Ticks". 1, Two, Oxford University Press. (2014).
- Raikhel, A. S., Balashov, Y. S. "An Atlas of Ixodid Tick Ultrastructure" (English Translation). , Entomology Society of America Entomological Society of America. (1983).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon