È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Neutralizzando le risposte dell'anticorpo al virus A(H3N2) in sieri umani di Microneutralization l'Influenza di misurazione analisi utilizzando cellule MDCK-SIAT1
In questo articolo
Riepilogo
Anticorpi neutralizzanti l'influenza correlano con protezione delle infezioni influenzali. Saggi microneutralization misurano anticorpi neutralizzanti nel siero umano e sono spesso utilizzati per sierologia umana dell'influenza. Descriviamo un dosaggio di microneutralization usando le cellule MDCK-SIAT1 per misurare i titoli dell'anticorpo neutralizzante ai virus contemporaneo di A(H3N2) di 3C.2a e 3C.3a dopo la vaccinazione contro l'influenza o infezione.
Abstract
Anticorpi neutralizzanti contro emoagglutinina (HA) di virus influenzali sono considerati il principale meccanismo immune che correla con protezione per infezioni influenzali. Saggi microneutralization (MN) sono spesso utilizzati per misurare le risposte anticorpali neutralizzanti nel siero umano dopo vaccinazione contro l'influenza o infezione. Cellule di rene canino Madine Darby (MDCK) sono il substrato di cella comunemente utilizzata per le analisi di MN. Tuttavia, l'influenza A(H3N2) 3C.2a e 3C.3a hanno acquisito il virus attualmente in circolazione alterata specificità di legame del recettore. La linea cellulare di MDCK-SIAT1 con moiety aumentata α-2,6 galattosio sialico sulla superficie ha dimostrato di fornire infettività migliorata e le repliche più fedeli rispetto alle cellule MDCK convenzionale per questi virus A(H3N2) contemporanei. Qui, descriviamo un saggio di MN usando le cellule MDCK-SIAT1 che è stato ottimizzato per quantificare i titoli dell'anticorpo neutralizzante a questi virus A(H3N2) contemporanei. In questo protocollo, calore inattivato sieri contenenti anticorpi neutralizzanti sono in primo luogo in serie diluiti, poi incubati con 100 TCID50/bene di A(H3N2) virus per consentire gli anticorpi nel siero da associare ai virus. Le cellule MDCK-SIAT1 sono poi aggiunto alla miscela di anticorpo del virus e incubate per 18-20 h a 37 ° C, 5% CO2 per consentire A(H3N2) virus di infettare le cellule MDCK-SIAT1. Dopo l'incubazione overnight, piastre sono fissi e la quantità di virus in ogni bene è quantificata mediante un'analisi enzima-collegata dell'immunosorbente (ELISA) utilizzando anti-influenzale un anticorpi monoclonali nucleoproteina (NP). Titolo dell'anticorpo di neutralizzazione è definita come il reciproco della più alta diluizione del siero che fornisce ≥ 50% inibizione dell'infettività del virus.
Introduzione
Virus influenzali continuano a causare morbilità e mortalità nella popolazione umana ogni anno. HA è la glicoproteina di superficie principale di virus influenzali. Anticorpi neutralizzanti HA di targeting sono il principale meccanismo immune che correla con la protezione di infezione influenzale1,2. Emoagglutinazione inibizione (HI) saggi e saggi di MN sono due metodi ampiamente usati per misurare le risposte dell'anticorpo in sieri umani dopo di infezione o la vaccinazione influenzale3. Il test HI misura l'inibizione dell'anticorpo di emagglutinazione virus dei globuli rossi ed è considerato un saggio di surrogati. A differenza di HI, il dosaggio di MN può direttamente misurare i livelli di anticorpi nel siero umano che neutralizzano l'infezione di riossidazione in colture cellulari. Le cellule MDCK sono spesso utilizzate nell'isolamento del virus dell'influenza e MN saggi4.
Virus influenzali sono sottoposti costantemente deriva antigenica e MAIUSC, l'acquisizione di mutazioni HA proteine che possono alterare la specificità di legame del recettore del virus. Dal 2014, nuovi cluster di A(H3N2) virus è emerso e continuò a circolare fino alla stagione corrente. La maggior parte di questi virus appartenga al gruppo genetico 3C.2a e 3C.3a basato su analisi filogenetica delle proteine HA. Molti dei virus circolanti 3C.2a aveva ridotto la capacità di hemagglutinate globuli rossi e quindi non possono essere caratterizzati da HI saggi5. Saggi di neutralizzazione devono essere utilizzati per misurare le risposte dell'anticorpo a questi virus che non hemagglutinate6. Inoltre, gli studi hanno indicato che questi virus A(H3N2) contemporanei hanno alterato le proprietà di binding recettoriale rispetto ai precedenti A(H3N2) virus e tendono ad accumulare mutazioni cultura adattato e polimorfismo quando attraversate in vitro delle cellule culture7,8,9. Rispetto ai convenzionali cellule MDCK, MDCK-SIAT1 è una linea cellulare sviluppata da Matrosovich et al. mediante trasfezione stabile di cellule MDCK con cDNA di umano α2, 6-sialtransferase (SIAT1). Questa linea cellulare esprime gli importi aumentati di α2, moiety 6-sialico galattosio e quantità in diminuzione di α2, acido 3-sialico moiety rispetto al padre di cellule MDCK10. Le cellule MDCK-SIAT1 sono indicate per migliorare i tassi di isolamento per A(H3N2) virus rispetto alle cellule MDCK11. Recentemente, Lin et al. ha riferito che di recente è emerso 3C.2a e 3C.3a A(H3N2) dell'influenza virus umani, più fedeli repliche di virus e meglio infettività del virus sono stati raggiunti quando virus sono stati coltivati in linee cellulari MDCK-SIAT1 rispetto a cellule MDCK7. Così, le cellule MDCK-SIAT1 sono più adatti in saggi di MN per caratterizzare le risposte dell'anticorpo ai recenti cluster di A(H3N2) virus.
Qui descriviamo un'analisi di MN usando cellule MDCK-SIAT1 per misurare le risposte dell'anticorpo a contemporanea 3C.2a e 3C.3a A(H3N2) virus in sieri umani. Virus coltivato in uova o da cellule può essere usato per questo test. Sieri di calore inattivato contenente anticorpi neutralizzanti sono prima diluiti serialmente, poi incubati con 100 TCID50/bene di A(H3N2) virus per consentire gli anticorpi nel siero da associare al virus. Le cellule MDCK-SIAT1 sono poi aggiunto alla miscela di anticorpo del virus e incubate per 18-20 h a 37 ° C, 5% CO2 per consentire il A(H3N2) virus di infettare le cellule MDCK-SIAT1 e replicare. Dopo l'incubazione durante la notte, le piastre sono fissate e la quantità di virus in ogni pozzetto è quantificata mediante un test ELISA che usando gli anticorpi monoclonali di anti-riossidazione A NP. La rilevazione di NP indica la presenza dell'infezione del virus e l'assenza di anticorpi neutralizzanti. Titolo dell'anticorpo di neutralizzazione è definita come il reciproco della più alta diluizione del siero che fornisce ≥ 50% inibizione dell'infettività del virus.
Protocollo
Tutti i virus influenzali devono essere gestiti secondo i requisiti del livello di biosicurezza appropriato (BSL-2 o superiore) come definito in Biosafety in Microbiological and Biomedical Laboratories (BMBL)12.
1. preparazione dei reagenti e materiale di partenza
-
Preparare le cellule MDCK-SIAT1 e terreno di coltura sterile cellulare
- Preparare il terreno di coltura delle cellule MDCK-SIAT1 utilizzando 500 mL di Dulbecco per volta Eagle Medium (DMEM) con alto glucosio, inattivato con calore di v/v di 10% siero bovino fetale (FBS), 2 mM L-Glutammina, piruvato di sodio di 1 mM, solfato di 1mg/mL G418 (ad es., Geneticin) e 100 U/ml di penicillina con 100 µ g/mL di streptomicina (opzionale). Sterilizzare mediante filtrazione attraverso una membrana di dimensioni dei pori di 0,2 µM. G418 solfato viene aggiunto per garantire la stabilità del plasmide nelle cellule MDCK-SIAT1.
- Preparare la linea di cellule MDCK-SIAT1 . Un 162 cm,2- flask(s) di coltura del tessuto contenente 30 mL di coltura di cellule sterili, boccette con 2-2.5 x 106 MDCK-SIAT1 cellule di semi e incubare a 37 ° C 5% CO2 per 2 giorni; Questo verrà utilizzato per la determinazione della dose infettiva (TCID) di coltura del tessuto e MN saggi.
Nota: Le cellule MDCK-SIAT1 sono stati gentilmente fornite da Dr. M. Matrosovich, Marburg, Germania10. Questa linea cellulare possa essere ottenuta anche commercialmente (Vedi la Tabella materiali).
- Preparare i mezzi di propagazione di virus sterile utilizzando 500 mL di glucosio elevato DMEM, 0,3% albumina di siero bovino (BSA) frazione V, 100 U/mL di penicillina e 100 µ g/mL di streptomicina e 20 mM HEPES. Sterilizzare mediante filtrazione attraverso una membrana di dimensioni dei pori di 0,2 µM.
- Preparare 0,75% (v/v) cellule di anima rosse di cavia (gpRBCs) in tampone fosfato salino (PBS) contenente PBS 0,01 M, a pH 7,2.
- Preparare il diluente sterile virus utilizzando DMEM alto glucosio, 1% frazione V di albumina bovina (BSA), 100 U/mL di penicillina, 100 µ g/mL di streptomicina e 20 mM HEPES. Sterilizzare mediante filtrazione con una membrana di formato del poro 0,2 µm e preparare fresco per ogni dosaggio.
- Preparare il fissativo cella fredda come 80% acetone freddo in PBS (PBS 0,01 M, pH 7,2).
- Preparare il tampone di lavaggio con PBS (0.01 M PBS pH 7.2) e 0,3% (v/v) di tween-20.
- Preparare il diluente anticorpo usando PBS (PBS 0,01 M, pH 7,2), 0,3% (v/v) di tween-20 e 5% scremato latte in polvere.
- Utilizzare clone dell'anticorpo monoclonale murino di anti-influenzale A NP A1 e A3 piscina come l' anticorpo primario. Diluire il diluente alla concentrazione ottima come determinato tramite la titolazione degli anticorpi.
- Capra di uso IgG anti-topo coniugata con perossidasi di rafano cavallo (HRP) come l' anticorpo secondario. Diluire il diluente alla concentrazione ottima come determinato tramite la titolazione degli anticorpi.
- Preparare il substrato perossidasi utilizzando o- fenilendiamina dicloridrato (OPD) in tampone citrato fosfato di 0,05 M a pH 5. Preparare il tampone citrato fosfato di 0,05 M sciogliendo 1 capsula/100 mL deionizzata H2O. sciogliere 1 OPD compressa (10 mg) / 20 mL di tampone citrato fosfato immediatamente prima dell'uso.
- Utilizzare 0,5 M di acido solforico come la soluzione di arresto OPD. Aggiungere 28 mL di brodo acido solforico 18 M a 972 mL deionizzata H2O in una cappa chimica.
-
Trattamento di sieri umani e animali
- Inattivare con il calore i sieri umani utilizzati nelle analisi MN in un bagno di acqua a 56 ° C per 30 min prima del dosaggio. Utilizzare immediatamente o se necessario, archivio scongelati sieri a 4 ° C per non più di 24 h; Se è necessario più lungo periodo di stoccaggio, memorizzare sieri congelati a-20 ° C o a temperature inferiori.
- Trattare i sieri animali utilizzati nelle analisi MN con recettore distruggendo enzima (RDE) e inattivare con il calore prima del dosaggio come indicato di seguito.
- Scongelare i sieri a bagnomaria 37 ˚ c e poi metterli su ghiaccio. Mescolare 1 volume di campione di siero animale con 3 volumi di RDE. Incubare a 37 ° c per 18-20 h.
- Inattivare con il calore i sieri RDE trattati a 56 ° c per 30 min. aggiungere 6 volumi di PBS, pH 7,2 a ciascun campione per una pre-diluizione finale di 01:10. Se necessario, archiviare scongelati sieri a 4 ° C per non più di 24 h; Se è necessario più lungo periodo di stoccaggio, memorizzare sieri congelati a-20 ° C o a temperature inferiori.
Nota: Sieri animali possono contenere vari glicani acido sialico che possono associare a HAs dei virus influenzali e inibire il legame di riossidazione specifici anti-HA gli anticorpi. Pertanto, è necessario RDE trattare tutti i sieri animali prima i saggi di MN per rimuovere questi leganti HA virali aspecifico nei campioni di siero.
2. passaggio della coltura delle cellule MDCK-SIAT1
Nota: Tutte le colture di cellule devono essere eseguite in un armadietto per prevenire la contaminazione di sicurezza biologica.
- Decantare il mezzo di coltura cellulare da strato monomolecolare della cellula in boccette2 162 cm. Tripsinizzano le cellule risciacquando il monostrato con tripsina-EDTA. Aggiungere 5 mL di tripsina-EDTA per coprire strato monomolecolare della cellula. Incubare a 37 ° C, 5% CO2 finché il monostrato si stacca (5-10 min). Aggiungere 15 mL di terreno di coltura delle cellule MDCK-SIAT1 ciascun matraccio contenente cellule tripsinizzate, pipetta su e giù per celle separate.
- Utilizzare un emocitometro e trypan blu per determinare che la cella conta e vitalità. Le cellule in nuove boccette di coltura del tessuto2 162 cm contenente 30 mL di terreno di coltura delle cellule con 2-2,5 x 106 cellule /flask di semi e incubare a 37 ° C con 5% CO2 per 2 giorni per l'uso nelle analisi TCID e MN. Celle a 4-5 x 106 cellule/boccetta di semi e incubare a 37 ° C con 5% CO2 per 2 giorni per l'uso nella propagazione di virus.
Nota: La densità di semina può essere regolata se periodi più o meno lunghi di cultura sono desiderati. Per la propagazione di virus, cellule dovrebbero raggiungere oltre 100% confluency. Per le analisi di coltura del tessuto infezione Dose (TCID) e MN, le cellule dovrebbero essere al confluency 75-95% (Figura 1).
3. la propagazione del virus A(H3N2) nelle cellule MDCK-SIAT1
Nota: A(H3N2) virus possono essere propagati in 10-11 giorno vecchia embrionate gallina uova secondo il protocollo standard3, o delle cellule MDCK-SIAT1 culture. Le cellule MDCK-SIAT1 dovrebbero raggiungere oltre 100% confluenza per inoculazione di virus. Scorte di sementi di virus può essere inoculate con diluizioni multiple di inoculo. Le diluizioni di inoculo con il miglior raccolto HA e l'infettività può essere utilizzati per ulteriori analisi di MN.
- Rimuovere il mezzo di coltura cellulare da strato monomolecolare della cellula (> 100% confluency) in boccette2 162 cm. Lavare il monostrato due volte con 15 mL di PBS sterile 0,01 M, pH 7,2 e decantare.
- Etichetta 1 boccetta come controllo e aggiungere 20 mL di mezzi di propagazione di virus. Incubare la beuta a 37 ° C con 5% CO2. Lasciare intatto questo matraccio e utilizzare per il confronto dopo 1 giorni di diffusione dei virus.
- Aggiungere 10 mL di mezzi di propagazione di virus sterile per la flask(s) e Incubare tutte le flask(s) a 37 ° C, 5% CO2. Scongelare una fiala di stock di virus di riossidazione a temperatura ambiente, poi mettere sul ghiaccio. Diluire con i media di propagazione del virus per le diluizioni di destinazione.
Nota: Destinazione diluizioni possono essere regolate sulla base HA i titoli delle scorte di virus. Per preparare un 1: 1000 diluito inoculo, aggiungere 100 µ l di virus a 9,9 mL di mezzi di propagazione di virus sterile. Invertire delicatamente, rimuovere 1 mL della diluizione 1: 100 a 9 mL di mezzi di propagazione di virus sterile, capovolgere delicatamente per un inoculo di 1: 1000 diluito. Mettere sul ghiaccio. - Rimuovere tutte le beute tranne boccetta di controllo dall'incubatrice. Rimuovere il supporto di propagazione di virus da flask(s) delle cellule MDCK-SIAT1 e inoculare ciascuna beuta con 10 mL di virus diluito. Incubare le beute a 37 ° C, 5% CO2 per 1 h, ruotando il flask(s) ogni 15 min.
- Togliere il flask(s) 5 mL di inoculo e sostituire con supporto di propagazione di virus 15 mL contenente 1 µ g/mL TPCK-tripsina. Incubare la flask(s) a 37 ° C, 5% CO2 per 16-18 h.
- Dopo l'incubazione overnight, osservare il flask(s) sotto 100 X ingrandimento del microscopio e guardare per gli effetti citopatici (CPE) sulle cellule. Togliere il flask(s) 15-17 mL di surnatante di virus e sostituire con 15-17 mL di terreno di propagazione di virus preparati al momento da 2 µ g/mL TPCK-tripsina.
- Incubare le beute a 37 ° C, 5% CO2. Monitorare il CPE di monostrato (confronto con il pallone di controllo cella) e verifica l'HA periodicamente (ogni 4 h) con 0,75% gpRBC fino alla raccolta.
- Impostare una piastra per microtitolazione HA 96 pozzetti. Aggiungere 50 µ l di PBS 0,01 M, pH 7,2 a pozzetti A2 - A12 e B2 - B12 (duplicati) e pozzetti H1 - H12 per controllo.
- Aggiungere 100 µ l del surnatante virus su A1 e A2, eseguire una diluizione seriale 2 volte da A1 e B1 e B12 e A12. Scartare 50 µ l dopo la miscelazione in A12 e B12. Aggiungere 50 µ l di gpRBCs 0,75% alle righe A, B e H.
- Toccare la piastra e incubare a temperatura ambiente per 1 h.
- Determinare HA di virus surnatante inclinando la micropiastra a 96 pozzetti a 45° di angolo di 60°.
- Leggere piastre per emagglutinazione; la più alta diluizione del virus che raggiunge emagglutinazione completa è considerata il punto finale di titolazione HA per il virus specifico. Registrare il reciproco della diluizione più alta virus con emagglutinazione completa, il titolo HA del virus.
Nota: Globuli rossi si stabilirono nella riga H (controlli) dovrebbero iniziare a "correre" e formare una piccola forma a goccia a causa della gravità. Aspetta che RBCs in controllo pozzi finire in esecuzione, quindi leggere i pulsanti di RBC nel virus pozzi di titolazione. Coloro che presentano i pulsanti RBC e "Esegui", non raggiungere emagglutinazione. Individuare la diluizione massima di virus che inibiscono completamente emagglutinazione come il punto finale HA titolo del virus.
- Raccogliere il virus quando l'HA della cultura virus altipiani o la cultura del virus raggiunge il bersaglio HA (ad esempio, > 16 HAU), ma prima della cella monostrato inizia a visualizzare CPE significativo.
- Centrifugare la coltura di virus surnatante a 300 x g per 10 min a 4 ° C per appallottolare i detriti cellulari. Trasferire il surnatante chiarificato che contiene la raccolta di virus in una provetta pulita. Aliquotare il supernatante contenente il virus raccolto in fiale criogeniche sterili monouso e congelare immediatamente a-70 ° C o più freddo.
Nota: Le scorte di virus utilizzate per le analisi di MN dovrebbero avere alto titolo infettiva e minime particelle difettose. La minima diluizione degli stock di virus per raggiungere 100 TCID5050 µ l per l'analisi di MN è 1: 100.
Nota: Le scorte di virus utilizzate nelle analisi MN non dovrebbero essere scongelate e ricongelate.
- Centrifugare la coltura di virus surnatante a 300 x g per 10 min a 4 ° C per appallottolare i detriti cellulari. Trasferire il surnatante chiarificato che contiene la raccolta di virus in una provetta pulita. Aliquotare il supernatante contenente il virus raccolto in fiale criogeniche sterili monouso e congelare immediatamente a-70 ° C o più freddo.
4. la determinazione della TCID del Virus
- 1 ° giorno: Titolazione del Virus
- Scongelare una fiala di virus a temperatura ambiente e collocare immediatamente sul ghiaccio.
- Testare il virus alle due diverse diluizioni di partenza: 10-2 e 10-3. Aggiungere 100 µ l di virus a 9,9 mL di diluente per la diluizione di 10-2 del virus. Aggiungere 1 mL di diluizione di 10-2 a 9,0 mL di diluente per la diluizione di 10-3 virus.
- Utilizzando due microtitolo piastre (piastra 1 per 10-2 diluizione) e 2 per la diluizione del 10-3 , aggiungere 100 µ l di diluente del virus in tutti i pozzetti tranne colonna 1 della piastra microtiter a 96 pozzetti.
- Eseguire diluizioni di10 ½log (10-2, 10-2,5, 10-3, ecc.). µ L 146 del virus a partire di diluizione in tutti i pozzetti nella colonna 1 e trasferire in serie 46 µ l dalla colonna 1 attraverso colonna 11 (Figura 2). Puntali per pipette cambiamento tra pozzetti. Dopo la miscelazione colonna 11, scartare le punte con il 46 µ l della diluizione.
- Utilizzare la colonna 12 come il controllo delle cellule (CC); Esso contiene solo diluente di virus. Incubare per 1h a 37 ° C, 5% CO2.
- Giorno 1: Preparazione delle cellule MDCK-SIAT1
Nota: Il monostrato di cellule MDCK-SIAT1 dovrebbe raggiungere confluency di 75-95% per saggi TCID e MN. Una boccetta di 162 cm2 al 95% confluency dovrebbe produrre abbastanza cellule per seme ~ 4-5 piastre di microtitolazione.- Lavare il monostrato confluente 75-95% con 20 mL di PBS per rimuovere i FB in terreni di coltura. Tripsinizzano le cellule come segue.
- Sciacquare il monostrato con PBS sterile, aggiungere 7 mL di tripsina-EDTA per coprire strato monomolecolare della cellula. Posare il pallone piatto e incubare a 37 ° C, 5% CO2 finché il monostrato si stacca (circa 5-10 min). Aggiungere 7 mL di diluente virus ciascun matraccio contenente le cellule trypsinized.
- Lavare le cellule con diluente di virus per rimuovere il FBS.
- Pipettare delicatamente su e giù per separare le cellule. Trasferire le cellule in una provetta conica da 50 mL; riempire il tubo con il diluente di virus.
- Centrifugare a 485 x g per 5 min, decantare il virus diluente, sostituire con fresca 50 mL di diluente di virus e centrifugare a 485 x g per 5 min, decantare il virus diluente e con diluente nuovi virus (10ml/boccetta) per risospendere il pellet.
- Utilizzare un emocitometro e blu di trypan per determinare il conteggio delle cellule e la vitalità. Regolare la concentrazione di cellule con il virus di diluente a 1,5 x 105 cellule/mL.
- Aggiungere 100 µ l di cellule MDCK-SIAT1 diluite in ciascun pozzetto di piastre di microtitolazione (1,5 x 104 cellule/pozzetto) e le piastre di copertura. Incubare a 37 ° C, 5% CO2 per 18-20 h.
- Lavare il monostrato confluente 75-95% con 20 mL di PBS per rimuovere i FB in terreni di coltura. Tripsinizzano le cellule come segue.
- Giorno 2: ELISA
-
Fissazione delle cellule
- Rimuovere il mezzo da piastre di microtitolazione. Lavare ogni bene con 200 µ l di PBS. Aggiungere 300 µ l freddo 80% di acetone in ogni pozzetto e incubare a temperatura ambiente per 10 minuti rimuovere il fissativo e lasciare asciugare le piastre all'aria.
-
ELISA
-
Aggiunta di anticorpo primario
Nota: Anticorpo monoclonale anti-influenzale A NP deve essere utilizzato in eccesso come l'anticorpo primario in ELISA. Determinare la diluizione di anticorpo ottimale per ogni sacco di anticorpi primari eseguendo titolazioni degli anticorpi in MN. Selezionare la concentrazione di anticorpo primario che è in eccesso e con il miglior rapporto segnale-sfondo.- Diluire l'anticorpo monoclonale anti-influenzale A NP (anticorpo primario) per la concentrazione target nell'anticorpo diluente (ad es. aggiungere 30 µ l di anticorpo primario a 30 mL di anticorpo diluente per una diluizione di destinazione di 1: 1000).
- Lavare 3 volte con 300 µ l di tampone di lavaggio le piastre. Aggiungere dell'anticorpo primario diluito 100 µ l a ciascun pozzetto. Incubare a temperatura ambiente per 1 h.
-
Aggiunta dell'anticorpo secondario
Nota: Capra anti mouse IgG coniugato al rafano perossidasi (HRP) dovrebbero essere usato in eccesso come l'anticorpo secondario in ELISA. Determinare la diluizione di anticorpo ottimale per ogni sacco di anticorpi secondari eseguendo titolazioni degli anticorpi. Selezionare la concentrazione di anticorpo secondario in eccesso e con il miglior rapporto segnale-sfondo.- Diluire il anti-topo di capra IgG coniugato all'anticorpo HRP (anticorpo secondario) per la concentrazione target nell'anticorpo diluente (ad es. aggiungere 7,5 µ l di anticorpo secondario a 30 mL di anticorpo diluente per una diluizione di 1: 4000 di destinazione).
- Lavare le piastre 3 volte con 300 µ l di tampone di lavaggio. Aggiunta dell'anticorpo secondario diluito 100 µ l a ciascun pozzetto. Incubare a temperatura ambiente per 1 h.
-
Aggiunta di substrato e lettura targhe
- Lavare le piastre 5 volte con 300 µ l di tampone di lavaggio e premete su un panno privo di lanugine.
- Aggiungere 100 µ l di substrato appena preparata in ogni pozzetto e incubare a temperatura ambiente fino a quando lo sviluppo di colori saturi e la densità ottica (OD) di pozzetti di controllo cella < 0.2.
- Aggiungere 100 µ l di soluzione bloccante ai pozzetti. Leggere il diametro esterno di pozzetti a 490 nm utilizzando uno spettrofotometro per micropiastre.
-
Aggiunta di anticorpo primario
-
Fissazione delle cellule
- Calcolo di TCID 50
- Calcolare la mediana OD490 dei controlli delle celle (colonna 12).
- Prendere in considerazione qualsiasi prova bene con un OD490 maggiore di due volte la mediana OD490 dei pozzetti CC come "positivo"; in caso contrario, è considerato "negativo".
- Calcolare il TCID50 del virus usando il metodo Reed-Muench13.
- Determinare il numero di lati positivi e negativi ad ogni diluizione.
- Calcolare il "cumulativo positivo", "Cumulativo negativo", "Rapporto" e "% positivo" come illustrato nella tabella 1.
- Calcolare la "distanza proporzionale" tra la proiezione di diluizione > 50% positivi e la proiezione di diluizione < 50% positivi utilizzando il seguente:
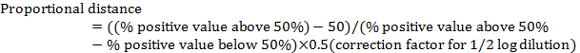
Nota: Il fattore di correzione per la diluizione del registro ½ è 0,5. Ad esempio, nella tabella 1: (80 − 50) /(80 − 20) x 0,5 = 0,25 - Calcolare il virus TCID50 aggiungendo la distanza proporzionale alla diluizione risultati positivi > 50%.
Nota: ad esempio, in tabella 2, TCID50 è 10-5+(-0.25) = 10-5.25. Nota che questo è il TCID50 del virus per 100 µ l (o 10-5.25100 µ l).
- Calcolare la diluizione del virus. Per i dosaggi MN, diluire il virus a 200 TCID50100 µ l (equivalente a 100 TCID5050 µ l per pozzetto).
Nota: Nell'esempio in tabella 1, 1 TCID50 è 10-5.25 in 100 µ l e la diluizione per ottenere 200 TCID50100 µ l è 1:891 sulla base dei calcoli: 200 x 10-5.25 = 10-2.95 = 1/102,95 = 1 / 891
5. MN analisi usando le cellule MDCK-SIAT1
-
Giorno 1: Test sieri preparazione e piastra di layout del controllo
- Scongelare i sieri in bagno di acqua di 37 ° C e rimuovere immediatamente dopo lo scongelamento. Aliquotare la quantità dei sieri che deve essere testato; un minimo di 10 µ l di sieri originale è necessario per testare con un virus in canottiera. Testare i sieri in duplicati se possibile.
- I sieri umani per 30 min in un bagno di acqua di 56 ° C come descritto al punto 1.12.1 inattivare con il calore. Sieri di posto sul ghiaccio post inattivazione termica, aggiungere il diluente di virus al sera per raggiungere un 01:10 pre-diluizione.
- RDE trattare e pre-diluire i sieri animali a 01:10 da utilizzare per i controlli al 1.12.2.
Nota: Scongelati e trattati sieri umani e animali possono essere conservati a 4 ° C per non più di 24 h. sieri devono essere conservati congelati a-20 ° C o più freddo se più lungo periodo di stoccaggio è necessario prima del dosaggio. - Per testare con un virus, aggiungere 100 µ l 01:10 diluito sieri alle colonne da A1 a A10 (Figura 4). Aggiungere 50 µ l di diluente del virus a righe B attraverso H, ad eccezione di colonne 11 e 12 (Figura 4). Eseguire una diluizione seriale 2 volte da riga A attraverso H e scartare le ultime 50 µ l a riga H (Figura 4).
Nota: Quando più virus devono essere testati, sieri possono essere diluiti in tubi di titolo. La diluizione del siero, per lo scopo di determinare il titolo del siero, in un pozzo è 01:10, Beh B 01:20, Beh C 01:40, ben D 1: 80, ben E 1: 160, Beh F 1: 320, ben 1: 640 G, ben H 1:1,280 (Figura 4). - Per il controllo di virus, aggiungere 50 µ l di diluente del virus a pozzetti A12, B12, C12 e D12 (no sera). Per il controllo di cella, aggiungere 100 µ l di diluente del virus a pozzetti E12, F12, G12 e H12 (nessun virus, nessun sieri).
- Per i sieri di controllo (ad esempio, controlli di sieri di furetto), aggiungere 100 µ l di sieri di controllo diluito per bene una colonna 11 e aggiungere 50 µ l di diluente del virus a pozzetti B11 - H11. Serie diluito giù.
- Le piastre di copertura, incubare a 37 ° C, 5% CO2 fino a che pronto per l'aggiunta di virus.
-
1 giorno: Aggiunta Virus
- Diluire il virus a 100 TCID5050 µ l con virus diluente.
- Aggiungere 50 µ l virus diluito in tutti i pozzetti, ad eccezione di colonna 11 sulle piastre di titolazione di ritorno (BT) e i pozzetti di controllo cella E12, F12, G12 e H12 su tutte le lastre (Figura 4). Per le matrici con sieri di controllo a colonna 11, aggiungere virus colonna 11.
- Titolazione di ritorno (BT)
- Includere un BT nella colonna 11 di un set di piastre duplicati (ad esempio, piastra 1A e 1B). Aggiungere 50 µ l di diluente del virus in tutti i pozzetti nella colonna 11. Aggiungere 50 µ l del virus a 100 TCID5050 µ l per il primo pozzo (A11). Mescolare pipettando su e giù.
- Mescolare e trasferire 50 µ l a pozzetti successivi per eseguire diluizioni seriali 2 volte. Cambiare le punte di pipetta tra pozzetti per evitare il riporto di virus. Scartare 50 µ l dal ben H11.
- Aggiungere 50 µ l di diluente a colonna 11 per portare il volume finale di 100 µ l. virus toccare le piastre per mescolare.
- Incubare le piastre a 37 ° C, 5% CO2 per 1 h.
- Eseguire l'aggiunta delle cellule MDCK-SIAT1 il giorno 1 ed ELISA il giorno 2 come descritto ai punti 4.2 e 4.3 (anche descritto ai punti 5.3 e 5.4)
-
1 ° giorno: Aggiunta di cellule MDCK-SIAT1
- Preparare le cellule MDCK-SIAT1 come descritto al punto 4.2. Aggiungere 100 µ l MDCK-SIAT1 celle a 1,5 x 105 cellule/mL in ciascun pozzetto (1,5 x 104 cellule/pozzetto). Incubare le piastre a 37 ° C, 5% CO2 per 18-20 h.
-
Giorno 2: ELISA
- Dopo l'incubazione overnight, il secondo giorno, è possibile fissare le cellule con 80% di acetone freddo come descritto al punto 4.3.
-
Aggiunta di anticorpo primario
- Lavare le piastre 3 volte con 300 µ l di tampone di lavaggio. Diluire l'anticorpo monoclonale anti-influenzale A NP (anticorpo primario) alla concentrazione ottima come determinato per titolazione in diluente per anticorpi (per esempio aggiungere 30 µ l di anticorpo primario a 30 mL di anticorpo diluente per una diluizione di destinazione di 1: 1000).
Nota: 100 µ l di anticorpo primario è necessaria per pozzetto. ~ 10 mL è necessario per ciascuna piastra. - Aggiungere dell'anticorpo primario diluito 100 µ l a ciascun pozzetto. Incubare a temperatura ambiente per 1 h.
- Lavare le piastre 3 volte con 300 µ l di tampone di lavaggio. Diluire l'anticorpo monoclonale anti-influenzale A NP (anticorpo primario) alla concentrazione ottima come determinato per titolazione in diluente per anticorpi (per esempio aggiungere 30 µ l di anticorpo primario a 30 mL di anticorpo diluente per una diluizione di destinazione di 1: 1000).
-
Aggiunta dell'anticorpo secondario
- Lavare 3 volte con 300 µ l di tampone di lavaggio le piastre. Diluire la capra anti mouse IgG coniugata con un anticorpo HRP (anticorpo secondario) per la concentrazione target nel diluente dell'anticorpo (ad esempio aggiungere 7,5 µ l di anticorpo secondario a 30 mL di anticorpo diluente per un obiettivo 1: 4000.)
Nota: 100 µ l di anticorpo secondario è richiesto a 96. ~ 10 mL è necessario per ciascuna piastra. - Aggiunta dell'anticorpo secondario diluito 100 µ l a ciascun pozzetto. Incubare a temperatura ambiente per 1 h.
- Lavare 3 volte con 300 µ l di tampone di lavaggio le piastre. Diluire la capra anti mouse IgG coniugata con un anticorpo HRP (anticorpo secondario) per la concentrazione target nel diluente dell'anticorpo (ad esempio aggiungere 7,5 µ l di anticorpo secondario a 30 mL di anticorpo diluente per un obiettivo 1: 4000.)
-
Aggiunta di substrato e lettura targhe
- Lavare le piastre 5 volte con 300 µ l di tampone di lavaggio e toccare il panno privo di lanugine.
- Aggiungere 100 µ l di substrato appena preparata in ogni pozzetto e incubare a temperatura ambiente fino a raggiungono i pozzetti di controllo del virus un OD490 = 0,8 - 3, con il controllo di cella a un livello basso sfondo OD490 < 0.2.
- Aggiungere 100 µ l della stop solution in tutti i pozzetti. Leggere il diametro esterno di pozzetti a 490 nm utilizzando uno spettrofotometro per micropiastre.
-
Analisi dei dati
Nota: I calcoli di MN sono determinati individualmente per ogni piatto.- Determinare il titolo anticorpale neutralizzante di ogni campione di siero.
- Calcolare il cut-off di490 OD di neutralizzazione del virus di 50% per ogni piatto usando la seguente equazione:
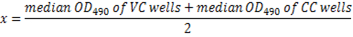
Nota: Qui x = 50% della linea di demarcazione neutralizzazione. La diluizione del siero reciproco corrispondente alla diluizione più alta con OD490 meno del 50% della linea di demarcazione (≥ 50% inibizione) è considerato il titolo dell'anticorpo di neutralizzazione per quel campione di siero (Figura 5).
- Calcolare il cut-off di490 OD di neutralizzazione del virus di 50% per ogni piatto usando la seguente equazione:
- Controllare che i pozzetti di controllo delle cellule hanno un OD490 < 0,2 e pozzetti di controllo del virus hanno un OD490 = 0,8 - 3.
- Verificare che l'infettività del virus in ogni dosaggio (100 x TCID50) da virus BT nell'intervallo accettabile di BT è 50, 100 o 200 TCID. Se si utilizza il cut-off stesso come definito al punto 4.4.2, la più alta diluizione del virus nella colonna BT con OD sopra il taglio dovrebbe essere a wells E11, F11, G11.
Nota: I controlli positivi del siero dovrebbero dare 2 volte i titoli all'interno dei valori ottenuti nei test precedenti. Il OD490 del controllo negativo del siero dovrebbe essere simile a quello osservato per il controllo di virus.
- Determinare il titolo anticorpale neutralizzante di ogni campione di siero.
Risultati
Determinazione di infettività delle scorte virus è il primo passo nell'analisi della MN. La figura 2 illustra il layout della piastra per determinare il TCID50 degli stock di virus. Per gli stock di virus con infettività sconosciuto, il virus può essere titolato da pre-diluizioni multiple, ad esempio 10-2 e 10-3, al fine di acquisire la migliore curva di titolazione per calcolare infettività del virus. La quantità di vir...
Discussione
Il dosaggio di MN è uno dei principali saggi utilizzati per i test sierologici dell'influenza per rilevare le risposte di anticorpi dell'influenza infezione o la vaccinazione. I titoli generati da saggi di MN sono spesso utilizzati come il risultato primario di molti studi di seroepidemiology di riossidazione. MN saggi sono anche ampiamente utilizzati per la sero-diagnosi e la valutazione dell'immunogenicità del vaccino. Internazionali inter-laboratorio studi sono stati condotti per confrontare saggi MN eseguite in pi?...
Divulgazioni
Gli autori non segnalano alcun conflitto di interessi. Le risultanze e le conclusioni in questa pubblicazione sono quelle degli autori e non rappresentano necessariamente le opinioni dei centri per controllo di malattia, la prevenzione e l'agenzia di finanziamento.
Riconoscimenti
Ringraziamo Dr. Xiuhua Lu, Dr. Feng Liu e MS. Ashley Burroughs dalla divisione dell'Influenza del CDC per la loro revisione critica e assistenza nella preparazione di questo manoscritto. Si ringrazia il Dr. Adrian Reber dalla divisione dell'Influenza del CDC per la sua assistenza nel preparare la grafica della Figura 3. Infine, desideriamo ringraziare il Dr. M. Matrosovich, Marburg, in Germania, per fornire le cellule MDCK-SIAT1.
Materiali
| Name | Company | Catalog Number | Comments |
| Dulbecco’s Modified Eagle Medium (DMEM) with high Glucose | Life Science | 11965 | A critical component of Sterile Cell Culture, Virus Propagation and Virus Diluent Media |
| Fetal bovine serum (FBS) | Hyclone | SH30070.03 | |
| Bovine Serum Albumin (BSA) Fraction V, Protase Free | Sigma-Aldrich | 3117332001 | |
| L-Glutamine | Life Science | 25030-081 | |
| Sodium pyruvate | Life Science | 11360-070 | |
| Geneticin G-418 disulfate salt | Sigma-Aldrich | A1720-5G | |
| HEPES | Life Science | 15630-080 | |
| Penicillin/Streptomycin | Life Science | 15140-122 | |
| Acetone | VWR | 67-64-1 | Used at an 80% concentration |
| Phosphate-Citrate Buffer with Sodium Perborate | Sigma-Aldrich | SLBF2806V | |
| O-Phenylenediamine Dihydrochloride tablet | Sigma-Aldrich | SLBQ1086V | 1 tablet per 100ml of cell culture grade water |
| Sulfuric Acid | Fisher Scientific | A510-P500 | Used 0.5M final concentration |
| Ethanol, Denatured, 4L | VWR | EM-AX0422-3 | Used at an 70% concentration |
| Trypsin-EDTA | Life Science | 1748048 | |
| RDE II "Seiken" | Denka Seiken | 370013 | |
| Tween 20 | Sigma-Aldrich | P1379-500ml | |
| Anti-NP mouse monoclonal Ab | Millipore pool | MAB 8257 MAB 8258 | |
| Anti-mouse IgG HRP | KPL | 074-1802 | |
| 96-well flat-bottom plates | Thermo Scientific | 3455 | |
| Plate reader | Molecular Device | Spectromax 384 plus | |
| Cell Culture Flask 162 cm2/Vent Cap | Corning/VWR | 3151 |
Riferimenti
- Reber, A., Katz, J. Immunological assessment of influenza vaccines and immune correlates of protection. Expert Rev of Vaccines. 12, 519-536 (2013).
- Li, C. K., Rappuoli, R., Xu, X. N. Correlates of protection against influenza infection in humans--on the path to a universal vaccine?. Curr Opin in Immunol. 25, 470-476 (2013).
- WHO Global Influenza Surveillance Network. . Manual for the labratory diagnosis and virological surveillence of influenza. , (2011).
- Govorkova, E. A., Kodihalli, S., Alymova, I. V., Fanget, B., Webster, R. G. Growth and immunogenicity of influenza viruses cultivated in Vero or MDCK cells and in embryonated chicken eggs. Dev Biological Stand. 98, 39-51 (1999).
- Levine, M. Z., et al. Neutralizing Antibody Responses to Antigenically Drifted Influenza A(H3N2) Viruses among Children and Adolescents following 2014-2015 Inactivated and Live Attenuated Influenza Vaccination. Clin Vaccine Immunol : CVI. 23, 831-839 (2016).
- Lin, Y., et al. The characteristics and antigenic properties of recently emerged subclade 3C.3a and 3C.2a human influenza A(H3N2) viruses passaged in MDCK cells. Influenza Other Respir Viruses. , (2017).
- Lin, Y. P., et al. Neuraminidase receptor binding variants of human influenza A(H3N2) viruses resulting from substitution of aspartic acid 151 in the catalytic site: a role in virus attachment?. J of Virol. 84, 6769-6781 (2010).
- Skowronski, D. M., et al. Mutations acquired during cell culture isolation may affect antigenic characterisation of influenza A(H3N2) clade 3C.2a viruses. Euro Surveillance. 21, 30112 (2016).
- Matrosovich, M., Matrosovich, T., Carr, J., Roberts, N. A., Klenk, H. D. Overexpression of the alpha-2,6-sialyltransferase in MDCK cells increases influenza virus sensitivity to neuraminidase inhibitors. J of Virol. 77, 8418-8425 (2003).
- Oh, D. Y., Barr, I. G., Mosse, J. A., Laurie, K. L. MDCK-SIAT1 cells show improved isolation rates for recent human influenza viruses compared to conventional MDCK cells. J Clin Microbiol. 46, 2189-2194 (2008).
- US Department of Health and Human Services. . Biosafety in Microbiological and Biomedical Laboratories. , (2009).
- Reed, H. M. A simple method of estimating fifty percent endpoints. The American Journal of Hygiene. 27, (1938).
- Laurie, K. L., et al. International Laboratory Comparison of Influenza Microneutralization Assays for A(H1N1)pdm09, A(H3N2), and A(H5N1) Influenza Viruses by CONSISE. Clinical Vaccine Immunol : CVI. 22, 957-964 (2015).
- Lin, Y., Gu, Y., McCauley, J. W. Optimization of a Quantitative Micro-neutralization Assay. J Vis Exp : JoVE. , (2016).
- van Baalen, C. A., et al. ViroSpot microneutralization assay for antigenic characterization of human influenza viruses. Vaccine. 35, 46-52 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon