Method Article
Una descrizione cartografica 3D della cellula mediante tomografia a raggi X Cryo Soft
In questo articolo
Riepilogo
Qui viene presentato un protocollo che descrive le fasi di preparazione del campione e di raccolta dei dati richieste nella tomografia a raggi X criomoscido morbido (SXT) per visualizzare l'ultrastruttura di intere cellule crioconservate con una risoluzione di 25 nm a mezzo passo.
Abstract
Le tecniche di imaging sono fondamentali per comprendere l'organizzazione cellulare e i macchinari nella ricerca biologica e nei campi correlati. Tra queste tecniche, la tomografia criomosa morbida a raggi X (SXT) consente di imaging di intere cellule crio-conservate nell'intervallo di energia a raggi X della finestra dell'acqua (284-543 eV), in cui le strutture in carbonio hanno un assorbimento intrinsecamente superiore rispetto all'acqua, consentendo la ricostruzione 3D del coefficiente di assorbimento lineare del materiale contenuto in ciascun voxel. Le informazioni strutturali quantitative a livello di intere celle fino a 10 μm di spessore sono quindi realizzabili in questo modo, con un elevato throughput e una risoluzione spaziale fino a 25-30 nm di mezzo passo. Cryo-SXT si è dimostrato rilevante per l'attuale ricerca biomedica, fornendo informazioni 3D sui processi di infezione cellulare (virus, batteri o parassiti), cambiamenti morfologici dovuti a malattie (come le malattie genetiche recessive) e aiutandoci a comprendere l'azione dei farmaci a livello cellulare o localizzando strutture specifiche nell'ambiente cellulare 3D. Inoltre, sfruttando la lunghezza d'onda sintonizzabile presso gli impianti di sincrotrone, la spettromicroscopia o la sua controparte 3D, la spettrotomografia, può essere utilizzata anche per visualizzare e quantificare elementi specifici nella cellula, come il calcio nei processi di biomineralizzazione. Cryo-SXT fornisce informazioni complementari ad altre tecniche di imaging biologico come la microscopia elettronica, la fluorescenza a raggi X o la fluorescenza a luce visibile ed è generalmente utilizzato come metodo partner per l'imaging correlato 2D o 3D in condizioni criogeniche al fine di collegare funzione, posizione e morfologia.
Introduzione
Cryo-SXT può svolgere un ruolo centrale nella ricerca sull'imaging biologico in quanto fornisce volumi 3D a media risoluzione (25-30 nm mezzo passo) di cellule intere idratate 1,2,3,4,5,6. Nell'intervallo di energia della finestra dell'acqua, tra i bordi K di assorbimento del carbonio e dell'ossigeno (4,4-2,3 nm), le strutture cellulari ricche di carbonio assorbono 10 volte di più del mezzo ricco di ossigeno che le permea e le circonda. In questo intervallo di energia, le celle vetrificate fino a 10 μm di spessore possono essere fotografate senza la necessità di sezionamento o colorazione, portando a proiezioni quantitative ad alto contrasto di assorbimento, che, combinate con le capacità di rotazione del campione, consentono la ricostruzione tomografica della struttura cellulare. Cryo-SXT riempie una nicchia in termini di dimensioni del campione e risoluzione spaziale che non è facilmente accessibile con nessun'altra tecnica di imaging.
In breve, il contrasto di assorbimento di cryo-SXT è quantitativo, in quanto l'attenuazione dei fotoni attraverso il campione di spessore t obbedisce alla legge di Beer-Lambert come segue: 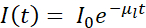 , dove I0 rappresenta l'intensità incidente e μl il coefficiente di assorbimento lineare, che dipende dalla lunghezza d'onda λ e dalla parte immaginaria β dell'indice di rifrazione del campione (
, dove I0 rappresenta l'intensità incidente e μl il coefficiente di assorbimento lineare, che dipende dalla lunghezza d'onda λ e dalla parte immaginaria β dell'indice di rifrazione del campione (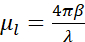 ). L'attenuazione è una funzione della composizione biochimica e dello spessore delle strutture che vengono fotografate, con ogni componente biochimico che ha uno specifico coefficiente di assorbimento lineare dei raggi X μl (LAC). Ciò significa che ogni valore di voxel per tomografia dipende dagli elementi chimici e dalla loro concentrazione in quel voxel7. Ciò consente la discriminazione naturale di diversi organelli come nuclei, nucleoli, corpi lipidici o mitocondri, o diversi stati di compattazione della cromatina esclusivamente in base ai loro valori LAC intrinseci ricostruiti 2,8,9.
). L'attenuazione è una funzione della composizione biochimica e dello spessore delle strutture che vengono fotografate, con ogni componente biochimico che ha uno specifico coefficiente di assorbimento lineare dei raggi X μl (LAC). Ciò significa che ogni valore di voxel per tomografia dipende dagli elementi chimici e dalla loro concentrazione in quel voxel7. Ciò consente la discriminazione naturale di diversi organelli come nuclei, nucleoli, corpi lipidici o mitocondri, o diversi stati di compattazione della cromatina esclusivamente in base ai loro valori LAC intrinseci ricostruiti 2,8,9.
Inoltre, cryo-SXT è una tecnica ad alto rendimento con tomogrammi raccolti in pochi minuti. Ciò consente specificamente l'imaging a mesoscala di popolazioni cellulari che possono essere catturate in punti temporali chiave come divisione, differenziazione e apoptosi, ma anche in diversi stati di risposta come quelli indotti dall'esposizione chimica a specifiche terapie farmacologiche o a infezioni patogene. I dati raccolti in questi punti chiave forniranno una descrizione 3D del sistema con una registrazione fedele dell'organizzazione spaziale dei diversi organelli cellulari in quei momenti specifici.
Di solito, cryo-SXT viene utilizzato in combinazione con altre tecniche che seguono approcci correlativi che consentono di localizzare caratteristiche, eventi o macromolecole specifiche all'interno dell'ambiente cellulare 3D 4,10,11,12,13,14,15,16 o dati di fluorescenza a raggi X duri17,18 . Gli approcci correlativi in condizioni criogeniche sono di fondamentale importanza al fine di ottenere il quadro più completo e prezioso del sistema di interesse. Un breve riassunto del tipico flusso di lavoro delle linee di fascio crio-SXT Mistral (Alba) e B24 (Diamond) è delineato nella Figura 1.
Inoltre, sfruttando la capacità di sintonizzazione della lunghezza d'onda presso gli impianti di sincrotrone, è possibile ottenere informazioni spettroscopiche in aggiunta a quella strutturale utilizzando l'assorbimento differenziale specifico di particolari elementi contenuti nel campione. Un esempio di questo potrebbe essere la posizione del calcio nello studio dei processi di biomineralizzazione nelle cellule 19,20,21. Scattando immagini 2D a diverse energie fotoniche (spettri) o tomogrammi sottostanti e sul bordo di assorbimento dei raggi X di interesse, è possibile identificare i pixel o i voxel contenenti l'elemento selezionato. Gli spettri consentono anche di differenziare gli stati chimici (cioè l'evoluzione del calcio amorfo in idrossiapatite come nel precedente esempio di biomineralizzazione20). La quantificazione di diversi elementi è possibile in 2D e 3D. L'imaging spettroscopico delle cellule vetrificate viene in genere eseguito nella finestra dell'acqua, ma è anche possibile in altri intervalli di energia se il contenuto di acqua è abbastanza basso o se vengono utilizzati altri protocolli di preparazione del campione, inclusa la disidratazione, 22. Un protocollo dettagliato di spettroscopia passo-passo è al di là del focus del protocollo qui.
In quanto segue, il protocollo si concentra sul riepilogo breve delle principali fasi di preparazione del campione, sebbene ogni sistema potrebbe aver bisogno di un perfezionamento individuale, seguito da una dettagliata procedura di raccolta dati passo-passo per la tomografia criomosola a raggi X.
Protocollo
1. Preparazione del campione
- Preparazione del supporto della rete
- Irradiare le griglie con UV per 3 ore con il film di carbonio rivolto verso l'alto per la sterilizzazione.
- Facoltativo: in caso di problemi con celle non collegate alla griglia, attenersi a una delle seguenti operazioni.
- Idrofilizzare la lamina di supporto al carbonio mediante trattamento al plasma delle griglie per aumentare la diffusione del campione e una migliore adesione cellulare.
- Posizionare le griglie di carbonio verso l'alto nell'apparecchiatura della camera di scarico a bagliore ed esporre la griglia al plasma per 30 s a 15 minuti (utilizzando Ar o / e O2) a seconda dell'apparecchiatura.
- Funzionalizzare le griglie con Poly-L-lysine (PLL)
- Aggiungere singole gocce di PLL da 60 μL in una capsula di Petri e posizionare la griglia sopra la goccia PLL con il film di carbonio rivolto verso il basso. Incubare per 30 minuti a 37 °C e tamponare il PLL con carta da filtro.
- Funzionalizzare le griglie con siero bovino fetale (FBS).
- Immergi le griglie in FBS durante la notte. Immergere le griglie in una soluzione tampone per lavare e lasciare la griglia su una carta da filtro per rimuovere il liquido in eccesso.
- Idrofilizzare la lamina di supporto al carbonio mediante trattamento al plasma delle griglie per aumentare la diffusione del campione e una migliore adesione cellulare.
- Cellule aderenti in crescita su griglie
- Far crescere le cellule in una capsula di coltura cellulare di 100 mm per raggiungere l'80% -90% di confluenza (Figura 2A).
- Semina 1-5 x 105 celle/mL (regola il valore al sistema) sopra le griglie Au in una capsula di Petri P60 (3 mL totali).
NOTA: L'aggiunta della sospensione cellulare deve essere fatta con molta attenzione con il film di carbonio della griglia rivolto verso l'alto in una parabola di coltura cellulare di 60 mm. Preparare diverse griglie per condizione, una condizione per capsula di Petri P60 (Figura 2B). - Lasciare che le celle si depositino fino a quando la confluenza sulla griglia raggiunge diverse celle (da 1 a 10 a seconda delle dimensioni della cella) in ogni quadrato di mesh (a seconda della linea cellulare, questo può richiedere fino a 24 ore).
NOTA: Prima del congelamento, le griglie devono essere controllate con un microscopio a luce visibile (VLM), valutando l'integrità del film di carbonio e la confluenza cellulare in ciascuna griglia (Figura 2C). Attendere fino a quando non viene raggiunta la densità di cella corretta sulla griglia. Se la griglia è troppo confluente o la lamina di carbonio è rotta, ricominciare da capo.
- Deposizione di celle in sospensione su griglie
- Scegliere una griglia Au o Cu preparata usando la pinzetta dal congelatore a immersione (vedere paragrafo 1.6).
- Preparare una sospensione di 1-5 x 105 celle (assorbanza della densità ottica di 0,3 in caso di batteri) e aggiungere 4 μL della sospensione preparata alla griglia.
- Incubare la griglia con la goccia per alcuni minuti orizzontalmente per consentire la deposizione delle cellule e quindi posizionare la pinzetta che tiene la griglia all'interno della camera di vetrificazione climatizzata impostata sulle condizioni di temperatura e umidità appropriate (passaggio 1.6.1).
- Opzionale: marcatura fluorescente dei campioni
NOTA: Può essere utile etichettare fluorescentemente alcuni organelli del campione. A seconda dell'interesse, possono essere utilizzati fluorofori specifici o cellule che esprimono stabilmente una proteina fluorescente o trasfettate per l'espressione transitoria di una proteina di interesse. Ciò consente un facile rilevamento delle cellule utilizzando la microscopia a crio-epifluorescenza e aiuta a trovare le cellule di interesse per la successiva imaging a raggi X. In quanto segue, il protocollo descrive solo l'uso dei fluorofori.- Preparare una soluzione di lavoro per il fluoroforo (vedere la raccomandazione del produttore) e seguire il protocollo specifico.
- Aggiungere i fluorofori alla capsula di Petri contenente le griglie e mescolare delicatamente.
- Lasciare incubare in un ambiente buio e andare direttamente al passaggio 1.6 al termine dell'incubazione.
NOTA: è importante essere pronti a vetrificare una volta terminata l'incubazione per evitare tag non specifici e di conseguenza segnali fluorescenti sfocati.
- Preparazione di nanoparticelle (NP) per l'allineamento della proiezione del tomogramma
- Prendere un'aliquota di 1mL della soluzione madre fiduciaria Au (100 nm a Mistral o 250 nm a B24) e centrifugare a bassa velocità (per evitare l'aggregazione) per 1 min per consentire alle NP di pellettizzare.
NOTA: Se possibile, si preferisce lasciare che i fiduciari si sistemino naturalmente durante la notte o più, per evitare l'aggregazione. - Rimuovere il surnatante. Immediatamente prima del congelamento, sospendere nuovamente le NP in un mezzo privo di siero da 20 μL o in una soluzione tampone per ottenere una soluzione omogenea.
NOTA: La sonicazione e il vortice sono raccomandati per aiutare a omogeneizzare la soluzione.
- Prendere un'aliquota di 1mL della soluzione madre fiduciaria Au (100 nm a Mistral o 250 nm a B24) e centrifugare a bassa velocità (per evitare l'aggregazione) per 1 min per consentire alle NP di pellettizzare.
- Griglie di congelamento a tuffo
ATTENZIONE: l'azoto liquido può causare ustioni da freddo e devono essere indossati adeguati dispositivi di protezione (cappotto da laboratorio lungo, occhiali di sicurezza, guanti, pantaloni lunghi e scarpe chiuse). L'etano è altamente esplosivo e dovrebbe essere tenuto lontano da scintille o fuoco aperto.- Seguire le istruzioni del produttore per preparare e utilizzare lo strumento di congelamento a tuffo.
NOTA: L'umidità è solitamente impostata su 80% -90%; la temperatura dipenderà dal tipo di cellula (lievito 30 °C massimo, cellule di mammifero 37 °C, cellule di insetti 28 °C, ecc.). - Prendi una griglia con le pinzette di montaggio dalla capsula di Petri vicino al bordo, facendo molta attenzione a non piegare la griglia e montarla sul dispositivo di congelamento a tuffo. Assicurarsi che le cellule siano rivolte lontano dalla carta assorbente.
- Opzionale: lavare la griglia tre volte in buffer nel caso di celle aderenti.
- Aggiungere 1,5 μL di fiduciali Au NP sulla parte superiore delle celle (attraverso il foro sul lato destro della camera) e lasciare riposare per 30 s prima di tamponare e immergere la griglia.
NOTA: Nel caso di celle in sospensione, aggiungere i fiduciali Au NP alla griglia mentre è ancora in posizione orizzontale prima di montare la pinzetta e lasciarli depositare per 30 s. Il blotting è fondamentale per griglie di alta qualità. La distanza di blotting deve essere calibrata prima di iniziare; la planarità della carta assorbente è importante per il blotting riproducibile (seguire le istruzioni del produttore). Il tempo di blotting deve essere valutato per ogni tipo di cellula. - Preparare diverse griglie per condizione. Trasferire la griglia e conservare a temperature criogeniche per preservare la vetrificazione.
- Seguire le istruzioni del produttore per preparare e utilizzare lo strumento di congelamento a tuffo.
- Griglie di screening con microscopia a luce criovisibile
- Trasferire le griglie sotto azoto liquido dalle crio-scatole a una crio-cassetta standard (tre posizioni per griglie TEM da 3 mm) all'interno di uno stadio crio-raffreddato (vedere le istruzioni del produttore).
- La criocassetta viene posizionata sul ponte criostadio e lo stadio criogenico è montato su un microscopio ottico a epifluorescenza ad ampio campo.
- Immagine delle griglie a -196,5 °C utilizzando un obiettivo a lunga distanza di lavoro (10x, 50x, 100x) per individuare le celle adatte per l'imaging crio-SXT e valutare la qualità della griglia (presenza di ghiaccio spesso, integrità del film di supporto del carbonio, presenza di segnale fluorescente, ecc.).
- Localizzare le cellule di interesse utilizzando il campo luminoso e/o l'imaging a fluorescenza.
NOTA: in questa fase, acquisire le immagini se sul microscopio è presente una fotocamera collegata. Usalo per la correlazione delle immagini. - Dopo lo screening, restituire le griglie alle crio-scatole in un dewar di stoccaggio di azoto liquido.
NOTA: Se non vengono trovati campioni validi, ripetere le fasi di preparazione del campione modificando uno qualsiasi dei parametri. Di solito, i parametri che richiedono modifiche sono il tempo di blotting, nel caso in cui il ghiaccio sia troppo spesso o la confluenza, se ci sono troppe celle per quadrato di mesh.
2. Caricamento nel microscopio a raggi X a trasmissione (TXM)
- Raffreddare la camera di trasferimento con azoto liquido fino a raggiungere <100 K, raffreddare la postazione di lavoro (Figura 3A) e accendere il riscaldatore del bordo della stazione di lavoro. Attendere fino a quando non smette di bollire.
- Inserire le crio-scatole contenenti griglie necessarie nelle posizioni corrispondenti nella workstation (Figura 3A). Presta attenzione a trasferirli in modo sicuro in condizioni criografiche.
- Caricare le griglie selezionate nei portacampioni precedentemente raffreddati (Figura 3B); caricare i supporti sulla navetta e proteggerli con i coperchi (Figura 3C).
- Caricare la navetta nella camera di trasferimento a <100 K e pomparla a basso vuoto (Figura 3D).
- Collegare la camera di trasferimento al TXM (Figura 4A) e caricare la navetta dalla camera di trasferimento al TXM seguendo la procedura di vuoto sullo schermo (Figura 4B).
- Una volta che la navetta è all'interno con i campioni, il braccio robotico TXM può portare un portacampioni alla volta allo stadio del campione (Figura 4C).
3. Imaging con il software TXM
NOTA: le griglie nella fase del campione vengono prima fotografate utilizzando un microscopio a luce visibile on-line (VLM) per mappare la griglia in modalità brightfield e/o fluorescenza prima di essere ripresi con i raggi X. Usa l'icona del joystick corrispondente alla scheda Motion Control nel pannello in alto a sinistra per aprire Motion Control.
- Imaging della griglia con il VLM on-line.
- Acquisizione di mosaici a campo luminoso
- Selezionare la fotocamera VLM (lente di ingrandimento > VLM) e attivare la sorgente led VLM per l'imaging a campo luminoso (Microscopio > acquisizione > Impostazioni di acquisizione > Impostazioni sorgente e selezionare Trasmissione).
- Ruotare il campione a -60 gradi per affrontare l'obiettivo VLM (Motion Control > Sample > Sample θ) e spostare il campione nelle posizioni centrate previste (Motion Control > Sample e modificare Sample X, Sample Y).
- Durante l'acquisizione di immagini in fasi da 20 a 50 μm prima di portare la griglia approssimativamente a fuoco (microscopio > acquisizione > impostazioni di acquisizione > modalità di acquisizione > continuous > start; Motion Control > Campione e modifica campione Z).
- Affinare la messa a fuoco con passi più piccoli fino a 5 μm fino a quando le celle e/o i fori della pellicola di supporto del carbonio sono a fuoco (microscopio > acquisizione > impostazioni di acquisizione > modalità di acquisizione > avvio continuo >; Motion Control > Sample and change Sample Z) e avviare l'acquisizione di una mappa a mosaico completa della griglia in modalità campo luminoso. Utilizzare i valori predefiniti per il mosaico. (Impostazioni di acquisizione > acquisizione > microscopio > modalità di acquisizione > mosaico > inizio).
NOTA: Una mappa a mosaico è fatta di singole immagini. Il numero di immagini (numero di colonne, righe e dimensione del passo in X e Y) deve essere impostato per visualizzare l'intera griglia. La dimensione del passo dipende dalla dimensione del campo visivo (FoV). I valori predefiniti vengono utilizzati qui. Se la griglia non è ben centrata nel piano X-Y rispetto al Mosaico completo FoV, interrompere l'acquisizione, correggere le coordinate X e Y del campione e ripetere l'acquisizione.
- Acquisizione mosaico in modalità fluorescenza
- Spegnere la sorgente guidata da VLM per l'imaging a campo luminoso (Impostazioni di acquisizione > > di acquisizione > microscopio > e deselezionare trasmissione) e selezionare manualmente la sorgente luminosa a LED corrispondente alla lunghezza d'onda di eccitazione desiderata (rosso, verde o blu) e il filtro ottico corrispondente durante la configurazione.
- Affinare l'attenzione sull'immagine di fluorescenza (microscopio > acquisizione > impostazioni di acquisizione > modalità di acquisizione > avvio continuo >; Motion Control > Sample and change Sample Z), quindi acquisire una mappa a mosaico che mantenga i parametri posizionali (X e Y) dal mosaico a campo luminoso (Microscope > Acquisition > Acquisition Settings > Acquisition Modes > Mosaic > Start).
- Spegnere la sorgente luminosa a LED
- Facoltativo: in base alle mappe del campo luminoso e del mosaico a fluorescenza, annotare le regioni di interesse (ROI) identificate in precedenza (passaggio 1.7.4) o il nuovo ROI (posizioni X-Y nell'immagine).
- Acquisizione di mosaici a campo luminoso
- Acquisizione di mosaici a raggi X
- Selezionare il rilevatore di raggi X CCD (lente d'ingrandimento > selezionare Pixis), portare il campione a una rotazione di 0 gradi (Motion Control > Sample e cambiare Sample θ) e spostare l'ottica dei raggi X (Condensatore e Piastra di Zona) nelle posizioni allineate (Motion Control > Condensatore > cambiare Condensatore z; Motion Control > Zone Plate e cambia Zone Plate Z).
NOTA: Raffreddare il chip CCD a -65 °C prima dell'imaging (Microscopio > Temperatura della telecamera > Pixis e modificare la temperatura impostata su -65 °C e fare clic su Applica). - Spostate il campione al centro del quadrato della mesh di uno dei ROI (Motion Control > Sample selezionato e modificate Sample X, Sample Y).
- Utilizzare il binning 2 e uscire dalla fessura a 5 μm per ridurre al minimo l'irradiazione e l'esposizione a 1 s a Mistral, e il binning 1 e 60 μm a B24, regolare la messa a fuoco utilizzando la traduzione Z del campione (microscopio > acquisizione > impostazioni di acquisizione > impostazioni della fotocamera e modificare il binning; Motion Control > XS e cambio XS; Impostazioni di acquisizione > di acquisizione > microscopio> modalità di acquisizione > avvio continuo > ; Motion Control > Campione e modifica campione Z). Iniziare a passi di 5 μm e perfezionarlo fino a passi di 0,5 μm, fino a quando la cella o i fori del foglio di carbonio sono ben a fuoco.
- Acquisire una mappa a mosaico del quadrato della mesh (Impostazioni di acquisizione > > di acquisizione del microscopio > modalità di acquisizione > mosaico > Start).
NOTA: I parametri di acquisizione del mosaico possono essere determinati allo stesso modo dei mosaici VLM (punto 3.1.1.4). La dimensione del passo dipende dall'ingrandimento preselezionato dal personale della beamline. - Spostare il campione in una posizione a campo piatto (FF) (un'area vuota all'interno della griglia, preferibilmente un foro nel supporto del carbonio) (Motion Control > Sample e modificare sample X, sample Y).
- Impostare il tempo di esposizione a 1 s a Mistral e 0,5 s a B24 (Microscopio > acquisizione > impostazioni di acquisizione > impostazioni della fotocamera e modificare il tempo di esposizione) e acquisire una singola immagine (microscopio > acquisizione > modalità di acquisizione > single > start).
- Normalizzare (dividere) il mosaico acquisito dall'immagine FF per ottenere la trasmissione (con valori compresi tra 0 e 1) e salvare il mosaico normalizzato (cliccare sulla prima icona dell'angolo in alto a destra sul lato destro dell'immagine per aprire il menu>scegli la scheda Riferimento> clicca su Riferimento singolo e sfoglia lo specifico FF).
- Selezionare il rilevatore di raggi X CCD (lente d'ingrandimento > selezionare Pixis), portare il campione a una rotazione di 0 gradi (Motion Control > Sample e cambiare Sample θ) e spostare l'ottica dei raggi X (Condensatore e Piastra di Zona) nelle posizioni allineate (Motion Control > Condensatore > cambiare Condensatore z; Motion Control > Zone Plate e cambia Zone Plate Z).
- Preparazione alla raccolta di una serie di inclinazioni a raggi X
NOTA: Trova l'asse di rotazione per ogni regione di interesse in fase di ripresa.- Fai una selezione di aree all'interno dei mosaici per eseguire la tomografia, ad esempio posizionando una forma quadrata (fai clic su Strumento sul lato sinistro della finestra dell'immagine) con le dimensioni del FoV nella mappa del mosaico a raggi X.
NOTA: Quando si selezionano le aree, considerare la distanza dal bordo (≥10 μm) e da altre celle, per evitare sovrapposizioni di celle durante la rotazione. Inoltre, controllare le condizioni della cellula (forma cellulare prevista, spessore del ghiaccio, successo della vetrificazione, diffusione fiduciale, ecc.). - Allineamento del campione sull'asse di rotazione
NOTA: La procedura segnalata è iterativa. L'iterazione converge più velocemente utilizzando ± angoli di 60° come angoli di rotazione massimi (θM). Se non sono accessibili, utilizzare angoli il più vicino possibile a ± 60°.- Impostare la fotocamera su binning 2, tempo di esposizione 1 s (Microscopio > Acquisizione > Impostazioni di acquisizione > Impostazioni fotocamera e modificare il tempo di esposizione; modificare Binning; Microscopio > modalità di acquisizione > acquisizione > avvio continuo >), impostare la fessura di uscita su 5 μm.
NOTA: Ridurre al minimo la dose il più possibile lavorando con un'apertura minima della fessura di uscita. - Con la rotazione a 0°, spostarsi nell'area precedentemente selezionata utilizzando la traslazione del campione X e del campione Y e concentrarsi sulla caratteristica della cella da inserire nell'asse di rotazione utilizzando la traslazione Z del campione (Motion Control > Sample e modificare Sample x; Campione y; Esempio z).
- Ruotare il campione a + θM (Motion Control > Sample e modificare Sample θ) e disegnare una linea (L+) (fare clic sul pulsante line tool sul lato destro della finestra dell'immagine) sulla feature della cella da inserire sull'asse di rotazione.
- Ruotare fino a -θM (Motion Control > Sample e modificare Sample θ) e disegnare una linea (L-) (fare clic sul pulsante dello strumento linea sul lato destro della finestra dell'immagine) sulla caratteristica della cella che si desidera inserire sull'asse di rotazione.
- A +θM o -θM, utilizzate la traslazione Z di esempio per spostare la feature selezionata nella posizione centrale tra entrambe le linee (Controllo movimento > Campione e modifica Campione Z).
- Ripetere la procedura dal punto 3.3.2.3 in modo iterativo fino a raggiungere una distanza minima da L+ a L-.
NOTA: La distanza tra le due linee L+ e L- dovrebbe essere inferiore rispetto all'iterazione precedente. - Al campione θ = 0 (Motion Control > Sample e change Sample θ), spostare il campione X il doppio della distanza necessaria per posizionare la feature selezionata al centro di entrambe le linee (Motion Control > Sample e modificare Sample X). Spostate la piastra di zona (ZP) X per riportare la feature al centro del FoV (Motion Control > ZP e modificare ZP X). Eseguire questo passaggio solo una volta per griglia.
- Rimilizza la posizione ZP Z rispetto al nuovo asse di rotazione registrando una serie focale ZP Z (raccolte di immagini in diverse posizioni ZP Z, di solito in passi di 0,3 μm) (Microscopio > acquisizione > impostazioni di acquisizione > modalità di acquisizione > serie focale > Start) e sposta lo ZP Z nella posizione in cui il campione è a fuoco (Motion Control > Zone Plate e cambia Zone Plate z).
- Impostare i parametri per l'acquisizione della serie tilt.
NOTA: L'intervallo angolare massimo è in definitiva limitato dalla lunghezza focale dello ZP (±70 o ±65 gradi per uno ZP di 40 nm o 25 nm per un campione piatto, rispettivamente). Ottimizzare il rapporto segnale-rumore (S/N) e i danni da radiazioni per definire il tempo di esposizione. Per la tomografia, utilizzare tempi di esposizione diversi a diversi intervalli angolari. - Determinare l'intervallo angolare di rotazione più elevato, nel caso in cui l'ombreggiatura si verifichi prima che venga raggiunto l'angolo di rotazione massimo.
- Impostare il binning della fotocamera su 1 (Microscope > Acquisition > Acquisition Settings > Camera Settings e modificare Binning), aprire la fessura di uscita a 15 μm a Mistral (Motion Control > XS e cambiare XS) e impostare la rotazione su 0° (Motion Control > Sample e change Sample θ).
- Acquisire una singola immagine con un tempo di esposizione di 1 s (Microscope > Acquisition > Acquisition Settings > Acquisition Modes > Single > Start) e stimare il tempo di esposizione richiesto per ogni angolo di inclinazione della tomografia.
- Impostare la fotocamera su binning 2, tempo di esposizione 1 s (Microscopio > Acquisizione > Impostazioni di acquisizione > Impostazioni fotocamera e modificare il tempo di esposizione; modificare Binning; Microscopio > modalità di acquisizione > acquisizione > avvio continuo >), impostare la fessura di uscita su 5 μm.
- Fai una selezione di aree all'interno dei mosaici per eseguire la tomografia, ad esempio posizionando una forma quadrata (fai clic su Strumento sul lato sinistro della finestra dell'immagine) con le dimensioni del FoV nella mappa del mosaico a raggi X.
- Acquisizione tomografia
- Passare all'angolo massimo negativo +0,1 (ad esempio, per ZP 25 nm andare a -65,1°) (Motion Control > Sample e modificare Sample θ).
- Impostare il numero di immagini come numero totale di angoli (tenendo conto dell'immagine con angolo 0) e l'intervallo angolare (Microscopio > Acquisizione > Impostazioni di acquisizione > Modalità di acquisizione > Tomografia e modificare il numero di immagini, quindi modificare Angolo Inizio e Angolo fine).
- Impostare il tempo di esposizione definito e avviare l'acquisizione (Microscopio > Acquisizione > Impostazioni di acquisizione > Impostazioni fotocamera e modificare il tempo di esposizione e quindi fare clic su Avvia).
- Passare alla posizione FF (Motion Control > Sample e cambiare Sample X; quindi, cambiare Sample Y) e acquisire 10 immagini FF (Microscope > Acquisition > Acquisition Modes > Average e modificare il numero di immagini e quindi fare clic su Start).
NOTA: A Mistral, la spettromicroscopia o la microscopia dipendente dall'energia è possibile quando si acquisiscono proiezioni durante la scansione dell'energia attraverso un bordo di assorbimento di interesse. L'output finale è una pila di proiezioni 2D, che conterrà uno spettro di assorbimento dei raggi X (XAS) in ogni pixel, cioè immagini con informazioni chimiche. L'estensione al 3D che combina la spettroscopia con la tomografia è in linea di principio possibile. La dose totale richiesta potrebbe essere una limitazione e quindi possono essere necessarie strategie specifiche come l'imaging ad assorbimento differenziale.
4. Analisi dei dati
NOTA: tutta l'analisi dei dati viene eseguita con software aperto disponibile e script sviluppati con pipeline automatizzate.
- A Mistral
- Pipeline converte gli stack di tomografia dall'estensione txrm (estensione software TXM) a hdf5 (formato di dati gerarchico open source) con tutti i metadati necessari, quindi normalizza lo stack in base alla media FF e alla corrente della macchina e infine deconvoca gli stack mediante la funzione di diffusione del punto misurato del sistema ottico per una specifica lente ZP (Fresnel zone plate) e l'energia23, 24. Per la deconvoluzione, trova il valore k = 1/SNR corretto (dipende dallo spessore del campione). Per lo script sviluppato presso Mistral, digitare il comando: txrm2deconv "input tomo" "input FF" -zp="ZP used" - e= " energy " - dx= " pixel size " - k= " 1/SNR " - t=-1.
- Per lo script sviluppato presso Mistral per l'allineamento automatico, digitare il comando: ctalignxcorr "normalized deconvolved stack.mrc" "normalized stack.hdf5".
NOTA: Un certo numero di applicazioni software possono essere utilizzate per l'allineamento delle proiezioni ad un asse di rotazione comune utilizzando i fiduciali Au NP25,26. L'allineamento delle proiezioni richiede una precisione subpixel. L'allineamento automatico può essere soddisfacente solo quando i fiduciali Au NP sono sufficienti (>7) e ben distribuiti sul campo visivo. L'allineamento manuale è spesso necessario a posteriori per correggere l'allineamento automatico per ottenere la massima precisione possibile. - Per ricostruire lo stack normalizzato allineato, utilizzare uno dei numerosi algoritmi disponibili.
NOTA: lo stack allineato può essere ricostruito utilizzando la proiezione posteriore ponderata (WBP) o le tecniche di ricostruzione iterativa simultanea (SIRT) in pochi minuti. Tuttavia, al fine di preservare i coefficienti di assorbimento lineare (LAC), si preferisce Algebraic Reconstruction Techniques (ART)27. ART richiede più tempo di calcolo, quindi SIRT28 viene eseguito per primo per una rapida ricostruzione della serie di inclinazioni allineata automaticamente (30 iterazioni in pochi minuti). Una volta che l'allineamento è soddisfacente, verrà utilizzata l'ART.
- Al B24
- Una pipeline personalizzata converte i file di dati txrm in Tiff standard con tutti i metadati necessari e quindi li invia a un flusso di lavoro runtomo batch che elabora i set di dati in tre modi: WBP, SIRT e patch.
Risultati
Preparare i campioni per cryo-SXT può essere difficile. Anche all'interno della stessa griglia di esempio, è possibile avere aree ideali e aree non ideali, come si può vedere nella Figura 5, che mostra due quadrati dalla stessa griglia Au finder. Il campione ideale dovrebbe avere singole celle al centro di una maglia quadrata, incorporate in un sottile strato di ghiaccio e circondate da marcatori fiduciali Au ben dispersi utilizzati per l'allineamento delle proiezioni di inclinazione prima della ricostruzione tomografica. La Figura 5A mostra una cellula simile ai fibroblasti (NIH 3T3) che soddisfa molti di questi criteri. Una singola sezione di una ricostruzione 3D utilizzando ART27 dell'area contrassegnata con la casella rossa che indica il campo visivo (FoV) è mostrata nella Figura 5B. Molti organelli diversi come i mitocondri (M), il reticolo endoplasmatico (ER), le vescicole (V) e il nucleo (N) possono essere distinti grazie alla ricostruzione quantitativa dei LAC. Inoltre, il rapporto segnale-rumore della ricostruzione è molto elevato consentendo di ottenere un elevato contrasto delle caratteristiche cellulari. D'altra parte, la Figura 5C mostra un quadrato con una maggiore densità di cellule. Per questo motivo, il blotting è solitamente meno efficiente, portando a uno strato di ghiaccio più spesso o persino a problemi di vetrificazione. In alcuni casi, questo può già essere osservato durante lo screening della griglia utilizzando la mappatura dell'epifluorescenza prima dell'imaging a raggi X, e tali griglie dovrebbero essere evitate ad ogni costo. Nella Figura 5C, si può osservare una fessura all'interno della griglia e il ghiaccio vetrificato che attraversano l'intero quadrato della rete (contrassegnato dalle frecce rosse). Qualsiasi imaging vicino a crepe dovrebbe essere evitato a causa della probabile instabilità della griglia quando esposto al raggio. Inoltre, le crepe possono essere un segno di ghiaccio spesso, come nel caso di quest'area. Una serie di inclinazioni è stata registrata nell'area contrassegnata con la casella rossa. Nella Figura 5D viene mostrata una singola sezione della ricostruzione 3D corrispondente. Anche se alcune strutture più grandi possono essere riconosciute, i dettagli fini si perdono all'interno del rumore e della consistenza granulosa a causa della scarsa qualità di vetrificazione del ghiaccio spesso, come si può vedere in particolare, ad esempio, sui mitocondri superiori puntati dalla freccia.
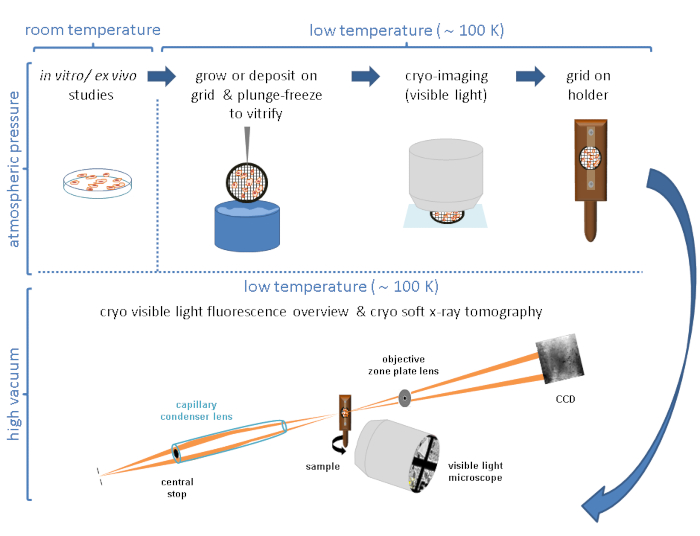
Figura 1: Flusso di lavoro. Flusso di lavoro schematico seguito prima della raccolta dei dati cryo-SXT. Fare clic qui per visualizzare una versione più grande di questa figura.
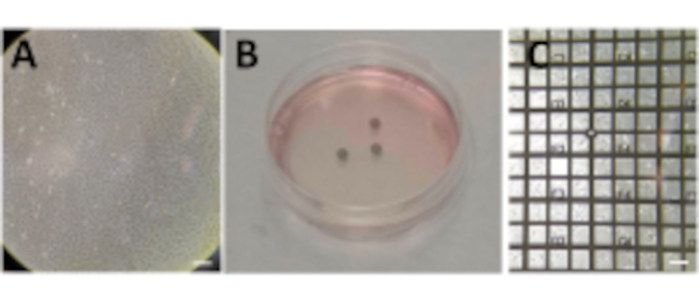
Figura 2: Cellule in crescita su griglie. (A) Cellule che crescono in una capsula di Petri P100 con una confluenza intorno all'80%-90%. (B) P60 Capsula di Petri con diverse griglie dopo la semina delle cellule. (C) Cellule che crescono sulla cima di una griglia dopo 24 ore. Barre di scala: 100 μm. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Griglie di carico sui portacampioni e nella camera di trasferimento. (A) Posto di lavoro riempito di azoto liquido con la navetta e le criobox pronte per il caricamento delle griglie. (B) Portacampioni inserito nel caricatore con la griglia caricata. (C) Navetta con il portacampioni in posizione 3 senza coperchio. (D) Posto di lavoro con la camera di trasferimento collegata. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Caricamento di campioni nel TXM (A) Collegamento della camera di trasferimento al TXM. (B) Navetta all'interno del TXM. (C) Braccio robotico TXM che inserisce il portacampioni nella fase del campione. Fare clic qui per visualizzare una versione più grande di questa figura.
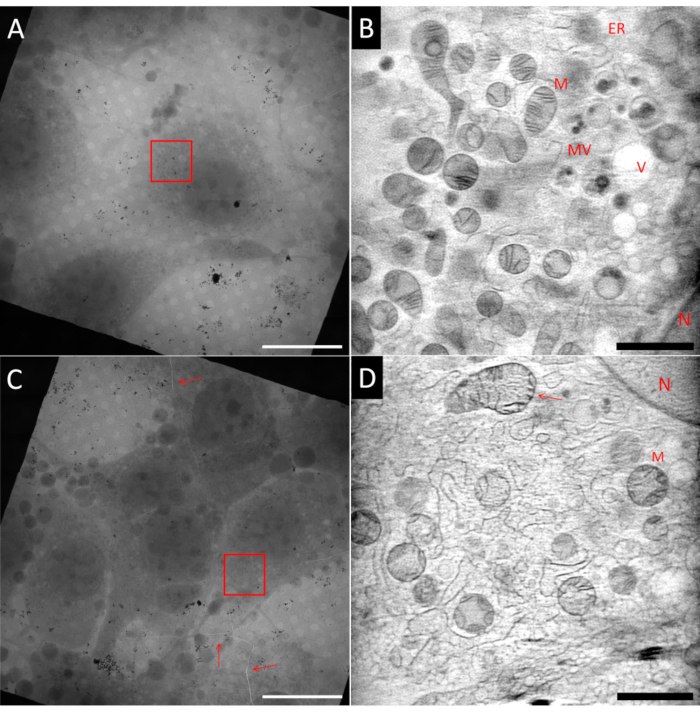
Figura 5: Esempio di tomogrammi a raggi X criomoscidi. Riga superiore: campione ideale, (A) vista a mosaico 2D di un quadrato della griglia che mostra una cella isolata al centro. (B) Una fetta del volume 3D ricostruito che mostra l'area contrassegnata con la casella rossa (A). Rispetto a (D) l'immagine è molto più liscia e sono visibili più dettagli. Riga inferiore: campioni non ideali, (C) vista a mosaico 2D di un quadrato della griglia che mostra una confluenza cellulare troppo elevata e crepe nel ghiaccio e nella lamina della griglia (frecce rosse). (D) Una fetta del volume 3D ricostruito che mostra l'area contrassegnata con la casella rossa in (C). La vetrificazione scarsa o non ottimale può essere identificata dalla trama granulosa dell'immagine. N: Nucleo; M: Mitocondri; ER: Reticolo endoplasmatico; MV: Corpi multivescicolari; V: Vacuolo; Barre di scala: A & C 20 μm; B & D 2 μm.VFare clic qui per visualizzare una versione più grande di questa figura.
Discussione
La preparazione del campione è un passaggio fondamentale per ottenere tomogrammi a raggi X morbidi di alta qualità, poiché la loro qualità dipende direttamente dalla qualità della vitrificazione del campione e dallo spessore dello strato di ghiaccio in cui è incorporata la cella. Le proiezioni con un elevato rapporto segnale-rumore saranno raccolte in regioni con sottile strato di ghiaccio, consentendo di ridurre al minimo la dose di radiazione necessaria per ottenere la massima risoluzione possibile. Inoltre, la confluenza cellulare influenzerà anche la qualità finale del tomogramma, poiché si dovrebbe evitare di avere cellule vicine che entrano nel FoV durante la rotazione. Infine, la giusta dispersione dei marcatori au fiduciali determinerà l'accuratezza dell'allineamento della proiezione e quindi determinerà infine la qualità del volume finale ricostruito in 3D. Si noti che una corretta diffusione di Au fiducials sulla griglia consente l'automatizzazione della fase di allineamento della proiezione, senza la quale è necessaria un'elevata competenza per un passaggio così critico.
Il protocollo qui descritto descrive solo una possibile strategia di preparazione del campione, che ha somiglianze con quelle utilizzate nella crioenografia elettronica (cryo-ET). In entrambi i casi, i protocolli che migliorano la preparazione del campione esigente per una migliore riproducibilità saranno fondamentali per il successo di queste tecniche, e si stanno compiendo sforzi verso questo obiettivo29. Vale la pena ricordare che oltre all'imaging di cellule isolate, è possibile visualizzare anche sezioni di tessuto a condizione che il segnale di trasmissione attraverso la sezione sia sufficiente ad angoli di inclinazione elevati. In genere, ciò implica sezioni di pochi micron (inferiori a 10 μm).
Per visualizzare una struttura o un evento specifico all'interno di una cella, è necessario assicurarsi che questa particolare caratteristica sia all'interno del FoV della serie tilt. Poiché il FoV in crio-SXT è limitato a 10 x 10 μmda 2 a 15 x 15 μm2 a seconda dell'obiettivo e tiene conto di un sovracampionamento in pixel della risoluzione ad almeno un fattore di 2, è spesso più piccolo dell'estensione dell'intera cella (vedi i quadrati rossi indicati nella Figura 5). Pertanto, il ROI deve essere trovato e correttamente etichettato. Questo di solito viene fatto per mezzo di tag fluorescenti e approcci correlativi alla luce visibile. Le strategie 2D che combinano l'epifluorescenza sono semplici in quanto il microscopio a trasmissione a raggi X morbidi ha un microscopio a fluorescenza a luce visibile on-line integrato, ma sono disponibili anche altri approcci per il segnale di fluorescenza 2D o 3D ad alta risoluzione 4,12,13,15,16 . In questi casi, la griglia deve essere prima fotografata in strumenti specifici come i microscopi a super risoluzione. Si noti che gli approcci correlativi più efficienti sono quelli che coinvolgono la raccolta di dati in condizioni criogeniche. Questo perché il lasso di tempo tra la temperatura ambiente (RT), l'imaging a fluorescenza della luce visibile e la vitrificazione del campione, ad esempio, ostacolerà la cattura dell'evento cellulare giusto in tempo; inoltre, la procedura di vetrificazione potrebbe staccare dalla griglia la cella di interesse che è stata ripresa a RT. Anche se la maggior parte degli approcci di imaging correlativi potrebbe implicare che le griglie del campione debbano essere manipolate e trasportate da uno strumento all'altro, e nonostante l'aumento del rischio di contaminazione o danno alla griglia che ciò comporta, la ricompensa è chiara: essere in grado di individuare eventi o molecole specifici all'interno del paesaggio cellulare.
Quando è richiesta l'imaging di cellule intere, è possibile cucire diversi tomogrammi a condizione che la dose totale applicata non superi il limite di danno da radiazioni. Di solito, la dose depositata per la raccolta di pochi tomogrammi sulla stessa cellula è ben al di sotto del limite alla risoluzione raggiungibile (109 Gy) e, pertanto, non è necessaria alcuna strategia specifica per abbassare la dose, sebbene questa sia dipendente dal campione e dall'esperimento. Nel caso di una raccolta intensiva di dati come la spettrotomografia, sarebbe effettivamente necessaria una riduzione al minimo della dose e sarebbe necessario applicare una comoda raccolta di dati e strategie di elaborazione specifiche.
Cryo-SXT ha diverse limitazioni, che dovrebbero essere menzionate qui. Il primo è il noto cuneo mancante, che è intrinseco all'utilizzo di supporti di campioni piatti. I supporti capillari per campioni che consentono una rotazione di 180 gradi sono stati utilizzati in passato e sono ancora utilizzati in alcune strutture, ma presentano anche inconvenienti come un contrasto impoverito dovuto all'assorbimento del vetro e alla restrizione dell'uso di cellule in sospensione. Un modo per diminuire l'effetto del cuneo mancante è eseguire la tomografia a doppia inclinazione. Questo è davvero possibile alla linea di fascio Mistral al giorno d'oggi. La seconda limitazione è impostata dalla lente a piastre a zona di Fresnel utilizzata in tali microscopi. Questo obiettivo imposta la risoluzione massima raggiungibile e la profondità di campo (DoF), entrambi strettamente correlati. Ciò implica che l'aumento della risoluzione diminuirà il DoF mentre lo spessore della cella sarà spesso maggiore. Ad esempio, una lente da 40 nm avrà in teoria un DoF di 3 μm e una risoluzione di 24,4 nm di mezzo passo. Il compromesso tra risoluzione e DoF è quindi strategico e la scelta dell'obiettivo dipenderà dal tipo di cella30,31. Infine, i TXM operativi in tutto il mondo sono ben lungi dall'essere microscopi ideali e si stanno compiendo sforzi per migliorare i sistemi ottici per raggiungere le aspettative teoriche. Infine, la visualizzazione e la segmentazione dei volumi ricostruiti può essere effettuata con strumenti software specifici 25,32,33,34.
In sintesi, cryo-SXT consente di eseguire l'imaging quantitativo delle cellule a media risoluzione (25-30 nm mezzo passo) e in numeri statistici (poche decine di tomogrammi al giorno). Ciò consente di ottenere l'organizzazione, la distribuzione e la dimensione degli organelli in condizioni specifiche, ad esempio durante l'infezione o le malattie patogene, in punti temporali precisi o dopo particolari trattamenti. Si tratta, quindi, di un'utile tecnica di imaging biologico complementare alle più comuni microscopie a elettroni e a luce visibile, ognuna delle quali affronta una specifica gamma di dimensioni e risoluzione del campione. Cryo-SXT è spesso usato in approcci correlativi che coinvolgono la fluorescenza della luce visibile, ma sono possibili anche altre strategie crio-correlateve.
Divulgazioni
Gli autori non dichiarano alcun conflitto di interessi.
Riconoscimenti
Questo progetto ha ricevuto finanziamenti dal progetto Horizon 2020 iNEXT-Discovery della Commissione europea e dal programma di ricerca e innovazione Horizon 2020 dell'Unione europea nell'ambito della convenzione di sovvenzione Marie Skłodowska-Curie n. 75439.
Materiali
| Name | Company | Catalog Number | Comments |
| Amira | Thermo Fisher | Software for segmentation | |
| Au Holey Carbon Films finder grids | (Quantifoil Micro Tools Gmb | R 2/2 Au G200F1 | Au Holey Carbon Films finder grids |
| Au nanoparticles | BBI Group, Cardiff, UK | Au nanoparticles 100nm | 100 nm Au nanoparticles (NPs) at Mistral (Alba) |
| Au nanoparticles | BBI Group, Cardiff, UK | Au nanoparticles 250nm | 250 nm Au nanoparticles (NPs) at B24 (Diamond) |
| Axio Scope A1 | Zeiss | 430035 9060 | Fluorescence microscope |
| Blotting No.1 filter paper | Whatman | WHA10010155 | Blotting filter |
| Bsoft | Software for projection alignment, reconstruction and visualization (Heymann et al., 2008) | ||
| Chimera | Software for segmentation (Pettersen et al. 2004) | ||
| Cryo-EM Glow Discharge Set | PELCO easiGlow | 91000S | Glow Discharge Cleaning System |
| Cu Holey Carbon Films finder grids | (Quantifoil Micro Tools Gmb | R 2/2Cu G200F1 | Cu Holey Carbon Films finder grids |
| Fetal calf serum | Sigma | F9665 | Heat Inactivated, sterile-filtered, suitable for cell culture |
| ImageJ | Software for image processing and analysis in Java (NIH & LOCI University of Wisconsin) | ||
| IMOD | Software for projection alignment, reconstruction and visualization (Kremer et al., 1996) | ||
| Leica EM GP Grid Plunger | Leica | 16706401 | Automatic Plunge Freezer EM |
| LINKAM cryo-stage | Linkam Scientific Instruments | CMS 196 | Cryo-Correlative Microscopy Stage |
| MIB | Software for segmentation (Belevich et al. 2016) | ||
| P100 Petri dish | Sigma | P6106 | Treated for cell culture and sterile |
| P60 Petri dish | Sigma | D8054 | Treated for cell culture and sterile |
| Polylysin | Sigma | P4707 | Poly-L-lysine solution 0.01%, sterile-filtered |
| Soft X-Ray microscope 0.25-1.2keV | Xradia | NCT-SB | Transmission soft X-Ray microscope |
| SURVOS | Software for segmentation (Luengo et al. 2017) | ||
| Tomo3d | Software for reconstruction (SIRT, WBP) (Agulleiro et al. 2011) | ||
| TomoJ | Software for reconstruction (ART) (Messaoudi et al., 2007) | ||
| XM Data Explorer | Zeiss | TXM software |
Riferimenti
- Carrascosa, J. L., et al. Cryo X-ray tomography of vaccinia virus membranes and inner compartments. Journal of Structural Biology. 168, 234-239 (2009).
- Uchida, M., et al. Soft X-ray tomography of phenotypic switching and the cellular response to antifungal peptoids in Candida albicans. PNAS. 106, 19375-19380 (2009).
- Schneider, G., et al. Three-dimensional cellular ultrastructure resolved by X-ray microscopy. Nature Methods. 10, 1-3 (2010).
- Chichón, F. J., et al. Cryo nano-tomography of vaccinia virus infected cells. Journal of Structural Biology. 177, 202-211 (2012).
- Groen, J., Conesa, J. J., Valcárcel, R., Pereiro, E. The cellular landscape by soft X-ray tomography. Biophysical Reviews. 11, 611-619 (2019).
- Kepsutlu, B., et al. Cells undergo major changes in the quantity of cytoplasmic organelles after uptake of gold nanoparticles with biologically relevant surface coatings. ACS Nano. 14, 2248-2264 (2020).
- Natterer, F. . The mathematics of computerized tomography. , (1986).
- Weiss, D., et al. Computed tomography of cryogenic biological specimens based on X-ray microscopic images. Ultramicroscopy. 84, 185-197 (2000).
- Clowney, E. J., et al. Nuclear aggregation of olfactory receptor genes governs their monogenic expression. Cell. 151, 724-737 (2012).
- Duke, E. M. H., et al. Imaging endosomes and autophagosomes in whole mammalian cells using correlative cryo-fluorescence and cryo-soft X-ray microscopy (cryo-CLXM). Ultramicroscopy. 143, 77-87 (2014).
- Cinquin, B., et al. Putting molecules in their place. Journal of Cellular Biochemistry. 115, 209-216 (2014).
- Hagen, C., et al. Multimodal nanoparticles as alignment and correlation markers in fluorescence/soft X-ray cryo-microscopy/tomography of nucleoplasmic reticulum and apoptosis in mammalian cells. Ultramicroscopy. 146, 46-54 (2014).
- Pérez-Berná, A. J., et al. Structural changes in cells images by soft X-ray cryo-tomography during Hepatitis C virus infection. ACS Nano. 10, 6597-6611 (2016).
- Chiappi, M., et al. Cryo-soft X-ray tomography as a quantitative three-dimensional tool to model nanoparticle:cell interaction. Journal of Nanobiotechnology. 14, 15 (2016).
- Varsano, N., et al. Development of correlative cryo-soft X-ray tomography and stochastic reconstruction microscopy. A study of cholesterol crystal early formation in cells. Journal of the American Chemical Society. 138, 14931-14940 (2016).
- Kounatidis, I., et al. Correlative cryo-structured illumination fluorescence microscopy and soft X-ray tomography elucidates reovirus intracellular release pathway. Cell. 182, 1-16 (2020).
- Kapishnikov, S., et al. Mode of action of quinoline antimalarial drugs in red blood cells infected by Plasmodium falciparum revealed in vivo. PNAS. 116 (46), 22946-22952 (2019).
- Conesa, J. J., et al. Unambiguous intracellular localization and quantification of a potent iridium anticancer compound by correlative 3D cryo X-ray imaging. Angewandte Chemie International Edition. 59, 1270-1278 (2020).
- Gal, A., et al. Native-state imaging of calcifying and noncalcifying microalgae reveals similarities in their calcium storage organelles. PNAS. 115 (43), 11000-11005 (2018).
- Procopio, A., et al. Chemical fingerprint of Zn-hydroxyapatite in the early stages of osteogenic differentiation. ACS Central Science. 5, 1449-1460 (2019).
- Kahil, K., et al. Cellular pathways of calcium transport and concentration toward mineral formation in sea urchin larvae. PNAS. 117 (49), 30957-30965 (2020).
- Conesa, J. J., et al. Intracellular nanoparticles mass quantification by near-edge absorption soft X-ray nanotomography. Scientific Reports. 6, 22354 (2016).
- Otón, J., Sorzano, C. O. S., Maribini, R., Pereiro, E., Carazo, J. M. Measurement of the modulation transfer function of an X-ray microscope based on multiple Fourier orders analysis of a Siemens star. Optics Express. 23 (8), 9567-9572 (2015).
- Otón, J., et al. Characterization of transfer function, resolution and depth of field of a soft X-ray microscope applied to tomography enhancement by Wiener deconvolution. Biomedical Optics Express. 7, 5092-5103 (2016).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116, 71-76 (1996).
- Heymann, J. B., Cardone, G., Winkler, D. C., Steven, A. C. Computational resources for cryo-electron tomography in Bsoft. Journal of Structural Biology. 161, 232-242 (2008).
- Messaoudi, C., et al. Three-dimensional chemical mapping by EFTEM-TomoJ including improvement of SNR by PCA and ART reconstruction of volume by noise supression. Microscopy Microanalysis. 19, 1669-1677 (2013).
- Agulleiro, J. I., Fernández, J. J. Fast tomographic reconstruction on multicore computers. Bioinformatics. 27, 582-583 (2011).
- Toro-Nahuelpan, M., et al. Tailoring cryo-electron microscopy grids by photo-micropatterning for in-cell structural studies. Nature Methods. 17, 50-54 (2020).
- Attwood, D. . Soft X-rays and Extreme Ultraviolet Radiation. Principles and Applications. , (2000).
- Howells, M., Jacobsen, C., Warwick, T. . Principles and Applications of Zone Plate X-ray. Microscopes. Science of Microscopy. , (2007).
- Pettersen, E. F., et al. UCSF Chimera--a visualization system for exploratory research and analysis. Journal of Computational Chemistry. 25, 1605-1612 (2004).
- Belevich, I., Joensuu, M., Kumar, D., Vihinen, H., Jokitalo, E. Microscopy image browser: a platform for segmentation and analysis of multidimensional datasets. PLOS Biology. 14 (1), 1002340 (2016).
- Luengo, I., et al. SuRVoS: Super-region volume segmentation workbench. Journal of Structural Biology. 198, 43-53 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon