Method Article
Preparazione e microlavorazione Cryo-FIB di Saccharomyces cerevisiae per Tomografia Crioeletnico
In questo articolo
Riepilogo
Presentiamo un protocollo per la preparazione lamellare di campioni biologici surgelati a tuffo mediante microlavorazione a fascio ionico crio-focalizzato per studi strutturali ad alta risoluzione di macromolecole in situ con tomografia crioeletnico. Il protocollo presentato fornisce linee guida per la preparazione di lamelle di alta qualità ad alta riproducibilità per la caratterizzazione strutturale di macromolecole all'interno del Saccharomyces cerevisiae.
Abstract
Oggi, la tomografia crioenonenica (cryo-ET) è l'unica tecnica in grado di fornire dati strutturali a risoluzione quasi atomica su complessi macromolecolari in situ. A causa della forte interazione di un elettrone con la materia, gli studi crio-ET ad alta risoluzione sono limitati a campioni con uno spessore inferiore a 200 nm, il che limita l'applicabilità della crio-ET solo alle regioni periferiche di una cellula. Un flusso di lavoro complesso che comprende la preparazione di sezioni trasversali cellulari sottili mediante microlavorazione a fascio ionico crio-focalizzato (cryo-FIBM) è stato introdotto nell'ultimo decennio per consentire l'acquisizione di dati crio-ET dall'interno di celle più grandi. Presentiamo un protocollo per la preparazione di lamelle cellulari da un campione vitrificato mediante congelamento a immersione utilizzando Saccharomyces cerevisiae come esempio prototipico di una cellula eucariotica con ampio utilizzo nella ricerca di biologia cellulare e molecolare. Descriviamo i protocolli per la vitrificazione di S. cerevisiae in patch isolate di poche cellule o un monostrato continuo delle cellule su una griglia TEM e forniamo un protocollo per la preparazione delle lamelle mediante crio-FIB per questi due campioni.
Introduzione
Recenti sviluppi tecnologici e software hanno reso la crio-microscopia elettronica (cryo-EM) di campioni biologici vetrificati una delle tecniche chiave nella ricerca di biologia strutturale nell'ultimo decennio1,2. La preparazione di un campione per crio-EM di solito consiste nell'applicazione di una proteina purificata o di un complesso di proteine con acido nucleico sul supporto del campione (griglia TEM), seguita dalla rimozione della maggior parte del liquido con una carta da filtro e dal congelamento a immersione della griglia con lo strato sottile residuo di un campione in etano liquido o propano3 . Il campione viene quindi fissato in uno strato sottile (tipicamente <80 nm) di tampone amorfo in uno stato completamente idratato, in condizioni quasi native e senza bisogno di alcuna fissazione chimica o contrasto di metalli pesanti. L'imaging del campione strutturalmente omogeneo nel microscopio elettronico a trasmissione si traduce quindi in dati che possono essere utilizzati per la determinazione della struttura tridimensionale della macromolecola a risoluzione quasi atomica utilizzando un protocollo di analisi delle singole particelle2. Tale struttura in vitro corrisponde alla rappresentazione della macromolecola nelle condizioni e nel trattamento utilizzati durante la preparazione del campione. Sebbene le strutture determinate nelle condizioni in vitro di solito corrispondano allo stato pienamente funzionale della macromolecola, la capacità di immaginare le relazioni spaziali tra le varie macromolecole all'interno della cellula fornirebbe un ulteriore contesto funzionale ai dati strutturali.
La tomografia crioenografica elettronica (cryo-ET) viene utilizzata per ricostruire volumi 3D di oggetti pleomorfi o complessi macromolecolari in situ4,5. Il vantaggio di cryo-ET è che l'informazione tridimensionale si ottiene mediante l'imaging di una singola entità. Tuttavia, la risoluzione alla quale si osservano singoli complessi macromolecolari o organelli è molto limitata. Pertanto, la media delle macromolecole (media sub-tomogramma, STA) con la stessa struttura da un numero maggiore di tomogrammi è necessaria per raggiungere modelli di risoluzione 4-8 Å dai dati crio-ET6,7. Recentemente è stato dimostrato che crio-ET e STA possono anche essere applicati per determinare strutture ad alta risoluzione di macchine macromolecolari come i ribosomi nel contesto dell'ambiente cellulare7. Tuttavia, l'utilizzo della microscopia elettronica a trasmissione è limitato dallo spessore del campione. In generale, questo non è un problema per la crio-EM a singola particella in cui l'ottimizzazione delle condizioni di vetrificazione può eventualmente comportare l'incorporazione del campione in un sottile strato di ghiaccio. D'altra parte, la maggior parte delle celle non sono in realtà trasparenti elettroniche per il fascio di elettroni da 300 keV. Il percorso libero medio anelastico nei campioni biologici vetrificati per gli elettroni a 300 keV è di circa 395 nm8, il che significa che gli studi crio-ET sono limitati alla periferia cellulare per la maggior parte delle cellule.
Sono state sviluppate diverse tecniche per assottigliare il campione fino a uno spessore sufficiente per la crio-ET. La crio-ultramicrotomia utilizza l'affettatura meccanica del campione con un coltello diamantato alla temperatura dell'azoto liquido (-196 °C) per fornire sezioni spesse 60-80 nm adatte per crio-ET9,10,11. Più sezioni possono essere preparate da una singola cella e l'analisi dei dati può eventualmente produrre informazioni strutturali 3D per la maggior parte della cella. Tuttavia, l'affettamento meccanico può causare diversi artefatti come sezioni curve, crepacci o compressione del campione, che possono influenzare la struttura risultante e polarizzare i dati crio-ET10,11,12. La microlavorazione a fascio ionico crio-focalizzato (cryo-FIBM) rappresenta un approccio alternativo in cui una sottile sezione cellulare viene preparata mediante ablazione graduale del campione utilizzando un fascio focalizzato di ioni Ga+ (FIB) in un processo a più fasi, che può risultare in una sezione trasversale cellulare spessa 80-300 nm (lamella)13,14,15 . A differenza della crio-ultramicrotomia, solo una lamella viene preparata da una singola cellula, che rappresenta ~ 0,3-3% del suo volume, e la microlavorazione di più celle è solitamente necessaria per trovare una regione di interesse nella sezione trasversale fresata. Inoltre, il throughput dell'intero flusso di lavoro è oggi ancora piuttosto basso, spesso limitato a 6-8 lamelle di alta qualità da una sessione crio-FIBM di 8 ore. D'altra parte, le sezioni trasversali crio-FIBM sono prive di artefatti di compressione e forniscono un input adatto per crio-ET ad alta risoluzione. Inoltre, il trasferimento della lamella al supporto del campione per crio-ET non è necessario in quanto il campione viene trattenuto sulla griglia TEM durante l'intero processo di preparazione della lamella e la stessa griglia può essere successivamente trasferita a TEM. Prevediamo che la produttività del crio-FIBM sarà presto significativamente migliorata, principalmente dalla disponibilità di software per la fresatura di lamelle non supervisionate16,17 e dall'utilizzo di FIB che operano secondo il principio del plasma a coppia di carica, che offrirà un'ablazione più rapida del materiale.
Saccharomyces cerevisiae (lievito) sono cellule eucariotiche di forma sferica e diametro di ~ 2-5 μm. Grazie alle sue dimensioni, accessibilità, genetica, tempo di generazione e semplice manipolazione, il lievito è ampiamente studiato come organismo modello eucariotico nella ricerca di biologia cellulare e molecolare simile a Escherichia coli, che è ben studiato come organismo modello procariotico in batteriologia. Il lievito può essere facilmente coltivato in sospensione e in breve tempo viene generata un'elevata quantità di cellule (tempo di raddoppio 1 – 2 ore). Ancora più importante, il lievito condivide una complessa struttura cellulare interna con cellule animali e vegetali, pur mantenendo un piccolo genoma composto da un basso contenuto di DNA non codificante. La caratterizzazione strutturale del proteoma del lievito dai dati in situ ad alta risoluzione può quindi aiutare a fornire una descrizione meccanicistica per l'ampia quantità di dati funzionali disponibili in letteratura.
Qui forniamo un protocollo completo per l'acquisizione di dati crio-ET in situ sul campione di lievito, che copre tutte le fasi dalla coltivazione del campione fino alla preparazione della lamella crio-FIBM e il trasferimento del campione a TEM per l'acquisizione dei dati crio-ET.
Protocollo
1. Coltivazione e preparazione di cellule saccharomyces cerevisiae per la vitrificazione
- Preparare il mezzo di crescita liquido per Saccharomyces cerevisiae.
- Autoclave una bottiglia di vetro da 500 ml per la preparazione del terreno di crescita.
- Pesare 2,2 g di estratto di lievito (1,1%) e 4,4 g di Peptone (2,2%) e mescolare in 200 ml di acqua.
- Sterilizzare in autoclave per 15 min a 121 °C.
- Pesare 10 g di glucosio in polvere e mescolare in 50 ml di acqua per ottenere una soluzione di glucosio al 20%. Passare la soluzione attraverso un filtro da 0,2 μm e conservarla a 4 °C.
- Preparare il mezzo solido per Saccharomyces cerevisiae.
- Pesare 4 g di polvere di agar e mescolare con 200 ml di terreno di coltura.
- Sterilizzare in autoclave per 15 minuti a 121 °C.
- Raffreddare il mezzo a 40-50 °C e aggiungere 20 ml di glucosio sterile al 20% (preparato nella fase precedente). Versare ~20 ml del mezzo completo nella capsula di Petri e lasciarlo solidificare a temperatura ambiente.
- Avvolgere le piastre di agar in parafilm per proteggerle dall'essiccazione e conservare a 4 °C.
- Cultura Saccharomyces cerevisiae in sospensione
NOTA: Il protocollo è ottimizzato per la preparazione di un campione di crio-FIBM da una sospensione della linea cellulare Saccharomyces cerevisiae ceppo BY4741 [ATCC 4040002] o ceppi simili.- Autoclave un pallone Erlenmeyer (o simile) da 50 ml.
- Lavora in una cappa o in una scatola a flusso laminare. Pipettare 10 mL del mezzo di crescita in un matraccio Erlenmeyer sterile da 50 mL.
- Integrare il mezzo con 1 mL di glucosio filtrato al 20%. Scegli una colonia di lievito da una piastra di agar con un anello di inoculazione sterile e monouso (1-10 μL).
- Posizionare il matraccio Erlenmeyer nell'incubatrice e la coltura a 30 °C con agitazione (150-200 giri/min) fino al raggiungimento della fase esponenziale (circa 7 ore).
NOTA: Abbiamo osservato che la fase esponenziale viene raggiunta dopo ~ 15 ore quando le colonie vengono prelevate da piastre di agar che sono state coltivate a temperatura ambiente per 4 settimane. Vedi anche Nota sotto.
- Coltura Saccharomyces cerevisiae su una piastra di agar da brodo di glicerolo.
- Utilizzare una nuova piastra di agar dalla conservazione a 4 °C.
- Prendere il brodo di S. cerevisiae dal congelatore a -80 °C e metterlo in un supporto di congelamento per evitare il completo scongelamento del brodo.
- Raschiare e trasferire una piccola coltura con un ciclo di inoculazione sterile (1-10 μL) a 50 μL di terreno di coltura. Mescolare correttamente.
- Trasferire l'intero volume della coltura mista di S. cerevisiae e disperdere con un bastoncino di diffusione sterile sulla superficie della piastra di agar.
- Incubare a 30 °C per circa 48 ore fino a formare colonie di 1,5-2 mm di diametro.
NOTA: Si consiglia di colturare le colonie appena prima dell'esperimento. Le colonie di età superiore a 1 settimana richiederanno un periodo prolungato per la coltivazione in mezzi liquidi per raggiungere la fase di crescita esponenziale.
- Cultura Saccharomyces cerevisiae su un piatto di agar.
- Utilizzare piatti di agar preparati con colonie di S. cerevisiae coltivate.
- Pipettare 10 mL di terreno di coltura sterile e 1 mL di glucosio filtrato al 20% in un matraccio Erlenmeyer da 50 mL.
- Scegli una colonia di coltura di S. cerevisiae con un ciclo di inoculazione sterile e mescola con un terreno di crescita in un pallone.
- Incubare 50 minuti a 30 °C con agitazione (150-200 giri/min).
- Diluire la coltura in sospensione dieci volte con il mezzo di crescita e disperdere 50 μL di sospensione su una piastra di agar con un bastoncino di diffusione sterile.
- Incubare a 30 °C per circa 48 ore fino a quando non si osservano colonie di 1,5-2 mm.
- Avvolgere i bordi della capsula di Petri con parafilm per evitare che si secchi. Conservare a temperatura ambiente e utilizzare per un massimo di 4 settimane.
- Preparare Saccharomyces cerevisiae per il congelamento a immersione in gruppi cellulari.
- Preparare la coltura cellulare di S. cerevisiae secondo il protocollo nella sezione Coltivazione di Saccharomyces cerevisiae in sospensione (Sezione 1.3) e incubare ~7 h a 30 °C con agitazione (150-200 rpm).
- Misurare l'OD della coltura in sospensione cellulare di S. cerevisiae a 600 nm utilizzando uno spettrofotometro UV/Vis.
- Concentrare la sospensione cellulare a OD600 = 1.
- Preparare S. cerevisiae per il congelamento a immersione in un monostrato cellulare.
- Preparare la coltura cellulare di S. cerevisiae secondo il protocollo nella sezione Coltivazione di Saccharomyces cerevisiae in sospensione e incubare ~7 h a 30 °C con agitazione (150-200 rpm).
- Misurare l'OD della coltura in sospensione cellulare di S. cerevisiae a 600 nm utilizzando uno spettrofotometro UV/Vis.
- Trasferire le cellule in mezzo al tubo della centrifuga e ruotare delicatamente per 2 minuti ad una forza centrifuga relativa (900 x g).
- Scartare il mezzo dal pellet cellulare mediante pipettaggio.
- Aggiungere il mezzo fresco al pellet cellulare. Calcola il volume del mezzo per ottenere una sospensione cellulare di OD600 uguale a 30-60.
- Aggiungere glicerolo (soluzione stock al 50%) alla sospensione cellulare ad una concentrazione finale del 5% poco prima della vetrificazione.
NOTA: Il glicerolo funziona come un agente protettivo, che migliora la qualità del ghiaccio nelle regioni tra le cellule. Il glicerolo viene aggiunto alle cellule solo poco prima della vetrificazione per ridurre al minimo il suo assorbimento nella cellula.
2. Vitrificazione dell'esemplare di Saccharomyces cerevisiae
- Griglie TEM a scarica luminosa con il lato del film di carbonio rivolto verso l'alto per 30-45 s (pressione: 6-9 Pa, corrente: 7 mA).
- Impostare il robot di vetrificazione sui seguenti parametri: temperatura: 18 °C, umidità: 100%, tempo di macchia: 6 s, tempo di attesa: 5 s, ciclo di blotting: 1x e forza della macchia: 5.
NOTA: la forza della macchia è un valore specifico dello strumento e i valori ottimali della forza della macchia possono differire tra le diverse macchine. Il valore ottimale per un particolare stantuffo deve essere confermato sperimentalmente. - Preparare l'etano liquido per la vetrificazione.
- Montare il cuscinetto superficiale non assorbente sul tampone di blotting rivolto verso il campione. Utilizzare la carta da filtro per l'altro tampone.
- Scegliere la griglia a scarica a bagliore con le pinzette e montare le pinzette sullo strumento di congelamento a tuffo.
- Applicare 3,5 μL di sospensione di S. cerevisiae sul lato carbonio della griglia all'interno della camera climatica dello stantuffo. Mescolare correttamente prima di ogni applicazione sulla griglia.
- Immergere congelare la griglia nell'etano liquido.
- Trasferire la griglia dall'etano liquido a LN2. Conservare le griglie con celle vetrificate in condizioni LN2 o montarle nella cartuccia a griglia TEM per caricarle nel microscopio FIB-SEM.
3. Montaggio delle griglie TEM nella cartuccia della griglia
NOTA: il flusso di lavoro qui descritto utilizza le griglie TEM montate nella cartuccia della griglia per facilitare la gestione e il trasferimento dei campioni tra microscopi SEM e TEM. Il gruppo cartuccia è costituito da un anello a C, una griglia TEM e un anello a clip a C. Altre opzioni sono disponibili quando si lavora con strumentazione di altri produttori di microscopi. La stazione di lavoro di assemblaggio della cartuccia griglia è riempita con LN2. Il livello LN2 copre la griglia con le griglie TEM, ma il montaggio delle griglie TEM nella cartuccia viene eseguito in vapori LN2. Si consiglia vivamente di indossare una maschera protettiva o uno scudo durante la procedura di vetrificazione per evitare che la contaminazione traspiri il campione. Non lavorare con gli strumenti che hanno accumulato contaminazione da ghiaccio.
- Mettere insieme la workstation di assemblaggio della cartuccia griglia e preparare gli utensili asciutti per il ritaglio.
- Raffreddare la postazione di lavoro di assemblaggio con LN2. Raffreddare gli strumenti di ritaglio alla temperatura LN2.
- Inserire una griglia con un campione vetrificato nella postazione di lavoro di assemblaggio.
- Posizionare una griglia TEM con il campione con le celle rivolte verso il basso verso l'anello A C e fissare con una clip a C.
- Posizionare le cartucce ritagliate nella griglia e chiudere correttamente.
- Conservare le cartucce nel LN2 Dewar o caricarle nel microscopio FIB-SEM.
4. Caricamento e manipolazione del campione al microscopio FIB-SEM
NOTA: Le istruzioni sono state scritte per l'utilizzo del microscopio a doppio fascio Versa 3D dotato del sistema di preparazione crio-FIB/SEM PP3010. Le soluzioni alternative possono richiedere diversi parametri specifici; tuttavia, il concetto generale del flusso di lavoro dovrebbe essere ancora valido.
- Raffreddare il microscopio fino alla temperatura dell'azoto liquido.
- Pompare la camera del microscopio e la camera di preparazione (se presente) ad un vuoto elevato prima dell'inizio del raffreddamento (< 4 x 10-4 Pa) per prevenire la crescita della contaminazione.
- Impostare il flusso di azoto gassoso a 5 L / min per la camera di preparazione, lo stadio del microscopio e l'anticontaminatore del microscopio. Attendere che tutti i componenti raggiungano una temperatura di < -180 °C.
NOTA: La configurazione del microscopio FIB-SEM utilizzata in questo studio utilizza gas azoto raffreddato per raffreddare il suo stadio e l'anticontaminatore. Altri sistemi possono utilizzare un metodo diverso per il raffreddamento dello stadio. La pressione della camera raggiunge ~ 3 x 10-5 Pa una volta che lo stadio e l'anticontaminatore sono a -190 ° C sullo strumento utilizzato qui. Il tasso di crescita dello strato contaminante di idrocarburi sulla superficie della lamella con la configurazione sperimentale utilizzata in questo studio è di ~ 15 nm / ora.
- Caricare la cartuccia della griglia con il campione al microscopio.
- Assemblare e raffreddare la stazione di carico alla temperatura LN2.
- Posizionare la navetta (portacampioni), la scatola della griglia con il campione, l'apriscatole della griglia e le pinzette nella stazione di carico raffreddata.
- Trasferire con attenzione la cartuccia della griglia con il campione rivolto verso l'alto nella navetta.
- Capovolgere la navetta all'interno della stazione di carico nella posizione di carico.
- Caricare nel microscopio.
- Facoltativamente, rivestire il campione con strati protettivi metallici.
NOTA: Un forte effetto di carica può essere osservato quando si immagina materiale biologico idratato congelato SEM. Inoltre, l'imaging di campioni biologici con FIB (anche a basse correnti) induce un rapido danno al campione. Pertanto, un rivestimento aggiuntivo del campione potrebbe essere eseguito all'interno del microscopio FIB-SEM, per proteggere la superficie del campione.- Depositare lo strato protettivo con platino organometallico mediante il sistema di iniezione del gas (GIS).
- Impostare il campione all'altezza eucentrica (punto di coincidenza per l'imaging con elettroni e ioni).
- Inclinare il palco a 45° (campione inclinato di 90° rispetto al fascio di elettroni).
- Spostare il palco nell'asse z di4 mm al di sotto dell'altezza eucentrica.
- Impostare l'ago GIS a 26-30 °C.
- Deposita ~ 300-1000 nm dello strato di platino organometallico sulla griglia con il campione biologico (corrisponde a 30-120 s della deposizione GIS).
NOTA: Di solito applichiamo GIS per 30 s per campioni con piccoli cluster cellulari e 45 s per campioni con un monostrato delle cellule.
- Sputter riveste la superficie del campione con uno strato metallico conduttivo.
- Depositare ~ 10 nm dello strato metallico (Ir, Au, Pt) sulla griglia con un campione biologico.
- Depositare lo strato protettivo con platino organometallico mediante il sistema di iniezione del gas (GIS).
- Impostare i parametri del microscopio per la preparazione della lamella
- Per FIB, utilizzare i seguenti parametri: alta tensione = 30 kV, corrente = 10 pA (imaging), 10 pA-3 nA (FIB-fresatura)
- Per SEM, utilizzare i seguenti parametri: alta tensione = 2-5 kV, dimensione spot = 4,5, corrente = 8-27 pA.
- Impostare la rotazione della scansione su 180° per entrambi i fasci.
- Impostare l'inclinazione del palco: angolo di fresatura 6-11° (corrisponde all'inclinazione dello stage di 13°–18° per la navetta del campione con pre-inclinazione di 45° e microscopio FIB/SEM con angolo di 52° tra colonna SEM e FIB).
5. Preparazione della lamella Saccharomyces cerevisiae
- Controllare la qualità della griglia e selezionare un cluster adatto di Saccharomyces cerevisiae.
- Verificare che la griglia TEM sia correttamente cancellata da entrambi i lati senza acqua aggiuntiva attorno al cluster di celle o sul lato posteriore della griglia.
NOTA: per controllare il retro della griglia, ruotare lo stage a -10° e visualizzare la griglia con FIB. - Selezionare i cluster cellulari ottimali per la preparazione delle lamelle in base alle seguenti raccomandazioni.
- Posizionare i cluster di celle al centro della griglia. L'area di fresatura non deve estendersi al di fuori del quadrato con dimensioni 1100 x 1100 μm posizionate al centro della griglia (550 μm in ogni direzione dal centro della griglia).
- Posizionare i cluster di celle nella parte centrale del quadrato della griglia senza sovrapposizioni alla barra della griglia.
- Posizionare i cluster di celle nel quadrato della griglia con un foglio di carbonio compatto e forato senza crepe.
- Assicurarsi che il cluster non sia circondato da contaminazione da ghiaccio.
- Verificare che la griglia TEM sia correttamente cancellata da entrambi i lati senza acqua aggiuntiva attorno al cluster di celle o sul lato posteriore della griglia.
- Selezionare la posizione di fresatura ottimale nel monostrato Saccharomyces cerevisiae.
- Assicurarsi che le barre della griglia che circondano il monostrato della cella sul quadrato della griglia selezionato siano visibili.
- Selezionare la posizione ottimale nel monostrato cellulare in base alle seguenti raccomandazioni. Le aree selezionate per la fresatura devono soddisfare i seguenti criteri:
- Avere la regione di interesse nel centro della griglia. L'area di fresatura non deve estendersi al di fuori del quadrato di dimensioni 1100 x 1100 μm posizionato al centro della griglia (550 μm in ogni direzione dal centro della griglia).
- Avere la regione di interesse nella parte centrale del quadrato della griglia senza sovrapposizioni con la barra della griglia.
- Non circondare il monostrato cellulare con contaminazione da ghiaccio.
- Preparare la lamella S. cerevisiae con crio-FIB.
NOTA: la serie di fresatura viene generata e centrata rispetto alla regione di interesse. Cryo-FIBM viene eseguito in sequenza con più fasi di fresatura eseguite a diverse impostazioni FIB. La lamella con uno spessore di circa 2 μm viene inizialmente fresata utilizzando corrente elevata (0,3-3 nA). La lamella viene quindi gradualmente assottigliata a 500 nm. La fase di fresatura fine a basse correnti (10-30 pA) viene utilizzata per finalizzare la lamella a ~ 100-200 nm di spessore.- Imposta una regione di interesse (ROI) all'altezza eucentrica e salva questa posizione.
NOTA: il punto di coincidenza deve essere determinato e salvato separatamente per ogni posizione. L'altezza eucentrica viene impostata inclinando il palco a 0° e centrando il ROI spostando il palco nella direzione x e y. Il palco viene quindi inclinato a 25° e il ROI viene riportato al centro dell'area scansionata modificando la posizione dell'asse zdello stage. Infine, il palco è inclinato all'indietro a 13°-18° per la fresatura. - Definite una serie di fresatura rettangolare al di sopra del ROI con una direzione di scansione dall'alto verso il basso.
- Definite una serie di fresatura rettangolare al di sotto del ROI con una direzione di scansione dal basso verso l'alto.
- Definite il modello di fresatura rettangolare inattivo che copre la regione di interesse per una stima approssimativa dello spessore della lamella. Questo modello non viene fresato durante la preparazione della lamella.
- Contrassegnare tutti i motivi e impostare la larghezza della lamella(dimensione x). La larghezza del modello di fresatura non deve superare i 2/3 della larghezza del cluster. Ciò corrisponde a 8-15 μm nella maggior parte dei casi.
- Impostare i parametri per le fasi di fresatura del sgrossatura
- Corrente FIB: 0,3–3,0 nA; spessore finale della lamella: 1,5–2 μm; larghezza dell'area FIBM: 8-12 um; inclinazione del palco: 13-17°; durata: 8 minuti; modelli di fresatura attivi: superiore e inferiore.
- Corrente FIB: 0,3–1,0 nA; spessore lamellare finale: 1 μm; larghezza dell'area FIBM: 7,5-11,5 μm; inclinazione del palco: 13-17°; durata: 8 minuti; modelli di fresatura attivi: superiore e inferiore.
- Corrente FIB: 100-300 pA; spessore lamellare finale: 0,5 μm ; larghezza dell'area FIBM: 7,5-11,5 μm; inclinazione del palco: 13-17°; durata: 8 minuti; modelli di fresatura attivi: superiore e inferiore.
- Corrente FIB: 30-100 pA; spessore lamellare finale: 0,3 μm ; larghezza dell'area FIBM: 7,5-11,5 μm; inclinazione del palco: 13-17°; durata: 8 minuti; modelli di fresatura attivi: superiore e inferiore.
- Impostare i parametri per la fase di fresatura fine:
- Corrente FIB: 10-30 pA; spessore lamellare finale: <0,2 um; larghezza dell'area FIBM: 7-11 μm; inclinazione del palco: 13-17° (+1°); durata: 12 minuti; modelli di fresatura attivi: superiore.
NOTA: Le fasi di fresatura approssimativa (5.3.6) vengono eseguite in sequenza per ogni lamella. Al contrario, la fase di fresatura fine (5.3.7) non segue direttamente la fresatura grezza, ma le fasi di fresatura fine vengono eseguite in sequenza per tutte le lamelle alla fine della sessione per ridurre al minimo la contaminazione da idrocarburi sulla superficie della lamella. Un'inclinazione extra dello stadio di +1° viene utilizzata durante la fase di fresatura fine per aumentare l'uniformità dello spessore della lamella su tutta la sua lunghezza.
- Corrente FIB: 10-30 pA; spessore lamellare finale: <0,2 um; larghezza dell'area FIBM: 7-11 μm; inclinazione del palco: 13-17° (+1°); durata: 12 minuti; modelli di fresatura attivi: superiore.
- Imposta una regione di interesse (ROI) all'altezza eucentrica e salva questa posizione.
6. Trasferimento della lamella Saccharomyces cerevisiae a cryo-TEM
- Preparare un Dewar correttamente essiccato e riempirlo con LN2.
- Scaricare i campioni con lamelle dal microscopio FIB/SEM in condizioni crio-condizioni, trasferirli in una scatola a griglia e conservarli in un Dewar di stoccaggio LN2 per la conservazione a lungo termine. In alternativa, caricare le griglie direttamente in crio-TEM.
- È importante il corretto orientamento della lamella rispetto all'asse di inclinazione dello stadio crio-TEM (vedere il video di accompagnamento alle 8:10 per maggiori dettagli). Assicurarsi che la direzione di fresatura delle lamelle preparate sia perpendicolare all'asse di inclinazione dello stadio crio-TEM.
- Pre-inclinare lo stadio crio-TEM per compensare l'inclinazione della lamella rispetto al piano della griglia e raccogliere la serie di inclinazione utilizzando lo schema dose-simmetrico19.
NOTA: la grandezza della pre-inclinazione (tipicamente 6-8°) è determinata dall'angolo tra il piano della griglia TEM e la direzione FIB durante la microlavorazione. La posizione del bordo anteriore della lamella nell'immagine in crio-TEM può essere utilizzata per determinare il segno dell'angolo di pre-inclinazione. Per questo, il senso della rotazione dello stadio del microscopio deve essere noto. Nella nostra configurazione sperimentale, la posizione del bordo anteriore della lamella sul lato destro dell'immagine scattata in modalità NANOSONDA CORRISPONDE ALLA PRE-inclinazionE NEGATIVA.
Risultati
La coltura di Saccharomyces cerevisiae è stata raccolta nel mezzo della fase di crescita esponenziale. Abbiamo preparato due tipi di campioni in cui le celle sono state distribuite come piccoli gruppi di più celle sulla superficie della griglia TEM(Figura 1A,C)o hanno formato un monostrato continuo su singoli quadrati della griglia TEM (Figura 1B, D). Il fattore discriminante per la preparazione del campione con isole cellulari distinte o con il monostrato cellulare è la concentrazione della coltura cellulare applicata alla griglia TEM. La coltura cellulare raccolta è stata concentrata a OD600 = 1,0 per il primo caso, o a OD600 = da 30 a 60 per il secondo caso, rispettivamente. Il campione per la preparazione del monostrato cellulare è stato ulteriormente integrato con glicerolo v/v al 5% prima della vetrificazione. Il glicerolo è fondamentale per la vitrificazione della soluzione tampone, che riempie lo spazio tra le cellule (Figura 2) in quanto i riflessi del tampone cristallino possono essere dannosi per il corretto tracciamento posizionale e la messa a fuoco durante la raccolta dei dati crio-ET.
Inoltre, la coltura della sospensione del lievito è stata cancellata contro il materiale non assorbente come il tampone in PTFE o il tampone personalizzato stampato in 3D in materiale FlexFill 98A. La carta assorbente è stata posizionata solo sul retro della griglia rispetto all'applicazione campione (back-blotting). La strategia di back-blotting è consigliata per il congelamento del tuffo della coltura in sospensione poiché il blotting con la carta da filtro da entrambi i lati provoca l'adesione delle cellule alla carta assorbente (Figura 1E).
Il protocollo qui descritto utilizza griglie TEM ritagliate nella cartuccia della griglia, che forma un supporto stabile per la griglia e facilita la gestione del campione dopo la vetrificazione. Ciò impone la necessità che altri porta campioni e navette nel microscopio FIB/SEM e TEM possano accettare una cartuccia a griglia di questo tipo.
Dopo il trasferimento del campione nel microscopio FIB/ SEM, il campione è stato prima rivestito con uno strato di 0,3-1,0 μm di metilciclopentandienil platino utilizzando il sistema di iniezione di gas del microscopio (GIS). Un ulteriore strato di iridio inorganico è stato sputtered sulla superficie del campione per indurire lo strato GIS e rendere la superficie conduttiva. Le lamelle sono state fresate in più fasi (Figura 3) dove (I) la corrente di fresatura, (II) la larghezza della lamella e (III) la distanza dell'area di fresatura sopra e sotto i campioni sono state ridotte in modo graduale. La fase finale di fresatura ("lucidatura") è stata effettuata a bassa corrente (10-30 pA) solo dal lato superiore della lamella e con il campione inclinato di un ulteriore 1° verso la trave Ga+. L'utilizzo del protocollo descritto ha portato in media a 8-10 lamelle preparate su due griglie TEM in una sessione di 6-8 ore.
Le griglie TEM con le lamelle sono state successivamente trasferite in un microscopio elettronico a trasmissione. Le lamelle sono state prima schermate e solo quelle che mostravano tende minime (artefatti derivanti da fresatura irregolare sulla superficie della lamella), basso livello di contaminazione superficiale e buon contrasto cellulare (di solito osservato per lamelle con spessore <200 nm) sono state selezionate per l'acquisizione dei dati crio-ET. Inoltre, le lamelle contenenti crepe su tutta la lunghezza sono state scartate dalla raccolta dei dati. In generale, circa il 50% delle lamelle trasferite a TEM erano adatte all'acquisizione dei dati. Le serie Tilt sono state raccolte sul rivelatore di elettroni diretti post-GIF K2 con la fessura di selezione dell'energia impostata a 20 eV. La raccolta dei dati è stata effettuata nel software SerialEM18 e le serie di inclinazione sono state raccolte utilizzando uno schema di simmetria di dose19 con l'intervallo di inclinazione di ±60° e l'incremento di 3°. I dati sono stati acquisiti all'ingrandimento corrispondente alla dimensione dei pixel di 3,47 A/px. La dose complessiva di 65 e/Å2 è stata uniformemente distribuita sui singoli sotto-fotogrammi. Le immagini di inclinazione sono state raccolte come un insieme di tre fotogrammi, che sono stati successivamente corretti per il movimento e il danno da radiazioni durante l'acquisizione dei dati utilizzando il programma MotionCor220. I parametri della funzione di trasferimento del contrasto sono stati stimati utilizzando Ctffind421. La serie tilt è stata elaborata in eTomo18. La routine di tracciamento delle patch è stata utilizzata per allineare le immagini. Il tomogramma è stato ricostruito utilizzando un algoritmo di retroproiezione ponderato dopo 2x binning delle immagini, e successivamente filtrato utilizzando un filtro simile a SIRT (impostato su 8 iterazioni) in IMOD18. La segmentazione del tomogramma è stata eseguita manualmente nel software Amira22. I tomogrammi ricostruiti forniscono una rappresentazione ad alta risoluzione dell'interno cellulare del lievito e ci consentono di osservare organelli come vacuoli o mitocondri ad un alto livello di dettaglio o studiare complessi macromolecolari come microtubuli o complessi di pori nucleari in situ e in condizioni quasi native (Figura 4).

Figura 1: Immagini FIB e SEM di S. cerevisiae vetrificate
Immagini FIB (A) e SEM (C) dei piccoli grappoli di lievito vetrificati sulla griglia TEM. Immagini FIB (B) e SEM (D) del lievito che formano un monostrato continuo sulla superficie della griglia. Il campione è stato rivestito con GIS e strato di irridio prima dell'imaging. Le barre di scala nei pannelli A-B corrispondono a 10 μm. Il campione di lievito viene cancellato contro materiale non assorbente come PTFE o FlexFill 98A (verde) e con la carta assorbente posizionata dal retro della griglia (bianco, E). Fare clic qui per visualizzare una versione più grande di questa figura.
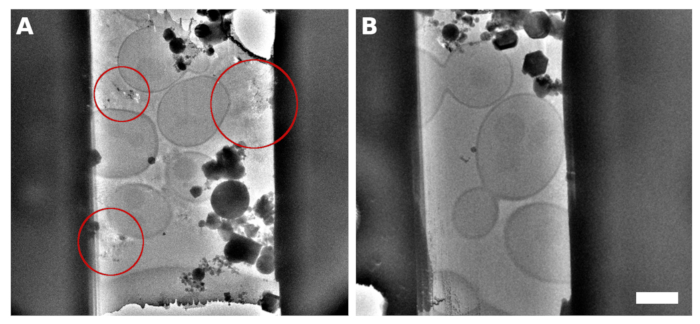
Figura 2: S. cerevisiae lamellae
Un'immagine TEM di una lamella microlavorato dal campione con lievito monostrato continuo sulla superficie della griglia. I riflessi osservati tra le cellule contenevano un mezzo/tampone impropriamente vetrificato(A,evidenziato con cerchi rossi). Un'immagine TEM di lamelle generate sul lievito vetrificate in un monostrato continuo con l'aggiunta del 5% di glicerolo nel mezzo/tampone(B). La barra della scala corrisponde a 2 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
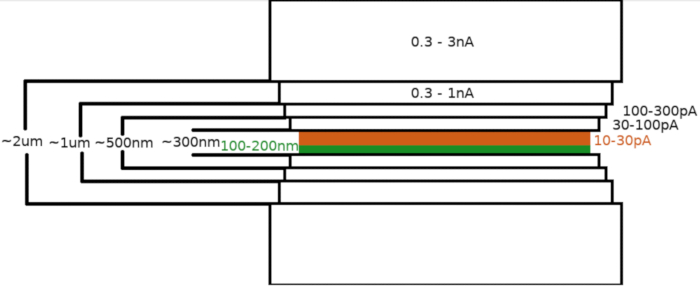
Figura 3: Flusso di lavoro Cryo-FIBM
Rappresentazione schematica del processo di fresatura delle lamelle. Le fasi iniziali di fresatura approssimativa vengono eseguite a correnti FIB elevate da entrambi i lati della posizione provvisoria della lamella (evidenziata in verde) mentre la fase finale di lucidatura viene eseguita solo dal lato superiore e a bassa corrente FIB (evidenziata in arancione, vedere il video di accompagnamento alle 6:33). Fare clic qui per visualizzare una versione più grande di questa figura.
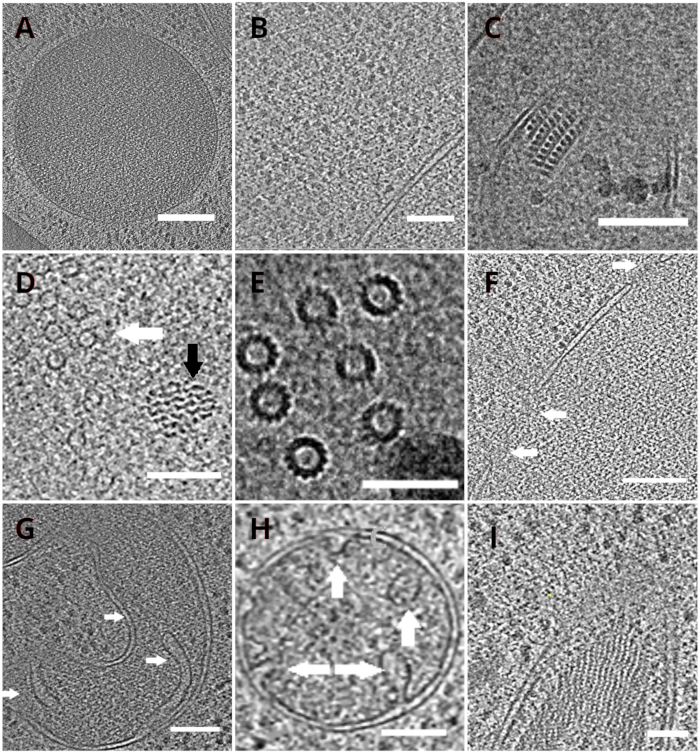
Figura 4: Organelli di lievito e complessi macromolecolari rappresentati da crio-ET
Fette dei tomogrammi ricostruiti raffiguranti un vacuolo (A, barra della scala: 200 nm), ribosomi (B, barra della scala: 200 nm), un nucleo paracristallino di perossisoma (C, barra della scala: 100 nm), microtubule (freccia bianca) in prossimità di struttura fibrosa non identificata (freccia nera, D, barra della scala: 100 nm), dettagli di più microtubuli (E, barra della scala: 50 nm), una membrana nucleare con pori indicati da frecce (F, scala bar 200nm), mitocondrio(G,H,barra scala: 100 nm, le frecce indicano singole cristae), un fascio di strutture filamentose non identificate(I,scala barra: 100 nm). I pannelli B, C, D, E, G contengono una sezione di tomogrammi preparati da piccoli gruppi di cellule mentre le sezioni di tomogrammi raccolti sulle lamelle da un monostrato delle cellule sono mostrate nei pannelli A, F, H, I. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
La preparazione dei campioni cellulari per crio-ET è un flusso di lavoro complesso che richiede l'utilizzo di diversi strumenti di fascia alta. La qualità del campione può essere compromessa durante ogni fase di preparazione che influenza il throughput dell'intero protocollo. Inoltre, la necessità del trasferimento del campione tra singoli strumenti presenta un ulteriore rischio di contaminazione o devitrificazione del campione. Pertanto, l'ottimizzazione delle singole fasi nel flusso di lavoro di preparazione del campione è di grande importanza per aumentare la produttività e la riproducibilità del flusso di lavoro di preparazione della lamella. Il protocollo qui presentato descrive la preparazione ottimizzata di Saccharomyces cerevisiae per la caratterizzazione strutturale di complessi macromolecolari in situ mediante crio-ET.
Il protocollo descrive la preparazione di due tipi di campioni di lievito che si differenziano principalmente per la concentrazione delle cellule sulla griglia TEM. Entrambi i campioni di lievito hanno prodotto lamelle di alta qualità per la crio-ET e la selezione del tipo di campione può essere effettuata in conformità con gli obiettivi del particolare studio. Il lievito forma gruppi isolati di poche cellule sparse casualmente sulla superficie della griglia nel primo caso, mentre un monostrato continuo di cellule è presente sulla superficie della griglia TEM per il secondo tipo di campione. Il primo è adatto per la preparazione rapida della lamella grazie al piccolo volume del materiale che deve essere fresato. La lamella finale è abbastanza corta e, quindi, contiene solo 2-4 sezioni trasversali cellulari. Le aree adatte per la preparazione del campione sono distribuite in modo casuale sulla superficie della griglia, compresi i quadrati della griglia, il che può limitare parzialmente l'automazione del flusso di lavoro di preparazione della lamella. Quest'ultimo tipo di campione richiede l'utilizzo di correnti più grandi durante la fase iniziale di fresatura per mantenere il tempo di fresatura complessivo. Inoltre, questo tipo di campione è più incline agli artefatti che derivano da fresatura irregolare (tendaggi). Pertanto, il GIS viene sputterato sulla superficie del campione per un periodo più lungo del 50% rispetto al campione con piccoli cluster cellulari per formare uno strato protettivo più spesso. Successivamente, il campione viene sputtered con uno strato aggiuntivo di iridio (in alternativa platino o oro) per polimerizzare lo strato GIS, renderlo più rigido e aumentare la conduttività superficiale del campione. FIBM di aree aggiuntive su ciascun lato della lamella (~ 2-5 μm dal bordo della lamella) durante la prima fase della fresatura della lamella ruvida è stato trovato utile per diminuire il numero di lamelle rotte molto probabilmente a causa della diminuzione della tensione nella sezione trasversale finale23. La lamella finale è lunga e contiene ~ 10 sezioni trasversali cellulari, il che aumenta il numero di regioni adatte alla crio-ET. La vetrificazione impropria del mezzo o del tampone tra le cellule può essere facilmente attenuata dall'aggiunta del crioprotente alla soluzione tampone (5% di glicerolo utilizzato in questo studio). Poiché la maggior parte dei quadrati sono adatti per la preparazione delle lamelle, il campione con cellule organizzate in un monostrato continuo è adatto per la preparazione di lamelle non supervisionate.
Un altro aspetto importante nel flusso di lavoro di preparazione della lamella è il trasferimento al microscopio elettronico a trasmissione e il corretto posizionamento della lamella sull'asse di inclinazione dello stadio del microscopio. In modo ottimale, l'asse principale della lamella è perpendicolare all'asse di inclinazione del microscopio che consente il tracciamento e la messa a fuoco all'altezza della regione ripresa e impedisce ai bordi della lamella di schermare il campo visivo ad angoli di inclinazione elevati. Quando si raccolgono i dati crio-ET utilizzando lo schema dose-simmetrico,18 il campione deve essere inizialmente ruotato al microscopio per compensare l'inclinazione della lamella rispetto al piano della griglia.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato supportato da Instruct-Ultra (Grant 731005), iNEXT-Discovery (Grant 871037) finanziato dal programma Horizon 2020 della Commissione Europea e dall'infrastruttura di ricerca CIISB, un Centro Instruct-ERIC (LM2018127). Riconosciamo il supporto ottenuto da Thermo Fisher Scientific Brno.
Materiali
| Name | Company | Catalog Number | Comments |
| Agar | Himedia | MB053 | |
| Glucose | PENTA | 12020-31000 | |
| Glycerol | Merck | G5516-1L | |
| ethane | Messer | 1007 | |
| LN2 | Lineq | LN2-1L | |
| Peptone | Merck | P5905-1KG | |
| Saccharomyces cerevisiae | ATCC | 201388 | strain BY4741 |
| Tweezers | Dumont | T539 | |
| Yeast extract | Duchefa Biochemie | Y1333.1000 | |
| Disposable | |||
| Blotting papers | Ted Pella | 47000-10 | |
| C-clip | ThermoScientific | 9432 909 97551 | |
| C-clip ring | ThermoScientific | 9432 909 97561 | |
| Spreading sticks | Merck | Z376779-1PAK | |
| Sterile inoculation loops | BRAND | BR452201-1000EA | |
| Sterile plastic Petri dishes | Sigma | SIAL0166 | |
| TEM grids | Quantifoil | 4420G-XA | |
| Equipment | |||
| Autoclave | Systec | 101291545 | |
| balances | BEL | M124A | |
| Cryo-FIB/SEM microscope | ThermoScientific | 1006123 | |
| Cryo-TEM microscope | ThermoScientific | 9432 057 03301 | |
| Laminar flow box | Telstar | AH5 | |
| Plasma cleaner | Gatan | 955.82001 | |
| Shaking incubator | New Brunswick | M1282-0002 | |
| UV/VIS spectrophotometer | WPA | S800 | |
| Vitrification robot | ThermoScientific | 9432 053 50621 |
Riferimenti
- McMullan, G., Faruqi, A., Clare, D., Henderson, R. Comparison of optimal performance at 300keV of three direct electron detectors for use in low dose electron microscopy. Ultramicroscopy. 147, 156-163 (2014).
- Zivanov, J., et al. New tools for automated high-resolution cryo-EM structure determination in RELION-3. eLife. 7, (2018).
- Dubochet, J., McDowall, A. W. Vitrification of pure water for electron microscopy. Journal of Microscopy. 124, 3-4 (1981).
- Villa, E., Schaffer, M., Plitzko, J. M., Baumeister, W. Opening windows into the cell: focused-ion-beam milling for cryo-electron tomography. Current Opinion in Structural Biology. 23, 771-777 (2013).
- Mahamid, J., et al. Visualizing the molecular sociology at the HeLa cell nuclear periphery. Science. 351 (6276), 969-972 (2016).
- Schur, F. K. Toward high-resolution in situ structural biology with cryo-electron tomography and subtomogram averaging. Current Opinion in Structural Biology. 58, 1-9 (2019).
- O'Reilly, F. J., et al. In-cell architecture of an actively transcribing-translating expressome. Science. 369, 554-557 (2020).
- Rice, W. J., et al. Routine determination of ice thickness for cryo-EM grids. Journal of Structural Biology. 204, 38-44 (2018).
- Al-Amoudi, A., Norlen, L. P. O., Dubochet, J. Cryo-electron microscopy of vitreous sections of native biological cells and tissues. Journal of Structural Biology. 148, 131-135 (2004).
- Al-Amoudi, A., Studer, D., Dubochet, J. Cutting artefacts and cutting process in vitreous sections for cryo-electron microscopy. Journal of Structural Biology. 150, 109-121 (2005).
- Pierson, J., et al. Improving the technique of vitreous cryo-sectioning for cryo-electron tomography: electrostatic charging for section attachment and implementation of an anti-contamination glove box. Journal of Structural Biology. 169, 219-225 (2010).
- Dubochet, J., et al. How to "read" a vitreous section. Methods in Cell Biology. 79, 385-406 (2007).
- Rigort, A., et al. Focused ion beam micromachining of eukaryotic cells for cryoelectron tomography. Proceedings of the National Academy of Sciences. 109, 4449-4454 (2012).
- Schaffer, M., et al. Cryo-focused Ion Beam Sample Preparation for Imaging Vitreous Cells by Cryo-electron Tomography. Bio-protocol. 5, (2015).
- Wagner, F. R., et al. Preparing samples from whole cells using focused-ion-beam milling for cryo-electron tomography. Nature Protocols. 15 (6), 2041-2070 (2020).
- Buckley, G., et al. Automated cryo-lamella preparation for high-throughput in-situ structural Biology. Journal of Structural Biology. 210 (2), 107488 (2020).
- Zachs, T., et al. Fully automated, sequential focused ion beam milling for cryo-eletron tomography. eLife. e52286, (2020).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152, 36-51 (2005).
- Hagen, W. J., Wan, W., Briggs, J. A. Implementation of a cryo-electron tomography tilt-scheme optimized for high resolution subtomogram averaging. Journal of Structural Biology. 197, 191-198 (2017).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14, 331-332 (2017).
- Rohou, A., Grigorieff, N. CTFFIND4: Fast and accurate defocus estimation from electron micrographs. Journal of Structural Biology. 192, 216-221 (2015).
- Stalling, D., Westerhoff, M., Hege, H. -. C. Amira: A highly interactive system for visual data analysis. The Visualization Handbook. , 749-767 (2005).
- Wolff, G., et al. Mind the gap: Micro-expansion joints drastically decrease the bending of FIB-milled cryo-lamellae. Journal of Structural Biology. 208, 107389 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon