Method Article
Quantificazione della competizione interbatterica utilizzando l'imaging a fluorescenza a singola cellula
In questo articolo
Riepilogo
Questo manoscritto descrive un metodo per utilizzare la microscopia a fluorescenza a singola cellula per visualizzare e quantificare la competizione batterica in cocoltura.
Abstract
La competizione interbatterica può avere un impatto diretto sulla struttura e sulla funzione dei microbiomi. Questo lavoro descrive un approccio di microscopia a fluorescenza che può essere utilizzato per visualizzare e quantificare le interazioni competitive tra diversi ceppi batterici a livello di singola cellula. Il protocollo qui descritto fornisce metodi per approcci avanzati nella preparazione di vetrini su microscopi a epifluorescenza sia verticale che invertita, tecniche di imaging a cellule vive e time-lapse e analisi quantitativa delle immagini utilizzando il software open source FIJI. L'approccio in questo manoscritto delinea la quantificazione delle interazioni competitive tra le popolazioni simbiotiche di Vibrio fischeri misurando il cambiamento di area nel tempo per due ceppi coincubati che esprimono diverse proteine fluorescenti da plasmidi stabili. Vengono descritti metodi alternativi per ottimizzare questo protocollo in sistemi modello batterico che richiedono condizioni di crescita diverse. Sebbene il saggio qui descritto utilizzi condizioni ottimizzate per V. fischeri,questo approccio è altamente riproducibile e può essere facilmente adattato per studiare la competizione tra isolati coltivabili da diversi microbiomi.
Introduzione
Questo articolo delinea un metodo per quantificare la competizione batterica a livello di singola cellula utilizzando la microscopia a fluorescenza. La struttura e la funzione delle comunità microbiche è spesso modellata dalle interazioni competitive tra microbi e in molti casi la caratterizzazione di queste interazioni richiede l'osservazione di diversi ceppi batterici nella coincubazione1,2,3,4,5,6,7,8 . Tradizionalmente, la competizione batterica è quantificata a livello di popolazione contando le unità formanti colonie (CFU) di ceppi inibitori e bersaglio prima e dopo un periodo di coincubazione2,9. I meccanismi di competizione microbica sono ampiamente distribuiti tra i batteri e possono fare affidamento sulla diffusione o sul contatto cellula-cellula per inibire le cellule bersaglio10,11, 12,13, 14,15,16,17,18,19.
Sebbene i ceppi batterici siano spesso osservati nella coincubazione a livello di popolazione, questo manoscritto delinea un test per la quantificazione a singola cellula della competizione batterica. Inoltre, questo lavoro include suggerimenti per adattare il protocollo per l'uso con altre specie batteriche. Mentre le tecniche specifiche in questo articolo sono utilizzate per studiare la competizione intraspecifica dipendente dal contatto tra ceppi del batterio simbiotico Vibrio fischeri20,21,22, possono essere adattati per la competizione tra molti organismi. Questo articolo fornisce istruzioni per la configurazione del vetrino su microscopi verticali e invertiti e tutte le analisi sono descritte utilizzando il software open source FIJI23 in modo che il metodo possa essere utilizzato dai ricercatori con accesso a diverse configurazioni di imaging e programmi di analisi. Data l'importanza di studiare la competizione microbica sia a livello di popolazione che a livello di singola cellula, questo metodo sarà una risorsa preziosa per i ricercatori per quantificare le interazioni competitive, in particolare quelle che non hanno accesso a software di analisi proprietari.
Protocollo
1. Ottimizzazione dei ceppi batterici
- Scegli due ceppi batterici per i test di competizione batterica a singola cellula. Qui vengono utilizzati due ceppi di V. fischeri: un ceppo bersaglio (ES11424) e un ceppo inibitore (MJ1125) che è noto per uccidere il ceppo bersaglio utilizzando il sistema di secrezione di tipo VI sul cromosoma II (T6SS2)1, che è un meccanismo di uccisione dipendente dal contatto.
- Determinare i controlli appropriati per l'esperimento. In questo esempio, il controllo appropriato consiste nell'incubare sia i ceppi inibitori mutanti wild-type che quelli mutanti T6SS con il ceppo bersaglio per quantificare l'effetto dell'uccisione mediata da T6SS.
NOTA: Ulteriori controlli possono includere un ceppo bersaglio che esprime i geni di immunità necessari per prevenire l'uccisione dipendente da T6SS o un ceppo mutante inibitore che esprime copie wild-type dei geni mutati in trans per ripristinare l'attività di T6SS1. - Quando possibile, trasformare ceppi con plasmidi stabili che codificano geni per diverse proteine fluorescenti (ad esempio, GFP o RFP) per distinguere visivamente i tipi di ceppo al microscopio. Qui, il ceppo inibitore è etichettato con un plasmide che codifica GFP (pVSV102) e il ceppo bersaglio è etichettato con un plasmide che codifica dsRed (pVSV208)26.
NOTA: Se non è possibile utilizzare plasmidi stabili, è possibile introdurre tag fluorescenti sul cromosoma batterico per lavisualizzazione 27,28. - Durante il periodo di ottimizzazione iniziale, immagini di colture clonali dei ceppi etichettati sotto ciascuno dei filtri fluorescenti che verranno utilizzati durante l'esperimento per garantire che le cellule siano visibili solo nel canale previsto. Ad esempio, assicurarsi che un ceppo con tag GFP sia visibile solo nel canale FITC.
2. Preparazione del tampone di agarose
- Preparare la soluzione di tampone di agarose sciogliendo l'agarose (p/v) al 2% a bassa fusione in mPBS. Riscaldare brevemente la soluzione nel microonde e vortice fino a quando l'agarose è completamente dissolto. Mantenere questa soluzione calda ponendola a bagnomaria a 55°C fino al momento dell'uso. Vedere la sezione Discussione per ulteriori informazioni sulla preparazione dei tamponi di agarose.
NOTA: Qui, mPBS è stato preparato aggiungendo 20 g / L NaCl allo standard 1x PBS. - Avvolgere un pezzo di nastro da laboratorio attorno a un vetrino cinque volte. Ripetere questo processo una seconda volta sulla stessa diapositiva in modo che la distanza tra i due pezzi di nastro sia leggermente inferiore alla larghezza di un coperchio (Figura 1A). Ad esempio, se si utilizzano coverslip da25 mm 2, i pezzi di nastro devono essere distanziati di circa 20 mm l'uno dall'altro.
NOTA: mentre il numero di volte in cui il nastro viene avvolto attorno alla diapositiva può essere modificato per regolare lo spessore del tampone di agarose, è importante che gli strati di nastro siano della stessa altezza su entrambi i lati del vetrino in modo che il tampone di agarose rimanga piatto. - Pipettare la soluzione calda di agarose tra i due pezzi di nastro e immediatamente sopra con una copertura in modo che si appoggi sui pezzi di nastro. Ciò garantirà che la superficie del tampone di agarose rimanga piatta. Il volume della soluzione di agarose pipettata in questa fase dovrebbe essere sufficiente affinché il coperchio entri in contatto con il liquido e spinga fuori eventuali bolle nella soluzione di agarose. Per questa particolare configurazione, sono sufficienti 200 μL di agarose caldo.
- Lasciare solidificare il tampone di agarosio a temperatura ambiente per almeno 1 ora prima del test di coincubazione. Il passo 2.2 produrrà un tampone di agarose di circa 20 mm2.
- Tagliare questo tampone di agarose con una lama di rasoio in quattro pad da 5 mm2 da utilizzare per l'imaging.
NOTA: I tamponi di agarose possono essere preparati fino a una settimana prima dell'esperimento e conservati a 4 ° C in una piastra di Petri vuota e sterile sigillata con parafilm per evitare l'essiccazione.
3. Preparare i ceppi per la co-incubazione
- Strisciare ogni ceppo da utilizzare nel saggio di coincubazione da -80 °C su piastre di agar LBS integrate con gli antibiotici appropriati e incubare durante la notte a 24 ° C. Per questo esempio, vengono utilizzati tre ceppi: il ceppo inibitore wild-type, il mutante del sistema di secrezione di tipo VI e il ceppo bersaglio.
- Il giorno successivo, iniziare le colture notturne in duplicato biologico raccogliendo due colonie da ciascun ceppo e riconsociendole in terreno LBS integrato con gli antibiotici appropriati e incubare durante la notte a 24 ° C con agitazione a 200 giri / min.
- La mattina seguente, la sottocoltura di ogni replica biologica 1:1000 in terreno LBS fresco senza antibiotici e incuba a 24 ° C con agitazione per 4-5 ore o fino a quando le cellule raggiungono un OD600 di ~ 1,5.
NOTA: potrebbe essere necessario ottimizzare i tempi dei passaggi 3.1, 3.2 e 3.3 per le diverse specie batteriche poiché il loro tasso di crescita può variare in modo sostanziale. Per questo test, le cellule dovevano essere in fase di mid-log all'inizio del test di coincubazione.
4. Ceppi batterici Coincubate
- A partire dalle colture mid-log dal passaggio 3.3, misurare e registrare la densità ottica a 600 nm (OD600) per tutti i campioni.
- Normalizzare ogni campione ad un OD600 = 1.0, che corrisponde a circa 109 CFU/mL per V. fischeri,diluendo la coltura con mezzo LBS.
- Mescolare i due ceppi concorrenti insieme in un rapporto 1:1 basato sul volume aggiungendo 30 μL di ciascun ceppo normalizzato a un tubo da 1,5 ml etichettato. Vortice la coltura a ceppo misto per 1-2 s.
NOTA: In alcuni casi, può essere opportuno mescolare le cocolture in rapporti diversi. Ad esempio, quando un ceppo cresce molto più velocemente dell'altro, potrebbe essere necessario iniziare il ceppo a crescita più lenta con un vantaggio numerico per osservare la concorrenza. L'ottimizzazione può anche essere necessaria se OD600 non corrisponde a CFU/mL simili per entrambi i ceppi. - Ripetere il passaggio 4.3 per ogni replicazione biologica e trattamento. Nell'esempio mostrato qui, ciò si tradurrà in un totale di quattro tubi a ceppo misto: due repliche biologiche con il ceppo inibitore wild-type mescolato con il ceppo bersaglio e due repliche biologiche con il ceppo mutante del sistema di secrezione di tipo VI mescolato con il ceppo bersaglio.
- Per garantire che le cellule concorrenti siano sufficientemente dense per l'uccisione dipendente dal contatto nella coincubazione sul cuscinetto di agar, concentrare ogni coltura mista 3 volte centrifugando la coltura mista in un tubo centrifugo standard da 1,5 mL per 1 minuto a 21.130 x g,scartando il surnatante e riutilizzando ogni pellet in un mezzo LBS da 20 μL. Ripetere l'operazione per ogni campione.
NOTA: Alcune cellule batteriche sono sensibili ai danni da centrifugazione ad alto rcf; in questi casi la coltura mista può essere centrifugata per 3 min a 4600 x g29. Inoltre, quando si quantifica la competizione dipendente dal contatto, è importante garantire una densità cellulare sufficiente sul vetrino per osservare l'uccisione. In questo articolo, i trattamenti "affollati", in cui si osserva l'uccisione, avevano circa 10 cellule / 20 μm2; vedere la sezione Discussione per ulteriori informazioni.
5. Configurazione delle diapositive
- Quando si utilizza un microscopio verticale, posizionare un tampone di agarose da ~ 5 mm2 su un vetrino standard da 1 mm. Individuare 2 μL di una coltura mista sul tampone di agarose e posizionare un coperchio #1.5 (25 mm2) sopra il punto. Vedere la Figura 1B per un esempio.
- Quando si utilizza un microscopio invertito, individuare 2 μL di una coltura mista sul fondo coverslip #1.5 di una capsula di Petri da 35 mm e posizionare un tampone di agarosio da ~ 5 mm2 sopra il punto di coincubazione. Posizionare una copertura circolare in vetro da 12 mm sopra il tampone di agarose. Vedere la Figura 1C per un esempio.
- Ripetere i passaggi 5.1 o 5.2, a seconda della configurazione del microscopio utilizzata, per le restanti tre colture miste, ottenendo quattro vetrini o piatti da immaginare.
- Lasciare che le diapositive rimangano sul banco per circa 5 minuti prima di procedere al passaggio 6. Ciò consente alle cellule di depositarsi sul cuscinetto di agar ed elimina il movimento durante il processo di imaging.
6. Microscopia a fluorescenza
- Inizia concentrandoti sulle cellule usando la luce bianca (contrasto di fase o DIC) per ridurre al minimo gli effetti del foto-sbiancamento. In base alle dimensioni medie di una singola cellula batterica, utilizzare un obiettivo di olio 60x o 100x.
- Regolare il tempo di esposizione e le impostazioni di acquisizione per ciascun canale in modo che le celle siano visibili nel canale appropriato con un rilevamento minimo dello sfondo.
NOTA: è opportuno utilizzare tempi di esposizione diversi per canali diversi, ma lo stesso tempo di esposizione deve essere utilizzato in tutte le repliche biologiche e trattamenti per un determinato canale. - Per ogni campione, selezionare almeno cinque campi di visualizzazione (FOV) e acquisire immagini in ciascun canale appropriato utilizzando le impostazioni di acquisizione del passaggio 6.2 (vedere esempi nella Figura 2). Salvate i punti XY da ciascun FOV in modo che lo stesso FOV possa essere ripreso durante il punto temporale finale. L'imaging dello stesso FOV in ogni punto temporale è necessario per determinare la proporzione di area occupata da cellule bersaglio o inibitorie durante le fasi di analisi.
NOTA: in questo esempio, la fluorescenza della GFP viene rilevata utilizzando un filtro con una lunghezza d'onda di eccitazione di 467 - 498 nm e un filtro di emissione di 513 - 556 nm ed è di falso colore verde. La fluorescenza di dsRed viene rilevata utilizzando un filtro con una lunghezza d'onda di eccitazione di 542 - 582 nm e un filtro di emissione di 603 - 678 nm ed è magenta di falso colore. - Dopo 2 ore, ripetere il passaggio 6.3 per ogni campione utilizzando i punti XY precedentemente salvati (Fig 2).
NOTA: Potrebbe essere necessario ottimizzare i tempi delle immagini successive per organismi con diversi tassi di crescita o meccanismi competitivi.
7. Analisi delle immagini nelle FIJI
- Scarica e installa il software di elaborazione delle immagini FIJI utilizzando le istruzioni disponibili qui: https://imagej.net/Fiji/Downloads
- Apri FIJI e importa i file di immagine per l'analisi.
NOTA: nella maggior parte dei casi . I file TIFF verranno utilizzati per l'analisi delle immagini, anche se alcuni software di acquisizione di immagini verranno esportati utilizzando tipi di file proprietari. FIJI è in grado di riconoscere la maggior parte dei tipi di file proprietari e le immagini possono essere importate e analizzate come segue. - Per ogni immagine acquisita nei passaggi 6.3 e 6.4, convertire l'immagine in scala di grigi, separare i canali e iniziare con la soglia (Ctrl + Maiusc + T) e creando una maschera binaria dell'immagine preelabota (Figura 3A,B).
NOTA: qui vengono utilizzate le impostazioni di soglia predefinite in FIJI. In alcuni casi, potrebbe essere necessario modificare tali impostazioni, nel qual caso le stesse impostazioni dovrebbero essere utilizzate per tutte le immagini dell'esperimento. - Impostare la scala sull'immagine (Analizza | Imposta scala) utilizzando i valori appropriati per l'impostazione al microscopio23.
- Impostare le misurazioni (Analizza | Imposta misure) e seleziona Area.
NOTA: è possibile aggiungere altre misurazioni se sono appropriate per l'esperimento. Per l'analisi di esempio mostrata qui è necessaria solo la misurazione dell'area dell'oggetto. - Analizza particelle (Analizza | Analizza particelle) utilizzando le impostazioni predefinite (Figura 3C). Se ci sono detriti nel campione, potrebbe essere necessario regolare la dimensione o la circolarità per filtrare le particelle non cellulari. Seleziona Mostra | Contorni in modo che l'output di questa analisi includa un contorno numerato di tutte le particelle analizzate (Figura 3D).
NOTA: il confronto tra il contorno nella Figura 3D e l'immagine iniziale è particolarmente importante nella fase di ottimizzazione per garantire che (1) tutte le celle vengano analizzate e (2) che eventuali detriti siano esclusi dall'analisi. - Esportare le misurazioni dal passaggio 7.4 (Figura 3E) in un software per fogli di calcolo per ulteriori analisi e grafici.
- Ripetere i passaggi 7.1 - 7.5 per tutti i canali e le immagini acquisite durante l'esperimento.
8. Calcolo della percentuale dell'area target iniziale nel tempo
- Per ogni campo visivo analizzato nella sezione 7, assicurarsi che il file esportato contenga una misurazione individuale dell'area per ogni particella analizzata. Partendo dal canale di fluorescenza della deformazione target, calcolare l'area della particella somma per ogni singolo campo visivo. Per due repliche biologiche con cinque FOV ciascuna, ciò dovrebbe comportare dieci aree di somma per trattamento in ogni punto temporale.
- Calcola la percentuale di area target iniziale nel tempo per ogni FOV usando la seguente equazione: (
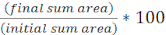 )
) - Ripetere questo calcolo per tutti i trattamenti e rappresentare graficamente la percentuale dell'area target iniziale (risultato dell'equazione dal passaggio 8.2) per ciascun trattamento (Figura 4A).
- Determinare se c'è un aumento netto della popolazione target (che indica la crescita), una diminuzione netta della popolazione target (che indica la morte) o nessun cambiamento (che indica nessuna crescita o morte) per ciascun trattamento.
NOTA: la percentuale dell'area target iniziale con valori superiori a 100 indica una crescita target netta e valori inferiori a 100 indicano la morte netta dell'obiettivo. La percentuale dei valori target iniziali che rimangono a 100 non indica alcun cambiamento netto nella popolazione target. Vedi la discussione per gli esperimenti di follow-up suggeriti.
9. Calcolo della percentuale di area dell'inibitore iniziale nel tempo
- Ripetere i passaggi da 8.1 a 8.3, questa volta utilizzando le misurazioni raccolte dal canale di fluorescenza del ceppo inibitore nella sezione 7 (Figura 4B).
- Determinare se c'è stato un aumento netto della popolazione di inibitori (crescita); una diminuzione netta della popolazione di inibitori (morte) o nessun cambiamento per ciascun trattamento. Valori superiori a 100 indicano una crescita netta dell'inibitore e valori inferiori a 100 indicano la morte netta dell'inibitore.
Risultati
Per visualizzare e quantificare le interazioni competitive tra batteri a livello di singola cellula, è stato sviluppato e ottimizzato un protocollo per V. fischeri modificando il nostro consolidato saggio basato su CFU1,2. Questo metodo utilizza plasmidi stabili con codifica GFP e dsRed per distinguere visivamente diversi ceppi di V. fischeri. Il risultato competitivo di queste interazioni può essere quantificato analizzando le immagini acquisite da questo test utilizzando il software open source FIJI. Ad esempio, il seguente esperimento è stato eseguito utilizzando isolati di V. fischeri. Un ceppo inibitore ospitava un plasmide che codifica GFP e un ceppo bersaglio ospitava un plasmide che codifica dsRed. Dato che il T6SS2 codificato dall'inibitore è un meccanismo di uccisione dipendente dal contatto, sono stati inclusi trattamenti in cui le cellule erano affollate (alto contatto cellula-cellula) o disperse (basso contatto cellula-cellula) su un vetrino per evidenziare l'impatto della configurazione sperimentale sui risultati finali di questo test. Nei dati del campione, i ceppi concorrenti sono stati miscelati in un rapporto 1:1 e incubati su un tampone di agarose per 2 ore, e sono state scattate sia immagini iniziali che finali (2 ore). Come controllo, un ceppo mutante T6SS2 è stato anche coincubato con il ceppo bersaglio sia in condizioni affollate che disperse. Le colture di ciascun ceppo sono state preparate e coincubate come descritto sopra e le diapositive sono state preparate come mostrato nella Figura 1.
La Figura 2 mostra immagini rappresentative al microscopio a fluorescenza di ciascun trattamento sperimentale con lo stesso campo visivo ripreso in un punto temporale iniziale e finale. Per ogni trattamento, un inibitore wild-type o un ceppo mutante T6SS che ospitava un plasmide che codifica GFP è stato miscelato in un rapporto 1:1 con il ceppo bersaglio che ospitava un plasmide che codifica dsRed. Durante un periodo di coincubazione di 2 ore con questa configurazione sperimentale, le cellule di V. fischeri in crescita possono passare attraverso 1-2 divisioni (Figura 2; frecce grigie). Nella Figura 2A,il contatto cellula-cellula è stato forzato tra il bersaglio e l'inibitore concentrando la coltura mista prima di individuare sul vetrino. Si osserva che più cellule bersaglio diventano arrotondate e/o scompaiono nel corso di 2 ore, coerentemente con l'eliminazione delle cellule bersaglio da parte dell'inibitore (Figura 2; frecce bianche). Vedere la sezione Discussione per ulteriori informazioni sull'interpretazione dell'arrotondamento o della lisi delle celle di destinazione. Nella Figura 2B, la stessa coincubazione è stata individuata su un vetrino, questa volta senza concentrare la coltura mista in modo che le cellule rimanessero disperse e ci fosse un contatto minimo tra i ceppi sul vetrino. Qui, non si osserva che nessuna cellule bersaglio scompaia o sia rotonda, suggerendo che il ceppo bersaglio non è stato inibito in questo trattamento. La Figura 2C e la Figura 2D mostrano gli stessi trattamenti affollati e dispersi sopra descritti, questa volta utilizzando un mutante T6SS come ceppo inibitore. Le cellule bersaglio non sono state osservate scomparire o arrotondarsi quando coincubate con un mutante T6SS in condizioni affollate o disperse, suggerendo ancora una volta che il bersaglio non era inibito in nessuno dei due trattamenti.
La Figura 3 mostra il flusso di lavoro di analisi FIJI utilizzato per quantificare la concorrenza in questo protocollo. È stata selezionata un'immagine rappresentativa dal canale di destinazione (Figura 3A) ed è stata creata una maschera binaria utilizzando le impostazioni di soglia predefinite in FIJI (Figura 3B). La scala dell'immagine è stata impostata in modo appropriato per questa configurazione di microscopia. Le particelle sono state analizzate utilizzando il parametro di dimensione = 0 - infinito, il parametro di circolarità = 0,00 - 1,00 e è stato selezionato Mostra contorni (Figura 3C). I risultati di questa analisi delle particelle sono mostrati sia come un contorno numerato di ciascuna particella (Figura 3D), sia come una tabella con colonne per il numero di particelle, il nome del file (etichetta) e l'area delle particelle inμm 2 (area) (Figura 3E).
Nella Figura 4, i dati ottenuti dalla Figura 3E sono rappresentati graficamente e analizzati. Nella Figura 4A,la percentuale di area target iniziale nel timepoint finale è presentata per ciascun trattamento secondo il passo 8.2. Se la percentuale dell'area target iniziale è maggiore di 100, ciò rappresenta un aumento netto dell'obiettivo (cioè la crescita) e si osserva in condizioni in cui la popolazione target non è significativamente inibita. Tuttavia, se la percentuale dell'area target iniziale è inferiore a 100, questo risultato indica una diminuzione netta del target (cioè la morte) e viene osservato in condizioni in cui la popolazione target è significativamente inibita. Quando il bersaglio è stato coincubato con un inibitore wild-type in condizioni affollate, i dati mostrano una diminuzione netta nell'area target. Al contrario, quando il bersaglio è stato coincubato con un inibitore wild-type in condizioni disperse o un mutante T6SS in condizioni affollate o disperse, i dati mostrano un aumento netto nell'area target. La percentuale di area target iniziale quando il bersaglio è stato coincubato con un inibitore wild-type in condizioni affollate era inferiore a 100 e significativamente inferiore a tutti gli altri trattamenti secondo un ANOVA unizionale seguito da un test di confronto multiplo di Tukey in tutti i trattamenti (p < 0,0001). Questi dati indicano che la morte delle cellule bersaglio dipende da un T6SS funzionale nell'inibitore e sottolineano l'importanza di una configurazione sperimentale che consenta un sufficiente contatto cellula-cellula, al fine di rilevare la morte cellulare da un meccanismo di uccisione dipendente dal contatto.
La Figura 4B presenta la percentuale di area iniziale dell'inibitore nel timepoint finale per ciascun trattamento. In questo esempio, è stata osservata una crescita netta del ceppo inibitore in tutti i trattamenti. Tuttavia, la percentuale di area iniziale dell'inibitore era significativamente più alta quando un inibitore wild-type era coincubato con il bersaglio in condizioni affollate rispetto a tutti gli altri trattamenti secondo un ANOVA unizionale seguito da un test di confronto multiplo di Tukey su tutti i trattamenti (p < 0,0001). Inizialmente, abbiamo considerato che l'aumento netto dell'area inibitore può essere guidato dall'aumento dello spazio disponibile in cui crescere man mano che le cellule bersaglio vengono eliminate. Tuttavia, questo stesso aumento della crescita degli inibitori non è stato osservato nei trattamenti dispersi, in cui le cellule inibitrici avevano spazio per crescere dall'inizio della coincubazione. In alternativa, questo risultato potrebbe suggerire che i nutrienti rilasciati dalla lisi delle cellule bersaglio consentono un maggiore aumento della popolazione di inibitori. Presi insieme, questi risultati suggeriscono che il ceppo inibitore elimina il bersaglio in modo dipendente da T6SS solo quando l'alto contatto cellula-cellula è forzato dall'affollamento delle cellule sul vetrino.
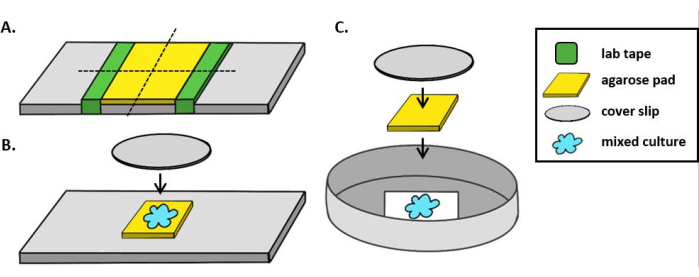
Figura 1: Preparazione del tampone di agarosio e configurazione del vetrino per i saggi di coincubazione. (A) Configurazione per la produzione di tamponi di agarosio al 2%. Cinque strati di nastro da laboratorio (verde) sono avvolti attorno a un coperchio a due punti a circa 20 mm di distanza. Successivamente, l'acarosio caldo al 2% in mPBS (giallo) viene pipettato tra i pezzi di nastro e immediatamente coperto con uno slittamento di coperturadi 25 mm 2 e lasciato solidificare per almeno 1 ora a temperatura ambiente. Utilizzare una lama di rasoio per tagliare il tampone di agarose in ~ 5 mm2 pezzi e utilizzare una pinzetta per trasferire il pad in una nuova diapositiva per l'imaging. (B) Quando si visualizza l'imaging su un microscopio verticale, posizionare il tampone di agarose da 5 mm2 direttamente sul vetrino, seguire la coltura mista (blu) e uno slittamento circolare di 12 mm #1.5. (C)Quando si visualizza l'imaging su un microscopio invertito, individuare la coltura mista direttamente sul fondo di copertura di vetro n. 1,5 di una capsula di Petri da 35 mm e posizionare un tampone di agarose sopra la coltura seguito da un secondo coperchio circolare di 12 mm per appiattire il tampone di agarose. Fare clic qui per visualizzare una versione più grande di questa figura.
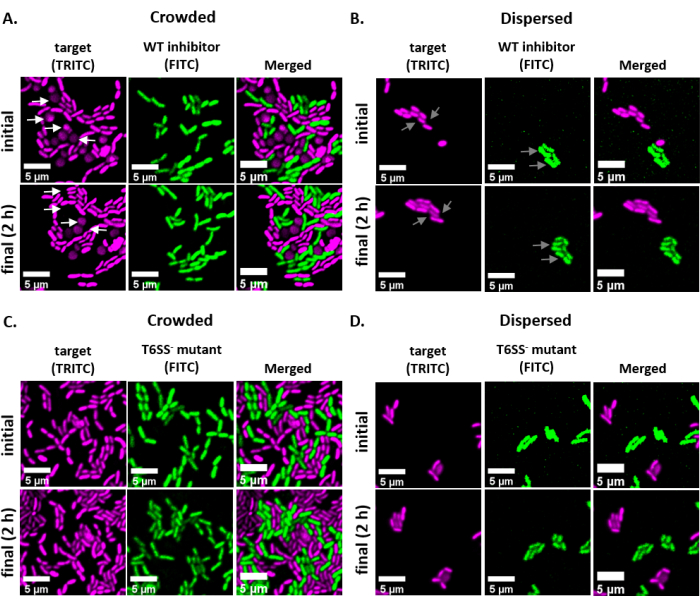
Figura 2: Immagini time lapse di punti di coincubazione in condizioni affollate o disperse. (A) Immagini rappresentative nei punti temporali iniziali e finali in cui una coltura mista di inibitore bersaglio e wild-type è stata concentrata 3 volte prima di individuare sul vetrino per forzare il contatto cellula-cellula tra ceppi. Le frecce bianche nel canale TRITC indicano esempi di cellule bersaglio che arrotondano o lisi nel corso dell'esperimento. (B) Immagini rappresentative in cui è stata individuata una coltura mista di inibitore bersaglio e wild-type senza concentrazione in modo che le cellule siano disperse e vi sia un contatto minimo cellula-cellula tra i ceppi. Le frecce grigie nei canali FITC e TRITC indicano esempi di divisione cellulare nel corso dell'esperimento. (C) Immagini rappresentative in cui la coltura mista di bersaglio e T6SS- mutante è stata concentrata 3 volte prima di essere individuata sul vetrino per forzare il contatto cellula-cellula tra ceppi. (D) Immagini rappresentative in cui è stata individuata una coltura mista di bersaglio e T6SS- mutante senza concentrazione in modo che le cellule si disperdano e vi sia un contatto minimo cellula-cellula tra i ceppi. Barre di scala = 5 μm e sono coerenti in tutte le immagini; Il canale TRITC è magenta di falso colore, il canale FITC è di falso colore verde. La deconvoluzione è stata eseguita su tutte le immagini; lo sfondo è stato sottratto e la luminosità/contrasto è stata regolata uniformemente su tutte le immagini. Fare clic qui per visualizzare una versione più grande di questa figura.
Figura 3: Flusso di lavoro di analisi FIJI. (A) Immagine rappresentativa per l'analisi. Questo flusso di lavoro viene ripetuto per entrambi i canali in tutti i campi di visualizzazione e gli esempi. Barre di scala = 5 μm e sono coerenti in tutte le immagini; Il canale TRITC è magenta di falso colore, il canale FITC è di falso colore verde. (B) Maschera binaria creata sogliendo l'immagine utilizzando le impostazioni predefinite in FIJI. (C) Esempio di impostazioni per l'analisi delle particelle utilizzate in questo manoscritto. Gamma di dimensioni = 0 - infinito μm2; circolarità = 0,00 - 1,00; show = contorni. (D) Profilo delle particelle creato come risultato dell'analisi delle particelle in (C). Il contorno delle particelle in (D) deve essere confrontato con l'immagine originale (A) per garantire che tutte le cellule siano state catturate nell'analisi delle particelle. (E) Tabella dei risultati creata come risultato dell'analisi delle particelle in (C). Il numero dell'oggetto (colonna 1) corrisponde alle singole particelle (una o più celle) delineate ed etichettate in rosso nel pannello (D). Label = nome file dell'immagine analizzata; Area = area totale delle particelle in μm2. Fare clic qui per visualizzare una versione più grande di questa figura.
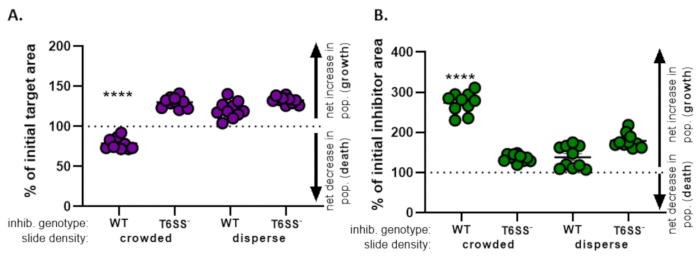
Figura 4: Dati di esempio per valutare se il ceppo bersaglio è inibito. La percentuale di area iniziale nei punti temporali finali per il ceppo bersaglio (A) e il ceppo inibitore (B), a diverse densità cellulari iniziali. La densità del vetrino indica una densità di cellule iniziali che è affollata (alto contatto cellulare tra ceppi) o più dispersa (basso contatto cellulare tra ceppi) come descritto nella Figura 2. Il genotipo inibitore indica che un ceppo wild-type o mutante T6SS (T6SS-) è stato coincubato con il ceppo bersaglio. Gli asterischi indicano una differenza significativa nella variazione percentuale rispetto a tutti i trattamenti (ANOVA unidirezionale seguito da un test di confronto multiplo di Tukey che confronta tutti i trattamenti; (p < 0,0001). La linea tratteggiata non indica alcun cambiamento netto nell'area di deformazione tra il punto temporale iniziale e quello finale; una variazione % > 100 indica un aumento netto (cioè una crescita) e una variazione % < 100 indica una diminuzione netta (cioè la morte cellulare). Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Il protocollo sopra descritto fornisce un potente strumento per quantificare e caratterizzare la competizione interbatterica a livello di singola cellula. Questo test, che è stato sviluppato modificando il nostro test di competizione basato su CFU su piastre di agar1,2 , ha permesso lavisualizzazionedella competizione a cella singola tra isolati di V. fischeri e suggerimenti sono forniti per ottimizzare il metodo per una vasta gamma di sistemi e configurazioni di microscopia. Sebbene il metodo qui descritto sia stato ottimizzato per il simbionte per organi leggeri V. fischeri,può essere facilmente modificato per ospitare molti microbi diversi e coltivabili. È importante notare che i meccanismi competitivi possono essere regolati da un numero qualsiasi di variabili ambientali, tra cui temperatura, salinità e viscosità30,31,32,33,34. Lavori precedenti hanno confermato che V. fischeri compete utilizzando un sistema di secrezione di tipo VI dipendente dal contatto che è attivo sulle superfici30, rendendo le condizioni descritte in questo test adatte allo studio della competizione tra i ceppi di esempio. È anche importante considerare la densità iniziale delle cellule sul vetrino quando si quantifica la competizione batterica. Dato che il contatto tra cellule bersaglio e cellule inibitrici è spesso necessario per l'uccisione, la coltura mista dovrebbe essere concentrata in modo tale che il contatto cellula-cellula sia massimizzato e le cellule rimangano su un unico piano sul vetrino. Le colture cellulari dovrebbero essere coltivate a una densità ottica simile (fase mid-log) e quindi concentrate per forzare il contatto piuttosto che semplicemente colture in crescita a una densità ottica più elevata a causa dei cambiamenti fisiologici delle cellule in diverse fasi di crescita. In altri sistemi, potrebbe essere necessario modificare le condizioni di coltura e la configurazione sperimentale per garantire che il meccanismo competitivo sia attivo e possa essere rilevato nella condizione di coincubazione.
I cuscinetti di agarose utilizzati in questo test offrono diversi vantaggi: forniscono stabilizzazione in modo che le cellule non si muovano liberamente e impediscono alla coltura di asciugarsi nel corso dell'esperimento. Inoltre, se per l'esperimento sono necessari induttori chimici, come l'isopropil-β-D-tiogalacoside (IPTG), possono essere facilmente aggiunti alla soluzione di agarose. Tuttavia, è importante notare che la preparazione dell'agarose dovrà probabilmente essere regolata per sistemi diversi. Nell'esempio sopra descritto, il tampone di agarose è stato preparato sciogliendo il 2% di agarose (w /v) in 20 psu mPBS, che è la salinità standard utilizzata nel terreno di crescita V. fischeri. Inoltre, in alcuni casi potrebbe essere necessario aggiungere una fonte di carbonio al cuscinetto di agarose affinché le cellule crescano e competano su esperimenti più lunghi. In tal caso, l'mPBS nei cuscinetti di agarose può essere sostituito con qualsiasi mezzo di crescita, sebbene i nutrienti nel mezzo di crescita possano venire con il compromesso di ulteriore fluorescenza di fondo.
Senza un software di analisi delle immagini proprietario, può essere molto difficile ottenere conteggi di singole cellule quando il contatto cellula-cellula è elevato, che come mostriamo qui è necessario per osservare l'uccisione dipendente dal contatto. Questo test è stato progettato per fornire un metodo alternativo per la quantificazione che non si basa sulla conta delle singole cellule. Invece, l'area cellulare totale per ciascun canale di fluorescenza viene utilizzata per quantificare l'entità dell'uccisione tra ceppi coincubati. Poiché questo metodo si basa sull'area anziché sul conteggio delle singole celle, le impostazioni di soglia predefinite sono in genere sufficienti per delineare l'area totale della cella. L'accuratezza della soglia può essere verificata dividendo l'area totale dell'oggetto per un campo visivo rappresentativo per la dimensione media delle cellule per l'organismo modello e confrontando questo numero di cellule stimato con un conteggio manuale delle cellule per la stessa immagine.
Nelle coincubazioni tra un inibitore e un ceppo bersaglio (non killer), è prevista una crescita netta dell'inibitore. Come si vede nella Figura 4,la crescita degli inibitori può essere significativamente più alta nei trattamenti in cui si osserva l'uccisione, rispetto ai trattamenti in cui non si osserva l'uccisione, forse perché i nutrienti rilasciati dalla lisi delle cellule bersaglio consentono al ceppo inibitore di crescere più rapidamente. Nell'esempio mostrato qui, si osserva la morte netta del bersaglio perché la competizione mediata da T6SS provoca la lisi cellulare bersaglio in cui il bersaglio viene eliminato fisicamente. Tuttavia, è importante notare che non tutti i meccanismi competitivi comportano l'eliminazione fisica delle cellule bersaglio. Se un bersaglio è incapace da una tossina che causa l'inibizione della crescita, il protocollo qui delineato può comportare che la popolazione target visibile rimanga stabile nel tempo poiché le cellule bersaglio non crescono più ma non si lyse. In tal caso, sarebbe opportuno confrontare i risultati di questo saggio con test di follow-up per la vitalità delle cellule bersaglio, come la placcatura per unità formanti colonie (CFU) o eseguendo saggi di morte viva mediante colorazione con ioduro di propidio o verde SYTOX35,36.
Rispetto ai saggi di coincubazione che si basano sui conteggi CFU, questo test consente di osservare e quantificare la struttura spaziale della competizione tra ceppi e di tracciare i cambiamenti nella morfologia delle cellule bersaglio nel tempo. Ad esempio, le cellule inibitrici che uccidono usando un T6SS sono note per codificare proteine del dominio LysM che degradano la parete cellulare bersaglio, con conseguente arrotondamento iniziale delle cellule e quindilisi 13, che abbiamo osservato nell'esempio mostrato in Figura 2A. Inoltre, questo protocollo può essere utilizzato per tracciare la concorrenza ad alta risoluzione su scale temporali molto brevi. Nell'esempio mostrato qui, si osserva una significativa diminuzione dell'area target dopo solo due ore quando le cellule sono affollate e il contatto cellula-cellula è forzato tra i ceppi (Figura 4). L'analisi delle immagini qui descritta potrebbe anche essere eseguita utilizzando la microscopia confocale, che consentirebbe di studiare la competizione batterica in vivo o in biofilm complessi, senza interrompere la distribuzione spaziale dei ceppi coincubati.
In sintesi, il test qui descritto mira a fornire un approccio accessibile e facilmente modificabile per visualizzare e quantificare la competizione batterica a livello di singola cellula utilizzando la microscopia a fluorescenza. Questo metodo può essere applicato a diversi isolati batterici e può essere utilizzato per visualizzare la competizione batterica anche in ambienti complessi come all'interno di un ospite o di una matrice di biofilm.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
A.N.S è stato supportato dalla sovvenzione NIGMS R35 GM137886 e S.N.S è stato supportato dal National Defense Science and Engineering Graduate Fellowship Program.
Materiali
| Name | Company | Catalog Number | Comments |
| 1.5 mL Microcentrifuge tube | Fisher | 05-408-129 | |
| 10 uL single channel pipette | |||
| 1000 uL single channel pipette | |||
| 20 uL single channel pipette | |||
| 200 uL single channel pipette | |||
| Agarose | Fisher | BP165-25 | Low melting agarose |
| Calculator | |||
| Cellvis 35 mm Dish | Fisher | NC0409658 | #1.5 cover glass bottom |
| Chloramphenicol | Sigma | C0378 | stock (20 mg/mL in Ethanol); final concentration in media (2 μg /mL LBS) |
| DAPI Nucleic Acid Stain | Fisher | EN62248 | optional (if not using stable plasmids) |
| FIJI image analysis sofware | ImageJ | https://imagej.net/Fiji/Downloads | open-source software |
| Fisherbrand Cover Glasses: Circles | Fisher | 12-545-81P | #1.5 cover glass; 12 mm diameter |
| Kanamycin Sulfate | Fisher | BP906-5 | stock (100 mg/mL in water, filter sterilize); final concentration in media (1 μg/mL LBS) |
| Lens Cleaning Tissue Paper | Fisher | S24530 | |
| Parafilm | Fisher | 13-374-12 | |
| Petri Plates | Fisher | FB0875713 | sterile with lid |
| Razor Blades | Fisher | S65921 | |
| Semi-micro Cuvettes | VWR | 97000-586 | |
| Spectrophotometer | |||
| SYBR Green Nucleic Acid Stain | Fisher | S7563 | optional (if not using stable plasmids) |
| Thermo Scientific Gold Seal Plain Microscope Slides | Fisher | 12-518-100B | |
| Thermo Scientific Richard-Allan Scientific Cover Glass | Fisher | 22-050-235 | #1.5 cover glass, 25 mm2 |
| Type F Immersion Oil | Fisher | NC0297589 | |
| Upright or inverted fluorescence microscope with camera and imaging software | Images in this article were acquired on a Nikon TI-2 inverted fluorescent microscope outfitted with an ORCA-Fusion Digital CMOS camera using NIS-Elements software. | ||
| Vortex | |||
| Water bath | Used to keep agarose warm prior to pipetting | ||
| LBS media | |||
| 1M Tris Buffer (pH ~7.5) | 50 mL 1 M stock buffer (62 mL HCl, 938 mL DI water, 121 g Trizma Base) | ||
| Agar Technical | Fisher | DF0812-17-9 | 15 g (Add only for plates) |
| DI water | 950 mL | ||
| Sodium Chloride | Fisher | S640-3 | 20 g |
| Tryptone | Fisher | BP97265 | 10 g |
| Yeast Extract | Fisher | BP9727-2 | 5 g |
| mPBS (marine PBS) | Phosphate buffered saline with marine salts added; used for making agarose pad | ||
| 10X PBS | Fisher | ICN1960454 | |
| Instant Ocean Sea Salt | Instant Ocean | SS1-160P | Adjust concentration to appropriate salinity; 20 psu used here |
| Sterile Vacuum Filter Units | Fisher | SCGVU01RE | Used to filter-sterilize mPBS |
| Vacuum pump | Used to filter-sterilize mPBS |
Riferimenti
- Speare, L., et al. Bacterial symbionts use a type VI secretion system to eliminate competitors in their natural host. Proceedings of the National Academy of Sciences. 115 (36), 8528-8537 (2018).
- Speare, L., Septer, A. N. Coincubation assay for quantifying competitive interactions between Vibrio fischeri isolates. Journal of Visualized Experiments. (149), e59759 (2019).
- Frost, I., et al. Cooperation, competition and antibiotic resistance in bacterial colonies. The ISME journal. 12 (6), 1582-1593 (2018).
- Stubbendieck, R. M., Vargas-Bautista, C., Straight, P. D. Bacterial communities: interactions to scale. Frontiers in Microbiology. 7, 1234 (2016).
- Souza, D. P., et al. Bacterial killing via a type IV secretion system. Nature Communications. 6 (1), 1-9 (2015).
- Anderson, M. C., Vonaesch, P., Saffarian, A., Marteyn, B. S., Sansonetti, P. J. Shigella sonnei encodes a functional T6SS used for interbacterial competition and niche occupancy. Cell Host and Microbe. 21 (6), 769-776 (2017).
- Basler, M., Ho, B., Mekalanos, J. Tit-for-tat: Type VI secretion system counterattack during bacterial cell-cell interactions. Cell. 152 (4), 884-894 (2013).
- Guillemette, R., Ushijima, B., Jalan, M., Häse, C. C., Azam, F. Insight into the resilience and susceptibility of marine bacteria to T6SS attack by Vibrio cholerae and Vibrio coralliilyticus. PloS One. 15 (1), 0227864 (2020).
- Hachani, A., Lossi, N. S., Filloux, A. A visual assay to monitor T6SS-mediated bacterial competition. Journal of Visualized Experiments. (73), e50103 (2013).
- Hibbing, M. E., Fuqua, C., Parsek, M. R., Peterson, S. B. Bacterial competition: surviving and thriving in the microbial jungle. Nature Reviews Microbiology. 8 (1), 15-25 (2010).
- Ruhe, Z. C., Low, D. A., Hayes, C. S. Bacterial contact-dependent growth inhibition. Trends in Microbiology. 21 (5), 230-237 (2013).
- Wood, D. W., Pierson, L. S. The phzI gene of Pseudomonas aureofaciens 30-84 is responsible for the production of a diffusible signal required for phenazine antibiotic production. Gene. 168 (1), 49-53 (1996).
- Smith, W. P., et al. The evolution of the type VI secretion system as a disintegration weapon. PLoS Biology. 18 (5), 3000720 (2020).
- Chen, L., Zou, Y., She, P., Wu, Y. Composition, function, and regulation of T6SS in Pseudomonas aeruginosa. Microbiological Research. 172, 19-25 (2015).
- Sana, T. G., Lugo, K. A., Monack, D. M. T6SS: The bacterial "fight club" in the host gut. PLoS Pathogens. 13 (6), 1006325 (2017).
- Basler, M. Type VI secretion system: secretion by a contractile nanomachine. Philosophical Transactions of the Royal Society B: Biological Sciences. 370 (1679), 20150021 (2015).
- Joshi, A., et al. Rules of engagement: the type VI secretion system in Vibrio cholerae. Trends in Microbiology. 25 (4), 267-279 (2017).
- Nadell, C. D., Drescher, K., Foster, K. R. Spatial structure, cooperation and competition in biofilms. Nature Reviews Microbiology. 14 (9), 589-600 (2016).
- Stubbendieck, R. M., Straight, P. D. Multifaceted interfaces of bacterial competition. Journal of Bacteriology. 198 (16), 2145-2155 (2016).
- Septer, A. N. The Vibrio-squid symbiosis as a model for studying interbacterial competition. Msystems. 4 (3), (2019).
- Tischler, A. H., Hodge-Hanson, K. M., Visick, K. L. Vibrio fischeri-squid symbiosis. eLS. , 1-9 (2019).
- Mandel, M. J., Dunn, A. K. Impact and influence of the natural Vibrio-squid symbiosis in understanding bacterial-animal interactions. Frontiers in Microbiology. 7, 1982 (2016).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Boettcher, K., Ruby, E. Depressed light emission by symbiotic Vibrio fischeri of the sepiolid squid Euprymna Scolopes. Journal of Bacteriology. 172 (7), 3701-3706 (1990).
- Doino, J. A., McFall-Ngai, M. J. A transient exposure to symbiosis-competent bacteria induces light organ morphogenesis in the host squid. The Biological Bulletin. 189 (3), 347-355 (1995).
- Dunn, A. K., Millikan, D. S., Adin, D. M., Bose, J. L., Stabb, E. V. New rfp-and pES213-derived tools for analyzing symbiotic Vibrio fischeri reveal patterns of infection and lux expression in situ. Applied and Environmental Microbiology. 72 (1), 802-810 (2006).
- Lambertsen, L., Sternberg, C., Molin, S. Mini-Tn7 transposons for site-specific tagging of bacteria with fluorescent proteins. Environmental Microbiology. 6 (7), 726-732 (2004).
- Koch, B., Jensen, L. E., Nybroe, O. A panel of Tn7-based vectors for insertion of the gfp marker gene or for delivery of cloned DNA into Gram-negative bacteria at a neutral chromosomal site. Journal of Microbiological Methods. 45 (3), 187-195 (2001).
- Peterson, B. W., Sharma, P. K., Van Der Mei, H. C., Busscher, H. J. Bacterial cell surface damage due to centrifugal compaction. Applied and Environmental Microbiology. 78 (1), 120-125 (2012).
- Speare, L., Smith, S., Salvato, F., Kleiner, M., Septer, A. N. Environmental viscosity modulates interbacterial killing during habitat transition. MBio. 11 (1), (2020).
- Salomon, D., Gonzalez, H., Updegraff, B. L., Orth, K. Vibrio parahaemolyticus type VI secretion system 1 is activated in marine conditions to target bacteria, and is differentially regulated from system 2. PloS One. 8 (4), 61086 (2013).
- Sana, T. G., et al. Salmonella Typhimurium utilizes a T6SS-mediated antibacterial weapon to establish in the host gut. Proceedings of the National Academy of Sciences. 113 (34), 5044-5051 (2016).
- Bachmann, V., et al. Bile salts modulate the mucin-activated type VI secretion system of pandemic Vibrio cholerae. PLoS Neglected Tropical Diseases. 9 (8), 0004031 (2015).
- Ishikawa, T., et al. Pathoadaptive conditional regulation of the type VI secretion system in Vibrio cholerae O1 strains. Infection and Immunity. 80 (2), 575-584 (2012).
- Johnson, M. B., Criss, A. K. Fluorescence microscopy methods for determining the viability of bacteria in association with mammalian cells. Journal of Visualized Experiments. (79), e50729 (2013).
- Stiefel, P., Schmidt-Emrich, S., Maniura-Weber, K., Ren, Q. Critical aspects of using bacterial cell viability assays with the fluorophores SYTO9 and propidium iodide. BMC Microbiology. 15 (1), 36 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon