Method Article
Rilevamento di rotture a doppio filamento di DNA in ovociti di topo
In questo articolo
Riepilogo
Mantenere l'integrità del genoma dell'ovocita è necessario per garantire la fedeltà genetica nell'embrione risultante. Qui, presentiamo un protocollo accurato per rilevare le rotture del doppio filamento del DNA nelle cellule germinali femminili dei mammiferi.
Abstract
Gli ovociti sono tra le cellule più grandi e longeve del corpo femminile. Si formano nelle ovaie durante lo sviluppo embrionale e rimangono arrestate alla profase della meiosi I. Lo stato di quiescenza può durare per anni fino a quando gli ovociti ricevono uno stimolo per crescere e ottenere la competenza per riprendere la meiosi. Questo stato di arresto prolungato le rende estremamente suscettibili all'accumulo di insulti dannosi per il DNA, che colpiscono l'integrità genetica dei gameti femminili e, quindi, l'integrità genetica del futuro embrione.
Di conseguenza, lo sviluppo di un metodo accurato per rilevare il danno al DNA, che è il primo passo per stabilire meccanismi di risposta al danno al DNA, è di vitale importanza. Questo articolo descrive un protocollo comune per testare la presenza e l'andamento del danno al DNA in ovociti arrestati dalla profase durante un periodo di 20 ore. In particolare, sezioniamo le ovaie di topo, recuperiamo i complessi cumulo-ovocita (COC), rimuoviamo le cellule del cumulo dai COC e coltiviamo gli ovociti in un terreno Μ2 contenente 3-isobutil-1-metilxantina per mantenere lo stato di arresto. Successivamente, gli ovociti vengono trattati con il farmaco antineoplastico citotossico, l'etoposide, per generare rotture a doppio filamento (DSB).
Utilizzando l'immunofluorescenza e la microscopia confocale, rileviamo e quantifichiamo i livelli della proteina core γH2AX, che è la forma fosforilata dell'istone H2AX. H2AX diventa fosforilato nei siti dei DSB dopo un danno al DNA. L'incapacità di ripristinare l'integrità del DNA in seguito a danni al DNA negli ovociti può portare a infertilità, difetti alla nascita e aumento dei tassi di aborti spontanei. Pertanto, la comprensione dei meccanismi di risposta al danno del DNA e, allo stesso tempo, la creazione di un metodo intatto per lo studio di questi meccanismi sono essenziali per la ricerca in biologia riproduttiva.
Introduzione
Il processo di meiosi nelle cellule germinali femminili dei mammiferi inizia nelle ovaie prima della nascita. Il numero totale di ovociti si stabilisce nelle ovaie principalmente durante l'embriogenesi. Gli ovociti entrano in meiosi e rimangono arrestati alla profase I1. Dopo l'inizio della pubertà e la produzione e l'azione endocrina dell'ormone follicolo-stimolante (FSH) e dell'ormone luteinizzante (LH), gli ovociti possono ricominciare e completare la meiosi2. Nell'uomo, l'arresto della profase può durare fino a 50 anni3. Le divisioni cellulari che seguono l'ingresso nella meiosi I sono asimmetriche, con conseguente produzione di un piccolo corpo polare e di un ovocita che mantiene le sue dimensioni. Pertanto, la maggior parte dei componenti citoplasmatici sono immagazzinati nell'ooplasma durante l'embriogenesi precoce4. Successivamente, gli ovociti entrano nella meiosi II, senza riformare il nucleo o decondensare i cromosomi, e rimangono arrestati alla metafase II fino alla fecondazione5.
Una caratteristica unica che distingue gli ovociti dalle cellule somatiche è lo stato di arresto nella profase I, quando l'ovocita possiede un nucleo intatto (arresto della vescicola germinale [GV]), indicato come stadioGV 6. In base all'organizzazione della cromatina, gli ovociti allo stadio GV sono classificati in due categorie: nucleolo non circondato (NSN) e nucleolo circondato (SN)7,8. Negli ovociti in stadio NSN GV, la cromatina si diffonde in tutta la regione nucleare e la trascrizione è attiva, mentre negli ovociti SN, la cromatina forma un anello compatto che circonda il nucleolo e la trascrizione è silente9. Entrambi i tipi di ovociti allo stadio GV mostrano competenza meiotica; entrano in meiosi alla stessa velocità, ma gli ovociti NSN presentano una bassa capacità di sviluppo e non possono svilupparsi oltre lo stadio embrionale10 a due cellule.
Lo stato prolungato di arresto della profase I aumenta l'incidenza dell'accumulo di danni al DNA11. Pertanto, i meccanismi di risposta al danno al DNA negli ovociti sono essenziali per consentire la produzione di gameti con integrità genetica e per garantire che l'embrione risultante abbia un contenuto cromosomico fisiologico.
Un aspetto centrale della risposta al danno del DNA è la riparazione del DNA. Le principali vie per la riparazione del DSB nelle cellule eucariotiche includono l'unione delle estremità non omologhe (NHEJ), la ricombinazione omologa (HR) e l'NHEJ alternativo12,13,14,15. NHEJ è un meccanismo più veloce ma più soggetto a errori, mentre HR richiede più tempo per essere completato, ma ha un'alta fedeltà16.
Non ci sono abbastanza conoscenze sui meccanismi che gli ovociti utilizzano per riparare i danni al DNA. Gli studi hanno dimostrato che il danno al DNA indotto negli ovociti di mammiferi adulti dall'uso di agenti genotossici, come l'etoposide, la doxorubicina o le radiazioni UVB o ionizzanti, non influisce sui tempi e sui tassi di uscita dall'arresto della profase I17. Gli ovociti possono andare incontro a rottura del GV (GVBD) anche in presenza di livelli elevati di danno. Questo danno può essere determinato dall'osservazione di γH2AX. Questa forma fosforilata di H2AX (γΗ2ΑΧ) è un marcatore DSB, che si trova nel sito delle rotture e funziona come un'impalcatura per aiutare i fattori di riparazione e le proteine ad accumularsi alle estremità rotte18.
L'assenza di arresto del ciclo cellulare a seguito di un danno al DNA è dovuta a un insufficiente checkpoint del danno al DNA che consente agli ovociti con DNA non riparato di rientrare in meiosi. A seguito di alti livelli di danno al DNA, un checkpoint può mantenere l'arresto della profase attraverso l'attivazione di una via dipendente da ATM/Chk1. La limitata risposta dei punti di controllo ai DSB è dovuta all'attivazione limitata di ATM17,19. Nella fase M della meiosi I, la ricerca ha dimostrato che il danno al DNA può attivare un checkpoint della meiosi I indotto dal checkpoint dell'assemblaggio del fuso (SAC), che impedisce l'attivazione del complesso/ciclosoma promotore dell'anafase dell'ubiquitina ligasi E3 (APC/C) e, quindi, l'uscita dalla fase M. Inoltre, l'ablazione delle proteine SAC supera lo stato di arresto della fase M, sottolineando così l'importanza della SAC nello stabilire il puntodi controllo 20 della meiosi I.
Come dimostrano chiaramente ricerche precedenti, i DSB non possono indurre un robusto checkpoint profasico negli ovociti di topo. Se tale danno non viene riparato, potrebbe portare a embrioni portatori di anomalie cromosomiche. Pertanto, è importante studiare la risposta al danno del DNA nelle diverse fasi della gametogenesi femminile per comprendere meglio i percorsi unici che gli ovociti utilizzano per far fronte a potenziali insulti genetici.
Protocollo
Tutti gli esperimenti sui topi sono stati approvati dalle autorità locali (Regione di Ioannina, Grecia) e condotti in conformità con le Direttive del Consiglio delle Comunità Europee 2010/63/UE. Gli esperimenti sono stati condotti nel rispetto dei principi delle 3R. Tutti i topi CD-1 utilizzati per gli esperimenti sono stati tenuti nella struttura della casa degli animali dell'Università di Ioannina, in Grecia, in una stanza a temperatura controllata (22 °C) e umidità (60%) e sono stati nutriti ad libitum. La casa per animali ha una licenza per gestire una struttura per l'allevamento (EL33-BIObr01), la fornitura (EL33-BIOsup01) e gli esperimenti (EL33BIO-exp01).
1. Preparazione dei reagenti
- Diluire la polvere di 3-isobutile-1-metilxantina (IBMX) (vedere la tabella dei materiali) in dimetilsolfossido (DMSO) (vedere la tabella dei materiali) fino a una concentrazione finale di 200 mM. Μake 10 μL aliquote e conservare a −20 °C. Utilizzare la soluzione entro 1 mese.
NOTA: La polvere IBMX viene mantenuta a −20 °C. - Preparare tutti i tamponi di immunofluorescenza e conservarli a 4 °C.
- Preparare soluzione salina sterile tamponata con fosfato (PBS) diluendo una compressa di PBS (vedere la tabella dei materiali) in 200 mL di ddH2Ο.
- Preparare il tampone PHEM aggiungendo 80 mL di ddH 2 Ο, 0,59575 g di HEPES, 1,81422 g di PIPES, 0,38035 g di EGTA e 0,04066 g di MgCl2(vedere la Tabella dei Materiali) agitando con un agitatore magnetico (vedere la Tabella dei Materiali) e contemporaneamente aggiungere NaOH (vedere la Tabella dei Materiali) fino a quando il pH raggiunge 6,9 (controllare utilizzando un misuratore di pH/ORP [vedere la Tabella dei Materiali]). Quindi, aggiungere ddH2Ο a un volume finale di 100 mL.
- Preparare il tampone paraformaldeide-Triton-X-100 (PFA-Tx-100) diluendo la polvere di PFA (vedere la tabella dei materiali) nel tampone PHEM agitando con un agitatore magnetico sotto riscaldamento a una concentrazione finale del 4% di PFA. Quindi, filtrare il tampone utilizzando una siringa e un filtro da 0,2 μm (vedere la Tabella dei materiali) e aggiungere lo 0,5% di Tx-100 (vedere la Tabella dei materiali). Preparare circa 10 mL di PFA-Tx-100 (0,4 g di PFA, 50 μL di Tx-100), sufficienti per un esperimento. Conservare a 4 °C per un massimo di 1 settimana.
ATTENZIONE: Indossare guanti per maneggiare PFA ed evitare il contatto con la pelle e gli occhi. - Preparare il tampone di lavaggio aggiungendo albumina sierica bovina (concentrazione finale: 0,5% p/v BSA) (vedere la tabella dei materiali) nel PBS e agitare meccanicamente. Aggiungere il 10% p/v di tampone NaN3 (sodio azide) a una diluizione 1:1.000 per ridurre al minimo il rischio di contaminazione fungina e batterica. Preparare un tampone NaN 3 al 10% p/v aggiungendo 1 g di polvere di NaN3 (vedere la tabella dei materiali) a 10 mL di ddH2O; conservare il tampone NaN3 a temperatura ambiente.
- Preparare il tampone bloccante aggiungendo BSA (concentrazione finale: 3% p/v) nel PBS e agitando meccanicamente. Aggiungere il 10% di tampone NaN3 a una diluizione 1:1.000.
2. Prelievo di ovociti GV da ovaie dissecate e induzione di DSB
NOTA: Tutti gli strumenti e le soluzioni devono essere sterili. La manipolazione degli ovociti viene effettuata utilizzando una pipetta a bocca sotto uno stereomicroscopio (vedere la Tabella dei materiali) e tutte le gocce sono ricoperte di olio minerale (vedere la Tabella dei materiali e la Figura 1E).
- Iniettare nei topi per via intraperitoneale 7 unità internazionali (UI) di gonadotropina sierica di cavalla gravida (PMSG) (vedere la tabella dei materiali) 46-48 ore prima di eliminare i topi mediante lussazione cervicale.
NOTA: Tutti i topi utilizzati devono avere un'età compresa tra 8 e 12 settimane. - Filtrare il terreno di coltura M2 (vedere la tabella dei materiali) con una siringa e un filtro da 0,2 μm e aggiungere IBMX 200 mM a una concentrazione finale di 200 μM in una provetta a fondo tondo da 14 mL (vedere la tabella dei materiali) per mantenere gli ovociti arrestati alla profase I. Quindi, preparare le gocce di terreno M2-IBMX in una piastra di coltura di tessuto plastico (vedere la Tabella dei Materiali) e posizionarle su un blocco caldo (vedere la Tabella dei Materiali) a 37 °C per almeno 30 minuti prima dell'isolamento degli ovociti. Conservare l'M2 a 4 °C.
- Sacrificare i topi mediante lussazione cervicale, sezionare le ovaie e metterli in una provetta a fondo tondo da 5 mL (vedere la tabella dei materiali) con M2-IBMX.
- Trasferire le ovaie in un coperchio di plastica contenente 1,5 mL di M2-IBMX, rimuovere eventuali segmenti di tessuto adiposo periovarico o delle tube di Falloppio e rilasciare i COC mediante perforazione meccanica delle ovaie con un ago da 27 G (vedere la tabella dei materiali e la figura 1A-C).
- Trasferire i COC in una piastra di coltura con gocce di M2-IBMX (circa 25-30 μL ciascuna) e rimuovere le cellule del cumulo mediante pipettaggio ripetuto utilizzando una pipetta Pasteur in vetro a foro stretto (vedere la Tabella dei materiali e la Figura 1D).
- Selezionare gli ovociti in stadio SN GV e trasferirli in una goccia (25 μL) di terreno M2-IBMX su un blocco caldo a 37 °C al riparo dalla luce (Figura 1F).
- Cerca gli ovociti SN in base alle loro dimensioni maggiori e ai nuclei posizionati centralmente in contrasto con gli ovociti NSN, in cui i nuclei sono posizionati perifericamente21. In ogni caso, osservare la configurazione del DNA al microscopio confocale prima di prendere la decisione finale sul tipo di ovocita GV (SN o NSN).
- Indurre DSB utilizzando etoposide (vedere la Tabella dei Materiali). Porre gli ovociti allo stadio GV in gocce (25 μL ciascuna) dell'agente genotossico per 1 ora sul blocco caldo a 37 °C in condizioni di oscurità.
NOTA: L'etoposide è un inibitore della topoisomerasi II che introduce DSB nel DNA22. Conservare l'etoposide in aliquote da 10 μL da 20 mg/mL a temperatura ambiente, al riparo dalla luce. Le concentrazioni che sono state testate sono 5 μg/mL, 20 μg/mL e 50 μg/mL. - Per mantenere gli ovociti allo stadio GV arrestati per un periodo prolungato, porre gli ovociti in gocce di terreno di coltura M16 (vedere la tabella dei materiali) integrata con 400 μM IBMX in un'incubatrice (vedere la tabella dei materiali) a 37 °C e 5% di CO2 . Conservare l'M16 a 4 °C, filtrare il terreno con una siringa e un filtro da 0,2 μm e incubarlo per almeno 1 ora prima dell'uso.
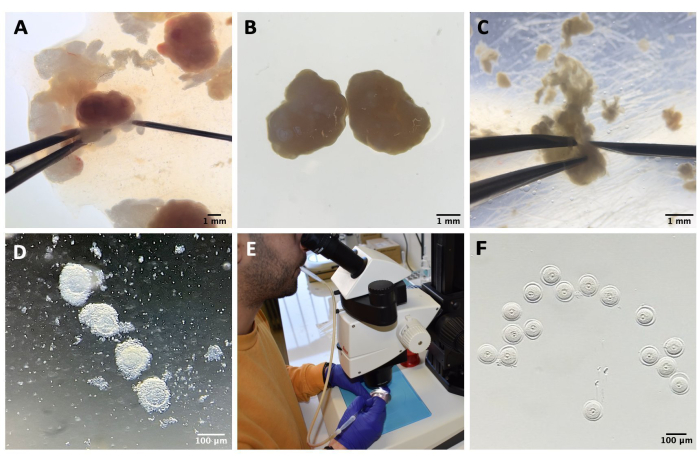
Figura 1: Processo di isolamento degli ovociti . (A) Rimozione del tessuto adiposo periovarico e dei segmenti residui delle tube di Falloppio dalle ovaie in mezzo M2 con IBMX. Fotografia ottenuta attraverso gli oculari dello stereomicroscopio. Barra della scala = 1 mm. (B) Ovaie isolate in terreno M2 con IBMX. Immagine ottenuta attraverso gli oculari dello stereomicroscopio. Barra della scala = 1 mm. (C) Perforazione meccanica delle ovaie utilizzando un ago da 27 G in terreno M2 con IBMX. Immagine ottenuta attraverso gli oculari dello stereomicroscopio. Barra della scala = 1 mm. (D) COC rilasciati dalle ovaie dopo la perforazione in terreno M2 con IBMX. Immagine ottenuta attraverso gli oculari dello stereomicroscopio. Barra graduata = 100 μm. (E) Prelievo di ovociti con pipetta a bocca. (F) Ovociti denudati, dopo la rimozione delle cellule del cumulo circostanti, in terreno M2 con IBMX. Immagine ottenuta attraverso gli oculari dello stereomicroscopio. Barra della scala = 100 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
3. Fissazione degli ovociti e immunofluorescenza
NOTA: La manipolazione degli ovociti viene condotta utilizzando una pipetta orale al microscopio stereoscopico e tutte le gocce sono ricoperte di olio minerale.
- Posizionare gli ovociti GV di controllo e trattati con etoposide in diverse piastre di coltura di tessuti plastici con tampone PFA-Tx-100 per 40 minuti a temperatura ambiente.
- Lavare gli ovociti in tre diverse gocce di tampone di lavaggio (50 μL ciascuna) a temperatura ambiente. Lasciare gli ovociti per 5 minuti in ogni goccia.
- Porre gli ovociti in gocce di tampone bloccante (25 μL ciascuna) per 1 ora su un blocco caldo a 37 °C.
- Preparare l'anticorpo primario che riconosce γH2AX (fosfo-Η2ΑΧ di coniglio) (Ser139) (vedere la tabella dei materiali) (soluzione madre: 1 mg/mL). Utilizzare una diluizione 1:200 nel tampone bloccante e porre gli ovociti in gocce di anticorpo primario (15 μL ciascuna) a 4 °C per tutta la notte.
NOTA: Phospho-Η2ΑΧ (γH2AX) è un marcatore comune per la rilevazione di DSB sia nelle cellule somatiche che negli ovociti GV18,23. - Il giorno seguente, lavare gli ovociti in tre diverse gocce di tampone di lavaggio (50 μL ciascuna) a temperatura ambiente. Lasciare gli ovociti per 5 minuti in ogni goccia.
- Preparare l'anticorpo secondario, Alexa Fluor 488-coniugato capra anti-coniglio (vedere la tabella dei materiali) (soluzione madre: 2 mg/mL). Utilizzare una diluizione 1:200 nel tampone bloccante e porre gli ovociti in gocce di anticorpo secondario (15 μL ciascuna) per 1 ora su un blocco caldo a 37 °C al riparo dalla luce.
- Trasferire gli ovociti in gocce di DRAQ7 (25 μL ciascuna) (soluzione madre: 0,3 mM; vedere la tabella dei materiali), che è un colorante di DNA fluorescente di colore rosso lontano che colora il DNA solo nelle cellule permeabilizzate. Utilizzare una diluizione 1:250 nel tampone di lavaggio per 10 minuti a temperatura ambiente in condizioni di oscurità.
- Lavare gli ovociti in tre diverse gocce di tampone di lavaggio (50 μL ciascuna) a temperatura ambiente. Lasciarli per 5 minuti in ogni goccia, quindi trasferirli in piccole gocce (circa 5 μL ciascuna) di tampone di lavaggio in una capsula di Petri con fondo di vetro da 35 mm (vedere la tabella dei materiali) per microscopia confocale (Figura 2A).
NOTA: Il lavaggio sia della colorazione del DNA che dell'anticorpo secondario viene eseguito contemporaneamente.
4. Microscopia confocale
NOTA: La microscopia confocale deve essere eseguita immediatamente per evitare la riduzione dell'intensità della fluorescenza dopo il posizionamento degli ovociti in piastre con fondo di vetro. È necessario l'accesso a un microscopio confocale (vedi Tabella dei materiali) con tavolino motorizzato.
- Configurazione del microscopio
- Nel sistema confocale, accendere il controller laser, i laser, il controller del microscopio, le lampade per la luce trasmessa e il PC (Figura 2B, D).
- Apri il software confocale e scegli la lente a olio 40x.
- Posizionate la capsula nel portacampioni e cercate di mettere a fuoco gli ovociti spostando il tavolino sugli assi XY e Z utilizzando il joystick (Figura 2C).
- Scansione degli ovociti
- Impostare la potenza del laser, il guadagno e la dimensione del foro stenopeico in modo indipendente per ogni esperimento al fine di ridurre al minimo la saturazione.
- Per ogni ovocita, impostare l'area di interesse, in particolare nel nucleo nell'area del DNA. Definire i bordi dell'area del DNA e regolare la dimensione del passo z a 3 μm. Quindi, avvia la scansione.
- Salvare le immagini per ogni cella della cartella selezionata.
- Al termine della scansione, uscire dal software, spegnere il computer e spegnere il controller laser, i laser, il controller del microscopio e le lampade per la luce trasmessa.
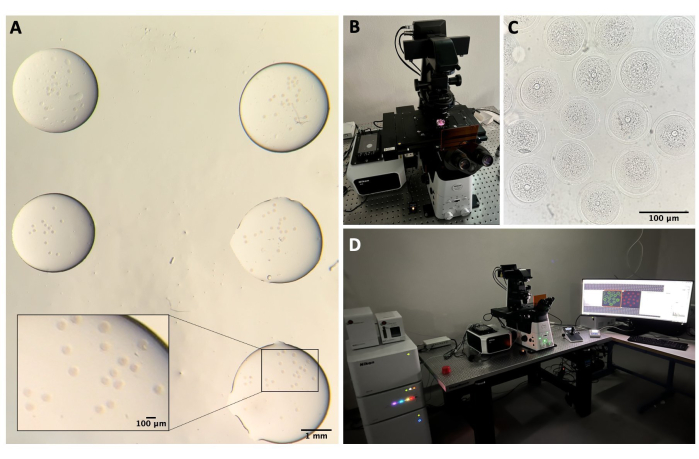
Figura 2: Microscopia confocale. (A) Ovociti fissati dopo l'esecuzione del protocollo di immunofluorescenza e della colorazione del DNA, che si trovano in gocce separate di tampone di lavaggio, ricoperti di olio minerale, posti in un piatto con fondo di vetro e preparati per l'imaging al microscopio confocale. Ogni goccia contiene una diversa categoria sperimentale. Immagine ottenuta attraverso gli oculari dello stereomicroscopio. Barra della scala = 1 mm/100 μm per la parte ingrandita. (B) Lastra con fondo di vetro posta sul tavolino del microscopio confocale. (C) Immagine in campo chiaro di ovociti ottenuta mediante microscopia confocale. Barra della scala = 100 μm. (D) Il sistema di microscopia confocale. Fare clic qui per visualizzare una versione più grande di questa figura.
5. Analisi di imaging
- Scarica Fiji ImageJ-win64 nel browser (https://imagej.net/software/fiji/downloads), aprilo e importa i dati come file stack TIFF.
NOTA: Aprire ogni file di ovociti separatamente. - Clicca sull'immagine | Colore | Dividi canali per dividere tutti i canali.
- Fare clic su LUT (Look up Table) e scegliere i colori preferiti per ciascun canale.
- Clicca sull'immagine | Colore | Unisci canali per unire i canali per γΗ2ΑΧ e DNA. Lasciare libero il canale in campo chiaro.
- Negli ovociti NSN e negli ovociti SN con bassi livelli di danno al DNA, γΗ2ΑΧ viene rilevato come focolai nella regione del DNA. In questo caso, fare clic sul comando " Multipunto" o punto e selezionare ogni fuoco γΗ2ΑΧ che coincide con il DNA. Ripeti questo passaggio per tutte le pile.
- Negli ovociti SN con alti livelli di danno al DNA, il segnale γΗ2ΑΧ è distribuito in tutta la regione del DNA. In questo caso, fare clic su Immagine | Pile | Z e, con il comando Selezioni a mano libera , selezionare l'intera area DNA.
- Per misurare la fluorescenza γΗ2ΑΧ, fare clic su Analizza | Misurare e copiare le misurazioni in un file .xlsx. Quindi, calcola la fluorescenza media, normalizza i valori e conta il numero di fuochi prima di creare qualsiasi grafico.
- Fare clic su Analizza | Imposta scala per impostare la scala e quindi su Analizza | Strumenti | Barra di scala per aggiungere una barra di scala ai canali.
Risultati
Utilizzando la procedura qui dimostrata, le ovaie di topo sono state sezionate, il grasso è stato rimosso e sono stati raccolti ovociti allo stadio GV completamente cresciuti. Quindi, le cellule del cumulo sono state rimosse mediante pipettaggio ripetitivo utilizzando una pipetta stretta e sono state poste in gocce fresche di terreno M2-IBMX e ricoperte di olio minerale su un blocco caldo (37 °C) (Figura 1A-F). Sono state preparate tre diverse concentrazioni di etoposide (5 μg/mL, 20 μg/mL e 50 μg/mL) utilizzando una concentrazione di etoposide di 20 mg/mL. Gli ovociti allo stadio GV sono stati posti in tre distinte concentrazioni di etoposide per 1 ora in gocce ricoperte di olio minerale e protette dalla luce a 37 °C. È stato quindi seguito il protocollo di immunofluorescenza, come descritto in dettaglio nella sezione del protocollo, e gli ovociti sono stati posti in piastre con fondo di vetro e osservati mediante microscopia confocale (Figura 2).
Negli ovociti in stadio SN GV, immediatamente dopo il danno al DNA, la presenza di γH2AX è aumentata a tutte le concentrazioni di etoposide (5 μg/mL, 20 μg/mL e 50 μg/mL) e il γH2AX è stato distribuito in tutta la regione del DNA (Figura 3). La quantificazione e la stima del DSB sono state eseguite osservando l'intensità della fluorescenza γH2AX nei siti del DNA. La fluorescenza γH2AX si è intensificata proporzionalmente all'aumentare delle concentrazioni di etoposide. Inoltre, dopo un arresto protraniato della profase (20 ore dopo il trattamento con etoposide), gli ovociti allo stadio GV hanno mostrato la capacità di ridurre il numero e l'intensità dei focolai γH2AX, il che implica la presenza di processi di riparazione attivi negli ovociti con arresto dello stadio GV (Figura 3E).
A differenza degli ovociti SN, in cui la fluorescenza γH2AX è stata distribuita attraverso il DNA, negli ovociti NSN, γH2AX è stato mostrato in focolai immediatamente dopo il trattamento con etoposide a 20 μg/mL. Abbiamo stimato il numero di focolai che coincidevano con l'area del DNA, calcolato la fluorescenza di ogni focolaio e presentato la fluorescenza media di tutti gli ovociti. Sia la fluorescenza che il numero di focolai hanno mostrato differenze statisticamente significative tra le due categorie di ovociti (Figura 4).
La microscopia confocale fornisce informazioni sul numero e l'intensità dei focolai in diverse pile Z, aiutando così a identificare la presenza di danni al DNA e le dinamiche di riparazione in punti temporali distinti. La scansione Galvano fornisce una scansione di precisione con uno sfondo basso e una migliore analisi delle immagini di scansione.
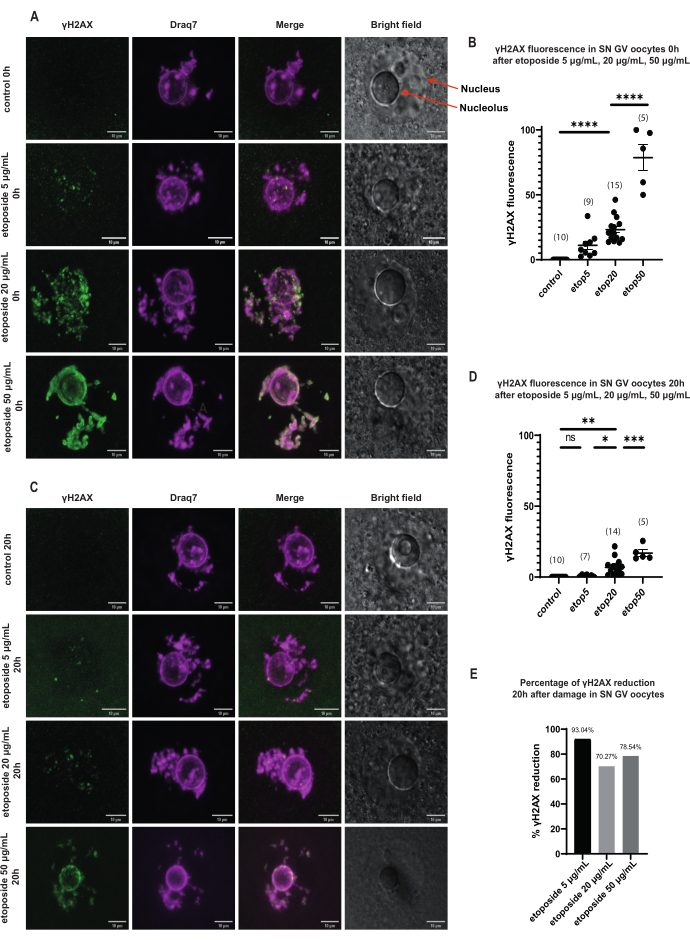
Figura 3: Riduzione di γH2AX in ovociti in stadio SN GV trattati con tre diverse concentrazioni di etoposide dopo arresto prolungato di GV. (A) Fluorescenza γH2AX in ovociti in stadio SN GV 0 h dopo il trattamento con etoposide. Il γH2AX aumenta immediatamente dopo l'esposizione a tutte le concentrazioni di etoposide e l'aumento è dipendente dalla concentrazione (verde: γΗ2ΑΧ, magenta: DNA). Le immagini sono proiezioni Z-stack e la luminosità/contrasto sono stati regolati per ciascun canale utilizzando Fiji / ImageJ. Barra della scala = 10 μm. (B) Grafico della fluorescenza γH2AX in ovociti in stadio SN GV 0 h dopo il trattamento con concentrazioni distinte di etoposidi. I dati rappresentano la media ± SEM. Ogni punto rappresenta un ovocita (il numero di ovociti è mostrato nel grafico), (ns = non significativo, ** p < 0,005, **** p < 0,0001, ANOVA unidirezionale con test di confronto multiplo di Tukey). (C) Fluorescenza γH2AX in ovociti in stadio SN GV 20 ore dopo il trattamento con etoposidi. γH2AX si riduce 20 ore dopo l'esposizione a tutte le concentrazioni di etoposide (verde: γΗ2ΑΧ, magenta: DNA). Le immagini sono proiezioni Z-stack e la luminosità/contrasto sono stati regolati per ciascun canale utilizzando Fiji/ImageJ. Barra della scala = 10 μm. (D) Grafico della fluorescenza γH2AX in ovociti in stadio SN GV 20 ore dopo il trattamento con concentrazioni distinte di etoposide. I dati rappresentano la media ± SEM. Ogni punto rappresenta un ovocita (il numero di ovociti è mostrato nel grafico), (ns = non significativo, * p < 0,05, ** p < 0,005, *** p < 0,0005, **** p < 0,0001, ANOVA unidirezionale con test di confronto multiplo di Tukey). (E) Grafico a barre della riduzione della fluorescenza γH2AX in ovociti in stadio SN GV dopo arresto della profase in ovociti trattati con etoposide. Il numero sopra ogni colonna indica la diminuzione percentuale della fluorescenza γH2AX. Fare clic qui per visualizzare una versione più grande di questa figura.
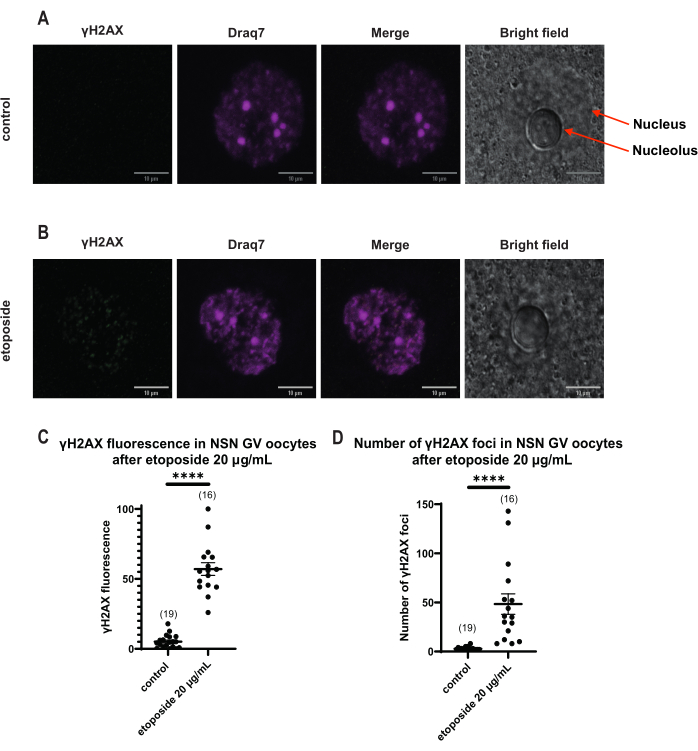
Figura 4: Fosforilazione di Η2ΑΧ in ovociti NSN in stadio GV dopo trattamento con etoposide a 20 μg/mL. (A) Immagini confocali rappresentative di un ovocita di controllo NSN in fase GV (verde: γΗ2ΑΧ, magenta: DNA). Le immagini sono proiezioni Z-stack e la luminosità/contrasto sono stati regolati per ciascun canale utilizzando Fiji/ImageJ. Barra della scala = 10 μm. (B) Immagini confocali rappresentative di un ovocita NSN in stadio GV trattato con etoposide (verde: γΗ2ΑΧ, magenta: DNA). Gli ovociti sono stati fissati 0 ore dopo il trattamento con etoposidi. Le immagini sono proiezioni Z-stack e la luminosità/contrasto sono stati regolati per ciascun canale utilizzando Fiji/ImageJ. Barra della scala = 10 μm. (C) La fluorescenza γΗ2ΑΧ normalizzata negli ovociti in stadio NSN GV dopo 20 μg/mL di trattamento con etoposide. I dati rappresentano la media ± SEM. Ogni punto rappresenta un ovocita (il numero di ovociti è mostrato nel grafico), tratto da due esperimenti indipendenti (**** p < 0,0001, t-test non parametrico spaiato, U-test di Mann-Whitney). (D) Numero di focolai di γΗ2ΑΧ negli ovociti in stadio NSN GV dopo il trattamento con etoposide da 20 μg/mL. I dati rappresentano la media ± SEM. Ogni punto rappresenta un ovocita (il numero di ovociti è mostrato nel grafico), tratto da due esperimenti indipendenti (**** p < 0,0001, t-test non parametrico spaiato, U-test di Mann-Whitney). Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Utilizzando il metodo qui descritto, abbiamo rilevato DSB negli ovociti di mammifero. Questo metodo consente di rilevare e studiare il processo di riparazione del DNA negli ovociti. Lo stesso protocollo potrebbe essere utilizzato anche per l'analisi di altre proteine che partecipano ai processi fisiologici negli ovociti di mammifero. È importante studiare come gli ovociti rispondono a potenziali danni al DNA per comprendere meglio la causa della subfertilità femminile nell'uomo.
Studiare la risposta al danno al DNA negli ovociti dei mammiferi può essere difficile a causa della sensibilità degli ovociti. La manipolazione degli ovociti richiede temperature specifiche e concentrazioni di CO 2 e O2 . Allo stesso tempo, gli ovociti devono essere protetti dalla luce. La manipolazione deve essere effettuata utilizzando pipette di vetro non troppo strette, in quanto ciò potrebbe essere dannoso per gli ovociti, ma anche non troppo larghe, in quanto ciò potrebbe causare la diluizione del terreno e, quindi, influire negativamente sulla procedura di fissazione. In ogni fase della fissazione, vengono utilizzate diverse gocce di tamponi per ridurre al minimo l'effetto di diluizione. Un modo alternativo per osservare i DSB è il saggio Comet24. Anche se questa tecnica è più sensibile, è più complicata. Allo stesso tempo, utilizzando il test Comet, non è possibile rilevare l'esatta regione del DNA in cui si verifica il danno e nelle cellule con abbondanti molecole di RNA, come gli ovociti in stadio GV25, il fondo potrebbe essere aumentato, portando a un falso segnale di danno al DNA26.
Utilizzando il protocollo di immunofluorescenza qui descritto, siamo in grado di rilevare i DSB con precisione e stimare il progresso della riparazione negli ovociti in stadio GV, come indicato dalla riduzione della fluorescenza γH2AX nel tempo. Tuttavia, una limitazione di questo metodo è che alcuni anticorpi possono presentare una distribuzione aspecifica in tutto l'ooplasma, portando così a immagini con un'elevata fluorescenza di fondo. Il tampone PFA-Tx-100 viene utilizzato al posto del PFA sequenziale e del Tx-100, in quanto abbiamo osservato che migliora il processo di fissazione consentendo la rilevazione di una minore fluorescenza di fondo e non specifica. Una seconda limitazione dell'uso di γH2AX per il rilevamento di DSB è che il danno non può essere stimato dopo GVBD a causa della fosforilazione spontanea di γH2AX nella meiosi23.
In questo protocollo di immunofluorescenza, gli ovociti rimangono in un tampone liquido e non possono essere conservati all'interno di vetrini. Questo fatto rende difficile conservare le cellule fisse per giorni dopo l'aggiunta dell'anticorpo secondario. Al fine di ottenere immagini di buona qualità e di non perdere il segnale, è preferibile eseguire l'imaging entro poche ore dall'aggiunta dell'anticorpo secondario. Va anche notato che la scansione dei nuclei attraverso l'asse Z potrebbe rendere il segnale più debole a causa della sovraesposizione. Per questo motivo, è preferibile ridurre la potenza del laser e aumentare la velocità di scansione.
Infine, un'altra limitazione del protocollo di immunofluorescenza è che può essere utilizzato solo per cellule fisse/non viventi. Pertanto, possiamo stimare solo la presenza e l'assenza di fattori in punti temporali specifici senza sapere se ci sono fluttuazioni nella loro concentrazione o cambiamenti nel loro comportamento nel tempo. Questo problema potrebbe essere superato utilizzando l'imaging di cellule vive e marcatori marcati con fluorescenza.
Divulgazioni
Gli autori non hanno conflitti di interesse.
Riconoscimenti
Riconosciamo il sostegno a questo lavoro da parte del progetto "Creazione di infrastrutture di 'capacity building' nella ricerca biomedica (BIOMED-20)" (MIS 5047236), che è implementato nell'ambito dell'azione "Rafforzamento dell'infrastruttura di ricerca e innovazione", finanziata dal programma operativo "Competitività, imprenditorialità e innovazione" (NSRF 2014-2020) e cofinanziata dalla Grecia e dall'Unione europea (Fondo europeo di sviluppo regionale).
Materiali
| Name | Company | Catalog Number | Comments |
| 3 mL Pasteur pipettes in LDPE, graduated | APTACA | 1502 | |
| 10 cc syringes | SoftCare | 114.104.21 | |
| Alexa Fluor 488-conjugated goat anti-rabbit Secondary Ab | Biotium | 20012 | |
| Anti-phospho-H2A.X (Ser139) | Merck Millipore | 07-164 | |
| ARE Heating Magnetic Stirrer | VELP Scientifica | F20500162 | |
| BD FALCON 5 mL Polystyrene Round-Bottom Tubes | BD Biosciences | 352054 | |
| BD Microlance 3 Needles 27 G - 0.40 x 13 mm | Becton Dickinson | 300635 | |
| Bovine Serum Albumin Fraction V | Roche | 10735078001 | |
| DMSO Anhydrous | Biotium | 90082 | |
| DRAQ7 DNA dye | BioStatus | DR71000 | |
| EGTA | Sigma-Aldrich | E4378-25G | |
| EMSURE MgCl2. 6H2O | Merck Millipore | 1058330250 | |
| Etoposide | CHEMIPHARM | L01CB01 | |
| FALCON 14 mL Polystyrene Round-Bottom Tubes | Corning Science | 532057 | |
| FALCON Tissue Culture Dishes, Easy-Grip, 35 x 10 mm Style | Corning Science | 353001 | |
| Glass Bottom Culture Dishes (35 mm Petri dish/ 14 mm Microwell, No. 0 coverglass) | MatTek Corporation | P35G-0-14-C | |
| HEPES | Sigma-Aldrich | H6147-25G | |
| HERACELL 150i CO2 Incubator | ThermoFisher Scientific | 50116048 | |
| IBMX powder | Sigma-Aldrich | I5879-100MG | |
| Leica M125 Stereo Microscope | Leica Microsystems | ||
| M16 Medium | Sigma-Aldrich | M7292 | |
| M2 Medium | Sigma-Aldrich | M7167 | |
| Mineral Oil | Sigma-Aldrich | M5310 | |
| NaN3 | Honeywell | 13412H | |
| NaOH | Merck Millipore | 1064981000 | |
| Nikon AX ECLIPSE Ti2 Confocal Microscope | Nikon Corporation | ||
| Nikon SMZ800N Stereo Microscope | Nikon Corporation | ||
| Paraformaldehyde | Sigma-Aldrich | 158127 | |
| Pasteur pippettes, glass, long form 230 mm | DURAN WHEATON KIMBLE | 357335 | |
| pH/ORP meter | Hanna Instruments Ltd | HI2211 | |
| Phosphate buffered saline tablets | Sigma-Aldrich | P4417-100TAB | |
| PIPES | Sigma-Aldrich | P1851 | |
| PMSG Protein Lyophilised | Genway Biotech (now AVIVA Systems Biology) | GWB-2AE30A (now OPPA01037) | |
| QBD4 Dry block heater | Grant Instruments (Cambridge) Ltd | A25218 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Whatman Puradisc 25 mm 0.2 μm filters | GE Healthcare | 6780-2502 |
Riferimenti
- Wang, X., Pepling, M. E. Regulation of meiotic prophase one in mammalian oocytes. Frontiers in Cell and Developmental Biology. 9, 667306(2021).
- Filatov, M., Khramova, Y., Semenova, M. Molecular mechanisms of prophase I meiotic arrest maintenance and meiotic resumption in mammalian oocytes. Reproductive Sciences. 26 (11), 1519-1537 (2019).
- Adhikari, D., et al. Inhibitory phosphorylation of Cdk1 mediates prolonged prophase I arrest in female germ cells and is essential for female reproductive lifespan. Cell Research. 26 (11), 1212-1225 (2016).
- Sun, S. C., Kim, N. H. Molecular mechanisms of asymmetric division in oocytes. Microscopy and Microanalysis. 19 (4), 883-897 (2013).
- Jones, K. T. Mammalian egg activation: From Ca2+ spiking to cell cycle progression. Reproduction. 130 (6), 813-823 (2005).
- Solc, P., Schultz, R. M., Motlik, J. Prophase I arrest and progression to metaphase I in mouse oocytes: comparison of resumption of meiosis and recovery from G2-arrest in somatic cells. Molecular Human Reproduction. 16 (9), 654-664 (2010).
- Parfenov, V., Potchukalina, G., Dudina, L., Kostyuchek, D., Gruzova, M. Human antral follicles: oocyte nucleus and the karyosphere formation (electron microscopic and autoradiographic data). Gamete Research. 22 (2), 219-231 (1989).
- Zuccotti, M., Piccinelli, A., Giorgi Rossi, P., Garagna, S., Redi, C. A. Chromatin organization during mouse oocyte growth. Molecular Reproduction and Development. 41 (4), 479-485 (1995).
- Sun, X., et al. Comprehensive analysis of nonsurrounded nucleolus and surrounded nucleolus oocytes on chromatin accessibility using ATAC-seq. Molecular Reproduction and Development. 90 (2), 87-97 (2023).
- Zuccotti, M., Bellone, M., Longo, F., Redi, C. A., Garagna, S. Fully-mature antral mouse oocytes are transcriptionally silent but their heterochromatin maintains a transcriptional permissive histone acetylation profile. Journal of Assisted Reproduction and Genetics. 28 (12), 1193-1196 (2011).
- Winship, A. L., Stringer, J. M., Liew, S. H., Hutt, K. J. The importance of DNA repair for maintaining oocyte quality in response to anti-cancer treatments, environmental toxins and maternal ageing. Human Reproduction Update. 24 (2), 119-134 (2018).
- Lieber, M. R. The mechanism of human nonhomologous DNA end joining. The Journal of Biological Chemistry. 283 (1), 1-5 (2008).
- Chiruvella, K. K., Liang, Z., Wilson, T. E. Repair of double-strand breaks by end joining. Cold Spring Harbor Perspectives in Biology. 5 (5), 012757(2013).
- Chang, H. H. Y., Pannunzio, N. R., Adachi, N., Lieber, M. R. Non-homologous DNA end joining and alternative pathways to double-strand break repair. Nature Reviews. Molecular Cell Biology. 18 (8), 495-506 (2017).
- Shibata, A., Jeggo, P. A. Roles for the DNA-PK complex and 53BP1 in protecting ends from resection during DNA double-strand break repair. Journal of Radiation Research. 61 (5), 718-726 (2020).
- Mohiuddin, I. S., Kang, M. H. DNA-PK as an emerging therapeutic target in cancer. Frontiers in Oncology. 9, 635(2019).
- Marangos, P., Carroll, J. Oocytes progress beyond prophase in the presence of DNA damage. Current Biology. 22 (11), 989-994 (2012).
- Paull, T. T., et al. A critical role for histone H2AX in recruitment of repair factors to nuclear foci after DNA damage. Current Biology. 10 (15), 886-895 (2000).
- Bakkenist, C. J., Kastan, M. B. DNA damage activates ATM through intermolecular autophosphorylation and dimer dissociation. Nature. 421 (6922), 499-506 (2003).
- Marangos, P., et al. DNA damage-induced metaphase I arrest is mediated by the spindle assembly checkpoint and maternal age. Nature Communications. 6, 8706(2015).
- Lavrentyeva, E. A., Shishova, K. V., Zatsepina, O. V. Differences in nuclear dynamics in mouse GV oocytes with a diverse chromatin configuration. Biology Bulletin Russian Academy of Sciences. 46, 332-341 (2019).
- Montecucco, A., Zanetta, F., Biamonti, G. Molecular mechanisms of etoposide. EXCLI Journal. 14, 95-108 (1998).
- Mayer, A., et al. DNA damage response during mouse oocyte maturation. Cell Cycle. 15 (4), 546-558 (2016).
- Olive, P., Banáth, J. The comet assay: A method to measure DNA damage in individual cells. Nature Protocols. 1, 23-29 (2006).
- Wu, D., Dean, J. EXOSC10 sculpts the transcriptome during the growth-to-maturation transition in mouse oocytes. Nucleic Acids Research. 48 (10), 5349-5365 (2020).
- Simon, L., Emery, B., Carrell, D. DNA damage: COMET assay. Manual of Sperm Function Testing in Human Assisted Reproduction. Agarwal, A., Henkel, R., Majzoub, A. , 202-212 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon