È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Virus pseudotipizzati come strumento molecolare per monitorare le risposte immunitarie umorali contro SARS-CoV-2 tramite test di neutralizzazione
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
I virus pseudotipizzati (PV) sono virioni difettosi di replicazione che vengono utilizzati per studiare le interazioni ospite-virus in condizioni più sicure rispetto alla manipolazione di virus autentici. Di seguito è presentato un protocollo dettagliato che mostra come i PV SARS-CoV-2 possono essere utilizzati per testare la capacità neutralizzante del siero dei pazienti dopo la vaccinazione COVID-19.
Abstract
I virus pseudotipizzati (PV) sono strumenti molecolari che possono essere utilizzati per studiare le interazioni ospite-virus e per testare la capacità neutralizzante di campioni di siero, oltre al loro uso più noto nella terapia genica per la veicolazione di un gene di interesse. I PV sono difettosi di replicazione perché il genoma virale è diviso in diversi plasmidi che non sono incorporati nei PV. Questo sistema sicuro e versatile consente l'uso di PV in laboratori di livello di biosicurezza 2. Qui, presentiamo una metodologia generale per produrre PV lentivirali basata su tre plasmidi come menzionato qui: (1) il plasmide della spina dorsale che trasporta il gene reporter necessario per monitorare l'infezione; (2) il plasmide impacchettatore che trasporta i geni per tutte le proteine strutturali necessarie per generare le PVs; (3) il plasmide di espressione della glicoproteina superficiale dell'involucro che determina il trofismo del virus e media l'ingresso virale nella cellula ospite. In questo lavoro, SARS-CoV-2 Spike è la glicoproteina dell'involucro utilizzata per la produzione di lentivirus pseudotipizzati SARS-CoV-2 non replicati.
In breve, le cellule di impacchettamento (HEK293T) sono state co-trasfettate con i tre diversi plasmidi utilizzando metodi standard. Dopo 48 ore, il surnatante contenente i PV è stato raccolto, filtrato e conservato a -80 °C. L'infettività dei PV di SARS-CoV-2 è stata testata studiando l'espressione del gene reporter (luciferasi) in una linea cellulare bersaglio 48 ore dopo l'infezione. Maggiore è il valore delle unità di luminescenza relativa (RLU), maggiore è il tasso di infezione/trasduzione. Inoltre, i PV infettivi sono stati aggiunti ai campioni di siero diluiti in serie per studiare il processo di neutralizzazione dell'ingresso degli pseudovirus nelle cellule bersaglio, misurato come riduzione dell'intensità dell'RLU: valori più bassi corrispondenti a un'elevata attività neutralizzante.
Introduzione
I virus pseudotipizzati (PV) sono strumenti molecolari utilizzati in microbiologia per studiare le interazioni ospite-virus e patogeno-patogeno 1,2,3,4. I PV sono costituiti da una parte interna, il nucleo virale che protegge il genoma virale, e da una parte esterna, le glicoproteine dell'involucro sulla superficie del virus che definisce il tropismo5. Uno pseudovirus è incompetente nella replicazione della cellula bersaglio perché non contiene tutte le informazioni genetiche per generare nuove particelle virali. Questa combinazione di caratteristiche peculiari rende i PV un'alternativa sicura a un virus wildtype. I virus wildtype, d'altra parte, sono altamente patogeni e non possono essere utilizzati nei laboratori BSL 2 per l'analisi6.
L'infettività delle PV può essere monitorata da un gene reporter, di solito codificante per una proteina fluorescente (GFP, RFP, YFP) o un enzima che produce prodotti chemiluminescenti (luciferasi). Questo è contenuto in uno dei plasmidi utilizzati per la produzione di PV e incorporato nel genoma dello pseudovirus7.
Attualmente esistono diversi tipi di nuclei fotovoltaici, comprese le particelle derivate da lentivirali basate sul genoma dell'HIV-1. Il grande vantaggio dei PV basati sull'HIV-1 rispetto ad altre piattaforme è il loro intrinseco processo di integrazione nel genoma della cellula bersaglio8. Sebbene l'HIV-1 sia un virus altamente contagioso e sia l'agente eziologico dell'AIDS, questi vettori lentivirali sono sicuri da usare grazie alle ampie fasi di ottimizzazione nel corso degli anni. Condizioni ottimali di sicurezza sono state raggiunte con l'introduzione di vettori lentivirali di seconda generazione, in cui i geni virali sono stati impoveriti senza influenzare le capacità di trasduzione9. La 3ae la 4a generazione hanno contribuito ad aumentare la sicurezza della manipolazione dei vettori lentivirali con l'ulteriore suddivisione del genoma virale in plasmidi separati10,11. Le ultime generazioni di PV sono generalmente impiegate per produrre vettori lentivirali per la terapia genica.
I PV possono essere utilizzati per studiare le interazioni tra virus e cellule ospiti, sia durante la fase di produzione che durante la fase di infezione. I PV sono particolarmente impiegati nei saggi di neutralizzazione degli pseudovirus (PVNA). I PVNA sono ampiamente validati per valutare il potenziale di neutralizzazione del siero o del plasma prendendo di mira la glicoproteina virale sull'involucro12,13 del PV. L'attività di neutralizzazione, espressa come concentrazione inibitoria 50 (IC50), è definita come la diluizione del siero/plasma che blocca il 50% dell'ingresso delle particelle virali14. In questo protocollo, abbiamo descritto la messa a punto di un PVNA per testare l'attività anticorpale contro la Sindrome Respiratoria Acuta Grave - Coronavirus 2 (SARS-CoV-2) in sieri raccolti prima e dopo aver ricevuto una dose di vaccino di richiamo.
Protocollo
Il presente protocollo è stato approvato e segue le linee guida del Comitato Etico dell'Università di Verona (protocollo di approvazione numero 1538). Il consenso scritto informato è stato ottenuto dai soggetti umani che hanno partecipato allo studio. I campioni di sangue intero sono stati raccolti da volontari operatori sanitari (HCW) che erano in procinto di ricevere vaccini anti-SARS-CoV-2. Questi campioni sono stati raccolti in provette di plastica contenenti anticoagulanti per il successivo isolamento del siero15.
Tutti i seguenti processi devono essere eseguiti in una cappa biologica di Classe 2, lavorando in condizioni sterili. La manipolazione dei virus deve essere eseguita con cura e tutti i prodotti di scarto devono essere neutralizzati in una soluzione di candeggina diluita. Una panoramica del protocollo è mostrata nella Figura 1.
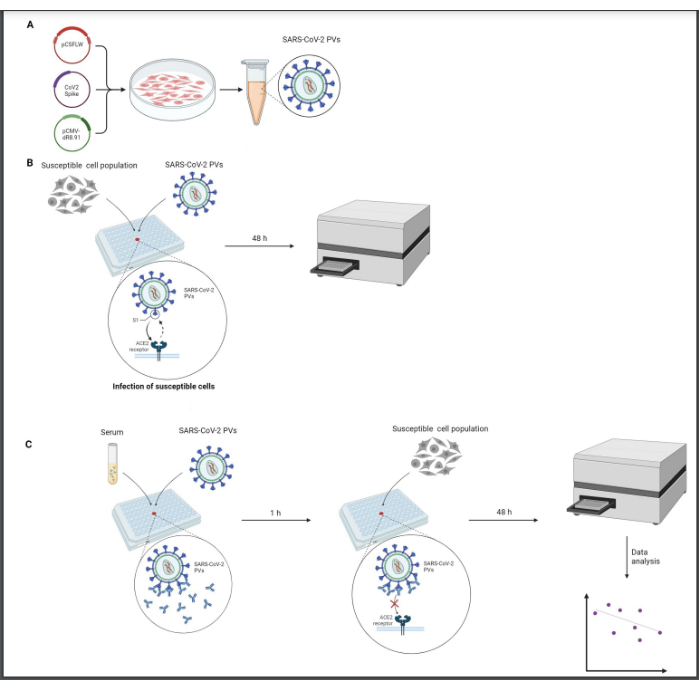
Figura 1: Rappresentazione grafica di un saggio di neutralizzazione. (A) produzione di PV, (B) titolazione di PV e (C) test di neutralizzazione. Tutte le procedure vengono eseguite in una cappa biologica di classe 2 in condizioni sterili. La fase di titolazione (B) deve essere eseguita per standardizzare i livelli di infettività dei PV prima dell'uso nel saggio di neutralizzazione (C). Questa figura è stata creata con BioRender. Fare clic qui per visualizzare una versione più grande di questa figura.
1. Test di produzione e infettività di SARS-CoV-2 PVs
- Seminare 5 x 10cellule da 5 HEK293T in Dulbecco's Modified Eagle Medium completo (DMEM, ad alto contenuto di glucosio, 10% siero fetale bovino (FBS), 1% L-glutammina, 1% penicillina/streptomicina) in una piastra a 6 pozzetti (6WP) per raggiungere una densità cellulare adeguata compatibile con il reagente di trasfezione utilizzato. Nel caso di esecuzione della trasfezione con polietilenimmina (PEI) (preparare il reagente seguendo le istruzioni del produttore), assicurarsi che le cellule raggiungano il 40-60% di densità il giorno della trasfezione (passaggio 1.3). Conservare le cellule in un incubatore umidificato a 37 °C e al 5% di CO2 .
- Prima della trasfezione, sostituire il terreno cellulare esaurito con terreno fresco senza antibiotici (DMEM, alto contenuto di glucosio, 10% FBS, 1% L-glutammina) per ottenere una maggiore efficienza di trasfezione.
NOTA: Il giorno dopo la semina, HEK293T cellule sono pronte per essere trasfettate. - Trasfettare le cellule HEK293T aderenti con un reagente di trasfezione adatto secondo le istruzioni del produttore. Se si utilizza il PEI, preparare due miscele e seguire i passaggi seguenti.
- Per preparare la miscela A, aggiungere 500 ng di plasmide16 per impacchettare pCMV-dR8.91, 750 ng di plasmide reporterpCSFLW 16 e 450 ng di plasmide esprimente SARS-CoV-2 Spike in 100 μL di terreno sierico ridotto.
- Per preparare la miscela B, aggiungere 17,5 μL di PEI (concentrazione: 1 mg/mL) a 100 μL di terreno sierico ridotto.
- Lasciare incubare entrambe le miscele a temperatura ambiente (RT) per 5 minuti. Successivamente, mescolare il contenuto di entrambe le provette aggiungendo la miscela PEI B alla miscela di DNA A.
- Incubare la provetta per 20-30 minuti a RT. Agitare delicatamente la provetta ogni 3-4 minuti per migliorare la miscelazione. Infine, aggiungere il composto alle celle HEK293T.
- 16-20 ore dopo la trasfezione, sostituire il terreno di coltura con DMEM fresco e completo. Incubare a 37 °C e 5% di CO2 per consentire la produzione di PV da parte di cellule trasfettate.
- 72 ore dopo la trasfezione, raccogliere il surnatante contenente PV. Quindi centrifugare a 1600 x g per 7 minuti a temperatura ambiente per rimuovere i detriti cellulari e le cellule morte e filtrarlo attraverso un filtro in acetato di cellulosa da 0,45 μm.
- FASE OPZIONALE: Per aumentare la resa finale del titolo PV, eseguire trasfezioni multiple, raggruppare i terreni cellulari contenenti PV e concentrarli utilizzando tubi di concentrazione.
- Procedere direttamente con le fasi successive ("titolazione PV", sezione 2) o aliquotare il mezzo contenente PV in provette adatte per conservarlo a -80 °C fino all'uso. Preparare un'aliquota aggiuntiva (400-500 μL) da utilizzare per la titolazione.
NOTA: La realizzazione di aliquote multiple garantirà la riproducibilità tra gli esperimenti evitando eccessivi cicli di disgelo-congelamento.
2. Titolazione PVs
- Utilizzare il terreno fresco contenente PV per le fasi successive o scongelare l'aliquota di prova (fase 1.7) per eseguire la titolazione del nuovo stock virale. Il congelamento di aliquote dello stesso stock fotovoltaico garantirà la riproducibilità.
- Aggiungere 50 μL di DMEM completo (o terreno completo compatibile con la linea cellulare target in uso) in tutti i pozzetti di una piastra a 96 pozzetti (96 WP) necessari per testare in duplicato lo stock PV, lasciando vuota la riga "A". Aggiungere 100 μL di PV alla riga "A". In base al numero di preparati da testare, lasciare una colonna senza il virus come controllo "solo cellulare" (Figura 2).
- Pipettare 50 μL dalla fila A alla fila B e ripetere questo processo fino alla fila G per ottenere diluizioni seriali della scorta iniziale. Eliminare il volume in eccesso dall'ultima riga.
- Staccare le cellule utilizzando tripsina/etilendiamminotetraacetico acido 1x (EDTA) nella soluzione salina tampone fosfato di Dulbecco 1x (DPBS 1x), dopo aver rimosso il terreno esausto e lavato le celle con DPBS 1x due volte. Preparare le cellule a una densità di 4 x 105 cellule/mL.
NOTA: In questo protocollo, l'infezione da PVs è stata testata sulla linea cellulare suscettibile HEK293T/ACE2; tali cellule sono state derivate da HEK293T, trasdotte utilizzando un vettore lentivirale per esprimere il recettore ACE2. - Aggiungere 50 μL di sospensione cellulare in ciascun pozzetto per garantire un numero di cellule di 2 x 104 cellule per pozzetto.
- Incubare a 37 °C e 5% di CO2 per 48 ore.
- Dopo l'incubazione, eseguire il test della luciferasi per ottenere la lettura secondo le istruzioni del produttore. Aggiungere 100 μL del reagente luciferasi ai pozzetti e incubare al buio a RT per 2 minuti. Spostare il contenuto di ciascun pozzetto su una piastra nera da 96 pozzetti (compatibile con il lettore di piastre disponibile) e leggere le piastre in un lettore di piastre da 96 pozzetti.
NOTA: Il luminometro utilizzato per la lettura della luciferasi produrrà un foglio di calcolo con i dati grezzi e non elaborati che verranno utilizzati per l'analisi a valle (in questo caso, un file Excel). L'infettività del virus sarà espressa come unità di luminescenza relativa (RLU) (descritte nel paragrafo 4.1).
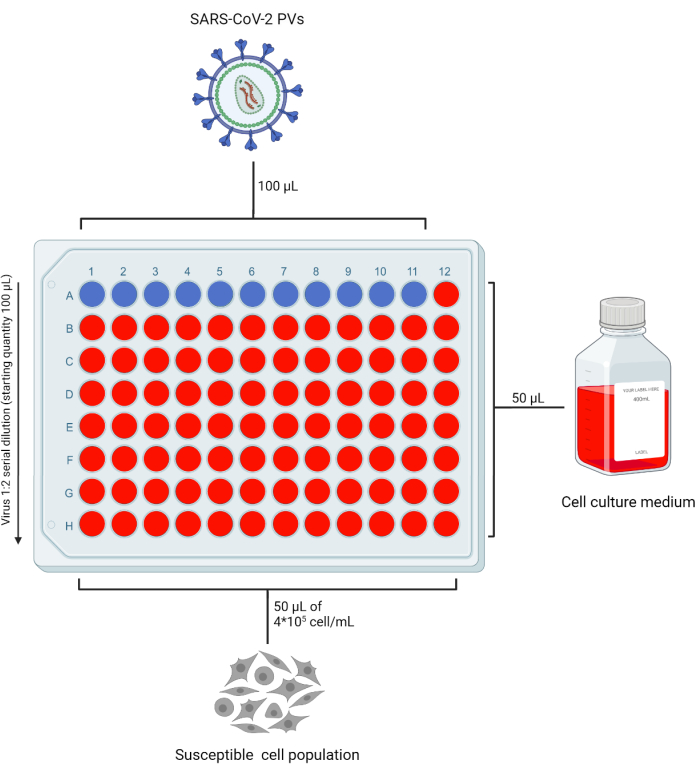
Figura 2: Layout rappresentativo di una piastra a 96 pozzetti per la titolazione di PV. Un volume fisso di surnatante contenente PV viene aggiunto alla riga A, colonne 1-11, e diluito in serie. L'ultima colonna viene lasciata come controllo "solo cella". Questa figura è stata creata con BioRender. Fare clic qui per visualizzare una versione più grande di questa figura.
3. Saggio di neutralizzazione
- Scongelare i sieri dei pazienti sul ghiaccio. Inattivare i campioni di siero incubandoli a 56 °C per 30 min.
- In una piastra da 96 pozzetti, aggiungere 50 μL di DMEM fresco e completo (o terreno completo compatibile con la linea cellulare bersaglio utilizzata) in ciascuno dei seguenti pozzetti: dalla riga B (colonne 1-10) alla riga H (colonne 1-10). Inserire 95 μL di DMEM fresco e completo nella riga A (colonne 1-10). Aggiungere rispettivamente 50 μL e 100 μL di DMEM completo nei pozzetti delle colonne 11 e 12. Si tratta rispettivamente dei controlli infetti (controllo del virus, o VC) e non infetti (solo cellule, o CC), (Figura 3).
- Aggiungere 5 μL di campioni di siero/plasma inattivati dal calore nella riga A (colonne 1-10). Ogni campione sarà in duplice copia. Con una pipetta multicanale, miscelare i campioni nella prima fila e spostare 50 μL di terreno contenente siero dalla fila A alla fila B. Ripetere questo processo fino all'ultima riga (Figura 3). Scartare i restanti 50 μL.
- Scongelare il numero necessario di aliquote di PV e diluire a ≥ 104 RLU/mL. Aggiungere 50 μL di terreno diluito contenente PV a ciascun pozzetto (dalla colonna 1 alla colonna 11) utilizzando una pipetta multicanale per ottenere una diluizione 1:1 del siero/plasma inattivato termicamente al virus. Incubare a 37 °C e al 5% di CO2 per 1 ora per consentire agli anticorpi nei campioni di siero di legarsi alla proteina spike del SARS-CoV-2 sui PV.
- Preparare almeno 5 mL di sospensione di cellule sensibili (HEK293T/ACE2) ad una densità cellulare di 4 x 105 cellule/mL. Aggiungere 50 μL di sospensione cellulare a ciascun pozzetto e incubare a 37 °C e 5% di CO2 per 48 ore.
- Dopo l'incubazione, eseguire la lettura del saggio della luciferasi secondo le istruzioni del produttore, come descritto al punto 2.7.
NOTA: Il luminometro utilizzato per la lettura della luciferasi produrrà un foglio di calcolo (in questo caso, .xlsx) con i dati grezzi e non elaborati che verranno utilizzati per l'analisi a valle (il file del saggio della luciferasi).
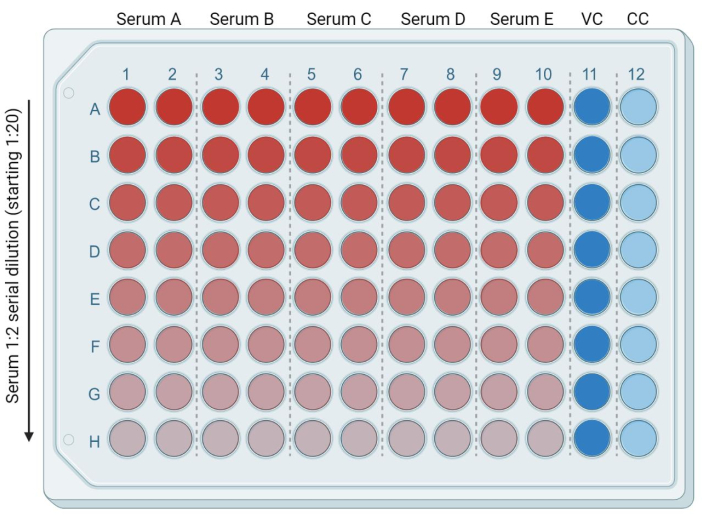
Figura 3: Rappresentazione della piastra basata sulla diluizione del siero. Il rosso brillante corrisponde a una maggiore quantità di siero e il blu brillante (colonna 11) corrisponde al controllo delle cellule infette (VC, controllo del virus). La corsia azzurra (colonna 12) corrisponde alle cellule non infette (CC, controllo cellulare). Questa figura è stata creata con BioRender. Fare clic qui per visualizzare una versione più grande di questa figura.
4. Analisi della titolazione
- Nel file del saggio della luciferasi, assegnare i nomi/titoli ai campioni corrispondenti.
- Moltiplicare la misura RLU per i fattori di diluizione (dall'alto verso il basso della griglia: 20x, 40x, 80x, 160x, 320x, 640x, 1,280x, 2,560x) per ottenere RLU/mL. Se si utilizzano fattori di diluizione diversi, modificare di conseguenza i fattori di moltiplicazione.
- Calcolare l'RLU/mL medio per ogni preparazione PV.
5. Analisi del saggio di neutralizzazione dei PV
- Nel file del foglio di calcolo del saggio della luciferasi (in questo caso, .xlsx), assegnare i titoli corrispondenti ai campioni testati. Inserire il fattore di diluizione del campione (40s, 80x, 160x, 320x, 640x, 1,280x, 2,560x, 5,120x). Calcolare il Log10 dei fattori di diluizione.
- Calcolare l'RLU media del controllo non infetto e infetto (Figura 3, colonne 11 e 12, rispettivamente). Questi valori saranno utili per la normalizzazione nel passaggio 5.5.
- Aprire un nuovo documento per l'analisi dei dati. Selezionare Analisi X/Y, immettere X come Numeri e Y come Immettere 2 valori di replica in sottocolonne affiancate.
- Immettere i valori Log10 (diluizione) come numeri X. Immettere l'RLU duplicato dei campioni.
- Vai a Analizza > Normalizza > Contrassegna tutti i campioni sullo stesso foglio. Immettere i valori medi VC e CC rispettivamente in Come viene definito lo 0%?, e Come viene definito il 100%?. Fare clic su OK.
- Nel foglio dati normalizzato, vai a Analizza analisi XY > > Analisi non lineari (adattamento curva). Contrassegnare tutti i campioni e fare clic su OK. Per Dose-risposta - Inibizione, selezionare log (inibitore) vs risposta normalizzata - pendenza variabile.
- In Vincola, modificare HillSlope in Deve essere minore di 0.
- In Output, seleziona Crea tabella di riepilogo e grafico. Fare clic su OK per ottenere le analisi finali. Un foglio di lavoro con un modello per l'analisi è fornito nel file supplementare 1.
Risultati
Questo protocollo descrive la produzione di PV di SARS-CoV-2 e un'applicazione a valle di questi PV per analizzare l'attività di neutralizzazione del siero/plasma di soggetti sottoposti a vaccinazione anti-COVID-1917. Inoltre, questo protocollo può essere applicato per produrre pseudotipi di ciascuna variante di SARS-CoV-2 che desta preoccupazione (VOC) per testare l'evoluzione della risposta neutralizzante. Nonostante questo protocollo faciliti lo studio della risposta immunitaria umorale dopo ...
Discussione
Sebbene l'utilizzo di un virus wildtype simuli l'infezione effettiva, i PV lentivirali sono un'opzione più sicura per studiare i meccanismi associati all'ingresso e all'infezione virale senza i severi requisiti di sicurezza necessari per lavorare con i virus patogeni 4,20,21. I PV sono composti da un nucleo virale difettoso di replicazione circondato dalla glicoproteina dell'involucro superficiale di un virus patogeno che è l'...
Divulgazioni
Gli autori dichiarano di non avere alcun conflitto di interessi.
Riconoscimenti
Riconosciamo il contributo dei volontari degli operatori sanitari. Questo progetto è stato sostenuto dal Dipartimento di Eccellenza 2023/2027, MUR, Italia. AR e DZ sono stati sostenuti da PRIN2022 (finanziamenti dell'UE; NextGenerationEU)
Materiali
| Name | Company | Catalog Number | Comments |
| 0.45 μm filter | SARSTEDT | 83 1826 | |
| 6-well plate | SARSTEDT | 83 3920 | |
| 96-well plate | SARSTEDT | 8,33,924 | |
| Amicon Ultra-15 Centrifugal Filter Units | Merck | 10403892 | |
| Black Opaque 96-well Microplate | Perkin Elmer | 60005270 | |
| Dulbecco's Modified Eagle Medium | SIGMA-ALDRICH | D6546 - 500ML | |
| Dulbecco's phosphate buffered saline (PBS 1x) | AUROGENE | AU-L0615-500 | |
| Foetal Bovine Serum | AUROGENE | AU-S1810-500 | |
| Graphpad Prism version 7 | graphpad dotmatics | NA | In the manuscript, we replace the commercial name with 'data analysis program' |
| HEK293T cells | ATCC | CRL-3216 | |
| HEK293T/ACE2 cells | ATCC | CRL-3216 | HEK293T has been transduced to overexpress ACE2 with a lentiviral vector. |
| L-glutamine | AUROGENE | AU-X0550-100 | |
| Luminometer - Victor3 | Perkin Elmer | HH35000500 | In the manuscript, we replace the commercial name with 'luminometer' |
| Opti-MEM | Thermo Fisher Scientific | 11058021 | In the manuscript, we replace the commercial name with 'reduced serum medium' |
| p8.91 packaging plasmid | Di Genova et al., 2021 | A kind gift from Prof. Nigel Temperton (ref 16.) | |
| pCSFLW reporter plasmid | Di Genova et al., 2021 | A kind gift from Prof. Nigel Temperton (ref 16.) | |
| Penicillin/streptomycin | AUROGENE | AU-L0022-100 | |
| Polyethylenimine, branched (PEI) (25 kDa) | SIGMA-ALDRICH | 408727 | |
| RRL.sin.cPPT.SFFV/Ace2.IRES-puro.WPRE (MT126) | Addgene | 145839 | This plasmid was used to generate HEK293Tcells/ACE2 |
| SARS-CoV-2 Spike expressing plasmid | Addgene | pGBW-m4137382 | |
| steadylite plus Reporter Gene Assay System | Perkin Elmer | 6066759 | In the manuscript, we replaced the commercial name with 'luciferase reading reagent' |
| Trypsin EDTA 1x | AUROGENE | AU-L0949-100 |
Riferimenti
- Ozaki, D. A., et al. International technology transfer of a GCLP-compliant HIV-1 neutralizing antibody assay for human clinical trials. Plos One. 7 (1), e30963 (2012).
- Pouget, M., et al. Generation of liposomes to study the effect of Mycobacterium tuberculosis lipids on HIV-1 cis- and trans-infections. International Journal of Molecular Sciences. 22 (4), 1945 (2021).
- McKay, L. G. A., et al. The HCV envelope glycoprotein down-modulates NF-κB signalling and associates with stimulation of the host endoplasmic reticulum stress pathway. Frontiers in Immunology. 13, 831695 (2022).
- Xiang, Q., Li, L., Wu, J., Tian, M., Fu, Y. Application of pseudovirus system in the development of vaccine, antiviral-drugs, and neutralizing antibodies. Microbiological Research. 258, 126993 (2022).
- Li, Q., Liu, Q., Huang, W., Li, X., Wang, Y. Current status on the development of pseudoviruses for enveloped viruses. Reviews in Medical Virology. 28, e1963 (2018).
- D'Apice, L., et al. Comparative analysis of the neutralizing activity against SARS-CoV-2 Wuhan-Hu-1 strain and variants of concern: Performance evaluation of a pseudovirus-based neutralization assay. Frontiers in Immunology. 13, 981693 (2022).
- Falzarano, D., Groseth, A., Hoenen, T. Development and application of reporter-expressing mononegaviruses: current challenges and perspectives. Antiviral Research. 103, 78-87 (2014).
- Gutierrez-Guerrero, A., Cosset, F. -. L., Verhoeyen, E. Lentiviral vector pseudotypes: Precious tools to improve gene modification of hematopoietic cells for research and gene therapy. Viruses. 12, 1016 (2020).
- Zufferey, R., Nagy, D., Mandel, R. J., Naldini, L., Trono, D. Multiply attenuated lentiviral vector achieves efficient gene delivery in vivo. Nature Biotechnology. 15 (9), 871-875 (1997).
- Dull, T. A third-generation lentivirus vector with a conditional packaging system. Journal of Virology. 72 (11), 8463-8471 (1998).
- Berkhout, B. A Fourth generation lentiviral Vector: Simplifying genomic gymnastics. Molecular Therapy. 25 (8), 1741-1743 (2017).
- Wu, X. Development and evaluation of a pseudovirus-luciferase assay for rapid and quantitative detection of neutralizing antibodies against Enterovirus 71. Plos One. 8 (6), e64116 (2013).
- Ferrara, F., et al. Development of lentiviral vectors pseudotyped with Influenza B hemagglutinins: application in vaccine immunogenicity, mAb potency, and sero-surveillance studies. Frontiers in Immunology. 12, 661379 (2021).
- Hu, J., et al. Development of cell-based pseudovirus entry assay to identify potential viral entry inhibitors and neutralizing antibodies against SARS-CoV-2. Genes & Diseases. 7 (4), 551-557 (2020).
- Dalle Carbonare, L., et al. Serology study after BTN162b2 vaccination in participants previously infected with SARS-CoV-2 in two different waves versus naïve. Communications Medicine. 1 (1), 38 (2021).
- Di Genova, C., et al. Production, titration, neutralisation, storage and lyophilisation of severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) lentiviral pseudotypes. Bio-protocol. 11 (21), e4236 (2021).
- Chmielewska, A. M., Czarnota, A., Bieńkowska-Szewczyk, K., Grzyb, K. Immune response against SARS-CoV-2 variants: The role of neutralization assays. NPJ Vaccines. 6 (1), 1-8 (2021).
- Chen, Q., et al. Development and optimization of a sensitive pseudovirus-based assay for HIV-1 neutralizing antibodies detection using A3R5 cells. Human Vaccines & Immunotherapeutics. 14 (1), 199-208 (2018).
- Gauger, P. C., Vincent, A. L. Serum virus neutralization assay for detection and quantitation of serum neutralizing antibodies to influenza A virus in swine. Methods in Molecular Biology (Clifton, N.J). 2123, 321-333 (2020).
- Miglietta, R., Pastori, C., Venuti, A., Ochsenbauer, C., Lopalco, L. Synergy in monoclonal antibody neutralization of HIV-1 pseudoviruses and infectious molecular clones. Journal of Translational Medicine. 12 (1), 346 (2014).
- Chen, M., Zhang, X. -. E. Construction and applications of SARS-CoV-2 pseudoviruses: A mini review. International Journal of Biological Sciences. 17 (6), 1574-1580 (2021).
- Zipeto, D., et al. Induction of human immunodeficiency virus neutralizing antibodies using fusion complexes. Microbes and Infection. 8 (6), 1424-1433 (2006).
- WHO Coronavirus (COVID-19) Dashboard. Available from: https://covid19.who.int (2022)
- Zhou, P. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. 579 (7798), 270-273 (2020).
- Chen, X., Huang, H., Ju, J., Sun, R., Zhang, J. Impact of vaccination on the COVID-19 pandemic in U.S. states. Scientific Reports. 12 (1), 1554 (2022).
- Stefani, C., Fantoni, T., Bissoli, M., Thomas, J., Ruggiero, A. HIV and SARS-CoV-2 Co-Infection: From Population Study Evidence to In Vitro Studies. Life. 12 (12), 2089 (2022).
- Watson, O. J., et al. Global impact of the first year of COVID-19 vaccination: a mathematical modelling study. The Lancet Infectious Diseases. 22 (9), 1293-1302 (2022).
- Cantoni, D. Analysis of antibody neutralisation activity against SARS-CoV-2 variants and seasonal human coronaviruses NL63, HKU1, and 229E induced by three different COVID-19 vaccine olatforms. Vaccines. 11 (1), 58 (2023).
- Siracusano, G., et al. Different decay of antibody response and VOC sensitivity in naïve and previously infected subjects at 15 weeks following vaccination with BNT162b2. Journal of Translational Medicine. 20 (1), 22 (2022).
- Ruggiero, A. SARS-CoV-2 vaccination elicits unconventional IgM specific responses in naïve and previously COVID-19-infected individuals. eBioMedicine. 77, (2022).
- Piubelli, C. Subjects who developed SARS-CoV-2 specific IgM after vaccination show a longer humoral immunity and a lower frequency of infection. eBioMedicine. 89, 104471 (2023).
- Zhang, G. F. Infectivity of pseudotyped SARS-CoV-2 variants of concern in different human cell types and inhibitory effects of recombinant spike protein and entry-related cellular factors. Journal of Medical Virology. 95 (1), e28437 (2023).
- da Costa, K. A. S. Influenza A (N1-N9) and Influenza B (B/Victoria and B/Yamagata) neuraminidase pseudotypes as tools for pandemic preparedness and improved influenza vaccine design. Vaccines. 10 (9), 1520 (2022).
- Condor Capcha, J. M. Generation of SARS-CoV-2 spike pseudotyped virus for viral entry and neutralization assays: a 1-week protocol. Frontiers in Cardiovascular Medicine. 7, 618651 (2021).
- Diomede, L., et al. Doxycycline inhibition of a pseudotyped virus transduction does not translate to inhibition of SARS-CoV-2 infectivity. Viruses. 13 (9), 1745 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon