È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Sistema di sintesi proteica cell-free per la costruzione di cellule sintetiche
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo descrive il sistema di sintesi proteica senza cellule (CFPS) utilizzato nella costruzione di cellule sintetiche. Delinea le fasi chiave con risultati rappresentativi in diversi micro-compartimenti. Il protocollo mira a stabilire le migliori pratiche per i diversi laboratori della comunità delle cellule sintetiche, facendo avanzare i progressi nello sviluppo delle cellule sintetiche.
Abstract
Il sistema di sintesi proteica senza cellule (CFPS) è stato ampiamente impiegato per facilitare l'assemblaggio dal basso verso l'alto di cellule sintetiche. Funge da ospite per il macchinario centrale del Dogma Centrale, rappresentando un telaio ottimale per l'integrazione e l'assemblaggio di diversi sistemi di mimetismo cellulare artificiale. Nonostante il suo uso frequente nella fabbricazione di celle sintetiche, la creazione di un sistema CFPS robusto e su misura per un'applicazione specifica rimane una sfida non banale. In questo articolo sui metodi, presentiamo un protocollo completo per il sistema CFPS, abitualmente impiegato nella costruzione di cellule sintetiche. Questo protocollo comprende le fasi chiave della preparazione del sistema CFPS, tra cui l'estratto cellulare, la preparazione del modello e l'ottimizzazione dell'espressione di routine utilizzando una proteina reporter fluorescente. Inoltre, mostriamo risultati rappresentativi incapsulando il sistema CFPS all'interno di vari micro-compartimenti, come goccioline monostrato, vescicole a doppia emulsione e camere situate sopra doppi strati lipidici supportati. Infine, chiariamo i passaggi critici e le condizioni necessarie per il corretto assemblaggio di questi sistemi CFPS in ambienti distinti. Ci aspettiamo che il nostro approccio faciliti la creazione di buone pratiche di lavoro tra i vari laboratori all'interno della comunità delle cellule sintetiche in continua espansione, accelerando così i progressi nel campo dello sviluppo delle cellule sintetiche.
Introduzione
La sintesi di cellule sintetiche o artificiali è emersa come un campo di ricerca interdisciplinare molto importante, attirando un notevole interesse da parte di scienziati nei settori della biologia sintetica, della chimica e della biofisica. Questi scienziati sono uniti dall'obiettivo comune di costruire una cellula vivente minima 1,2,3. La rapida crescita di questo campo è andata di pari passo con i progressi significativi nelle tecnologie critiche, come la manipolazione del DNA ricombinante4, i materiali biomimetici5 e le tecniche di microfabbricazione per la compartimentazione6, incluso il metodo di sintesi proteica libera da cellule (CFPS)7. I sistemi CFPS comprendono il macchinario cellulare essenziale per la trascrizione e la traduzione, fornendo il quadro di base per lo sviluppo e l'integrazione di cellule artificiali multifunzionali.
Sebbene le tecniche CFPS siano spesso utilizzate nell'assemblaggio di celle sintetiche, lo sviluppo di un sistema CFPS robusto e su misura per l'assemblaggio di vari sistemi di celle sintetiche rimane una sfida complessa. Attualmente, sono disponibili numerosi sistemi CFPS, derivati sia da organismi modello di procarioti che di eucarioti8, ciascuno specializzato per particolari applicazioni nella sintesi cellulare sintetica. Oltre al loro ruolo centrale nella trascrizione e nella traduzione, i sistemi CFPS variano nei loro componenti principali e nelle procedure di preparazione associate. Queste variazioni, che includono differenze negli estratti cellulari, nelle RNA polimerasi, nei metodi di preparazione dei modelli e nelle composizioni dei tamponi, sono in gran parte dovute alle distinte traiettorie di sviluppo perseguite dai gruppi di ricerca che hanno ottimizzato intensamente i loro sistemi per la massima resa proteica.
Tra i vari componenti del sistema CFPS, l'estratto cellulare è un pool enzimatico critico per la trascrizione e la traduzione, e quindi un determinante chiave delle prestazioni della CFPS9. La CFPS a base di Escherichia coli (E. coli) è il sistema più comunemente utilizzato grazie al suo status di organismo procariotico meglio conosciuto. Inoltre, il gruppo di ricerca di Ueda ha sviluppato un sistema CFPS completamente ricostituito che comprende proteine e ribosomi purificati individualmente, noto come PURE10, particolarmente adatto per applicazioni che richiedono un background chiaro. Oggi, anche i sistemi CFPS basati su E. coli si sono diversificati, soprattutto in termini di ceppi sorgente per l'extrac11 e i metodi di preparazione12,13, RNA polimerasi14,15, fonti di energia16,17 e sistemi tampone18,19. I ceppi più frequentemente utilizzati includono i derivati dei ceppi K12 e B, come A1920, JM10921, BL21 (DE3)22 e Rossetta223, insieme alle loro controparti geneticamente modificate.
Inizialmente, sono stati scelti ceppi di E. coli con ridotte attività di RNasi e proteasi per migliorare la stabilità dell'mRNA e la stabilità delle proteine ricombinanti di nuova sintesi, portando a un aumento delle rese proteiche finali24. Successivamente, gli estratti di E. coli sono stati ingegnerizzati per facilitare specifiche modifiche post-traduzionali, tra cui la glicosilazione25, la fosforilazione26 e la lipidazione27, sono stati sviluppati per ottenere le modifiche post-traduzionali di cui sopra. Inoltre, è stata incorporata una serie di additivi come chaperon molecolari28 e stabilizzanti chimici per aiutare il ripiegamento delle proteine bersaglio, contribuendo alla diversificazione dei sistemi CFPS. La batteriofago T7 RNA polimerasi, nota per la sua elevata processività, è prevalentemente impiegata per la trascrizione, sebbene siano state utilizzate anche altre polimerasi come SP629. L'RNA polimerasi endogena di E. coli è stata adattata per la prototipazione di circuiti genetici sfruttando i fattori sigma30. Infine, una varietà di precursori energetici 31,32,33 e diversi sali e componenti tampone 19,34,35 sono stati sistematicamente ottimizzati per migliorare la produttività.
Oltre al sistema CFPS stesso, anche i metodi di incapsulamento e i materiali di compartimentazione sono vitali per il successo dell'assemblaggio di celle sintetiche. Vari sistemi che sono stati sviluppati per incapsulare con successo la reazione CFPS includono goccioline di acqua/olio stabilizzate con tensioattivo, lipidi/polimero e le loro vescicole unilamellari ibride (con diametri che vanno da 50 nm a diversi μm), nonché doppi strati lipidici supportati da planari. Tuttavia, a causa del contenuto di molecole complessate del sistema CFPS, il tasso di successo dell'incapsulamento dipende da casi specifici, in particolare per la formazione di vescicole. Per migliorare il tasso di successo e l'efficienza dell'incapsulamento della CFPS, sono stati sviluppati vari chip di microfluidi per facilitare la formazione di goccioline e vescicole36. Tuttavia, sarà necessario stabilire chip e dispositivi aggiuntivi.
Questo protocollo delinea un sistema CFPS di E. coli che utilizza il ceppo BL21(DE3), che è un ospite comunemente impiegato per la produzione di proteine ricombinanti. Il protocollo comprende un resoconto dettagliato della preparazione dell'estratto cellulare, della preparazione del modello e dell'ottimizzazione dell'espressione standard utilizzando una proteina reporter fluorescente. Inoltre, presentiamo risultati esemplari ottenuti incapsulando il sistema CFPS all'interno di diversi micro-compartimenti, tra cui goccioline monostrato, vescicole a doppia emulsione e camere situate sopra doppi strati lipidici supportati. Infine, vengono illustrati gli elementi procedurali fondamentali e le condizioni necessarie per il successo dell'istituzione di questi sistemi di PCPS in contesti ambientali distinti.
Protocollo
1. Preparazione dell'estratto
- Strisciare il ceppo di E. coli BL21 (DE3) da uno stock di glicerolo su una piastra di agar Luria Bertani (LB) e incubare per almeno 15 ore a 37 °C.
- Preparare una precoltura notturna inoculando una singola colonia dalla piastra LB appena preparata in un pallone da 50 ml di terreno Luria Bertani (LB).
- Inoculare 5 mL di precoltura in 500 mL di terreno 2xYTPG in un pallone di Erlenmeyer da 3 L sconcertato. Coltivatela a 37 °C agitandola vigorosamente (tra 220 giri/min e 250 giri/min) e raccogliete le cellule quando raggiungono la fase di metà log (OD600 ~ 3). Quindi, immergere le colture cellulari in acqua fredda e ghiacciata per 10 minuti e centrifugare a 8.000 × g per 15 minuti a 4 °C.
NOTA: In questa fase, il glucosio nel terreno 2xYTPG può essere omesso per applicazioni specifiche 9,12. - Risospendere i pellet di cellule risultanti in 35 mL di tampone S30 A preraffreddato, quindi centrifugare a 8.000 × g per 15 minuti a 4 °C. Scartare il surnatante.
- Ripetere il passaggio 1.4 due volte.
NOTA: I pellet della cella possono essere conservati a -80 °C se non vengono utilizzati immediatamente. - Risospendere i pellet finali nel tampone S30 B (ad esempio, utilizzare 1,1 mL di tampone S30 B per 1 g di pellet cellulare) e disgregare le celle con un passaggio attraverso la French Press a 17.000 psi.
- Per l'utilizzo della French Press, seguire il manuale dell'utente e trasferire la sospensione della cella nella camera di disgregazione metallica della French Press, assicurandosi che tutti i componenti siano assemblati e che la valvola inferiore della camera di disgregazione sia chiusa.
- Trasferire la camera di disgregazione assemblata sulla piattaforma idraulica e fissare il blocco di sicurezza.
- Accendere la pompa idraulica e avviare l'interruzione.
- Controllare la valvola di uscita per consentire alla sospensione cellulare di defluire dal tubo di uscita in una nuova provetta da 50 mL. Regola la velocità del flusso per garantire un'interruzione efficiente, idealmente una goccia alla volta. Mantenere la pressione al di sopra del limite inferiore di 15.000 psi.
- Centrifugare il lisato risultante a 30.000 × g per 30 minuti a 4 °C e raccogliere il surnatante. Ripetere la centrifugazione una volta e raccogliere il surnatante. Aggiungere 0,3 volumi di tampone di preincubazione nel surnatante raccolto e incubare agitando delicatamente a 37 °C per 80 minuti.
NOTA: L'uso del tampone di preincubazione potrebbe aumentare la resa finale, anche se è stato riportato che un run-off vuoto (senza tampone di preincubazione) potrebbe anche migliorare l'efficienza37,38, che dipende dal ceppo sorgente corrispondente. - Dializzare la miscela di deflusso risultante per 2 ore contro 2 L di tampone S30 C, sostituire una volta il tampone di dialisi con 2 L di tampone S30 C fresco e dializzare per una notte a 4 °C39.
NOTA: La seconda fase della dialisi notturna può essere ridotta a circa 3 ore. - Raccogliere il campione dializzato e centrifugare a 30.000 × g per 30 minuti a 4 °C. Raccogliere il surnatante e l'aliquota in volumi appropriati e congelare immediatamente in azoto liquido.
NOTA: Il campione congelato può essere conservato a -80 °C almeno per 6 mesi o 1 anno senza perdita di efficienza.
2. RNA polimerasi T7
- Trasformare il plasmide pAR1219 in cellule competenti di E. coli stella BL21(DE3)40.
- Inoculare 10 ml di una coltura notturna in 1 litro di terreno LB (contenente 100 μg/mL di ampicillina). Far crescere le cellule a 37 °C fino a quando OD600 raggiunge 0,6-0,8.
- Iniziare l'induzione aggiungendo una concentrazione finale di 1 mM IPTG. Indurre le celle per altre 5 ore e raccogliere per centrifugazione a 8.000 × g per 15 minuti a 4 °C. Conservare i pellet di celle a -80 °C per un massimo di diverse settimane.
- Risospendere i pellet cellulari in 30 mL di tampone T7 A e disgregare le cellule con un passaggio attraverso la French Press a 15.000 psi. Rimuovere i detriti cellulari mediante centrifugazione a 20.000 × g per 30 minuti a 4 °C.
- Aggiungere streptomicina solfato goccia a goccia al surnatante del passaggio precedente, raggiungendo una concentrazione finale del 4% (p/v). Dopo una breve incubazione su ghiaccio, centrifugare a 20.000 × g per 30 minuti a 4 °C.
- Filtrare il surnatante attraverso una membrana filtrante da 0,45 μm e caricare il campione filtrato su una colonna a scambio anionico forte (volume della colonna di 40 mL), che è stata prebilanciata con 10 volumi di colonna del tampone T7 B, tramite un sistema cromatografo liquido automatizzato.
- Lavare la colonna caricata con 50 volumi di tampone T7 B ed eluire il campione con 10 volumi di colonne di miscele di tampone C T7 a basso contenuto di sale (50 mM NaCl) e ad alto contenuto di sale (500 mM), stabilendo un gradiente di concentrazione lineare di NaCl da 50 a 500 mM a una velocità di flusso di ~3 mL/min41.
- Raccogli le frazioni di picco e analizzale con SDS-PAGE.
NOTA: La polimerasi T7 mostra una banda predominante a ~100 kDa, mentre quantità significative di impurità appaiono ancora sul gel SDS-PAGE. - Raggruppare le frazioni contenenti T7 polimerasi e dializzare contro 2 L di tampone T7 C durante la notte.
- Aggiungere glicerolo per raggiungere una concentrazione finale del 10% e concentrare la miscela risultante fino a una concentrazione finale di 3-4 mg/mL mediante ultrafiltrazione42.
NOTA: Se durante il processo di concentrazione compaiono precipitazioni, arrestare immediatamente e rimuovere i precipitati mediante centrifugazione. - Regolare la concentrazione di glicerolo al 50%. Aliquotare e congelare il campione con azoto liquido. Conservare a −80 °C per un periodo più lungo.
NOTA: Una piccola aliquota può anche essere conservata a -20 °C per almeno 1 mese senza perdita di efficienza.
3. Preparazione del tampone
- Preparare i tamponi come indicato nella Tabella 1 un giorno prima dell'uso.
4. Progettazione e preparazione del modello
- Clonare il gene di interesse in un vettore basato sul promotore T7 o generare un prodotto di PCR lineare contenente il gene di interesse.
NOTA: Vedere la sezione di discussione per i principi di progettazione. - Preparare il plasmide da una coltura notturna utilizzando un kit di estrazione del plasmide.
NOTA: Si consiglia di selezionare un kit plasmidico che includa una fase di precipitazione dell'isopropanolo seguita da una procedura di lavaggio con etanolo al 70%. - Sciogliere il DNA essiccato in un piccolo volume di acqua ultrapura a una concentrazione compresa tra 200 μg/mL e 500 μg/mL, come determinato da uno spettrofotometro a micro volume.
- Opzionale: Esprimere direttamente le proteine dai prodotti di PCR lineare.
NOTA: In questo caso, è necessaria una fase di purificazione PCR.
5. Ottimizzazione di Mg2+ e K+
- Preparare una miscela master per lo screening della concentrazione di Mg2+ come indicato nella tabella 2 o per lo screening della concentrazione di K+ come indicato nella tabella 3.
- Trasferire la miscela master in provette per microfusibili individuali o in una piastra a 96 pozzetti a forma di V.
- Pipettare il volume esatto delle soluzioni madre di Mg2+ o K+ in provette per microfusi individuali o nella piastra a 96 pozzetti a forma di V e completare le reazioni CFPS con acqua ultrapura. Incubare le reazioni per almeno 2 ore.
NOTA: Se si utilizza una piastra a 96 pozzetti a forma di V, sigillare la piastra con un coperchio di plastica per evitare l'evaporazione. - Prelevare 2 μl della miscela di reazione e trasferirla in una piastra nera a 96 pozzetti per le misure di fluorescenza con un lettore di piastre.
NOTA: Utilizziamo un lettore di piastre fluorescenti con la seguente configurazione: eccitazione/emissione: 485/528, guadagno: 50, altezza di lettura: 7,00 mm. Tuttavia, è necessario regolare il lettore di piastre per generare una curva di calibrazione specifica utilizzando proteine fluorescenti purificate.
6. Incapsulamento
- Gocciolina
- Preparare un olio fluorurato contenente tensioattivo (2% PFPE-PEG in HFE7500) o una soluzione di olio lipidico-minerale sciogliendo il lipide in olio minerale (fare riferimento al passaggio 6.2.1)
- Preparare un volume totale di 100 μl di reazione CFPS combinando i corrispondenti reagenti 2-18 della Tabella 4. Aggiungere la reazione CFPS a 500 μl di olio precedentemente preparato in una provetta da 1,5 mL, quindi strofinare energicamente la provetta sulla rastrelliera per provette 50 volte per formare goccioline fini (acqua nell'olio). Incubare la provetta a 30 °C per eseguire la reazione.
NOTA: Semplici chip microfluidici potrebbero anche essere utilizzati per generare goccioline omogenee43.
- Vescicole unilamellari giganti (GUV)
- Preparazione della soluzione di olio lipidico-minerale
- Aggiungere 57 μL di cloroformio in un flaconcino di vetro da 4 mL e quindi aggiungere 18 μL di lipidi 25 mg/mL di 1-palmitoil-2-oleoil-glicerolo-3-fosfocolina (POPC) per formare la soluzione lipidica di cloroformio, raggiungendo una concentrazione finale di 8 mM (utilizzare altri lipidi con concentrazioni finali simili).
NOTA: Questa fase di miscelazione ha lo scopo di garantire una miscelazione omogenea di diversi lipidi. - Far evaporare il cloroformio sotto un flusso di argon per 15 minuti, quindi evaporare ulteriormente sotto vuoto per 1 ora.
- Sciogliere i lipidi secchi risultanti in 1.500 μL di olio minerale, raggiungendo una concentrazione finale di 400 μM. Incubare la miscela per una notte a temperatura ambiente.
- Aggiungere 57 μL di cloroformio in un flaconcino di vetro da 4 mL e quindi aggiungere 18 μL di lipidi 25 mg/mL di 1-palmitoil-2-oleoil-glicerolo-3-fosfocolina (POPC) per formare la soluzione lipidica di cloroformio, raggiungendo una concentrazione finale di 8 mM (utilizzare altri lipidi con concentrazioni finali simili).
- Formazione di uno strato lipidico interfacciale
- Aggiungere 500 μl della soluzione esterna (vedere Tabella 5) in una provetta da 1,5 mL e sovrapporre lentamente 250 μl di soluzione di olio lipidico-minerale sopra la soluzione esterna. Incubare a temperatura ambiente per 30 minuti per formare uno strato lipidico interfacciale stabile.
- Preparazione della soluzione interna
- Mescolare tutti i reagenti della Tabella 6 per formare la soluzione interna e tenerla in ghiaccio fino all'uso. Completare la soluzione interna aggiungendo T7RNAP, estratto S30 e stampo di DNA nella soluzione preinterna come reagenti 16-18 elencati nella Tabella 4.
NOTA: L'aumento della concentrazione di saccarosio incapsulato al di sopra di 250 mM potrebbe inibire la traduzione senza cellule44.
- Mescolare tutti i reagenti della Tabella 6 per formare la soluzione interna e tenerla in ghiaccio fino all'uso. Completare la soluzione interna aggiungendo T7RNAP, estratto S30 e stampo di DNA nella soluzione preinterna come reagenti 16-18 elencati nella Tabella 4.
- Formazione di GUV
- Aggiungere 50 μl di soluzione interna a una nuova provetta da 1,5 mL contenente 500 μl di olio lipidico-minerale, pipettare rapidamente su e giù e agitare energicamente.
- Lasciare la provetta in ghiaccio per 10 minuti e aggiungere lentamente 20 μl della miscela di emulsione alla parte superiore della fase oleosa nella provetta da 1,5 mL del passaggio 6.2.2.
- Centrifugare a 800 × g (utilizzare una velocità di centrifugazione compresa tra 100 × g e 1000 × g) per 10 minuti a 4 °C e osservare la formazione di GUV sul fondo della provetta.
NOTA: Potrebbe essere utilizzata una velocità di centrifugazione specifica più elevata; tuttavia, si potrebbe considerare che la velocità di centrifugazione potrebbe influenzare la dimensione dei GUV formati45. - Rimuovere la fase superiore dell'olio.
- Aspirare con cura 30 μl di GUV sul fondo della provetta. Incubare i GUV raccolti a 30 °C.
NOTA: La temperatura di incubazione ottimale varia a seconda delle proteine. - Utilizzare la microscopia confocale a scansione laser (LSM) per monitorare l'espressione delle proteine fluorescenti.
- Preparazione della soluzione di olio lipidico-minerale
- Doppio strato lipidico supportato (SLB)
- Preparazione delle camere SLB
- Piranha-clean 24 x 24 mm #1.5 vetrini coprioggetti aggiungendo sette gocce di acido solforico e due gocce di perossido di idrogeno al 50% al centro di ciascun vetrino di copertura. Incubare i reagenti sui vetrini coprioggetti per almeno 45 minuti; Sciacquare abbondantemente con acqua ultrapura.
- Assemblare la camera di ricostituzione applicando una provetta per microfuge da 0,5 mL sui vetrini coprioggetti puliti utilizzando colla ottica polimerizzata sotto una lampada a raggi ultravioletti (365 nm) per 10 minuti per formare una camera di reazione.
- Formazione di SLB
- Sciogliere l'80 mol% di 1,2-dioleoil-sn-glicerolo-3-fosfocolina (DOPC), il 19,95 mol% di 1,2-dioleoil-sn-glicerolo-3-fosfo-L-serina (DOPS) e lo 0,05 mol% di Atto488-DOPE (1,2-dioleoyl-sn-glicero-3-fosfoetanolammina = DOPE), mescolare in cloroformio, asciugare con un flusso di azoto delicato e aspirare per 1 ora per rimuovere completamente il solvente.
- Reidratare il film lipidico essiccato nel tampone SLB A, ottenendo una concentrazione lipidica finale di 4 mg/mL.
- Vorticare e sonicare i campioni risultanti fino a quando non diventano chiari.
- Diluire 4 mg/mL di SUV con 130 μL di tampone SLB A, trasferire 75 μL della sospensione nelle camere di reazione preformate e incubarla su un blocco termico a 37 °C per 1 minuto. Aggiungere 150 μl di tampone SLB A alla camera di reazione per un'ulteriore incubazione per 2 minuti.
- Lavare la camera con 2 mL di tampone SLB B (tampone SLB A senza MgCl2) prima di sostituire il tampone S30 C con BSA allo 0,4% (p/v) per le reazioni CFPS, lasciando 100 μL di tampone all'interno della camera per evitare che l'SLB formato si secchi.
- Rimuovere il tampone S30 C residuo e aggiungere con cautela la miscela di reazione CFPS nella camera SLB. Incubare la camera a 30 °C e monitorare l'espressione al microscopio confocale a scansione laser.
- Preparazione delle camere SLB
Risultati
Per ogni nuovo lotto di estratto cellulare e di RNA polimerasi T7, si raccomanda di eseguire uno screening di base delle concentrazioni di Mg2+ e K+ per garantire le prestazioni ottimali del sistema CFPS. La fluorescenza della supercartella GFP può fungere da indicatore della resa complessiva del sistema CFPS in condizioni variabili, come illustrato nella Figura 1A, B. Inoltre, nella Figura 1C
Discussione
Questo manoscritto delinea un sistema di sintesi proteica libera da cellule (CFPS) modificato progettato per l'uso in vari micro-compartimenti attraverso piattaforme cellulari sintetiche, tra cui goccioline di acqua in olio, GUV e SLB. Abbiamo utilizzato il ceppo ospite standard di espressione proteica ricombinante di E. coli, BL21(DE3), come estratto di origine per la costruzione di sistemi cellulari sintetici incentrati sulle proteine. Questo approccio ha prodotto circa 0,5 mg...
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti noti o relazioni personali che possano aver influenzato il lavoro riportato in questo articolo.
Riconoscimenti
M. Y. riconosce il finanziamento del Postgraduate Research & Practice Innovation Program della provincia di Jiangsu, Cina (Grant No. KYCX22_2803). L.K. è grato per il sostegno della ricerca sulle scienze naturali degli istituti di istruzione superiore di Jiangsu in Cina, Cina (sovvenzione n. 17KJB180003), della Fondazione per le scienze naturali dell'Università normale di Jiangsu, Cina (sovvenzione n. 17XLR037), dello sviluppo del programma accademico prioritario degli istituti di istruzione superiore di Jiangsu, in Cina, e del programma di professori appositamente nominati di Jiangsu, in Cina.
Materiali
| Name | Company | Catalog Number | Comments |
| 1,2-dioleoyl-sn-glycero-3-phosphocholine(DOPC) | Avanti | 850375P | |
| 1,2-dioleoyl-sn-glycero-3-phospho-L-serine (sodium salt)(DOPS) | Avanti | 840035P | |
| 1,4 dithiothreitol (DTT) | Sigma-Aldrich | 1.11474 | |
| 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine (POPC) | Avanti | 850457P | |
| 3,5-cyclic AMP (cAMP) | Sigma-Aldrich | A9501 | |
| 50 mL tubes | Eppendorf | Eppendorf Tubes BioBased | |
| 50% hydrogen peroxide | Sigma-Aldrich | 516813 | |
| Acetate | Sigma-Aldrich | A6283 | |
| Agar powder | Sigma-Aldrich | 05040 | |
| Alanin | Sigma-Aldrich | A4349 | |
| Amicon Stirred Cells | MerckMillipore | UFSC05001 | |
| Ammonium acetate (NH4OAc) | Sigma-Aldrich | A7262 | |
| Arginin | Sigma-Aldrich | A4474 | |
| Asparagin | Sigma-Aldrich | A0884 | |
| Aspartat | Sigma-Aldrich | A5474 | |
| ATP | Roche | 11140965001 | |
| Atto 488 DOPE | Sigma-Aldrich | 67335 | |
| Atto 647N DOPE | Sigma-Aldrich | 42247 | |
| Baffled Erlenmeyer flask | Shuniu | 250 mL, 1000mL | |
| Bovine Serum Albumin(BSA) | Roche | 10711454001 | |
| Centrifugetube | Eppendorf | Eppendorf Tubes 3810X | |
| Centrifugetube rack | Eppendorf | 0030119819 | |
| Chemiluminescence and epifluorescence imaging system | Uvitec | Alliance Q9 Advanced | |
| Chloroform | Sigma-Aldrich | 288306 | |
| Confocal Laser Scanning Microscopy (LSM) | ZEISS | LSM 780 | |
| Countess Cell Counting Chamber Slides | Thermo Fisher Scientific | C10283 | |
| Coverslip | Thermo Scientific | Menzel BB02400500A113MNZ0 | |
| creatine kinase (CK) | Roche | 10127566001 | |
| Creatine phosphate (CP) | Sigma-Aldrich | 10621714001 | |
| Culture dish | Huanqiu | 90 mm | |
| Cystein | Sigma-Aldrich | C5360 | |
| Cytidine 5'-triphosphate disodium salt (CTP) | aladdin | C101487 | |
| Dialysis membrane | Spectrum | Standard RC Tubing MWCO: 12-14 kD | |
| E.Z.N.A. Cycle Pure Kit | Omega Bio-Tek | D6492-01 | |
| Electro-Heating Standing-Temperature Cultivator | Yiheng instrument | DHP-9602 | |
| Ethylenediaminetetraacetic acid(EDTA) | Biosharp | 1100027 | |
| Fluorescent plate reader | BioTek | Synergy 2 | |
| Fluorinated oil | Suzhou CChip scientific instrument | 2%HFE7500 | |
| Folinic acid | Sigma-Aldrich | 47612 | |
| French Press | G.Heinemann | HTU-DIGI-Press | |
| Glucose | Sigma-Aldrich | G7021 | |
| Glutamat | Sigma-Aldrich | G5667 | |
| Glutamin | Sigma-Aldrich | G5792 | |
| Glycerol | Sigma-Aldrich | G5516 | |
| Glycin | Sigma-Aldrich | G7126 | |
| Guanosine 5'-triphosphate sodium salt hydrate(GTP) | Roche | 10106399001 | |
| HEPES | Sigma-Aldrich | H3375 | |
| HiPrep Q FF 16/10 | Cytiva | 28936543 | |
| Histidin | Sigma-Aldrich | H6034 | |
| Isoleucin | Sigma-Aldrich | I5281 | |
| Isopropyl-β-D-thiogalactopyranoside (IPTG) | Sigma-Aldrich | I5502 | |
| K2HPO4 | Sigma-Aldrich | P8281 | |
| KH2PO4 | Sigma-Aldrich | P5655 | |
| Leucin | Sigma-Aldrich | L6914 | |
| Lysin | Sigma-Aldrich | L5501 | |
| Magnesium acetate tetrahydrate (Mg(OAc)2 ) | Sigma-Aldrich | M5661 | |
| Magnesium chloride(MgCl2) | Sigma-Aldrich | M2670 | |
| Methionin | Sigma-Aldrich | M8439 | |
| Microcentrifuge | Eppendorf | 5424 R | |
| Mineral oil | Sigma-Aldrich | M5904 | |
| Mini-PROTEAN Tetra Cell Systems | Bio-Rad | 1645050 | |
| Multipurpose Centrifuge | Eppendorf | 5810 R | |
| NaN3 | Sigma-Aldrich | S2002 | |
| Nucleic Acid & Protein UV-Assay Measurements | IMPLEN | NanoPhotometer N60 | |
| NucleoBond Xtra Maxi kit for transfection-grade plasmid DNA | MACHEREY-NAGEL | 740414.5 | |
| Nunc-Immuno MicroWell 96 well polystyrene plates | Sigma-Aldrich | P8616 | |
| PCR Thermal Cycler | Eppendorf | Mastercycler nexus | |
| Peptone | Sigma-Aldrich | 83059 | |
| Phenylalanin | Sigma-Aldrich | P8740 | |
| Phosphoenolpyruvat (PEP) | GLPBIO | GC44635 | |
| PMSF | Sigma-Aldrich | PMSF-RO | |
| Polyethylene glycol 8000 (PEG 8000) | Sigma-Aldrich | 89510 | |
| Potassium Acetate(KOAc) | Sigma-Aldrich | P5708 | |
| Potassium chloride(KCl) | Sigma-Aldrich | P9541 | |
| Potassium glutamate (K-glutamate) | Sigma-Aldrich | G1501 | |
| Potassium hydroxide(KOH) | Sigma-Aldrich | 221473 | |
| Prolin | Sigma-Aldrich | P8865 | |
| Pyruvate kinase (PK) | Sigma-Aldrich | P9136 | |
| Serin | Sigma-Aldrich | S4311 | |
| Shaker | Zhichushakers | ZQZY-AF8 | |
| Sodium chloride(NaCl) | Sigma-Aldrich | S5886 | |
| Sodium hydroxide(NaOH) | Sigma-Aldrich | S5881 | |
| Sucrose | aladdin | S112226 | |
| Sulfuric acid | Sigma-Aldrich | 339741 | |
| Syringe Filters | Jinteng | 0.45 μm | |
| Test tube | Shuniu | 20 mL | |
| TGX FastCast Acrylamide Kit, 12% | Bio-Rad | #1610175 | |
| ThermoMixer | Eppendorf | ThermoMixer C | |
| Threonin | Sigma-Aldrich | T8441 | |
| Tris base | Sigma-Aldrich | V900483 | |
| tRNA | Roche | 10109550001 | |
| Tryptone | Sigma-Aldrich | T7293 | |
| Tryptophan | Sigma-Aldrich | T8941 | |
| Tyrosin | Sigma-Aldrich | T8566 | |
| UTP Trisodium salt (UTP) | aladdin | U100365 | |
| Vacuum Pump with Circulated Water System | Zhengzhou Greatwall Scientific Industrial and Trade Co.Ltd | SHB-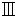 | |
| Valin | Sigma-Aldrich | V4638 | |
| Vortex Mixers | Kylin-Bell | Vortex QL-861 | |
| Water purification system | MerckMillipore | Direct ultrapure water (Type 1) | |
| Yeast extract | Sigma-Aldrich | 70161 | |
| β-mercaptoethanol | Sigma-Aldrich | 444203 |
Riferimenti
- Elani, Y. Interfacing living and synthetic cells as an emerging frontier in synthetic biology. Angew Chem Int Ed Engl. 60 (11), 5602-5611 (2021).
- Cho, E., Lu, Y. Compartmentalizing cell-free systems: toward creating life-like artificial cells and beyond. ACS Synth Biol. 9 (11), 2881-2901 (2020).
- Silverman, A. D., Karim, A. S., Jewett, M. C. Cell-free gene expression: an expanded repertoire of applications. Nat Rev Genet. 21 (3), 151-170 (2020).
- Zhang, L., Guo, W., Lu, Y. Advances in cell-free biosensors: principle, mechanism, and applications. Biotechnol J. 15 (9), e2000187 (2020).
- Lu, Y., Allegri, G., Huskens, J. Vesicle-based artificial cells: materials, construction methods and applications. Mater Horiz. 9 (3), 892-907 (2022).
- Fu, Z., Ochsner, M. A., De Hoog, H. P., Tomczak, N., Nallani, M. Multicompartmentalized polymersomes for selective encapsulation of biomacromolecules. Chem Commun (Camb). 47 (10), 2862-2864 (2011).
- Ayoubi-Joshaghani, M. H., et al. Cell-free protein synthesis: the transition from batch reactions to minimal cells and microfluidic devices. Biotechnol Bioeng. 117 (4), 1204-1229 (2020).
- Yue, K., Chen, J. Y., Li, Y. Q., Kai, L. Advancing synthetic biology through cell-free protein synthesis. Comput Struct Biotechnol J. 21, 2899-2908 (2023).
- Kwon, Y. C., Jewett, M. C. High-throughput preparation methods of crude extract for robust cell-free protein synthesis. Sci Rep. 5, 8663 (2015).
- Shimizu, Y., et al. Cell-free translation reconstituted with purified components. Nat Biotechnol. 19 (8), 751-755 (2001).
- Schwarz, D., et al. Preparative scale expression of membrane proteins in Escherichia coli-based continuous exchange cell-free systems. Nat Protoc. 2 (11), 2945-2957 (2007).
- Levine, M. Z., Gregorio, N. E., Jewett, M. C., Watts, K. R., Oza, J. P. Escherichia coli-based cell-free protein synthesis: protocols for a robust, flexible, and accessible platform technology. J Vis Exp. (144), e58882 (2019).
- Cole, S. D., Miklos, A. E., Chiao, A. C., Sun, Z. Z., Lux, M. W. Methodologies for preparation of prokaryotic extracts for cell-free expression systems. Synth Syst Biotechnol. 5 (4), 252-267 (2020).
- Shin, J., Noireaux, V. Efficient cell-free expression with the endogenous E. coli RNA polymerase and sigma factor 70. J Biol Eng. 4, 8 (2010).
- Mcmanus, J. B., Emanuel, P. A., Murray, R. M., Lux, M. W. A method for cost-effective and rapid characterization of engineered T7-based transcription factors by cell-free protein synthesis reveals insights into the regulation of T7 RNA polymerase-driven expression. Arch Biochem Biophys. 674, 108045 (2019).
- Kim, T. W., et al. Prolonged cell-free protein synthesis using dual energy sources: combined use of creatine phosphate and glucose for the efficient supply of ATP and retarded accumulation of phosphate. Biotechnol Bioeng. 97 (6), 1510-1515 (2007).
- Kim, D. M., Swartz, J. R. Regeneration of adenosine triphosphate from glycolytic intermediates for cell-free protein synthesis. Biotechnol Bioeng. 74 (4), 309-316 (2001).
- Liu, Y., Fritz, B. R., Anderson, M. J., Schoborg, J. A., Jewett, M. C. Characterizing and alleviating substrate limitations for improved in vitro ribosome construction. ACS Synth Biol. 4 (4), 454-462 (2015).
- Rasor, B. J., et al. Mechanistic insights into cell-free gene expression through an integrated-omics analysis of extract processing methods. ACS Synth Biol. 12 (2), 405-418 (2023).
- Kim, D. M., Kigawa, T., Choi, C. Y., Yokoyama, S. A highly efficient cell-free protein synthesis system from Escherichia coli. Eur J Biochem. 239 (3), 881-886 (1996).
- Li, J. J., Li, P. X., Liu, Q., Li, J. J., Qi, H. Translation initiation consistency between in vivo and in vitro bacterial protein expression systems. Front Bioeng Biotechnol. 11, 1201580 (2023).
- Shrestha, P., Holland, T. M., Bundy, B. C. Streamlined extract preparation for Escherichia coli-based cell-free protein synthesis by sonication or bead vortex mixing. Biotechniques. 53 (3), 163-174 (2012).
- Sun, Z. Z., et al. Protocols for implementing an Escherichia coli based tx-tl cell-free expression system for synthetic biology. J Vis Exp. (79), e50762 (2013).
- Zubay, G. In vitro synthesis of protein in microbial systems. Annu Rev Genet. 7, 267-287 (1973).
- Jaroentomeechai, T., et al. Single-pot glycoprotein biosynthesis using a cell-free transcription-translation system enriched with glycosylation machinery. Nat Commun. 9 (1), 2686 (2018).
- Katsura, K., et al. Phosphorylated and non-phosphorylated HCK kinase domains produced by cell-free protein expression. Protein Expr Purif. 150, 92-99 (2018).
- Suzuki, T., et al. N-terminal protein modifications in an insect cell-free protein synthesis system and their identification by mass spectrometry. Proteomics. 6 (16), 4486-4495 (2006).
- Chi, H., Wang, X., Li, J., Ren, H., Huang, F. Chaperonin-enhanced Escherichia coli cell-free expression of functional CXCR4. J Biotechnol. 231, 193-200 (2016).
- Endo, Y., Sawasaki, T. High-throughput, genome-scale protein production method based on the wheat germ cell-free expression system. Biotechnol Adv. 21 (8), 695-713 (2003).
- Shin, J., Noireaux, V. An E. coli cell-free expression toolbox: Application to synthetic gene circuits and artificial cells. ACS Synth Biol. 1 (1), 29-41 (2012).
- Jewett, M. C., Swartz, J. R. Mimicking the Escherichia coli cytoplasmic environment activates long-lived and efficient cell-free protein synthesis. Biotechnol Bioeng. 86 (1), 19-26 (2004).
- Ryabova, L. A., Vinokurov, L. M., Shekhovtsova, E. A., Alakhov, Y. B., Spirin, A. S. Acetyl phosphate as an energy source for bacterial cell-free translation systems. Anal Biochem. 226 (1), 184-186 (1995).
- Guzman-Chavez, F., et al. Constructing cell-free expression systems for low-cost access. ACS Synth Biol. 11 (3), 1114-1128 (2022).
- Jewett, M. C., Swartz, J. R. Substrate replenishment extends protein synthesis with an in vitro translation system designed to mimic the cytoplasm. Biotechnol Bioeng. 87 (4), 465-472 (2004).
- Caschera, F., et al. High-throughput optimization cycle of a cell-free ribosome assembly and protein synthesis system. ACS Synth Biol. 7 (12), 2841-2853 (2018).
- Yue, K., et al. Bottom-up synthetic biology using cell-free protein synthesis. Adv Biochem Eng Biotechnol. 185, 1-20 (2023).
- Liu, D. V., Zawada, J. F., Swartz, J. R. Streamlining Escherichia coli S30 extract preparation for economical cell-free protein synthesis. Biotechnol Prog. 21 (2), 460-465 (2005).
- Kim, T. W., et al. Simple procedures for the construction of a robust and cost-effective cell-free protein synthesis system. J Biotechnol. 126 (4), 554-561 (2006).
- McPhie, P. [4] Dialysis. Methods Enzymol. 22, 23-32 (1971).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. J Vis Exp. (6), e253 (2007).
- Kai, L., et al. Systems for the cell-free synthesis of proteins. Methods Mol Biol. 800, 201-225 (2012).
- Lobb, R. J., et al. Optimized exosome isolation protocol for cell culture supernatant and human plasma. J Extracell Vesicles. 4, 27031 (2015).
- Hori, Y., Kantak, C., Murray, R. M., Abate, A. R. Cell-free extract based optimization of biomolecular circuits with droplet microfluidics. Lab Chip. 17 (18), 3037-3042 (2017).
- Takahashi, H., Ogawa, A. Preparation of a millimeter-sized supergiant liposome that allows for efficient, eukaryotic cell-free translation in the interior by spontaneous emulsion transfer. ACS Synth Biol. 9 (7), 1608-1614 (2020).
- Adir, O., et al. Preparing protein producing synthetic cells using cell free bacterial extracts, liposomes and emulsion transfer. J Vis Exp. (158), e60829 (2020).
- Kai, L., Dotsch, V., Kaldenhoff, R., Bernhard, F. Artificial environments for the co-translational stabilization of cell-free expressed proteins. PLoS One. 8 (2), e56637 (2013).
- Kai, L., Sonal, H. T., Schwille, P. Reconstitution of a reversible membrane switch via prenylation by one-pot cell-free expression. ACS Synth Biol. 12 (1), 108-119 (2023).
- Foshag, D., et al. The E. Coli S30 lysate proteome: a prototype for cell-free protein production. N Biotechnol. 40, 245-260 (2018).
- Garenne, D., Beisel, C. L., Noireaux, V. Characterization of the all-E. coli transcription-translation system mytxtl by mass spectrometry. Rapid Commun Mass Spectrom. 33 (11), 1036-1048 (2019).
- Puigbo, P., Guzman, E., Romeu, A., Garcia-Vallve, S. Optimizer: A web server for optimizing the codon usage of DNA sequences. Nucleic Acids Res. 35, W126-W131 (2007).
- Wiegand, D. J., Lee, H. H., Ostrov, N., Church, G. M. Cell-free protein expression using the rapidly growing bacterium vibrio natriegens. J Vis Exp. (145), e59495 (2019).
- Calhoun, K. A., Swartz, J. R. An economical method for cell-free protein synthesis using glucose and nucleoside monophosphates. Biotechnol Prog. 21 (4), 1146-1153 (2005).
- Kim, D. M., Swartz, J. R. Prolonging cell-free protein synthesis with a novel ATP regeneration system. Biotechnol Bioeng. 66 (3), 180-188 (1999).
- Nishimura, K., et al. Cell-free protein synthesis inside giant unilamellar vesicles analyzed by flow cytometry. Langmuir. 28 (22), 8426-8432 (2012).
- Teh, S. Y., Khnouf, R., Fan, H., Lee, A. P. Stable, biocompatible lipid vesicle generation by solvent extraction-based droplet microfluidics. Biomicrofluidics. 5 (4), 044113 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon