このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
古典的なホジキンリンパ腫のリードステルベルグ細胞のフローソーティングとExome配列決定
要約
ここでは、古典的なホジキンリンパ腫(HL)細胞から高品質、全exomeデータを生成するように設計された、フローサイトメトリーによる細胞ソーティングと低入力、次世代ライブラリー構築プロトコルを説明します。
要約
古典的ホジキンリンパ腫のホジキンリード - スターンバーグ細胞は、炎症性リンパ球のバックグラウンド内にまばらに分布しており、典型的には腫瘍質量の1%未満を構成する。バルク腫瘍に由来する物質は、特徴付けに不十分な濃度で腫瘍含量を含む。したがって、8個の抗体を用いた蛍光活性化細胞選別、ならびに側方および前方散乱は、後の研究のために腫瘍から高純度のHRS細胞を迅速に分離および濃縮する方法としてここに記載されている。同時に、exomeシークエンシングの標準プロトコールでは、通常、フローソーティングでも高すぎることが多い100〜1,000 ngの入力DNAが必要であるため、高品質のライブラリー構築プロトコールを最適化して提供しますわずか10 ngの入力DNAからのデータ。この組み合わせは、全eのハイブリダイゼーション捕捉に適した次世代ライブラリーを作製することができるxome baitsまたはより集中したターゲットパネルを必要に応じて選択します。 HRS細胞のある種の配列決定は、健常な腫瘍内のT細胞またはB細胞と比較した場合、突然変異、挿入および欠失、およびコピー数の変化を含む体細胞変化を同定することができる。これらの知見は、HRS細胞の分子生物学を解明し、標的薬物治療の道筋を明らかにする可能性がある。
概要
次世代シークエンシングの結果としての癌ゲノミクスの進歩は、治療標的の同定および多くの血液学的および非血液学的新生物の予後診断において顕著なブレークスルーをもたらした。特定のゲノム改変に基づく新たな個別治療戦略は、多くの腫瘍型で急速に導入されている(参考文献1,2参照)。リンパ腫ゲノミクスの著しい進歩にもかかわらず、古典的ホジキンリンパ腫(CHL)における腫瘍性HRS細胞のゲノムは、過小発現していた。この研究は、反応性微小環境内の腫瘍性HRS細胞の不足により妨げられており、精製HRS細胞集団を単離することは困難である3 。
原発性腫瘍から生存可能なHRS細胞を単離するための方法は、Fromm et al。 図4に示すように 、この方法は、CD30、CD15、CD40、CD95、CD45 CD20、CD5およびCD64からなる8抗体カクテルを使用して、CHL腫瘍懸濁液からHRS細胞を明白に同定する。少なくとも10 7個の細胞(約10mgの組織)からなる腫瘍生検材料からの新鮮または凍結細胞懸濁液から少なくとも1,000個の生存可能なHRS細胞を単離することができる(フローサイトメトリー分析により90%以上であり、 10個の連続した症例のexomeゲノム分析によって少なくとも80%。
私たちは、フローサイトメトリーによる細胞単離技術を改良し、このプロセスを大幅に簡素化し、原発性CHL腫瘍から数千の生存可能なHRS細胞を迅速に単離することを可能にしました7 。本発明者らは、ホジキンリンパ腫の原発例における腫瘍細胞の最初の完全なエキソーム配列であると考えられるものを産生するためにこの技術を利用した。私たちの研究は、個々のCHL症例のゲノムワイドな高スループット研究の可能性を示しており、すでにCHL発症の側面を説明する可能性のある新規なゲノム改変の同定につながっている。
我々はさらに、ハイスループットゲノム研究のために抽出されたDNAを利用するためのパイプラインを開発した。 HRS細胞数が1,000個以下のHRS細胞からの信頼性の高い結果を得るために、アダプターライゲーション効率を高め、DNA断片ライブラリーを作製するための改変された次世代DNAライブラリー作製手順8を開発しました過度の増幅なしに。この方法は、ルーチンの臨床サンプルの分析と、再発性の突然変異および染色体の変化の検出を可能にする。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1.組織の処理と凍結
- リン酸緩衝生理食塩水(PBS)またはロズウェルパークメモリアルインスティチューション(RPMI)培地でリンパ節組織を収集し、収集24時間以内に処理する。摘出したリンパ節組織9を、2%ウシ胎仔血清(FCS)を含むRPMI 10mLを含むペトリ皿に移し、新鮮なメス刃で細かく切り刻む。組織をさらに粉砕/解離させるために、10mLシリンジプランジャーの背面を使用します。
- 液体を100μmセルストレーナーを通して50mLコニカルチューブに移す。ペトリ皿およびフィルターをRPMI 2%FCS 10mLでさらに洗浄する。
- 自動細胞計数器または血球計算器のいずれかを使用して細胞計数を得る。
注:一般に、約5mm 3のCHLリンパ節組織から少なくとも2×10 7個の細胞が予想される。 80%を超える生存率が期待されるが、サンプルによって異なる可能性がある。 - 400 xgで10分間細胞をスピンダウンし、aspira上清をすすぐ。細胞ペレットを含むチューブを氷上に置く。
- 凍結前の凍結媒体。低温凍結培地に2×10 7 / mLの濃度で細胞を再懸濁し、ピペットで再懸濁する。ボルテックスしないでください。懸濁液を氷上で10分間インキュベートする。
- 試料1mL /クライオバイアル(試料を氷上で予め冷却)に等分する。バイアルを-80℃の冷凍庫に1〜2日間移し、再び液体窒素に移す。
細胞分取のための細胞懸濁液の調製
注:各ロットの抗体は、300μAの染色容量で1000万個の細胞を使用して適切に滴定しなければなりません。末梢血は、末梢血にスパイクされたKMH2細胞株が滴定に使用され得るCD30以外のすべての抗体に使用され得る10 。我々は、一般的に、製造業者が推奨する抗体量で始まり、2倍の2倍希釈および1倍の2倍増加(4データ点)抗体滴定の各ロットについて。例えば、製造業者が10μLの量を推奨する場合、2μL、5μL、10μLおよび20μLの容量を使用して滴定を行います。
- 水浴を37℃に設定する。ダークガラスバイアルに抗体の滴定量をプレミックスし、PBS + 2%BSAを加えて100μLの総カクテル量にします。
注:抗体の滴定を推奨しますが、以下の量を出発点として使用することができます:CD64,20μL; CD95,5μL; CD30,20μL; CD5,10μL; CD20,10μL; CD15,20μL; CD40,5μL;およびCD45,10μL。 - バイアルを液体窒素からドライアイスが入った氷バケツに移し、融解を防ぐ。 RPMI / 20%FCS / DNase(100μg/ mL)を含有する解凍培地50mLを、37℃の水浴中の50mLコニカルチューブ中で予熱する。
- 解凍培地45 mLを新鮮なチューブに移し、37℃に保つ。極低温バイアルを37℃のウォーターバットに入れて急速に細胞を解凍する非常に小さい凍結部分のみが残るまで繰り返す。
- バイアルの内容物を、45mLの解凍媒体を含むチューブに注ぎます。空の極低温バイアルを1mLの解凍媒体で2回すすぎ、すすぎ液を合わせる。
- DNase消化および細胞再平衡化を可能にするために、細胞を室温で15分間インキュベートする。細胞を500xgで10分間遠心し、上清を吸引する。
- 約200μLの解凍培地(5mL)に細胞を再懸濁し、室温に2〜3分間平衡化させる。凍結した生細胞の70%超の回収が期待される。
注:必要に応じて、添付のT細胞によってロゼットされるソーティングされたHRS細胞の純度を高めるために、このステップで非標識抗体のカクテルを添加することができます(オプションプロトコルを参照)。 - 100μLの抗体カクテルを添加し、室温(RT)で15分間インキュベートし、光から保護する。選別用培地3 mLを加え、細胞を500 xgでスピンダウンする。10分間インキュベートし、上清を吸引する。
- 選別用培地1 mLに細胞を再懸濁し、5 mLフローチューブトップストレーナに移す。
- 50 mLコニカルチューブと細胞ストレーナーの両方を、さらに1 mLの選別用培地で濯ぎ、細胞を氷上に置きます。
3.(オプションのプロトコル)T Cell Rosette Blocking
注:HRS細胞は、組織切片および細胞懸濁液中のT細胞によってロゼットされ、これらのT細胞は、ソートされたHRS画分を潜在的に汚染する可能性がある。これらの相互作用は、T細胞上のLFA-1およびCD2に結合するHRS細胞上のCD54およびCD58によって媒介される4,11。これらの相互作用は、これらの接着分子に対する非標識抗体でブロックすることができる。
- 100μLのRPMI中に100,000〜500,000個の細胞を分注する。
- 氷上で、CD2、CD54、CD58、およびLFA-1(それぞれ10μL)への非標識抗体と細胞懸濁液を1時間インキュベートする。 T彼は細胞懸濁液を蛍光抗体で標識することができます。
4.細胞選別によるHRS-、B-、T-細胞の単離
注:我々は5つのレーザーを使用した特別な研究オーダー器具(材料スプレッドシートを参照)を使用しましたが、抗体パネルで使用される蛍光色素を検出する能力を持つソーターで十分です。以下のステップを実行するには、ソフトウェア12の機能とセルソーター動作の基本知識に精通している必要があります。詳細な手順については、オンラインソフトウェアマニュアルを参照してください。
- サイトメーター設定:
- コンピュータの電源を入れ、ログインします。 BSC(およびBSCコンセント)の電源を入れ、サイトメータの電源を入れます。サイトメータの内部CPUが起動するのを少なくとも90秒待ってから、レーザー制御ソフトウェアを開き、すべてのレーザーの電源が入っていることを確認します。サイトメーターソフトウェアを起動し、ログインします。。
- サイトメーターソフトウェア内で、「Cytometer→View Configurations」をクリックします。コンフィグレーションサブプログラムのダイアログボックスが表示されたら、130μmのカスタム構成を選択し、[設定の設定]と[OK]をクリックします。構成サブプログラムを終了します。
- サイトメーターソフトウェアのダイアログボックスを見て、 "Use CS&T settings"をクリックしてください。 130μmのノズルを機器に取り付け、ストリームウィンドウの赤い「X」ボタンをクリックしてストリームをオンにします。少なくとも30分間ウォーミングアップしてください。
- 目的の方法を使用して機器の性能チェックを実行します(ここで説明したソーターには性能追従ソフトウェアモジュールが含まれています。ソフトウェアマニュアル(pp。117-122)およびリファレンススタンダードを参照してくださいこの設定)。
- "Cytometer→CST"をクリックし、 "Characterize"フィールドが "Chec"に設定されていることを確認しますk Performance "をクリックし、" Run "をクリックします。ソフトウェアによってプロンプトが表示されたら、リファレンス粒子のチューブをサンプルステージにロードし、「OK」をクリックします。
- 実行が完了したら、[完了]をクリックしてソフトウェアモジュールを閉じます。サイトメーターソフトウェアがサイトメーターに再接続し終わったら、表示されるダイアログボックスの "Use CS&T settings"をクリックします。
- サイトメーターソフトウェアの自動滴下遅延機能を使用して、正しいドロップ遅延を決定します(ソフトウェアマニュアルの154-161ページを参照)。
- サイトメーターソフトウェアの「ブラウザ」ウィンドウで「ドロップディレイ」実験を開き、サンプルステージにキャリブレーション粒子のチューブを取り付け、「アクイジションダッシュボード」ウィンドウで「ロード」をクリックします。 [ストリーム]ウィンドウの[スイートスポット]ボタンをクリックして、自動ストリーム監視機能をオンにします。すべてのフォローインでこの機能を常時オンにしておくgステップ。
- サイドストリームウィンドウの "Voltage"ボタンをクリックして偏向板電圧をオンにし、 "Voltage"ボタンのすぐ隣の "Test Sort"ボタンをクリックしてテストソートをオンにします。
- 左側のストリームを除き、すべてのサイドストリーム設定をゼロに調整します。サイドストリームウィンドウで2つのストリームスポットが表示されるように、左側のストリーム設定を調整します。 [光学フィルタ]ボタンをクリックし、左側のストリームのスポットがサイドストリームウィンドウの黒い領域に表示される左のボックスに入ることを確認します。
- 必要に応じて、左側のストリーム設定を調整します。 「ソートのテスト」ボタンを再度クリックしてテストソートをオフにします。 [ブラウザ]ウィンドウで、[+]をクリックして[ドロップ遅延]実験の[グローバルワークシート]要素を展開します。
- "Sort Layout_001"をダブルクリックしてソートレイアウトを開き、目視検査で確認します左ソート集団に「P1」が割り当てられていることを確認し、「ソートレイアウト」ウィンドウで「ソート」をクリックします。 [レイアウトの並べ替え]ウィンドウで、[自動遅延]をクリックし、[実行]をクリックします。
- 実行が完了したら、[終了]をクリックします。サンプルステージに滅菌脱イオン水のチューブを取り付け、「Acquisition Dashboard」ウィンドウの「Load」をクリックします。先に進む前に、少なくとも5分間水管を動かして残留試験粒子を装置から除去する。
- ソーターソフトウェアの内蔵補正設定を使用して補償コントロールを実行します(詳細は、ソフトウェアマニュアルの131〜137ページを参照してください)。
- [Experiment New Experiment]をクリックして新しい実験を作成します。 "Instrument Status"ウィンドウの "Parameters"タブで、未使用のパラメータがあれば削除します。 [Experiment Compensat]をクリックします。イオンセットアップは補償コントロールを作成します。
- ブラウザのウィンドウで、 "+"記号をクリックして "Compensation Controls"標本を展開します。データを記録せずに補正コントロールチューブを実行し、必要に応じて各蛍光色素の陽性に染色されたビーズ集団がチャンネル10,000〜100,000の間にあるように検出器の電圧を調整します(「機器ステータス」の「パラメータ」タブ)それらの主要検出チャネルにおいて最も明るい。
- 各チューブの各パラメータに必要な電圧を書き留めます。左に1回クリックして "補償コントロール"の下にある "Unstained Control"チューブをハイライトします。無染色のコントロールチューブをサンプルステージにセットし、 "Acquisition Control"ウィンドウの "Load"をクリックします。
- 個々の補償制御をすべてのパラメータの検出器電圧フィールドに実行することによって、決定された電圧を手動で入力します。「Acquisition Control」ウィンドウで「Record」をクリックします。残りの補償コントロールをすべて実行し、検出器の設定を変更せずにデータを記録します。処理する前に、滅菌脱イオン水のチューブを5分間設置し、機器の残留物をすべて除去する。
- HRS細胞ゲーティング:
- 最初のゲーティングのために少なくとも100,000のイベントを取得して記録し、3,000-4,000イベントを取得するために流量を調整します(ステップ4.1を参照)。
注:細胞濃度が高すぎる場合は、細胞を希釈するために追加の選別培地を追加する必要があります。取得を停止します。 - 図1で概説した手順を使用してゲートHRS細胞
注:ほとんどのCHL症例では、0.01%〜0.1%の細胞がHRS細胞になります。
- 最初のゲーティングのために少なくとも100,000のイベントを取得して記録し、3,000-4,000イベントを取得するために流量を調整します(ステップ4.1を参照)。
- B細胞およびT細胞ゲーティング:
- CD20とCD5のゲーティングによって、体細胞コントロール(B細胞とT細胞)を同定し、respe(CD45 / SSHによるゲッティングリンパ球)、その後のCD20対CD5( 図1参照)。
- あらかじめ冷却された2方向または4方向収集ラック内の収集チューブへのターゲット収集ストリーム。流管または15 mL遠心分離管のいずれかを採集培地で少なくとも途中まで満たしてください。
- 仕入先の指示に従ってソート設定でHRSおよびコントロール集団を適切な採取チューブに割り当てます。セルの取得を再開し、並べ替えを開始します。
- すべてのHRS細胞と最大100万のB細胞とT細胞を収集する。あらかじめ冷蔵された4方向(または2方向)収集ラックで収集チューブを収集チューブに向けます。
5. DNA抽出
- ペレットは、1.5mLコニカルチューブで3,000×gで10分間遠心分離して細胞を集め、1mLのPBSで1回再懸濁して細胞を洗浄した。
- 再度ペレットを3,000×gで10分間遠心分離し、上清を除去する。妨害しないように非常に注意する小さなペレット。
- 洗浄した細胞に150μLの溶解バッファー(または使用するキットの適切な容量)を加え、上下にピペッティングして混合します。
注:必要に応じて、この時点で-70℃で細胞ライセートを保存することができます。 - フィルターカラムをチューブの内側に配置してカラムアセンブリを構築し、ステップ5.5のライセートをカラムに加える。アセンブリを13,000 xgで3分間スピンします。
- アセンブリからミニカラムを取り出し、コレクションチューブ内の液体を捨てます。コレクションチューブのミニカラムを交換します。
- 650μLのカラム洗浄液を各アセンブリに添加する。 13,000 x gで1分間遠心分離する。回収チューブから液体を捨てる。この手順を合計4回繰り返す。
- 回収チューブから液体を捨て、ミニカラムアセンブリを再組み立てします。 13,000 xgで2分間遠心分離して結合マトリックスを乾燥させます。
- ミニカラムを新しい1.5mLチューブに移し、25μLの10mM Tris-Clを加える。その後の超音波処理工程のために好ましいか、または65℃に加熱されたヌクレアーゼフリー水である。室温で2分間インキュベートし、13,000×gで1分間遠心分離する。 25μLでもう一度繰り返し、合計50μLとします。
- ピペットで上下に分けてDNA溶出液を混合し、蛍光定量法を用いて定量する14 。
6.図書館の建設
- 開始前の準備:
- 水位12、強度5、サイクル/バースト200、および温度7でソニケーターを設定します。
- 蛍光測定法および実験設計によって決定された利用可能なDNA量に基づいて、ライブラリー構築に使用するDNAの量を決定する。
注記:DNAが少なすぎてライブラリの品質が損なわれた場合、確認のために残されている材料の使用はほとんどない可能性があることに注意してください。標準以下の入力量の場合、入力質量が大きければ大きいほど、得られたシークエンシングライブラリーのレクシー。このプロトコルのガイドラインのいくつかは次のとおりです.10 ngで良好な結果が得られ、50 ngは概算最大値とみなされます。 - アダプターを決定する: 表1の値にゆるやかに基づいてモル比を挿入する。
- 使用するアダプタの量を次のように計算します。
注:DNA入力のモル数、 n
私。 n = 1.54e-12 -12 *入力質量(単位:ng)/(平均フラグメントサイズ)
ii。 平均断片サイズが200bpである場合、これは以下のように単純化される:
n = 7.7e-15 *入力質量(ng)
含まれるアダプターのモル数、 a
私。 a = n * r
アダプターライゲーションステップで使用するアダプターストックの量v
私。 v( μL ) = a / [ 10 -12 * アダプタストック濃度(μM )]
注:アダプタの濃度が低い場合、必要な量のアダプタが含まれていることを確認するために、アダプタ溶液を使用して、水の代わりにエンド修復試薬を希釈することができます。
例:所望のモルアダプター:インサート比15:1およびアダプターストック濃度2μMについて、200bpの平均断片サイズを有する100ngの入力DNAについて、使用するアダプターの推奨容量は5.8μLである。 - インデックス付きバーコードをサンプルに割り当てます。
注:ハイブリダイゼーション反応またはシーケンサーのフローセル上のレーンに一緒にプールされるライブラリーは、重複するインデックスを含んではいけません。
- ライブラリー構築 - DNA切断:
- 使用するDNAの全量を超音波処理チューブに加える。入力DNAのみを含むサンプルの容量が50μL未満の場合は、Buffer EBを50μLまで添加して混合してください。
- 3のためのSonicate0秒。
- チューブを取り外し、マイクロチューブの壁上部からスプレーを採取するのに十分なだけ、ミニ遠心分離機でクイックスピンを行います。
- 合計210秒間の超音波処理のために、合計7回の30秒間の超音波処理セッションについて、ステップ6.2.2〜6.2.3を繰り返す。
注: 210秒をいくつかのセッションに分けて試してみてください。
- ライブラリー構築 - エンド修復、A-テーリングおよびアダプターライゲーション:
注:ライブラリーPCR増幅ステップの前に、サイズ選択を行わないでください。- サンプルDNAを超音波処理した後、エンド修復およびAテーリングについては、製造元の説明書19に従ってください。
- A-テーリングの後、反応中の適切なモル数のアダプター(ステップ6.1.3で計算)を使用してください。アダプター、A-tailed DNA断片、酵素、バッファーを合わせ、20℃で一晩約16時間インキュベートする。
- リブ再構築 - ライブラリー増幅:
- アダプターライゲーション反応のビーズクリーンアップを行います。ビーズをPCR産物に添加した後、室温で5分間待つ。磁気スタンドに置き、液体を除去し、200μLの80%エタノールで2回洗浄し、ビーズを過剰乾燥せずにほとんど乾燥させ、25μLのピペットでDNAをビーズから溶出させるチューブを磁気スタンドに当てたまま、ビーズにヌクレアーゼフリーの水を加えます。
注:ビーズをポリエチレングリコール中に保存することを実験することが可能であり得る
(PEG)を捨てる代わりに、増幅の後まで除去するが、これは試験されていない。 - 50μLのPCRマスターミックスにつき1:1,000希釈のGreen dye0.6μLを加えます。あるいは、機器の適切な容量で、リアルタイムPCR互換の選択色素を使用してください。
- 98℃でPCR反応をプログラムする初期変性のために45秒間、次に変性、アニーリング、および98℃で15秒間、60℃で30秒間、および72℃で30秒間の伸長のサイクルを行うか、または代替物を用いる場合は適切なプログラムを選択するポリメラーゼ酵素。
- 各サイクルで72℃で蛍光データを取得するように設定します。最後の伸長を72℃で1分間プログラムし、続いて4℃で無期限に保持する。補足表の試薬を使用してアダプター結合ライブラリーを増幅するためのPCRプライマーは、Oligo 1、AATGATACGGCGACCACCGAGAおよびOligo 2、CAAGCAGAAGACGGCATACGAG
- 指数関数的増殖期の終了直前に停止するqPCRソフトウェアでリアルタイムに蛍光強度値を観察しながら、上記のPCR条件を用いてライブラリーを増幅する。
- 増幅後、増幅反応の容量の0.8倍のrecovを使用して、標準のビーズクリーンアップ(6.4.1項参照)を行います典型的には、0.8×50 =40μLのビーズである。ビーズをPCR産物に添加し、室温で5分間待つ。
- 磁気スタンド上に置き、液体を除去し、200μLの80%エタノールで2回洗浄し、乾燥したビーズにヌクレアーゼフリーの水を添加することによって溶出しないでほとんどの液体を除去するのに十分な時間ビーズを乾燥させる。
- 蛍光測定を用いて得られたDNAを定量する。サイズのライブラリフラグメントを視覚化する。詳細については、 データ品質管理の期待値に関するセクションを参照してください。
- アダプターライゲーション反応のビーズクリーンアップを行います。ビーズをPCR産物に添加した後、室温で5分間待つ。磁気スタンドに置き、液体を除去し、200μLの80%エタノールで2回洗浄し、ビーズを過剰乾燥せずにほとんど乾燥させ、25μLのピペットでDNAをビーズから溶出させるチューブを磁気スタンドに当てたまま、ビーズにヌクレアーゼフリーの水を加えます。
7.エクソンハイブリダイゼーション
- 4つのライブラリーを個別のアダプターバーコードと組み合わせてください。
注:蛍光測定法による質量およびゲルによるサイズを用いてモル濃度を計算し、次いで、ライブラリーをプールしたライブラリーの合計1,000ng質量に対して等モル量で合わせる。すべての腫瘍 - 正常対を別々のプールに分けるのではなく、一緒に保つことが最善です。 - exomeキャプチャプロトコルを適用する up class = "xref"> 15に設定し、キャプチャクリーンアップ後に8 PCRサイクルを実行します。捕捉ターゲットの他の選択肢も可能である。
8.マルチプレックスシーケンシング
- 配列は、材料スプレッドシート16で参照されているシークエンシングプラットフォーム上の単一のレーンに4つの多重化ライブラリを含む単一のハイブリダイゼーションキャプチャをシーケンスします。
注記:ターゲットの読み取り深度をさらに計画して最適化する上級ユーザーは、別の構成も可能です。
9.分析(必要に応じて代替パイプラインで置換可能)
- スネップと小さなindels:
- Burrows-Wheeler Aligner(BWA) 17または選択可能な代替アルゴリズムを使用して、生データをヒト参照ゲノム、UCSC hg19にマッピングする。 Samtools 18またはPicardを使用して、マッピング品質スコアが20以下で、PCR複製がフィルタリングまたはマークされているref "> 19。
- Strelka 20または選択した変異体呼び出し者を用いたT細胞体細胞対照と比較して、HRS試料中の体細胞性ヌクレオチド変異体および小indelsを検出する。 snpEff 21を適用して、Strelka出力に注釈を付けます。必要に応じて、統合ゲノムビューア(IGV) 22,23を使用して異型遺伝子座を系統的に検査する。
- 番号の変更をコピーする:
- 次のようにして、腫瘍内の正常化された読取りカウントの腫瘍内の正常化された読取りカウントのすべてのexome標的間隔「 i 」について対数変換比「 ltr 」を計算する:
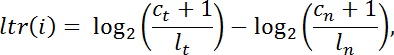
注: cは所与の捕捉間隔にマッピングされる読み取りの数であり、 lは総ライブラリサイズであり、 tは腫瘍を表し、 nは正常を表す。 - 十分なカバレッジ( C t + C n <100読み込み)のインターバルをフィルタリングしてさらに分析します。 RのBioconductorのDNAcopy v.1.0 24を使用してパン間隔の分割を行います。
注:平均ltrの絶対値が0.5未満であるセグメントをコピー中立にすることを検討してください。残りのセグメントは、平均ltrの符号が正の場合(換言すれば、正規化後の正常サンプルよりも腫瘍サンプルの読み込みが有意に多い)、コピー数の増加として指定することができ、平均ltrの値は負である。
- 次のようにして、腫瘍内の正常化された読取りカウントの腫瘍内の正常化された読取りカウントのすべてのexome標的間隔「 i 」について対数変換比「 ltr 」を計算する:
Access restricted. Please log in or start a trial to view this content.
結果
バイオアナライザープロットは、ライブラリー増幅および0.8xビーズクリーンアップの後に行う必要があります。所望の範囲内のフラグメントサイズの「通常のような」分布を見るはずです( 図2a )。曲線の目に見える「肩」のようなこの形状からの逸脱は、高分子量または低分子量の人工物の存在を示す。例えば、 図2b〜2d
Access restricted. Please log in or start a trial to view this content.
ディスカッション
この技術を習得した後のアプリケーションまたは方向
この研究により、少なくとも10ngのDNAを含むサンプルからのexomeシークエンシングが可能になります。臨床的な文脈において、この限界は、不十分な材料のために大部分の細針吸引試料を除外するが、適切な中核生検および切除生検試料を含む。これにより、可能なサンプルのより大きなセットからのデ?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者は何も開示することはない。
謝辞
このプロジェクト方法の開発は、Weill Cornell Medical Collegeの病理学および実験医学部門によって資金提供されました。私たちは、部分的な資金調達のために、計算生物学と医学の3つの機関のトレーニングプログラムを認めています。時間と知識を私たちと共有した科学者、特にMaryke Appelに感謝します。ダンバージェス; Iwanka Kozarewa;チャド・ロックリー; Jenny Zhang、Xiaobo(Shawn)Liang、Dong Xu、Wei Zhang、Huimin Shang、Tatiana Batson、Tuo Zhangを含むWeill Cornell Medical CollegeのGenomics Core Facilityの皆さんです。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Petri or Cell Culture Dish (sterile) | |||
| RPMI-1640 Media | Roswell Park Memorial Institute | ||
| Fetal Calf Serum (FCS), (heat inactivated) | |||
| Freezing Media (RPMI, 20% FCS, 10% dimethylsulfoxide (DMSO))-make fresh and keep sterile | |||
| RPMI with 2% FCS (make fresh or store for up to 1 month) | |||
| scalpel with fresh blade | |||
| 10 mL syringe (no needle) | |||
| Cryogenic vials | |||
| 50 mL conical centrifuge tubes, force | |||
| Centrifuge | capable of handling 50 mL conical centrifuge tubes and providing 400g | ||
| Hepes buffer(1 M, cell culture grade) | |||
| phosphate buffered saline (PBS) | |||
| Pluoronic-F68 | Thermo-Fisher | 24040-032 | |
| DNAase-I | Sigma-Aldrich, St. Louis, MO | D4527-10KU | store as 5 mg/mL in RPMI in -200 °C |
| Bovine Serum Albumin (BSA) | |||
| Sort Media (PBS + 2% BSA + 25 mM HEPES + Pluoronic –F68 (1x)) | |||
| CD64-FITC (22) | Beckman Coulter, Miami, FL | 20 μL suggested starting volume; Titering is suggested | |
| CD30-PE (BerH83) | BD Biosciences, San Jose, CA | 20 μL suggested starting volume; Titering is suggested | |
| CD5-ECD (BL1a) | Beckman Coulter, Miami, FL | 10 μL suggested starting volume; Titering is suggested | |
| CD40-PerCP-eFluor 710 (1C10) | Ebiosciences, San Diego, CA | 5 μL suggested starting volume; Titering is suggested | |
| CD20-PC7 (B9E9) | Beckman Coulter, Miami, FL | 10 μL suggested starting volume; Titering is suggested | |
| CD15-APC (HI98) | BD Biosciences, San Jose, CA | 20 μL suggested starting volume; Titering is suggested | |
| CD45 APC-H7 (2D1) | BD Biosciences, San Jose, CA | Can be substituted with 10 μL suggested volume of CD45-Krome Orange (J.33, Beckman Coulter); Titering is suggested | |
| CD95-Pacific Blue (DX2) | Life Technologies, Grand Island, NY | 5 μL suggested starting volume; Titering is suggested | |
| CD2 (5 μg; clone RPA-2.10) | Biolegend, San Diego, CA | For optional protocol; Titering is suggested | |
| CD54 (10 μg; clone 84H10) | Serotec, Oxford, United Kingdom | For optional protocol; Titering is suggested | |
| CD58 (10 μg; clone TS2/9) | eBioscience, San Diego, CA | For optional protocol; Titering is suggested | |
| LFA-1 (12 μg; clone MHM23) | Novus Biologicals, Littleton, CO | For optional protocol; Titering is suggested | |
| BD CS&T Beads | BD Biosciences, San Jose, CA | ||
| BD Accudrop Beads | BD Biosciences, San Jose, CA | ||
| BC Versa Comp antibody capture beads | Beckman Coulter, Miami, FL | Compensation Beads | |
| BD-FACS ARIA special research order instrument using 5 lasers | BD Biosciences, San Jose, CA | any BD-FACS aria with capabilities to detect the fluorochromes in the antibody panel should be sufficient | |
| Wizard | Promega | A2360 | |
| 10 mM Tris-Cl buffer | NA | ||
| Qubit dsDNA HS Assay kit | Life Technologies, Carlsbad, CA | ||
| S2 Sonicator | Covaris, Woburn, MA | Alternatives may be substituted | |
| microTUBE | Covaris, Woburn, MA | ||
| Low-Throughput Library Preparation Kit | Kapa Biosystems, Wilmington, MA | KK8221 | |
| Sybr Green | Sigma-Aldrich, St. Louis, MO | S9430 | |
| Agencourt AMPure XP Beads | Beckman Coulter, Miami, FL | ||
| Bioanalyzer | Agilent Technologies, Santa Clara, CA | ||
| SeqCap EZ Exome v.3.0 | Roche Nimblegen | 6465684001 | |
| HiSeq | Illumina | ||
| TruSeq-style Universal adapter | Integrated DNA Technologies (IDT), Coralville, Iowa | HPLC purification; AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGAT*C*T | |
| TruSeq-style index adapter | Integrated DNA Technologies (IDT), Coralville, Iowa | HPLC purification; /5Phos/GATCGGAAGAGCACACGTCTGAACTCCAGTCACNNNNNNATCTCGTATGCCGTCTTCTGCTTG | |
| TruSeq-style PCR primer 1 | Integrated DNA Technologies (IDT), Coralville, Iowa | AATGATACGGCGACCACCGAGA | |
| TruSeq-style PCR primer 2 | Integrated DNA Technologies (IDT), Coralville, Iowa | CAAGCAGAAGACGGCATACGAG | |
| Nuclease Free Duplex Buffer | Integrated DNA Technologies (IDT), Coralville, Iowa | ||
| BD FACSDIVA software | BD Biosciences, San Jose, CA | ||
| BD Falcon Tubes | BD Biosciences, San Jose, CA | ||
| BD Flow Tubes | BD Biosciences, San Jose, CA |
参考文献
- Abrams, J. National Cancer Institute's Precision Medicine Initiatives for the new National Clinical Trials Network. American Society of Clinical Oncology educational book / ASCO. American Society of Clinical Oncology. Meeting. , 71-76 (2014).
- Gagan, J., Van Allen, E. M. Next-generation sequencing to guide cancer therapy. Genome medicine. 7, 80(2015).
- Matsuki, E., Younes, A. Lymphomagenesis in Hodgkin lymphoma. Seminars in cancer biology. 34, 14-21 (2015).
- Fromm, J. R., Kussick, S. J., Wood, B. L. Identification and purification of classical Hodgkin cells from lymph nodes by flow cytometry and flow cytometric cell sorting. Am J Clin Pathol. 126 (5), 764-780 (2006).
- Fromm, J. R., Thomas, A., Wood, B. L. Flow cytometry can diagnose classical hodgkin lymphoma in lymph nodes with high sensitivity and specificity. Am J Clin Pathol. 131 (3), 322-332 (2009).
- Roshal, M., Wood, B. L., Fromm, J. R. Flow cytometric detection of the classical hodgkin lymphoma: clinical and research applications. Advances in hematology. 2011, 387034(2011).
- Reichel, J., et al. Flow sorting and exome sequencing reveal the oncogenome of primary Hodgkin and Reed-Sternberg cells. Blood. 125 (7), 1061-1072 (2015).
- Kozarewa, I. A Modified Method for Whole Exome Resequencing from Minimal Amounts of Starting DNA. PLoS ONE. 7 (3), e32617(2012).
- Brunicardi, F. C. Schwartz's Principles of Surgery. , 10th ed, McGraw-Hill Education / Medical. (2014).
- Kantor, A. B., Roederer, M. FACS analysis of leukocytes. Handbook of Experimental Immunology. 2, Blackwell Scientific. (1996).
- Sanders, M. E. Molecular pathways of adhesion in spontaneous rosetting of T-lymphocytes to the Hodgkin's cell line L428. Cancer Res. 48 (1), 37-40 (1988).
- Biosciences. FACSDiva Software v6.0. , Available from: http://www.bdbiosciences.com/in/instruments/software/facsdiva/resources/overview.jsp (2007).
- Biosciences. FACSAria II User's Guide. Part No. 644832, Revision A. , (2009).
- Life Technologies. Qubit 3.0 Fluorometer, Catalog Number Q33216. , Available from: https://www.thermofisher.com/order/catalog/product/Q33216 (2017).
- Roche Nimblegen. SeqCap EZ Library SR User's Guide ver. 4.1. , (2013).
- Illumina. HiSeq High-Throughput Sequencing System. , Available from: http://www.illumina.com/systems/hiseq_2500_1500.html (2016).
- Li, H., Durbin, R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics. 25 (14), 1754-1760 (2009).
- Li, H. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- The Broad Institute. Picard Tools. , Available from: http://broadinstitute.github.io/picard/ (2016).
- Saunders, C. T. Strelka: accurate somatic small-variant calling from sequenced tumor-normal sample pairs. Bioinformatics. 28 (14), 1811-1817 (2012).
- Cingolani, P., et al. A program for annotating and predicting the effects of single nucleotide polymorphisms, SnpEff: SNPs in the genome of Drosophila melanogaster strain w1118; iso-2; iso-3. Fly (Austin). 6 (2), 80-92 (2012).
- Dobin, A. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Thorvaldsdottir, H., Robinson, J. T., Mesirov, J. P. Integrative Genomics Viewer (IGV): high-performance genomics data visualization and exploration. Briefings in bioinformatics. 14 (2), 178-192 (2013).
- DNA copy number data analysis. v.R package version 1.34.0. , Available from: https://omictools.com/dnacopy-tool (2013).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved