需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
经典霍奇金淋巴瘤的芦苇细胞的流分选和外显子测序
摘要
在这里,我们描述了一种组合流式细胞分选和低投入的下一代图书馆建设协议,旨在从古典霍奇金淋巴瘤(CHL)的霍奇金Reed-Sternberg(HRS)细胞产生高质量的全外显子数据。
摘要
经典霍奇金淋巴瘤的霍奇金Reed-Sternberg细胞在炎性淋巴细胞的背景下稀疏分布,通常包含少于肿瘤块的1%。衍生自大块肿瘤的材料含有不足以表征的浓度的肿瘤含量。因此,使用八种抗体以及侧向和前向散射的荧光激活细胞分选在此被描述为一种快速分离和浓缩来自肿瘤的高纯度数千个HRS细胞的方法,用于随后的研究。同时,由于外源序列测序的标准协议通常需要100-1,000 ng的输入DNA,即使使用流分类方法也常常过高,因此我们还提供了优化的低输入库构建协议,能够生产高品质数据少于10 ng的输入DNA。这种组合能够生产适合于全e杂交捕获的下一代文库根据需要,xome诱饵或更专注于目标的面板。当与健康肿瘤T细胞或B细胞比较时,HRS细胞的外显子序列可以鉴定体细胞变异,包括突变,插入和缺失以及拷贝数变化。这些发现阐明HRS细胞的分子生物学,并可能揭示靶向药物治疗的途径。
引言
由于下一代测序,癌症基因组学的进展导致了治疗靶点的鉴定和许多血液学和非血液学肿瘤的预后的重大突破。基于特定基因组改变的新的个体化治疗策略正在迅速引入许多肿瘤类型(参考文献1,2 )。尽管在淋巴瘤基因组学方面取得了显着的进展,经典霍奇金淋巴瘤(CHL)中肿瘤HRS细胞的基因组未被发现。研究受到反应性微环境中肿瘤性HRS细胞稀缺的阻碍,使得难以分离纯化的HRS细胞群体3 。
从原始肿瘤分离可行的HRS细胞的方法由Fromm 等人开发 如图4所示 ,该方法使用由CD30,CD15,CD40,CD95,CD45 CD20,CD5和CD64组成的八抗体混合物,从CHL肿瘤悬浮液中明确鉴定HRS细胞,使用这种方法,我们能够从由至少10 7个细胞(约10毫克组织)组成的肿瘤活组织检查的新鲜或冷冻细胞悬浮液中分离出至少1,000个可行的HRS细胞,通过流式细胞术分析纯度大于90%,估计为至少80%通过exome基因组分析连续十例。
我们已经完善了流式细胞分离技术,大大缓解了该过程,从而能够从原发性CHL肿瘤中快速分离成千上万个可行的HRS细胞。我们已经利用这种技术来产生被认为是霍奇金淋巴瘤初级病例中肿瘤细胞的第一个全部外显子序列。我们的研究证明了这一点高通量,全基因组研究个体CHL病例的可行性,已经导致鉴定新的基因组改变,具有解释CHL发病机理的潜力。
我们进一步开发了一条管道,以利用提取的DNA进行高通量基因组研究。为了从少至1,000个分选的HRS细胞(从顺序病例获得的最小值)获得可靠的结果,我们进一步开发了修改的下一代DNA文库构建程序8 ,其使得我们能够增加衔接子连接效率并产生DNA片段文库没有过多的放大。这种方法允许对常规临床样品的分析和复发性突变和染色体变异的检测7 。
Access restricted. Please log in or start a trial to view this content.
研究方案
组织加工和冷冻
- 收集磷酸盐缓冲盐水(PBS)或罗斯维尔公园纪念研究所培养基(RPMI)中的淋巴结组织,并收集24小时内的过程。将切除的淋巴结组织9转移到含有10mL具有2%胎牛血清(FCS)的RPMI的培养皿中,并用新鲜的手术刀刀片精细切碎。使用10 mL注射器柱塞的背面进一步研磨/解离组织。
- 将液体转移到一个50毫升锥形管通过一个100微米的细胞过滤器。用额外的10 mL RPMI 2%FCS冲洗培养皿和过滤器。
- 使用自动细胞计数器或血细胞计数器获取细胞计数。
注意:一般来说,预计约5毫米3的CHL淋巴结组织至少有2×10 7个细胞。预计有超过80%的可行性,但可能会因样品而异。 - 将细胞以400 x g的速度旋转10分钟,并将其吸收上清液。将含有细胞沉淀的管放在冰上。
- 在冰上预冷冻冷冻培养基。将细胞以2×10 7 / mL的浓度重悬于冷冻培养基中,并通过移液重悬。不要旋涡将悬浮液在冰上孵育10分钟。
- 将样品1 mL /冷冻小瓶(冰上预冷)等分。将小瓶转移到-80°C冷冻箱中1-2天,然后再次将其转移到液氮中。
2.准备细胞分选细胞悬液
注意:每批抗体必须使用1000微升染色体积中的1000万个细胞进行适当滴定。外周血可用于除了CD30之外的所有抗体,其中加入外周血的KMH2细胞系可用于滴定10 。我们通常从制造商推荐的抗体体积开始,并进行两次两倍稀释和两倍增加(四个数据点)对于每次大量的抗体滴定。例如,如果制造商建议使用10μL体积,我们会使用2,5,10和20μL的体积进行滴定。
- 将水浴设置为37°C。在黑色玻璃小瓶中预混合滴定量的抗体,并加入PBS + 2%BSA,总体积为100μL。
注意:虽然我们建议滴定抗体,但以下体积可能用作起始点:CD64,20μL; CD95,5μL; CD30,20μL; CD5,10μL; CD20,10μL; CD15,20μL; CD40,5μL;和CD45,10μL。 - 将小瓶从液氮转移到含有干冰的冰桶中,以防止融化。在37℃水浴中,在50mL锥形管中预热50ml含RPMI / 20%FCS / DNase(100μg/ mL)的解冻培养基。
- 将45 mL解冻培养基转移到新鲜管中,并保持在37°C。通过在37°C水蝙蝠中保存一个低温小瓶快速解冻细胞h,直到只剩下很小的冻结部分。
- 将小瓶内容物倒入含有45 mL解冻介质的管中。用1 mL解冻培养基冲洗空的低温瓶2次,并混合冲洗液。
- 在室温下孵育细胞15分钟以允许DNA酶消化和细胞重新平衡。将细胞以500xg的速度旋转10分钟并吸出上清液。
- 将细胞重悬在大约200μL的解冻介质中(5mL)中,使其平衡至室温2-3分钟。预计恢复> 70%的冷冻活细胞。
注意:任选地,为了更高纯度的可能被附着的T细胞引起的分选的HRS细胞,可以在该步骤添加未标记抗体的混合物(参见任选的方案)。 - 加入100μL抗体混合液,室温(RT)孵育15 min,避光保存。加入3 mL分选培养基,以500 xg的速度旋转细胞10分钟,并吸出上清液。
- 将细胞重悬于1 mL分选培养基中,并将其转移至5 mL流管顶部过滤器。
- 用另外1 mL分选培养基冲洗50mL锥形管和细胞过滤器,并将细胞置于冰上。
3.(可选协议)T Cell Rosette Blocking
注意:HRS细胞在组织切片和细胞悬浮液中被T细胞吞噬,并且这些T细胞可能潜在地污染分选的HRS级分。这些相互作用由CD54和CD58在T细胞4,11上结合LFA-1和CD2的HRS细胞介导。这些相互作用可以用对这些粘附分子的未标记的抗体来阻断。
- 在100μLRPMI中将100,000至500,000个细胞等分。
- 用冰标记的CD2,CD54,CD58和LFA-1(各10μL)的细胞悬浮液孵育1小时。 Ť他现在可以用荧光抗体标记细胞悬浮液。
4.使用细胞分选的HRS-,B-和T细胞分离
注意:虽然我们使用了一个使用5个激光器的特殊研究订单仪器(参见材料电子表格),但任何能够检测抗体面板中使用的荧光染料的分选机应该是足够的。执行以下步骤需要熟悉软件12功能和细胞分选器操作的基本知识。有关详细说明,请参阅在线软件手册。
- 细胞仪设置:
- 打开电脑并登录。打开BSC(和BSC出口)电源,然后打开细胞计数器。等待至少90秒钟,以便启动细胞计数器的内部CPU,然后打开激光控制软件,并验证所有激光器是否通电。启动细胞仪软件13并登录。
- 在细胞仪软件中,点击"Cytometer→查看配置"。当配置子程序的对话框打开时,突出显示130-μm自定义配置,然后单击"设置配置"和"确定"。退出配置子程序。
- 观察细胞仪软件中的对话框,然后单击"使用CS&T设置"。在仪器中安装一个130-μm的喷嘴,点击流窗口中的红色"X"按钮打开流。让仪器预热至少30分钟。
- 使用所需的方法对仪器进行性能检查(本文所述分拣机附带的性能跟踪软件模块,请参见软件手册(第117-122页)和参考标准(参考标准中使用的材料这个设置)。
- 单击"Cytometer→CST",确保"Characterize"字段设置为"Chec"k性能",然后单击"运行"。当软件提示时,将一个参考粒子管加载到样品台上,然后单击"确定"。
- 运行完成后,单击"完成"并关闭软件模块。细胞计数器软件重新连接到细胞仪后,点击出现的对话框中的"使用CS&T设置"。
- 使用细胞仪软件的自动丢失延迟功能确定正确的丢失延迟(请参见软件手册的第154 - 161页)。
- 在细胞仪软件的"浏览器"窗口中打开"延迟延迟"实验,在样品台上安装一组校准颗粒,然后在"采集仪表板"窗口中单击"加载"。点击"流"窗口中的"甜点"按钮打开自动流监视功能。随时关闭此功能g步。
- 通过点击侧流窗口中的"电压"按钮打开偏转板电压,然后点击紧邻"电压"按钮旁边的"测试排序"按钮打开测试排序。
- 将所有侧面流设置调整为零,除了左侧流。调整左侧流设置,使得在侧面流窗口中可以看到两个流点。单击"光学滤波器"按钮,确认左侧流点将落在侧边窗口黑色区域中的左侧框中。
- 如果需要,调整左侧流设置。再次点击"测试排序"按钮关闭测试排序。在"浏览器"窗口中,点击"+",展开"拖延延迟"实验的"全局工作表"元素。
- 双击"排序布局_001"打开排序布局,通过目视检查进行验证离开左排序群体具有"P1",并在"排序布局"窗口中单击"排序"。在"排序布局"窗口中,单击"自动延迟",然后单击"运行"。
- 运行完成后,单击"退出"。在样品台上安装无菌去离子水管,并在"采集仪表板"窗口中单击"加载"。运行水管至少5分钟,以清除仪器中残留的测试颗粒,然后继续。
- 使用分拣机软件中的内置补偿设置运行补偿控制(使用材料电子表格或等效物的补偿珠)(有关其他详细信息,请参见软件手册的第131 - 137页)。
- 点击"实验新实验"创建一个新的实验。在"仪器状态"窗口的"参数"选项卡中,删除未使用的参数(如果有)。点击"实验补偿"离子设置创建补偿控制"。
- 在"浏览器"窗口中,点击"+"标志展开"补偿控制"标本。运行补偿控制管,而不记录数据,如有必要,调整检测器电压(在"仪器状态"窗口的"参数"选项卡中),以便每个荧光染料的正染色珠粒数在10,000至100,000之间,最主要的检测渠道是最亮的。
- 记下每个管的每个参数所需的电压。在"补偿控制"样本下单击左侧单击,突出显示"无污染控制"管。将未染色的控制管装载到样品台上,并在"采集控制"窗口中单击"加载"。
- 通过将各个补偿控制运行到所有参数的检测器电压范围内,手动输入确定的电压然后点击"采集控制"窗口中的"记录"。运行所有剩余的补偿控制,记录数据而不更改任何检测器设置。安装一根无菌的去离子水5分钟,以清除仪器中的任何残余物质,然后再继续操作。
- HRS细胞门控:
- 获取和记录至少100,000个事件用于初始门控,同时调整流量以获得3,000-4,000个事件/秒(参见步骤4.1)。
注意:如果细胞浓度过高,可能需要添加额外的分选培养基来稀释细胞。停止收购。 - 使用图1中概述的步骤门HRS细胞
注意:在大多数CHL病例中,0.01%至0.1%的细胞将是HRS细胞。
- 获取和记录至少100,000个事件用于初始门控,同时调整流量以获得3,000-4,000个事件/秒(参见步骤4.1)。
- B-和T细胞门控:
- 通过CD20和CD5的门控识别体细胞控制(B和T细胞)(通过CD45 / SSH门控淋巴细胞),然后是CD20与CD5(参见图1 )。
- 目标收集流到预冷双向或四向收集架中的收集管。至少一半的采集介质,填充流管或15-mL离心机式管。
- 在按照供应商说明进行的排序设置中,将HRS和控制种群分配到适当的收集管。重新启动细胞采集并开始排序。
- 收集所有HRS细胞和高达100万B和T细胞。将收集流定向到预冷的四通(或双向)收集架中的收集管。
DNA提取
- 通过在1.5mL锥形管中以3,000xg离心10分钟来收集细胞,并用1mL PBS重悬一次以洗涤细胞。
- 再次以3000xg离心10分钟,除去上清液;要小心不要打扰小颗粒。
- 加入150μL裂解缓冲液(或使用的试剂盒的适当体积)到洗涤的细胞中,并通过上下移液混合。
注意:如果需要,可以在-70°C下储存细胞裂解物。 - 通过将过滤柱放置在管内并将溶液从步骤5.5添加到柱中来构建柱组件。以13,000 xg旋转组件3分钟。
- 从组件中取出小柱,并丢弃收集管中的液体。更换收集管中的小柱。
- 向每个装配中加入650μL柱洗涤溶液。以13,000 x g离心1分钟。从收集管中丢弃液体。重复此步骤总共4次洗涤。
- 从收集管中丢弃液体并重新组装小柱组件。以13,000 xg离心2分钟以干燥结合基质。
- 将小柱转移到新的1.5 mL管中,加入25μL10 mM Tris-Cl优选用于随后的超声处理步骤,或加热至65℃的无核酸酶水。在室温下孵育2分钟,并以13,000 xg离心组装1分钟。再次重复25μL,总共50μL。
- 通过上下移液混合DNA洗脱液,并使用荧光测定法14进行定量。
图书馆建设
- 开始前准备:
- 在水位12,强度5,循环/爆破200和温度7下设置超声波发生器。
- 基于通过荧光测定法和实验设计确定的可用DNA量,确定用于文库构建的DNA的量。
注意:请记住,如果DNA的使用太少,并且库的质量受到损害,则为了验证目的留下的材料可能没有什么应用。对于不合标准输入量,输入质量越大,化合物越高所得测序文库的词汇。该协议的一些准则如下:10ng应该产生良好的结果,50ng可能被认为是最大的。 - 确定适配器:将摩尔比基于表1中的数值松散计算。
- 以下列方式计算要使用的适配器数量:
注意:DNA输入的摩尔数, n
一世。 n = 1.54e-12 -12 *输入质量(ng)/(平均碎片大小)
II。 当平均片段大小为200bp时,这简化为:
n = 7.7e-15 *输入质量(ng)
要包括的适配器的摩尔数, a
一世。 a = n * r
适配器库存量在适配器连接步骤中使用, v
一世。 v(以 μL计) = a / [ 10 -12 * 适配器库存浓度(μM )]
注意:如果适配器浓度较低,为确保必需量的适配器,适配器溶液可用于稀释终端修复试剂以代替水。
实施例:对于100ng平均片段大小为200bp的输入DNA,对于所需的摩尔转化子:插入比为15:1,衔接原料浓度为2μM,适配器的推荐体积为5.8μL。 - 将索引的条形码分配给样品。
注意:将汇集到杂交反应或定序器流动池中的通道的库不能包含任何冗余索引。
- 图书馆建设 - DNA剪切:
- 将全部使用的DNA添加到超声处理管中。如果含有单独输入DNA的样品的体积小于50μL,则将缓冲液EB加入总体积为50μL并混合。
- 超声处理30秒。
- 取出管子并在微型离心机中进行快速旋转,足以从微管壁的上部收集任何喷雾。
- 重复步骤6.2.2-6.2.3,总共有三十个超声处理,共超过二十秒超声处理。
注意:随意尝试将210秒分成较少的会话。
- 图书馆建设 - 终端修复,A拖尾和适配器结扎:
注意:避免在库PCR扩增步骤之前进行任何大小选择。- 在样品DNA超声处理后,按照制造商的说明书19进行末端修复和A-tailing。
- A拖尾后,在反应中使用适当数量的适配器(在步骤6.1.3中计算)。将适配器,A尾DNA片段,酶和缓冲液合并,并在20℃下孵育过夜约16小时。
- 库图书馆扩建:
- 对接头连接反应进行珠清洗。向PCR产物中加入珠子后,在室温下等待5分钟。将其放置在磁性支架上并取出液体,在200μL80%乙醇中洗涤两次,将珠子干燥足够长的时间以除去大部分液体,而不会过度干燥,并通过吸取25μL的如所建议的那样,将无核酸酶的水保持在磁珠上,同时保持在磁性支架上。
注意:可能试验保存聚乙二醇中的珠粒
(PEG),直到扩增,而不是丢弃它们,但是这还没有被测试。 - 每50μLPCR主混合物包含0.6μL的1:1,000稀释绿色染料。或者,使用适用于设备的体积中选择的实时PCR兼容的交联染料。
- 在98℃进行PCR反应45s用于初始变性,随后是98℃变性,退火和延伸循环15秒,60℃30秒和72℃30秒的循环,或者如果使用替代物选择适当的程序聚合酶。
- 将机器设置为在每个周期72摄氏度的荧光数据。在72℃下进行1分钟的最终延伸,然后在4℃保持一段无限期的时间。用于在补充表中使用试剂扩增衔接子连接的文库的PCR引物是:Oligo 1,AATGATACGGCGACCACCGAGA和Oligo 2,CAAGCAGAAGACGGCATACGAG
- 使用上述PCR条件扩增文库,同时使用qPCR软件实时观察荧光强度值,在指数生长期结束之前停止。
- 扩增后,使用0.8倍体积的扩增反应进行标准珠清洗(参见步骤6.4.1)通常为0.8×50 =40μL珠。将小珠加入PCR产物,并在室温下等待5分钟。
- 将其放置在磁性支架上并取出液体,在200μL80%乙醇中洗涤两次,将珠子干燥足够长的时间以除去大部分液体,而不会过度干燥,并通过向干珠中加入无核酸酶的水洗脱。
- 使用荧光测定法定量得到的DNA。可视化库片段大小;有关更多详细信息,请参阅Data QC期望部分。
- 对接头连接反应进行珠清洗。向PCR产物中加入珠子后,在室温下等待5分钟。将其放置在磁性支架上并取出液体,在200μL80%乙醇中洗涤两次,将珠子干燥足够长的时间以除去大部分液体,而不会过度干燥,并通过吸取25μL的如所建议的那样,将无核酸酶的水保持在磁珠上,同时保持在磁性支架上。
外来杂交
- 组合四个库与不同的适配器条形码。
注意:通过荧光测定和通过凝胶的大小的质量用于计算摩尔浓度,然后将文库以等摩尔量组合,总共为1,000-ng质量的合并文库。最好将所有肿瘤 - 正常对保持在一起,而不是将它们分离成单独的池。 - 应用exome捕获协议 up class ="xref"> 15,并在捕获清理后进行8个PCR循环。捕获目标的其他选择也许是可能的。
8.多路测序
- 在"材料"电子表格16中引用的测序平台上,在单个泳道中进行单个杂交捕获,包含四个多重文库。
注意:对于希望进一步规划和优化其目标读取覆盖深度的高级用户,可以进行其他配置。
9.分析(如果需要,可以用替代管道替代)
- Sn ps::
- 将原始数据映射到人类参考基因组UCSC hg19,使用Burrows-Wheeler Aligner(BWA) 17或其他选择的算法。过滤或标记读数,映射质量得分低于20,PCR重复使用Samtools 18或Picard参考"> 19。
- 使用Strelka 20或选择的变体呼叫者,与T细胞体细胞对照相比,检测HRS样本中的体细胞核苷酸变体和小indels。应用snpEff 21来注释Strelka输出。如果需要,使用集成基因组查看器(IGV) 22,23系统地检查变体位点的伪像。
- 复制号码变更:
- 按照以下方式计算肿瘤内图书馆内归一化阅读计数的每个外显子目标间隔" i "与正常值的对数转换比率" ltr "
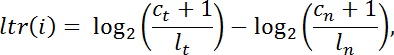
注意: c是映射到给定捕获间隔的读数, l是总库大小, t表示肿瘤, n表示正常。 - 过滤间隔不足( C t + C n <100读)进行进一步分析。使用来自Bioconductor的DNAcopy v.1.0 24进行泛区间分割
注意:考虑平均值ltr的绝对值低于0.5的片段为复制中性。如果平均值ltr的符号为正(换句话说,在标准化后肿瘤样本中的读数大于正常样本中的读数),或者拷贝数丢失,则符号可能被指定为拷贝数增益的平均值ltr为负数。
- 按照以下方式计算肿瘤内图书馆内归一化阅读计数的每个外显子目标间隔" i "与正常值的对数转换比率" ltr "
Access restricted. Please log in or start a trial to view this content.
结果
在文库扩增和0.8倍珠粒清理后应采用生物分析仪。人们应该看到片段大小的"正常"分布在所需的范围内( 图2a )。与该形状的偏差,例如曲线中可见的"肩"表示存在高分子量或低分子量的假象。例如, 图2b -2d显示了包含可理想的未被排序的可见伪像的库的示例。如果图书馆受到严重影响,如果DNA可用和/或细胞分选?...
Access restricted. Please log in or start a trial to view this content.
讨论
掌握此技术后的未来应用或方向
该工作允许从含有至少10ng DNA的样品进行外显子测序。在临床情况下,由于材料不足,此限制不包括大多数细针抽吸样品,但包括足够的核心活检和切除活检样本。这将使得能够从更大的可能样本集中获取数据。
协议中的关键步骤
适当的冷冻和解离技术对于实验的成功至关?...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者没有什么可以披露的。
致谢
该项目方法的开发由威尔康奈尔医学院病理与实验室医学系资助。我们承认计算生物学和医学三部门培训计划的部分资助。我们要感谢与我们分享他们时间和知识的科学家,特别是玛丽亚·安德尔;丹·伯吉斯伊万卡·科扎雷瓦乍得Locklear以及来自威尔康奈尔医学院基因组学核心设施的所有人,包括张珍珍,肖波(肖恩)梁,董旭,魏章,惠民尚,塔蒂亚娜·贝森和托章。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| Petri or Cell Culture Dish (sterile) | |||
| RPMI-1640 Media | Roswell Park Memorial Institute | ||
| Fetal Calf Serum (FCS), (heat inactivated) | |||
| Freezing Media (RPMI, 20% FCS, 10% dimethylsulfoxide (DMSO))-make fresh and keep sterile | |||
| RPMI with 2% FCS (make fresh or store for up to 1 month) | |||
| scalpel with fresh blade | |||
| 10 mL syringe (no needle) | |||
| Cryogenic vials | |||
| 50 mL conical centrifuge tubes, force | |||
| Centrifuge | capable of handling 50 mL conical centrifuge tubes and providing 400g | ||
| Hepes buffer(1 M, cell culture grade) | |||
| phosphate buffered saline (PBS) | |||
| Pluoronic-F68 | Thermo-Fisher | 24040-032 | |
| DNAase-I | Sigma-Aldrich, St. Louis, MO | D4527-10KU | store as 5 mg/mL in RPMI in -200 °C |
| Bovine Serum Albumin (BSA) | |||
| Sort Media (PBS + 2% BSA + 25 mM HEPES + Pluoronic –F68 (1x)) | |||
| CD64-FITC (22) | Beckman Coulter, Miami, FL | 20 μL suggested starting volume; Titering is suggested | |
| CD30-PE (BerH83) | BD Biosciences, San Jose, CA | 20 μL suggested starting volume; Titering is suggested | |
| CD5-ECD (BL1a) | Beckman Coulter, Miami, FL | 10 μL suggested starting volume; Titering is suggested | |
| CD40-PerCP-eFluor 710 (1C10) | Ebiosciences, San Diego, CA | 5 μL suggested starting volume; Titering is suggested | |
| CD20-PC7 (B9E9) | Beckman Coulter, Miami, FL | 10 μL suggested starting volume; Titering is suggested | |
| CD15-APC (HI98) | BD Biosciences, San Jose, CA | 20 μL suggested starting volume; Titering is suggested | |
| CD45 APC-H7 (2D1) | BD Biosciences, San Jose, CA | Can be substituted with 10 μL suggested volume of CD45-Krome Orange (J.33, Beckman Coulter); Titering is suggested | |
| CD95-Pacific Blue (DX2) | Life Technologies, Grand Island, NY | 5 μL suggested starting volume; Titering is suggested | |
| CD2 (5 μg; clone RPA-2.10) | Biolegend, San Diego, CA | For optional protocol; Titering is suggested | |
| CD54 (10 μg; clone 84H10) | Serotec, Oxford, United Kingdom | For optional protocol; Titering is suggested | |
| CD58 (10 μg; clone TS2/9) | eBioscience, San Diego, CA | For optional protocol; Titering is suggested | |
| LFA-1 (12 μg; clone MHM23) | Novus Biologicals, Littleton, CO | For optional protocol; Titering is suggested | |
| BD CS&T Beads | BD Biosciences, San Jose, CA | ||
| BD Accudrop Beads | BD Biosciences, San Jose, CA | ||
| BC Versa Comp antibody capture beads | Beckman Coulter, Miami, FL | Compensation Beads | |
| BD-FACS ARIA special research order instrument using 5 lasers | BD Biosciences, San Jose, CA | any BD-FACS aria with capabilities to detect the fluorochromes in the antibody panel should be sufficient | |
| Wizard | Promega | A2360 | |
| 10 mM Tris-Cl buffer | NA | ||
| Qubit dsDNA HS Assay kit | Life Technologies, Carlsbad, CA | ||
| S2 Sonicator | Covaris, Woburn, MA | Alternatives may be substituted | |
| microTUBE | Covaris, Woburn, MA | ||
| Low-Throughput Library Preparation Kit | Kapa Biosystems, Wilmington, MA | KK8221 | |
| Sybr Green | Sigma-Aldrich, St. Louis, MO | S9430 | |
| Agencourt AMPure XP Beads | Beckman Coulter, Miami, FL | ||
| Bioanalyzer | Agilent Technologies, Santa Clara, CA | ||
| SeqCap EZ Exome v.3.0 | Roche Nimblegen | 6465684001 | |
| HiSeq | Illumina | ||
| TruSeq-style Universal adapter | Integrated DNA Technologies (IDT), Coralville, Iowa | HPLC purification; AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGAT*C*T | |
| TruSeq-style index adapter | Integrated DNA Technologies (IDT), Coralville, Iowa | HPLC purification; /5Phos/GATCGGAAGAGCACACGTCTGAACTCCAGTCACNNNNNNATCTCGTATGCCGTCTTCTGCTTG | |
| TruSeq-style PCR primer 1 | Integrated DNA Technologies (IDT), Coralville, Iowa | AATGATACGGCGACCACCGAGA | |
| TruSeq-style PCR primer 2 | Integrated DNA Technologies (IDT), Coralville, Iowa | CAAGCAGAAGACGGCATACGAG | |
| Nuclease Free Duplex Buffer | Integrated DNA Technologies (IDT), Coralville, Iowa | ||
| BD FACSDIVA software | BD Biosciences, San Jose, CA | ||
| BD Falcon Tubes | BD Biosciences, San Jose, CA | ||
| BD Flow Tubes | BD Biosciences, San Jose, CA |
参考文献
- Abrams, J. National Cancer Institute's Precision Medicine Initiatives for the new National Clinical Trials Network. American Society of Clinical Oncology educational book / ASCO. American Society of Clinical Oncology. Meeting. , 71-76 (2014).
- Gagan, J., Van Allen, E. M. Next-generation sequencing to guide cancer therapy. Genome medicine. 7, 80(2015).
- Matsuki, E., Younes, A. Lymphomagenesis in Hodgkin lymphoma. Seminars in cancer biology. 34, 14-21 (2015).
- Fromm, J. R., Kussick, S. J., Wood, B. L. Identification and purification of classical Hodgkin cells from lymph nodes by flow cytometry and flow cytometric cell sorting. Am J Clin Pathol. 126 (5), 764-780 (2006).
- Fromm, J. R., Thomas, A., Wood, B. L. Flow cytometry can diagnose classical hodgkin lymphoma in lymph nodes with high sensitivity and specificity. Am J Clin Pathol. 131 (3), 322-332 (2009).
- Roshal, M., Wood, B. L., Fromm, J. R. Flow cytometric detection of the classical hodgkin lymphoma: clinical and research applications. Advances in hematology. 2011, 387034(2011).
- Reichel, J., et al. Flow sorting and exome sequencing reveal the oncogenome of primary Hodgkin and Reed-Sternberg cells. Blood. 125 (7), 1061-1072 (2015).
- Kozarewa, I. A Modified Method for Whole Exome Resequencing from Minimal Amounts of Starting DNA. PLoS ONE. 7 (3), e32617(2012).
- Brunicardi, F. C. Schwartz's Principles of Surgery. , 10th ed, McGraw-Hill Education / Medical. (2014).
- Kantor, A. B., Roederer, M. FACS analysis of leukocytes. Handbook of Experimental Immunology. 2, Blackwell Scientific. (1996).
- Sanders, M. E. Molecular pathways of adhesion in spontaneous rosetting of T-lymphocytes to the Hodgkin's cell line L428. Cancer Res. 48 (1), 37-40 (1988).
- Biosciences. FACSDiva Software v6.0. , Available from: http://www.bdbiosciences.com/in/instruments/software/facsdiva/resources/overview.jsp (2007).
- Biosciences. FACSAria II User's Guide. Part No. 644832, Revision A. , (2009).
- Life Technologies. Qubit 3.0 Fluorometer, Catalog Number Q33216. , Available from: https://www.thermofisher.com/order/catalog/product/Q33216 (2017).
- Roche Nimblegen. SeqCap EZ Library SR User's Guide ver. 4.1. , (2013).
- Illumina. HiSeq High-Throughput Sequencing System. , Available from: http://www.illumina.com/systems/hiseq_2500_1500.html (2016).
- Li, H., Durbin, R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics. 25 (14), 1754-1760 (2009).
- Li, H. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- The Broad Institute. Picard Tools. , Available from: http://broadinstitute.github.io/picard/ (2016).
- Saunders, C. T. Strelka: accurate somatic small-variant calling from sequenced tumor-normal sample pairs. Bioinformatics. 28 (14), 1811-1817 (2012).
- Cingolani, P., et al. A program for annotating and predicting the effects of single nucleotide polymorphisms, SnpEff: SNPs in the genome of Drosophila melanogaster strain w1118; iso-2; iso-3. Fly (Austin). 6 (2), 80-92 (2012).
- Dobin, A. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Thorvaldsdottir, H., Robinson, J. T., Mesirov, J. P. Integrative Genomics Viewer (IGV): high-performance genomics data visualization and exploration. Briefings in bioinformatics. 14 (2), 178-192 (2013).
- DNA copy number data analysis. v.R package version 1.34.0. , Available from: https://omictools.com/dnacopy-tool (2013).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。