Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Klasik Hodgkin Lenfomalı Reed-Sternberg Hücrelerinin Akış-Sıralama ve Ekstraksiyon Sıralaması
Bu Makalede
Özet
Burada, klasik Hodgkin lenfoma (HRS) hücrelerinden Hodgkin Reed-Sternberg (HRS) hücrelerinden yüksek kaliteli, tamamen exome veri üretmek üzere tasarlanmış, kombine bir akış sitometrik hücre sıralama ve düşük girişli, yeni nesil kütüphane yapılandırma protokolünü açıklıyoruz.
Özet
Klasik Hodgkin lenfomasının Hodgkin Reed-Sternberg hücreleri, inflamatuvar lenfositlerden oluşan bir arka plan içinde seyrek dağılır ve tipik olarak tümör kütlesinin% 1'inden daha azını oluştururlar. Toplu tümörden türetilen materyal, karakterizasyon için yetersiz konsantrasyonda tümör içeriği içerir. Bu nedenle, sekiz antikorun yanısıra yanal ve öne saçılan floresanla aktive edilmiş hücre ayırımı burada daha sonraki çalışmalarda tümörden yüksek saflıkta binlerce HRS hücresi ile hızlı bir şekilde ayrılma ve konsantrasyon yöntemi olarak tanımlanmaktadır. Aynı zamanda, ekspres sıralama için standart protokoller genellikle 100-1000 ng girdi DNA gerektirir, bu da genellikle akış sıralama ile çok yüksektir, ayrıca yüksek kaliteli üretilebilen optimize edilmiş, düşük girişli bir kütüphane yapım protokolü sağlarız En az 10 ng giriş DNA'sı verileri. Bu kombinasyon, yeni nesil kütüphaneler üretebilir; bu kütüphaneler, bütün eXome yemleri veya daha fazla odaklanmış hedef paneller. HRS hücrelerinin ekspres dizilimi, sağlıklı tümör içi T veya B hücreleriyle karşılaştırıldığında, mutasyonlar, eklemeler ve silmeler ve kopya sayısı değişiklikleri dahil olmak üzere somatik değişiklikleri tanımlayabilir. Bu bulgular, HRS hücrelerinin moleküler biyolojisini aydınlatmakta ve hedefli ilaç tedavilerinin yollarını gösterebilir.
Giriş
Yeni jenerasyon sekanslamanın bir sonucu olarak kanser genomolojisindeki ilerlemeler, birçok hematolojik ve non hematolojik neoplazm için terapötik hedeflerin tanımlanmasında ve prognostikasyonda önemli gelişmelere yol açtı. Spesifik genomik değişikliklere dayalı yeni bireyselleştirilmiş tedavi stratejileri, birçok tümör tipinde hızla ortaya çıkmaktadır (referanslar 1 , 2'de gözden geçirilmiştir). Lenfoma genomiklerinde önemli gelişmelere rağmen, klasik Hodgkin lenfoma (CHL) neoplastik HRS hücrelerinin genomu az açıklanmıştır. Soruşturmalar, reaktif mikro ortamda neoplastik HRS hücrelerinin azlığı nedeniyle engellendi ve saf HRS hücre popülasyonlarının izole edilmesi güçleşti.
Primer tümörlerden canlı HRS hücrelerinin izole edilmesi yöntemi Fromm ve ark. Tarafından geliştirilmiştir . 4 ,Ref "> 5 , 6. Bu yöntem, bir CHL tümör süspansiyonundan kesin olarak HRS hücrelerini tanımlamak için, CD30, CD15, CD40, CD95, CD45 CD20, CD5 ve CD64'den oluşan bir sekiz antikorlu kokteyl kullanmaktadır. En azından 107 hücre (yaklaşık 10 mg doku) içeren tümör biyopsilerinden taze veya dondurulmuş hücre süspansiyonlarından en az 1000 yaşayan HRS hücresini izole edebilmektedir Saflık, akış sitometrik analizle% 90'dan büyüktür ve tahmin edildiği üzere En az% 80, ardışık on vakanın ekzojen genomik analizi.
Birincil CHL tümörlerinden binlerce canlı HRS hücresinin hızla izole edilmesini sağlayan, işlemi büyük ölçüde hafifleten bir akış sitometrik hücre izolasyon tekniğini rafine ettik. Birincil Hodgkin lenfoma vakalarında tümör hücrelerinin ilk tüm eksende serisinin olduğuna inananların üretilmesi için bu tekniği kullandık. Çalışmalarımız,Tekli CHL vakaları için yüksek verimli, genom çapında çalışmaların fizibilitesi ve CHL patogenezinin özelliklerini açıklama potansiyeline sahip yeni genomik değişikliklerin belirlenmesine zaten yol açtı.
Yüksek verimli genomik çalışmalar için çıkarılan DNA'yı kullanmak için bir boru hattı daha geliştirdik. 1.000'e kadar sıralanmış HRS hücrelerinden (en azından ardışık vakalardan elde edilen) güvenilir sonuçlar elde etmek için, adaptör ligasyon verimliliğini arttırmamıza ve DNA fragmanı kütüphaneleri üretmemize izin veren modifiye yeni nesil DNA kütüphanesi oluşturma prosedürü8 daha da geliştirdik Aşırı amplifikasyon olmaksızın. Bu yöntem, rutin klinik örneklerin analizi ve tekrar eden mutasyonların ve kromozomal değişikliklerin saptanmasına imkan verir 7 .
Access restricted. Please log in or start a trial to view this content.
Protokol
1. Doku İşleme ve Dondurma
- Lenf düğümü dokusunu fosfat tamponlu salin (PBS) veya Roswell Park Memorial Enstitüsü ortamı (RPMI) içinde toplayın ve toplama işleminin 24 saati içinde işleme tabi tutun. Çıkarılan lenf nodu dokusunu 9 ,% 2 fetal buzağı serumu (FCS) ile 10 mL RPMI içeren bir Petri kabına aktarın ve taze bir neşter bıçağı ile ince bir şekilde kıyplayın. Dokuyu öğütmek / parçalamak için 10 mL şırınga pistonunun arkasını kullanın.
- Sıvıyı, 100 μm'lik bir hücre süzgeci yoluyla 50 mL'lik bir konik tüpe aktarın. Petri kabını ve filtreyi ilave 10 mL RPMI% 2 FCS ile yıkayın.
- Otomatik bir hücre sayacı veya bir hemositometre kullanarak bir hücre sayısı elde edin.
NOT: Genel olarak, yaklaşık 5 mm3 CHL lenf nodu dokusundan en az 2 x 107 hücre beklenir. % 80'in üzerinde bir canlılık bekleniyor, ancak örnek göre değişebilir. - Hücreleri 400 xg'de 10 dakika döndürün ve aspirasyon yapınSüpernatanı test edin. Hücre pelletini içeren tüpü buz üzerine koyun.
- Buz üzerinde önceden soğutulmuş dondurucu madde. 2 x 10 7 / mL konsantrasyonda soğuk dondurma ortamında hücreleri tekrar süspansiyon ve pipetleme ile tekrar süspansiyon haline getirin. Vorteks etme. Süspansiyonu 10 dakika boyunca buz üzerinde inkübe edin.
- Örnek 1 mL / cryo flakon (önceden buz soğutmalı) alıntılayın. Şişeleri 1-2 gün boyunca -80 ° C derin dondurucuya aktarın ve tekrar sıvı nitrojene aktarın.
2. Hücre Ayırma için Hücre Süspansiyonlarının Hazırlanması
NOT: Her bir antikor lotu, 300 μΛ boyama hacminde 10 milyon hücre kullanılarak titre edilmelidir. Periferik kan CD30 dışındaki tüm antikorlar için kullanılabilir, bunun için titrasyon 10 için periferik kana eklenen KMH2 hücre hattı kullanılabilir. Genellikle üretici tarafından tavsiye edilen antikor hacmi ile başlar ve iki iki kat seyreltme ve iki kat artış (dört veri noktası) gerçekleştiririz.Her bir lot antikor titrasyonu için. Örneğin, üretici 10 μL hacim önerdiğinde, titrasyonu 2, 5, 10 ve 20 μL hacimlerde gerçekleştiririz.
- Bir su banyosu 37 ° C'ye ayarlayın. Titreşimli antikor miktarını karanlık bir cam şişe içerisinde önceden karıştırın ve toplam 100 μL'lik bir kokteyl hacmi için PBS +% 2 BSA ekleyin.
NOT: Antikorların titrasyonlanmasını öneriyoruz, ancak aşağıdaki ciltler bir başlangıç noktası olarak kullanılabilir: CD64, 20 uL; CD95, 5 uL; CD30, 20 uL; CD5, 10 uL; CD20, 10 uL; CD15, 20 uL; CD40, 5 uL; Ve CD45, 10 uL. - Çözünmeyi önlemek için flakonu sıvı nitrojenden kuru buz içeren bir buz kovasına aktarın. 37 mL'lik bir su banyosunda 50 mL'lik konik bir tüpte RPMI /% 20 FCS / DNase (100 μg / mL) içeren 50 mL eritici maddenin önceden ısıtın.
- 45 mL eritme maddesini yeni bir tüpe aktarın ve 37 ° C'de tutun. Hücreleri, 37 ° C'lik su saksısında kriyojenik bir viyal tutarak hızla çözülürSaat kadar çok az donmuş kısım kalır.
- Şişe içeriğini, 45 mL eritici maddenin bulunduğu tüpe boşaltın. Boş kriyojenik flakonu 1 mL eritme maddesi ile 2 kez durulayın ve durulayıcıları birleştirin.
- DNaz sindirimi ve hücre yeniden dengeleme için izin vermek için hücreleri oda sıcaklığında 15 dakika inkübe edin. Hücreleri 500 xg'de 10 dakika döndürün ve süpernatantı aspire edin.
- Çözülen yaklaşık 200 mcL çözülme hücresini (5 mL) tekrar süspanse edin ve 2-3 dakika oda sıcaklığında dengeye getirmesine izin verin. Donmuş canlı hücrelerin>% 70'inin iyileştirilmesi beklenmektedir.
NOT: İsteğe bağlı olarak, bağlı T hücreleri tarafından roze yapılabilen sıralanmış HRS hücrelerinin saflığı için bu aşamada etiketlenmemiş antikorlardan oluşan bir kokteyl eklenebilir (isteğe bağlı protokole bakınız). - Bir antikor kokteyli 100 mcL ekleyin ve ışığa karşı korunan oda sıcaklığında (RT) 15 dakika inkübe edin. 3 mL ayırma aracı ekleyin, hücreleri 500 xg'de döndürün10 dakika boyunca karıştırın ve süpernatanı aspire edin.
- Hücreleri, ayırma ortamının 1 mL'sinde süspanse edin ve bunları bir 5 mL akış tüpü üst süzgeçine aktarın.
- 50 mL konik tüp ve hücre süzgecini, ilave bir 1 mL'lik ayırma aracı ile durulayın ve buz üzerine yerleştirin.
3. (Opsiyonel Protokol) T Hücre Roset Bloklama
NOT: HRS hücreleri, doku kesitleri ve hücre süspansiyonundaki T hücreleri tarafından rozetlenir ve bu T hücreleri, sıralanmış HRS fraksiyonunu potansiyel olarak kirletebilir. Bu etkileşimler, T hücreleri 4 , 11 üzerinde LFA-1 ve CD2'ye bağlanan HRS hücresi üzerindeki CD54 ve CD58 aracılıdır. Bu etkileşimler, bu adezyon moleküllerine yönelik işaretlenmemiş antikorlarla bloke edilebilir.
- RPMI 100 mcL'de 100.000 ila 500.000 hücreye bölün.
- CD2, CD54, CD58 ve LFA-1'e (her biri 10 mcL) etiketlenmemiş antikorlarla hücre süspansiyonunu 1 saat boyunca buz üzerinde buz koyun. THücre süspansiyonu floresan antikorlarla etiketlenebilir.
4. Hücre Ayırma Yöntemi Kullanılarak HRS-, B- ve T-hücresi İzolasyonu
NOT: 5 lazer kullanan özel bir araştırma düzenini kullanmış olsak da (bkz. Malzeme cetveli), antikor panelinde kullanılan florokromları tespit edebilen herhangi bir ayırıcı yeterli olmalıdır. Aşağıdaki adımların uygulanması, yazılım 12 işlevine aşinalık ve hücre sıralayıcı işlemleri hakkında temel bilgi gerektirir. Ayrıntılı talimatlar için lütfen çevrimiçi yazılım kılavuzuna bakın.
- Sitometre kurulumu:
- Bilgisayarı açın ve oturum açın. BSC'ye (ve BSC çıkışlarına) güç verin ve ardından sitometrenin gücünü açın. Başlamak için sitometrenin dahili CPU'su için en az 90 saniye bekleyin ve ardından lazer kontrol yazılımını açın ve tüm lazerlere güç verildiğini doğrulayın. Sitometre yazılımını 13 başlatın ve oturum açın..
- Sitometre yazılımında "Sitometre → Yapılandırmaları Görüntüle" yi tıklayın. Yapılandırma alt programının iletişim kutusu açıldığında, 130 μm özel yapılandırmayı vurgulayın ve "yapılandırmayı ayarla" ve "Tamam" ı tıklayın. Yapılandırma alt programından çıkın.
- Sitometre yazılımındaki diyalog kutusuna dikkat edin ve "CS & T ayarlarını kullan" ı tıklayın. Cihaza 130 μm'lik bir meme takın ve akış penceresindeki kırmızı "X" düğmesini tıklayarak akışı açın. Cihazın en az 30 dakika ısınmasına izin verin.
- İstenilen yöntemi kullanarak cihaz üzerinde bir performans kontrolü yapın (burada açıklanan sıralayıcıya bir performans izleme yazılım modülü dahildir, yazılım kılavuzuna bakın (sf. 117-122) ve referans standardı (bkz. Standartta kullanılan malzemeler) Bu kurulum).
- "Cytometer → CST" i tıklayın, "Characterize" alanının "Chec" olarak ayarlandığından emin olunK Performans "ı tıklayın ve" Çalıştır "ı tıklayın. Yazılım tarafından istendiğinde, örnek partisine bir referans partikülleri tüpü yükleyin ve" Tamam "ı tıklayın.
- İşlem tamamlandıktan sonra, "Son" düğmesine tıklayın ve yazılım modülünü kapatın. Sitometre yazılımı sitometrenin tekrar bağlantısını tamamladıktan sonra, beliren iletişim kutusunda "CS & T ayarlarını kullan" ı tıklayın.
- Sitometre yazılımının otomatik düşme geciktirme özelliğini kullanarak doğru bırakma gecikmesini belirleyin (yazılım kılavuzunun s. 154 - 161'e bakın).
- Sitometre yazılımının "Tarayıcı" penceresindeki "Bırakma Gecikmesi" deneyini açın, numune aşamasında bir kalibrasyon parçacıkları tüpü yerleştirin ve "Edinme Kontrol Paneli" penceresinde "Yükle" yi tıklayın. "Akış" penceresindeki "Tatlı Nokta" düğmesini tıklayarak otomatik akış izleme özelliğini açın. Bu özelliği, takip etmek için her zaman açık bırakmaG adımlar.
- Yan akış penceresindeki "Gerilim" düğmesini tıklayarak saptırma plakası voltajını açın ve ardından "Gerilim" düğmesine hemen bitişikteki "Test Sıralama" düğmesini tıklayarak test sıralama özelliğini açın.
- Sol taraf akışı hariç tüm yan akış ayarlarını sıfıra ayarlayın. Sol taraf akışı ayarını, yan akış penceresinde iki akış noktasının görülebileceği şekilde ayarlayın. "Optik Filtre" düğmesini tıklayın ve sol yan akış spotunun, yan akış penceresinin siyah alanında görünen sol kutunun içine düştüğünü doğrulayın.
- Gerekirse sol yan akım ayarını yapın. "Sıralama Testi" düğmesine tekrar basarak test sıralama özelliğini kapatın. "Tarayıcı" penceresinde, "Bırakma Gecikmesi" denemesinin "Genel Çalışma Yaprakları" öğesini "+." Tıklayarak genişletin.
- Sıralama düzenini açmak için "Düzen Düzeni _001" e çift tıklayın, görsel denetleme ile doğrulayınSol sıralama popülasyonunun "P1" e ait olduğunu ve "Sıralama Düzeni" penceresinde "Sıralama" yı tıkladığını belirtir. "Düzen Düzeni" penceresinde "Otomatik Gecikme" ve ardından "Çalıştır" ı tıklayın.
- İşlem tamamlandığında, "Çıkış" ı tıklayın. Örnek sahnesine steril deiyonize su tüpü takın ve "Edinme Gösterge Tablosu" penceresinde "Yükle" yi tıklayın. Devam etmeden önce aletten kalan test parçacıklarını temizlemek için su tüpünü en az 5 dakika çalıştırın.
- Sıralayıcı yazılımında dahili bir kompanzasyon ayarını kullanarak kompanzasyon kontrollerini (Malzeme elektronik tablosundan veya eşdeğerlerinden kompozisyon boncukları kullanarak) çalıştırın (ek ayrıntılar için yazılım kılavuzunun s. 131 - 137'ye bakınız).
- "Yeni Denemeyi Deney" i tıklayarak yeni bir deneme oluşturun. "Alet Durumu" penceresinin "Parametreler" sekmesinde varsa kullanılmayan parametreleri silin. "Deneme Karşıtlığı" nı tıklayınIyon Ayarı Kompanzasyon Kontrolleri Oluşturun. "
- "Tarayıcı" penceresinde "+" işaretini tıklayarak "Telafi Kontrolleri" örneğini genişletin. Telafi kontrol tüplerini verileri kaydetmeden çalıştırın ve gerekirse dedektör voltajlarını ("Enstrüman Durumu" penceresinin "Parametreler" sekmesinde ayarlayın), böylece her florokrom için pozitif boyanmış boncuk popülasyonları 10.000 ve 100.000 kanal arasında olacak şekilde ayarlayın ve Birincil algılama kanalında en parlak olanı.
- Her bir tüp için her parametre için gereken voltajları yazın. "Tazminat Kontrolleri" örneğinin altında bulunan "Unstained Control" tüpünü, tekli sol tıklama ile vurgulayın. Lekesiz kontrol tüpünü numune aşamasına yerleştirin ve "Satın Alma Kontrolü" penceresindeki "Yükle" yi tıklayın.
- Tüm parametreler için dedektör voltaj alanlarına tek tek kompanzasyon kontrolleri uygulayarak belirlenen voltajları manuel olarak girin veArdından "Edinme Kontrolü" penceresindeki "Kaydet" düğmesini tıklayın. Kalan telafi kontrollerini çalıştırın, dedektör ayarlarını değiştirmeden verileri kaydedin. Devam etmeden önce cihazdaki artık maddeleri temizlemek için 5 dk boyunca steril, deiyonize su tüpü takın.
- HRS hücresi kapısı:
- 3.000-4.000 olay / s edinmek için akış oranını ayarlarken başlangıç kapısı açma işlemi için en az 100.000 olayı edinin ve kaydedin (bkz. Adım 4.1).
NOT: Hücre konsantrasyonu çok yüksekse, hücreleri sulandırmak için ilave sınıflandırma aracı eklemek gerekebilir. Edinimi durdurun. - Gate HRS hücrelerini, Şekil 1'de
NOT: Çoğu CHL vakasında, hücrelerin% 0.01 ila% 0.1'i HRS hücreleri olacaktır.
- 3.000-4.000 olay / s edinmek için akış oranını ayarlarken başlangıç kapısı açma işlemi için en az 100.000 olayı edinin ve kaydedin (bkz. Adım 4.1).
- B ve T hücresi geçidi:
- CD20 ve CD5 kapısı ile somatik kontrolleri (B ve T hücreleri) tanımlayın, respe(Lenfositleri CD45 / SSH ile kapamaktadır), ardından CD20'ye karşı CD20 (bkz. Şekil 1 ).
- Hedef toplama, önceden soğutulmuş iki yönlü veya dört yönlü toplama rafında toplama tüplerine akar. Akış tüplerine veya 15 mL santrifüj tüplerine en az yarım toplama aracı ile doldurun.
- Satıcı talimatlarını takip ederek hRS'yi ve kontrol popülasyonlarını uygun toplama tüplerine atayın. Hücre edinimi yeniden başlatın ve sıralama başlatın.
- Tüm HRS hücrelerini ve 1 milyondan fazla B ve T hücresini toplayın. Toplama akışlarını önceden soğutulmuş dört yönlü (veya iki yönlü) toplama rafında toplama tüplerine hedefleyin.
5. DNA Ekstraksiyonu
- Pellet, 1.5 mL konik tüplerde 10 dakika süreyle 3.000 xg'de santrifüjle hücreleri toplar ve hücreleri yıkamak için 1 mL PBS ile tekrar süspanse edilir.
- 10 dakika 3,000 xg'de bir kez daha peletleyin ve süpernatanı çıkarın; Rahatsız etmemek için çok dikkatli olKüçücük pelet
- Yıkanan hücrelere 150 μL liziz tamponu (veya kit için uygun bir hacim) ilave edin ve pipetleme ile yukarı ve aşağı karıştırın.
NOT: Gerekirse, hücre lizatı, bu noktada -70 ° C'de saklanabilir. - Filtre sütununu tüpün içine yerleştirerek bir kolon düzeneği oluşturun ve adım 5.5'teki lisatı sütuna ekleyin. Montajı 13.000 xg'de 3 dk olarak çevirin.
- Mini sütun grubundan çıkarın ve toplama borusundaki sıvıyı atın. Toplama borusundaki minik sütunu değiştirin.
- Her bir düzene 650 uL kolon yıkama çözeltisi ekleyin. 13.000 x g'de 1 dakika boyunca santrifüjleyin. Toplama tüpündeki sıvıyı atın. Toplam 4 yıkama için bu adımı tekrarlayın.
- Sıvıyı toplama borusundan atın ve minik sütun grubunu tekrar monte edin. Bağlama matrisini kurutmak için 13.000 xg'de 2 dakika boyunca santrifüjleyin.
- Mini kolonu yeni bir 1.5 mL tüp içine aktarın ve 25 mcL 10 mM Tris-Cl ekleyin.Sonraki sonication basamağı için veya 65 ° C'ye ısıtılmış Nuclease-Free Water için tercih edilir. Oda sıcaklığında 2 dakika inkübe edin ve 1 dakika boyunca 13.000 xg'de santrifüjleyin. Toplam 50 mcL için bir kez daha 25 mcL ile tekrarlayın.
- DNA elüsyonunu yukarıya ve aşağıya doğru pipetleyerek karıştırın ve florimetri kullanarak nicelendirin 14 .
6. Kütüphane Yapısı
- Başlamadan önce hazırlama:
- Sonikatörü su seviyesi 12, Yoğunluk 5, Döngüler / patlama 200 ve Sıcaklık 7'de ayarlayın.
- Florimetre ile belirlenen mevcut DNA miktarına ve deneysel tasarıma dayanarak, kütüphane yapımı için kullanılacak DNA miktarını belirleyin.
NOT: Çok az DNA kullanılıyorsa ve kütüphane kalitesi tehlikeye atılırsa, doğrulama amacıyla geride bırakılan materyal için çok az uygulama olabilir. Standart olmayan giriş miktarları için, giriş kütlesi ne kadar büyük olursa, compElde edilen sıralama kitaplığının sözlüğü. Bu protokol için bazı kurallar şunlardır: 10 ng iyi sonuçlar vermelidir ve 50 ng zorlu maksimum kabul edilebilir. - Bağdaştırıcıya karar verin: Molar oranı, Tablo 1'deki değerlere göre gevşekçe yerleştirin.
- Kullanılacak bağdaştırıcı miktarını aşağıdaki biçimde hesaplayın:
NOT: DNA girişi mol sayısı, n
ben. N = 1.54e-12 -12 * giriş kütlesi (inç) / (ortalama parça boyutu)
ii. Ortalama fragman boyutu 200 bp olduğunda, bu basitleştirir:
N = 7.7e-15 * giriş kütlesi (inç)
Dahil edilecek adaptörün mol sayısı, a
ben. A = n * r
Adaptör ligasyon adımında kullanılacak adaptör stok hacmi, v
ben. V ( μL cinsinden ) = a / [ 10-12 * adaptör stoğuKonsantrasyon (μM cinsinden )]
NOT: Adaptör konsantrasyonu düşükse, gerekli miktarda bağdaştırıcının bulunmasını sağlamak için, son tamir reaktiflerini su yerine sulandırmak için adaptör çözeltisi kullanılabilir.
Örnek: Arzulanan bir molar adaptör: insert oranı 15: 1 ve adaptör stok konsantrasyonu 2 μM için ortalama fragman boyutu 200 bp olan 100 ng giriş DNA'sı için önerilen adaptör hacmi 5.8 μL'dir. - Örneklere dizinli barkodlar atayın.
NOT: Bir hibridizasyon reaksiyonuna veya bir sekansörün akış hücresindeki bir şeritte bir araya toplanacak olan kütüphaneler, gereksiz indisler içermemelidir.
- Kütüphane yapımı - DNA makaslama:
- Sonikasyon tüpüne kullanılacak DNA miktarını ekleyin. Yalnızca giriş DNA'sı içeren numune hacmi 50 μL'den düşükse, Tampon EB'yi toplam hacme 50 mcL'ye kadar ekleyin ve karıştırın.
- Sonic için 30 s.
- Tüpü çıkarın ve mini tüp süpürgesinde, mikro tüpün duvarlarının üst kısmından herhangi bir sprey toplayacak kadar çabuk sıkın.
- 6.2.2-6.2.3 arası adımları toplam yirmi saniyelik sonication için yedi 30 saniyelik sonication seansları için tekrarlayın.
NOT : 210 s'lik oturum sayısını daha aza bölmekle çekinmeden deneyin.
- Kütüphane yapımı - Tamir, A-tailing ve adaptör ligasyonu:
NOT: Kitaplık PCR çoğaltma adımından önce herhangi bir boyut seçimi yapmaktan kaçının.- Örnek DNA sonikasyonundan sonra son onarım ve A-kuyruklama için üreticinin talimatlarını izleyin 19 .
- A-tailing'ten sonra, reaksiyonda uygun sayıda mol adaptör (adım 6.1.3'te hesaplanmıştır) kullanın. Adaptörü, A-kuyruklu DNA fragmanlarını, enzimi birleştirin ve yaklaşık 16 saat boyunca 20 ° C'de gece boyunca inkübe edin ve inkübe edin.
- libRary construction - Kütüphane büyütme:
- Adaptör ligasyonu reaksiyonunda boncuk temizleme yapın. PCR ürününe boncuk ekledikten sonra, oda sıcaklığında 5 dakika bekleyin. Manyetik bir stand karşı yerleştirin ve sıvıları çıkarın, 200 μL% 80 etanol 200 μL yıkayın, boncukları aşırı kurutma olmadan sıvının çoğunu çıkarmak için yeterince uzun süre kurutun ve 25 mcL pipetleme ile boncuklardan DNA'yı elute edin Önerilen şekilde nükleaz içermeyen su, tüp bir manyetik standa karşı kalırken boncuklara yapıştırın.
NOT: Boncukların polietilen glikol içinde muhafaza edilmesiyle deney yapılabilir
(PEG), amplifikasyon sonrasına kadar atmak yerine test edildi, ancak bu test edilmemiştir. - PCR master karışımının 50 μL'si başına 0,6 uL, 1: 1000 oranında Green dye ile sulandırınız. Alternatif olarak, ekipman için uygun hacimde gerçek zamanlı PCR ile uyumlu bir ara renk boya kullanın.
- PCR reaksiyonunu 98 ° C'de programlayın.Denatürasyon, tavlama ve 15 saniye süreyle 98 ° C'de, 30 saniye süreyle 60 ° C'de ve 30 saniye boyunca 72 ° C'de uzatma işlemi izledi. Alternatif bir yöntem kullanılıyorsa uygun programı seçin Polimeraz enzim.
- Makineyi, her döngü için 72 ° C'de flüoresan verileri alacak şekilde ayarlayın. Son uzantıyı 72 ° C'de 1 dakika boyunca programlayın, ardından belirsiz bir süre 4 ° C'de tutun. Ek tablodaki reaktifleri kullanarak bağdaştırıcıya bağlanmış kütüphanenin amplifikasyonu için PCR primerleri şunlardır: Oligo 1, AATGATACGGCGACCACCGAGA ve Oligo 2, CAAGCAGAAGACGGCATACGAG
- QPCR yazılımı ile gerçek zamanlı olarak floresans yoğunluk değerlerini gözlemleyerek, üstel büyüme evresinin bitiminden hemen önce dururken yukarıda açıklanan PCR koşullarını kullanarak kütüphaneyi genişletin.
- Büyütmeden sonra, 0.8x amplifikasyon reaksiyonu hacmi hacmini kullanarak standart bir boncuk temizleme yapın (bakınız adım 6.4.1)Tipik olarak 0.8 x 50 = 40 μL boncuk. Boncukları PCR ürününe ekleyin ve oda sıcaklığında 5 dakika bekleyin.
- Manyetik bir standın üzerine yerleştirin ve sıvıları çalkalayın, 200 μL% 80 etanol ile yıkayın, fazla kuruyan olmadan sıvının çoğunu çıkaracak kadar boncukları kurutun ve kuru boncuklara nükleaz içermeyen su ilave ederek elute edin.
- Oluşan DNA'yı fluorimetri kullanarak ölçün. Boyut için kitaplık parçalarını görselleştirin; Daha fazla bilgi için Veri QC beklentileri bölümüne bakın.
- Adaptör ligasyonu reaksiyonunda boncuk temizleme yapın. PCR ürününe boncuk ekledikten sonra, oda sıcaklığında 5 dakika bekleyin. Manyetik bir stand karşı yerleştirin ve sıvıları çıkarın, 200 μL% 80 etanol 200 μL yıkayın, boncukları aşırı kurutma olmadan sıvının çoğunu çıkarmak için yeterince uzun süre kurutun ve 25 mcL pipetleme ile boncuklardan DNA'yı elute edin Önerilen şekilde nükleaz içermeyen su, tüp bir manyetik standa karşı kalırken boncuklara yapıştırın.
7. Exodim Hibridizasyon
- Dört kitaplıkları farklı bağdaştırıcı çubuk kodlarıyla birleştirin.
NOT: Molariteyi hesaplamak için florimetre ile kütle ve jel ile boyut kullanılır ve daha sonra toplamda 1.000 ng'lik bir kütle havuzu için kütüphaneler eşit mololar halinde birleştirilir. Bütün tümör-normal çiftleri ayrı havuzlara ayırmak yerine bir arada tutmak en iyisidir. - Exome yakalama protokolünü uygula Up class = "xref"> 15 ve yakalama temizlendikten sonra 8 PCR döngüsü yapın. Yakalama hedeflerinden başka seçenekler olabilir.
8. Çoklama Sıralı
- Materyaller çizelgesinde atıf yapılan sıralama platformunda tek bir şeritte dört çoklu kütüphane içeren tek bir hibridizasyon yakalama dizisi 16 .
NOT: Alternatif yapılandırmalar, hedef kitlenin kapsama derinliğini planlamak ve optimize etmek isteyen gelişmiş kullanıcılar için mümkündür.
9. Analiz (İstenirse Alternatif Boru Hattı (ları ile değiştirilebilir)
- Snps ve küçük silâhlar:
- Ham veriyi, insan referans genomuna, UCSC hg19'a, Burrows-Wheeler Aligner (BWA) 17'yi kullanarak veya alternatif bir algoritma ile eşleyin. Okumaları, 20'nin altında bir eşleme kalitesi skoruyla filtreleyin veya işaretleyin ve Samtools 18 veya Picard'ı kullanarak PCR çoğaltır.Ref "> 19.
- HRS örneklerinde somatik nükleotid varyantları ve küçük indeller, Strelka 20'yi veya seçilen bir varyant çağıranı kullanan T hücre somatik kontrolleriyle karşılaştırılır. Strelka çıktısına açıklama yapmak için snpEff 21'i uygulayın. İsterseniz, Entegre Genom Görüntüleyicisi (IGV) 22 , 23 kullanarak eserler için varyant loküsleri sistematik olarak inceleyin.
- Numara değişikliklerini kopyala:
- Tümördeki kitap içi normalize edilmiş okuma sayımlarının her "ex" hedef aralığı " i " için günlük dönüşümü " ltr " oranını aşağıdaki şekilde normalinkine karşı hesaplayın:
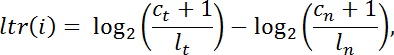
NOT: c , belirli bir yakalama aralığına eşlenen okuma sayısıdır, l toplam kütüphane boyutudur, t tümörü belirtir ve n normal gösterir. - Daha fazla analiz için yetersiz kapsama alanı ( C t + C n <100 okuma) aralıklarını filtreleyin. R. Bioconductor'tan DNA kopyası v.1.0 24 kullanarak pan-interval segmentasyon uygulayın.
NOT: Kopyalama nötr olmak için ortalama ltr'nin mutlak değerinin 0.5'in altındaki kesimleri göz önünde bulundurun . Ortalama ltr'nin işareti pozitif ise (diğer bir deyişle, normalizasyon sonrası normal numunedeki numunelerden daha fazla okumalar varsa) veya kopya sayısı kayıpları varsa, kalan bölümler kopya sayısı kazançları olarak belirtilebilir Ortalama ltr negatiftir .
- Tümördeki kitap içi normalize edilmiş okuma sayımlarının her "ex" hedef aralığı " i " için günlük dönüşümü " ltr " oranını aşağıdaki şekilde normalinkine karşı hesaplayın:
Access restricted. Please log in or start a trial to view this content.
Sonuçlar
Kütüphane çoğaltma ve 0.8x boncuk temizlemesinden sonra bir biyoanalizör arsa alınmalıdır. İstenilen aralıktaki parçacık boyutlarının "normal benzeri" bir dağılımını görmelidir ( Şekil 2a ). Eğrilikte görünür bir "omuz" gibi bu şekilden sapmalar, yüksek veya düşük molekül ağırlıklı bir eser bulunduğunu gösterir. Örneğin, Şekil 2b- 2d , ideal olarak dizi...
Access restricted. Please log in or start a trial to view this content.
Tartışmalar
Bu tekniğe hakim olduktan sonra gelecekteki uygulamalar veya yönler
Bu çalışma, en az 10 ng DNA ihtiva eden örneklerden ekspres sıralama yapılmasına olanak tanır. Klinik bağlamda, bu sınır, yetersiz materyal nedeniyle en ince iğne aspirasyon numunelerini dışlar, ancak yeterli çekirdek biyopsi ve eksizyonel biyopsi örnekleri içerir. Bu, daha geniş bir olası örnek kümesinden veri edinmesini sağlayacaktır.
Protokol d...
Access restricted. Please log in or start a trial to view this content.
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Bu proje yönteminin geliştirilmesi, Weill Cornell Tıp Fakültesi, Patoloji ve Laboratuvar Tıp Anabilim Dalı tarafından finanse edildi. Kısmi finansman için Hesaplamalı Biyoloji ve Tıp Alanında Üçlü Kurumsal Eğitim Programını kabul ediyoruz. Zaman ve bilgilerini bizimle paylaşan bilim adamlarına, özellikle Maryke Appel'e teşekkür ediyoruz; Dan Burgess; Iwanka Kozarewa; Chad Locklear; Jenny Zhang, Xiaobo (Shawn) Liang, Dong Xu, Wei Zhang, Huimin Shang, Tatiana Batson ve Tuo Zhang gibi Weill Cornell Tıp Fakültesi Genomik Çekirdek Tesisi'nden herkes.
Access restricted. Please log in or start a trial to view this content.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Petri or Cell Culture Dish (sterile) | |||
| RPMI-1640 Media | Roswell Park Memorial Institute | ||
| Fetal Calf Serum (FCS), (heat inactivated) | |||
| Freezing Media (RPMI, 20% FCS, 10% dimethylsulfoxide (DMSO))-make fresh and keep sterile | |||
| RPMI with 2% FCS (make fresh or store for up to 1 month) | |||
| scalpel with fresh blade | |||
| 10 mL syringe (no needle) | |||
| Cryogenic vials | |||
| 50 mL conical centrifuge tubes, force | |||
| Centrifuge | capable of handling 50 mL conical centrifuge tubes and providing 400g | ||
| Hepes buffer(1 M, cell culture grade) | |||
| phosphate buffered saline (PBS) | |||
| Pluoronic-F68 | Thermo-Fisher | 24040-032 | |
| DNAase-I | Sigma-Aldrich, St. Louis, MO | D4527-10KU | store as 5 mg/mL in RPMI in -200 °C |
| Bovine Serum Albumin (BSA) | |||
| Sort Media (PBS + 2% BSA + 25 mM HEPES + Pluoronic –F68 (1x)) | |||
| CD64-FITC (22) | Beckman Coulter, Miami, FL | 20 μL suggested starting volume; Titering is suggested | |
| CD30-PE (BerH83) | BD Biosciences, San Jose, CA | 20 μL suggested starting volume; Titering is suggested | |
| CD5-ECD (BL1a) | Beckman Coulter, Miami, FL | 10 μL suggested starting volume; Titering is suggested | |
| CD40-PerCP-eFluor 710 (1C10) | Ebiosciences, San Diego, CA | 5 μL suggested starting volume; Titering is suggested | |
| CD20-PC7 (B9E9) | Beckman Coulter, Miami, FL | 10 μL suggested starting volume; Titering is suggested | |
| CD15-APC (HI98) | BD Biosciences, San Jose, CA | 20 μL suggested starting volume; Titering is suggested | |
| CD45 APC-H7 (2D1) | BD Biosciences, San Jose, CA | Can be substituted with 10 μL suggested volume of CD45-Krome Orange (J.33, Beckman Coulter); Titering is suggested | |
| CD95-Pacific Blue (DX2) | Life Technologies, Grand Island, NY | 5 μL suggested starting volume; Titering is suggested | |
| CD2 (5 μg; clone RPA-2.10) | Biolegend, San Diego, CA | For optional protocol; Titering is suggested | |
| CD54 (10 μg; clone 84H10) | Serotec, Oxford, United Kingdom | For optional protocol; Titering is suggested | |
| CD58 (10 μg; clone TS2/9) | eBioscience, San Diego, CA | For optional protocol; Titering is suggested | |
| LFA-1 (12 μg; clone MHM23) | Novus Biologicals, Littleton, CO | For optional protocol; Titering is suggested | |
| BD CS&T Beads | BD Biosciences, San Jose, CA | ||
| BD Accudrop Beads | BD Biosciences, San Jose, CA | ||
| BC Versa Comp antibody capture beads | Beckman Coulter, Miami, FL | Compensation Beads | |
| BD-FACS ARIA special research order instrument using 5 lasers | BD Biosciences, San Jose, CA | any BD-FACS aria with capabilities to detect the fluorochromes in the antibody panel should be sufficient | |
| Wizard | Promega | A2360 | |
| 10 mM Tris-Cl buffer | NA | ||
| Qubit dsDNA HS Assay kit | Life Technologies, Carlsbad, CA | ||
| S2 Sonicator | Covaris, Woburn, MA | Alternatives may be substituted | |
| microTUBE | Covaris, Woburn, MA | ||
| Low-Throughput Library Preparation Kit | Kapa Biosystems, Wilmington, MA | KK8221 | |
| Sybr Green | Sigma-Aldrich, St. Louis, MO | S9430 | |
| Agencourt AMPure XP Beads | Beckman Coulter, Miami, FL | ||
| Bioanalyzer | Agilent Technologies, Santa Clara, CA | ||
| SeqCap EZ Exome v.3.0 | Roche Nimblegen | 6465684001 | |
| HiSeq | Illumina | ||
| TruSeq-style Universal adapter | Integrated DNA Technologies (IDT), Coralville, Iowa | HPLC purification; AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGAT*C*T | |
| TruSeq-style index adapter | Integrated DNA Technologies (IDT), Coralville, Iowa | HPLC purification; /5Phos/GATCGGAAGAGCACACGTCTGAACTCCAGTCACNNNNNNATCTCGTATGCCGTCTTCTGCTTG | |
| TruSeq-style PCR primer 1 | Integrated DNA Technologies (IDT), Coralville, Iowa | AATGATACGGCGACCACCGAGA | |
| TruSeq-style PCR primer 2 | Integrated DNA Technologies (IDT), Coralville, Iowa | CAAGCAGAAGACGGCATACGAG | |
| Nuclease Free Duplex Buffer | Integrated DNA Technologies (IDT), Coralville, Iowa | ||
| BD FACSDIVA software | BD Biosciences, San Jose, CA | ||
| BD Falcon Tubes | BD Biosciences, San Jose, CA | ||
| BD Flow Tubes | BD Biosciences, San Jose, CA |
Referanslar
- Abrams, J. National Cancer Institute's Precision Medicine Initiatives for the new National Clinical Trials Network. American Society of Clinical Oncology educational book / ASCO. American Society of Clinical Oncology. Meeting. , 71-76 (2014).
- Gagan, J., Van Allen, E. M. Next-generation sequencing to guide cancer therapy. Genome medicine. 7, 80(2015).
- Matsuki, E., Younes, A. Lymphomagenesis in Hodgkin lymphoma. Seminars in cancer biology. 34, 14-21 (2015).
- Fromm, J. R., Kussick, S. J., Wood, B. L. Identification and purification of classical Hodgkin cells from lymph nodes by flow cytometry and flow cytometric cell sorting. Am J Clin Pathol. 126 (5), 764-780 (2006).
- Fromm, J. R., Thomas, A., Wood, B. L. Flow cytometry can diagnose classical hodgkin lymphoma in lymph nodes with high sensitivity and specificity. Am J Clin Pathol. 131 (3), 322-332 (2009).
- Roshal, M., Wood, B. L., Fromm, J. R. Flow cytometric detection of the classical hodgkin lymphoma: clinical and research applications. Advances in hematology. 2011, 387034(2011).
- Reichel, J., et al. Flow sorting and exome sequencing reveal the oncogenome of primary Hodgkin and Reed-Sternberg cells. Blood. 125 (7), 1061-1072 (2015).
- Kozarewa, I. A Modified Method for Whole Exome Resequencing from Minimal Amounts of Starting DNA. PLoS ONE. 7 (3), e32617(2012).
- Brunicardi, F. C. Schwartz's Principles of Surgery. , 10th ed, McGraw-Hill Education / Medical. (2014).
- Kantor, A. B., Roederer, M. FACS analysis of leukocytes. Handbook of Experimental Immunology. 2, Blackwell Scientific. (1996).
- Sanders, M. E. Molecular pathways of adhesion in spontaneous rosetting of T-lymphocytes to the Hodgkin's cell line L428. Cancer Res. 48 (1), 37-40 (1988).
- Biosciences. FACSDiva Software v6.0. , Available from: http://www.bdbiosciences.com/in/instruments/software/facsdiva/resources/overview.jsp (2007).
- Biosciences. FACSAria II User's Guide. Part No. 644832, Revision A. , (2009).
- Life Technologies. Qubit 3.0 Fluorometer, Catalog Number Q33216. , Available from: https://www.thermofisher.com/order/catalog/product/Q33216 (2017).
- Roche Nimblegen. SeqCap EZ Library SR User's Guide ver. 4.1. , (2013).
- Illumina. HiSeq High-Throughput Sequencing System. , Available from: http://www.illumina.com/systems/hiseq_2500_1500.html (2016).
- Li, H., Durbin, R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics. 25 (14), 1754-1760 (2009).
- Li, H. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- The Broad Institute. Picard Tools. , Available from: http://broadinstitute.github.io/picard/ (2016).
- Saunders, C. T. Strelka: accurate somatic small-variant calling from sequenced tumor-normal sample pairs. Bioinformatics. 28 (14), 1811-1817 (2012).
- Cingolani, P., et al. A program for annotating and predicting the effects of single nucleotide polymorphisms, SnpEff: SNPs in the genome of Drosophila melanogaster strain w1118; iso-2; iso-3. Fly (Austin). 6 (2), 80-92 (2012).
- Dobin, A. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Thorvaldsdottir, H., Robinson, J. T., Mesirov, J. P. Integrative Genomics Viewer (IGV): high-performance genomics data visualization and exploration. Briefings in bioinformatics. 14 (2), 178-192 (2013).
- DNA copy number data analysis. v.R package version 1.34.0. , Available from: https://omictools.com/dnacopy-tool (2013).
Access restricted. Please log in or start a trial to view this content.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır