Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Поток-сортировка и экзомерная секвенирование клеток Рида-Штернберга классической лимфомы Ходжкина
В этой статье
Резюме
Здесь мы описываем комбинированную сортировку по проточной цитометрической ячейке и протокол построения библиотеки нижнего уровня следующего поколения, предназначенный для получения высококачественных данных с полным объемом данных из клеток Ходжкина Рида-Штернберга (HRS) классической лимфомы Ходжкина (КХЛ).
Аннотация
Клетки Ходжкина Рида-Штернберга классической лимфомы Ходжкина редко распределяются на фоне воспалительных лимфоцитов и обычно составляют менее 1% от массы опухоли. Материал, полученный из объемной опухоли, содержит содержание опухоли при концентрации, недостаточной для характеристики. Таким образом, флуоресцентная активированная клеточная сортировка с использованием восьми антител, а также рассеяние на стороне и в прямом направлении описывается здесь как способ быстрого отделения и концентрирования с высокой степенью чистоты тысяч клеток HRS из опухоли для последующего исследования. В то же время, поскольку стандартные протоколы для секвенирования exome обычно требуют 100-1000 нг входной ДНК, которая зачастую слишком высока, даже при сортировке потока, мы также предоставляем оптимизированный протокол построения библиотеки с низким входным потоком, способный производить высококачественные Данные из всего лишь 10 нг входной ДНК. Эта комбинация способна создавать библиотеки следующего поколения, подходящие для захвата гибридизации целых eПриманки для xome или более целенаправленные целевые панели, по желанию. Секвенирование Exome клеток HRS при сравнении с здоровыми внутриутробными T или B клетками может идентифицировать соматические изменения, включая мутации, вставки и делеции, а также изменения количества копий. Эти результаты подтверждают молекулярную биологию клеток HRS и могут выявлять пути для целенаправленного лечения наркотиками.
Введение
Прогресс в геномике рака в результате секвенирования следующего поколения привел к значительным прорывам в идентификации терапевтических целей и прогнозировании многих гематологических и негематологических новообразований. Новые индивидуальные стратегии лечения, основанные на специфических геномных изменениях, быстро внедряются во многих типах опухолей (см. Ссылки 1 , 2 ). Несмотря на значительный прогресс в геномике лимфомы, геном неопластических клеток HRS в классической лимфоме Ходжкина (КХЛ) был недостаточно изучен. Исследованиям мешала нехватка неопластических клеток HRS в реактивном микроокружении, что затрудняло выделение популяций очищенных клеток HRS 3 .
Метод выделения жизнеспособных клеток HRS из первичных опухолей был разработан Фроммом и др. 4 ,Ref "> 5 , 6. В этом методе используется коктейль с восемью антителами, состоящий из CD30, CD15, CD40, CD95, CD45 CD20, CD5 и CD64, для однозначной идентификации клеток HRS из суспензии опухоли CHL. Способны изолировать по меньшей мере 1000 жизнеспособных клеток HRS из свежих или замороженных клеточных суспензий из биопсий опухоли, состоящих из по меньшей мере 10 7 клеток (приблизительно 10 мг ткани). Чистота превышает 90% путем проточного цитометрического анализа и, по оценкам, Менее 80% по геномному анализу экзома в десяти последовательных случаях.
Мы усовершенствовали метод выделения проточной цитометрической клетки, который значительно облегчил процесс, позволяя быстро изолировать тысячи жизнеспособных клеток HRS от первичных опухолей CHL 7 . Мы использовали эту технику для получения того, что считается первой целой экзотической последовательностью опухолевых клеток в первичных случаях лимфомы Ходжкина. Наши исследования демонстрируютE) возможность высокопроизводительных, геномических исследований отдельных случаев КХЛ и уже привели к идентификации новых геномных изменений с возможностью объяснить аспекты патогенеза ХГЛ.
Мы также разработали трубопровод для использования извлеченной ДНК для высокопроизводительных геномных исследований. Чтобы получить достоверные результаты из 1000 отсортированных клеток HRS (минимальный из последовательных случаев), мы также разработали модифицированную процедуру построения библиотеки ДНК следующего поколения 8, которая позволила нам повысить эффективность лигирования адаптера и создать библиотеки фрагментов ДНК Без чрезмерного усиления. Этот метод позволяет анализировать обычные клинические образцы и выявлять повторяющиеся мутации и хромосомные изменения. 7 .
Access restricted. Please log in or start a trial to view this content.
протокол
1. Переработка и замораживание тканей
- Соберите ткань лимфатических узлов в забуференном фосфатом физиологическом растворе (PBS) или среде Мемориального института Розуэлла (RPMI) и процесс в течение 24 часов сбора. Перенесите вырезанную ткань лимфатического узла 9 в чашку Петри, содержащую 10 мл RPMI с 2% фетальной телячьей сывороткой (FCS), и мелко нарезать ее новым лезвием скальпеля. Используйте заднюю часть 10-мл шприцевого плунжера для дальнейшего измельчения / диссоциации ткани.
- Перенесите жидкость в коническую трубку объемом 50 мл через сетчатый фильтр размером 100 мкм. Промойте чашку Петри и фильтр дополнительными 10 мл RPMI 2% FCS.
- Получите количество клеток, используя либо автоматический счетчик клеток, либо гемоцитометр.
ПРИМЕЧАНИЕ. Как правило, ожидается, что, по крайней мере, 2 × 10 7 клеток будут от приблизительно 5 мм 3 ткани лимфоузла CHL. Ожидается более 80% жизнеспособности, но она может варьироваться в зависимости от образца. - Прокрутите клетки до 400 мкг в течение 10 мин и аспираСупернатант. Поместите пробирку, содержащую клеточный осадок, на лед.
- Предварительно охладительная среда на льду. Ресуспендируют клетки в холодной морозильной среде в концентрации 2 × 10 7 / мл и ресуспендируют ее пипетированием. Не вихрь. Инкубируйте суспензию на льду в течение 10 мин.
- Аликвоту образца 1 мл / крио-флакон (предварительно охлажденный на льду). Перенесите флаконы в морозильник -80 ° C в течение 1-2 дней и снова переведите их в жидкий азот.
2. Подготовка суспензий клеток для сортировки ячеек
ПРИМЕЧАНИЕ. Каждая партия антител должна быть правильно титрована с использованием 10 миллионов клеток в 300-μL-окрашивании. Периферическую кровь можно использовать для всех антител, за исключением CD30, для которых клеточная линия KMH2, высунутая в периферическую кровь, может использоваться для титрования 10 . Обычно мы начинаем с рекомендованного производителем объема антитела и выполняем два двухкратных разведения и одно двукратное увеличение (четыре точки данных)Для каждой партии титрования антител. Например, если производитель рекомендует объем в 10 мкл, мы проводим титрование с использованием объемов 2, 5, 10 и 20 мкл.
- Установите водяную баню до 37 ° C. Премиксное количество антител в темном стеклянном флаконе и добавление PBS + 2% BSA для общего объема коктейля 100 мкл.
ПРИМЕЧАНИЕ. Хотя мы рекомендуем титрование антител, в качестве отправной точки можно использовать следующие объемы: CD64, 20 мкл; CD95, 5 мкл; CD30, 20 мкл; CD5, 10 мкл; CD20, 10 мкл; CD15, 20 мкл; CD40, 5 мкл; И CD45, 10 мкл. - Перенесите флакон из жидкого азота в ведро для льда, содержащее сухой лед, чтобы предотвратить оттаивание. Предварительно нагрейте 50 мл оттаивающей среды, содержащей RPMI / 20% FCS / ДНКазу (100 мкг / мл) в 50-миллилитровой конической трубке на 37 ° C водяной бане.
- Переместите 45 мл оттаивающей среды на свежую пробирку и держите ее при 37 ° C. Быстро оттаять клетки, удерживая криогенный флакон в водяной бане 37 ° CЧ, пока не останется только очень маленькая замороженная часть.
- Налейте содержимое флакона в пробирку, содержащую 45 мл талой среды. Промойте пустой криогенный флакон 2 раза 1 мл талой среды и объедините промывки.
- Инкубируйте клетки при комнатной температуре в течение 15 минут, чтобы обеспечить переваривание ДНКазы и повторное уравновешивание клеток. Прокрутите клетки до 500 мкг в течение 10 мин и аспирируйте супернатант.
- Ресуспендируют клетки примерно в 200 мкл оттаивающей среды, удерживаемой назад (5 мл), и позволяют уравновешивать ее до комнатной температуры в течение 2-3 минут. Ожидается извлечение> 70% замороженных жизнеспособных клеток.
ПРИМЕЧАНИЕ. Необязательно, для большей чистоты сортированных клеток HRS, которые могут быть установлены с помощью прикрепленных Т-клеток, на этой стадии может быть добавлен коктейль немеченых антител (см. Дополнительный протокол). - Добавить 100 мкл коктейля с антителами и инкубировать в течение 15 мин при комнатной температуре (RT), защищенном от света. Добавить 3 мл сортирующей среды, согнуть клетки по 500 мкгВ течение 10 мин и аспирируют супернатант.
- Повторно суспендируйте клетки в 1 мл сортирующей среды и перенесите их в 5 мл расходомер верхней трубки.
- Промыть как 50 мл коническую трубку, так и сетчатый фильтр с добавлением 1 мл сортирующей среды и разместить клетки на льду.
3. (Факультативный протокол) Блокировка розеток T-клеток
ПРИМЕЧАНИЕ. Клетки HRS собирают с помощью Т-клеток в тканевых срезах и клеточной суспензии, и эти Т-клетки могут потенциально загрязнять отсортированную фракцию HRS. Эти взаимодействия опосредуются CD54 и CD58 на связывание HRS-клеток с LFA-1 и CD2 на Т-клетках 4 , 11 . Эти взаимодействия могут быть блокированы немечеными антителами к этим молекулам адгезии.
- Аликвот 100 000 - 500 000 клеток в 100 мкл RPMI.
- Инкубируйте клеточную суспензию с немечеными антителами к CD2, CD54, CD58 и LFA-1 (по 10 мкл каждый) на льду в течение 1 часа. TКлеточная суспензия теперь может быть помечена флуоресцентными антителами.
4. Изоляция HRS-, B- и T-клеток с использованием сортировки клеток
ПРИМЕЧАНИЕ. Хотя мы использовали специальный инструмент для заказа на исследование с использованием 5 лазеров (см. Таблицу материалов), любой сортировщик, обладающий способностью обнаруживать флуорохромы, используемые в панели антител, должен быть достаточным. Для выполнения описанных ниже шагов требуется знание функции программного обеспечения 12 и базовые знания о работе сортировщика клеток. Подробные инструкции см. В онлайн-руководстве по программному обеспечению.
- Установка цитометра:
- Включите компьютер и войдите в систему. Включите BSC (и выходы BSC), а затем включите цитометр. Подождите не менее 90 с для запуска внутреннего ЦП цитометра, а затем откройте программное обеспечение для управления лазером и убедитесь, что все лазеры включены. Запустите программное обеспечение цитометра 13 и войдите в систему,
- В программном обеспечении цитометра нажмите «Цитометр → Просмотр конфигураций». Когда откроется диалоговое окно для подпрограммы конфигурации, выделите настраиваемую конфигурацию 130 мкм и нажмите «Установить конфигурацию» и «ОК». Выйдите из подпрограммы конфигурации.
- Обратите внимание на диалоговое окно в программном обеспечении цитометра и нажмите «использовать настройки CS & T». Установите сопло 130 мкм в инструмент и включите поток, нажав красную кнопку «X» в окне потока. Дайте прибору разогреться не менее 30 минут.
- Запустите проверку производительности на инструменте с использованием желаемого метода (программный модуль отслеживания производительности включен в описанный здесь сортировщик, см. Руководство по программному обеспечению (стр. 117 - 122) и эталонный стандарт (см. Материалы для стандарта, используемого в Эта настройка).
- Нажмите «Цитометр → CST», убедитесь, что для поля «Characterize» установлено значение «ChecK Performance "в раскрывающемся меню и нажмите« Запустить ». Когда появится запрос по программному обеспечению, загрузите трубку контрольных частиц на образец и нажмите« ОК ».
- По завершении запуска нажмите «Готово» и закройте программный модуль. После того как программное обеспечение цитометра закончит повторное подключение к цитометру, нажмите «использовать настройки CS & T» в появившемся диалоговом окне.
- Определите правильную задержку задержек, используя функцию автоматического замедления для программы цитометра (см. Стр. 154 - 161 руководства по программному обеспечению).
- Откройте эксперимент «Задержка на отсрочку» в окне «Браузер» программного обеспечения цитометра, установите пробку калибровочных частиц на этапе выборки и нажмите «Загрузить» в окне «Панель приборов сбора данных». Включите функцию автоматического контроля потока, нажав кнопку «Sweet Spot» в окне «Stream». Оставляйте эту функцию в любое время для всехГ шагов.
- Включите напряжение отклоняющей пластины, нажав кнопку «Напряжение» в окне бокового потока, а затем включите проверку сортировки, нажав кнопку «Test Sort», сразу же примыкающую к кнопке «Напряжение».
- Отрегулируйте все настройки бокового потока до нуля, кроме левого потока. Отрегулируйте настройку левого бокового потока так, чтобы в окне бокового потока были видны два потока. Нажмите кнопку «Оптический фильтр» и убедитесь, что пятно левого бокового потока попадет в левое поле, которое появляется в черной области окна бокового потока.
- При необходимости отрегулируйте настройку левого бокового потока. Выключите тестовую сортировку, снова нажав кнопку «Test Sort». В окне «Браузер» разверните элемент «Глобальные рабочие листы» в эксперименте «Задержка с задержкой», нажав «+».
- Дважды щелкните «Сортировка Layout_001», чтобы открыть макет сортировки, проверьте с помощью визуального осмотраЧтобы левая сортировка была присвоена ему «P1» и нажмите «Сортировка» в окне «Сортировка». В окне «Сортировка» выберите «Автоматическая задержка», а затем «Выполнить».
- Когда выполнение завершено, нажмите «Выход». Установите пробку стерильной деионизированной воды на ступеньку образца и нажмите «Загрузить» в окне «Панель приборов сбора данных». Запустите трубку воды в течение не менее 5 минут, чтобы очистить оставшиеся тестовые частицы от инструмента до продолжения.
- Выполнить управление компенсацией (используя компенсационные бусины из таблицы материалов или аналогичные данные), используя встроенную настройку компенсации в программном обеспечении сортировщика (см. Стр. 131 - 137 руководства по программному обеспечению для получения дополнительной информации).
- Создайте новый эксперимент, нажав «Эксперимент нового эксперимента». На вкладке «Параметры» окна «Состояние прибора» удалите неиспользуемые параметры, если они есть. Нажмите «Компенсация эксперимента»Ионная настройка Создание элементов управления компенсацией. "
- В окне «Браузер» разверните образец «Элементы управления компенсацией», щелкнув знак «+». Управляйте трубами управления компенсацией без записи данных и, при необходимости, отрегулируйте напряжения детектора (на вкладке «Параметры» окна «Состояние прибора»), чтобы положительно окрашенные популяции бортов для каждого флуорохрома находились между каналами 10000 и 100000 и были Самый яркий в своем первичном канале обнаружения.
- Запишите напряжение, необходимое для каждого параметра для каждой трубки. Выделите пробирку «Неисповеданный контроль» под образцом «Контроль за компенсацией» одним щелчком левой кнопки мыши. Загрузите неокрашенную контрольную трубку на ступень образца и нажмите «Загрузить» в окне «Контроль захвата».
- Вручную введите заданные напряжения, запустив отдельные регуляторы компенсации в поля напряжения детектора для всех параметров иЗатем нажмите «Запись» в окне «Контроль доступа». Запустите все оставшиеся элементы управления компенсацией, записывая данные без изменения настроек детектора. Установите трубу из стерильной деионизированной воды в течение 5 минут, чтобы очистить остаточный материал от инструмента перед продолжением.
- HRS-ячейка:
- Приобретите и запишите не менее 100 000 событий для первоначального стробирования, отрегулировав скорость потока, чтобы получить 3000-4000 событий / с (см. Шаг 4.1).
ПРИМЕЧАНИЕ. Может потребоваться добавить дополнительную сортирующую среду для разбавления клеток, если концентрация клеток слишком высока. Прекратите приобретение. - Gate HRS с использованием шагов, описанных на рисунке 1
ПРИМЕЧАНИЕ. В большинстве случаев КХЛ от 0,01 до 0,1% клеток будут клетки HRS.
- Приобретите и запишите не менее 100 000 событий для первоначального стробирования, отрегулировав скорость потока, чтобы получить 3000-4000 событий / с (см. Шаг 4.1).
- Строение B- и Т-клеток:
- Определите соматические элементы управления (B и Т-клетки) с помощью стробирования CD20 и CD5, respe(Стробирующие лимфоциты CD45 / SSH), затем CD20 и CD5 (см . Рисунок 1 ).
- Целевые потоки сбора в сборные трубки в предварительно охлажденной двухсторонней или четырехходовой сборной стойке. Заполните либо расходомерные трубки, либо 15-миллиметровые трубки в форме центрифуг, по крайней мере, на полпути с коллекционной средой.
- Назначьте HRS и контролируйте популяции соответствующими сборными трубами в настройке сортировки, следуя инструкциям поставщика. Перезапустите захват ячейки и начните сортировку.
- Соберите все клетки HRS и до 1 миллиона B и T клеток. Направляйте потоки сбора в сборные трубки в предварительно охлажденную четырехстороннюю (или двустороннюю) сборную стойку.
5. Извлечение ДНК
- Гранулы собирали клетки центрифугированием в 1,5 мл конических пробирках при 3000 мкг в течение 10 мин и повторно суспендировали один раз с помощью 1 мл PBS для промывки клеток.
- Пелле еще раз при 3000 мкг в течение 10 мин и удалите супернатант; Будьте очень осторожны, чтобы не беспокоитьКрошечный осадок.
- Добавьте 150 мкл буфера для лизиса (или соответствующий объем для используемого набора) к промытым клеткам и перемешайте пипетированием вверх и вниз.
ПРИМЕЧАНИЕ. В этом случае, если необходимо, можно хранить клеточный лизат при -70 ° C. - Постройте узел столбца, поместив колонку фильтра внутри трубки и добавив лизат из шага 5.5 в колонку. Вращайте узел на 13000 xg в течение 3 мин.
- Снимите миниколонну со сборки и выбросьте жидкость в сборную трубку. Замените миниколонну в трубке для сбора.
- Добавьте в каждую сборку 650 мкл раствора для промывки колонны. Центрифуга в течение 1 мин при 13000 х g. Удалите жидкость из трубки для сбора. Повторите этот шаг в общей сложности 4 раза.
- Сбросьте жидкость из сборной трубы и соберите узел миниколонны. Центрифугируют в течение 2 мин при 13000 мкг, чтобы высушить матрицу связывания.
- Перенесите миниколонну в новую пробирку объемом 1,5 мл и добавьте 25 мкл 10 мМ Трис-Cl, которыйЯвляется предпочтительным для последующей стадии обработки ультразвуком, или Nuclease-Free Water, нагретой до 65 ° C. Инкубировать в течение 2 мин при комнатной температуре и центрифугировать сборку при 13000 мкг в течение 1 мин. Повторите еще раз с 25 мкл в общей сложности 50 мкл.
- Смешайте элюирование ДНК пипетированием вверх и вниз и количественно с помощью флуориметрии 14 .
6. Строительство библиотеки
- Подготовка перед началом:
- Настройте звуковой сигнал на уровне воды 12, интенсивность 5, циклы / взрыв 200 и температуру 7.
- Основываясь на доступной величине ДНК, определяемой флуориметрией и на экспериментальной схеме, определите количество ДНК, которое будет использоваться для построения библиотеки.
ПРИМЕЧАНИЕ. Имейте в виду, что если используется слишком мало ДНК и качество библиотеки скомпрометировано, может быть мало применений для материалов, которые оставлены для целей проверки. Для нестандартных входных величин, чем больше входная масса, тем выше compЛексика полученной библиотеки секвенирования. Некоторые рекомендации для этого протокола заключаются в следующем: 10 нг должны давать хорошие результаты, а 50 нг можно считать грубым максимумом. - Определите адаптер: вставьте молярное соотношение, основанное на значениях в таблице 1 .
- Рассчитайте количество адаптера, используемого следующим образом:
ПРИМЕЧАНИЕ. Количество молей ввода ДНК, n
я. N = 1,54e-12 -12 * входная масса (в ng) / (средний размер фрагмента)
II. Когда средний размер фрагмента составляет 200 п.о., это упрощает:
N = 7,7e-15 * входная масса (в ng)
Количество молей адаптера для включения,
я. A = n * r
Объем запаса адаптера для использования на этапе лигирования адаптера, v
я. V (в μL ) = a / [ 10 -12 * адаптерный запасКонцентрация (в мкМ )]
ПРИМЕЧАНИЕ. Если концентрация адаптера низкая, чтобы включить необходимое количество адаптера, адаптер-раствор может использоваться для разбавления реагентов для восстановления после замены вместо воды.
Пример: для 100 нг входной ДНК со средним размером фрагмента 200 б.п. для желаемого молярного адаптера: соотношение вставки 15: 1 и концентрация адаптера 2 мкМ, рекомендуемый объем адаптера для использования составляет 5,8 мкл. - Присвоить индексированные штрих-коды к образцам.
ПРИМЕЧАНИЕ. Библиотеки, которые будут объединены вместе в реакцию гибридизации или полосу движения в ячейке потока секвенсора, не должны содержать избыточных индексов.
- Строительство библиотеки - стрижка ДНК:
- Добавьте полное количество ДНК, которое будет использоваться в трубке для обработки ультразвуком. Если объем образца, содержащий только входную ДНК, составляет менее 50 мкл, добавьте буфер EB до общего объема 50 мкл и смешайте.
- Сонайт для 30 с.
- Удалите трубку и выполните быстрое вращение в мини-центрифуге, чтобы собрать любой аэрозоль из верхней части стенок микротрубочки.
- Повторите шаги 6.2.2-6.2.3 в течение всего семи сеансов обработки ультразвуком в течение 30 секунд в течение всего 210 с обработки ультразвуком.
ПРИМЕЧАНИЕ . Не стесняйтесь экспериментировать с делением 210 с на меньшее количество сеансов.
- Строительство библиотеки - Окончательный ремонт, A-tailing и перевязка адаптеров:
ПРИМЕЧАНИЕ. Избегайте делать выбор любого размера до этапа амплификации ПЦР библиотеки.- Следуйте инструкциям производителя 19 для окончательного ремонта и A-tailing после обработки ультразвуком ДНК образца.
- После A-tailing используйте соответствующее количество молей адаптера (рассчитанное на этапе 6.1.3) в реакции. Объедините адаптер, фрагменты ДНК A-хвоста, фермент и буфер и инкубируйте в течение ночи при 20 ° C в течение примерно 16 часов.
- LibRary construction - Расширение библиотеки:
- Выполните очистку борта реакции лигирования адаптера. После добавления шариков в продукт ПЦР подождите 5 мин при комнатной температуре. Поместите его на магнитную подставку и удалите жидкости, дважды вымывайте в 200 мкл 80% этанола, высушите гранулы достаточно долго, чтобы удалить большую часть жидкости без чрезмерной сушки, и элюировать ДНК из гранул путем пипетирования 25 мкл Безреакционной воды, как было предложено, на шарики, в то время как трубка остается на магнитной подставке.
ПРИМЕЧАНИЕ. Возможно проведение экспериментов с сохранением гранул в полиэтиленгликоле
(ПЭГ) до тех пор, пока усиление не будет отбрасываться, но это не было проверено. - Включите 0,6 мкл разведения 1: 1000 зеленого красителя на 50 мкл основной смеси ПЦР. Альтернативно, используйте совместимый с PCR интерколирующий краситель в соответствующем объеме для оборудования.
- Запрограммируйте реакцию ПЦР при 98 ° C для45 с для начальной денатурации с последующим циклом денатурации, отжигом и растяжением при 98 ° С в течение 15 с, 60 ° С в течение 30 с и 72 ° С в течение 30 с или выбрать подходящую программу, если использовать альтернативу Полимеразы.
- Установите машину для получения данных флуоресценции при 72 ° C для каждого цикла. Запрограммируйте окончательное удлинение при 72 ° C в течение 1 минуты с последующим удерживанием при 4 ° C в течение неопределенного промежутка времени. ПЦР-праймеры для амплификации библиотеки, адаптированной к адаптерам, с использованием реагентов в дополнительной таблице: Oligo 1, AATGATACGGCGACCACCGAGA и Oligo 2, CAAGCAGAAGACGGCATACGAG
- Усилить библиотеку, используя описанные выше условия ПЦР, наблюдая значения интенсивности флуоресценции в реальном времени с помощью программного обеспечения qPCR, останавливаясь непосредственно перед окончанием фазы экспоненциального роста.
- После амплификации выполните стандартную очистку шарика (см. Шаг 6.4.1), используя 0,8 × объем реакции амплификацииОбычно 0,8 × 50 = 40 мкл шариков. Добавьте шарики в продукт ПЦР и подождите 5 минут при комнатной температуре.
- Поместите его на магнитную подставку и удалите жидкости, дважды вымывайте в 200 мкл 80% этанола, высушите бусины достаточно долго, чтобы удалить большую часть жидкости без чрезмерной сушки, и элюировать, добавив воду без сушки в сухие гранулы.
- Количественную оценку полученной ДНК с помощью флуориметрии. Визуализировать фрагменты библиотеки для размера; Более подробную информацию см. В разделе «Ожидания качества данных».
- Выполните очистку борта реакции лигирования адаптера. После добавления шариков в продукт ПЦР подождите 5 мин при комнатной температуре. Поместите его на магнитную подставку и удалите жидкости, дважды вымывайте в 200 мкл 80% этанола, высушите гранулы достаточно долго, чтобы удалить большую часть жидкости без чрезмерной сушки, и элюировать ДНК из гранул путем пипетирования 25 мкл Безреакционной воды, как было предложено, на шарики, в то время как трубка остается на магнитной подставке.
7. Гибридизация Exome
- Объедините четыре библиотеки с отдельными штрих-кодами адаптера.
ПРИМЕЧАНИЕ. Масс по флуориметрии и размеру с помощью геля используют для вычисления молярности, а затем библиотеки объединяются в эквимолярных количествах в общей сумме 1000-нг-массы объединенной библиотеки. Лучше всего держать все пары нормальных опухолей, а не разделять их на отдельные пулы. - Применение протокола захвата exome Up class = "xref"> 15 и выполнить 8 циклов PCR после очистки захвата. Возможны другие варианты захвата.
8. Мультиплексированная последовательность
- Последовательность представляет собой единый захват гибридизации, содержащий четыре мультиплексированных библиотеки в одной полосе на платформе секвенирования, упомянутую в таблице 16 материалов.
ПРИМЕЧАНИЕ. Альтернативные конфигурации возможны для продвинутых пользователей, которые желают дополнительно планировать и оптимизировать свою целевую глубину охвата.
9. Анализ (может быть заменен альтернативным трубопроводом (трубопроводами), если это необходимо)
- Snps и небольшие индексы:
- Сопоставьте необработанные данные с человеческим эталонным геномом, UCSC hg19, используя Burrows-Wheeler Aligner (BWA) 17 или альтернативный алгоритм выбора. Фильтр или отметка читает с оценкой качества отображения ниже 20 и дублированием ПЦР с использованием Samtools 18 или PicardRef "> 19.
- Обнаруживать варианты соматического нуклеотида и небольшие индексы в образцах HRS по сравнению с соматическим контролем Т-клеток с использованием Strelka 20 или варианта выбора. Примените snpEff 21 для аннотации вывода Strelka. При желании систематически проверяйте варианты локусов для артефактов, используя средство Integrated Genome Viewer (IGV) 22 , 23 .
- Копирование номеров:
- Вычислить логарифмически-преобразованное отношение « ltr » для каждого интервала « i » конечного интервала нормированных отсчетов чтения внутри опухоли в опухоли по сравнению с нормальным следующим образом:
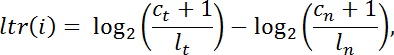
ПРИМЕЧАНИЕ: c - количество отображаемых изображений для заданного интервала захвата, l - общий размер библиотеки, t обозначает опухоль, а n обозначает нормальный. - Периодичность фильтрации с недостаточным покрытием ( C t + C n <100 просмотров) для дальнейшего анализа. Проведите интервальную сегментацию с использованием DNAcopy v.1.0 24 из Bioconductor в R.
ПРИМЕЧАНИЕ. Рассмотрим сегменты, где абсолютное значение среднего значения ltr ниже 0,5 для нейтрализации копии. Оставшиеся сегменты могут быть обозначены как увеличение числа копий, если знак среднего ltr положителен (другими словами, в образце опухоли значительно больше прочтений, чем в нормальном образце после нормализации) или потери количества копий, если знак От среднего ltr является отрицательным.
- Вычислить логарифмически-преобразованное отношение « ltr » для каждого интервала « i » конечного интервала нормированных отсчетов чтения внутри опухоли в опухоли по сравнению с нормальным следующим образом:
Access restricted. Please log in or start a trial to view this content.
Результаты
График биоанализатора следует принимать после усиления библиотеки и очистки шариков 0.8x. Нужно увидеть «нормальное» распределение размеров фрагментов в желаемом диапазоне ( рис. 2а ). Отклонения от этой формы, такие как видимое «плечо» на кривой, указыв...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Будущие приложения или направления после освоения этой техники
Эта работа позволяет секвенировать exome из образцов, содержащих по меньшей мере 10 нг ДНК. В клиническом контексте этот предел исключает большинство тонкоигольных аспирационных образцов из-за недос...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Разработка этого метода проекта финансировалась Департаментом патологии и лабораторной медицины медицинского колледжа Вайля Корнелла. Мы признаем трехуровневую программу обучения в области вычислительной биологии и медицины для частичного финансирования. Мы хотели бы поблагодарить ученых, которые делились своим временем и знаниями с нами, особенно с Маркеем Аппелем; Дэн Берджесс; Иванка Козарева; Чад Локлир; И всех из Медицинского Колледжа Уэлла Корнелла, Геномического Главного Фонда, в том числе Дженни Чжан, Сяобо (Шон) Лян, Донг Сюй, Вэй Чжан, Хуйминь Шан, Татьяна Бэтсон и Туо Чжан.
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| Petri or Cell Culture Dish (sterile) | |||
| RPMI-1640 Media | Roswell Park Memorial Institute | ||
| Fetal Calf Serum (FCS), (heat inactivated) | |||
| Freezing Media (RPMI, 20% FCS, 10% dimethylsulfoxide (DMSO))-make fresh and keep sterile | |||
| RPMI with 2% FCS (make fresh or store for up to 1 month) | |||
| scalpel with fresh blade | |||
| 10 mL syringe (no needle) | |||
| Cryogenic vials | |||
| 50 mL conical centrifuge tubes, force | |||
| Centrifuge | capable of handling 50 mL conical centrifuge tubes and providing 400g | ||
| Hepes buffer(1 M, cell culture grade) | |||
| phosphate buffered saline (PBS) | |||
| Pluoronic-F68 | Thermo-Fisher | 24040-032 | |
| DNAase-I | Sigma-Aldrich, St. Louis, MO | D4527-10KU | store as 5 mg/mL in RPMI in -200 °C |
| Bovine Serum Albumin (BSA) | |||
| Sort Media (PBS + 2% BSA + 25 mM HEPES + Pluoronic –F68 (1x)) | |||
| CD64-FITC (22) | Beckman Coulter, Miami, FL | 20 μL suggested starting volume; Titering is suggested | |
| CD30-PE (BerH83) | BD Biosciences, San Jose, CA | 20 μL suggested starting volume; Titering is suggested | |
| CD5-ECD (BL1a) | Beckman Coulter, Miami, FL | 10 μL suggested starting volume; Titering is suggested | |
| CD40-PerCP-eFluor 710 (1C10) | Ebiosciences, San Diego, CA | 5 μL suggested starting volume; Titering is suggested | |
| CD20-PC7 (B9E9) | Beckman Coulter, Miami, FL | 10 μL suggested starting volume; Titering is suggested | |
| CD15-APC (HI98) | BD Biosciences, San Jose, CA | 20 μL suggested starting volume; Titering is suggested | |
| CD45 APC-H7 (2D1) | BD Biosciences, San Jose, CA | Can be substituted with 10 μL suggested volume of CD45-Krome Orange (J.33, Beckman Coulter); Titering is suggested | |
| CD95-Pacific Blue (DX2) | Life Technologies, Grand Island, NY | 5 μL suggested starting volume; Titering is suggested | |
| CD2 (5 μg; clone RPA-2.10) | Biolegend, San Diego, CA | For optional protocol; Titering is suggested | |
| CD54 (10 μg; clone 84H10) | Serotec, Oxford, United Kingdom | For optional protocol; Titering is suggested | |
| CD58 (10 μg; clone TS2/9) | eBioscience, San Diego, CA | For optional protocol; Titering is suggested | |
| LFA-1 (12 μg; clone MHM23) | Novus Biologicals, Littleton, CO | For optional protocol; Titering is suggested | |
| BD CS&T Beads | BD Biosciences, San Jose, CA | ||
| BD Accudrop Beads | BD Biosciences, San Jose, CA | ||
| BC Versa Comp antibody capture beads | Beckman Coulter, Miami, FL | Compensation Beads | |
| BD-FACS ARIA special research order instrument using 5 lasers | BD Biosciences, San Jose, CA | any BD-FACS aria with capabilities to detect the fluorochromes in the antibody panel should be sufficient | |
| Wizard | Promega | A2360 | |
| 10 mM Tris-Cl buffer | NA | ||
| Qubit dsDNA HS Assay kit | Life Technologies, Carlsbad, CA | ||
| S2 Sonicator | Covaris, Woburn, MA | Alternatives may be substituted | |
| microTUBE | Covaris, Woburn, MA | ||
| Low-Throughput Library Preparation Kit | Kapa Biosystems, Wilmington, MA | KK8221 | |
| Sybr Green | Sigma-Aldrich, St. Louis, MO | S9430 | |
| Agencourt AMPure XP Beads | Beckman Coulter, Miami, FL | ||
| Bioanalyzer | Agilent Technologies, Santa Clara, CA | ||
| SeqCap EZ Exome v.3.0 | Roche Nimblegen | 6465684001 | |
| HiSeq | Illumina | ||
| TruSeq-style Universal adapter | Integrated DNA Technologies (IDT), Coralville, Iowa | HPLC purification; AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGAT*C*T | |
| TruSeq-style index adapter | Integrated DNA Technologies (IDT), Coralville, Iowa | HPLC purification; /5Phos/GATCGGAAGAGCACACGTCTGAACTCCAGTCACNNNNNNATCTCGTATGCCGTCTTCTGCTTG | |
| TruSeq-style PCR primer 1 | Integrated DNA Technologies (IDT), Coralville, Iowa | AATGATACGGCGACCACCGAGA | |
| TruSeq-style PCR primer 2 | Integrated DNA Technologies (IDT), Coralville, Iowa | CAAGCAGAAGACGGCATACGAG | |
| Nuclease Free Duplex Buffer | Integrated DNA Technologies (IDT), Coralville, Iowa | ||
| BD FACSDIVA software | BD Biosciences, San Jose, CA | ||
| BD Falcon Tubes | BD Biosciences, San Jose, CA | ||
| BD Flow Tubes | BD Biosciences, San Jose, CA |
Ссылки
- Abrams, J. National Cancer Institute's Precision Medicine Initiatives for the new National Clinical Trials Network. American Society of Clinical Oncology educational book / ASCO. American Society of Clinical Oncology. Meeting. , 71-76 (2014).
- Gagan, J., Van Allen, E. M. Next-generation sequencing to guide cancer therapy. Genome medicine. 7, 80(2015).
- Matsuki, E., Younes, A. Lymphomagenesis in Hodgkin lymphoma. Seminars in cancer biology. 34, 14-21 (2015).
- Fromm, J. R., Kussick, S. J., Wood, B. L. Identification and purification of classical Hodgkin cells from lymph nodes by flow cytometry and flow cytometric cell sorting. Am J Clin Pathol. 126 (5), 764-780 (2006).
- Fromm, J. R., Thomas, A., Wood, B. L. Flow cytometry can diagnose classical hodgkin lymphoma in lymph nodes with high sensitivity and specificity. Am J Clin Pathol. 131 (3), 322-332 (2009).
- Roshal, M., Wood, B. L., Fromm, J. R. Flow cytometric detection of the classical hodgkin lymphoma: clinical and research applications. Advances in hematology. 2011, 387034(2011).
- Reichel, J., et al. Flow sorting and exome sequencing reveal the oncogenome of primary Hodgkin and Reed-Sternberg cells. Blood. 125 (7), 1061-1072 (2015).
- Kozarewa, I. A Modified Method for Whole Exome Resequencing from Minimal Amounts of Starting DNA. PLoS ONE. 7 (3), e32617(2012).
- Brunicardi, F. C. Schwartz's Principles of Surgery. , 10th ed, McGraw-Hill Education / Medical. (2014).
- Kantor, A. B., Roederer, M. FACS analysis of leukocytes. Handbook of Experimental Immunology. 2, Blackwell Scientific. (1996).
- Sanders, M. E. Molecular pathways of adhesion in spontaneous rosetting of T-lymphocytes to the Hodgkin's cell line L428. Cancer Res. 48 (1), 37-40 (1988).
- Biosciences. FACSDiva Software v6.0. , Available from: http://www.bdbiosciences.com/in/instruments/software/facsdiva/resources/overview.jsp (2007).
- Biosciences. FACSAria II User's Guide. Part No. 644832, Revision A. , (2009).
- Life Technologies. Qubit 3.0 Fluorometer, Catalog Number Q33216. , Available from: https://www.thermofisher.com/order/catalog/product/Q33216 (2017).
- Roche Nimblegen. SeqCap EZ Library SR User's Guide ver. 4.1. , (2013).
- Illumina. HiSeq High-Throughput Sequencing System. , Available from: http://www.illumina.com/systems/hiseq_2500_1500.html (2016).
- Li, H., Durbin, R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics. 25 (14), 1754-1760 (2009).
- Li, H. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- The Broad Institute. Picard Tools. , Available from: http://broadinstitute.github.io/picard/ (2016).
- Saunders, C. T. Strelka: accurate somatic small-variant calling from sequenced tumor-normal sample pairs. Bioinformatics. 28 (14), 1811-1817 (2012).
- Cingolani, P., et al. A program for annotating and predicting the effects of single nucleotide polymorphisms, SnpEff: SNPs in the genome of Drosophila melanogaster strain w1118; iso-2; iso-3. Fly (Austin). 6 (2), 80-92 (2012).
- Dobin, A. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Thorvaldsdottir, H., Robinson, J. T., Mesirov, J. P. Integrative Genomics Viewer (IGV): high-performance genomics data visualization and exploration. Briefings in bioinformatics. 14 (2), 178-192 (2013).
- DNA copy number data analysis. v.R package version 1.34.0. , Available from: https://omictools.com/dnacopy-tool (2013).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены