É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Classificação de fluxo e sequenciação Exome das células de Reed-Sternberg do linfoma de Hodgkin clássico
Neste Artigo
Resumo
Aqui, descrevemos uma classificação de células de citometria de fluxo combinado e um protocolo de construção de bibliotecas de próxima geração, de baixa entrada, projetado para produzir dados de alta qualidade e total de exomas das células Hodgkin Reed-Sternberg (HRS) do linfoma de Hodgkin clássico (CHL).
Resumo
As células de Hodgkin Reed-Sternberg do linfoma de Hodgkin clássico são distribuídas escassamente em um fundo de linfócitos inflamatórios e tipicamente compreendem menos de 1% da massa tumoral. O material derivado do tumor a granel contém conteúdo de tumor em uma concentração insuficiente para caracterização. Portanto, a classificação de células ativadas por fluorescência usando oito anticorpos, bem como a dispersão lateral e frontal, é descrita aqui como um método de separação e concentração rápida com milhares de células HRS de alta pureza do tumor para estudo posterior. Ao mesmo tempo, porque os protocolos padrão para o sequenciamento exoma tipicamente requerem 100-1000 ng de DNA de entrada, que muitas vezes é muito alto, mesmo com a classificação de fluxo, também fornecemos um protocolo de construção de biblioteca otimizado e de baixa entrada capaz de produzir alta qualidade Dados de até 10 ng de DNA de entrada. Esta combinação é capaz de produzir bibliotecas de próxima geração adequadas para a captura de hibridação de todo o mundoIscas xome ou painéis direcionados focados, conforme desejado. Exome seqüenciamento das células HRS, quando comparado contra células T ou B intratumor saudáveis, pode identificar alterações somáticas, incluindo mutações, inserções e deleções, e alterações no número de cópias. Essas descobertas elucidam a biologia molecular das células HRS e podem revelar vias para tratamentos medicamentosos direcionados.
Introdução
Os avanços na genómica do câncer como resultado da sequenciação da próxima geração levaram a avanços significativos na identificação de alvos terapêuticos e na prognóstico para muitas neoplasias hematológicas e não hematológicas. Novas estratégias de tratamento individualizado baseadas em alterações genômicas específicas estão sendo introduzidas rapidamente em muitos tipos de tumores (revisado nas referências 1 , 2 ). Apesar do progresso significativo na genômica do linfoma, o genoma das células neoplásicas de HRS no linfoma de Hodgkin clássico (CHL) foi subexplorado. As investigações foram prejudicadas pela escassez de células neoplásicas de HRS dentro de um microambiente reativo, tornando difícil isolar populações de células HRS purificadas 3 .
O método para isolar células HRS viáveis de tumores primários foi desenvolvido por Fromm et al. 4 ,O método utiliza um cocktail de oito anticorpos, composto por CD30, CD15, CD40, CD95, CD45 CD20, CD5 e CD64, para identificar inequivocamente células HRS de uma suspensão de tumor CHL. Usando esta metodologia, nós São capazes de isolar pelo menos 1.000 células HRS viáveis de suspensões celulares frescas ou congeladas de biópsias tumorais consistindo em pelo menos 10 7 células (aproximadamente 10 mg de tecido). A pureza é superior a 90% por análise citométrica de fluxo e é estimada como sendo Pelo menos 80% pela análise genômica exoma de dez casos consecutivos.
Nós refinamos uma técnica de isolamento de células citométricas de fluxo que facilitou o processo, permitindo o isolamento rápido de milhares de células HRS viáveis de tumores primários de CHL 7 . Utilizamos a técnica para produzir o que se acredita ser a primeira sequência de exomas inteiras das células tumorais em casos primários de linfoma de Hodgkin. Nossos estudos demonstram queE viabilidade de estudos de alto rendimento, em todo o genoma, de casos individuais de CHL e já levaram à identificação de novas alterações genômicas com o potencial de explicar aspectos da patogênese de CHL.
Desenvolvemos ainda um pipeline para utilizar o DNA extraído para estudos genômicos de alto rendimento. A fim de obter resultados confiáveis de cerca de 1.000 células HRS classificadas (o mínimo obtido em casos seqüenciais), desenvolvemos um procedimento de construção de biblioteca de DNA de próxima geração modificado 8 que nos permitiu aumentar a eficiência da ligação de adaptador e gerar bibliotecas de fragmentos de DNA Sem amplificação excessiva. Este método permite a análise de amostras clínicas de rotina e a detecção de mutações recorrentes e alterações cromossômicas 7 .
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Processamento e congelamento de tecidos
- Recolher o tecido dos linfonodos em solução salina tamponada com fosfato (PBS) ou no meio do Instituto Roswell Park Memorial (RPMI) e processar dentro de 24 h de coleta. Transferir o tecido ganglionar excisado 9 para uma placa de Petri contendo 10 mL de RPMI com 2% de soro de vitelo fetal (FCS) e picá-lo finamente com uma lâmina de bisturi fresca. Use a parte traseira de um êmbolo de seringa de 10 mL para moer / dissociar o tecido.
- Transfira o líquido para um tubo cônico de 50 mL através de um filtro de células de 100 μm. Enxague a placa de Petri e o filtro com 10 mL adicionais de RPMI 2% FCS.
- Obtenha uma contagem de células usando um contador de células automatizado ou um hemocitômetro.
NOTA: Geralmente, pelo menos 2 x 10 7 células seriam esperadas de aproximadamente 5 mm 3 de tecido de linfonodo de CHL. É esperada uma viabilidade superior a 80%, mas pode variar de acordo com a amostra. - Gire as células a 400 xg por 10 min e aspiraO sobrenadante. Coloque o tubo contendo pellet celular no gelo.
- Meio de congelação pré-frio no gelo. Ressuspender as células em meio de congelação a frio a uma concentração de 2 x 10 7 / mL e ressuspendê-la por pipetagem. Não vortex. Incube a suspensão sobre gelo durante 10 min.
- Alíquota da amostra 1 mL / frasco de cryo (pré-refrigerado no gelo). Transfira os frascos para um congelador de -80 ° C durante 1-2 dias e transfira-os novamente para nitrogênio líquido.
2. Preparando suspensões de células para classificação de células
NOTA: Cada lote de anticorpo deve ser apropriadamente titered usando 10 milhões de células em um volume de coloração de 300 μΛ. O sangue periférico pode ser usado para todos os anticorpos, exceto CD30, para o qual a linha celular KMH2 introduzida no sangue periférico pode ser usada para titulação 10 . Em geral, começamos com o volume de anticorpo recomendado pelo fabricante e realizamos duas diluições de duas vezes e um aumento de duas vezes (quatro pontos de dados)Por cada quantidade de titulação de anticorpos. Por exemplo, se o fabricante recomenda um volume de 10 μL, realizamos a titulação usando volumes de 2, 5, 10 e 20 μL.
- Coloque um banho de água a 37 ° C. Premixar a quantidade de anticorpos em um frasco de vidro escuro e adicionar PBS + 2% de BSA para um volume total de coquetel de 100 μL.
NOTA: Embora nós recomendamos a titulação dos anticorpos, os seguintes volumes podem ser usados como ponto de partida: CD64, 20 μL; CD95, 5 μL; CD30, 20 μL; CD5, 10 μL; CD20, 10 μL; CD15, 20 μL; CD40, 5 μL; E CD45, 10 μL. - Transfira o frasco de nitrogênio líquido para um balde de gelo contendo gelo seco para evitar a descongelação. Pré-aqueça 50 ml de meio de descongelação contendo RPMI / 20% de FCS / DNase (100 μg / mL) num tubo cónico de 50 mL num banho de água a 37 ° C.
- Transfira 45 ml de meio de descongelamento para um tubo fresco e mantenha-o a 37 ° C. Descongelar rapidamente as células segurando um frasco criogênico em um morcego de água a 37 ° CH até que apenas uma porção congelada muito pequena permaneça.
- Despeje o conteúdo do frasco para dentro do tubo contendo 45 mL de meio de descongelamento. Enxague o frasco criogênico vazio 2 vezes com 1 mL de meio de descongelamento e combine os enxaguamentos.
- Incubar as células à temperatura ambiente durante 15 minutos para permitir a digestão com DNase e o reequilíbrio celular. Gire as células a 500 xg durante 10 min e aspirar o sobrenadante.
- Ressuspender as células em aproximadamente 200 μL do meio de descongelação retido (5 mL) e deixar equilibrar até a temperatura ambiente durante 2-3 minutos. Recomenda-se a recuperação de> 70% das células viáveis congeladas.
NOTA: Opcionalmente, para maior pureza de células HRS classificadas que podem ser rosetadas por células T anexas, um coquetel de anticorpos não marcados pode ser adicionado nesta etapa (veja o protocolo opcional). - Adicionar 100 μL de um coquetel de anticorpos e incubar durante 15 minutos à temperatura ambiente (RT), protegido da luz. Adicione 3 mL de meio de triagem, centrifugue as células a 500 xgPor 10 min, e aspirar o sobrenadante.
- Ressuspender as células em 1 mL de meio de triagem e transferi-las para um filtro de tubo de fluxo de 5 mL.
- Enxaguar tanto o tubo cônico de 50 mL como o filtro celular com 1 mL adicional de meio de triagem e colocar as células em gelo.
3. (Protocolo opcional) Bloqueio de Rosetas de células T
NOTA: As células HRS são rosetadas por células T em seções de tecido e suspensão celular, e essas células T podem potencialmente contaminar a fração HRS classificada. Essas interações são mediadas por CD54 e CD58 na ligação da célula HRS a LFA-1 e CD2 nas células T 4 , 11 . Essas interações podem ser bloqueadas com anticorpos não marcados para essas moléculas de adesão.
- Alíquotas de 100.000 a 500.000 células em 100 μL de RPMI.
- Incubar a suspensão celular com anticorpos não marcados para CD2, CD54, CD58 e LFA-1 (10 μL cada) em gelo durante 1 h. TA suspensão celular pode agora ser marcada com anticorpos fluorescentes.
4. HRS-, B- e Isolação de células T usando a classificação de células
NOTA: Embora usemos um instrumento de ordem de pesquisa especial usando 5 lances (veja a planilha de Materiais), qualquer classificador com a capacidade de detectar os fluoroquromos utilizados no painel de anticorpos deve ser suficiente. A execução das etapas abaixo requer uma familiaridade com a função do software 12 e um conhecimento básico das operações de classificador de células. Consulte o manual do software on-line para obter instruções detalhadas.
- Configuração do citómetro:
- Ligue o computador e faça o login. Ligue as tomadas BSC (e BSC) e, em seguida, ligue o citómetro. Aguarde pelo menos 90 s para a CPU interna do citómetro começar, e abra o software de controle a laser e verifique se todos os laser estão ligados. Inicie o software citométrico 13 e faça o login.
- Dentro do software do citometro, clique em "Citómetro → Configurações de visualização". Quando a caixa de diálogo do subprograma de configuração for aberta, destaque a configuração personalizada de 130 μm e clique em "configurar configuração" e "OK". Saia do subprograma de configuração.
- Observe a caixa de diálogo no software do citometro e clique em "usar as configurações do CS & T". Instale um bocal de 130 μm no instrumento e ligue o fluxo clicando no botão vermelho "X" na janela do fluxo. Deixe o instrumento aquecer durante pelo menos 30 min.
- Execute uma verificação de desempenho no instrumento usando o método desejado (um módulo de software de rastreamento de desempenho é incluído com o classificador descrito aqui, consulte o manual do software (pp. 117 - 122) e padrão de referência (consulte os Materiais para o padrão usado em Esta configuração).
- Clique em "Citómetro → CST", assegure-se de que o campo "Caracterizar" esteja definido como "ChecK Performance "no menu suspenso e clique em" Executar ". Quando solicitado pelo software, carregue um tubo de partículas de referência no estágio da amostra e clique em" OK ".
- Após a conclusão da execução, clique em "Concluir" e feche o módulo de software. Após o software do citómetro ter terminado de se reconectar ao citómetro, clique em "usar as configurações de CS & T" na caixa de diálogo que aparece.
- Determine o atraso de queda correto usando o recurso de atraso automático de queda do software do citometro (ver págs. 154 a 161 do manual do software).
- Abra o experimento "Drop Delay" na janela "Navegador" do software do citometro, instale um tubo de partículas de calibração no estágio da amostra e clique em "Carregar" na janela "Painel do Aquisição". Ative o recurso de monitoramento automático de fluxo, clicando no botão "Sweet Spot" na janela "Stream". Deixe este recurso em qualquer momento para todos os seguidoresG passos.
- Ative a tensão da placa de deflexão clicando no botão "Tensão" na janela de fluxo lateral e depois ative a classificação do teste clicando no botão "Testar classificação" imediatamente adjacente ao botão "Voltagem".
- Ajuste todas as configurações de fluxo lateral para zero, exceto para o fluxo do lado esquerdo. Ajuste a configuração do fluxo do lado esquerdo de modo que dois pontos de fluxo sejam visíveis na janela de fluxo lateral. Clique no botão "Filtro óptico" e verifique se o ponto do lado esquerdo do fluxo aparecerá dentro da caixa esquerda que aparece na área preta da janela do fluxo lateral.
- Ajuste a configuração do lado esquerdo se necessário. Desative a classificação do teste clicando no botão "Test Sort" novamente. Na janela "Navegador", expanda o elemento "Global Worksheets" do experimento "Drop Delay", clicando no botão "+".
- Clique duas vezes em "Ordenar Layout_001" para abrir o layout de classificação, verifique por inspeção visualQue a população de ordenação esquerda tenha "P1" atribuída a ele, e clique em "Classificar" na janela "Ordenar Layout". Na janela "Ordenar Layout", clique em "Atraso automático" e depois em "Executar".
- Quando a execução for concluída, clique em "Sair". Instale um tubo de água desionizada estéril no estágio da amostra e clique em "Carregar" na janela "Painel do Aquisição". Corra o tubo de água por pelo menos 5 minutos para limpar as partículas de teste residual do instrumento antes de prosseguir.
- Execute controles de compensação (usando contas de compensação da planilha Material ou equivalente) usando uma configuração de compensação integrada no software classificador (consulte as páginas 131 a 137 do manual do software para obter detalhes adicionais).
- Crie uma nova experiência clicando em "Experiência nova Experiência". Na guia "Parâmetros" da janela "Status do Instrumento", exclua os parâmetros não utilizados, se houver. Clique em "Experiment CompensatIon Setup Create Compensation Controls ".
- Na janela "Navegador", expanda a amostra "Controles de Compensação" clicando no sinal "+". Execute os tubos de controle de compensação sem registrar os dados e, se necessário, ajuste as tensões do detector (na guia "Parâmetros" da janela "Status do Instrumento") para que as populações de grânulos com corantes positivos para cada fluorochrome estejam entre o canal 10.000 e 100.000 e sejam Os mais brilhantes em seu canal de detecção primário.
- Anote as tensões necessárias para cada parâmetro para cada tubo. Destaque o tubo "Controle não mantido" sob a amostra "Compensação de controles", soltando um clique esquerdo. Coloque o tubo de controle não manchado no estágio da amostra e clique em "Carregar" na janela "Controle de aquisição".
- Digite manualmente as tensões determinadas executando controles de compensação individuais nos campos de tensão do detector para todos os parâmetros, eEm seguida, clique em "Gravar" na janela "Controle de Aquisição". Execute todos os controles de compensação restantes, gravando os dados sem alterar as configurações do detector. Instale um tubo de água destilada estéril durante 5 minutos para limpar qualquer material residual do instrumento antes de prosseguir.
- HAR-cell gating:
- Adquirir e gravar pelo menos 100.000 eventos para gating inicial enquanto ajusta a taxa de fluxo para adquirir 3.000-4.000 eventos / s (veja o passo 4.1).
NOTA: Pode ser necessário adicionar um meio de classificação adicional para diluir as células se a concentração celular for muito alta. Pare a aquisição. - Coloque as células HRS usando as etapas descritas na Figura 1
NOTA: Na maioria dos casos de CHL, entre 0,01% e 0,1% das células serão células HRS.
- Adquirir e gravar pelo menos 100.000 eventos para gating inicial enquanto ajusta a taxa de fluxo para adquirir 3.000-4.000 eventos / s (veja o passo 4.1).
- Abertura de células B e T:
- Identificar controles somáticos (células B e T) pelo gating de CD20 e CD5, respeitar(Gating lymphocytes by CD45 / SSH), seguido de CD20 versus CD5 (ver Figura 1 ).
- Fluxos de coleta de destino para tubos de coleta em um rack de coleta pré-refrigerado de duas vias ou quatro vias. Preencha tubos de fluxo ou tubos de estilo centrífugo de 15 mL pelo menos a meio caminho com o meio de coleta.
- Atribua HRS e controle as populações aos tubos de coleta adequados na configuração de classificação, seguindo as instruções do fornecedor. Reinicie a aquisição da célula e inicie o ordenamento.
- Recolher todas as células HRS e até 1 milhão de células B e T. Aponte os fluxos de coleta aos tubos de coleta em um rack de coleta pré-refrigerado de quatro vias (ou duas vias).
5. Extração de DNA
- As células coletadas por pastilhas por centrifugação em 1,5 ml de tubos cônicos a 3 000 xg durante 10 min e ressuspender uma vez com 1 mL de PBS para lavar as células.
- Pellet mais uma vez a 3.000 xg durante 10 min e remover o sobrenadante; Tenha muito cuidado para não incomodarO minúsculo grânulo.
- Adicione 150 μL de tampão de lise (ou um volume apropriado para o kit usado) nas células lavadas e misture por pipetagem para cima e para baixo.
NOTA: Um pode armazenar lisado celular a -70 ° C neste ponto, se necessário. - Construa um conjunto de colunas colocando a coluna de filtro dentro do tubo e adicione o lisado do passo 5.5 à coluna. Gire a montagem em 13.000 xg por 3 min.
- Remova a minicoluna da montagem e descarte o líquido no tubo de recolha. Substitua a minicoluna no tubo de recolha.
- Adicione 650 μL de solução de lavagem em coluna para cada montagem. Centrifugar durante 1 min a 13,000 x g. Descarte o líquido do tubo de recolha. Repita este passo para um total de 4 lavagens.
- Descarte o líquido do tubo de recolha e volte a montar a montagem da minicoluna. Centrifugar durante 2 min a 13,000 xg para secar a matriz de ligação.
- Transfira a minicoluna para um novo tubo de 1,5 mL e adicione 25 μL de Tris-Cl 10 mM, queÉ preferido para o passo de sonicação subsequente, ou água livre de nuclease aquecida a 65 ° C. Incubar durante 2 min a temperatura ambiente e centrifugar a montagem a 13 000 xg durante 1 min. Repita mais uma vez com 25 μL para um total de 50 μL.
- Misture a eluição do DNA por pipetagem para cima e para baixo e quantificar usando fluorimetria 14 .
6. Construção de bibliotecas
- Preparação antes do início:
- Configure o sonicador no nível de água 12, Intensidade 5, Ciclos / explosão 200 e Temp 7.
- Com base na quantidade de DNA disponível determinada pela fluorimetria e no desenho experimental, determine a quantidade de DNA a ser usada para a construção da biblioteca.
NOTA: Tenha em mente que, se muito pouco DNA for usado e a qualidade da biblioteca estiver comprometida, pode haver pouca aplicação para o material que é deixado para fins de validação. Para quantidades de insumos de qualidade inferior, quanto maior a massa de entrada, maior será o compLexidade da biblioteca de sequenciação resultante. Algumas diretrizes para este protocolo são as seguintes: 10 ng devem produzir bons resultados, e 50 ng podem ser considerados um máximo aproximado. - Decida sobre o adaptador: insira uma relação molar baseada vagamente nos valores da Tabela 1 .
- Calcule a quantidade de adaptador para usar da seguinte maneira:
NOTA: Número de moles de entrada de DNA, n
Eu. N = 1,54e-12 -12 * massa de entrada (em ng) / (tamanho médio do fragmento)
Ii. Quando o tamanho médio do fragmento é de 200 pb, isso simplifica:
N = 7.7e-15 * massa de entrada (em ng)
Número de moles de adaptador para incluir, um
Eu. A = n * r
Volume do material adaptador para usar no passo de ligação do adaptador, v
Eu. V (em μL ) = um / [ 10 -12 * adaptador de estoqueConcentração (em μM )]
NOTA: se a concentração do adaptador for baixa, para garantir que a quantidade necessária de adaptador esteja incluída, a solução do adaptador pode ser usada para diluir os reagentes de reparo final em vez de água.
Exemplo: Para 100 ng de DNA de entrada com um tamanho médio de fragmento de 200 pb, para um adaptador molar desejado: proporção de inserção de 15: 1 e uma concentração de estoque de adaptador de 2 μM, o volume de adaptador recomendado para usar é de 5,8 μL. - Atribua códigos de barras indexados às amostras.
NOTA: As bibliotecas que serão reunidas em uma reação de hibridação ou uma pista na célula de fluxo de um seqüenciador não devem conter nenhum índice redundante.
- Construção da biblioteca - cisalhamento do DNA:
- Adicione a quantidade total de DNA a ser utilizada em um tubo de sonicação. Se o volume de amostra contendo o DNA de entrada sozinho for inferior a 50 μL, adicione Buffer EB até um volume total de 50 μL e misture.
- Sonicate para 30 s.
- Remova o tubo e execute uma rotação rápida em uma mini centrífuga apenas o suficiente para coletar qualquer spray da parte superior das paredes do microtube.
- Repita as etapas 6.2.2-6.2.3 para um total de sete sessões de sonicação de 30 s para um total de 210 s de sonicação.
NOTA : Sinta-se livre para experimentar com a divisão dos 210 s em menos sessões.
- Construção da biblioteca - Reparação final, A-tailing e ligadura do adaptador:
NOTA: Evite fazer qualquer seleção de tamanho antes do passo de amplificação de PCR da biblioteca.- Siga as instruções do fabricante 19 para o reparo final e A-tailing após a sonicação do DNA da amostra.
- Após A-tailing, use o número apropriado de moles de adaptador (calculado no passo 6.1.3) na reação. Combine o adaptador, fragmentos de DNA de cola em A, enzima e tampão e incube durante a noite a 20 ° C durante aproximadamente 16 h.
- LibConstrução de Rary - Amplificação da biblioteca:
- Execute uma limpeza de talão da reação de ligação do adaptador. Após a adição de pérolas ao produto de PCR, aguarde 5 min à temperatura ambiente. Coloque-o contra um suporte magnético e remova os líquidos, lave duas vezes em 200 μL de etanol a 80%, retire as contas o suficiente para remover a maioria do líquido sem secagem excessiva e elute o DNA das contas, pipeteando 25 μL de Água livre de nuclease, como sugerido, sobre as contas enquanto o tubo permanece contra um suporte magnético.
NOTA: pode ser possível experimentar com a preservação das pérolas em polietilenoglicol
(PEG) até depois da amplificação em vez de descartá-los, mas isso não foi testado. - Inclua 0,6 μL de uma diluição 1: 1000 de corante verde por 50 μL de mistura mestre de PCR. Alternativamente, use um corante intercolando compatível com PCR em tempo real de escolha no volume apropriado para o equipamento.
- Programe a reação de PCR a 98 ° C para45 s para a desnaturação inicial, seguido de um ciclo de desnaturação, recozimento e extensão a 98 ° C durante 15 s, 60 ° C durante 30 s e 72 ° C durante 30 s, ou escolha o programa apropriado se estiver usando uma alternativa Enzima polimerase.
- Defina a máquina para levar dados de fluorescência a 72 ° C para cada ciclo. Programe uma extensão final a 72 ° C durante 1 min seguido por uma retenção a 4 ° C por um período indeterminado. Os iniciadores de PCR para a amplificação da biblioteca ligada por adaptador usando reagentes na tabela suplementar são: Oligo 1, AATGATACGGCGACCACCGAGA e Oligo 2, CAAGCAGAAGACGGCATACGAG
- Amplifique a biblioteca usando as condições de PCR descritas acima, observando os valores de intensidade de fluorescência em tempo real com o software qPCR, parando antes do final da fase de crescimento exponencial.
- Após a amplificação, faça uma limpeza de talão padrão (veja o passo 6.4.1) utilizando 0.8x o volume da reação de reação de amplificaçãoGeralmente 0,8 x 50 = 40 μL de pérolas. Adicione as contas ao produto de PCR e aguarde 5 min à temperatura ambiente.
- Coloque-o contra um suporte magnético e remova os líquidos, lave duas vezes em 200 μL de etanol a 80%, retire as contas o suficiente para remover a maior parte do líquido sem secar e elute adicionando água isenta de nuclease às esferas secas.
- Quantificar o DNA resultante usando fluorimetria. Visualize os fragmentos da biblioteca para o tamanho; Consulte a seção sobre expectativas de QC de dados para obter mais detalhes.
- Execute uma limpeza de talão da reação de ligação do adaptador. Após a adição de pérolas ao produto de PCR, aguarde 5 min à temperatura ambiente. Coloque-o contra um suporte magnético e remova os líquidos, lave duas vezes em 200 μL de etanol a 80%, retire as contas o suficiente para remover a maioria do líquido sem secagem excessiva e elute o DNA das contas, pipeteando 25 μL de Água livre de nuclease, como sugerido, sobre as contas enquanto o tubo permanece contra um suporte magnético.
7. Exome Hybridization
- Combine quatro bibliotecas com códigos de barras de adaptador distintos.
NOTA: A massa por fluorimetria e o tamanho por gel são usados para calcular a molaridade e, em seguida, as bibliotecas são combinadas em quantidades equimolares para um total de uma massa de 1.000 ng de biblioteca agrupada. É melhor manter todos os pares tumor-normal em conjunto, em vez de separá-los em pools separados. - Aplicar o protocolo de captura exome Up class = "xref"> 15 e fazer 8 ciclos de PCR após a limpeza da captura. Outras opções de alvos de captura podem ser possíveis.
8. Seqüenciamento multiplexado
- Seqüência de uma única captura de hibridização contendo quatro bibliotecas multiplexadas em uma única faixa na plataforma de seqüenciamento referenciada na planilha de materiais 16 .
NOTA: configurações alternativas são possíveis para usuários avançados que desejam planejar e otimizar a profundidade de cobertura de leitura alvo.
9. Análise (Pode ser Substituído com Pipeline Alternativo (s) se desejado)
- Snps e pequenos indels:
- Mapeie dados brutos para o genoma de referência humano, UCSC hg19, utilizando Burrows-Wheeler Aligner (BWA) 17 ou um algoritmo de escolha alternativo. Filtrar ou marcar lê com um índice de qualidade de mapeamento abaixo de 20 e duplicatas de PCR usando Samtools 18 ou PicardRef "> 19.
- Detectar variantes de nucleotídeos somáticos e indiais pequenos em amostras de HRS em comparação com os controles somáticos de células T usando Strelka 20 ou um variante de escolha. Aplique snpEff 21 para anotar a saída Strelka. Se desejar, inspecione sistematicamente os loci variantes para artefatos usando o Visor Integrado do Genoma (IGV) 22 , 23 .
- Alterações do número de cópias:
- Calcule a relação log-transformada " ltr " para cada intervalo de destino " i " de contagens de leitura normalizadas intra-biblioteca no tumor contra as do normal da seguinte maneira:
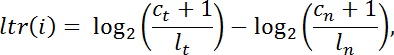
NOTA: c é o número de mapeamentos de leitura para um determinado intervalo de captura, l é o tamanho total da biblioteca, t indica tumor e n indica normal. - Filtra intervalos com cobertura insuficiente ( C t + C n <100 leituras) para análise posterior. Conduzir a segmentação pan-interval com DNAcopy v.1.0 24 de Bioconductor em R.
NOTA: Considere os segmentos onde o valor absoluto do ltr médio é inferior a 0,5 para ser neutro em cópias. Os segmentos remanescentes podem ser designados como ganhos de números de cópias, se o sinal do ltr médio for positivo (em outras palavras, há significativamente mais leituras na amostra de tumor do que na amostra normal após a normalização) ou as perdas de número de cópias, se o sinal Do ltr médio é negativo.
- Calcule a relação log-transformada " ltr " para cada intervalo de destino " i " de contagens de leitura normalizadas intra-biblioteca no tumor contra as do normal da seguinte maneira:
Access restricted. Please log in or start a trial to view this content.
Resultados
Um plano de bioanalyzer deve ser tomado após a ampliação da biblioteca e a limpeza do talão de 0,8x. Deveria ver uma distribuição "normal-like" de tamanhos de fragmentos no intervalo desejado ( Figura 2a ). Desvios dessa forma, como um "ombro" visível na curva, indicam a presença de um artefato de alto ou baixo peso molecular. Por exemplo, a Figura 2b -2d mostra exemplos de biblioteca...
Access restricted. Please log in or start a trial to view this content.
Discussão
Futuras aplicações ou direções depois de dominar esta técnica
Este trabalho permite a sequenciação exoma de amostras contendo pelo menos 10 ng de DNA. No contexto clínico, este limite exclui a maioria das amostras de aspiração com agulhas finas devido ao material insuficiente, mas inclui biópsias básicas adequadas e amostras de biópsia excisional. Isso permitirá a aquisição de dados de um conjunto maior de possíveis amostras.
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
O desenvolvimento deste método de projeto foi financiado pelo Departamento de Patologia e Medicina de Laboratório da Weill Cornell Medical College. Reconhecemos o Programa de Treinamento Tri-Institucional em Biologia Computacional e Medicina para financiamento parcial. Gostaríamos de agradecer aos cientistas que compartilharam seu tempo e conhecimento conosco, especialmente Maryke Appel; Dan Burgess; Iwanka Kozarewa; Chad Locklear; E todos do Weill Cornell Medical College Genomics Core Facility, incluindo Jenny Zhang, Xiaobo (Shawn) Liang, Dong Xu, Wei Zhang, Huimin Shang, Tatiana Batson e Tuo Zhang.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Petri or Cell Culture Dish (sterile) | |||
| RPMI-1640 Media | Roswell Park Memorial Institute | ||
| Fetal Calf Serum (FCS), (heat inactivated) | |||
| Freezing Media (RPMI, 20% FCS, 10% dimethylsulfoxide (DMSO))-make fresh and keep sterile | |||
| RPMI with 2% FCS (make fresh or store for up to 1 month) | |||
| scalpel with fresh blade | |||
| 10 mL syringe (no needle) | |||
| Cryogenic vials | |||
| 50 mL conical centrifuge tubes, force | |||
| Centrifuge | capable of handling 50 mL conical centrifuge tubes and providing 400g | ||
| Hepes buffer(1 M, cell culture grade) | |||
| phosphate buffered saline (PBS) | |||
| Pluoronic-F68 | Thermo-Fisher | 24040-032 | |
| DNAase-I | Sigma-Aldrich, St. Louis, MO | D4527-10KU | store as 5 mg/mL in RPMI in -200 °C |
| Bovine Serum Albumin (BSA) | |||
| Sort Media (PBS + 2% BSA + 25 mM HEPES + Pluoronic –F68 (1x)) | |||
| CD64-FITC (22) | Beckman Coulter, Miami, FL | 20 μL suggested starting volume; Titering is suggested | |
| CD30-PE (BerH83) | BD Biosciences, San Jose, CA | 20 μL suggested starting volume; Titering is suggested | |
| CD5-ECD (BL1a) | Beckman Coulter, Miami, FL | 10 μL suggested starting volume; Titering is suggested | |
| CD40-PerCP-eFluor 710 (1C10) | Ebiosciences, San Diego, CA | 5 μL suggested starting volume; Titering is suggested | |
| CD20-PC7 (B9E9) | Beckman Coulter, Miami, FL | 10 μL suggested starting volume; Titering is suggested | |
| CD15-APC (HI98) | BD Biosciences, San Jose, CA | 20 μL suggested starting volume; Titering is suggested | |
| CD45 APC-H7 (2D1) | BD Biosciences, San Jose, CA | Can be substituted with 10 μL suggested volume of CD45-Krome Orange (J.33, Beckman Coulter); Titering is suggested | |
| CD95-Pacific Blue (DX2) | Life Technologies, Grand Island, NY | 5 μL suggested starting volume; Titering is suggested | |
| CD2 (5 μg; clone RPA-2.10) | Biolegend, San Diego, CA | For optional protocol; Titering is suggested | |
| CD54 (10 μg; clone 84H10) | Serotec, Oxford, United Kingdom | For optional protocol; Titering is suggested | |
| CD58 (10 μg; clone TS2/9) | eBioscience, San Diego, CA | For optional protocol; Titering is suggested | |
| LFA-1 (12 μg; clone MHM23) | Novus Biologicals, Littleton, CO | For optional protocol; Titering is suggested | |
| BD CS&T Beads | BD Biosciences, San Jose, CA | ||
| BD Accudrop Beads | BD Biosciences, San Jose, CA | ||
| BC Versa Comp antibody capture beads | Beckman Coulter, Miami, FL | Compensation Beads | |
| BD-FACS ARIA special research order instrument using 5 lasers | BD Biosciences, San Jose, CA | any BD-FACS aria with capabilities to detect the fluorochromes in the antibody panel should be sufficient | |
| Wizard | Promega | A2360 | |
| 10 mM Tris-Cl buffer | NA | ||
| Qubit dsDNA HS Assay kit | Life Technologies, Carlsbad, CA | ||
| S2 Sonicator | Covaris, Woburn, MA | Alternatives may be substituted | |
| microTUBE | Covaris, Woburn, MA | ||
| Low-Throughput Library Preparation Kit | Kapa Biosystems, Wilmington, MA | KK8221 | |
| Sybr Green | Sigma-Aldrich, St. Louis, MO | S9430 | |
| Agencourt AMPure XP Beads | Beckman Coulter, Miami, FL | ||
| Bioanalyzer | Agilent Technologies, Santa Clara, CA | ||
| SeqCap EZ Exome v.3.0 | Roche Nimblegen | 6465684001 | |
| HiSeq | Illumina | ||
| TruSeq-style Universal adapter | Integrated DNA Technologies (IDT), Coralville, Iowa | HPLC purification; AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGAT*C*T | |
| TruSeq-style index adapter | Integrated DNA Technologies (IDT), Coralville, Iowa | HPLC purification; /5Phos/GATCGGAAGAGCACACGTCTGAACTCCAGTCACNNNNNNATCTCGTATGCCGTCTTCTGCTTG | |
| TruSeq-style PCR primer 1 | Integrated DNA Technologies (IDT), Coralville, Iowa | AATGATACGGCGACCACCGAGA | |
| TruSeq-style PCR primer 2 | Integrated DNA Technologies (IDT), Coralville, Iowa | CAAGCAGAAGACGGCATACGAG | |
| Nuclease Free Duplex Buffer | Integrated DNA Technologies (IDT), Coralville, Iowa | ||
| BD FACSDIVA software | BD Biosciences, San Jose, CA | ||
| BD Falcon Tubes | BD Biosciences, San Jose, CA | ||
| BD Flow Tubes | BD Biosciences, San Jose, CA |
Referências
- Abrams, J. National Cancer Institute's Precision Medicine Initiatives for the new National Clinical Trials Network. American Society of Clinical Oncology educational book / ASCO. American Society of Clinical Oncology. Meeting. , 71-76 (2014).
- Gagan, J., Van Allen, E. M. Next-generation sequencing to guide cancer therapy. Genome medicine. 7, 80(2015).
- Matsuki, E., Younes, A. Lymphomagenesis in Hodgkin lymphoma. Seminars in cancer biology. 34, 14-21 (2015).
- Fromm, J. R., Kussick, S. J., Wood, B. L. Identification and purification of classical Hodgkin cells from lymph nodes by flow cytometry and flow cytometric cell sorting. Am J Clin Pathol. 126 (5), 764-780 (2006).
- Fromm, J. R., Thomas, A., Wood, B. L. Flow cytometry can diagnose classical hodgkin lymphoma in lymph nodes with high sensitivity and specificity. Am J Clin Pathol. 131 (3), 322-332 (2009).
- Roshal, M., Wood, B. L., Fromm, J. R. Flow cytometric detection of the classical hodgkin lymphoma: clinical and research applications. Advances in hematology. 2011, 387034(2011).
- Reichel, J., et al. Flow sorting and exome sequencing reveal the oncogenome of primary Hodgkin and Reed-Sternberg cells. Blood. 125 (7), 1061-1072 (2015).
- Kozarewa, I. A Modified Method for Whole Exome Resequencing from Minimal Amounts of Starting DNA. PLoS ONE. 7 (3), e32617(2012).
- Brunicardi, F. C. Schwartz's Principles of Surgery. , 10th ed, McGraw-Hill Education / Medical. (2014).
- Kantor, A. B., Roederer, M. FACS analysis of leukocytes. Handbook of Experimental Immunology. 2, Blackwell Scientific. (1996).
- Sanders, M. E. Molecular pathways of adhesion in spontaneous rosetting of T-lymphocytes to the Hodgkin's cell line L428. Cancer Res. 48 (1), 37-40 (1988).
- Biosciences. FACSDiva Software v6.0. , Available from: http://www.bdbiosciences.com/in/instruments/software/facsdiva/resources/overview.jsp (2007).
- Biosciences. FACSAria II User's Guide. Part No. 644832, Revision A. , (2009).
- Life Technologies. Qubit 3.0 Fluorometer, Catalog Number Q33216. , Available from: https://www.thermofisher.com/order/catalog/product/Q33216 (2017).
- Roche Nimblegen. SeqCap EZ Library SR User's Guide ver. 4.1. , (2013).
- Illumina. HiSeq High-Throughput Sequencing System. , Available from: http://www.illumina.com/systems/hiseq_2500_1500.html (2016).
- Li, H., Durbin, R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics. 25 (14), 1754-1760 (2009).
- Li, H. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- The Broad Institute. Picard Tools. , Available from: http://broadinstitute.github.io/picard/ (2016).
- Saunders, C. T. Strelka: accurate somatic small-variant calling from sequenced tumor-normal sample pairs. Bioinformatics. 28 (14), 1811-1817 (2012).
- Cingolani, P., et al. A program for annotating and predicting the effects of single nucleotide polymorphisms, SnpEff: SNPs in the genome of Drosophila melanogaster strain w1118; iso-2; iso-3. Fly (Austin). 6 (2), 80-92 (2012).
- Dobin, A. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Thorvaldsdottir, H., Robinson, J. T., Mesirov, J. P. Integrative Genomics Viewer (IGV): high-performance genomics data visualization and exploration. Briefings in bioinformatics. 14 (2), 178-192 (2013).
- DNA copy number data analysis. v.R package version 1.34.0. , Available from: https://omictools.com/dnacopy-tool (2013).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados