Method Article
自己組み立てヒトタンパク質マイクロアレイにおけるキナーゼ阻害剤スクリーニング
要約
キナーゼ阻害剤のスクリーニングのための自己組み立てヒトタンパク質マイクロアレイの生成のための詳細なプロトコルが提示される。
要約
キナーゼ阻害剤のスクリーニングは、薬物の特性をよりよく理解し、臨床的に影響を及ぼす可能性のある新しい標的の同定のために重要である。このようなスクリーニングを達成するためにいくつかの方法論が報告されている。しかし、それぞれに独自の制限があります(例えば、ATP類似体のみのスクリーニング、精製キナーゼドメインの使用の制限、一度に数個以上のキナーゼの試験に関連する多大なコスト、およびタンパク質キナーゼのスクリーニングにおける柔軟性の欠如新しい突然変異)。ここでは、これらの制限の一部を克服し、キナーゼ阻害剤の公平なスクリーニングに使用することができる新しいプロトコルが提示される。この方法の強みは、異なるキナーゼまたは同じキナーゼの異なる変異体間で、複数のタンパク質にわたってキナーゼ阻害剤の活性を比較する能力です。ヒトベースのインビトロ転写および翻訳システム(IVTT)によるタンパク質キナーゼの発現によって生成される自己組み立てタンパク質マイクロアレイが採用されている。マイクロアレイに表示されるタンパク質は活性であり、キナーゼ阻害剤の効果の測定を可能にする。次の手順では、マイクロアレイの生成とスクリーニングからデータ分析まで、プロトコルステップについて詳しく説明します。
概要
タンパク質キナーゼは、その標的のリン酸化を担い、多くの細胞機能(すなわち、細胞増殖、分化、細胞死および生存)を制御する複雑な分子経路を調節することができる。キナーゼ活性の自由化は400以上の疾患に関連しており、キナーゼ阻害剤は、癌、心血管および神経疾患、炎症性疾患を含むいくつかの疾患の治療に利用できる薬物の主要なクラスの1つである。および自己免疫疾患1,2,3.
精密医療の出現により、新しい治療法、特にキナーゼ阻害剤の同定は、薬学的および臨床的に大きな魅力を持っています。キナーゼ阻害剤のデノボ設計や既存のFDA承認薬の新しい標的の同定など、キナーゼ/キナーゼ阻害剤の可能な新しいペアの同定にはいくつかのアプローチを使用できます。診療所でこれらの薬剤を実装するために必要な時間とお金は、以前の臨床試験データの可用性のために大幅に削減されているので、後者は、特に魅力的です。キナーゼ阻害剤の転用の正規例は、イマチニブであり、当初はBCR-Ablの阻害を通じて慢性骨髄性白血病(CML)の治療のために設計され、c-Kit過剰発現の治療にも成功する消化管間質腫瘍 (GISTs)4,5,6,7.
キナーゼ阻害剤のスクリーニングは、結合アッセイまたは酵素ベースのアッセイで行うことができる。アッセイの最初のクラスは、タンパク質と薬物の相互作用に焦点を当て、ライゲーション部位や親和性などの情報を提供することができます。これらのアッセイ時のキナーゼの活性は不明であるため、タンパク質の立体構造変化により多くの相互作用が見逃されたり、誤って同定されたりする可能性があります。一方、酵素ベースのアッセイでは、タンパク質キナーゼが活性であり、阻害剤が酵素活性に及ぼす影響に関する貴重な情報を提供する必要がありますが、このタイプのスクリーニングは一般的に時間と費用がかかります。現在、両方のタイプのアッセイは、いくつかのソースから市販されています。彼らは、いくつかの制限を持つキナーゼ阻害剤のスクリーニングのための信頼性の高いオプションを表します: I) 方法のほとんどは、個々の複数のキナーゼのテストを含む, これは、タンパク質の大規模なセットのスクリーニングを高価にすることができます;II)試験されるキナーゼのセットは、事前に選択された野生型キナーゼおよびいくつかのよく知られた変異バージョンのいくつかのキナーゼのリストに限定され、多くの新しい変異型アイソフォームの試験を妨げる。
この文脈では、タンパク質マイクロアレイは、市販の技術によって提示される限界のいくつかを克服することができる強力なプラットフォームです。目的の任意の配列の全長、活性タンパク質を使用してハイスループットスクリーニングで酵素ベースのアッセイを行うのに適しています。マイクロアレイは、NAPPA(核酸プログラマブルタンパク質アレイ)のような自己組み立てアプローチによって生成され、アッセイに間に合うようにタンパク質が発現され、アレイに表示されるものが実際に活性である可能性が高くなります。NAPPAに表示されるタンパク質は、自然な折りたたみおよび活性の可能性を改善するために、ヒト由来のリボソームおよびシャペロンタンパク質を使用して産生される。
タンパク質は、最初に、捕捉タグと融合した目的の遺伝子のコードをマイクロアレイ表面に印刷することによってプログラムされます。その後、インビトロ転写および翻訳(IVTT)システムを使用してマイクロアレイ上でタンパク質が生成され、新たに発現されたタンパク質は捕捉剤によってマイクロアレイ表面に固定化されます。発現されたNAPPA配列は、アレイに表示されるタンパク質の研究に、公平で高スループットの方法8,9で使用することができる。
以前は、NAPPAアレイに表示されるタンパク質が既知のパートナーと相互作用するために適切に折り畳まれたことが示されていました10;さらに、その酵素活性は2018年に初めて利用され、タンパク質キナーゼがマイクロアレイ自動リン酸化11に表示されることが示された。現在までに、NAPPAの方法論は、バイオマーカー発見12、13、14、15、16、17を含む多くの異なるアプリケーションに使用されてきました。タンパク質-タンパク質相互作用10、18、基質同定19、および薬物スクリーニング11。その柔軟性は、各アプリケーションへの適応を可能にするプラットフォームの主要な特性の1つです。
ここでは、自己組み立てNAPPAアレイにおけるチロシンキナーゼ阻害剤のスクリーニングのためのプロトコルが提示される。このプラットフォームは、活性ヒトタンパク質キナーゼの表示および低バックグラウンドおよび高ダイナミックレンジのタンパク質キナーゼ活性の分析に最適化されています。キナーゼ阻害剤のスクリーニングにNAPPAを使用するために実装された修飾の中には、すなわち、印刷化学の変化、II)キナーゼ阻害剤スクリーニング前のタンパク質マイクロアレイの脱リン酸化、およびIII)の検出の最適化が含まれる。アレイ上のリン酸化タンパク質。このプロトコルは、その種の最初であり、NAPPAマイクロアレイにおけるキナーゼ研究に関するユニークな情報を提供します。
プロトコル
1. 使用する共通のバッファとソリューション
- 調用結核媒体:素晴らしいスープ(24 g/L酵母エキス、20 g/Lトリプトン、4 mL/Lグリセロール、0.017 M KH2PO4;および0.072 M K2HPO4)。0.017 M KH2PO4および 0.072 M K2HPO4ソリューションは、10 倍のリン酸バッファー (0.17 M KH2PO4および 0.72 M K2HPO4)として購入できます。
- LB培地を調製: ルリア・ベルタニ (5 g/L 酵母エキス; 10 g/L トリプトン; 10 g/L NaCl).pH を 5 M NaOH で 7.0 に調整します。
- 1x TBSを準備:トリスバッファド生理生理生理(TBS:50 mMトリス-Cl、pH = 7.5;150 mM NaCl)。
- 1x TBSTを準備:TBSは0.1%の100%を補充した。
2. DNA製剤
注: NAPPA アレイに使用される DNA は非常に純粋でなければなりません。したがって、市販のDNAミニプレップはお勧めしません。現在、DNA調製のための2つのプロトコルが使用されています:ハウスハイスループットミニプレップ(ここに記載)または市販のMidi-またはマキシ準備で。社内ミニプレッププロトコルの平均スループットは、1人あたり1,500サンプルです。
- 社内のハイスループットミニプレップのための細菌の成長
- LB/寒天オムニプレートを準備します。LB寒天の30-40 mL(陽性クローンの選択のための抗生物質を補充したLB培地の細菌学的寒天1.5%)を各単一のウェルプレートに注ぎます。
- LB/寒天プレート上のスポットグリセロールストック。LB培地中のグリセロールストックを希釈する(1:300、通常は600μLのLBで2μL)。希釈したストックのスポット3 μLをLB/寒天プレートに10分間振ります。37 °C、上下逆さま、一晩でインキュベートします。
- 培養を予防接種する。80%のエタノールと炎で殺菌した96ピン装置を用いて、抗生物質を補充したTB培地のウェル当たり1.5mLの深部ブロックで寒天プレートから培養物を接種する。
- 文化をインキュベートする。ガス透過性シールでブロックを覆い、シェーカーに応じて37°C、300-800 rpmで22〜24時間インキュベートします。
注:800 rpmに設定されたシェーカーは、このインキュベーションに最適です。より遅い速度シェーカーを使用すると、より密度の高い培養物と低いDNA精製収率をもたらす可能性があります。 - ペレット培養。3,800 x gおよび4 °Cで30分間のスピンブロックは上清を捨てます。
- 社内のハイスループットミニプレップ
注:マルチチャンネルピペッターまたは自動ディスペンサーを使用して、社内のハイスループットミニプレップを実行できます。自動ディスペンサーを使用する場合は、使用前およびソリューション間でシステムをクリーニングしてください。- ミニ準備中に使用されるすべてのソリューションを準備します。
- 溶液1を準備する:TE再懸濁液バッファ(50 mMトリス、pH = 8.0;10 mM EDTA、pH = 8.0;0.1 mg/mL RNAE)。4 °Cで保存します。
- ソリューション 2 を準備する: NaOH/SDS 溶解バッファー (0.2 M NaOH; 1% SDS)より良い結果を出すには、新たに作られた溶液を使用する必要があります。
- ソリューション 3: KOAC 中和バッファー (2.8 M KOAc) を準備します。溶液のpHを氷河酢酸で5.1に調整します。4 °Cで保存します。
- 溶液N2を調製する:平衡バッファ(100 mMトリス、900 mM KCl;15%EtOH;0.15%トリトンX-100)。溶液のpHをリン酸で6.3に調整します。
- ソリューション N3 を準備する: bash バッファ (100 mM トリス; 1.15 M KCl; 15% EtOH)溶液のpHをリン酸で6.3に調整します。
- 溶液N5を調出す:溶出バッファー(100 mMトリス、1M KCl;15%EtOH)。溶液のpHをリン酸で8.5に調整します。
注:アニオン交換中のDNA結合、洗浄、溶出の制御が成功することは、バッファーKCl濃度およびpH値に大きく依存します。慎重なバッファコンポーネントの測定とpH調整が不可欠です。記載された測定値からの偏差が小さいと、歩留まりが著しなくなる可能性があります。
- ペレットを再サスペンドします。溶液1の200 μLを追加し、RTで5分間2,000rpmで振ります。必要に応じてブロックを渦にします。
- ライゼバクテリア。溶液2の200 μLを追加し、アルミニウムシールでプレートをシールし、5倍反転します。この手順は、ソリューション 2 の追加の最初から慎重に時間を取ります。5分を超えなす。
- ソリューションを中和します。溶液3の200 μLを追加し、アルミニウムシールでプレートをシールし、5倍反転します。シールは、リシス/中和バッファーが原因で緩んでいる可能性があるため、反転時には注意が必要です。溶液がシールに触れない部分的な反転は、サンプル間のクロス汚染を防ぐために推奨されます。
- リサートをクリアします。3,800 x gと 4 °C でプレートを遠心分離し、30 分間使用します。
- リサートペレット遠心分離工程中にアニオン交換樹脂スラリーを調記する。1Lボトルを使用して、300 mLマークに達するまでアニオン交換樹脂で満たし、900 mLまで溶液N2を追加します。
注意:このステップは、シリカ吸入から保護するためにフードで行う必要があります。 - アニオン交換樹脂プレートを用意します。フィルタープレートを深い井戸ブロックの上に積み重ねて、廃棄物回収容器として機能させます。同種になるまでアニオン交換スラリーを混ぜ、ガラスの谷に注ぎます。広い退屈なP1000の先端を使用して、スラリーの450 μLをフィルタープレートの各ウェルに移します。
- 遠心分離機積層プレート(樹脂板/深井戸板)を130×gとRTで5分間の遅い加速で積み重ね、流れを捨てます。
- 樹脂板/深井戸ブロックスタックに上清を移します。積み重ねられたプレートを30 x gで5分間回転させ、ゆっくりとしたランプアップ速度を持ちます。
- コラムを洗う。溶液N3(洗浄バッファー)を各ウェルに400μLを加えます。洗浄バッファーを除去するために真空マニホールドに樹脂プレートを転送します。洗浄手順を3回繰り返します。最後の洗濯時に、すべての井戸が適切に空にされていることを確認してください。スタックプレートを150 x gで5分間回転し、残留バッファーを除去します。
- 溶出DNA樹脂プレートをきれいな800μLの回収プレートの上に置きます。各ウェルに300 μLの溶液N5を加えます。RTに10分間座ってから、ゆっくりとしたランプアップ速度で20 x gで5分間積み重ねられたプレートを回転させます。積み重ねられたプレートを 233 x gで 1 分間回転させます。
- DNAを定量し、さらに使用するか、DNA沈殿にまっすぐに進むまで-20°Cでプレートを保存します。
注:サンプルあたり30 μg以上のDNAが必要です。DNA収率が低い場合は、DNAミニ調プリープを繰り返すか、降水ステップ中に2枚のプレートを組み合わせることをお勧めします(セクション2.3)。
- ミニ準備中に使用されるすべてのソリューションを準備します。
- DNA沈殿
- プレートを解凍し、DNA溶液を均質化する渦を、230 x gで30sで回転させ、ウェルの底部にすべての溶液を集める。
- 各ウェルに3 M NaOAcの40 μLと240 μLのイソプロパノールを加えます。アルミシールでプレートを覆い、3倍反転して混ぜます。
- 3,800 x gおよび25 °Cで30分間プレートを遠心分離する。上清を慎重に捨てます。
注:2つのプレートを組み合わせるには、2番目のプレートから最初のプレートにDNAを移し、ステップ2.3.2-2.3.3を繰り返します。 - DNAを洗浄して沈殿させる。各ウェルに80%エタノールの400 μLを追加します。アルミシール付きシールプレートを30分間1,000rpmで30分間振り、25°Cで30分間3,800 x g。上清を捨てます。
- DNAペレットを乾燥させます。プレートをペーパータオルの角度で逆さまに置き、井戸の底にアルコールが存在しないまで1〜2時間乾燥させます。任意のペレットをダウンさせるために2分間230 x gでシールと遠心分離機。
- プレートが乾燥したら、アルミシールでシールし、後で使用するために-20 °Cで凍結するか、DNAを再中断し続けます(ステップ4.1)。
3. アミノシランスライドコーティング
- ガラススライドを金属製のラックに置きます。各スライドを目視で検査し、傷や欠陥がないことを確認します。
- ロッキング中に15分間、コーティング溶液(アセトン中2%アミノシラン試薬)にスライドを沈める。アミノシラン溶液は、廃棄する前に、それぞれ30枚のスライドの2つのラックをコーティングするために使用することができます。
- すすりステップ。スライドラックをアセトン洗浄(99%アセトン)で水没させ、前後に振り、その後、すばやく5倍上下します。1つのコーナーに傾けて滴り落ち、ウルトラピュアウォーターを素早く上下5倍に沈めます。滴下に傾け、ナプキンの上に置く。
注:アセトン洗浄は2回使用できますが、超純水は毎回交換する必要があります。 - 圧力空気を使用して乾燥スライドを、すべての水滴が除去されるまで約3分間、すべての角度からそれらを吹き飛ばします。コーティングされたスライドは、密閉された箱の中の金属製のラックにRTで保管してください。
4. アレイサンプルの準備
- 20μLの超純水で社内ミニプレップ(ステップ2.3.6)からDNAペレットを再ステーペンし、2時間1,000rpmで振ります。ミディ/最大準備DNAの場合は、各サンプルを1.5 μg/μLの最終濃度に希釈し、20 μLを800μLの回収プレートに移します。
- 印刷ミックスを準備します。1つの96ウェルプレートの場合は、印刷ミックスの1 mL[237.5 μLの超純水;500 μLのポリリジン(0.01%);BS3の187.5 μL(ビス・スルホスクシニミジル、DMSOで50mg/mL)、および75 μLのポリクローナル抗水フラグを調製します。
注:化学物質は、沈殿を避けるために指定された順序で追加する必要があります。 - 各サンプルに10μLの印刷ミックスを追加し、アルミ箔でプレートをシールし、1,000rpmで90分間RTで振ります。プレートを一晩(約16時間)4°Cに保管します。
- 印刷日に、簡単に渦とプレートを回転させます。各サンプルの28 μLを384アレイプレートに移します。この転送は、オートメーションまたはマルチチャンネルピペットを使用して行うことができます。384アレイプレート内のサンプルの位置を追跡することが重要です。
- プレートを短時間回転して泡を取り除きます。プレートをホイルで密封します。
5. NAPPAアレイの生成:マイクロアレイ印刷
注:すべての印刷条件は、機器と材料の表に記載されている機器に最適化されました。別の配列を使用する場合は、さらに最適化が必要な場合があります。
- アレイラークリーンアップ。開始する前に、すべての廃棄物タンクを空にし、必要に応じて、超純水または80%エタノールで貯水池を補充します。糸くずのないワイプと超純水でピンを一つずつきれいにします。糸くずのフリーワイプでピンを乾かし、慎重にアレイザーヘッドに戻します。
- Arrayer のセットアップ: 印刷仕様 [インクあたりのスタンプの最大数: 1; 1; マルチスタンプタイミング: --; スタンプ時間 (ms): 0 ミリ秒; インク時間 (ms): 0 ms; 印刷深度調整: 90 ミクロン; タッチオフ数: 0].殺菌プロトコル:乾燥時間の0ミリ秒と待ち時間の500ミリ秒で2,000ミリ秒の超純水洗浄。これらの手順を 6x 繰り返します。その後、80%のエタノールを2,000ミリ秒、乾燥時間1,200ミリ秒、待ち時間500msで洗浄します。これらの手順を 6 倍繰り返します。
- スライド設計: 必要な配列パターンでアレイアを設定します。設計では、サンプルごとのレプリカ数、コントロールフィーチャの位置と数、配列レイアウト (1 ブロック、複数の同一ブロック)、印刷する配列の数、実行の長さなど、いくつかの要因を考慮する必要があります。
- アレイラーデッキにアミノシランコーティングされたスライド(ステップ3.4)を配置します。すべてのスライドがしっかりと保持されているかどうかを確認します。加湿器を起動します(60%に設定する必要があります)。
- 384ウェルプレートをアレイザーデッキに置きます。プログラムを起動します。
- マイクロアレイにラベルを付けます。印刷が完了したら、各スライドの下部(印刷されていない)側にスライドラベルを配置します。デッキ上のスライド印刷順序を数値順に維持します。
- 印刷されたアレイをRTの金属ラックに保管し、シリカパケットを入れた密閉された箱の中に保管してください。乾燥した環境で保たれるスライドは1年までの貯蔵寿命を有する。
- (オプション): 90 枚のスライドの 2 番目のバッチは、同じサンプルを使用して印刷できます。そうするには、プレートの印刷が完了したらすぐにアレイヤデッキから384ウェルプレートを取り外します。プレートを4°Cでシールして保管してください。配列の最初のバッチが完全に完了したら、デッキからそれらを削除し、新しいアミノシランコーティングされたスライドを配置し、新しい実行を開始します。各384ウェルプレートは、使用前に30分間RTであることを確認してください。サンプル1個につき4個以上のレプリカがスライドのバッチで印刷される場合は、384ウェルプレートを2つのプレートに分割して、アレイヤデッキに費やす時間を減らすことでサンプル蒸発を減少させることをお勧めします。
注: 2 回目の実行の開始前にすべてのリザーバを確認してください。
6. NAPPAスライド上のDNAの検出
- スライドをブロックします。スライドをピペットボックスに入れ、30 mLのブロッキングバッファを追加します。ロッキングシェーカーで1時間RTでインキュベートします。
- スライドを汚します。ブロッキング溶液を廃棄し、20 mLのブロッキングバッファーと33μLの蛍光DNAインターカリング色素を添加する。攪拌で15分間インキュベートします。その後、素早く超純水でスライドをすすいで、圧力空気で乾燥させます。スキャンを続行します (セクション 11)。
7. NAPPA スライドの表現
- RTでロッキングシェーカーのブロッキングバッファでスライドをブロックし、4つのスライド用のピペットボックスに約30 mLを使用します。
- 超純水でスライドをすすり、濾過された圧縮空気で乾燥させます。製造元の指示に従って各スライドにシールガスケットを適用します。
- IVTTミックスを追加します。各スライドはIVTTミックスの150 μLを必要とする。HeLaの82.5 μLをDEPC水の33μLで希釈し、16.5 μLのアクセサリタンパク質と33 μLの反応ミックスを補充します。非標識または非試料端部からIVTTミックスを添加する。ミックスをゆっくりとピペットします(入り込み端で一時的にビーズアップした場合は許容されます)。IVTTミックスが広がり、アレイの全領域をカバーできるように、シールガスケットを静かにマッサージします。両方のポートに小さな丸いポート シールを適用します。
- スライドをサポートに配置し、プログラム可能な冷却インキュベーターに転送します。タンパク質発現のために30°Cで90分間インキュベートし、続いて15°Cで30分間、照会タンパク質の固定化を行う。
- スライドを洗ってブロックします。シールガスケットを取り出し、約30 mLの1x TBSTのピペットボックスにスライドを取り出し、タンパク質表示用の5%ミルク(セクション8)またはキナーゼアッセイまたは薬物スクリーニング用の3%ウシ血清アルブミン(BSA)を補充した1x TBSTを補充します(セクション9)。RTで20分間攪拌してインキュベートし、このステップ2倍を繰り返します。
8. NAPPAアレイ上のタンパク質の検出
- 一次抗体を添加する。ブロッキング溶液(ステップ7.5)からスライドを取り外し、ペーパータオルを使用して裏面(印刷されていない側)を静かに乾かします。スライドを支持体に置き、1x TBST + 5%ミルクで希釈した一次抗体(マウスアンチフラグ)の600 μLを適用します。RTで1時間インキュベートします。
- ロッキングシェーカー(各5分間3x)に1x TBST + 5%ミルクでスライドを洗います。
- 二次抗体を添加する。洗濯液からスライドを取り出し、ペーパータオルで裏面をやさしく乾かします。スライドを支持体に置き、1x TBST + 5%ミルクで希釈した2次抗体(cy3-labbeled抗マウス抗体)の600 μLを塗布します。ライトからスライドを保護し、RTで1時間インキュベートします。
- ロッキングシェーカー(各5分間3x)に1x TBSTでスライドを洗います。超純水でスライドを素早くすすいで、圧力空気を使って乾燥させます。スキャンを続行します (セクション 11)。
9. NAPPAアレイにおけるチロシンキナーゼ阻害剤スクリーニング
注: 同じテストでは複数のスライドを処理できますが、各ステップで 1 つのスライドが一度に処理され、ステップ間で乾燥していないことを確認してください。スライドの非ラベルまたは非標本の端にすべての溶液を追加します。
- 薬物スクリーニング中に使用されるすべてのソリューションを準備します。
- 以下を組み合わせてホスファターゼ/DNase溶液を調製する:1xタンパク質メタロホスファターゼバッファー(50 mM HEPES、100 mM NaCl、2 mM DTT、0.01% Brij 35 pH = 7.5);1 mM MnCl2;ラムダタンパク質ホスファターゼの8,000単位;DNase I. 2単位のDNase I. 各マイクロアレイに対して400 μLの溶液を準備します。使用直前にホスファターゼとDNaseを追加します。
- 薬物希釈を行う。薬物は、10 mMの最終濃度にDMSOで再構成される。アレイでテストされたすべての薬物濃度を保証するには、濃度ごとに同じ体積のDMSO(DMSOの10,000倍のストック)が作成され、-80°Cに保たれていることを確認してください。使用時に、薬物は水中で1:100希釈される。
- 以下を組み合わせて薬剤/キナーゼ溶液を調製する: 1xキナーゼバッファー (pH = 7.5の25 mMトリス-HCl);5 mM β-グリセロリン酸;2 mM DTT;0.1 mM Na3VO4;10 mM MgCl2;500 μM ATP;そして2 μLの薬物(水中で1:100を希釈した)。マイクロアレイごとに200μLの溶液を準備します。
- ホスファターゼおよびDNase処理を行う。ブロッキング溶液(ステップ7.5)からスライドを取り外し、ペーパータオルで裏面を静かに乾かします。スライドをサポート上に置き、200 μLのホスファターゼ/DNase溶液を適用します。蒸発を避けるためにマイクロアレイカバースリップを配置します。オーブンで45分間30°Cでインキュベートします。
- ホスファターゼおよびDNase処理II:オーブンからアレイを除去し、カバースリップを破棄し、余分な溶液を除去し、作りたてのホスファターゼおよびDNase溶液の200 μLを適用する。マイクロアレイをカバースリップで覆い、オーブンで30°Cでさらに45分間インキュベートします。
- ロッキングシェーカーに1x TBST + 0.2 M NaClでスライドを洗います(各5分間3x)。
- 薬物治療およびキナーゼ反応を行う。洗濯液からスライドを取り出し、ペーパータオルで裏面をやさしく乾かします。スライドをサポート上に置き、200 μLの薬剤/キナーゼ溶液を適用します。蒸発を避けるために、カバースリップを上に置きます。オーブンで30°Cで1時間インキュベートします。
- ロッキングシェーカーに1x TBST + 0.2 M NaClでスライドを洗います(各5分間3x)。
- 1:100を希釈した一次抗体抗体抗体として使用して8.1〜8.4を繰り返す。1x TBST + 5% ミルクを 1x TBST + 3% BSA ですべてのステップで置き換えます。
10. 自動ハイブリダイゼーションプロトコル
注: また、ハイブリダイゼーション ステーションを使用して、NAPPA アレイ (セクション 7 から 9) 上のすべてのハイブリダイゼーションと洗面所を自動化し、プロトコルを補足ファイル 1として提供します。
11. 画像取得
注:マイクロアレイ画像は、20ミクロン以上の解像度で取得する必要があります。
- マイクロアレイをスライドホルダーマガジンにロードし、タンパク質を上向きにします。マガジンをマイクロアレイスキャナにロードします。
- 575/30 nm発光フィルタを使用して緑色のレーザーを選択し、cy-3標識付き二次抗体からの信号をスキャンします。別の蛍光色素を使用する場合は、蛍光色素からの信号を検出するために正しいレーザー/波長を選択します。
- 各イメージの名前と保存場所を定義します。
- (オプション):新しい蛍煙管ごとに、信号強度の線形範囲を検出するためにスキャン条件の最適化が推奨されます。これを行うには、光増倍量(PMT)の範囲を使用してマイクロアレイをスキャンし、不飽和信号と低い背景で鮮明な画像が得られるまでゲインします。
- 最適化された設定ですべてのマイクロアレイをスキャンし、自動ゲインをオフにすることを忘れないでください。
注: データ分析の場合、すべてのマイクロアレイを同じスキャン設定でスキャンする必要があります。cy3を蛍光色素として使用するキナーゼアッセイでは、装置および材料の表に記載されているスキャナを使用して、画像を20%PMT、25%のレーザー強度、および10ミクロンの分解能でスキャンします。
12. データ処理・分析
注: 同様の機能を持つマイクロアレイデータの定量化には、いくつかのソフトウェアパッケージを用意しています。ここで説明する手順は、機器および材料の表に記載されているソフトウェア用に設計されています。
- 定量化するTIFFファイルをロードし、マイクロアレイレイアウトに合わせてグリッドを設計し、可能な限り最小領域で信号全体を組み込むためにスポットのサイズを調整します。隣接するスポットは重なり合ってはなりません。ソフトウェアのパフォーマンスを視覚的に検査し、必要に応じてグリッドを手動で調整します。
- マイクロアレイの信号強度を定量します。スポットに異常(非特異的結合、ほこりなど)がないか目視で検査し、データ分析から取り除きます。
- スポットが存在しないアレイ上の隣接エリアの信号を使用して、バックグラウンドをローカルで修正します。
- データを正規化します。異なるアレイ間で信号を比較するには、各マイクロアレイの信号強度を正規化する必要があります。外れ値を除外するには、脱リン酸化マイクロアレイの正対照(IgGスポット)からの信号の30%をトリミングしてデータを正規化します。
注:IgGスポットからの信号は、マイクロアレイのリン酸化および脱リン酸化中に変化せず、正規化に適しています。 - アクティブなキナーゼを識別します。マイクロアレイに表示される各フィーチャーについて、自動リン酸化アレイと脱リン酸化アレイにおける正規化された信号強度の比率を計算します。活性キナーゼの同定に対して1.5倍の変更のしきい値を設定し、他のすべての機能を自動リン酸化(N/A)を受けることができないとしてマークします。
- ステップ12.5で識別された各キナーゼの活性を、調整された信号のパーセンテージ(正規化された正対照アレイ(DMSO)の信号強度)を正規化された負の制御アレイ(脱リン酸化)によって差し引いたものとして計算する。
結果
自己組織化されたNAPPAマイクロアレイは、バイオマーカーの発見、タンパク質とタンパク質の相互作用、基板同定、薬物スクリーニング10、11など、多くの異なる用途に使用できる固体プラットフォームを提供します。,12,13,14,15,16,17,18,19,20.
NAPPAマイクロアレイ上のチロシンキナーゼ阻害剤のキナーゼ活性およびスクリーニングの研究に採用された全体的な方法論は、図1に概略的に表される。まず、NAPPAマイクロアレイは、コーティングされたマイクロアレイ上にcDNAおよび捕捉剤を固定することによって生成される。cDNは、ヒトベースのIVTTシステムを使用してタンパク質の転写および翻訳のテンプレートとして使用され、新たに合成されたタンパク質は捕捉剤9によって固定化される。印刷されたマイクロアレイの品質は、アレイに表示されるDNAのレベル(一貫した印刷を確認する)またはタンパク質(タンパク質発現および捕捉を確認する)によって監視することができる。図 1)バックグラウンド信号を減少させ、実験のダイナミックレンジを増加させるために、マイクロアレイは1)ラムダホスファターゼで処理され、Ser/Thr/Tyr残基からリン酸化を除去し、その後2)DNaseでその場での化学を簡素化し、減少させる。背景 (図 1)
次のステップは、ATP(負対照アレイ、脱リン酸化マイクロアレイと呼ばれる)が存在しない場合にキナーゼバッファーでマイクロアレイをインキュベートし、キナーゼバッファーをATP(陽性対照、正対照)で補う自動リン酸化反応です。自動リン酸化アレイ)またはATP +DMSO(車両制御)と呼ばれます。この手順では、キナーゼは追加されません。したがって、マイクロアレイに表示される各キナーゼの本質的活性は、パン抗ホスホチロシン抗体を用いてそのリン酸化レベルの測定を通じて定量され、続いてcy3-labbeled二次抗体が続く(図1)。
4ドルプリケートで印刷されたヒトタンパク質キナーゼのパネルを表示するNAPPA-キナーゼ配列の品質管理を図2に示す。固定化されたDNAのレベルはDNA染色によって測定され、マイクロアレイ全体に均一なシグナルを示し、アレイに印刷されたDNAの量が均一であることを示唆した。また、DNA染色なしでいくつかの特徴を観察することも可能である。これらの機能は、DNAが印刷ミックスから省略されたいくつかのコントロールに対応しています[すなわち、空のスポット(何も印刷されなかった)、水の斑点、精製されたIgGスポット(ポリリジン、架本リンカーおよび精製IgG)、印刷ミックスのみ(完全な印刷ミックス:ポリリジン)プラスクロスリンカーおよび抗フラグ抗体、任意のDNAなし)。.NAPPA-キナーゼマイクロアレイに表示されるタンパク質のレベルは、抗タグ抗体を用いたIVTT反応後に評価した。
キナーゼスクリーニングでは、フラグを選択のタグとして使用し、マイクロアレイに表示されるタンパク質のレベルを抗フラグ抗体を用いて測定した。図示すように、cDNAを含むスポットの大半は、検出可能なタンパク質レベルを正常に表示しました。cDNAを持たない対照スポットの一部は、抗フラグ抗体を用いたシグナルを明らかにした:IgGスポット(二次抗体の活性を検出するために使用される)および空のベクタースポット(タグのみのcDNAコード)(図2)。NAPPA-キナーゼマイクロアレイは、0.88より高い異なる印刷バッチ間のタンパク質表示レベルの相関を用いて、スライド間で良好な再現性を示した(図2)。同じバッチ内の相関はさらに高く、0.92に近い(データは示されていない)。
次に、アレイに表示されるタンパク質のキナーゼ自動リン酸化活性を、抗ホスホチロシン抗体を用いて測定した(図3)。アレイに表示されるタンパク質は、発現後の高レベルのタンパク質リン酸化を示した(図3、左)が、配列に表示されるタンパク質の固有のキナーゼ活性またはIVTTミックス中に存在する活性キナーゼによって引き起こされる可能性がある。このリン酸化はラムダホスファターゼ処理で完全に除去され、これらのマイクロアレイはキナーゼアッセイに使用された。脱リン酸化後、ATPなしで行われた自動リン酸化反応は、予想通り有意なレベルのリン酸化を示さなかったが、ATPの存在下でキナーゼバッファーでインキュベートされたマイクロアレイは、タンパク質リン酸化を15分(同15分と示した)図 3)薬物スクリーニングのために、キナーゼ活性を60分後に自動リン酸化反応の後に測定し、試験したキナーゼの数を最大化した。
タンパク質発現直後にリン酸化レベルを測定したマイクロアレイ(図3、左)と自動リン酸化反応の60分後(図3、右)の比較を示した: i) タンパク質リン酸化のみ発現後、IVTTミックスに存在するタンパク質によって外因性リン酸化され得ることを示唆するが、自動リン酸化はできない。ii)タンパク質は、自動リン酸化反応の後にのみリン酸化され、これらのタンパク質はタンパク質発現後に活性でないこと、およびキナーゼバッファーに存在する必要な共因子が活性であることを示唆した。またはiii)両方のアレイ上でリン酸化されたタンパク質は、両方の設定で活性であったことを示唆している(図3)。
NAPPA-キナーゼ配列上のチロシンキナーゼ阻害剤のスクリーニングのために得られた結果の一例として、タンパク質キナーゼ間で明確な選択性を持つ3つのキナーゼ阻害剤が使用された:スタウロスポリン、イマチニブおよびイブルチニブ。すべてのスクリーニングについて、脱リン酸化NAPPAマイクロアレイは、自動リン酸化反応中にTKI(100nMから10uMまで)の濃度を増加させたインキュベートした。最初に試験されたTKIは、グローバルタンパク質キナーゼ阻害剤であるスタウロスポリンであり、事実上すべてのキナーゼを試験した11を超えるマイクロアレイ上で強力なキナーゼ阻害を示した。
次に、イマチニブを試験し、慢性骨髄性白血病および消化管間質腫瘍4、5、6、7の治療に用いられるABLおよびc-Kit阻害剤を用いて行った。NAPPA-キナーゼアレイでは、イマチニブはAbl1およびBCR-Abl1活性の有意な減少を示したが、他のキナーゼはほとんど影響を受けなかった(図4A)。キナーゼ活性のデータ定量は、脱リン酸化配列に対して正規化され、正対制御マイクロアレイのパーセンテージとして表された(車両のみ)。TNK2 (非関連キナーゼ)、Abl1 および BCR-Abl1 のデータを図 4Bに示します。予想通り、イマチニブはAbl1およびBCR-ABl1に対する選択的阻害を示した。c-Kit のデータは、正のコントロールアレイのアクティビティが不足しているため、決定的ではありませんでした。
最後に、ブルトンのチロシンキナーゼ(BTK)のFDA承認共価阻害剤であるイブルチニブを試験した。イブルチニブは現在、慢性リンパ球性白血病(CLL)、マントル細胞リンパ腫、およびウォルデンストロームのマクログロブリン血症21、22を含む、過活動BTKを有するいくつかの血液関連癌の治療に使用されている。図4Cは、イブルチニブスクリーニングのために得られた典型的な結果を代表する。ABL1(非関連キナーゼ)およびBTK(正規標的)およびERBB4(潜在的な新しい標的)のキナーゼ活性を図4Dに示す。データは、ERBB4が用量特異的な方法でイブルチニブによって阻害されうる可能性を示唆している。この阻害は、インビトロおよび細胞ベースのアッセイ11で確認され、このプラットフォームの力を実証した。
まとめると、NAPPA-キナーゼマイクロアレイプラットフォームは、TK阻害剤の公平なスクリーニングに使用できることを示唆している。さらに、スクリーニングは速く、目的のタンパク質キナーゼのあらゆる変化を含むように容易にカスタマイズすることができる。
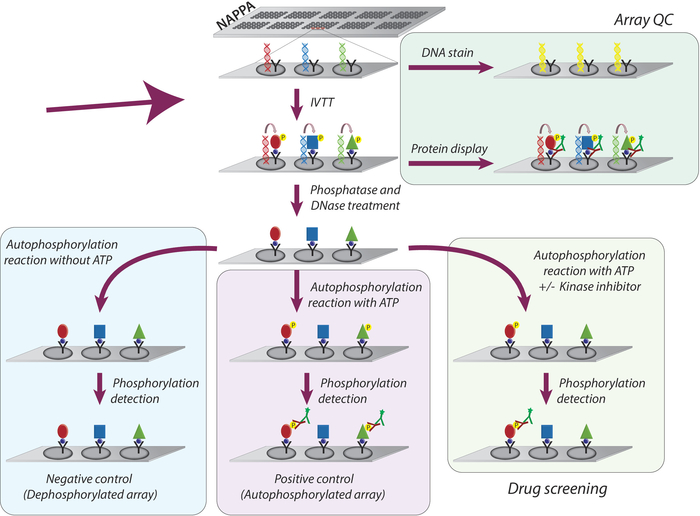
図1:NAPPAアレイにおけるチロシンキナーゼ阻害剤の品質管理とスクリーニングの概略表現NAPPAアレイは、タグと捕捉抗体と融合した目的のタンパク質に対するcDNAコードで印刷されます。インビトロ転写および翻訳反応(IVTT)の間に合成されたタンパク質は捕獲抗体によってタグを通してマイクロアレイ表面に捕獲される。アレイの品質管理(QC)は、蛍光DNAインターカリング色素を用いてスライド上に印刷されたDNAのレベルと、タグ特異的抗体を用いてアレイに表示されるタンパク質のレベルの測定によって行われます。キナーゼスクリーニングの場合、マイクロアレイはDNaseおよびホスファターゼで処理され、IVTT反応後、タンパク質合成中に生じた可能性のある印刷されたDNAおよび全てのリン酸化を除去する。脱リン酸化アレイは、薬物スクリーンに使用する準備が整いました。各アッセイでは、(I)脱リン酸化配列の3つのコントロールセットが日常的に使用され、自動リン酸化反応はATPなしで行われます。(II)自動リン酸化マイクロアレイは、ATPの存在下で自動リン酸化反応が行われる。(III)DMSO処理アレイ(車両)は、ATPおよびDMSOで自動リン酸化反応が行われる。異なる濃度のキナーゼ阻害剤で処理されたスライドは、DMSO処理アレイに使用される全く同じプロトコルに従います。この図のより大きなバージョンを表示するには、ここをクリックしてください。
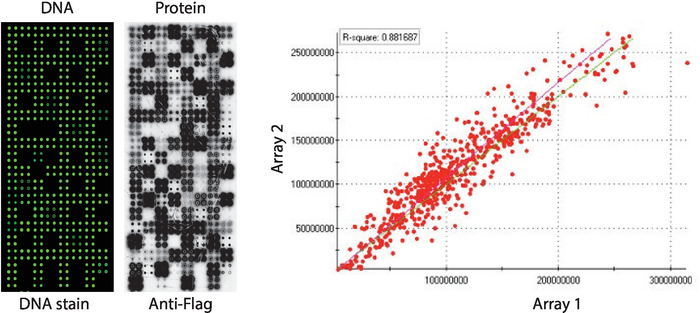
図2:自己組み立てNAPPA-キナーゼアレイの品質管理の代表的な結果蛍光DNAインターカレート色素(左)と抗フラグ抗体(中央)によって測定されたマイクロアレイ上に表示されるタンパク質のレベルによって測定されたDNA含有量が示されている。右側には、別々のバッチで印刷された2つのNAPPA-キナーゼ配列に表示されるタンパク質のレベルの相関プロットがあります。この図のより大きなバージョンを表示するには、ここをクリックしてください。
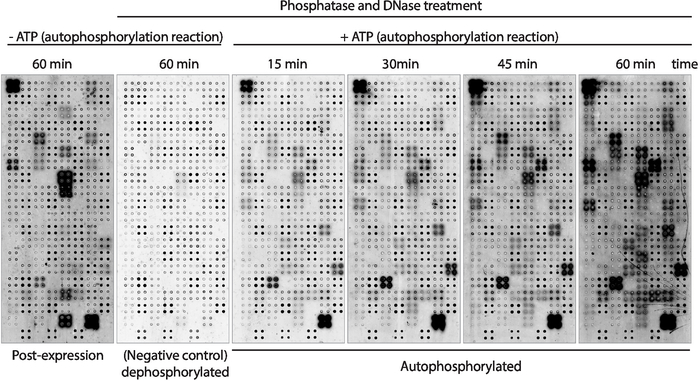
図3:NAPPA-キナーゼ配列におけるキナーゼ活性の代表的な結果四足歩行でタンパク質キナーゼを表示するマイクロアレイは、抗pTyr抗体を用いたタンパク質リン酸化の測定を通じてアレイ上のタンパク質キナーゼ活性を研究するために使用され、続いてcy3標識抗マウス抗体が続いた。ホスファターゼ/DNase処理を伴わず、自動リン酸化反応中にATPを使用せずに対照配列を使用し、タンパク質発現後のバックグラウンドリン酸化(ポスト発現)を測定した。残りのマイクロアレイをホスファターゼ/DNAで処理し、自動リン酸化反応はATP(脱リン酸化マイクロアレイ、陰性対照)またはATP(自動ホスホリン化マイクロアレイ)なしで行った。自動リン酸化マイクロアレイの場合、オートリン酸化反応は、図に示すように15分、30分、45分、または60分間行った。この図のより大きなバージョンを表示するには、ここをクリックしてください。
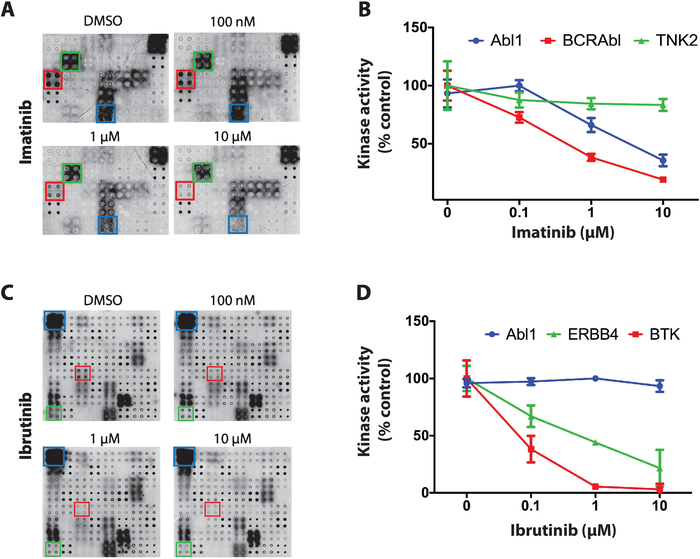
図4:NAPPA-キナーゼアレイ上のチロシンキナーゼスクリーンからの代表的なデータ。(A)ホスファターゼ/DNase処理NAPPA-キナーゼアレイは、自動リン酸化反応中にイマチニブの濃度を増加させる中でインキュベートし、キナーゼ活性を抗ホスホチル抗体で測定した。(B)イマチニブに曝露されたNAPPA-キナーゼアレイ上で観察されたキナーゼ活性の定量化。データは、負の対照アレイの信号に対して正規化された(脱リン酸化)、陽性対照アレイのパーセンテージとして示される(DMSOの存在下で行われる自動ホスポリル反応)。同様のデータは、イブルチニブ(C,D)のスクリーニングのために示されている。この図は、ラウフら11から変更されています。この図のより大きなバージョンを表示するには、ここをクリックしてください。
補足ファイル 1.自動ハイブリダイゼーションステーションを用いたNAPPAアレイにおけるチロシンキナーゼ阻害剤のスクリーニングのための代替プロトコル。このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
変更とトラブルシューティング
NAPPAアレイ上のキナーゼ活性の最適化段階では、観察された背景および低ダイナミックレンジの主なソースの1つは、印刷ミックスで使用されるBSAでした。BSAはアミノシラン表面との架橋に必要な一次アミンを提供し、DNAと捕捉抗体をその場で捕捉した。しかし、BSAはリン酸化が非常に高く、バックグラウンドノイズの上のアレイ上の自動リン酸化信号の検出が困難です。この問題を解決するために、印刷ミックス内のBSAのいくつかの代替案を試験し、ポリリジンが良好な代替として同定された。ポリリジンは、任意のリン酸化部位を欠いている;したがって、非表現配列の背景は非常に最小限です。さらに、ポリリジンで印刷されたマイクロアレイは再現性が高く、良好なレベルのタンパク質を表示します(図2)。
標準的なNAPPAアッセイに対して行われた次の重要な修飾は、ホスファターゼ/DNase処理ステップの添加であった。ホスファターゼを用いたマイクロアレイの処理により、タンパク質合成および捕捉時にIVTTミックスで発生したリン酸化物の除去が可能になります(図3)。このリン酸化の源は、本質的な自動リン酸化活性またはIVTTミックス中に存在するキナーゼの活性からである可能性がある。すべてのリン酸化後発現の除去により、活性であり、自動リン酸化を受けることができるキナーゼの容易な同定が可能となった(図3)。
プロトコル内の重要な手順
NAPPA は堅牢なテクノロジですが、予想どおり、いくつかの重要な手順があります。第1は、適切な濃度で高品質のDNAを獲得することです。品質の悪いDNAや低濃度のDNAを使用すると、いくつかの特徴が適切なレベルで発現および表示されない低品質のマイクロアレイを生成し、アレイ上で分析されるタンパク質の数を減少させます。第2の重要なステップは、マイクロアレイ上のタンパク質の発現です。高レベルの機能性タンパク質を発現するIVTTシステムの使用は、アレイ上のキナーゼ活性を研究するために不可欠です。
TKI スクリーニングの次の重要なステップは、マイクロアレイの処理方法です。マイクロアレイは、プロトコルのどのステップでも乾燥させるべきではなく、バックグラウンド信号を増加させる傷を防ぐために穏やかな取り扱いが推奨されます。実験全体の配列は互いに比較されるので、すべてのインキュベーションステップがすべてのスライドにわたって均等であることを保証することが重要です。たとえば、20 個の配列のバッチを処理して、配列間のインキュベーションの長さの違いを防ぐには、1 つの配列で 1 つのステップを実行するのに必要な時間を考慮する必要があります。
最後に、実験の設計と正と負の両方のコントロールを含めることは、品質管理とデータ分析に不可欠です。コントロールの最初のセットは、各配列に印刷されるもので、負のコントロール(すなわち、空のスポット(印刷された材料なし)、水または空のベクトル(タグのみを発現する)、および正のコントロール(すなわち、精製されたIgG、によって検出される)二次抗体であり、リン酸化レベルの変化に不活性である)。集合的に、マイクロアレイのバックグラウンドレベル、印刷中の持ち越しが可能、検出方法の信号強度を測定します。
コントロールの次のセットは、薬物スクリーニング制御であり、(DMSOの有無に)脱リン酸化および自動リン酸化マイクロアレイを含む。前述のように、脱リン酸化マイクロアレイは、ホスファターゼ処理後のリン酸化のレベルを測定し、したがって、他のすべての実験のベースラインレベルを測定します。ベースラインレベルが低いほど、アッセイのダイナミックレンジが高くなります。自動リン酸化アレイは、すべてのアレイの最大リン酸化レベルを示し、信号は強く明確でなければなりません。これは、データ分析だけでなく、すべての反応がアレイ上で正常に実行されたことを制御として使用されます。
技術の限界
現時点では、ここで提示される薬物スクリーニングの制限の1つは、自動リン酸化することができるタンパク質キナーゼのみをスクリーニングする能力である。これを克服する1つの可能な方法は、同じ場所にキナーゼと既知の基板を印刷することです。2つの異なるタンパク質のDNAの共同印刷は15に成功し、このアプローチの実現可能性を示唆した。さらに、アレイに表示されるタンパク質が正しく折りたたまれないと、不活性なタンパク質が生じる可能性があります。人間ベースの発現システムの使用は、アレイ上で測定されたキナーゼ活性を有意に改善した。しかし、一部のタンパク質は、その非活性のためにアレイ上でまだ分析することができません。
第2の制限は、パン抗ホスホチル抗体を用いたリン酸化の測定である。リン酸化部位のモチーフに関する非特異性にもかかわらず、すべての測定されたリン酸化はチロシン残基で起こり、セリンとスレオニンおよびそれぞれのキナーゼを残した。現在までに、インキュベーションと洗浄条件を最適化する試みが何度か試みられたにもかかわらず、10以上のパンホスホサー/Thr抗体が成功せずに試験されています。抗体に依存しない新しい検出システムは、薬物阻害のためにスクリーニングできるタンパク質キナーゼの数を拡大する最良の選択肢である可能性がある。このコンテキストでは、クリック結合などの放射能や化学的アプローチを含むいくつかのオプションを使用できます。バックグラウンド信号を最小限に抑え、アッセイに良好なダイナミックレンジを提供するには、一連の最適化が必要です。
3 つ目の制限は、アレイに印刷する cDNA クローンの取得です。cDNAクローンは、クリエイターやゲートウェイ23などのサイト固有の組み換えシステムを含む任意のクローン作成技術を使用して生成することができます。もう一つのオプションは、で見つかったDNAsuライブラリからクローンを購入することです。.
第4の制限は、すべての実験室が独自のNAPPAアレイを製造し、スクリーニングするための適切な機器が装備されていないことです。このプロトコルは、ハイスループット機器を必要とせずにマイクロアレイ上に印刷するDNAを生成する代替方法と、すべてのハイブリダイゼーションステップを手動で実行するプロトコルを提供します。ただし、アレイアレイスキャナとマイクロアレイスキャナへのアクセスは依然として必要です。この問題を克服する1つのオプションは、カスタマイズされたNAPPAマイクロアレイを非営利の学術価格で配布するNAPPAコアサービスと施設を使用することです。最後に、任意のスクリーニング方法論の時点で、配列上で得られたデータはアーティファクト(正または負のいずれか)に影響を受けやすいため、直交アッセイを使用して検証する必要があります。
既存の方法に関する意義
タンパク質キナーゼのスクリーニングのためにいくつかのプラットフォームが市販されています。日常的に使用されるアプローチの1つは、タンパク質断片、キナーゼドメイン、キナーゼドメインおよび一部の調節領域を有するより大きなタンパク質断片、さらには全長タンパク質で行うことができる結合アッセイです。タンパク質は、通常、発現および精製プロトコルのコストと簡潔さのために細菌系で発現される。目的の薬物とタンパク質との相互作用は、例えば、蛍光またはタグの存在のような何らかのタイプのレポートアッセイで測定される。このアプローチの主な制限は、タンパク質が薬物との相互作用中に必ずしも活性ではないという事実であり、偽陽性および偽陰性相互作用の同定をもたらす可能性がある。タンパク質断片は、特に立体構造の変化や活性の欠如に対して脆弱であり、得られたすべてのデータは、好ましくは全長の形態で、活性タンパク質を使用して検証されるべきである。一部のプラットフォームのもう 1 つの制限は、ATP アナログのみをスクリーニングする機能で、全体的な使用を制限することです。
酵素ベースのアプローチを使用してTKIのスクリーニングのために市販されているサービスのほとんどは、関心のあるキナーゼの野生型バージョンのみを使用し、時には選択された少数の変異体のみを使用します。薬剤耐性がTKIで治療された患者において非常に一般的であることを知ることは、最も適切な阻害剤の選択のために、異なる変異体における薬物応答を測定できることが重要である。NAPPAの性質上、キナーゼ変異体のスクリーニングは簡単で簡単に行うことができ、必要な唯一のツールは、例えば、サイト特異的変異体によって行うことができるNAPPA cDNAコレクションにキナーゼ変異体を組み込むことです。
今後のアプリケーション
キナーゼ阻害剤を用いたがん治療における治療の最も一般的な形態の1つは、治療コース中の薬物標的における変異の獲得である。キナーゼ阻害剤に対する応答に関するこれらの変異体のスクリーニングは、各患者に対する個別化された治療を達成するために、TKIの第2/第3世代の選択にとって極めて重要である。ここで提示される薬物スクリーニングアプローチは、ヒトゲノムに存在するチロシンキナーゼのパネルに対して任意のチロシンキナーゼ阻害剤を試験することができる公平なスクリーニングプラットフォームを提供する。NAPPAアレイに表示されるタンパク質は、スライドに印刷されたcDNAからインビトロで発現されるので、任意の変異変異体は、配列に表示されるcDNAコレクションに容易に組み込むことができます。NAPPA技術のハイスループットパワーと組み合わせて、アレイ上でキナーゼ変異体を生成・発現できる施設は、キナーゼ変異体の研究と薬物に対する応答に特有の環境を提供し、NAPPAを適当なものにします。精密医療の目標の一つであるパーソナライズされた薬物スクリーニング。
開示事項
著者は利益相反を宣言しない。
謝辞
著者たちは、プロジェクトの開発中に彼らの助けと批判のためにLaBaerの研究室のすべての人に感謝したいと思います。このプロジェクトは、NIH助成金U01CA117374、U01AI077883、バージニアG.パイパー財団によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| 364 well plates (for arraying) | Genetix | x7020 | |
| 800 µL 96-well collection plate | Abgene | AB-0859 | |
| 96-pin device | Boekel | 140500 | |
| Acetic Acid | Millipore-Sigma | 1.00066 | |
| Acetone 99.9% | Millipore Sigma | 650501 | |
| Aluminum seal for 96 well plates | VWR | 76004-236 | |
| Aminosilane (3-aminopropyltriethoxysilane) | Pierce | 80370 | |
| ANTI-FLAG M2 antibody produced in mouse | Millipore Sigma | F3165 | |
| Anti-Flag rabbit Antibody (polyclonal) | Millipore Sigma | F7425 | |
| ATP 10 mM | Cell Signaling | 9804S | |
| β-Glycerophosphate disodium salt hydrate | Millipore-Sigma | G9422 | |
| bacteriological agar | VWR | 97064-336 | |
| Blocking Buffer | ThermoFisher/Pierce | 37535 | |
| Brij 35 | ThermoFisher/Pierce | BP345-500 | |
| BS3 (bis-sulfosuccinimidyl) | ThermoFisher/Pierce | 21580 | |
| BSA (bovine serum albumin) | Millipore Sigma | A2153 | |
| Coverslip 24 x 60 mm | VWR | 48393-106 | |
| Cy3 AffiniPure Donkey Anti-Mouse IgG (H+L) | Jackson ImmunoResearch | 715-165-150 | |
| DeepWell Block, case of 50 | ThermoFisher/AbGene | AB-0661 | |
| DEPC water | Ambion | 9906 | |
| DMSO (Dimethyl Sulfoxide) | Millipore-Sigma | D8418 | |
| DNA-intercalating dye | Invitrogen | P11495 | |
| DNase I | Millipore-Sigma | AMPD1-1KT | |
| DTT | Millipore-Sigma | 43816 | |
| EDTA | Millipore-Sigma | EDS | |
| Ethanol 200 proof | Millipore-Sigma | E7023 | |
| Filter plates | Millipore-Sigma | WHA77002804 | |
| Gas Permeable Seals, box of 50 | ThermoFisher/AbGene | AB-0718 | |
| Glass box | Wheaton | 900201 | |
| Glass slides | VWR | 48300-047 | |
| Glycerol | Millipore-Sigma | G5516 | |
| HCl (Hydrochloric acid) | Millipore-Sigma | H1758 | |
| HEPES Buffer Solution | Millipore-Sigma | 83264 | |
| Human-based IVTT system | Thermo Scientific | 88882 | |
| ImmunoPure Mouse IgG whole molecule | ThermoFisher/Pierce | 31202 | |
| Isopropanol | Millipore-Sigma | I9516 | |
| KCl (Potassium chloride) | Millipore-Sigma | P9333 | |
| KH2PO4(Potassium phosphate monobasic) | Millipore-Sigma | P5655 | |
| Kinase buffer | Cell Signaling | 9802 | |
| KOAc (Potassium acetate) | Millipore-Sigma | P1190 | |
| Lambda Protein Phosphatase | new england biolabs | P0753 | |
| Lifterslips, 24 x 60 mm | ThermoFisher Scientific | 25X60I24789001LS | |
| Metal 30-slide rack with no handles | Wheaton | 900234 | |
| MgCL2 (Magnesium chloride) | Millipore-Sigma | M8266 | |
| Na3VO4 (Sodium orthovanadate) | Millipore-Sigma | S6508 | |
| NaCl (Sodium Chloride) | Millipore-Sigma | S3014 | |
| NaOAc (Sodium acetate) | Millipore-Sigma | S2889 | |
| NaOH (Sodium hydroxide) | Millipore-Sigma | S8045 | |
| NucleoBond Xtra Midi / Maxi | Macherey-Nagel | 740410.10 / 740414.10 | |
| Nucleoprep Anion II | Macherey Nagel | 740503.1 | |
| Phosphoric Acid | Millipore-Sigma | 79617 | |
| Poly-L-Lysine Solution (0.01%) | Millipore-Sigma | A-005-C | |
| Protein Phosphatase (Lambda) | New England Biolabs | P0753 | |
| RNAse | Invitrogen | 12091021 | |
| SDS (Sodium dodecyl sulfate) | Millipore-Sigma | L6026 | |
| SDS (Sodium dodecyl sulfate) | Millipore-Sigma | 05030 | |
| Sealing gasket | Grace Bio-Labs, Inc | 44904 | |
| Silica packets | VWR | 100489-246 | |
| Single well plate | ThermoFisher/Nalge Nunc | 242811 | |
| Sodium acetate (3M, pH 5.5) | Millipore-Sigma | 71196 | |
| TB media (Terrific Broth) | Millipore-Sigma | T0918 | |
| Tris | IBI scientific | IB70144 | |
| Triton X-100 | Millipore-Sigma | T8787 | |
| Tryptone | Millipore-Sigma | T7293 | |
| Tween 20 | Millipore-Sigma | P9416 | |
| Yeast Extract | Millipore-Sigma | Y1625 | |
| Name | Company | Catalog number | Comments |
| Equipments | Maker/model | ||
| Programmable chilling incubator | Torrey Pines IN30 Incubator with Cooling | ||
| Shaker for bacterial growth | ATR Multitron shaker | ||
| Vacuum manifold with liquid waste trap | MultiScreenVacuum Manifold 96 well | ||
| 96 well autopippetor/liquid handler | Genmate or Biomek FX | ||
| Liquid dispenser | Wellmate | ||
| DNA microarrayer | Genetix QArray2 | ||
| Automatic hybridization station | Tecan HS4800 Pro Hybridization Station | ||
| Microarray scanner | Tecan PowerScanner | ||
| Microarray data quantification | Tecan Array-ProAnalyzer 6.3 |
参考文献
- Melnikova, I., Golden, J. Targeting protein kinases. Nature Review Drug Discovery. 3 (12), 993-994 (2004).
- Patterson, H., Nibbs, R., McInnes, I., Siebert, S. Protein kinase inhibitors in the treatment of inflammatory and autoimmune diseases. Clinical and Experimental Immunology. 176 (1), 1-10 (2014).
- Wu, P., Nielsen, T. E., Clausen, M. H. FDA-approved small-molecule kinase inhibitors. Trends Pharmacological Sciencies. 36 (7), 422-439 (2015).
- Druker, B. J., et al. Effects of a selective inhibitor of the Abl tyrosine kinase on the growth of Bcr-Abl positive cells. Nature Medicine. 2 (5), 561-566 (1996).
- Heinrich, M. C., et al. Inhibition of c-kit receptor tyrosine kinase activity by STI 571, a selective tyrosine kinase inhibitor. Blood. 96 (3), 925-932 (2000).
- Stagno, F., et al. Imatinib mesylate in chronic myeloid leukemia: frontline treatment and long-term outcomes. Expert Review Anticancer Therapy. 16 (3), 273-278 (2016).
- Ben Ami, E., Demetri, G. D. A safety evaluation of imatinib mesylate in the treatment of gastrointestinal stromal tumor. Expert Opinions in Drug Safety. 15 (4), 571-578 (2016).
- Ramachandran, N., et al. Self-assembling protein microarrays. Science. 305 (5680), 86-90 (2004).
- Festa, F., et al. Robust microarray production of freshly expressed proteins in a human milieu. Proteomics Clinical Applications. 7 (5-6), 372-377 (2013).
- Yazaki, J., et al. Mapping transcription factor interactome networks using HaloTag protein arrays. Proceedings of the National Academy of Sciences of the United States of America. 113 (29), E4238-E4247 (2016).
- Rauf, F., et al. Ibrutinib inhibition of ERBB4 reduces cell growth in a WNT5A-dependent manner. Oncogene. 37 (17), 2237-2250 (2018).
- Anderson, K. S., et al. Protein microarray signature of autoantibody biomarkers for the early detection of breast cancer. Journal of Proteome Research. 10 (1), 85-96 (2011).
- Wang, J., et al. Plasma Autoantibodies Associated with Basal-like Breast Cancers. Cancer Epidemiol Biomarkers Prevention. 24 (9), 1332-1340 (2015).
- Bian, X., et al. Tracking the Antibody Immunome in Type 1 Diabetes Using Protein Arrays. Journal of Proteome Research. 16 (1), 195-203 (2017).
- Song, L., et al. Identification of Antibody Targets for Tuberculosis Serology using High-Density Nucleic Acid Programmable Protein Arrays. Molecular and Cellular Proteomics. 16 (4 suppl 1), S277-S289 (2017).
- Wang, J., et al. Comparative Study of Autoantibody Responses between Lung Adenocarcinoma and Benign Pulmonary Nodules. Journal of Thoracic Oncology. 11 (3), 334-345 (2016).
- Montor, W. R., et al. Genome-wide study of Pseudomonas aeruginosa outer membrane protein immunogenicity using self-assembling protein microarrays. Infection and Immunity. 77 (11), 4877-4886 (2009).
- Tang, Y., Qiu, J., Machner, M., LaBaer, J. Discovering Protein-Protein Interactions Using Nucleic Acid Programmable Protein Arrays. Current Protocols in Cell Biology. 74, 11-15 (2017).
- Yu, X., et al. Copper-catalyzed azide-alkyne cycloaddition (click chemistry)-based detection of global pathogen-host AMPylation on self-assembled human protein microarrays. Molecular and Cellular Proteomics. 13 (11), 3164-3176 (2014).
- Anderson, K. S., et al. Autoantibody signature for the serologic detection of ovarian cancer. Journal of Proteome Research. 14 (1), 578-586 (2015).
- Woyach, J. A., Johnson, A. J., Byrd, J. C. The B-cell receptor signaling pathway as a therapeutic target in CLL. Blood. 120 (6), 1175-1184 (2012).
- Smith, M. R. Ibrutinib in B lymphoid malignancies. Expert Opinion on Pharmacotherapy. 16 (12), 1879-1887 (2015).
- Festa, F., Steel, J., Bian, X., Labaer, J. High-throughput cloning and expression library creation for functional proteomics. Proteomics. 13 (9), 1381-1399 (2013).
- Seiler, C. Y., et al. DNASU plasmid and PSI:Biology-Materials repositories: resources to accelerate biological research. Nucleic Acids Research. 42, D1253-D1260 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved