Method Article
자체 조립된 인간 단백질 마이크로어레이에서 키나제 억제제 스크리닝
요약
키나아제 억제제의 스크리닝을 위한 자가 조립된 인간 단백질 마이크로어레이의 생성을 위한 상세한 프로토콜이 제시된다.
초록
키나아제 억제제의 스크리닝은 약물의 특성을 더 잘 이해하고 임상적 의미를 갖는 잠재적으로 새로운 표적을 식별하는 데 매우 중요합니다. 몇몇 방법론은 그 같은 검열을 달성하기 위하여 보고되었습니다. 그러나, 각각은 그것의 자신의 한계가 있습니다 (예를 들면, 유일한 ATP 유사체의 검열, 정제된 키나아제 도메인을 사용하는 제한, 한 번에 몇몇 키나아제 이상 시험과 관련되었던 중요한 비용, 및 단백질 키나아제를 가진 검열에 있는 융통성 의 부족 새로운 돌연변이). 여기서, 이러한 제한사항 중 일부를 극복하고 키나아제 억제제의 편견 없는 스크리닝에 사용될 수 있는 새로운 프로토콜이 제시된다. 이 방법의 강점은 여러 단백질에 걸쳐 키나아제 억제제의 활성을 비교하는 능력입니다, 다른 키나아제 또는 동일한 키나아제의 다른 변이체 사이. 인간 기반의 체외 전사 및 번역 시스템(IVTT)에 의한 단백질 키나아제의 발현을 통해 생성된 자가 조립 단백질 마이크로어레이가 채용된다. 마이크로어레이에 표시된 단백질은 활성상태이며, 키나아제 억제제의 효과를 측정할 수 있습니다. 다음 절차에서는 마이크로어레이 생성 및 스크리닝에서 데이터 분석에 이르는 프로토콜 단계를 자세히 설명합니다.
서문
단백질 키나아제는 그들의 표적의 인산화에 책임 있고 많은 세포 기능을 통제하는 복잡한 분자 통로를 조절할 수 있습니다 (즉, 세포 증식, 분화, 세포 죽음 및 생존). 키나아제 활성의 규제 완화는 400개 이상의 질병과 관련이 있으며, 키나아제 억제제가 암, 심혈관 및 신경 장애뿐만 아니라 염증성 질환을 포함한 여러 질병의 치료에 사용할 수 있는 주요 약물 중 하나로 손대습니다. 및 자가 면역 질환1,2,3.
정밀 의학의 출현으로 새로운 치료법, 특히 키나아제 억제제의 식별은 제약 및 임상적으로 큰 매력을 가지고 있습니다. 키나아제 억제제의 de novo 설계 및 기존 FDA 승인 약물에 대한 새로운 표적의 식별을 포함하여 가능한 새로운 쌍의 키나아제/키나아제 억제제의 식별을 위해 여러 가지 접근법을 사용할 수 있습니다. 후자는 특히 매력적이다, 병원에서 이러한 약물을 구현하는 데 필요한 시간과 돈이 크게 이전 임상 시험 데이터의 가용성으로 인해 감소되기 때문에. 키나아제 억제제의 용도 변경의 정식 예는 imatinib, 처음에 BCR-Abl의 억제를 통해 만성 골수성 백혈병 (CML)의 치료를 위해 설계, 이는 또한 성공적으로 c-키트 과발현의 치료에 사용될 수있다 위장 기질 종양 (GISTs)4,5,6,7.
키나아제 억제제의 스크리닝은 결합 성 세포 또는 효소 계 세포계 검정에서 수행될 수 있다. 1차 검사는 단백질-약물 상호 작용에 초점을 맞추고 결찰 부위 및 친화성과 같은 정보를 제공할 수 있습니다. 이러한 해석의 시점에서 키나아제의 활성은 알려지지 않았기 때문에, 단백질의 형태 변화로 인해 다수의 상호작용이 놓치거나 잘못 규명될 수 있다. 한편, 효소 계 세포계 세포는 단백질 키나아제가 활성화되고 효소 활성에 대한 억제제의 효과에 관한 귀중한 정보를 제공해야 하지만, 이러한 유형의 스크리닝은 일반적으로 더 많은 시간과 비용이 소요된다. 현재, 두 가지 유형의 암세는 여러 소스에서 상업적으로 사용할 수 있습니다. 그(것)들은 몇몇 한계를 가진 키나아제 억제제의 검열을 위한 믿을 수 있는 선택권을 나타냅니다: I) 방법의 대부분은 다중 키나아제의 시험을 개별적으로 관련시킵니다, 이는 많은 단백질 세트의 검열을 비용이 많이 들게 할 수 있습니다; II) 시험되는 키나아제 세트는 미리 선택된 야생형 키나아제 및 몇몇 잘 알려진 일부 키나아제의 목록으로 제한되어 많은 새로운 돌연변이 이소폼의 시험을 방해한다.
이러한 맥락에서, 단백질 마이크로어레이는 시판되는 기술에 의해 제시된 몇 가지 한계를 극복할 수 있는 강력한 플랫폼이다. 관심 있는 임의의 서열의 전신, 활성 단백질을 사용하여 고처리량 스크리닝에서 효소 기반 검술을 수행하는 데 적합합니다. 마이크로어레이는 단백질이 세포에 적시에 발현되는 NAPPA (핵산 프로그래밍 가능한 단백질 배열)와 같은 자가 조립 된 접근법에 의해 생성 될 수 있으며, 어레이에 표시된 단백질이 실제로 활성화 될 가능성을 증가시다. NAPPA에 표시 하는 단백질 자연 접기 및 활동의 가능성을 개선 하기 위해 인간 파생 된 리보솜과 샤페론 단백질을 사용 하 여 생산.
단백질은 처음에 포착 제와 함께, 마이크로 어레이 표면에, 포획 태그와 융합관심의 유전자를 위해 코딩 하는 cDNAs를 인쇄하여 프로그램됩니다. 단백질은 체외 전사 및 번역(IVTT) 시스템을 사용하여 마이크로어레이상에서 생산되고, 갓 발현된 단백질은 포획제에 의해 마이크로어레이 표면에 고정된다. 발현 된 NAPPA 어레이는 어레이에 표시되는 단백질의 연구에 사용할 수 있습니다, 높은 처리량 방식8,9.
이전에는 NAPPA 어레이에 표시된 단백질이 알려진 파트너10과상호 작용하기 위해 제대로 접혀 있음을 보여주었습니다. 더욱이, 이들의 효소 활성은 2018년에 처음 악용되었고, 그 때 는 마이크로어레이 자기포스포릴레이트11에표시되는 단백질 키나아제들이 나타났다. 현재까지, NAPPA 방법론은 바이오 마커 발견12,13,14, 15,16,17, 등 많은 뚜렷한 응용 프로그램에 사용되어 왔다. 단백질-단백질 상호작용10,18,기질 식별19,약물 스크리닝11. 유연성은 각 응용 프로그램에 적응할 수 있는 플랫폼의 주요 특성 중 하나입니다.
여기서, 자가 조립된 NAPPA 어레이에서 티로신 키나아제 억제제의 스크리닝을 위한 프로토콜이 제시된다. 이 플랫폼은 활성 인간 단백질 키나아제의 표시와 낮은 배경 및 높은 동적 범위의 단백질 키나아제 활성 분석에 최적화되어 있습니다. 키나아제 억제제의 스크리닝을 위해 NAPPA를 사용하도록 구현된 변형 중에는: I) 인쇄 화학의 변화, II) 키나아제 억제제 스크리닝 전에 단백질 마이크로어레이의 탈인광, 및 III) 검출의 최적화가 포함된다. 어레이에 인산화 단백질. 이 프로토콜은 동종 최초이며 NAPPA 마이크로어레이의 키나아제 연구에 대한 고유한 정보를 제공합니다.
프로토콜
1. 일반적인 버퍼 및 솔루션 사용
- 결핵 매체를 준비: 훌륭한 국물 (24 g/L 효모 추출 물; 20 g/L 트립톤; 4 mL/L 글리세롤; 0.017 M KH2PO4;그리고 0.072 M K2HPO4). 0.017 M KH2PO4 및 0.072 M K2HPO4 솔루션은 10x 인산완충충(0.17 M KH2PO4 및 0.72 MK2HPO4)으로구매할 수 있다.
- LB 배지 준비: 루리아-베르타니 (5 g/L 효모 추출물; 10 g/L 트립톤; 10 g/L NaCl). 5 M NaOH로 pH를 7.0으로 조정합니다.
- 1x TBS 준비: 트리스 버퍼링 식염수 (TBS: 50 mM Tris-Cl, pH = 7.5; 150 mM NaCl).
- 1x TBST 준비: 0.1% 트웬 20으로 보충된 TBS.
2. DNA 준비
참고 : NAPPA 어레이에 사용되는 DNA는 매우 순수해야합니다. 따라서 상업용 DNA 미니 프렙은 권장되지 않습니다. 현재, DNA 준비를 위한 2개의 프로토콜이 이용됩니다: 집에서 높은 처리량 미니 준비 (여기에 기술된) 또는 상업적인 Midi- 또는 Maxi 준비. 사내 미니 준비 프로토콜의 평균 처리량은 1인당 하루 1,500개의 샘플입니다.
- 사내 고처리량 미니 준비를 위한 세균 성장
- LB /한천 옴니 플레이트를 준비합니다. LB 한천 30-40 mL을 부어 (LB 미디어에 1.5 % 양성 클론 선택을위한 항생제로 보충 된 세균 한 천) 각 단일 웰 플레이트에.
- LB/한천 플레이트에 글리세롤 스톡을 발견하십시오. LB 매체에 글리세롤 스톡을 희석하십시오 (1 :300, 보통 2 μL에서 600 μL의 LB). 10 분 동안 흔들어. 희석 된 스톡의 3 μL을 LB / 한천 접시에 놓습니다. 37 °C에서 배양, 거꾸로, 하룻밤.
- 문화를 접종합니다. 80% 에탄올 및 화염으로 멸균된 96핀 장치를 사용하여, 항생제로 보충된 결핵 배지의 웰당 1.5 mL로 한천판에서 배양을 접종하였다.
- 배양 배양. 가스 투과성 씰로 블록을 덮고 셰이커에 따라 37 °C, 300-800 rpm에서 22-24 시간 동안 배양하십시오.
참고: 800rpm으로 설정된 셰이커는 이 배양에 최적입니다. 느린 속도 셰이커를 사용하면 조밀한 배양이 줄어들고 DNA 정화 수율이 낮아질 수 있습니다. - 펠릿 문화. 3,800 x g및 4 °C에서 30 분 동안 블록을 스핀하십시오.
- 사내 고처리량 미니 준비
참고: 멀티 채널 파이펫터 또는 자동 디스펜서를 사용하여 사내 고처리량 미니 프렙을 수행할 수 있습니다. 자동 디스펜서를 사용하는 경우 사용하기 전에 그리고 솔루션 사이에 시스템을 청소해야 합니다.- 미니 준비 중에 사용되는 모든 솔루션을 준비합니다.
- 솔루션 준비 1: TE 재서스펜션 버퍼(50 mM Tris, pH = 8.0; 10 mM EDTA, pH = 8.0; 0.1 mg/mL RNAe). 4 °C에서 보관하십시오.
- 솔루션 준비 2: NaOH/SDS 용해 버퍼(0.2 M NaOH; 1% SDS). 더 나은 결과를 얻으려면 갓 만든 솔루션을 사용해야합니다.
- 솔루션 준비 3: KOAC 중화 버퍼(2.8M KOAc). 빙하 아세트산으로 용액의 pH를 5.1로 조정합니다. 4 °C에서 보관하십시오.
- 솔루션 N2 준비: 평형 버퍼 (100 mM Tris; 900 mM KCl; 15% EtOH; 0.15% 트리톤 X-100). 인산으로 용액의 pH를 6.3으로 조정합니다.
- 솔루션 N3 준비: 배쉬 버퍼 (100 mM Tris; 1.15 M KCl; 15% EtOH). 인산으로 용액의 pH를 6.3으로 조정합니다.
- 용액 N5 준비: 용출 버퍼 (100 mM Tris; 1 M KCl; 15% EtOH). 인산으로 용액의 pH를 8.5로 조정합니다.
참고: 음이온 교환 동안 DNA 결합, 세척 및 용출의 성공적인 제어는 완충된 KCl 농도 및 pH 값에 크게 의존한다. 신중한 버퍼 부품 측정 및 pH 조정이 필수적입니다. 설명된 측정값의 작은 편차는 수율의 상당한 손실을 초래할 수 있습니다.
- 펠릿을 다시 일시 중단합니다. 용액 1의 200 μL을 추가하고 RT에서 5 분 동안 2,000 rpm에서 흔들어 주세요. 성공적인 용해를 위해 펠릿의 완전한 재 현탁액이 필요합니다. 필요한 경우 블록을 소용돌이.
- 박테리아를 lyse. 용액 2의 200 μL을 추가하고 알루미늄 씰로 플레이트를 밀봉하고 5 x를 반전하십시오. 신중하게 솔루션 2 추가의 시작 부분에서이 단계를 시간. 5분을 초과하지 마십시오.
- 솔루션을 중화합니다. 용액 3의 200 μL을 추가하고 알루미늄 씰로 플레이트를 밀봉하고 5 x를 반전하십시오. 물개는 용해/중화 버퍼로 인해 느슨할 수 있으므로 반전할 때는 주의하십시오. 용액이 씰에 닿지 않는 부분 반전은 시료 간의 교차 오염을 방지하는 것이 좋습니다.
- 명확한 리세이트. 3,800 x g 및 4 °C에서 30 분 동안 플레이트를 원심 분리합니다.
- 용해 펠릿 원심 분리 단계 동안 용해 교환 수지 슬러리를 준비합니다. 1L 병을 사용하여 음이온 교환 수지로 300 mL 마크에 도달 할 때까지 채운 다음 용액 N2를 최대 900 mL까지 추가하십시오.
주의 : 이 단계는 실리카 흡입으로부터 보호하기 위해 후드에서 수행해야합니다. - 아니온 교환 수지 플레이트를 준비합니다. 폐기물 수거 용기역할을 하기 위해 깊은 우물 블록 위에 필터 플레이트를 쌓습니다. 음이온 교환 슬러리가 균일 할 때까지 혼합 한 다음 유리 트로프에 부어 넣습니다. 넓은 지루한 P1000 팁을 사용하여 슬러리의 450 μL을 필터 플레이트의 각 우물에 전달합니다.
- 원심 분리기 는 130 x g 및 RT에서 5 분 동안 느린 가속으로 플레이트 (수지 플레이트 / 깊은 우물 플레이트)를 쌓아 놓습니다.
- 수지 플레이트 / 깊은 우물 블록 스택에 리세이트 상판을 전송합니다. 느린 램프 업 속도로 30 x g에서 5 분 동안 적층 된 플레이트를 회전시킵니다.
- 열을 씻으시고. 400 μL의 용액 N3(세척 버퍼)을 각 웰에 추가합니다. 수지 플레이트를 진공 매니폴드로 이송하여 세척 버퍼를 제거합니다. 세탁 단계를 3x 반복합니다. 마지막 세척시, 모든 우물이 제대로 비워졌는지 확인하십시오. 스택 플레이트를 150 x g에서 5분 동안 회전하여 잔류 버퍼를 제거합니다.
- DNA를 용해. 수지 플레이트를 깨끗한 800 μL 컬렉션 플레이트에 놓습니다. 각 우물에 300 μL의 용액 N5를 추가합니다. RT에 10 분 동안 앉은 다음 느린 램프 업 속도로 20 x g에서 5 분 동안 쌓인 플레이트를 회전시킵니다. 233 x g에서1 분 동안 적층 된 접시를 돌이되다.
- DNA를 정량화하고 플레이트를 -20°C에서 저장하여 추가 사용하거나 DNA 침전으로 바로 진행합니다.
참고: 샘플당 최소 30 μg의 DNA가 필요합니다. DNA 수율이 낮은 경우, DNA 미니 프렙을 반복하거나, 또는 침전 단계(섹션 2.3) 동안 두 판을 조합하는 것이 좋습니다.
- 미니 준비 중에 사용되는 모든 솔루션을 준비합니다.
- DNA 침전
- 플레이트를 해동하고, DNA 용액을 균질화하기 위해 소용돌이를 만들고, 우물 바닥에 있는 모든 용액을 수집하기 위해 30s동안 230 x g에서 회전한다.
- 각 우물에 40 μL의 3 M NaOAc 및 240 μL의 이소프로판올을 추가합니다. 알루미늄 씰로 접시를 덮고 3x를 뒤집어 섞습니다.
- 3,800 x g 및 25 °C에서 30 분 동안 플레이트를 원심 분리합니다. 조심스럽게 상급을 폐기하십시오.
참고 : 두 판을 결합하려면 두 번째 플레이트에서 DNA를 첫 번째 플레이트에서 펠릿으로 옮기고 2.3.2-2.3.3 단계를 반복합니다. - DNA를 씻고 침전시. 각 우물에 80% 에탄올의 400 μL을 추가하십시오. 알루미늄 씰로 접시를 밀봉하고 30 분 동안 1,000rpm에서 흔들어 줍니다. 25 °C에서 30 분 동안 3,800 x g에서 원심 분리기. 상급제는 버리십시오.
- DNA 펠릿을 건조시. 접시를 종이 타월에 비스듬히 거꾸로 놓고 우물 바닥에 알코올이 없을 때까지 1-2 시간 동안 건조시키십시오. 230 x g에서 2 분 동안 밀봉 및 원심 분리기를 사용하여 펠릿을 내려 오십시오.
- 플레이트가 건조되면 알루미늄 씰로 밀봉하고 나중에 사용하기 위해 -20 °C에서 얼리거나 DNA를 계속 일시 중단하십시오 (4.1 단계).
3. 아미노일란 슬라이드 코팅
- 유리 슬라이드를 금속 랙에 놓습니다. 각 슬라이드를 시각적으로 검사하여 긁힘이나 결함이 없는지 확인합니다.
- 흔들면서 15 분 동안 코팅 용액 (아세톤에 2 % 아미노 슬레인 시약)에 잠긴 슬라이드. 아미노일란 용액은 폐기되기 전에 각각 30개의 슬라이드 두 랙을 코팅하는 데 사용할 수 있습니다.
- 헹위 합니다. 슬라이드 랙을 아세톤 워시(99% 아세톤)에 담그고 앞뒤로 흔들어 서 빠르게 5배 위아래로 움직입니다. 한 쪽 모서리로 기울여 물방울을 떨어뜨린 다음 울트라퓨어 워터에 빠르게 5배 빠르게 담급전히 담급합니다. 기울어서 떨어뜨립니다.
참고: 아세톤 워시는 두 번 사용할 수 있으며, 초순수는 매번 교체해야 합니다. - 모든 물방울이 제거 될 때까지 약 3 분 동안 모든 각도에서 불어, 압력 공기를 사용하여 건조 슬라이드. 코팅된 슬라이드를 RT에 단단히 밀봉된 상자 안에 금속 랙에 보관하십시오.
4. 배열 샘플 준비
- 20 μL의 초순수에서 사내 미니 프렙(단계 2.3.6)에서 DNA 펠릿을 다시 일시 중단하고 2시간 동안 1,000 rpm에서 흔들어 줍니다. midi/max 준비 DNA의 경우 각 샘플을 1.5 μg/μL의 최종 농도로 희석하고 20 μL을 800 μL 수집 플레이트로 옮긴다.
- 인쇄 믹스를 준비합니다. 96개의 웰 플레이트에 대해 인쇄 믹스 1mL[초순수 237.5 μL, 폴리리신 500 μL(0.01%); BS3 의 187.5 μL(비스-설포수니미딜, DMSO에서 50 mg/mL)과 75 μL의 폴리클로날 항항항을 준비합니다.
참고 : 화학 물질은 강수량을 피하기 위해 지정된 순서로 추가해야합니다. - 각 샘플에 10 μL의 인쇄 믹스를 추가하고 알루미늄 호일로 접시를 밀봉하고 RT에서 1,000 rpm에서 90 분 동안 흔들어줍니다. 플레이트를 4°C에서 밤새(~16시간) 보관합니다.
- 인쇄 일에, 잠시 소용돌이와 접시를 회전. 각 샘플의 28 μL을 384 어레이 플레이트로 이송합니다. 이 전송은 자동화 또는 다중 채널 파이펫을 사용하여 수행 할 수 있습니다. 384 어레이 플레이트에서 샘플의 위치를 추적하는 것이 중요합니다.
- 접시를 잠시 아래로 돌리면 거품이 제거됩니다. 호일로 접시를 밀봉하십시오.
5. NAPPA 어레이 생성 : 마이크로 어레이 인쇄
참고: 모든 인쇄 조건은 장비 및 재료 표에나열된 기기에 최적화되었습니다. 다른 배열을 사용하는 경우 추가 최적화가 필요할 수 있습니다.
- 배열기 정리. 시작하기 전에 모든 폐기물 탱크를 비우고 필요한 경우 초순수 또는 80 % 에탄올로 저수지를 리필하십시오. 보풀이 없는 물티슈와 초순수로 핀을 하나씩 청소합니다. 보풀이 없는 물티슈로 핀을 말려서 어레이헤드에 조심스럽게 넣습니다.
- 배열자 설정: 인쇄 사양 [잉크당 최대 스탬프 수: 1; 스팟당 스탬프 수: 1; 멀티 스탬프 타이밍: --; 스탬프 시간(ms): 0 ms; 수동 입력 시간(ms): 0 ms: 0 ms; 인쇄 깊이 조정: 90 미크론; 터치 오프 수: 0]. 살균 프로토콜 : 건조 시간 0 ms와 대기 시간 500 ms와 2,000 ms에 대한 초순수 물 세척; 이 단계를 6x 반복합니다. 건조 시간 1,200 ms와 대기 시간의 500 ms와 2,000 ms에 대한 80 % 에탄올세척 다음; 이 단계를 6x 반복합니다.
- 슬라이드 디자인: 원하는 배열 패턴으로 어레이거를 설정합니다. 디자인은 여러 가지 요소 (즉, 각 샘플에 대한 복제본 수, 위치 및 컨트롤 기능 수, 배열 레이아웃 (한 블록, 여러 개의 동일한 블록), 인쇄 할 배열 수, 실행 길이 등)을 고려해야합니다.
- 아미노일레인 코팅 슬라이드(단계 3.4)를 어레이 데크에 놓습니다. 진공이 모든 슬라이드를 제자리에 단단히 고정하고 있는지 확인합니다. 가습기를 시작합니다 (60 %로 설정해야합니다)
- 배열 갑판에 384 웰 플레이트를 놓습니다. 프로그램을 시작합니다.
- 마이크로어레이에 레이블을 지정합니다. 인쇄가 완료되면 각 슬라이드의 아래쪽(인쇄되지 않은) 쪽에 슬라이드 레이블을 놓습니다. 데크에서 슬라이드 인쇄 순서를 숫자 순서로 유지합니다.
- 인쇄된 어레이를 RT에 실리카 패킷이 있는 단단히 밀봉된 상자 안에 금속 랙에 보관하십시오. 건조한 환경에서 보관된 슬라이드의 유효 기간은 최대 1년까지입니다.
- (선택 사항): 동일한 샘플을 사용하여 90개의 슬라이드의 두 번째 배치를 인쇄할 수 있습니다. 이렇게 하려면 플레이트 인쇄가 완료되는 즉시 어레이 데크에서 384 웰 플레이트를 제거합니다. 접시를 4 °C에서 밀봉하고 보관하십시오. 배열의 첫 번째 배치가 완전히 완료 된 후, 갑판에서 그들을 제거, 새로운 아미노 일레인 코팅 슬라이드를 배치하고 새로운 실행을 시작합니다. 각 384 웰 플레이트는 사용 전에 30 분 동안 RT에 있는지 확인하십시오. 샘플당 4개 이상의 복제본이 슬라이드 배치로 인쇄되는 경우 384개의 웰 플레이트를 두 개의 플레이트로 분할하여 어레이 데크에 소요되는 시간을 줄여 샘플 증발을 줄이는 것이 좋습니다.
참고: 두 번째 실행이 시작되기 전에 모든 저장소를 확인합니다.
6. NAPPA 슬라이드에 DNA의 검출
- 슬라이드를 차단합니다. 슬라이드를 파이펫 상자에 넣고 블로킹 버퍼 30mL를 추가합니다. 흔들쉐이커에서 RT에서 1시간 동안 인큐베이션합니다.
- 슬라이드에 얼룩을 지십시오. 차단 용액을 버리고 20 mL의 차단 버퍼와 형광 DNA 간질 염료 33 μL을 추가합니다. 교반으로 15 분 동안 배양하십시오. 그런 다음 초순수로 슬라이드를 빠르게 헹구고 압력 공기로 건조시십시오. 스캔을 진행합니다(섹션 11).
7. NAPPA 슬라이드의 표현
- RT의 흔들 쉐이커에서 블로킹 버퍼로 슬라이드를 1시간 동안 차단합니다.
- 초순수로 슬라이드를 헹구고 여과된 압축 공기로 건조시면 됩니다. 제조업체의 지침에 따라 각 슬라이드에 밀봉 개스킷을 적용합니다.
- IVTT 믹스를 추가합니다. 각 슬라이드에는 150 μL의 IVTT 혼합물이 필요합니다. DEPC 물 33 μL에서 82.5 μL의 HeLa 용해물을 희석하고 16.5 μL의 액세서리 단백질과 33 uL의 반응 혼합물로 보충하십시오. 비 라벨 또는 비 시편 끝에서 IVTT 혼합물을 추가합니다. 천천히 믹스를 파이펫 (그것은 입구 끝에서 일시적으로 구슬 경우 허용). IVTT 혼합물이 퍼지고 어레이의 전체 영역을 덮을 수 있도록 밀봉 개스킷을 부드럽게 마사지하십시오. 두 포트모두에 작은 원형 포트 씰을 적용합니다.
- 슬라이드를 지지대위에 놓고 프로그래밍 가능한 냉각 인큐베이터로 옮김을 옮김으로 옮김을 배치합니다. 단백질 발현을 위해 30°C에서 90분 동안 배양한 다음, 질의 단백질의 고정화를 위해 15°C에서 30분씩 배양하였다.
- 슬라이드를 씻고 차단합니다. 밀봉 개스킷을 제거하고 단백질 디스플레이를 위해 5% 우유로 보충된 1x TBST의 약 30 mL로 파이펫 상자에 슬라이드를 담그거나(섹션 8) 또는 키나아제 검사 또는 약물 스크리닝을 위해 3% 소 혈청 알부민(BSA)으로 보충된 1x TBST를 보충하였다. RT에서 20 분 동안 교반하여 배양하고이 단계를 2x 반복하십시오.
8. NAPPA 어레이에서 단백질 검출
- 1 차 적인 항체를 추가합니다. 차단 용액에서 슬라이드를 제거하고(7.5단계) 종이 타월을 사용하여 뒷면(인쇄되지 않은 면)을 부드럽게 건조시고 있습니다. 슬라이드를 지지체위에 놓고 1x TBST + 5% 우유에 1:200희석된 1차 항체(마우스 항플래그)의 600 μL을 적용한다. RT에서 1 시간 동안 배양하십시오.
- 흔들쉐이커에 1x TBST + 5% 우유로 슬라이드를 씻으십시오(각 5분당 3x).
- 이차 항체를 추가합니다. 세탁용액에서 슬라이드를 제거하고 종이 타월로 뒷면을 부드럽게 건조시면 됩니다. 슬라이드를 지지체위에 놓고 1x TBST + 5% 우유에 1:200을 희석한 이차 항체(cy3-labbeled 항마우스 항체)의 600 μL을 적용합니다. 빛으로부터 슬라이드를 보호하고 RT에서 1 시간 동안 배양하십시오.
- 흔들쉐이커에서 1x TBST로 슬라이드를 세척합니다(각 5분당 3x). 초순수로 슬라이드를 빠르게 헹구고 압력 공기를 사용하여 건조시십시오. 스캔을 진행합니다(섹션 11).
9. NAPPA 어레이에 티로신 키나아제 억제제 스크리닝
참고: 동일한 실험에서 여러 슬라이드를 처리할 수 있지만 각 단계에서 한 번에 하나의 슬라이드가 처리되고 단계 간에 건조되지 않도록 해야 합니다. 슬라이드의 비레이블 또는 비시편한 끝에 모든 솔루션을 추가합니다.
- 약물 스크리닝 중에 사용되는 모든 솔루션을 준비하십시오.
- 다음을 결합하여 인산염/DNase 용액을 준비한다: 1x 단백질 메탈로-인산염 완충제(50 mM HEPES, 100 mM NaCl, 2 mM DTT, 0.01% Brij 35 에서 pH=7.5); 1 mM MnCl2; 람다 단백질 인산염의 8,000 단위; 및 DNase I. 각 마이크로어레이에 대해 용액의 400 μL을 준비한다. 사용하기 직전에 인산염과 DNase를 추가하십시오.
- 약물 희석을 하십시오. 약물은 10 mM의 최종 농도로 DMSO에서 재구성된다. 어레이에서 테스트된 모든 약물 농도를 보장하기 위해, 동일한 부피의 DMSO(DMSO의 10,000x 스톡)가 각 농도에 대해 생성되고 -80°C로 유지되도록 한다. 사용 시, 약물은 물에 1:100 희석됩니다.
- 다음을 결합하여 약물/키나아제 용액을 준비한다: 1x 키나아제 완충제(pH=7.5의 25 mM Tris-HCl); 5 mM 베타-글리세로인산염; 2 mM DTT; 0.1 mM Na3VO4; 10 mM MgCl2; 500 μM ATP; 그리고 2 μL의 약물 (물에 희석 1 :100). 각 마이크로어레이에 대해 용액의 200 μL을 준비합니다.
- 인산염 및 DNase 치료를 수행합니다. 차단 용액에서 슬라이드를 제거하고(7.5단계) 종이 타월을 사용하여 뒷면을 부드럽게 말립니다. 슬라이드를 지지대위에 놓고 200 μL의 인산염/DNase 용액을 적용합니다. 증발을 방지하기 위해 마이크로어레이 커버슬립을 놓습니다. 오븐에서 45 분 동안 30 °C에서 배양하십시오.
- 인산염 및 DNase 처리 II: 오븐에서 어레이를 제거하고, 커버슬립을 버리고, 과잉 용액을 제거하고, 갓 만든 인산염 및 DNase 용액 200 μL을 적용합니다. 커버 슬립으로 마이크로어레이를 덮고 오븐에서 30°C에서 45분 간 배양합니다.
- 흔들쉐이커에 1x TBST + 0.2 M NaCl로 슬라이드를 세척하십시오(각 5분당 3x).
- 약물 치료 및 키나아제 반응을 수행한다. 세탁용액에서 슬라이드를 제거하고 종이 타월로 뒷면을 부드럽게 말립니다. 슬라이드를 지지대위에 놓고 200 μL의 약물/키나아제 용액을 적용합니다. 증발을 피하기 위해 덮개 슬립을 위에 놓습니다. 오븐에서 30 °C에서 1 시간 동안 배양하십시오.
- 흔들쉐이커에 1x TBST + 0.2 M NaCl로 슬라이드를 세척하십시오(각 5분당 3x).
- 1차 항체인 포쇼-티르 항체를 사용하여 8.1-8.4단계를 반복하여 1:100을 희석한다. 모든 단계에서 1x TBST + 5% 우유를 1x TBST + 3% BSA로 교체하십시오.
10. 자동화된 하이브리드화 프로토콜
참고: 또는 하이브리드화 스테이션을 사용하여 NAPPA 어레이(섹션 7-9)의 모든 하이브리드화 및 세차기를 자동화할 수 있으며 프로토콜은 보충 파일 1로제공됩니다.
11. 이미지 획득
참고: 마이크로어레이 이미지는 20미크론 이상의 해상도로 획득해야 합니다.
- 마이크로어레이를 슬라이드 홀더 매거진에 넣고 단백질을 위로 향합니다. 매거진을 마이크로어레이 스캐너에 로드합니다.
- 575/30 nm 방출 필터가 있는 녹색 레이저를 선택하여 cy-3 라벨이 부착된 이차 항체에서 신호를 스캔합니다. 다른 형광섬유를 사용하는 경우 올바른 레이저/파장을 선택하여 형광 염료의 신호를 감지합니다.
- 각 이미지의 이름과 저장할 위치를 정의합니다.
- (선택 사항): 각각의 새로운 형광에 대해 스캐닝 조건의 최적화를 통해 신호 강도의 선형 범위를 감지하는 것이 좋습니다. 이렇게 하려면 다양한 광증선(PMT)을 사용하여 마이크로어레이를 스캔하고 채도가 낮은 신호와 낮은 배경으로 선명한 이미지를 얻을 때까지 이득합니다.
- 최적화된 설정으로 모든 마이크로어레이를 스캔하고 자동 게인을 끄는 것을 기억하십시오.
참고: 데이터 분석을 위해 모든 마이크로어레이는 동일한 스캔 설정을 사용하여 스캔해야 합니다. cy3를 형광포로 사용하는 키나아제 검소의 경우, 장비 및 재료 표에 나열된 스캐너를 사용하여 20% PMT, 레이저 강도 25%, 해상도 10미크론으로 이미지를 스캔합니다.
12. 데이터 처리 및 분석
참고: 유사한 기능을 가진 마이크로어레이 데이터의 정량화를 위해 여러 소프트웨어 패키지를 사용할 수 있습니다. 여기에 설명된 절차는 장비 및 재료 표에나열된 소프트웨어를 위해 설계되었습니다.
- 정량화할 TIFF 파일을 로드하고, 마이크로어레이 레이아웃과 일치하도록 그리드를 설계하고, 스팟의 크기를 조정하여 전체 신호를 가능한 최소 영역으로 통합합니다. 인접한 지점이 겹치지 않아야 합니다. 소프트웨어가 얼마나 잘 수행되고 있는지 시각적으로 검사하고 필요한 경우 수동으로 그리드를 조정합니다.
- 마이크로어레이의 신호 강도를 정량화합니다. 비정상(비특이적 바인딩, 먼지 등)이 있는지 를 육안으로 검사하고 데이터 분석에서 제거합니다.
- 자리가 없는 배열에서 인접 영역의 신호를 사용하여 배경을 로컬로 수정합니다.
- 데이터를 정규화합니다. 서로 다른 어레이에서 신호를 비교하려면 각 마이크로어레이의 신호 강도를 정규화해야 합니다. 이상값을 제외하려면, 탈인된 마이크로어레이의 양성 대조군(IgG spots)으로부터 신호의 평균 30%를 트리밍하여 데이터를 정규화한다.
참고: IgG 스팟의 신호는 마이크로어레이의 인산화 및 탈인 동안 변경되지 않으며 정규화에 적합합니다. - 활성 키나아제 식별. 마이크로어레이에 표시되는 각 피쳐에 대해 자동 인포스포릴레이션 어레이와 탈인화물 배열의 정규화된 신호 강도 사이의 비율을 계산합니다. 활성 키나아제 식별을 위해 임계값을 1.5배 변경하여 설정하고 다른 모든 피처를 자동 인산화(N/A)를 수행할 수 없음으로 표시합니다.
- 12.5단계에서 확인된 각 키나아제의 활성을 조정된 신호의 백분율로 계산한다(정규화된 포지티브 어레이(DMSO)의 신호 세기는 정규화된 음극 대조군 어레이(dephosphorylated)의 신호 강도에 의해 감산된다.
결과
자가 조립된 NAPPA 마이크로어레이는 바이오마커 발견, 단백질-단백질 상호 작용, 기판 식별 및 약물 스크리닝10,11을 포함한 많은 뚜렷한 응용 분야에 사용될 수 있는 견고한 플랫폼을 제공합니다. ,12,13,14,15,16 ,17,18,19,20.
NAPPA 마이크로어레이에 대한 티로신 키나아제 억제제의 키나아제 활성 및 스크리닝에 대한 연구를 위해 채택된 전체 방법론은 도 1에개략적으로 나타난다. 첫째, NAPPA 마이크로어레이는 코팅된 마이크로어레이상에 cDNA 및 포획제의 고정화에 의해 생성된다. 이들은 인간 기반 IVTT 시스템을 사용하여 단백질의 전사 및 번역을 위한 템플릿으로서 사용된 cdDNA를 사용하고, 새로 합성된 단백질은 포획제9에의해 고정화된다. 인쇄된 마이크로어레이의 품질은 어레이에 표시되는 DNA 수준(일관된 인쇄 확인) 또는 단백질(단백질 발현 및 포획 확인)을 측정하여 모니터링할 수 있다. 그림 1). 배경 신호를 줄이고 실험의 동적 범위를 증가시키기 위해 마이크로 어레이는 1) 람다 인산염으로 처리되어 Ser/Thr/Tyr 잔류물에서 인산화를 제거한 다음 2) DNase를 사용하여 그 자리에서 화학을 단순화하고 감소시 배경(그림 1).
다음 단계는 마이크로어레이가 ATP(탈인식 마이크로어레이라고 함)가 없는 상태에서 키나아제 버퍼로 배양되는 오토포스포릴레이션 반응이며, 키나아제 버퍼는 ATP(양성 대조군, 자동 인포릴레이트 어레이 또는 ATP + DMSO(차량 제어)라고 합니다. 이 단계에서는 키나아제가 첨가되지 않는다는 점을 강조해야 합니다. 따라서, 마이크로어레이상에 표시된 각 키나아제의 본질활성은 팬 항인-티로신 항체를 이용한 그의 인산화 수준의 측정을 통해 정량화되고 그 뒤에 cy3-labbeled 이차 항체가 뒤따른다(도1).
네 배로 인쇄된 인간 단백질 키나아제 패널을 표시하는 NAPPA-키나아제 어레이의 품질 관리는 도 2에나타내고 있다. 고정화 된 DNA의 수준은 DNA 염색에 의해 측정되었고 마이크로 어레이에 걸쳐 균일 한 신호를 보였으며 어레이에 인쇄 된 DNA의 양이 균일했음을 시사했습니다. DNA 염색없이 여러 가지 특징을 관찰 할 수도 있습니다. 이러한 특징은 DNA가 인쇄 믹스에서 생략된 일부 컨트롤에 해당합니다 [즉, 빈 반점 (아무것도 인쇄되지 않았습니다), 물 반점, 정제 된 IgG 스팟 (폴리 리신, 가오리너 및 정제 IgG), 인쇄 믹스 만 (전체 인쇄 믹스 : 폴리 리신 플러스 크로스 링커 및 안티 플래그 항체, 어떤 DNA없이)].]. NAPPA-키나아제 마이크로어레이에 표시된 단백질의 수준은 항태그 항체를 사용하여 IVTT 반응 후에 평가되었다.
키나아제 스크리닝의 경우, 플래그는 항플래그 항체를 사용하여 마이크로어레이상에 표시된 단백질의 수준을 선택및 표기의 태그로서 사용하였으며 측정되었다. 도시된 바와 같이, cDNA를 포함하는 반점의 대다수는 성공적으로 단백질의 검출 가능한 수준을 표시했습니다. cDNA가 없는 몇몇 대조군 은 또한 항기 항체를 가진 신호를 밝혔다: IgG 스팟(이차 항체의 활성을 검출하는 데 사용) 및 빈 벡터 반점(태그전용 cDNA 코드)(그림2). NAPPA-키나아제 마이크로어레이는 슬라이드 들 사이에서 좋은 재현성을 보였으며, 뚜렷한 인쇄 배치 들 간의 단백질 표시 수준의 상관 관계가 0.88보다 높았습니다(그림 2). 동일한 배치 내에서 상관 관계는 0.92에 가깝게 훨씬 더 높았습니다(데이터가 표시되지 않음).
다음으로, 어레이상에 표시된 단백질의 키나아제 자가포스포릴화 활성을 항인-티로신 항체를 사용하여 측정하였다(도3). 어레이 상에 표시된 단백질은 발현 후 높은 수준의 단백질 인산화를 나타내었으며(도3,왼쪽), 이는 IVTT 혼합물에 존재하는 어레이 또는 활성 키나아제에 의해 서식상에 표시된 단백질의 본질적인 키나아제 활성에 의해 야기될 수 있다. 이러한 인산화는 람다 인산아제 처리로 완전히 제거되었고 이들 마이크로어레이는 키나아제 아세에 사용되었다. 탈인 후, ATP 없이 수행된 자가포스포릴화 반응은 예상대로 상당한 수준의 인산화를 보이지 않았으며, ATP가 있는 키나아제 버퍼로 배양된 마이크로어레이는 15분 빠른 단백질 인산화를 보였다( 그림 3). 약물 스크리닝의 경우, 키나아제 활성은 시험된 키나아제의 수를 최대화하기 위해 60분 의 자가포스포릴화 반응 후에 측정되었다.
단백질 발현 직후인 인산화 수준을 측정한 마이크로어레이(그림3,왼쪽)와 60분 후의 자가포스포릴화반응(그림 3,오른쪽)의 비교는 단백질 인산화만을 보였다. 발현 후, 그들은 IVTT 혼합물에 존재하는 단백질에 의해 외인성 인산화 될 수 있음을 시사하지만, 자가 포설 될 수 없다; ii) 단백질 인산화 반응 후에만 인산화, 이들 단백질이 단백질 발현 후 활성상태가 아니었으며, 활성이 되기 위해 키나아제 완충액에 존재하는 공동 인자가 필요함; 또는 iii) 두 어레이에 인산화된 단백질은 두 설정 모두에서 활성상태였다는 것을시사한다(도 3).
NAPPA-키나아제 어레이에 티로신 키나아제 억제제의 스크리닝을 위해 얻은 결과의 예로서 단백질 키나아제 전반에 걸쳐 뚜렷한 선택성을 가진 3개의 키나아제 억제제가 사용되었다: 스타우로스포린, 이마티닙 및 이브루티닙. 모든 스크리닝에 대해, 탈인성 NAPPA 마이크로어레이는 자가포스포릴화 반응 동안 TKI(100 nM에서 10 uM까지)의 증가농도로 배양하였다. 시험된 첫 번째 TKI는 글로벌 단백질 키나아제 억제제인 스타우로스포린으로, 거의 모든 키나아제 시험에서 마이크로어레이에 강력한 키나아제 억제를 보였다11.
다음으로, imatinib는 만성 골수성백혈병 및 위장기질 종양4,5,6,7의치료에 사용되는 ABL 및 c-Kit 억제제를 시험했다. NAPPA-키나아제 어레이에서 imatinib는 Abl1 및 BCR-Abl1 활성에서 유의한 감소를 보였고 다른 키나아제는 대부분 영향을 받지 않은 채로 남아있었다(그림 4A). 키나아제 활성에 대한 데이터 정량화는 탈인성 어레이에 대해 정규화되었고 양성 대조마이크로어레이의 백분율로 나타났다(비히클 만). TNK2(비관련 키나아제), Abl1 및 BCR-Abl1에 대한 데이터는 도 4B에나타내었다. 예상대로, imatinib는 Abl1 및 BCR-ABl1을 향한 선택적 억제를 보였다. c-Kit에 대한 데이터는 포지티브 컨트롤 어레이에 대한 활동 부족으로 인해 결정적이지 않았습니다.
마지막으로, ibrutinib, 브루톤의 티로신 키나제의 FDA 승인 공유 억제제 (BTK), 테스트. Ibrutinib는 현재 만성 림프구성 백혈병 (CLL), 맨틀 세포 림프종 및 발덴스트롬의 거시 글로볼린증21,22를포함하는 과민성 BTK를 가진 몇몇 혈액 관련 암의 처리에서 이용됩니다. 도 4C는,ibrutinib 스크리닝을 위해 얻어진 전형적인 결과를 대표한다. ABL1(비관련 키나아제) 및 BTK(정식 표적) 및 ERBB4(잠재적 신규 표적)의 키나아제 활성은 도 4D에도시되어 있다. 데이터는 ERBB4가 특정 한 방식으로 ibrutinib에 의해 억제 될 수 있음을 시사한다. 이러한 억제는 시험관내 및 세포계세포계세포계 세포계 세포계 세포계 세포에서 확인되었으며, 이 플랫폼의 힘을 입증했다.
종합하면, 데이터는 NAPPA-키나아제 마이크로어레이 플랫폼이 TK 억제제의 편견 없는 스크리닝에 사용될 수 있음을 시사한다. 더욱이, 스크리닝은 빠르며 관심 있는 단백질 키나제의 임의의 변이를 포함하도록 용이하게 맞춤화될 수 있다.
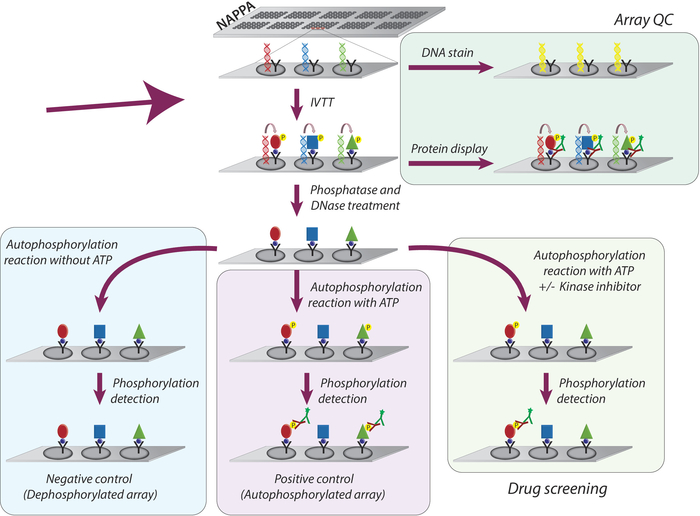
그림 1: NAPPA 어레이에서 티로신 키나제 억제제의 품질 관리 및 스크리닝의 개략적 표현. NAPPA 어레이는 태그 및 포획 항체와 융합된 관심 단백질에 대한 cDNA 코딩으로 인쇄됩니다. 시험관 내 전사 및 번역 반응(IVTT) 동안 합성된 단백질은 포획 항체에 의해 태그를 통해 마이크로어레이 표면에서 포착된다. 어레이의 품질 관리(QC)는 슬라이드에 인쇄된 DNA 수준의 측정에 의해 수행되며, 형광 DNA 간칼화 염료를 사용하고, 태그 특이적 항체를 사용하여 어레이에 표시되는 단백질 의 수준을 이용하여 수행된다. 키나아제 스크리닝을 위해, 마이크로어레이는 IVTT 반응 후 DNase 및 인산염으로 처리되고, 단백질 합성 중에 발생할 수 있는 인쇄된 DNA 및 모든 인산화를 제거한다. 탈인식 어레이는 이제 약물 화면에 사용할 준비가 되었습니다. 각 분석에 대해, 컨트롤의 세 세트는 일상적으로 사용된다: (I) dephosphorylated 배열, 있는 autophosphorylation 반응은 ATP 없이 수행; (II) 자가포릴화 마이크로어레이는 ATP의 존재 에서 자가 포스포릴화 반응이 수행되는; 및 (III) 자동 포스포릴화 반응이 ATP 및 DMSO로 수행되는 DMSO 처리 어레이(비히클). 키나아제 억제제의 상이한 농도로 처리된 슬라이드는 DMSO 처리어레이에 사용되는 것과 동일한 프로토콜을 따른다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
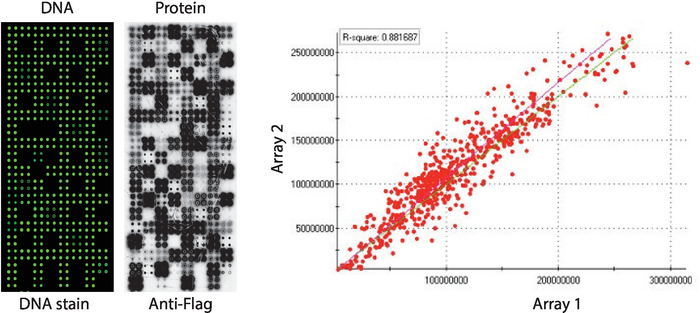
그림 2: 자체 조립된 NAPPA-키나아제 어레이에 대한 품질 관리의 대표적인 결과. 형광 DNA-인터칼레이팅 염료(왼쪽)에 의해 측정된 DNA 함량(왼쪽)과 항기 항체(가운데)에 의해 측정된 마이크로어레이상에 표시되는 단백질의 수준이 도시된다. 오른쪽에는 별도의 배치로 인쇄된 두 개의 NAPPA-키나아제 어레이에 표시되는 단백질 수준의 상관 관계 플롯이 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
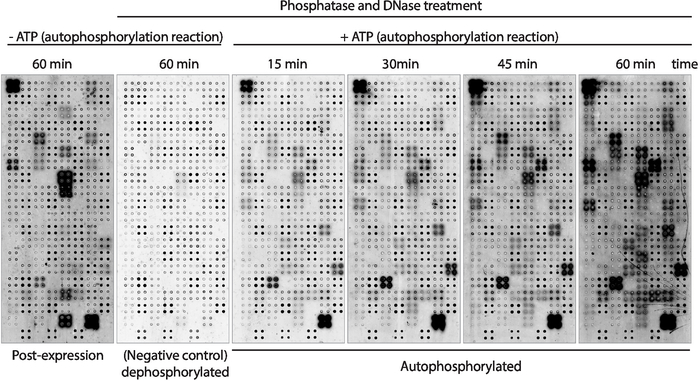
그림 3: NAPPA-키나아제 어레이에서키나제 활성의 대표적인 결과. 4배의 단백질 키나아제들을 표시하는 마이크로어레이는 항-pTyr 항체를 이용한 단백질 인산화의 측정을 통해 어레이상 단백질 키나아제 활성을 연구하고, 이어서 cy3 표지된 항마우스 항체를 사용하였다. 포스포리화 반응 동안 ATP 없이 인산염/DNase 처리 없이 대조군 어레이를 단백질 발현 후 의 배경 인산화를 측정하는데 사용하였다(발현 후). 나머지 마이크로어레이는 인산염/DNA로 처리되었고, 자가포스포릴화 반응은 ATP(탈인마이크로어레이, 음성 대조군) 또는 ATP(자가포스포릴화된 마이크로어레이)로 수행하였다. 자가포스포릴화된 마이크로어레이의 경우, 도시된 바와 같이 15분, 30분, 45분, 또는 60분 동안 오토포스포릴화 반응을 수행하였다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
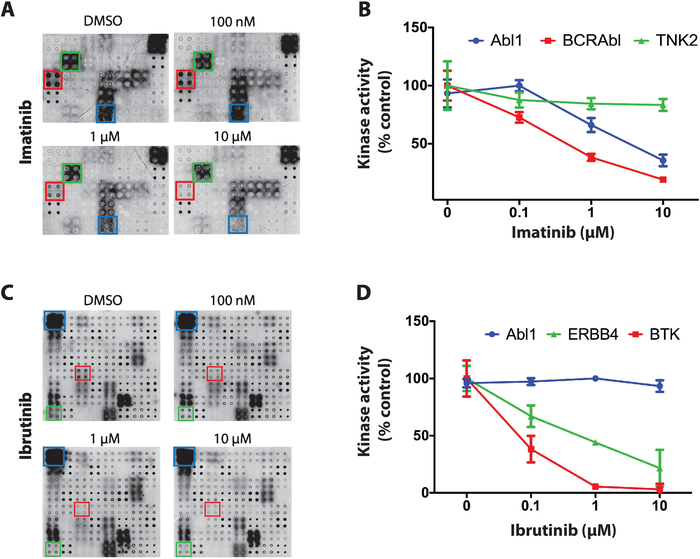
그림 4: NAPPA-키나아제 어레이의 티로신 키나아제 화면에서 대표적인 데이터. (a)포스파타제/DNase 처리NAPPA-키나아제 어레이는 자가포스포릴화 반응 동안 이마티닙의 농도를 증가시켜 배양하고 키나아제 활성을 항인-장타항체로 측정하였다. (B)이마티닙에 노출된 NAPPA-키나아제 어레이에서 관찰된 키나아제 활성의 정량화. 데이터는 음성 대조군 어레이(dephosphorylated)의 신호에 대해 정규화되었고 양성 대조군 어레이(DMSO의 존재 에서 수행된 autophosporylation 반응)의 백분율로 나타내고 있다. 이브루티닙(C,D)의스크리닝을 위해 유사한 데이터가 도시된다. 이 그림은 Rauf 등 에서수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 파일 1. 자동화된 혼성화 스테이션을 사용하여 NAPPA 어레이에서 티로신 키나아제 억제제의 스크리닝을 위한 대체 프로토콜. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
수정 및 문제 해결
NAPPA 어레이에 대한 키나아제 활성 연구의 최적화 단계 동안, 관찰된 배경 및 낮은 동적 범위의 주요 소스 중 하나는 인쇄 믹스에 사용된 BSA였다. BSA는 아미노실란 표면과 가교하는 데 필요한 1차 아민을 제공하고 DNA 및 포획 항체를 그 자리에서 트래핑했다. 그러나 BSA는 고광화되어 배경 잡음 위의 어레이에서 자가 포스포릴화 신호의 검출이 어렵습니다. 이 문제를 해결하기 위해, 인쇄 믹스에서 BSA에 대한 몇 가지 대안을 테스트하고, 폴리 리신은 좋은 대용으로 확인되었다. 폴리-리신은 인산화 부위가 부족합니다. 따라서 표현되지 않는 배열의 배경은 매우 최소화됩니다. 또한 폴리 리신으로 인쇄 된 마이크로 어레이는 재현 가능하고 양호한 수준의 단백질을 표시합니다(그림 2).
표준 NAPPA 분석법에서 수행된 다음 중요한 변형은 인산염/DNase 처리 단계의 첨가였다. 인산파타제와 마이크로어레이의 치료는 단백질 합성 및 포획 동안 IVTT 혼합물에서 발생한 임의의 인산화를 제거할 수있다(도 3). 이러한 인산화의 근원은 본질적인 자가포스포릴화 활성 또는 IVTT 혼합물에 존재하는 키나아제의 활성으로부터 일 수 있다. 모든 인산화 후 발현의 제거는 활성 및 자가포스포릴화를 겪을 수 있는 키나아제의 용이한 식별을 허용하였다(도3).
프로토콜 내의 중요한 단계
NAPPA는 강력한 기술이지만 예상대로 몇 가지 중요한 단계가 있습니다. 첫 번째는 적절한 농도에서 고품질 DNA를 획득하는 것입니다. 품질이 좋지 않거나 농도가 낮은 DNA를 사용하면 적절한 수준으로 표현및 표시되지 않는 여러 가지 특징을 가진 품질이 좋지 않은 마이크로어레이가 생성되어 어레이에서 분석된 단백질의 수가 줄어듭니다. 두 번째 중요한 단계는 마이크로어레이에 단백질의 발현이다. 높은 수준의 기능성 단백질을 발현하는 IVTT 시스템의 사용은 어레이에서 키나아제 활성을 연구하는 데 필수적입니다.
TKI 스크리닝의 다음 중요한 단계는 마이크로어레이를 처리하는 방법입니다. 마이크로어레이는 프로토콜의 모든 단계에서 건조되지 않아야 하며, 배경 신호를 증가시킬 수 있는 스크래치를 방지하기 위해 부드러운 취급을 권장합니다. 전체 실험의 배열을 서로 비교하므로 모든 인큐베이션 단계가 모든 슬라이드에 걸쳐 균일하게 되도록 하는 것이 중요합니다. 예를 들어, 배열 간 인큐베이션 길이의 차이를 방지하기 위해 20개의 배열 일괄 처리가 처리될 때 단일 배열에서 한 단계를 수행하는 데 필요한 시간을 고려해야 합니다.
마지막으로, 실험 설계와 양수 및 음성 제어의 포함은 품질 관리 및 데이터 분석에 매우 중요합니다. 컨트롤의 첫 번째 세트는 각 배열에 인쇄된 컨트롤(즉, 빈 반점(인쇄된 재료 없이), 물 또는 빈 벡터(태그만 표현)]뿐만 아니라 포지티브 컨트롤(즉, 정제된 IgG)을 포함합니다. 이차 항체는 인산화 수준에서 변경에 불활성이다). 전체적으로, 마이크로어레이의 배경 수준, 인쇄 중 가능한 이월 및 검출 방법의 신호 강도를 측정합니다.
제어의 다음 세트는 약물 스크리닝 제어 및 탈인성 및 자가 포설마이크로어레이레이(DMSO의 존재 또는 부재)를 포함한다. 앞서 언급했듯이, 탈인성 마이크로어레이는 인산염 처리 후 인산화 수준을 측정하고 따라서 다른 모든 실험에 대한 기준선 수준을 측정한다. 기준선 수준이 낮을수록, 아시스의 동적 범위가 높아집니다. 자동 인포릴화 어레이는 모든 어레이의 최대 인산화 수준을 나타내며 신호는 강력하고 명확해야 합니다. 데이터 분석에 사용되지만 모든 반응이 어레이에서 성공적으로 수행되었다는 제어로도 사용됩니다.
기술의 한계
현재, 여기에 제시된 약물 스크리닝의 한계 중 하나는 자가 포설될 수 있는 단백질 키나아제만을 스크리닝하는 능력이다. 이를 극복하는 한 가지 가능한 방법은 동일한 자리에서 키나아제 및 공지된 기판을 인쇄하는 것이다. 2개의 명백한 단백질을 위한 DNA의 공동 인쇄는 성공적으로 이 접근의 타당성을 건의하는15를달성했습니다. 더욱이, 어레이에 표시되는 단백질은 비활성 단백질의 결과로 정확하게 접히지 않을 수 있습니다. 인간 기반 발현 시스템의 사용은 어레이상에서 측정된 키나아제 활성에서 현저한 개선을 이루었다; 그러나, 몇몇 단백질은 아직도 그것의 비활동 때문에 배열에 분석될 수 없습니다.
제2의 제한은 팬 항인광-타이터 항체를 이용한 인산화의 측정이다. 인산화 부위의 모티프에 관한 비 특이성에도 불구하고, 모든 측정 된 인산화는 티로신 잔류물에서 발생했으며 세린과 트레오닌 및 각각의 키나아제를 남겼습니다. 현재까지 10개 이상의 팬 포스포-Ser/Thr 항체는 인큐베이션 및 세척 조건을 최적화하기 위한 여러 시도에도 불구하고 성공하지 못하고 테스트되었습니다. 항체와 무관한 새로운 검출 시스템은 약물 억제를 위해 스크리처될 수 있는 단백질 키나아제의 수를 확장하는 최선의 선택이 될 수 있다. 이러한 맥락에서, 클릭 컨쥬게이션과 같은 방사능 또는 화학적 접근법을 포함하여 몇 가지 옵션을 사용할 수 있습니다. 배경 신호를 최소화하고 검역에 적합한 동적 범위를 제공하기 위해서는 일련의 최적화가 필요합니다.
세 번째 제한사항은 어레이에 인쇄할 cDNA 클론을 획득하는 것입니다. cDNA 클론은 크리에이터 또는 게이트웨이23과같은 사이트별 재조합 시스템을 포함하는 임의의 복제 기술을 사용하여 생성될 수 있다. 또 다른 옵션은 DNAsu 라이브러리에서 클론을 구입하는 것입니다,에서 발견 , 여기서 이상 17,000 cDNA 클론, 전체 인간의 키노메를 포함, NAPPA 배열의 건설에 사용할 수24 .
네 번째 제한사항은 모든 실험실에 자체 NAPPA 어레이를 제작하고 선별할 수 있는 적절한 장비가 장착되어 있는 것은 아닙니다. 이 프로토콜은 처리량이 높은 장비없이 마이크로어레이에 인쇄되는 DNA를 생성하는 대체 방법과 모든 혼성화 단계를 수동으로 수행하는 프로토콜을 제공합니다. 그러나 어레이 및 마이크로어레이 스캐너에 대한 액세스는 여전히 필요합니다. 이 문제를 극복 하는 한 가지 옵션은 NAPPA 핵심 서비스 및 시설을 사용 하는 것입니다., 에서 발견 , 비영리 학술 가격에 사용자 정의 NAPPA 마이크로 어레이를 배포 하는. 마지막으로, 어떤 선별 방법론으로, 배열에 장악된 데이터는 유물 (양성 또는 부정)에 영향을 받기 쉬므로 직교 분석법을 사용하여 검증되어야합니다.
기존 방법에 대한 중요성
몇몇 플랫폼은 단백질 키나아제의 스크리닝을 위해 상업적으로 이용가능합니다. 일상적으로 사용되는 한 가지 접근법은 단백질 단편, 키나아제 도메인, 키나아제 도메인 및 일부 조절 영역을 가진 더 큰 단백질 단편, 심지어 전체 길이 단백질로 수행 될 수있는 결합 검사입니다. 단백질은 일반적으로 발현 및 정제 프로토콜의 비용 및 단순성 으로 인해 세균 시스템에서 발현된다. 관심 있는 약물과 단백질 사이의 상호작용은 예를 들어 형광 또는 태그의 존재와 같은 일부 유형의 보고서 분석으로 측정됩니다. 이 접근법 세트의 주요 제한은 단백질이 약물과의 상호 작용 중에 반드시 활성화되지 않는다는 사실이며, 이는 거짓 양성 및 거짓 음성 상호 작용의 식별을 초래할 수 있습니다. 단백질 단편은 형태 및 활성 의 부족에 있는 변경에 특히 취약하고 얻어진 모든 데이터는 활성 단백질을 사용하여, 바람직하게는 그들의 전체 길이 양식에서 검증되어야 합니다. 일부 플랫폼의 또 다른 제한사항은 ATP 아날로그만 스크리닝하여 전체 사용을 제한하는 기능입니다.
효소 기반 접근법을 사용하여 TKIs의 스크리닝을 위한 상업적으로 이용 가능한 대부분의 서비스는 관심 있는 키나아제의 야생형 버전만을 이용하고, 때로는 선택된 소수의 돌연변이만을 이용한다. 약물 내성이 TKI로 치료된 환자에서 매우 흔하다는 것을 아는 것은, 가장 적합한 억제제의 선택을 위해 상이한 돌연변이체에서 약물 반응을 측정할 수 있는 것이 중요하다. NAPPA의 특성으로 인해 키나아제 돌연변이체의 스크리닝은 간단하고 쉽게 수행 될 수 있으며, 유일한 필요한 도구는 사이트 별 돌연변이 발생에 의해 수행 될 수있는 NAPPA cDNA 컬렉션에 키나아제 돌연변이체의 통합입니다.
향후 응용 프로그램
키나아제 억제제를 이용한 암 치료에서 가장 흔한 치료 경과 의 한 가지는 치료 과정 동안 약물 표적에서 돌연변이를 획득하는 것이다. 키나아제 억제제에 대한 그들의 반응에 관하여 이 돌연변이의 검열은 각 환자를 위한 개인화한 처리를 달성하기 위하여 TKIs의 2/3 세대의 선택을 위한 생명 중요성입니다. 여기에 제시된 약물 스크리닝 접근법은, 임의의 티로신 키나아제 억제제가 인간 게놈에 존재하는 티로신 키나아제 패널에 대해 시험될 수 있는 편견 없는 스크리닝 플랫폼을 제공한다. NAPPA 어레이에 표시된 단백질은 슬라이드에 인쇄된 cDNA로부터 시험관 내에서 표현되기 때문에, 임의의 돌연변이 변이체는 어레이에 표시될 cDNA 컬렉션에 쉽게 통합될 수 있다. NAPPA 기술의 높은 처리량 파워와 결합된 키나아제 돌연변이체가 어레이상에서 생성되고 발현될 수 있는 시설은 키나아제 돌연변이체와 약물에 대한 반응을 연구할 수 있는 독특한 환경을 제공하여 NAPPA에 적합합니다. 정밀 의학의 목표 중 하나인 맞춤형 약물 스크리닝.
공개
저자는 이해 상충을 선언하지 않습니다.
감사의 말
저자는 프로젝트 개발 과정에서 LaBaer의 연구실에 있는 모든 사람들에게 도움과 비판에 감사드립니다. 이 프로젝트는 NIH 교부금 U01CA117374, U01AI077883 및 버지니아 G. 파이퍼 재단에 의해 지원되었다.
자료
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| 364 well plates (for arraying) | Genetix | x7020 | |
| 800 µL 96-well collection plate | Abgene | AB-0859 | |
| 96-pin device | Boekel | 140500 | |
| Acetic Acid | Millipore-Sigma | 1.00066 | |
| Acetone 99.9% | Millipore Sigma | 650501 | |
| Aluminum seal for 96 well plates | VWR | 76004-236 | |
| Aminosilane (3-aminopropyltriethoxysilane) | Pierce | 80370 | |
| ANTI-FLAG M2 antibody produced in mouse | Millipore Sigma | F3165 | |
| Anti-Flag rabbit Antibody (polyclonal) | Millipore Sigma | F7425 | |
| ATP 10 mM | Cell Signaling | 9804S | |
| β-Glycerophosphate disodium salt hydrate | Millipore-Sigma | G9422 | |
| bacteriological agar | VWR | 97064-336 | |
| Blocking Buffer | ThermoFisher/Pierce | 37535 | |
| Brij 35 | ThermoFisher/Pierce | BP345-500 | |
| BS3 (bis-sulfosuccinimidyl) | ThermoFisher/Pierce | 21580 | |
| BSA (bovine serum albumin) | Millipore Sigma | A2153 | |
| Coverslip 24 x 60 mm | VWR | 48393-106 | |
| Cy3 AffiniPure Donkey Anti-Mouse IgG (H+L) | Jackson ImmunoResearch | 715-165-150 | |
| DeepWell Block, case of 50 | ThermoFisher/AbGene | AB-0661 | |
| DEPC water | Ambion | 9906 | |
| DMSO (Dimethyl Sulfoxide) | Millipore-Sigma | D8418 | |
| DNA-intercalating dye | Invitrogen | P11495 | |
| DNase I | Millipore-Sigma | AMPD1-1KT | |
| DTT | Millipore-Sigma | 43816 | |
| EDTA | Millipore-Sigma | EDS | |
| Ethanol 200 proof | Millipore-Sigma | E7023 | |
| Filter plates | Millipore-Sigma | WHA77002804 | |
| Gas Permeable Seals, box of 50 | ThermoFisher/AbGene | AB-0718 | |
| Glass box | Wheaton | 900201 | |
| Glass slides | VWR | 48300-047 | |
| Glycerol | Millipore-Sigma | G5516 | |
| HCl (Hydrochloric acid) | Millipore-Sigma | H1758 | |
| HEPES Buffer Solution | Millipore-Sigma | 83264 | |
| Human-based IVTT system | Thermo Scientific | 88882 | |
| ImmunoPure Mouse IgG whole molecule | ThermoFisher/Pierce | 31202 | |
| Isopropanol | Millipore-Sigma | I9516 | |
| KCl (Potassium chloride) | Millipore-Sigma | P9333 | |
| KH2PO4(Potassium phosphate monobasic) | Millipore-Sigma | P5655 | |
| Kinase buffer | Cell Signaling | 9802 | |
| KOAc (Potassium acetate) | Millipore-Sigma | P1190 | |
| Lambda Protein Phosphatase | new england biolabs | P0753 | |
| Lifterslips, 24 x 60 mm | ThermoFisher Scientific | 25X60I24789001LS | |
| Metal 30-slide rack with no handles | Wheaton | 900234 | |
| MgCL2 (Magnesium chloride) | Millipore-Sigma | M8266 | |
| Na3VO4 (Sodium orthovanadate) | Millipore-Sigma | S6508 | |
| NaCl (Sodium Chloride) | Millipore-Sigma | S3014 | |
| NaOAc (Sodium acetate) | Millipore-Sigma | S2889 | |
| NaOH (Sodium hydroxide) | Millipore-Sigma | S8045 | |
| NucleoBond Xtra Midi / Maxi | Macherey-Nagel | 740410.10 / 740414.10 | |
| Nucleoprep Anion II | Macherey Nagel | 740503.1 | |
| Phosphoric Acid | Millipore-Sigma | 79617 | |
| Poly-L-Lysine Solution (0.01%) | Millipore-Sigma | A-005-C | |
| Protein Phosphatase (Lambda) | New England Biolabs | P0753 | |
| RNAse | Invitrogen | 12091021 | |
| SDS (Sodium dodecyl sulfate) | Millipore-Sigma | L6026 | |
| SDS (Sodium dodecyl sulfate) | Millipore-Sigma | 05030 | |
| Sealing gasket | Grace Bio-Labs, Inc | 44904 | |
| Silica packets | VWR | 100489-246 | |
| Single well plate | ThermoFisher/Nalge Nunc | 242811 | |
| Sodium acetate (3M, pH 5.5) | Millipore-Sigma | 71196 | |
| TB media (Terrific Broth) | Millipore-Sigma | T0918 | |
| Tris | IBI scientific | IB70144 | |
| Triton X-100 | Millipore-Sigma | T8787 | |
| Tryptone | Millipore-Sigma | T7293 | |
| Tween 20 | Millipore-Sigma | P9416 | |
| Yeast Extract | Millipore-Sigma | Y1625 | |
| Name | Company | Catalog number | Comments |
| Equipments | Maker/model | ||
| Programmable chilling incubator | Torrey Pines IN30 Incubator with Cooling | ||
| Shaker for bacterial growth | ATR Multitron shaker | ||
| Vacuum manifold with liquid waste trap | MultiScreenVacuum Manifold 96 well | ||
| 96 well autopippetor/liquid handler | Genmate or Biomek FX | ||
| Liquid dispenser | Wellmate | ||
| DNA microarrayer | Genetix QArray2 | ||
| Automatic hybridization station | Tecan HS4800 Pro Hybridization Station | ||
| Microarray scanner | Tecan PowerScanner | ||
| Microarray data quantification | Tecan Array-ProAnalyzer 6.3 |
참고문헌
- Melnikova, I., Golden, J. Targeting protein kinases. Nature Review Drug Discovery. 3 (12), 993-994 (2004).
- Patterson, H., Nibbs, R., McInnes, I., Siebert, S. Protein kinase inhibitors in the treatment of inflammatory and autoimmune diseases. Clinical and Experimental Immunology. 176 (1), 1-10 (2014).
- Wu, P., Nielsen, T. E., Clausen, M. H. FDA-approved small-molecule kinase inhibitors. Trends Pharmacological Sciencies. 36 (7), 422-439 (2015).
- Druker, B. J., et al. Effects of a selective inhibitor of the Abl tyrosine kinase on the growth of Bcr-Abl positive cells. Nature Medicine. 2 (5), 561-566 (1996).
- Heinrich, M. C., et al. Inhibition of c-kit receptor tyrosine kinase activity by STI 571, a selective tyrosine kinase inhibitor. Blood. 96 (3), 925-932 (2000).
- Stagno, F., et al. Imatinib mesylate in chronic myeloid leukemia: frontline treatment and long-term outcomes. Expert Review Anticancer Therapy. 16 (3), 273-278 (2016).
- Ben Ami, E., Demetri, G. D. A safety evaluation of imatinib mesylate in the treatment of gastrointestinal stromal tumor. Expert Opinions in Drug Safety. 15 (4), 571-578 (2016).
- Ramachandran, N., et al. Self-assembling protein microarrays. Science. 305 (5680), 86-90 (2004).
- Festa, F., et al. Robust microarray production of freshly expressed proteins in a human milieu. Proteomics Clinical Applications. 7 (5-6), 372-377 (2013).
- Yazaki, J., et al. Mapping transcription factor interactome networks using HaloTag protein arrays. Proceedings of the National Academy of Sciences of the United States of America. 113 (29), E4238-E4247 (2016).
- Rauf, F., et al. Ibrutinib inhibition of ERBB4 reduces cell growth in a WNT5A-dependent manner. Oncogene. 37 (17), 2237-2250 (2018).
- Anderson, K. S., et al. Protein microarray signature of autoantibody biomarkers for the early detection of breast cancer. Journal of Proteome Research. 10 (1), 85-96 (2011).
- Wang, J., et al. Plasma Autoantibodies Associated with Basal-like Breast Cancers. Cancer Epidemiol Biomarkers Prevention. 24 (9), 1332-1340 (2015).
- Bian, X., et al. Tracking the Antibody Immunome in Type 1 Diabetes Using Protein Arrays. Journal of Proteome Research. 16 (1), 195-203 (2017).
- Song, L., et al. Identification of Antibody Targets for Tuberculosis Serology using High-Density Nucleic Acid Programmable Protein Arrays. Molecular and Cellular Proteomics. 16 (4 suppl 1), S277-S289 (2017).
- Wang, J., et al. Comparative Study of Autoantibody Responses between Lung Adenocarcinoma and Benign Pulmonary Nodules. Journal of Thoracic Oncology. 11 (3), 334-345 (2016).
- Montor, W. R., et al. Genome-wide study of Pseudomonas aeruginosa outer membrane protein immunogenicity using self-assembling protein microarrays. Infection and Immunity. 77 (11), 4877-4886 (2009).
- Tang, Y., Qiu, J., Machner, M., LaBaer, J. Discovering Protein-Protein Interactions Using Nucleic Acid Programmable Protein Arrays. Current Protocols in Cell Biology. 74, 11-15 (2017).
- Yu, X., et al. Copper-catalyzed azide-alkyne cycloaddition (click chemistry)-based detection of global pathogen-host AMPylation on self-assembled human protein microarrays. Molecular and Cellular Proteomics. 13 (11), 3164-3176 (2014).
- Anderson, K. S., et al. Autoantibody signature for the serologic detection of ovarian cancer. Journal of Proteome Research. 14 (1), 578-586 (2015).
- Woyach, J. A., Johnson, A. J., Byrd, J. C. The B-cell receptor signaling pathway as a therapeutic target in CLL. Blood. 120 (6), 1175-1184 (2012).
- Smith, M. R. Ibrutinib in B lymphoid malignancies. Expert Opinion on Pharmacotherapy. 16 (12), 1879-1887 (2015).
- Festa, F., Steel, J., Bian, X., Labaer, J. High-throughput cloning and expression library creation for functional proteomics. Proteomics. 13 (9), 1381-1399 (2013).
- Seiler, C. Y., et al. DNASU plasmid and PSI:Biology-Materials repositories: resources to accelerate biological research. Nucleic Acids Research. 42, D1253-D1260 (2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유