Method Article
Dépistage de l'inhibiteur de kinase dans les microréseaux de protéines humaines auto-assemblés
Dans cet article
Résumé
Un protocole détaillé pour la génération de microarrays de protéines humaines auto-assemblés pour le dépistage des inhibiteurs de la kinase est présenté.
Résumé
Le dépistage des inhibiteurs de la kinase est crucial pour mieux comprendre les propriétés d'un médicament et pour l'identification de cibles potentiellement nouvelles ayant des implications cliniques. Plusieurs méthodologies ont été signalées pour effectuer un tel dépistage. Cependant, chacun a ses propres limites (p. ex., le dépistage de seulement analogues ATP, la restriction à l'utilisation de domaines de kinase purifiés, les coûts importants associés aux tests de plus de quelques kinases à la fois, et le manque de flexibilité dans le dépistage des kinases protéiques avec mutations nouvelles). Ici, un nouveau protocole qui surmonte certaines de ces limitations et peut être employé pour le criblage impartial des inhibiteurs de kinase est présenté. Une force de cette méthode est sa capacité à comparer l'activité des inhibiteurs de kinase à travers les protéines multiples, soit entre différentes kinases ou différentes variantes de la même kinase. Des microréseaux de protéines auto-assemblés générés par l'expression de kinases protéiques par un système de transcription et de traduction in vitro (IVTT) à base humaine sont utilisés. Les protéines affichées sur le microarray sont actives, permettant de mesurer les effets des inhibiteurs de la kinase. La procédure suivante décrit les étapes du protocole en détail, de la génération de microréseaux et le dépistage à l'analyse des données.
Introduction
Les kinases protéiques sont responsables de la phosphorylation de leurs cibles et peuvent moduler des voies moléculaires complexes qui contrôlent de nombreuses fonctions cellulaires (c.-à-d. prolifération cellulaire, différenciation, mort cellulaire et survie). La déréglementation de l'activité kinase est associée à plus de 400 maladies, faisant des inhibiteurs de la kinase l'une des principales classes de médicaments disponibles pour le traitement de plusieurs maladies, y compris le cancer, les troubles cardiovasculaires et neurologiques ainsi que les inflammatoires et les maladies auto-immunes1,2,3.
Avec l'avènement de la médecine de précision, l'identification de nouvelles thérapies, en particulier les inhibiteurs de la kinase, ont un grand attrait pharmaceutique et clinique. Plusieurs approches peuvent être utilisées pour l'identification de nouvelles paires possibles d'inhibiteurs de la kinase/kinase, y compris la conception de novo des inhibiteurs de la kinase et l'identification de nouvelles cibles pour les médicaments approuvés par la FDA. Ce dernier est particulièrement attrayant, puisque le temps et l'argent nécessaires à la mise en œuvre de ces médicaments dans les cliniques sont considérablement réduits en raison de la disponibilité des données d'essais cliniques antérieures. Un exemple canonique de la réutilisation d'un inhibiteur de kinase est l'imatinib, initialement conçu pour le traitement de la leucémie myélogène chronique (LMC) par l'inhibition de BCR-Abl, qui peut également être utilisé avec succès pour le traitement de c-Kit sur-exprimant tumeurs stromales gastro-intestinales (GISTs)4,5,6,7.
Le criblage des inhibiteurs de kinase peut être exécuté dans des essais contraignants ou des essais enzymatiques-basés. La première classe d'essais se concentre sur les interactions protéines-médicaments et peut fournir des informations telles que le site de ligature et l'affinité. Puisque l'activité de la kinase au moment de ces essais est inconnue, un certain nombre d'interactions peuvent être manquées ou faussement identifiées en raison des changements conformationnels dans la protéine. D'autre part, les essais à base d'enzymatiques exigent que les kinases protéiques soient actives et fournissent des informations précieuses sur l'effet de l'inhibiteur sur l'activité enzymatique, cependant, ce type de dépistage est généralement plus long et coûteux. À l'heure actuelle, les deux types d'essais sont disponibles dans le commerce auprès de plusieurs sources. Ils représentent une option fiable pour le dépistage des inhibiteurs de la kinase avec quelques limitations, y compris: I) la plupart des méthodes impliquent l'essai de kinases multiples individuellement, ce qui peut rendre le dépistage d'un grand ensemble de protéines coûteux; II) l'ensemble de kinases à tester se limite à une liste de kinases présélectionnées de type sauvage et à plusieurs versions mutées bien connues de certaines kinases, ce qui entrave l'essai de nombreux nouveaux isoformes mutés.
Dans ce contexte, les microréseaux protéiques sont une plate-forme puissante capable de surmonter certaines des limites présentées par les techniques disponibles dans le commerce. Il convient d'effectuer des essais à base d'enzymatiques dans le dépistage à haut débit à l'aide de protéines actives pleine longueur de toute séquence d'intérêt. Les microréseaux peuvent être générés par une approche auto-assemblée comme NAPPA (tableau de protéines programmables à l'acide nucléique), dans lequel les protéines sont exprimées juste à temps pour les essais, augmentant la probabilité que ceux affichés sur le tableau sont en effet actifs. Les protéines affichées sur NAPPA sont produites à l'aide de ribosomes d'origine humaine et de protéines chaperon afin d'améliorer la probabilité de pliage naturel et d'activité.
Les protéines sont initialement programmées en imprimant des cDNA codant pour les gènes d'intérêt fusionnés avec une étiquette de capture, avec un agent de capture, sur la surface du microarray. Les protéines sont ensuite produites sur les microréseaux à l'aide d'un système de transcription et de traduction in vitro (IVTT), et les protéines fraîchement exprimées sont immobilisées sur la surface du microréseau par l'agent de capture. Les tableaux NAPPA exprimés peuvent être utilisés pour l'étude des protéines affichées sur le tableau d'une manière impartiale et à haut débit8,9.
Auparavant, il a été démontré que les protéines affichées sur les tableaux NAPPA sont pliées correctement pour interagir avec des partenaires connus10; en outre, leur activité enzymatique a été exploitée pour la première fois en 2018, lorsqu'il a été démontré que les kinases protéiques affichées sur l'autophosphorylate microarray11. À ce jour, la méthodologie NAPPA a été utilisée pour de nombreuses applications distinctes, y compris la découverte de biomarqueurs12,13,14,15,16,17, interactions protéines-protéines10,18, identification de substrat19, et dépistage des médicaments11. Sa flexibilité est l'une des caractéristiques clés de la plate-forme qui permet de s'adapter à chaque application.
Ici, un protocole pour le criblage des inhibiteurs de kinase de tyrosine dans les tableaux auto-assemblés de NAPPA est présenté. La plate-forme est optimisée pour l'affichage des kinases actives de protéine humaine et pour l'analyse de l'activité de kinase de protéine, avec le fond bas et la gamme dynamique élevée. Parmi les modifications mises en œuvre pour utiliser nAPPA pour le dépistage des inhibiteurs de la kinase, mentionnons : I) les changements dans la chimie de l'impression, II) la déphosphorylation du microréseau protéique avant le criblage des inhibiteurs de la kinase, et III) l'optimisation de la détection de protéines phosphorylées sur le tableau. Ce protocole est le premier du genre et fournit des informations uniques sur l'étude kinase dans les microréseaux NAPPA.
Protocole
1. Les tampons et les solutions communs à utiliser
- Préparer le milieu de tb: Bouillon terrible (24 g/L extrait de levure; 20 g/L tryptone; 4 mL/L de glycérol; 0,017 M KH2PO4;et 0,072 M K2HPO4). Les 0,017 M KH2PO4 et 0,072 M K2HPO4 solutions peuvent être achetés comme un tampon de phosphate 10x (0,17 M KH2PO4 et 0,72 M K2HPO4).
- Préparer le milieu LB : Luria-Bertani (extrait de levure de 5 g/L; 10 g/L tryptone; et 10 g/L NaCl). Ajuster le pH à 7,0 avec 5 M NaOH.
- Préparer 1x SCT : Saline tamponnée Tris (TbS : 50 mM Tris-Cl, pH 7,5; 150 mM NaCl).
- Préparer 1x TBST: TBS complété avec 0,1% Tween 20.
2. Préparation de l'ADN
REMARQUE : L'ADN utilisé pour les tableaux NAPPA doit être très pur ; par conséquent, les mini-préparations commerciales d'ADN ne sont pas recommandées. Actuellement, deux protocoles pour la préparation de l'ADN sont utilisés: dans la maison à haut débit mini-préparation (décrit ici) ou commerciale Midi- ou Maxi-prep. Le débit moyen du protocole de mini-préparation interne est de 1 500 échantillons par jour et par personne.
- Croissance bactérienne pour la mini-préparation interne à haut débit
- Préparer la plaque omni LB/Agar. Verser 30 à 40 ml d'agar LB (1,5 % d'agar bactériologique dans le média LB complété d'antibiotiques pour la sélection de clones positifs) dans chaque plaque de puits unique.
- Repérez le bouillon de glycérol sur la plaque LB/agar. Diluer le stock de glycérol dans les médias LB (1:300, habituellement 2 l dans 600 l de LB). Agiter pendant 10 min. Spot 3 'L du bouillon dilué sur la plaque LB/agar. Incuber à 37 oC, à l'envers, toute la nuit.
- Inoculer les cultures. À l'aide de l'appareil à 96 broches qui a été stérilisé dans 80% d'éthanol et de flamme, inoculer la culture de la plaque d'agar dans un bloc de puits profondavec 1,5 ml par puits de tb moyen complété d'antibiotiques.
- Incuber les cultures. Couvrir le bloc d'un joint perméable au gaz et couver de 22 à 24 h à 37 oC, 300 à 800 tr/min selon le shaker.
REMARQUE : Les shakers réglés à 800 tr/min sont optimaux pour cette incubation. L'utilisation d'un shaker à vitesse plus lente peut entraîner des cultures moins denses et des rendements de purification de l'ADN plus faibles. - Cultures pellet. Spin blocs à 3 800 x g et 4 oC pendant 30 min. Jeter le supernatant.
- Mini-préparation interne à haut débit
REMARQUE : Les pipettors multicanaux ou distributeurs automatiques peuvent être utilisés pour effectuer la mini-préparation à haut débit interne. Si vous utilisez un distributeur automatique, assurez-vous de nettoyer le système avant d'utiliser et entre les solutions.- Préparer toutes les solutions utilisées lors de la mini-préparation :
- Préparer la solution 1 : tampon de résuspension TE (50 mM Tris, pH 8,0; 10 mM EDTA, pH 8,0; 0,1 mg/mL RNAse). Conserver à 4 oC.
- Préparer la solution 2 : tampon de lyse NaOH/SDS (0,2 M NaOH; 1 % SDS). Pour de meilleurs résultats, une solution fraîchement faite devrait être utilisée.
- Préparer la solution 3 : tampon de neutralisation KOAC (2,8 M KOAc). Ajuster le pH de la solution à 5,1 avec de l'acide acétique glaciaire. Conserver à 4 oC.
- Préparer la solution N2 : tampon d'équilibre (100 mM Tris; 900 mM KCl; 15 % EtOH; 0,15 % Triton X-100). Ajuster le pH de la solution à 6,3 avec de l'acide phosphorique.
- Préparer la solution N3 : tampon bash (100 mM Tris; 1,15 M KCl; 15 % EtOH). Ajuster le pH de la solution à 6,3 avec de l'acide phosphorique.
- Préparer la solution N5 : tampon d'élution (100 mM Tris; 1 M KCl; 15% EtOH). Ajuster le pH de la solution à 8,5 avec de l'acide phosphorique.
REMARQUE : Le contrôle réussi de la liaison, du lavage et de l'élution de l'ADN pendant l'échange d'anion dépend fortement de la concentration et des valeurs de pH tampondeles. Des mesures soigneuses des composants tampons et un ajustement du pH sont essentiels. De petits écarts par rapport aux mesures décrites peuvent entraîner une perte importante de rendements.
- Suspendre à nouveau les granulés. Ajouter 200 l de la solution 1 et secouer à 2 000 tr/min pendant 5 min à RT. Une nouvelle suspension complète de la pastille est nécessaire pour réussir la lyse. Vortex le bloc si nécessaire.
- Lyse bactéries. Ajouter 200 l de la solution 2, sceller la plaque avec un joint en aluminium et inverser 5x. Temps soigneusement cette étape à partir du début de la solution 2 ajout. Ne pas dépasser 5 min.
- Neutraliser la solution. Ajouter 200 l de la solution 3, sceller la plaque avec un joint en aluminium et inverser 5x. Le joint peut être lâche en raison de la lyse / tampons de neutralisation, alors faites preuve de prudence lors de l'inversion. Une inversion partielle, dans laquelle la solution ne touche jamais le joint, est recommandée pour prévenir la contamination croisée entre les échantillons.
- Lysate clair. Centrifuger les plaques à 3 800 x g et 4 oC pendant 30 min.
- Préparer la boue de réinsine d'échange d'anion pendant l'étape de centrifugation de granule de lysate. À l'aide d'une bouteille de 1 L, remplissez-la de résine d'échange d'anions jusqu'à ce qu'elle atteigne la marque de 300 ml, puis ajoutez la solution N2 jusqu'à 900 ml.
CAUTION: Cette étape doit être fait dans le capot pour se protéger contre l'inhalation de silice. - Préparer des plaques de résine d'échange d'anion. Empilez les plaques de filtre sur un bloc de puits profond pour agir comme un récipient de collecte des déchets. Mélanger la boue d'échange d'anion jusqu'à ce qu'elle soit homogène, puis verser dans une auge de verre. À l'aide de conseils P1000 à gros frais, transférer 450 ll de boue dans chaque puits des plaques de filtre.
- Plaques empilées centrifuges (plaque de résine/plaque de puits profond) à une accélération lente pendant 5 min à 130 x g et RT. Jetez le flux à travers.
- Transférer le supernatant de lysate sur la plaque de résine/les piles de blocs de puits profonds. Faites tourner les plaques empilées pendant 5 min à 30 x g avec une vitesse de montée en puissance lente.
- Laver la colonne. Ajouter 400 l de solution N3 (tampon de lavage) à chaque puits. Transférer la plaque de résine dans le collecteur sous vide pour enlever le tampon de lavage. Répétez les étapes de lavage 3x. Lors du dernier lavage, assurez-vous que tous les puits sont bien vidés. Faites tourner les plaques de pile à 150 x g pendant 5 min pour enlever tout tampon résiduel.
- L'ADN d'Elute. Placer la plaque de résine sur une plaque de collecte propre de 800 ll. Ajouter 300 l de solution N5 à chaque puits. Laisser s'asseoir à RT pendant 10 min, puis faire tourner les plaques empilées pendant 5 min à 20 x g avec une vitesse de montée en puissance lente. Faire tourner les assiettes empilées pendant 1 min à 233 x g.
- Quantifier l'ADN et stocker les plaques à -20 oC jusqu'à ce qu'ils soient utilisés ou procéder directement aux précipitations d'ADN.
REMARQUE : Un minimum de 30 g d'ADN par échantillon est nécessaire. Si le rendement de l'ADN est faible, il est recommandé de répéter la mini-préparation de l'ADN, ou de combiner deux plaques pendant l'étape de précipitation (section 2.3).
- Préparer toutes les solutions utilisées lors de la mini-préparation :
- Précipitations d'ADN
- Décongeler les plaques, vortex pour homogénéiser la solution d'ADN, et tourner à 230 x g pour 30 s pour recueillir toutes les solutions dans le fond du puits.
- Ajouter 40 OL de 3 M NaOAc et 240 oL d'isopropanol à chaque puits. Couvrir la plaque d'un joint en aluminium et mélanger en inversant 3x.
- Centrifuger les plaques à 30 min à 3 800 x g et 25 oC. Jetez soigneusement le supernatant.
REMARQUE : Pour combiner deux plaques, transférer l'ADN de la deuxième plaque dans la pastille de la première plaque et répéter les étapes 2.3.2-2.3.3. - Laver et précipiter l'ADN. Ajouter 400 l'éthanol à 80 % de chaque puits. Plaques d'étanchéité avec joint d'aluminium et secouer à 1 000 tr/min pendant 30 min. Centrifugeà 3 800 x g pendant 30 min à 25 oC. Jetez le supernatant.
- Séchez les granules d'ADN. Placez les assiettes à l'envers à un angle sur des essuie-tout et laissez-les sécher pendant 1 h et 2 h, jusqu'à ce qu'il n'y ait pas d'alcool au fond du puits. Sceller et centrifuger à 230 x g pendant 2 min pour abaisser les granulés.
- Une fois les plaques sèches, sceller avec du joint d'aluminium et congeler à -20 oC pour une utilisation ultérieure ou continuer à suspendre l'ADN (étape 4.1).
3. Manteau de lamelle Aminosilane
- Placer les lames de verre dans un support métallique. Inspectez visuellement chaque glissière pour s'assurer qu'il n'y a pas d'égratignures ou d'imperfections.
- Immerger les lames dans la solution de revêtement (2% de réactif aminosilane en acétone) pendant 15 min tout en se balançant. La solution aminosilane peut être utilisée pour enrober deux racks de 30 diapositives chacun avant d'avoir besoin d'être jeté.
- Rincez l'étape. Immerger le toboggan dans le lavage d'acétone (99% d'acétone), secouer d'avant en arrière, puis monter et descendre rapidement 5x. Inclinez-vous vers un coin pour vous égoutter, puis immerger dans l'eau Ultrapure de haut en bas rapidement 5x. Inclinez-vous pour s'égoutter, puis placez-les sur des serviettes.
REMARQUE : Le lavage d'acétone peut être utilisé deux fois, tandis que l'eau ultrapure doit être changée à chaque fois. - Des toboggans secs à l'aide d'air sous pression, soufflant sur eux sous tous les angles pendant environ 3 min jusqu'à ce que toutes les gouttelettes d'eau aient été enlevées. Conservez les toboggans enduits à RT dans un support métallique à l'intérieur d'une boîte hermétiquement scellée.
4. Préparation de l'échantillon array
- Resuspendre le granule d'ADN de la mini-préparation interne (étape 2.3.6) dans 20 L d'eau ultrapure et secouer à 1000 tr/min pendant 2 h. Pour l'ADN de préparation midi/max, diluer chaque échantillon à une concentration finale de 1,5 g/L et transférer 20 l à une plaque de collecte de 800 l.
- Préparer le mélange d'impression. Pour une plaque de puits de 96, préparer 1 mL de mélange d'impression [237,5 l d'eau ultrapure; 500 l de polylysine (0,01 %); 187,5 l de BS3 (bis-sulfosuccinimidyl, 50 mg/mL en DMSO); et 75 l d'anticorps anti-lapin polyclonal)].
REMARQUE : Les produits chimiques doivent être ajoutés dans l'ordre spécifié pour éviter les précipitations. - Ajouter 10 l de mélange d'impression à chaque échantillon, sceller les plaques avec du papier d'aluminium, et secouer à RT pendant 90 min à 1 000 tr/min. Conserver les assiettes pendant la nuit (16 h) à 4 oC.
- Le jour de l'impression, brièvement vortex et faire tourner les plaques. Transférer 28 ll de chaque échantillon sur une plaque de tableau de 384. Ce transfert peut se faire à l'aide d'automatisation ou d'une pipette multicanal. Il est crucial de garder une trace de la position des échantillons dans la plaque de tableau 384.
- Faites tourner la plaque brièvement pour enlever les bulles. Sceller les assiettes avec du papier d'aluminium.
5. Génération de tableaux NAPPA : impression microarray
REMARQUE : Toutes les conditions d'impression ont été optimisées pour l'instrument figurant dans la Table des équipements et des matériaux. Si vous utilisez un tableau différent, une optimisation supplémentaire peut être nécessaire.
- Arrayer nettoyer. Avant de commencer, videz tous les réservoirs de déchets et remplissez les réservoirs avec de l'eau ultrapure ou 80% d'éthanol, si nécessaire. Nettoyez les épingles une par une avec des lingettes sans matière de laine et de l'eau ultrapure. Séchez les broches avec des lingettes sans matière de résineuse et placez-les soigneusement dans la tête du tableau.
- Tableau mis en place: spécifications d'impression [nombre maximum de timbres par encre: 1; nombre de timbres par place: 1; timing multi-timbres: --; temps d'timbre (ms): 0 ms; temps d'encrage (ms): 0 ms; réglage de profondeur d'impression: 90 microns; nombre de touch-offs: 0]. protocole de stérilisation : lavage à l'eau ultrapure pour 2 000 ms avec 0 ms de temps de séchage et 500 ms de temps d'attente; répéter ces étapes 6x; suivi d'un lavage à 80 % d'éthanol pour 2 000 ms avec 1 200 ms de temps de séchage et 500 ms de temps d'attente; répéter ces étapes 6x.
- Conception de diapositive : configurez le tableau avec le modèle de tableau désiré. La conception doit tenir compte de plusieurs facteurs [c.-à-d., le nombre de répliques pour chaque échantillon, l'emplacement et le nombre des fonctions de contrôle, la disposition du tableau (un bloc, plusieurs blocs identiques), le nombre de tableaux à imprimer, la durée de la course, etc.].
- Placez les toboggans enduits d'aminosilane (étape 3.4) sur le pont de l'tableau. Vérifiez si le vide maintient toutes les glissières en place en toute sécurité. Démarrer l'humidificateur (il doit être fixé à 60%).
- Placez la plaque de puits 384 sur le pont de l'escalier. Démarrez le programme.
- Étiqueter les microréseaux. Lorsque l'impression est terminée, placez les étiquettes de diapositives sur le côté inférieur (non imprimé) de chaque diapositive. Maintenir l'ordre d'impression de diapositives sur le pont dans l'ordre numérique.
- Rangez les tableaux imprimés à RT dans un support métallique à l'intérieur d'une boîte hermétiquement scellée avec un paquet de silice. Les glissières conservées dans un environnement sec ont une durée de conservation pouvant aller jusqu'à un an.
- (Facultatif) : un deuxième lot de 90 diapositives peut être imprimé à l'aide des mêmes échantillons. Pour ce faire, retirez la plaque de puits 384 de la plate-forme de l'escalier dès que l'impression de la plaque est terminée. Sceller et conserver l'assiette à 4 oC. Une fois que le premier lot de tableaux est complètement terminé, retirez-les du pont, placez de nouveaux toboggans enduits d'aminosilane et commencez une nouvelle course. Assurez-vous que chaque plaque de puits 384 est à RT pendant 30 minutes avant son utilisation. Si plus de quatre répliques par échantillon sont imprimées dans un lot de diapositives, il est recommandé de diviser les 384 plaques de puits en deux plaques pour diminuer l'évaporation de l'échantillon en diminuant le temps passé sur le pont de l'escalier.
REMARQUE : Vérifiez tous les réservoirs avant le début de la deuxième manche.
6. Détection de l'ADN sur les diapositives NAPPA
- Bloquez les diapositives. Placez les diapositives dans une boîte de pipette et ajoutez 30 ml de tampon de blocage. Incuber à RT pendant 1 h sur un shaker à bascule.
- Tachez les diapositives. Jetez la solution de blocage et ajoutez 20 ml de tampon de blocage et 33 l de colorant fluorescent intercalateur d'ADN. Incuber pendant 15 min avec agitation. Ensuite, rincez rapidement les glissières à l'eau ultrapure et séchez-les avec de l'air sous pression. Procéder à la numérisation (article 11).
7. Expression des diapositives NAPPA
- Bloquer les glissières avec un tampon de blocage sur un shaker à bascule à RT pendant 1 h. Utilisez environ 30 ml dans une boîte de pipette pour quatre diapositives.
- Rincer les toboggans à l'eau ultrapure et sécher à l'air comprimé filtré. Appliquer le joint d'étanchéité sur chaque diapositive selon les instructions du fabricant.
- Ajouter le mélange IVTT. Chaque diapositive nécessitera 150 ll de mélange IVTT. Diluer 82,5 l de lysate HeLa dans 33 l'eau DEPC et compléter avec 16,5 l de protéines accessoires et 33 uL de mélange de réaction. Ajouter le mélange IVTT de l'extrémité non-étiquette ou non-spécimen. Pipette le mélange dans lentement (il est acceptable si elle perle temporairement à l'extrémité de l'enlet). Masser délicatement le joint d'étanchéité afin que le mélange IVTT s'étende et couvre toute la zone du tableau. Appliquer les petits joints de port ronds sur les deux ports.
- Placez les glissières sur un support et transférez-les à l'incubateur de refroidissement programmable. Incuber pendant 90 min à 30 oC pour l'expression des protéines, suivi de 30 min à 15 oC pour l'immobilisation de la protéine requête.
- Laver et bloquer les diapositives. Retirez le joint d'étanchéité et plongez les glissières dans une boîte à pipettes avec environ 30 ml de 1x TBST complétés par 5 % de lait pour l'affichage des protéines (section 8) ou 1x TBST complétés par de l'albumine de sérum bovin (BSA) de 3 % pour les analyses de kinase ou le dépistage des médicaments (section 9). Incuber à RT avec agitation pendant 20 min et répéter cette étape 2x.
8. Détection des protéines sur les tableaux NAPPA
- Ajouter l'anticorps primaire. Retirez les diapositives de la solution de blocage (étape 7.5) et séchez doucement le côté arrière (côté non imprimé) à l'aide d'un essuie-tout. Placez les diapositives sur un support et appliquez 600 l d'anticorps primaires (anti-drapeau de souris) dilués 1:200 dans 1x TBST et 5% de lait. Incuber pour 1 h à RT.
- Laver les lames avec 1x TBST et 5% de lait sur un shaker à bascule (3x pour 5 min chacun).
- Ajouter l'anticorps secondaire. Retirer les lames de la solution de lavage et sécher doucement le côté arrière à l'aide d'essuie-tout. Placez les diapositives sur un support et appliquez 600 l d'anticorps secondaires (anticorps antisouris cy3-labbeled) dilués 1:200 dans 1x TBST et 5% de lait. Protégez les glissières de la lumière et incuber pendant 1 h à RT.
- Laver les lames avec 1x TBST sur un shaker à bascule (3x pour 5 min chacun). Rincer rapidement les glissières à l'eau ultrapure et sécher à l'aide d'air sous pression. Procéder à la numérisation (article 11).
9. Dépistage des inhibiteurs de la kinase tyrosine sur les tableaux NAPPA
REMARQUE : Plusieurs diapositives peuvent être traitées dans la même expérience, cependant, assurez-vous que dans chaque étape, une diapositive est traitée à la fois et qu'ils ne sèchent pas entre les étapes. Ajoutez toutes les solutions à l'extrémité non-étiquette ou non-spécimen de la diapositive.
- Préparer toutes les solutions utilisées lors du dépistage des médicaments :
- Préparer la solution phosphatase/DNase en combinant les éléments suivants : 1x protéine metallo-phosphatases tampon (50 mM HEPES, 100 mM NaCl, 2 mM DTT, 0,01% Brij 35 à pH - 7,5); 1 mM Mm MnCl2; 8 000 unités de phosphatase de protéine lambda; et 2 unités de DNase I. Préparez 400 L de la solution pour chaque microarray. Ajouter la phosphatase et le DNase juste avant l'utilisation.
- Faire la dilution de la drogue. Les médicaments sont reconstitués dans DMSO à une concentration finale de 10 mM. Pour s'assurer que toutes les concentrations de médicaments testées sur le tableau, assurez-vous que le même volume de DMSO (un stock de 10 000x dans le DMSO) est créé pour chaque concentration et maintenu à -80 oC. Au moment de l'utilisation, les médicaments sont dilués 1:100 dans l'eau.
- Préparer la solution médicament/kinase en combinant ce qui suit : 1x tampon kinase (25 mM Tris-HCl de pH à 7,5); 5 mM de bêta-glycérophosphate; 2 mM DTT; 0,1 mM Na3VO4; 10 mM MgCl2; 500 M ATP; et 2 L de médicament (dilué 1:100 dans l'eau). Préparer 200 L de la solution pour chaque microarray.
- Effectuer le traitement de la phosphatase et de la DNase. Retirez les glissières de la solution de blocage (étape 7.5) et séchez doucement le dos à l'aide d'un essuie-tout. Placez les diapositives sur le support et appliquez 200 l de la solution phosphatase/DNase. Placez un bordereau de microarray pour éviter l'évaporation. Incuber à 30 oC pendant 45 min au four.
- Traitement de la phosphatase et de la DNase II : retirer les tableaux du four, jeter la glissière de couverture, enlever la solution excédentaire et appliquer 200 ll de la solution de phosphatase et de DNase fraîchement fabriquée. Couvrir les microréseaux de la couverture et incuber pendant 45 min de plus à 30 oC au four.
- Laver les toboggans avec 1x TBST et 0,2 M NaCl sur un shaker à bascule (3x pour 5 min chacun).
- Effectuer le traitement médicamenteux et la réaction kinase. Retirer les glissières de la solution de lavage et sécher doucement le dos à l'aide d'un essuie-tout. Placez les diapositives sur le support et appliquez 200 L de solution médicament/kinase. Placez un bordereau sur le dessus pour éviter l'évaporation. Incuber pendant 1 h à 30 oC au four.
- Laver les toboggans avec 1x TBST et 0,2 M NaCl sur un shaker à bascule (3x pour 5 min chacun).
- Répéter les étapes 8.1-8.4 en utilisant comme anticorps anti-phosho-Tyr d'anticorps primaires dilués 1:100. Remplacer 1x TBST et 5 % de lait à toutes les étapes par 1 x TBST et 3 % de BSA.
10. Protocole d'hybridation automatisé
REMARQUE : Alternativement, une station d'hybridation peut être utilisée pour automatiser toutes les hybridations et lavages sur les tableaux NAPPA (sections 7 à 9) et le protocole est fourni sous forme de fichier supplémentaire 1.
11. Acquisition d'images
REMARQUE : Les images microarray doivent être acquises à une résolution de 20 microns ou plus.
- Chargez les microréseaux dans le chargeur de curseur avec les protéines orientées vers le haut. Chargez le chargeur dans le scanner de microarray.
- Sélectionnez le laser vert avec un filtre d'émission de 575/30 nm pour scanner le signal de l'anticorps secondaire étiqueté cy-3. Si un fluorophore différent est utilisé, sélectionnez le laser/longueur d'onde correct pour détecter le signal du colorant fluorescent.
- Définissez le nom de chaque image et l'emplacement qu'elles seront enregistrées.
- (Facultatif) : pour chaque nouveau fluorophore, l'optimisation des conditions de balayage est recommandée pour détecter la plage linéaire de l'intensité du signal. Pour ce faire, scanner un microréseau à l'aide d'une gamme de photomultiplicateurs (PMT) et gagner jusqu'à ce qu'une image claire soit obtenue avec un signal non saturé et un fond faible.
- Scannez tous les microréseaux avec les paramètres optimisés et n'oubliez pas d'éteindre l'autogain.
REMARQUE : Pour l'analyse des données, tous les microréseaux doivent être numérisés à l'aide des mêmes paramètres de numérisation. Pour les essais de kinase utilisant le cy3 comme fluorophore, les images sont numérisées avec un PMT de 20%, une intensité laser de 25%, et 10 microns de résolution, à l'aide du scanner répertorié dans le Tableau de l'équipement et des matériaux.
12. Traitement et analyse des données
REMARQUE : Plusieurs progiciels sont disponibles pour la quantification des données de microarray avec des capacités similaires. La procédure décrite ici a été conçue pour le logiciel figurant dans la Table des équipements et des matériaux.
- Chargez les fichiers TIFF pour être quantifiés, concevez la grille pour correspondre à la disposition du microréseau et ajustez la taille des taches pour incorporer l'ensemble du signal avec la surface minimale possible. Les endroits voisins ne doivent pas se chevaucher. Inspectez visuellement la performance du logiciel et ajustez la grille manuellement, si nécessaire.
- Quantifier l'intensité du signal du microréseau. Inspectez visuellement les taches pour décevoir toute anomalie (liaison non spécifique, poussière, etc.) et retirez-les de l'analyse des données.
- Corriger l'arrière-plan localement en utilisant le signal des zones voisines sur le tableau dans lequel aucun endroit n'est présent.
- Normaliser les données. Pour comparer le signal entre différents tableaux, l'intensité du signal de chaque microréseau doit être normalisée. Pour exclure les valeurs aberrantes, normaliser les données à l'aide de la moyenne de 30% du signal du contrôle positif (taches IgG) des microréseaux déphosphorylés.
REMARQUE : Le signal de la tache d'IgG ne change pas pendant la phosphorylation et la déphosphorylation des microarrays et convient à la normalisation. - Identifiez les kinases actives. Pour chaque entité affichée sur le microréseau, calculez le rapport entre l'intensité du signal normalisé edans les tableaux autophosphorylés et les tableaux déphosphorylés. Fixez un seuil de 1,5 fois pour l'identification des kinases actives et marquez toutes les autres caractéristiques comme incapables de subir une autophosphorylation (N/A).
- Calculer l'activité de chaque kinase identifiée à l'étape 12.5 en pourcentage du signal ajusté (intensité du signal du tableau de contrôle positif normalisé (DMSO) soustrait par l'intensité du signal du tableau de contrôle négatif normalisé (déphosphoryé).
Résultats
Les microréseaux NAPPA auto-assemblés fournissent une plate-forme solide qui peut être utilisée pour de nombreuses applications distinctes, y compris la découverte de biomarqueurs, les interactions protéines-protéines, l'identification du substrat et le dépistage des médicaments10,11 ,12,13,14,15,16,17,18,19,20.
La méthodologie globale adoptée pour l'étude de l'activité kinase et le dépistage des inhibiteurs des kinases tyrosines sur les microréseaux NAPPA est représentée de façon schématique à la figure 1. Tout d'abord, les microréseaux NAPPA sont générés par l'immobilisation de l'ADNc et de l'agent de capture sur les microréseaux enduits. Les CDNA sont ensuite utilisés comme modèle pour la transcription et la traduction des protéines, à l'aide d'un système IVTT à base humaine, et les protéines nouvellement synthétisées sont immobilisées par l'agent de capture9. La qualité du microréseau imprimé peut être surveillée en mesurant les niveaux d'ADN (confirmant une impression cohérente) ou de protéines affichées sur le tableau (confirmant l'expression et la capture des protéines; Figure 1). Pour diminuer le signal de fond et augmenter la plage dynamique de l'expérience, les microréseaux sont traités avec 1) lambda phosphatase pour éliminer la phosphorylation des résidus de Ser/Thr/Tyr, puis avec 2) DNase pour simplifier la chimie sur place et diminuer (figure1).
L'étape suivante est la réaction d'autophosphorylation, dans laquelle les microréseaux sont incubés avec un tampon kinase en l'absence d'ATP (tableau de contrôle négatif, appelé microarrays déphosphorylés), et le tampon kinase est complété par l'ATP (contrôle positif, appelés tableaux autophosphorés) ou ATP et DMSO (contrôle des véhicules). Il convient de souligner qu'au cours de cette étape, aucune kinase n'est ajoutée; par conséquent, l'activité intrinsèque de chaque kinase affichée sur le microarray est quantifiée par la mesure de ses niveaux de phosphorylation à l'aide d'un anticorps pan antiphopho-tyrosine suivi d'un anticorps secondaire cy3-labbeled (figure 1).
Le contrôle de la qualité des tableaux NAPPA-kinase affichant un panneau de kinases de protéines humaines imprimés en quadruplicate est montré dans la figure 2. Les niveaux d'ADN immobilisé ont été mesurés par la coloration de l'ADN et il a montré un signal pair à travers le microarray, suggérant que la quantité d'ADN imprimée sur le tableau était uniforme. Il est également possible d'observer plusieurs caractéristiques sans aucune coloration d'ADN. Ces caractéristiques correspondent à certaines commandes dans lesquelles l'ADN a été omis du mélange d'impression [c.-à-d., taches vides (rien n'a été imprimé), taches d'eau, tache IgG purifiée (poly-lysine, crosslinker et IgG purifié), mélange d'impression seulement (mélange d'impression complet : poly-lysine plus crosslinker et anticorps anti-drapeau, sans ADN)]. Les niveaux de protéine affichés sur les microarrays nAPPA-kinase ont été évalués après la réaction d'IVTT utilisant l'anticorps anti-tag.
Pour le dépistage de la kinase, Flag a été utilisé comme étiquette de choix et le niveau de protéines affiché sur le microarray a été mesuré à l'aide d'un anticorps anti-drapeau. Comme indiqué, la majorité des taches contenant cDNA avec succès affiché des niveaux détectables de protéines. Certains des points de contrôle sans ADNc ont également révélé un signal avec l'anticorps anti-drapeau : igG spot (utilisé pour détecter l'activité de l'anticorps secondaire) et des taches vectorielles vides (codes d'ADNc pour l'étiquette seulement) (Figure 2). Les microréseaux nAPPA-kinase ont montré une bonne reproductibilité entre les diapositives, avec la corrélation des niveaux d'affichage des protéines parmi les lots d'impression distincts supérieurs à 0,88 (figure 2). Dans le même lot, la corrélation était encore plus élevée, proche de 0,92 (données non affichées).
Ensuite, l'activité d'autophosphorylation de kinase des protéines affichées sur le tableau a été mesurée à l'aide d'anticorps antiphospho-tyrosine (figure 3). Les protéines affichées sur le tableau présentaient des niveaux élevés de phosphorylation protéique après expression (Figure 3, gauche), qui peuvent être causés par l'activité intrinsèque de kinase de la protéine affichée sur le tableau ou par des kinases actives présentes dans le mélange IVTT. Cette phosphorylation a été complètement enlevée avec le traitement de phosphatase de lambda et ces microarrays ont été employés pour les essais de kinase. Après la déphosphorylation, les réactions d'autophosphorylation effectuées sans ATP n'ont montré aucun niveau significatif de phosphorylation, comme prévu, tandis que les microarrays incubés avec le tampon de kinase en présence de l'ATP ont montré la phosphorylation de protéine aussi rapide que 15 min ( Figure 3). Pour le dépistage des médicaments, l'activité de kinase a été mesurée après 60 min de réaction d'autophosphorylation pour maximiser le nombre de kinases testées.
La comparaison entre les microréseaux dans lesquels les niveaux de phosphorylation ont été mesurés juste après l'expression des protéines (Figure 3, à gauche) et après 60 minutes de réaction d'autophosphorylation(figure 3, droite) a montré : i) protéines phosphorylées seulement après expression, suggérant qu'ils peuvent être exogènement phosphorylés par les protéines présentes sur le mélange IVTT, mais ne peuvent pas être autophosphorylated ; ii) protéine phosphorylated seulement après la réaction d'autophosphorylation, suggérant que ces protéines n'étaient pas actives après l'expression de protéine et ont exigé des co-facteurs présents dans le tampon de kinase pour être actifs ; ou iii) de protéines phosphorylées sur les deux tableaux, ce qui suggère qu'ils étaient actifs dans les deux milieux (figure 3).
À titre d'exemple des résultats obtenus pour le dépistage des inhibiteurs de la tyrosine kinase sur les tableaux de nAPPA-kinase, trois inhibiteurs de la kinases ayant une sélectivité distincte entre les kinases protéiques ont été utilisés : staurosporine, imatinib et ibrutinib. Pour tous les dépistages, les microréseaux NAPPA déphosphorylés ont été incubés avec des concentrations croissantes de TKI (allant de 100 nM à 10 uM) pendant la réaction d'autophosphorylation. Le premier TKI testé était la staurosporine, un inhibiteur mondial de la kinase protéique, qui a montré une inhibition puissante de la kinase sur le microarray à travers pratiquement toutes les kinases testées11.
Ensuite, l'imatinib a été testé, un inhibiteur ABL et c-Kit utilisé pour le traitement de la leucémie myélogène chronique et les tumeurs stromales gastro-intestinales4,5,6,7. Sur les tableaux NAPPA-kinase, l'imatinib a montré une réduction significative de l'activité Abl1 et BCR-Abl1 alors que d'autres kinases sont restées pour la plupart inchangées (figure 4A). La quantification des données pour l'activité de kinase a été normalisée par rapport au tableau déphosphorylé et représentée en pourcentage du microréseau de contrôle positif (véhicule seulement). Les données relatives à TNK2 (kinase non pertinente), Abl1 et BCR-Abl1 sont présentées à la figure 4B. Comme prévu, l'imatinib a montré l'inhibition sélective vers Abl1 et BCR-ABl1. Les données pour c-Kit n'étaient pas concluantes en raison du manque d'activité sur les tableaux de contrôle positifs.
Enfin, l'ibrutinib, un inhibiteur covalent approuvé par la FDA de la tyrosine kinase de Bruton (BTK), a été testé. L'ibrutinib est actuellement utilisé dans le traitement de plusieurs cancers liés au sang avec la BTK hyperactive, y compris la leucémie lymphocytique chronique (LCL), le lymphome de cellules de manteau, et la macroglobulinemia de Waldenstrâm21,22. Figure 4C, est représentatif des résultats typiques obtenus pour le dépistage de l'ibrutinib. L'activité kinase d'ABL1 (kinase non pertinente) et de BTK (cible canonique) et d'ERBB4 (nouvelle cible potentielle) est indiquée à la figure 4D. Les données suggèrent ERBB4 peut être inhibé par l'ibrutinib d'une manière spécifique dose. Cette inhibition a été confirmée in vitro et dans les essais cellulaires11, démontrant la puissance de cette plate-forme.
Prises ensemble, les données suggèrent que la plate-forme de microréseau NAPPA-kinase pourrait être utilisée pour le dépistage impartial des inhibiteurs des atS. En outre, le dépistage est rapide et peut être facilement personnalisé pour inclure toute variation des kinases protéiques d'intérêt.
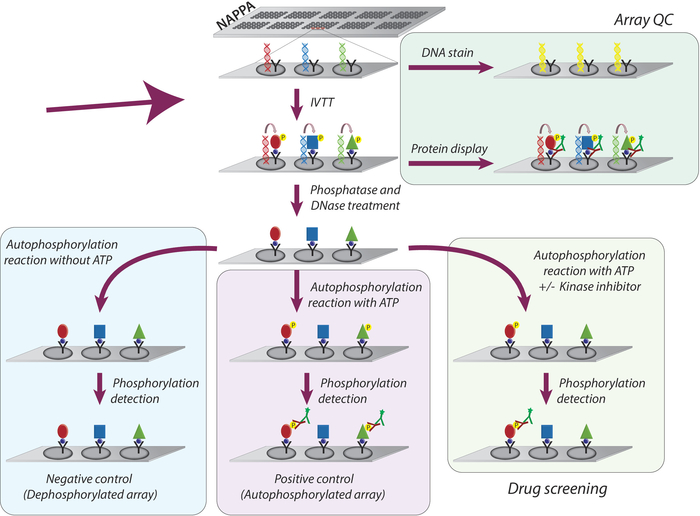
Figure 1 : Représentation schématique du contrôle de la qualité et du criblage des inhibiteurs de la tyrosine kinase dans les tableaux NAPPA. Les tableaux NAPPA sont imprimés avec un codage de cDNA pour la protéine d'intérêt fusionnée avec une étiquette et un anticorps de capture. Pendant la transcription in vitro et la réaction de traduction (IVTT), les protéines synthétisées sont capturées sur la surface du microarray à travers l'étiquette par l'anticorps de capture. Le contrôle de la qualité (QC) des tableaux est effectué par la mesure des niveaux d'ADN imprimés sur la diapositive, à l'aide d'un colorant fluorescent intercalant l'ADN, et les niveaux de protéines affichées sur le tableau à l'aide d'anticorps spécifiques aux étiquettes. Pour le criblage de kinase, les microarrays sont traités avec le DNase et la phosphatase, après la réaction d'IVTT, pour enlever l'ADN imprimé et toute la phosphorylation qui a pu s'être produite pendant la synthèse de protéine. Les tableaux déphosphorylés sont maintenant prêts à être utilisés pour l'écran de drogue. Pour chaque test, trois ensembles de contrôles sont couramment utilisés : (I) tableaux déphosphorylés, dans lesquels la réaction d'autophosphorylation est effectuée sans ATP; (II) microréseaux autophosphorés, dans lesquels la réaction d'autophosphorylation est effectuée en présence de l'ATP; et (III) tableau traité DMSO (véhicule), dans lequel la réaction d'autophosphorylation est effectuée avec ATP et DMSO. Les diapositives traitées avec une concentration différente d'inhibiteurs de la kinase suivent exactement le même protocole utilisé pour les tableaux traités Par DMSO. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
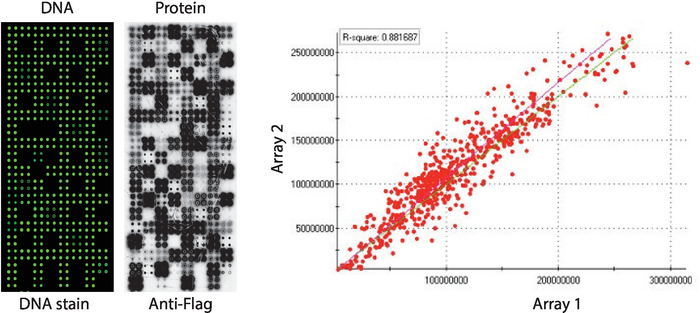
Figure 2 : Résultats représentatifs du contrôle de la qualité pour les tableaux NAPPA-kinase auto-assemblés. La teneur en ADN mesurée par un colorant fluorescent intercalant l'ADN (à gauche) et les niveaux de protéines affichés sur le microréseau mesuré par l'anticorps anti-Flag (au milieu) sont montrés. Sur le côté droit se trouve une parcelle de corrélation des niveaux de protéines affichées sur deux tableaux NAPPA-kinase imprimés en lots distincts. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
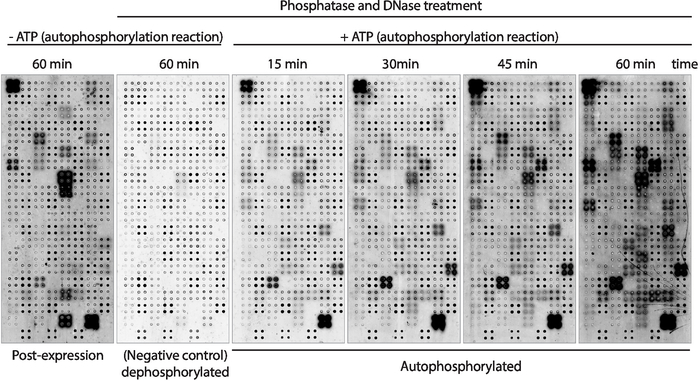
Figure 3 : Résultats représentatifs de l'activité kinase dans les tableaux NAPPA-kinase. Des microarrays affichant des kinases de protéine dans le quadruplicate ont été employés pour étudier l'activité de kinase de protéine sur le réseau par la mesure de la phosphorylation de protéine utilisant l'anticorps anti-pTyr, suivi de l'anticorps anti-souris cy3-étiqueté. Des tableaux de contrôle sans traitement de phosphatase/DNase et sans ATP pendant la réaction d'autophosphorylation ont été employés pour mesurer la phosphorylation de fond après expression de protéine (post-expression). Les microréseaux restants ont été traités avec la phosphatase/ADN, et la réaction d'autophosphorylation a été exécutée sans ATP (microarray déphosphorylé, contrôle négatif) ou avec ATP (microarrays autophosphorylated). Pour les microréseaux autophosphorylated la réaction d'autophosphorylation a été exécutée pendant 15 min, 30 min, 45 min, ou 60 min, comme indiqué. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
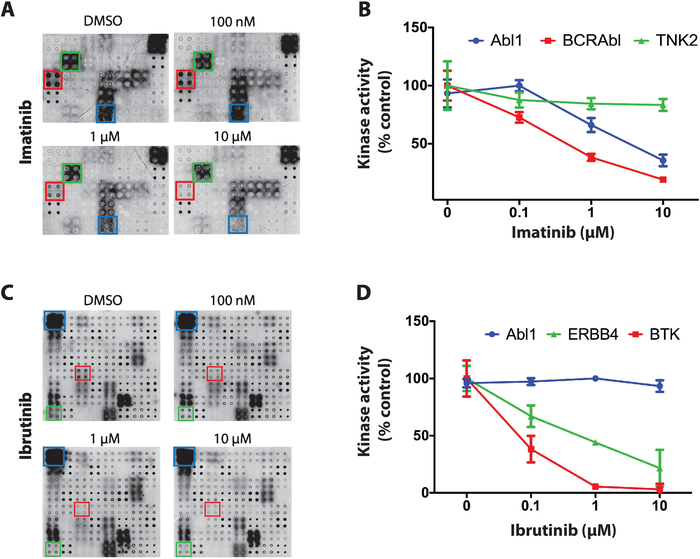
Figure 4 : Données représentatives de l'écran tyrosine kinase sur les tableaux NAPPA-kinase. (A) Les tableaux traités par phosphatase/DNase ont été incubés dans des concentrations croissantes d'imatinib pendant la réaction d'autophosphorylation et l'activité de kinase a été mesure avec l'anticorps anti-phospho-tyr. (B) Quantification de l'activité kinase observée sur les tableaux NAPPA-kinase exposés à l'imatinib. Les données ont été normalisées par rapport au signal des tableaux de contrôle négatifs (déphosphoryés) et elles sont indiquées en pourcentage des tableaux de contrôle positifs (réaction d'autophosporylation effectuée en présence de DMSO). Des données similaires sont présentées pour le dépistage de l'ibrutinib (C,D). Ce chiffre a été modifié à partir de Rauf et coll.11. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Dossier supplémentaire 1. Protocole alternatif pour le criblage des inhibiteurs de la tyrosine kinase dans les tableaux NAPPA à l'aide d'une station d'hybridation automatisée. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Modifications et dépannage
Au cours de la phase d'optimisation de l'étude de l'activité kinase sur les tableaux NAPPA, l'une des principales sources de fond et de faible plage dynamique observée était le BSA utilisé sur le mélange d'impression. BSA fournissait les principales amines nécessaires pour croiser la surface de l'aminosilane et piégeait l'ADN et l'anticorps de capture sur place. Cependant, le BSA est fortement phosphorylé, ce qui rend difficile la détection du signal d'autophosphorylation sur le tableau au-dessus du bruit de fond. Pour résoudre ce problème, plusieurs alternatives pour BSA dans le mélange d'impression ont été testées, et la polylysine a été identifiée comme bon substitut. La polylysine n'a aucun site de phosphorylation; par conséquent, l'arrière-plan des tableaux non exprimés est très minime. De plus, les microréseaux imprimés en polylysine sont reproductibles et présentent de bons niveaux de protéines (figure 2).
La modification critique suivante effectuée sur l'analyse standard de NAPPA était l'addition d'une étape de traitement de Phosphatase/DNase. Le traitement des microréseaux avec de la phosphatase permet d'éliminer toute phosphorylation qui s'est produite dans le mélange IVTT lors de la synthèse et de la capture des protéines (Figure 3). La source de cette phosphorylation pourrait provenir de l'activité intrinsèque d'autophosphorylation ou de l'activité des kinases présentes dans le mélange IVTT. L'élimination de toute post-expression de phosphorylation a permis d'identifier facilement les kinases qui sont actives et peuvent subir une autophosphorylation (figure 3).
Étapes critiques du protocole
NAPPA est une technologie robuste, mais comme prévu, il ya plusieurs étapes critiques. La première est l'acquisition d'ADN de haute qualité dans la concentration appropriée. L'utilisation d'ADN de mauvaise qualité ou en faibles concentrations générera des microréseaux de mauvaise qualité, plusieurs caractéristiques n'étant pas exprimées et affichées dans les niveaux appropriés, ce qui diminuera le nombre de protéines analysées sur le tableau. La deuxième étape critique est l'expression des protéines sur le microarray. L'utilisation d'un système IVTT qui exprimera des niveaux élevés de protéines fonctionnelles est essentielle pour étudier l'activité kinase sur le tableau.
La prochaine étape critique sur le dépistage DelI est la façon dont les microréseaux sont gérés. Les microréseaux ne doivent pas sécher pendant n'importe quelle étape du protocole, et la manipulation douce est recommandée pour empêcher des rayures qui peuvent augmenter le signal de fond. Étant donné que les tableaux de toute l'expérience seront comparés les uns aux autres, il est important de s'assurer que chaque étape d'incubation est même sur toutes les diapositives. Par exemple, le temps nécessaire pour effectuer une étape dans un seul tableau doit être pris en considération lorsqu'un lot de 20 tableaux est traité pour prévenir les différences dans la durée d'incubation entre les tableaux.
Enfin, la conception de l'expérience et l'inclusion de contrôles positifs et négatifs sont essentielles pour le contrôle de la qualité et l'analyse des données. La première série de contrôles sont celles imprimées dans chaque tableau et comprend des contrôles négatifs [c.-à-d. des taches vides (sans aucun matériau imprimé), de l'eau ou un vecteur vide (n'exprimez que l'étiquette)], ainsi qu'un contrôle positif (c.-à-d. IgG purifié, qui est détecté par le anticorps secondaires et est inerte à l'altération des niveaux de phosphorylation). Collectivement, ils mesurent les niveaux de fond du microarray, le report possible lors de l'impression et l'intensité du signal de la méthode de détection.
La prochaine série de contrôles sont les contrôles de dépistage des médicaments et comprennent les microréseaux déphosphorylés et autophosphorylés (en présence ou en absence de DMSO). Comme mentionné précédemment, le microarray déphosphorylated mesure le niveau de phosphorylation après le traitement de phosphatase et donc le niveau de base pour toutes les autres expériences. Plus le niveau de base est bas, plus la plage dynamique des essais est élevée. Les tableaux autophosphorés présentent les niveaux maximums de phosphorylation de tous les tableaux et le signal doit être fort et clair. Il est utilisé pour l'analyse des données, mais aussi comme un contrôle que toutes les réactions ont été effectuées avec succès sur le tableau.
Limites de la technique
À partir de maintenant, l'une des limites du dépistage des médicaments présenté ici est sa capacité à dépister uniquement les kinases protéiques qui peuvent être autophosphorylated. Une façon possible de surmonter cela est d'imprimer un kinase et substrat connu au même endroit. La co-impression de l'ADN pour deux protéines distinctes a été accomplie avec succès15, suggérant la faisabilité de cette approche. En outre, la protéine affichée sur le tableau peut ne pas être pliée correctement résultant en une protéine inactive. L'utilisation du système d'expression basé sur l'homme a apporté une amélioration significative de l'activité kinase mesurée sur le tableau; cependant, certaines protéines ne peuvent toujours pas être analysées sur le tableau en raison de son inactivité.
Une deuxième limitation est la mesure de la phosphorylation à l'aide d'un anticorps pan antiphospho-tyr. En dépit de sa non-spécificité concernant le motif du site de phosphorylation, toutes les phosphorylations mesurées se sont produites sur des résidus de tyrosine, laissant derrière eux des serines et des thréonines et leurs kinases respectives. À ce jour, plus de 10 anticorps pan phospho-Ser/Thr ont été testés sans succès, malgré plusieurs tentatives pour optimiser les conditions d'incubation et de lavage. Un nouveau système de détection indépendant des anticorps peut être la meilleure option pour augmenter le nombre de kinases protéiques qui peuvent être dépistées pour l'inhibition des médicaments. Dans ce contexte, quelques options sont disponibles, y compris la radioactivité ou des approches chimiques comme la conjugaison par clic. Une série d'optimisations est nécessaire pour minimiser le signal d'arrière-plan et fournir une bonne plage dynamique pour les essais.
La troisième limitation est l'acquisition de clones d'ADNc à imprimer sur le tableau. Les clones d'ADNc peuvent être générés à l'aide de n'importe quelle technique de clonage, y compris des systèmes de recombinaison spécifiques au site, tels que Creator ou Gateway23. Une autre option est d'acheter les clones de la bibliothèque DNAsu, trouvé à lt;https://dnasu.org/DNASU/Home.do-gt;, où plus de 17.000 clones cDNAs, y compris l'ensemble du kinome humain, est facilement disponible pour être utilisé pour la construction de tableaux NAPPA24 .
La quatrième limite est que tous les laboratoires ne sont pas équipés d'équipements appropriés pour fabriquer et filtrer leurs propres tableaux NAPPA. Ce protocole fournit des méthodes alternatives pour générer l'ADN à imprimer sur le microréseau, sans avoir besoin d'équipement à haut débit, et des protocoles pour effectuer manuellement toutes les étapes d'hybridation. Cependant, l'accès à un scanner de tableau et de microarray est toujours nécessaire. Une option pour surmonter ce problème est d'utiliser le service de base NAPPA et l'installation, trouvé à lt;http://nappaproteinarray.org / 'gt;, qui distribue personnalisé salan iemn à un prix académique à but non lucratif. Enfin, comme de toute méthode de dépistage, les données obtenues sur les tableaux sont susceptibles d'être des artefacts (positifs ou négatifs) et devraient donc être validées à l'aide d'analyses orthogonales.
Importance par rapport aux méthodes existantes
Plusieurs plates-formes sont disponibles dans le commerce pour le dépistage des kinases protéiques. Une approche couramment utilisée est l'analyse de liaison, qui peut être effectuée avec des fragments de protéines, domaine kinase, de plus grands fragments de protéines avec le domaine kinase et certaines régions de régulation, et même des protéines pleine longueur. Les protéines sont généralement exprimées dans les systèmes bactériens en raison du coût et de la simplicité dans les protocoles d'expression et de purification. L'interaction entre le médicament d'intérêt et la protéine est ensuite mesurée avec un certain type d'évaluation de rapport comme la fluorescence ou la présence d'étiquettes, par exemple. La principale limitation de cet ensemble d'approches est le fait que la protéine n'est pas nécessairement active lors de l'interaction avec le médicament, ce qui peut entraîner l'identification de fausses interactions positives et fausses et négatives. Les fragments de protéines sont particulièrement vulnérables aux changements dans la conformation et le manque d'activité et toutes les données obtenues doivent être validées à l'aide de protéines actives, de préférence sous leur forme intégrale. Une autre limitation de certaines plates-formes est la capacité de filtrer uniquement les analogues ATP, limitant son utilisation globale.
La plupart des services disponibles dans le commerce pour le dépistage des ITC à l'aide d'approches enzymatiques n'utilisent que des versions sauvages de la kinase d'intérêt, et parfois seulement quelques mutants sélectionnés. Sachant que la résistance aux médicaments est très fréquente chez les patients traités par TKI, il est important d'être en mesure de mesurer la réponse médicamentelle chez différents mutants, pour la sélection de l'inhibiteur le plus approprié. En raison de la nature de nAPPA, le criblage des mutants de kinase est simple et peut être facilement accompli, et le seul outil exigé est l'incorporation du mutant de kinase dans la collection de cDNA de NAPPA, qui peut être faite par mutagénèse spécifique au site, par exemple.
Applications futures
L'une des formes les plus courantes de traitement s'écoule ntlapse dans le traitement du cancer à l'aide d'inhibiteurs de la kinase est l'acquisition de mutations dans la cible médicamenteuse au cours d'un cours de traitement. Le dépistage de ces mutants concernant leur réponse aux inhibiteurs de la kinase est d'une importance vitale pour la sélection de la deuxième/troisième génération de TKIs afin d'obtenir un traitement personnalisé pour chaque patient. L'approche de dépistage des médicaments présentée ici, fournit une plate-forme de dépistage impartiale dans laquelle tout inhibiteur de la tyrosine kinase peut être testé contre un panel de kinases tyrosine présents dans le génome humain. Étant donné que les protéines affichées sur les tableaux NAPPA sont exprimées in vitro à partir de l'ADNc imprimé sur la diapositive, n'importe quelle variante mutante peut facilement être incorporée dans la collection d'ADNc pour être affichée sur le tableau. L'installation dans laquelle les mutants kinase peuvent être générés et exprimés sur le tableau, combiné avec la puissance à haut débit de la technique NAPPA, fournit un environnement unique pour l'étude des mutants kinase et leur réponse aux médicaments, ce qui rend NAPPA approprié pour dépistage personnalisé des médicaments, l'un des objectifs de la médecine de précision.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d'intérêts.
Remerciements
Les auteurs tient à remercier tout le monde au laboratoire de LaBaer pour leur aide et leurs critiques lors du développement du projet. Ce projet a été soutenu par la subvention des NIH U01CA117374, U01AI077883 et Virginia G. Piper Foundation.
matériels
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| 364 well plates (for arraying) | Genetix | x7020 | |
| 800 µL 96-well collection plate | Abgene | AB-0859 | |
| 96-pin device | Boekel | 140500 | |
| Acetic Acid | Millipore-Sigma | 1.00066 | |
| Acetone 99.9% | Millipore Sigma | 650501 | |
| Aluminum seal for 96 well plates | VWR | 76004-236 | |
| Aminosilane (3-aminopropyltriethoxysilane) | Pierce | 80370 | |
| ANTI-FLAG M2 antibody produced in mouse | Millipore Sigma | F3165 | |
| Anti-Flag rabbit Antibody (polyclonal) | Millipore Sigma | F7425 | |
| ATP 10 mM | Cell Signaling | 9804S | |
| β-Glycerophosphate disodium salt hydrate | Millipore-Sigma | G9422 | |
| bacteriological agar | VWR | 97064-336 | |
| Blocking Buffer | ThermoFisher/Pierce | 37535 | |
| Brij 35 | ThermoFisher/Pierce | BP345-500 | |
| BS3 (bis-sulfosuccinimidyl) | ThermoFisher/Pierce | 21580 | |
| BSA (bovine serum albumin) | Millipore Sigma | A2153 | |
| Coverslip 24 x 60 mm | VWR | 48393-106 | |
| Cy3 AffiniPure Donkey Anti-Mouse IgG (H+L) | Jackson ImmunoResearch | 715-165-150 | |
| DeepWell Block, case of 50 | ThermoFisher/AbGene | AB-0661 | |
| DEPC water | Ambion | 9906 | |
| DMSO (Dimethyl Sulfoxide) | Millipore-Sigma | D8418 | |
| DNA-intercalating dye | Invitrogen | P11495 | |
| DNase I | Millipore-Sigma | AMPD1-1KT | |
| DTT | Millipore-Sigma | 43816 | |
| EDTA | Millipore-Sigma | EDS | |
| Ethanol 200 proof | Millipore-Sigma | E7023 | |
| Filter plates | Millipore-Sigma | WHA77002804 | |
| Gas Permeable Seals, box of 50 | ThermoFisher/AbGene | AB-0718 | |
| Glass box | Wheaton | 900201 | |
| Glass slides | VWR | 48300-047 | |
| Glycerol | Millipore-Sigma | G5516 | |
| HCl (Hydrochloric acid) | Millipore-Sigma | H1758 | |
| HEPES Buffer Solution | Millipore-Sigma | 83264 | |
| Human-based IVTT system | Thermo Scientific | 88882 | |
| ImmunoPure Mouse IgG whole molecule | ThermoFisher/Pierce | 31202 | |
| Isopropanol | Millipore-Sigma | I9516 | |
| KCl (Potassium chloride) | Millipore-Sigma | P9333 | |
| KH2PO4(Potassium phosphate monobasic) | Millipore-Sigma | P5655 | |
| Kinase buffer | Cell Signaling | 9802 | |
| KOAc (Potassium acetate) | Millipore-Sigma | P1190 | |
| Lambda Protein Phosphatase | new england biolabs | P0753 | |
| Lifterslips, 24 x 60 mm | ThermoFisher Scientific | 25X60I24789001LS | |
| Metal 30-slide rack with no handles | Wheaton | 900234 | |
| MgCL2 (Magnesium chloride) | Millipore-Sigma | M8266 | |
| Na3VO4 (Sodium orthovanadate) | Millipore-Sigma | S6508 | |
| NaCl (Sodium Chloride) | Millipore-Sigma | S3014 | |
| NaOAc (Sodium acetate) | Millipore-Sigma | S2889 | |
| NaOH (Sodium hydroxide) | Millipore-Sigma | S8045 | |
| NucleoBond Xtra Midi / Maxi | Macherey-Nagel | 740410.10 / 740414.10 | |
| Nucleoprep Anion II | Macherey Nagel | 740503.1 | |
| Phosphoric Acid | Millipore-Sigma | 79617 | |
| Poly-L-Lysine Solution (0.01%) | Millipore-Sigma | A-005-C | |
| Protein Phosphatase (Lambda) | New England Biolabs | P0753 | |
| RNAse | Invitrogen | 12091021 | |
| SDS (Sodium dodecyl sulfate) | Millipore-Sigma | L6026 | |
| SDS (Sodium dodecyl sulfate) | Millipore-Sigma | 05030 | |
| Sealing gasket | Grace Bio-Labs, Inc | 44904 | |
| Silica packets | VWR | 100489-246 | |
| Single well plate | ThermoFisher/Nalge Nunc | 242811 | |
| Sodium acetate (3M, pH 5.5) | Millipore-Sigma | 71196 | |
| TB media (Terrific Broth) | Millipore-Sigma | T0918 | |
| Tris | IBI scientific | IB70144 | |
| Triton X-100 | Millipore-Sigma | T8787 | |
| Tryptone | Millipore-Sigma | T7293 | |
| Tween 20 | Millipore-Sigma | P9416 | |
| Yeast Extract | Millipore-Sigma | Y1625 | |
| Name | Company | Catalog number | Comments |
| Equipments | Maker/model | ||
| Programmable chilling incubator | Torrey Pines IN30 Incubator with Cooling | ||
| Shaker for bacterial growth | ATR Multitron shaker | ||
| Vacuum manifold with liquid waste trap | MultiScreenVacuum Manifold 96 well | ||
| 96 well autopippetor/liquid handler | Genmate or Biomek FX | ||
| Liquid dispenser | Wellmate | ||
| DNA microarrayer | Genetix QArray2 | ||
| Automatic hybridization station | Tecan HS4800 Pro Hybridization Station | ||
| Microarray scanner | Tecan PowerScanner | ||
| Microarray data quantification | Tecan Array-ProAnalyzer 6.3 |
Références
- Melnikova, I., Golden, J. Targeting protein kinases. Nature Review Drug Discovery. 3 (12), 993-994 (2004).
- Patterson, H., Nibbs, R., McInnes, I., Siebert, S. Protein kinase inhibitors in the treatment of inflammatory and autoimmune diseases. Clinical and Experimental Immunology. 176 (1), 1-10 (2014).
- Wu, P., Nielsen, T. E., Clausen, M. H. FDA-approved small-molecule kinase inhibitors. Trends Pharmacological Sciencies. 36 (7), 422-439 (2015).
- Druker, B. J., et al. Effects of a selective inhibitor of the Abl tyrosine kinase on the growth of Bcr-Abl positive cells. Nature Medicine. 2 (5), 561-566 (1996).
- Heinrich, M. C., et al. Inhibition of c-kit receptor tyrosine kinase activity by STI 571, a selective tyrosine kinase inhibitor. Blood. 96 (3), 925-932 (2000).
- Stagno, F., et al. Imatinib mesylate in chronic myeloid leukemia: frontline treatment and long-term outcomes. Expert Review Anticancer Therapy. 16 (3), 273-278 (2016).
- Ben Ami, E., Demetri, G. D. A safety evaluation of imatinib mesylate in the treatment of gastrointestinal stromal tumor. Expert Opinions in Drug Safety. 15 (4), 571-578 (2016).
- Ramachandran, N., et al. Self-assembling protein microarrays. Science. 305 (5680), 86-90 (2004).
- Festa, F., et al. Robust microarray production of freshly expressed proteins in a human milieu. Proteomics Clinical Applications. 7 (5-6), 372-377 (2013).
- Yazaki, J., et al. Mapping transcription factor interactome networks using HaloTag protein arrays. Proceedings of the National Academy of Sciences of the United States of America. 113 (29), E4238-E4247 (2016).
- Rauf, F., et al. Ibrutinib inhibition of ERBB4 reduces cell growth in a WNT5A-dependent manner. Oncogene. 37 (17), 2237-2250 (2018).
- Anderson, K. S., et al. Protein microarray signature of autoantibody biomarkers for the early detection of breast cancer. Journal of Proteome Research. 10 (1), 85-96 (2011).
- Wang, J., et al. Plasma Autoantibodies Associated with Basal-like Breast Cancers. Cancer Epidemiol Biomarkers Prevention. 24 (9), 1332-1340 (2015).
- Bian, X., et al. Tracking the Antibody Immunome in Type 1 Diabetes Using Protein Arrays. Journal of Proteome Research. 16 (1), 195-203 (2017).
- Song, L., et al. Identification of Antibody Targets for Tuberculosis Serology using High-Density Nucleic Acid Programmable Protein Arrays. Molecular and Cellular Proteomics. 16 (4 suppl 1), S277-S289 (2017).
- Wang, J., et al. Comparative Study of Autoantibody Responses between Lung Adenocarcinoma and Benign Pulmonary Nodules. Journal of Thoracic Oncology. 11 (3), 334-345 (2016).
- Montor, W. R., et al. Genome-wide study of Pseudomonas aeruginosa outer membrane protein immunogenicity using self-assembling protein microarrays. Infection and Immunity. 77 (11), 4877-4886 (2009).
- Tang, Y., Qiu, J., Machner, M., LaBaer, J. Discovering Protein-Protein Interactions Using Nucleic Acid Programmable Protein Arrays. Current Protocols in Cell Biology. 74, 11-15 (2017).
- Yu, X., et al. Copper-catalyzed azide-alkyne cycloaddition (click chemistry)-based detection of global pathogen-host AMPylation on self-assembled human protein microarrays. Molecular and Cellular Proteomics. 13 (11), 3164-3176 (2014).
- Anderson, K. S., et al. Autoantibody signature for the serologic detection of ovarian cancer. Journal of Proteome Research. 14 (1), 578-586 (2015).
- Woyach, J. A., Johnson, A. J., Byrd, J. C. The B-cell receptor signaling pathway as a therapeutic target in CLL. Blood. 120 (6), 1175-1184 (2012).
- Smith, M. R. Ibrutinib in B lymphoid malignancies. Expert Opinion on Pharmacotherapy. 16 (12), 1879-1887 (2015).
- Festa, F., Steel, J., Bian, X., Labaer, J. High-throughput cloning and expression library creation for functional proteomics. Proteomics. 13 (9), 1381-1399 (2013).
- Seiler, C. Y., et al. DNASU plasmid and PSI:Biology-Materials repositories: resources to accelerate biological research. Nucleic Acids Research. 42, D1253-D1260 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon