Method Article
Triagem de inibidores da quinase em Microarrays de proteínas humanas Automontados
Neste Artigo
Resumo
Um protocolo detalhado para a geração de Microarrays humanos Self-assembled da proteína para a seleção de inibidores da quinase é apresentado.
Resumo
A triagem de inibidores da quinase é crucial para uma melhor compreensão das propriedades de um fármaco e para a identificação de alvos potencialmente novos com implicações clínicas. Várias metodologias têm sido relatadas para realizar tal triagem. No entanto, cada um tem suas próprias limitações (por exemplo, a triagem de apenas análogos de ATP, restrição ao uso de domínios da quinase purificada, custos significativos associados ao teste de mais de algumas quinases de cada vez, e falta de flexibilidade no rastreamento de proteínas quinases com mutações novas). Aqui, um novo protocolo que supera algumas dessas limitações e pode ser usado para a triagem imparcial de inibidores da quinase é apresentado. A força deste método é a sua capacidade de comparar a atividade dos inibidores da quinase através de múltiplas proteínas, quer entre diferentes cinases ou variantes diferentes da mesma quinase. Os microarrays automontados da proteína gerados com a expressão de quinases da proteína por um sistema in vitro da transcrição e da tradução do humano-baseado (ivtt) são empregados. As proteínas exibidas no microarray são ativas, permitindo a mensuração dos efeitos dos inibidores da quinase. O procedimento a seguir descreve os passos de protocolo em detalhes, da geração de Microarray e triagem para a análise de dados.
Introdução
As proteínas quinases são responsáveis pela fosforilação de seus alvos e podem modular vias moleculares complexas que controlam muitas funções celulares (i.e., proliferação celular, diferenciação, morte celular e sobrevida). A desregulação da atividade da quinase está associada a mais de 400 doenças, tornando os inibidores da quinase uma das principais classes de medicamentos disponíveis para o tratamento de várias doenças, incluindo câncer, distúrbios cardiovasculares e neurológicos, bem como inflamatória e doenças auto-imunes1,2,3.
Com o advento da medicina de precisão, a identificação de novas terapias, especialmente os inibidores da quinase, têm grande apelo farmaceuticamente e clinicamente. Várias abordagens podem ser usadas para a identificação de possíveis novos pares de inibidores da quinase/quinase, incluindo o design de novo de inibidores da quinase e identificação de novos alvos para os medicamentos aprovados pela FDA existentes. Este último é especialmente atraente, uma vez que o tempo e o dinheiro necessários para implementar essas drogas em clínicas são drasticamente reduzidos devido à disponibilidade de dados de ensaios clínicos anteriores. Um exemplo canônico do Repurposing de um inibidor da quinase é imatinib, projetado inicialmente para o tratamento da leucemia Myelogenous crônica (LMC) com a inibição de BCR-abl, que pode igualmente com sucesso ser usado para o tratamento do c-kit que sobreexpressa tumores estromais gastrointestinais (GISTs)4,5,6,7.
A triagem de inibidores da quinase pode ser realizada em ensaios de ligação ou em ensaios enzimáticos. A primeira classe de ensaios centra-se em interações proteína-droga e pode fornecer informações como local de ligadura e afinidade. Desde que a atividade da quinase na altura destes ensaios é desconhecida, um número de interações pode ser faltado ou identificado falsa devido às mudanças conformacional na proteína. Por outro lado, os ensaios enzimáticos exigem que as proteínas cinases sejam ativas e forneçam informações valiosas sobre o efeito do inibidor na atividade enzimática, entretanto, esse tipo de triagem é geralmente mais demorado e dispendioso. Atualmente, ambos os tipos de ensaios estão disponíveis comercialmente a partir de várias fontes. Eles representam uma opção confiável para a triagem de inibidores da quinase com algumas limitações, incluindo: I) a maioria dos métodos envolvem testes de múltiplas cinases individualmente, o que pode tornar a triagem de um grande conjunto de proteínas dispendiosas; II) o conjunto de quinases a serem testados limita-se a uma lista de cinases pré-selecionadas, de tipo selvagem e de várias versões mutadas conhecidas de algumas cinases, dificultando o teste de muitas novas isoformas mutadas.
Neste contexto, os microarrays proteicos são uma plataforma poderosa capaz de superar algumas das limitações apresentadas pelas técnicas comercialmente disponíveis. É apropriado executar ensaios enzimáticos na triagem de alta taxa de transferência usando proteínas ativas de comprimento total de qualquer sequência de interesse. Os microarrays podem ser gerados por uma abordagem automontada como a NAPPA (matriz de proteínas programáveis de ácido nucleico), na qual as proteínas são expressas apenas a tempo para os ensaios, aumentando a probabilidade de que aqueles exibidos na matriz estejam realmente ativos. As proteínas exibidas no NAPPA são produzidas com ribossomas e proteínas de Chaperona derivadas de humanos, a fim de melhorar a probabilidade de dobramento e atividade natural.
As proteínas são programadas inicialmente imprimindo cDNAs que codificam para os genes do interesse fundidos com uma etiqueta da captação, junto com um agente da captação, na superfície do Microarray. As proteínas são produzidas então nos Microarrays usando um sistema in vitro da transcrição e da tradução (IVTT), e as proteínas recentemente expressas são imobilizadas na superfície do microarray pelo agente da captação. Os arrays Nappa expressos podem ser usados para o estudo das proteínas exibidas na matriz de forma imparcial e de altataxa de transferência8,9.
Anteriormente, mostrou-se que as proteínas exibidas em matrizes NAPPA são dobradas corretamente para interagir com parceiros conhecidos10; Além disso, sua atividade enzimática foi explorada pela primeira vez em 2018, quando foi demonstrado que as proteínas quinases exibidas no autofoslato de Microarray11. Até o momento, a metodologia Nappa tem sido utilizada para muitas aplicações distintas, incluindo a descoberta de biomarcadores12,13,14,15,16,17, interações proteína-proteína10,18, identificação do substrato19e triagem de fármacos11. Sua flexibilidade é uma das principais características da plataforma que permite a adaptação a cada aplicação.
Aqui, um protocolo para a seleção de inibidores da tirosina quinase em matrizes Self-assembled de NAPPA é apresentado. A plataforma é otimizada para a exibição de proteínas humanas ativas e para a análise da atividade da proteína quinase, com baixo nível de fundo e alta amplitude dinâmica. Entre as modificações implementadas para o uso de NAPPA para a triagem de inibidores da quinase incluem: I) alterações na química da impressão, II) de-fosforilação do Microarranjo proteico antes da triagem do inibidor da quinase, e III) otimização da detecção de proteínas fosforiladas na matriz. Este protocolo é o primeiro de seu tipo e fornece a informação original sobre o estudo da quinase em Microarrays de NAPPA.
Protocolo
1. buffers comuns e soluções a serem usadas
- Prepare o meio de TB: caldo fantástico (24 g/L extrato de levedura; 20 g/L de triptona; 4 mL/L de glicerol; 0, 17 M KH2po4; e 0, 72 m K2HPO4). As soluções 0, 17 MKH 2po4 e 0, 72 m k2HPO4 podem ser adquiridas como tampão fosfato 10x (0,17 m KH2po4 e 0,72 m k2HPO4).
- Prepare o meio LB: Luria-Bertani (5 g/L extrato de levedura; 10 g/L de triptona; e 10 g/L NaCl). Ajuste o pH a 7,0 com NaOH de 5 M.
- Prepare 1x TBS: solução salina tampão Tris (TBS: 50 mM Tris-Cl, pH = 7,5; 150 mM NaCl).
- Prepare 1x TBST: TBS suplementado com 0,1% Tween 20.
2. preparação do ADN
Nota: o DNA utilizado para matrizes NAPPA deve ser altamente puro; Conseqüentemente, os mini-Preps comerciais do ADN não são recomendados. Atualmente, dois protocolos para a preparação do ADN são usados: na mini-preparação high-throughput da casa (descrita aqui) ou MIDI-ou Maxi-Prep comercial. A taxa de transferência média do protocolo de mini-Prep em casa é de 1.500 amostras por dia por pessoa.
- Crescimento bacteriano para a mini-preparação da elevado-produção da em-casa
- Prepare a placa Omni LB/agar. Despeje 30 – 40 mL de agar LB (1,5% de agar bacteriológico em LB mídia suplementada com antibiótico para seleção de clones positivos) em cada placa de poço único.
- Manchar o estoque de glicerol na placa LB/agar. Diluir o estoque de glicerol no meio LB (1:300, geralmente 2 μL em 600 μL de LB). Agitar por 10 min. ponto 3 μL do estoque diluído na placa LB/agar. Incubar a 37 ° c, de cabeça para baixo, durante a noite.
- Inocular culturas. Usando o dispositivo 96-PIN que foi esterilizado em 80% etanol e chama, inocular a cultura da placa de agar em um bloco de poço profundo com 1,5 mL por poço de meio de TB suplementado com antibiótico.
- Incubar culturas. Cubra o bloco com um selo permeável a gás e incubar por 22 – 24 h a 37 ° c, 300 – 800 rpm, dependendo do Shaker.
Nota: os abanadores ajustados em 800 rpm são ideais para esta incubação. O uso de um agitador de velocidade mais lento pode resultar em culturas menos densas e menores rendimentos de purificação de DNA. - Culturas de pellets. Blocos da rotação em 3.800 x g e 4 ° c para o sobrenadante do descarte de 30 min.
- Mini-Prep em casa de alta taxa de transferência
Nota: pipetadores multicanal ou dispensadores automáticos podem ser usados para realizar a mini-preparação de alta taxa de transferência em casa. Se estiver usando um dispensador automático, certifique-se de limpar o sistema antes de usar e entre as soluções.- Prepare todas as soluções utilizadas durante a minipreparação:
- Preparar solução 1: tampão de ressuscição TE (50 mM Tris, pH = 8,0; 10 mM EDTA, pH = 8,0; 0,1 mg/mL RNAse). Conservar a 4 ° c.
- Preparar solução 2: tampão de Lise NaOH/SDS (0,2 M NaOH; 1% SDS). Para melhores resultados, uma solução recém-feita deve ser usada.
- Prepare a solução 3: tampão de neutralização KOAC (2,8 M KOAc). Ajuste o pH da solução para 5,1 com ácido acético glacial. Conservar a 4 ° c.
- Preparar solução N2: tampão de equilíbrio (100 mM TRIS; 900 mM KCl; 15% EtOH; 0,15% Triton X-100). Ajuste o pH da solução para 6,3 com ácido fosfórico.
- Prepare a solução N3: buffer bash (100 mM TRIS; 1,15 M KCl; 15% EtOH). Ajuste o pH da solução para 6,3 com ácido fosfórico.
- Prepare a solução N5: tampão de eluição (100 mM TRIS; 1 M KCl; 15% EtOH). Ajuste o pH da solução para 8,5 com ácido fosfórico.
Nota: o controle bem sucedido da ligação, da lavagem, e da eluição do ADN durante a troca do aníon é altamente-dependente da concentração do tampão KCl e dos valores de pH. As medidas cuidadosas do componente tampão e o ajuste do pH são essenciais. Pequenos desvios das medições descritas podem resultar em perda significativa de rendimentos.
- Re-suspender pellet. Adicionar 200 μL de solução 1 e agitar a 2.000 rpm durante 5 min em RT. a re-suspensão completa da pelota é necessária para o lysis bem sucedido. Vortex o bloco, se necessário.
- Bactérias lyse. Adicione 200 μL de solução 2, sele a placa com um selo de alumínio e inverta 5x. Cuidadosamente tempo este passo desde o início da solução 2 adição. Não exceda 5 min.
- Neutralize a solução. Adicione 200 μL de solução 3, sele a placa com um selo de alumínio e inverta 5x. O selo pode ser solto devido aos buffers de Lise/neutralização, portanto, tenha cuidado ao inverter. Uma inversão parcial, na qual a solução nunca toca o selo, é recomendada para evitar a contaminação cruzada entre as amostras.
- Lisado claro. Centrifugar as placas a 3.800 x g e 4 ° c durante 30 min.
- Prepare o chorume da resina da troca do aníon durante a etapa da centrifugação do lisate granulação. Usando um frasco de 1 L, encha-o com a resina da troca do aníon até que alcangue a marca de 300 mL, a seguir adicione a solução N2 até 900 mL.
Cuidado: esta etapa deve ser feita no capô para proteger contra a inalação de sílica. - Prepare placas de resina de troca de ânions. Empilhe as placas de filtro em cima de um bloco de poço profundo para atuar como um recipiente de coleta de resíduos. Misture a pasta de troca de ânions até que seja homogênea, em seguida, despeje em uma calha de vidro. Usando pontas largo-furadas de P1000, transfira 450 μL da pasta em cada poço das placas de filtro.
- Centrifugue placas empilhadas (placa da resina/placa profunda do poço) na aceleração lenta por 5 minutos em 130 x g e em RT. descarte o fluxo completamente.
- Transfira o sobrenadante do lisado à placa da resina/pilhas profundas do bloco do poço. Gire as placas empilhadas por 5 min a 30 x g com velocidade de rampa lenta.
- Coluna de lavagem. Adicionar 400 μL de solução N3 (tampão de lavagem) a cada poço. Transfira a placa de resina para o colector de vácuo para remover o tampão de lavagem. Repita as etapas de lavagem 3x. Na última lavagem, certifique-se de que todos os poços estão devidamente esvaziados. Gire as placas de pilha em 150 x g por 5 min para remover qualquer tampão residual.
- DNA de elute. Coloque a placa de resina sobre uma placa de recolha de 800 μL limpa. Adicionar 300 μL de solução N5 a cada poço. Deixe-o sentar-se no RT por 10 minutos, a seguir gire as placas empilhadas por 5 minutos em 20 x g com velocidade lenta da rampa-acima. Gire as chapas empilhadas por 1 min a 233 x g.
- Quantifique o DNA e armazene as placas a-20 ° c até o uso posterior ou prossiga direto para a precipitação do DNA.
Nota: é necessário um mínimo de 30 μg de ADN por amostra. Se o rendimento do DNA for baixo, recomenda-se repetir a mini-preparação do DNA ou, alternativamente, combinar duas placas durante o passo de precipitação (secção 2,3).
- Prepare todas as soluções utilizadas durante a minipreparação:
- Precipitação do ADN
- Descongelar placas, Vortex para homogeneizar a solução de DNA, e girar em 230 x g para 30 s para coletar toda a solução no fundo do poço.
- Adicionar 40 μL de NaOAc de 3 M e 240 μL de isopropanol a cada poço. Cubra a placa com um selo de alumínio e misture invertendo 3x.
- Centrifugue as placas por 30 min a 3.800 x g e 25 ° c. Elimine cuidadosamente o sobrenadante.
Nota: para combinar duas placas, transfira o DNA da segunda placa para o pellet da primeira placa e repita os passos 2.3.2 – 2.3.3. - Lave e precipeie o DNA. Adicionar 400 μL de 80% de etanol a cada poço. Placas de vedação com vedação de alumínio e agitar a 1.000 rpm por 30 min. centrifugador a 3.800 x g durante 30 min a 25 ° c. Descarte o sobrenadante.
- Seque as pelotas de DNA. Coloque as placas de cabeça para baixo em um ângulo em toalhas de papel e deixe-os secar por 1 – 2 h, até que nenhum álcool esteja presente na parte inferior do poço. Sele e Centrifugue em 230 x g por 2 minutos para trazer todas as pelotas para baixo.
- Uma vez que as placas estão secas, ou vedação com vedação de alumínio e congelar a-20 ° c para uso posterior ou continuar a ressuscitaro o DNA (passo 4,1).
3. revestimento da corrediça de aminosilane
- Coloc as corrediças de vidro na cremalheira do metal. Inspecione visualmente cada corrediça para assegurar nenhuns riscos ou as imperfeições estão atuais.
- Submerge slides em solução de revestimento (2% de reagente de de em acetona) durante 15 min durante o balanço. A solução do de pode ser usada para revestir duas cremalheiras de 30 corrediças cada um antes que precise de ser Descartado.
- Enxaguar a etapa. Mergulhe o rack de slides na lavagem de acetona (99% acetona), agitar para frente e para trás, em seguida, para cima e para baixo rapidamente 5x. Incline para um canto para pingar, em seguida, submergir em água ultrapura para cima e para baixo rapidamente 5x. Incline para pingar fora, em seguida, coloque em guardanapos.
Nota: a lavagem de acetona pode ser usada duas vezes, enquanto a água ultrapura deve ser mudada de cada vez. - Seque as corrediças usando o ar pressionado, soprando neles de todos os ângulos por aproximadamente 3 minutos até que todas as gotas de água estejam removidas. Armazene os slides revestidos em RT em uma cremalheira de metal dentro de uma caixa firmemente selada.
4. preparação da amostra da matriz
- Ressuspender o pellet de DNA da mini-Prep em casa (passo 2.3.6) em 20 μL de água ultrapura e agitar a 1.000 rpm por 2 h. Para o DNA de preparação MIDI/Max, diluir cada amostra para uma concentração final de 1,5 μg/μL e transferir 20 μL para uma placa de coleta de 800 μL.
- Prepare a mistura de impressão. Para 1 96 placa de poço, prepare 1 mL de mistura de impressão [237,5 μL de água ultrapura; 500 μL de poli-lisina (0, 1%); 187,5 μL de BS3 (BIS-sulfosuccinimidyl, 50 mg/mL em DMSO); e 75 μL de anticorpo policlonico de coelho antibandeira)].
Nota: os produtos químicos devem ser adicionados na ordem especificada para evitar a precipitação. - Adicionar 10 μL de mistura de impressão para cada amostra, placas de vedação com folha de alumínio, e agitar em RT para 90 min em 1.000 rpm. Guarde as placas durante a noite (~ 16 h) a 4 ° c.
- No dia da impressão, brevemente vórtice e girar as placas. Transfira 28 μL de cada amostra para uma placa de 384 matriz. Esta transferência pode ser feita usando a automatização ou uma pipeta multicanal. É crucial manter o controle da posição das amostras na placa da matriz 384.
- Gire a placa para baixo brevemente para remover quaisquer bolhas. Selar as placas com folha.
5. geração de matrizes de NAPPA: impressão do microarray
Nota: todas as condições de impressão foram otimizadas para o instrumento listado na tabela de equipamentos e materiais. Se estiver usando uma matriz diferente, pode ser necessária uma otimização adicional.
- Arrayer limpar. Antes de começar, esvazie todos os tanques de resíduos e reabasteça os reservatórios com água ultrapura ou 80% de etanol, se necessário. Limpe os pinos um-por-um com toalhetes sem fiapos e água ultrapura. Seque os pinos com toalhetes sem fiapos e coloque-os cuidadosamente de volta na cabeça do arrayer.
- Arrayer configurado: especificações de impressão [número máximo de selos por tinta: 1; número de selos por ponto: 1; cronometragem de carimbo:--; tempo de carimbo (MS): 0 ms; tempo de escrita à tinta (MS): 0 ms; ajuste de profundidade de impressão: 90 mícrons; número de retoques: 0]. em seguida, siga as Protocolo de esterilização: lavagem de água ultrapura para 2.000 MS com 0 ms de tempo de secagem e 500 MS de tempo de espera; Repita estes passos 6x; seguido de lavagem com 80% de etanol para 2.000 MS com 1.200 MS de tempo de secagem e 500 MS de tempo de espera; Repita estes passos 6x.
- Projeto da corrediça: ajuste o arrayer com o teste padrão de arraying desejado. O projeto deve levar em consideração vários fatores [ou seja, o número de réplicas para cada amostra, localização e número de recursos de controle, layout de matriz (um bloco, vários blocos idênticos), número de matrizes a serem impressos, comprimento da execução, etc.].
- Coloc os slides revestidos de (etapa 3,4) no deck do arrayer. Verifique se o aspirador está segurando todos os slides firmemente no lugar. Inicie o humidificador (deve ser fixado em 60%).
- Coloc a placa do poço 384 na plataforma do arrayer. Inicie o programa.
- Rotule os microarrays. Quando a impressão é feita, coloque rótulos de slides no lado inferior (não impresso) de cada slide. Mantenha a ordem de impressão dos slides no deck em ordem numérica.
- Armazene as matrizes impressas em RT em uma cremalheira de metal dentro de uma caixa firmemente selada com um pacote de sílica. As corrediças mantidas em um ambiente seco têm uma vida útil até um ano.
- (Opcional): um segundo lote de 90 slides pode ser impresso usando as mesmas amostras. Para fazer isso, retire a placa 384 bem do deck arrayer assim que a impressão da placa é feita. Sele e guarde a placa a 4 ° c. Após o primeiro lote de matrizes são completamente feitos, removê-los do convés, coloque novos slides revestidos de e iniciar uma nova corrida. Certifique-se de que cada 384 placa de poço está em RT por 30 min antes de seu uso. Se mais de quatro réplicas por amostra forem impressas em um lote de slides, é recomendável dividir as placas de 384 poços em duas placas para diminuir a evaporação da amostra diminuindo a quantidade de tempo gasto no deck do arrayer.
Nota: Verifique todos os reservatórios antes do início da segunda execução.
6. detecção de DNA em slides NAPPA
- Bloqueie os slides. Coloque os slides em uma caixa de pipeta e adicione 30 mL de buffer de bloqueio. Incubar em RT por 1 h em um agitador de balanço.
- Manchar os slides. Elimine a solução de bloqueio e adicione 20 mL de tampão de bloqueio e 33 μL de corante fluorescente de ADN intercalante. Incubar por 15 min com agitação. Em seguida, enxague rapidamente as lâminas com água ultrapura e seque com ar pressuriado. Prossiga com a digitalização (secção 11).
7. expressão das lâminas NAPPA
- Bloqueie os slides com buffer de bloqueio em um agitador de balanço na RT por 1 h. Use aproximadamente 30 mL em uma caixa de pipeta para quatro lâminas.
- Enxague as lâminas com água ultrapura e seque com ar comprimido filtrado. Aplique a junta de vedação a cada slide de acordo com as instruções do fabricante.
- Adicionar IVTT Mix. Cada slide exigirá 150 μL de mistura de IVTT. Diluir 82,5 μL de lisado de HeLa em 33 μL de água DEPC e complementar com 16,5 μL de proteínas acessórias e 33 uL de mistura de reacção. Adicione a mistura de IVTT da extremidade non-Label ou da não-amostra. Pipetar a mistura lentamente (é aceitável se ele se Perla temporariamente na extremidade de entrada). Massageie suavemente a junta de vedação para que a mistura IVTT se espalhe e cubra toda a área da matriz. Aplique os selos pequenos da porta redonda a ambas as portas.
- Coloque os slides em um suporte e transferi-los para a incubadora de refrigeração programável. Incubar por 90 min a 30 ° c para expressão protéica, seguido de 30 min a 15 ° c para imobilização da proteína de consulta.
- Lave e bloqueie os slides. Retire a junta de vedação e mergulhe as lâminas numa caixa de pipeta com cerca de 30 mL de 1x TBST suplementado com 5% de leite para exposição proteica (secção 8) ou 1x TBST suplementado com 3% de albumina sérica bovina (BSA) para ensaios de quinase ou rastreio de medicamentos (secção 9). Incubar em RT com agitação por 20 min e repita este passo 2x.
8. detecção de proteínas em matrizes NAPPA
- Adicione o anticorpo preliminar. Remova os slides da solução de bloqueio (etapa 7,5) e seque suavemente o verso (lado não impresso) usando uma toalha de papel. Coloque os slides em um suporte e aplique 600 μL de anticorpo primário (mouse anti-Flag) diluído 1:200 em 1x TBST + 5% de leite. Incubar por 1 h em RT.
- Lave as lâminas com 1x TBST + 5% de leite em um agitador de balanço (3x por 5 min cada).
- Adicione o anticorpo secundário. Retire os slides da solução de lavagem e seque suavemente o verso usando a toalha de papel. Coloque os slides em um suporte e aplique 600 μL de anticorpo secundário (anticorpo Cy3-labbeled anti-mouse) diluído 1:200 em 1x TBST + 5% leite. Proteja os slides da luz e incubar por 1 h em RT.
- Lave as lâminas com 1x TBST em um agitador de balanço (3x por 5 min cada). Enxague rapidamente as lâminas com água ultrapura e seque utilizando ar pressuriado. Prossiga com a digitalização (secção 11).
9. triagem do inibidor da tirosina quinase em matrizes NAPPA
Observação: vários slides podem ser processados no mesmo experimento, no entanto, certifique-se de que em cada etapa, um slide é processado por vez e que eles não secam entre as etapas. Adicione todas as soluções à extremidade não-rótulo ou não-espécime do slide.
- Prepare todas as soluções utilizadas durante a triagem de drogas:
- Prepare a solução de fosfatase/DNase combinando o seguinte: tampão das metaloffosfatases da proteína 1x (50 milímetros HEPES, 100 milímetros NaCl, 2 milímetros DTT, 0, 1% Brij 35 em pH = 7,5); 1 mM MnCl2; 8.000 unidades de proteína fosfatase lambda; e 2 unidades de DNase I. Prepare 400 μL da solução para cada microarray. Adicionar fosfatase e DNase pouco antes de usar.
- Faça a diluição da droga. As drogas são reconstituídas em DMSO para uma concentração final de 10 mM. Para garantir que todas as concentrações de fármacos testadas na matriz, assegure-se de que o mesmo volume de DMSO (um estoque de 10.000 x no DMSO) seja criado para cada concentração e mantido em-80 ° c. No momento do uso, as drogas são diluídas 1:100 na água.
- Prepare a solução da droga/quinase combinando o seguinte: tampão da quinase 1x (Tris-HCl de 25 milímetros do pH = 7,5); Beta-glicerofosfato de 5 mM; 2 mM DTT; 0,1 mM na3vo4; 10 mM MgCl2; 500 μM ATP; e 2 μL de fármaco (diluído 1:100 em água). Prepare 200 μL da solução para cada microarray.
- Realize o tratamento com fosfatase e DNase. Remova os slides da solução de bloqueio (etapa 7,5) e seque suavemente a parte traseira usando a toalha de papel. Coloque as lâminas em suporte e aplique 200 μL de solução de fosfatase/DNase. Coloque uma lamínula de Microarray para evitar a evaporação. Incubar a 30 ° c por 45 min no forno.
- Fosfatase e tratamento com DNase II: Retire as matrizes do forno, descarte a lamínula, retire o excesso de solução e aplique 200 μL de fosfatase recém-feita e solução de DNase. Cubra Microarrays com lamínula e incubar por mais 45 min a 30 ° c no forno.
- Lave os slides com 1x TBST + 0,2 M NaCl em um agitador de balanço (3x por 5 min cada).
- Realize o tratamento medicamentoso e a reação da quinase. Retire os slides da solução de lavagem e seque suavemente a parte traseira usando uma toalha de papel. Coloque as lâminas em suporte e aplique 200 μL de solução de fármaco/quinase. Coloque uma lamínula na parte superior para evitar a evaporação. Incubar por 1 h a 30 ° c no forno.
- Lave os slides com 1x TBST + 0,2 M NaCl em um agitador de balanço (3x por 5 min cada).
- Repita as etapas 8.1 – 8.4 usando como anticorpo primário anti-phosho-Tyr anticorpo diluído 1:100. Substitua 1x TBST + 5% leite em todas as etapas com 1x TBST + 3% BSA.
10. protocolo de hibridação automatizado
Nota: Alternativamente, uma estação de hibridação pode ser usada para automatizar todas as hibridizações e lavagens nas matrizes NAPPA (seções 7 – 9) e o protocolo é fornecido como arquivo complementar 1.
11. aquisição de imagens
Nota: as imagens de Microarray devem ser adquiridas com uma resolução de 20 mícrons ou mais.
- Carregue Microarrays no compartimento do porta-lâmina com as proteínas voltadas para cima. Carregue a revista no scanner de Microarray.
- Selecione o laser verde com um filtro de emissão de 575/30 nm para digitalizar o sinal do anticorpo secundário rotulado com CY-3. Se for usado um fluoróforo diferente, selecione o laser/comprimento de onda corretos para detectar o sinal do corante fluorescente.
- Defina o nome para cada imagem e o local que eles serão salvos.
- (Opcional): para cada novo fluoróforo, recomenda-se a otimização das condições de digitalização para detectar a amplitude linear da intensidade do sinal. Para fazer isso, digitalizar um microarray usando uma gama de fotomultiplicador (PMT) e ganho até que uma imagem clara é obtida com sinal não saturado e fundo baixo.
- Analise todos os microarrays com as configurações otimizadas e lembre-se de desativar o autogain.
Observação: para análise de dados, todos os microarrays devem ser verificados usando as mesmas configurações de digitalização. Para os ensaios de quinase usando Cy3 como um fluoróforo, as imagens são digitalizadas com 20% de PMT, intensidade de laser de 25% e 10 mícrons de resolução, usando o scanner listado na tabela de equipamentos e materiais.
12. processamento e análise de dados
Nota: vários pacotes de software estão disponíveis para a quantificação de dados de Microarray com capacidades semelhantes. O procedimento descrito aqui foi projetado para o software listado na tabela de equipamentos e materiais.
- Carregue os arquivos TIFF a serem quantificados, projete a grade para corresponder ao layout do Microarray e ajuste o tamanho dos pontos para incorporar todo o sinal com a área mínima possível. Os pontos vizinhos não devem se sobrepor. Inspecione visualmente o quão bem o software está realizando e ajuste a grade manualmente, se necessário.
- Quantificar a intensidade do sinal do Microarray. Inspecione visualmente os pontos para qualquer anormalidade (ligação não específica, poeira, etc.) e remova-os da análise de dados.
- Corrija o plano de fundo localmente usando o sinal de áreas vizinhas na matriz em que nenhum ponto está presente.
- Normalize os dados. Para comparar o sinal entre matrizes diferentes, a intensidade do sinal de cada microarray deve ser normalizada. Para excluir quaisquer outliers, normalizar os dados usando a média aparada 30% do sinal do controle positivo (pontos de IgG) dos Microarrays dephosphorylated.
Nota: o sinal do ponto de IgG não muda durante a fosforilação e desfosforilação dos Microarrays e é apropriado para a normalização. - Identifique as cinases ativas. Para cada recurso exibido no microarray, calcule a razão entre a intensidade do sinal normalizado nas matrizes autofosforiladas e dephosphorylated. Definir um limiar de 1,5-dobre a mudança para a identificação das cinases ativas e marcar todas as outras características como incapaz de submeter-se a autofosforilação (N/A).
- Calcule a atividade de cada quinase identificada na etapa 12,5 como uma porcentagem do sinal ajustado (intensidade do sinal da matriz de controle positivo normalizado (DMSO) subtraída pela intensidade do sinal da matriz de controle negativo normalizado (dephosphorylated).
Resultados
Os microarrays de Nappa automontados fornecem uma plataforma contínua que possa ser usada para muitas aplicações distintas, incluindo a descoberta do biomarcador, as interações proteína-proteína, a identificação do substrato, e a seleção da droga10,11 ,12,13,14,15,16,17,18,19,20.
A metodologia geral adotada para o estudo da atividade da quinase e o rastreamento de inibidores da tirosina cinases em Microarrays NAPPA são representados esquematicamente na Figura 1. Primeiramente, os microarrays de NAPPA são gerados pela imobilização do cDNA e pelo agente da captação nos Microarrays revestidos. Os cDNAs são então utilizados como um modelo para a transcrição e tradução de proteínas, utilizando um sistema de IVTT humano-baseado, e as proteínas recém-sintetizadas são imobilizados pelo agente de captura9. A qualidade do microarray impresso pode ser monitorada medindo os níveis de ADN (confirmando a impressão consistente) ou a proteína indicada na disposição (confirmando a expressão e a captação da proteína; Figura 1). Para diminuir o sinal de fundo e aumentar o intervalo dinâmico do experimento, os microarrays são tratados com 1) fosfatase lambda para remover a fosforilação de resíduos de ser/thr/Tyr, depois com 2) DNase para simplificar a química no local e diminuir fundo (Figura 1).
O próximo passo é a reação de autofosforilação, na qual os microarrays são incubados com tampão quinase na ausência de ATP (matriz de controle negativo, referida como Microarrays dephosphorylated), e o tampão da quinase é suplementado com ATP (controle positivo, denominados matrizes autofosforiladas) ou ATP + DMSO (controlo do veículo). Deve-se enfatizar que, durante esta etapa, não é adicionada quinase; Conseqüentemente, a atividade intrínseca de cada quinase indicada no microarray é quantificada com a medida de seus níveis do fosforilação usando um anticorpo da anti-phospho-tyrosine da bandeja seguido por um anticorpo secundário Cy3-labbeled (Figura 1).
O controle de qualidade das matrizes da NAPPA-quinase exibindo um painel de proteínas humanas quinases impressas em quadruplicado é mostrado na Figura 2. Os níveis de DNA imobilizado foram medidos por coloração de DNA e mostrou um sinal uniforme em todo o Microarranjo, sugerindo que a quantidade de DNA impressa na matriz era uniformizada. Também é possível observar várias características sem qualquer coloração de DNA. Essas características correspondem a alguns controles em que o DNA foi omitido da mistura de impressão [ou seja, manchas vazias (nada foi impresso), manchas de água, ponto de IgG purificada (poli-lisina, chapéu cabeado e IgG purificada), apenas mistura de impressão (mistura de impressão completa: poli-lisina mais chapéu cabeado e anticorpo antisinalizador, sem qualquer DNA)]. Os níveis de proteína indicados nos Microarrays da NAPPA-quinase foram avaliados após a reação de IVTT usando o anticorpo antitag.
Para a triagem da quinase, a bandeira foi usada como a tag de escolha e o nível de proteína exibido no microarray foi medido usando um anticorpo antisinalizador. Como mostrado, a maioria dos pontos que contêm cDNA exibiu com sucesso níveis detectáveis de proteína. Alguns dos pontos do controle sem cDNA igualmente revelaram o sinal com o anticorpo da anti-bandeira: ponto de IgG (usado para detectar a atividade do anticorpo secundário) e pontos vazios do vetor (códigos do cDNA para o Tag somente) (Figura 2). Os Microarrays da NAPPA-quinase mostraram boa reprodutibilidade entre as lâminas, com a correlação dos níveis de exposição da proteína entre lotes de impressão distintos superiores a 0,88 (Figura 2). Dentro do mesmo lote a correlação foi ainda maior, perto de 0,92 (dados não mostrados).
Em seguida, a atividade de autofosforilação da quinase das proteínas exibidas na matriz foi mensurada com anticorpo antifosfotirosina (Figura 3). A proteína indicada na matriz mostrou altos níveis de fosforilação protéica após a expressão (Figura 3, esquerda), que pode ser causada pela atividade da quinase intrínseca da proteína indicada na matriz ou por cinases ativas presentes na mistura de ivtt. Este fosforilação foi removido completamente com tratamento da fosfatase do lambda e estes Microarrays foram usados para os ensaios da quinase. Após dephosphorylation, as reações do autophosphorylation executadas sem ATP não mostraram nenhuns níveis significativos de fosforilação, como esperado, quando os microarrays incubadas com tampão da quinase na presença de ATP mostraram o fosforilação da proteína tão rapidamente quanto 15 minutos ( Figura 3). Para a triagem medicamentosa, a atividade da quinase foi mensurada após 60 min de reação de autofosforilação para maximizar o número de quinases testadas.
A comparação entre os microarrays nos quais os níveis de fosforilação foram medidos logo após a expressão protéica (Figura 3, esquerda) e após 60 min de reação de autofosforilação (Figura 3, direita) mostraram: i) proteínas fosforiladas apenas após a expressão, sugerindo que eles podem ser exogenamente fosforilado por proteínas presentes na mistura de IVTT, mas não podem ser autofosforilados; II) proteína fosforilada somente após a reação de autofosforilação, sugerindo que essas proteínas não eram ativas após a expressão protéica e os cofatores necessários presentes no tampão da quinase estivessem ativos; ou III) proteínas fosforiladas em ambas as matrizes, sugerindo que estivessem ativas em ambos os ajustes (Figura 3).
Como exemplo dos resultados obtidos para a triagem de inibidores da tirosina quinase nas matrizes da NAPPA-quinase foram utilizados três inibidores de cinases com seletividade distinta em proteínas quinases: staurosporina, imatinib e Ibrutinib. Para todas as seleções, os microarrays dephosphorylated de NAPPA foram incubados com concentrações crescentes de TKI (que variam de 100 nanômetro a 10 uM) durante a reação do autophosphorylation. A primeira TKI testada foi a staurosporina, um inibidor global da proteína quinase, que demonstrou inibição potente da quinase no microarray em praticamente todas as cinases testadas11.
Em seguida, foi testado o imatinib, um inibidor de ABL e c-kit utilizado para o tratamento de leucemia mielóide crônica e tumores estromais gastrointestinais4,5,6,7. Nas matrizes de NAPPA-quinase o imatinib mostrou uma redução significativa na atividade de Abl1 e de BCR-Abl1 visto que outras quinases permaneceram na maior parte não afetadas (figura 4a). A quantificação dos dados para a atividade da quinase foi normalizada contra a matriz dephosphorylated e representada como uma porcentagem do microarray positivo do controle (veículo somente). Os dados para TNK2 (quinase não relevante), Abl1 e BCR-Abl1 são mostrados na Figura 4B. Como esperado, o imatinib demonstrou inibição seletiva para Abl1 e BCR-ABl1. Os dados para o c-kit foram inconclusivos devido à falta de atividade nos arrays de controle positivo.
Finalmente, o Ibrutinib, um inibidor covalente aprovado pela FDA da tirosina quinase de Bruton (BTK), foi testado. O Ibrutinib é actualmente utilizado no tratamento de vários tipos de cancro relacionados com o sangue com BTK hiperactivo, incluindo leucemia linfocítica crónica (LLC), linfoma de células do manto e macroglobulinemia de Waldenström21,22. A Figura 4Cé representativa dos resultados típicos obtidos para a triagem do Ibrutinib. A atividade da quinase de ABL1 (quinase não relevante) e BTK (alvo canônico) e ERBB4 (potencial novo alvo) é mostrada na Figura 4D. Os dados sugerem que o ERBB4 pode ser inibido pelo Ibrutinib em uma forma específica da dose. Esta inibição foi confirmada in vitro e em ensaios baseados em células11, demonstrando o poder desta plataforma.
Tomados em conjunto, os dados sugerem que a plataforma de Microarray NAPPA-quinase poderia ser usada para a triagem imparcial de inibidores de TK. Além disso, a triagem é rápida e pode ser facilmente personalizada para incluir qualquer variação das proteínas quinases de interesse.
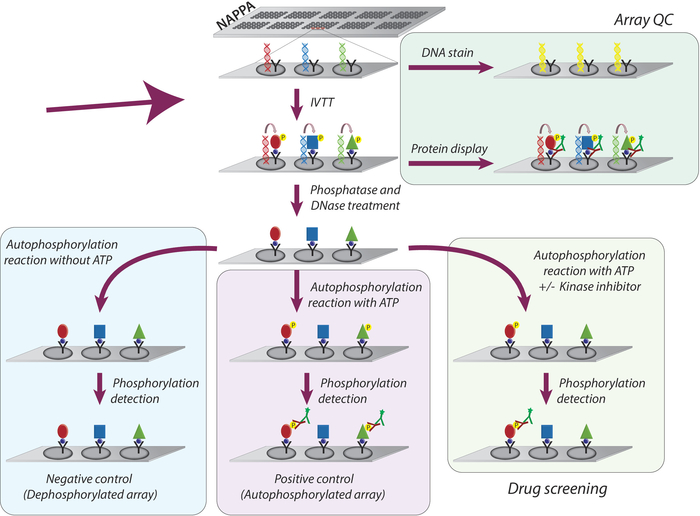
Figura 1: representação esquemática do controle de qualidade e triagem de inibidores da tirosina quinase em matrizes NAPPA. As matrizes de NAPPA são imprimidas com o cDNA que codifica para a proteína do interesse fundido com uma etiqueta e um anticorpo da captação. Durante a transcrição in vitro e a reação da tradução (IVTT) as proteínas sintetizadas são capturadas na superfície do microarray através da etiqueta pelo anticorpo da captação. O controle da qualidade (QC) das matrizes é executado pela medida dos níveis de ADN imprimidos na corrediça, usando um corante DNA-intercalating fluorescente, e os níveis de proteína indicados no array usando anticorpos tag-specific. Para a triagem da quinase, os microarrays são tratados com DNase e fosfatase, após a reação do IVTT, para remover o DNA impresso e toda a fosforilação que pode ter ocorrido durante a síntese proteica. As matrizes dephosphorylated estão agora prontas para ser usado para a tela da droga. Para cada ensaio, três conjuntos de controles são usados rotineiramente: (I) matrizes dephosphorylated, em que a reação do autophosphorylation é executada sem ATP; (II) Microarrays autofosforilados, nos quais a reação de autofosforilação é realizada na presença de ATP; e (III) matriz tratada com DMSO (veículo), na qual a reação de autofosforilação é realizada com ATP e DMSO. As lâminas tratadas com diferentes concentrações de inibidores da quinase seguem exatamente o mesmo protocolo utilizado para as matrizes tratadas com DMSO. Por favor clique aqui para ver uma versão maior desta figura.
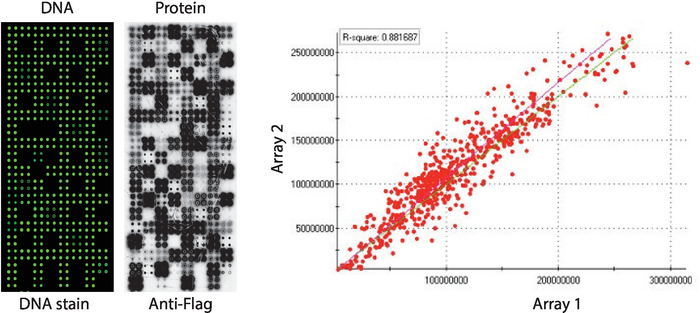
Figura 2: resultados representativos do controle de qualidade para matrizes de NAPPA-quinase automontadas. O índice do ADN medido por um corante DNA-intercalating fluorescente (esquerdo) e os níveis de proteína indicados no microarray medido pelo anticorpo da anti-bandeira (meio) são mostrados. No lado direito é um gráfico de correlação dos níveis de proteína exibido em dois arrays NAPPA-quinase impressos em lotes separados. Por favor clique aqui para ver uma versão maior desta figura.
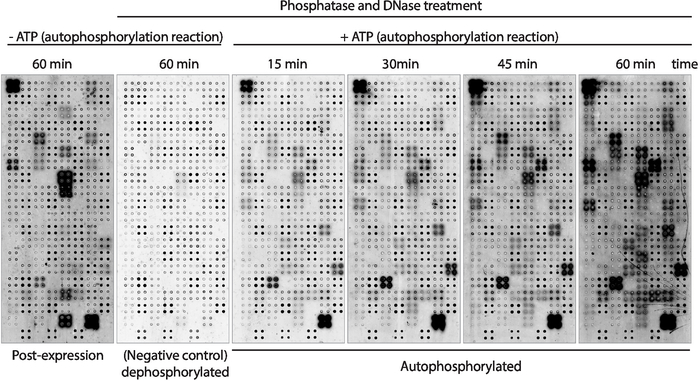
Figura 3: resultados representativos da atividade da quinase em matrizes da NAPPA-quinase. Microarrays exibindo proteínas quinases em quadruplicado foram usados para estudar a atividade da proteína kinase na matriz através da medição de fosforilação protéica usando anticorpo anti-pTyr, seguido pelo anticorpo anticamundongo Cy3-rotulado. As matrizes de controle sem o tratamento da fosfatase/DNase e sem ATP durante a reação do autophosphorylation foram usadas para medir o fosforilação do fundo após a expressão da proteína (borne-expressão). Os microarrays restantes foram tratados com a fosfatase/ADN, e a reação do autophosphorylation foi executada sem ATP (microarray dephosphorylated, controle negativo) ou com ATP (microarrays autophosphorylated). Para os microarrays autofosforilados, a reação de autofosforilação foi realizada por 15 min, 30 min, 45 min ou 60 min, como mostrado. Por favor clique aqui para ver uma versão maior desta figura.
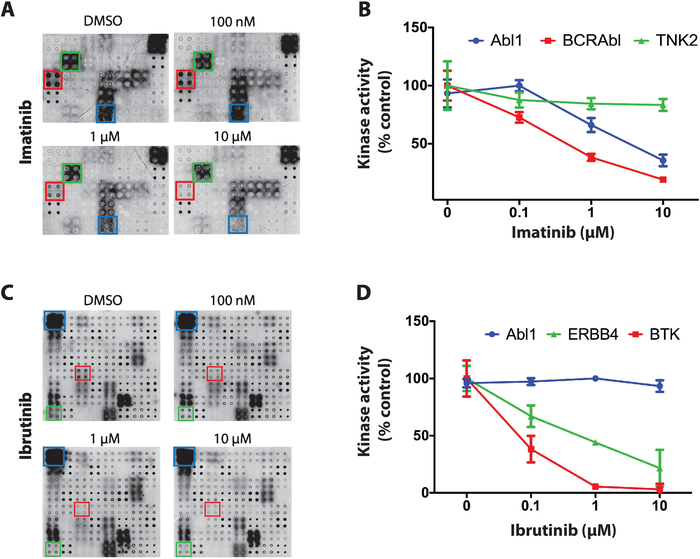
Figura 4: dados representativos da tela da tirosina quinase em matrizes NAPPA-quinase. (A) as matrizes de Nappa-quinase tratadas com fosfatase/DNase foram incubadas em concentrações crescentes de imatinib durante a reação de autofosforilação e a atividade da quinase foi medida com anticorpo antifosfotyr. (B) quantificação da atividade quinase observada nas matrizes da Nappa-quinase expostas ao imatinib. Os dados foram normalizados contra o sinal das matrizes de controle negativo (dephosphorylated) e é mostrado como uma porcentagem das matrizes positivas do controle (reação do autophosporylation executada na presença de DMSO). Dados semelhantes são mostrados para a triagem de Ibrutinib (C, D). Este número foi modificado de Rauf et al.11. Por favor clique aqui para ver uma versão maior desta figura.
Arquivo complementar 1. Protocolo alternativo para a seleção de inibidores da tirosina quinase em matrizes de NAPPA usando uma estação automatizada da hibridação. Por favor, clique aqui para baixar este arquivo.
Discussão
Modificações e solução de problemas
Durante a fase de otimização do estudo da atividade quinase em matrizes NAPPA, uma das principais fontes de fundo e baixa faixa dinâmica observada foi a BSA utilizada na mistura de impressão. BSA estava fornecendo as aminas primárias necessárias para a reticulação com a superfície do de e estava prendendo o DNA e o anticorpo de captura no local. No entanto, a BSA é altamente fosforilada, dificultando a detecção do sinal de autofosforilação na matriz acima do ruído de fundo. Para resolver este problema, foram testadas várias alternativas para a BSA na mistura de impressão, e a poli-lisina foi identificada como boa substituta. A poli-lisina carece de qualquer local de fosforilação; Portanto, o plano de fundo de matrizes não expressas é muito mínimo. Além disso, os microarrays impressos com poli-lisina são reprodutíveis e exibem bons níveis de proteínas (Figura 2).
A modificação crítica seguinte executada no ensaio padrão de NAPPA era a adição de uma etapa do tratamento da fosfatase/DNase. O tratamento dos Microarrays com fosfatase permite a remoção de qualquer fosforilação ocorrida na mistura de IVTT durante a síntese protéica e captação (Figura 3). A fonte desta fosforilação pode ser proveniente da atividade de autofosforilação intrínseca ou da atividade das cinases presentes na mistura de IVTT. A remoção de toda a pós-expressão de fosforilação permitiu a fácil identificação das cinases ativas e podem sofrer autofosforilação (Figura 3).
Etapas críticas dentro do protocolo
NAPPA é uma tecnologia robusta, mas como esperado, existem várias etapas críticas. A primeira é a aquisição de DNA de alta qualidade na concentração apropriada. O uso de DNA de má qualidade ou em baixas concentrações gerará Microarrays de baixa qualidade com diversas características que não estão sendo expressas e exibidas nos níveis adequados, diminuindo o número de proteínas analisadas na matriz. A segunda etapa crítica é a expressão de proteínas no microarray. O uso de um sistema de IVTT que expresse níveis elevados de proteína funcional é vital para estudar a atividade da quinase na disposição.
O próximo passo crítico na triagem TKI é como os microarrays são manipulados. Os microarrays não devem secar durante nenhuma etapa do protocolo, e a manipulação delicada é recomendada impedir os riscos que podem aumentar o sinal do fundo. Como as matrizes de todo o experimento serão comparadas entre si, é importante garantir que cada etapa de incubação seja mesmo em todos os slides. Por exemplo, o tempo necessário para executar uma etapa em uma única matriz deve ser levado em consideração quando um lote de 20 matrizes é processado para evitar diferenças no comprimento de incubação entre matrizes.
Por fim, o delineamento do experimento e a inclusão de controles positivos e negativos são críticos para o controle de qualidade e a análise dos dados. O primeiro conjunto de controles são aqueles impressos em cada matriz e inclui controles negativos [ou seja, manchas vazias (sem qualquer material impresso), água ou vetor vazio (expressar apenas a marca)], bem como um controle positivo (i.e., IgG purificada, que é detectado pelo anticorpo secundário e é inerte para alteração nos níveis de fosforilação). Coletivamente, eles medem os níveis de fundo do microarray, possível transição durante a impressão e a intensidade do sinal do método de detecção.
O próximo conjunto de controles são os controles de triagem de drogas e incluem os microarrays dephosphorylated e autophosphorylated (na presença ou ausência de DMSO). Como mencionado mais cedo, o microarray dephosphorylated mede o nível de fosforilação após o tratamento da fosfatase e conseqüentemente o nível da linha de base para todos experimentos restantes. Quanto menor o nível basal, maior o alcance dinâmico dos ensaios. Os arrays autofosforilados apresentam os níveis máximos de fosforilação de todas as matrizes e o sinal deve ser forte e claro. Ele é usado para análise de dados, mas também como um controle que todas as reações foram executadas com êxito na matriz.
Limitações da técnica
A partir de agora, uma das limitações da triagem de drogas aqui apresentada é a sua capacidade de tela apenas proteínas quinases que podem ser autofosforiladas. Uma maneira possível de superar isso é imprimir uma quinase e substrato conhecido no mesmo local. A co-impressão do ADN para duas proteínas distintas foi realizada com sucesso15, sugerindo a viabilidade desta aproximação. Além disso, a proteína indicada na matriz pode não ser dobrada corretamente, resultando em uma proteína inativa. O uso do sistema de expressão com base humana fez uma melhora significativa na atividade da quinase medida na matriz; no entanto, algumas proteínas ainda não podem ser analisadas na matriz devido à sua inatividade.
Uma segunda limitação é a medida da fosforilação utilizando um anticorpo antifosho-Tyr Pan. Apesar de sua não especificidade em relação ao motivo do local da fosforilação, todas as fosforilações medidas ocorreram em resíduos de tirosina, deixando para trás serinas e treoninas e suas respectivas quinases. Até à data, mais de 10 anticorpos Pan fosho-ser/thr foram testados sem sucesso, apesar de várias tentativas de otimizar a incubação e as condições de lavagem. Um novo sistema de detecção que é independente de anticorpos pode ser a melhor opção para expandir o número de proteínas quinases que podem ser rastreadas para inibição de drogas. Neste contexto, algumas opções estão disponíveis, incluindo radioatividade ou abordagens químicas, como a conjugação de clique. Uma série de otimizações é necessária para minimizar o sinal de fundo e fornecer uma boa faixa dinâmica para os ensaios.
A terceira limitação é a aquisição de clones de cDNA a serem impressos no array. Os clones de cDNA podem ser gerados usando qualquer técnica de clonagem, incluindo sistemas de recombinação específicos do site, como Creator ou gateway23. Outra opção é comprar os clones da biblioteca DNAsu, encontrados em < https://Dnasu.org/dnasu/Home.do >, onde mais de 17.000 clones de cDNAs, incluindo todo o kinome humano, está prontamente disponível para ser usado para a construção de matrizes NAPPA24 .
A quarta limitação é que nem todos os laboratórios estão equipados com equipamentos adequados para fabricar e fazer a tela de seus próprios arrays NAPPA. Este protocolo fornece métodos alternativos para gerar o DNA a ser imprimido no microarray, sem a necessidade de equipamento da elevado-produção, e protocolos para executar manualmente todas as etapas da hibridação. Entretanto, o acesso a um varredor do arrayer e do microarray é ainda necessário. Uma opção para superar esse problema é usar o serviço e a facilidade do NAPPA Core, encontrados em < http://nappaproteinarray.org/>, que distribui Microarrays NAPPA personalizados a um preço acadêmico sem fins lucrativos. Por fim, a partir de qualquer metodologia de triagem, os dados obtidos nas matrizes são suscetíveis a serem artefatos (positivos ou negativos) e, portanto, devem ser validados por meio de ensaios ortogonais.
Significância em relação aos métodos existentes
Várias plataformas estão disponíveis comercialmente para a triagem de proteínas quinases. Uma abordagem rotineiramente utilizada é a de ensaios vinculativos, que podem ser realizados com fragmentos proteicos, domínio quinase, maiores fragmentos proteicos com o domínio quinase e algumas regiões reguladoras, e até mesmo proteínas de comprimento total. As proteínas são geralmente expressas em sistemas bacterianos devido ao custo e simplicidade nos protocolos de expressão e purificação. A interação entre a droga do interesse e a proteína é medida então com algum tipo de ensaio do relatório como a fluorescência ou a presença de tag, por exemplo. A principal limitação deste conjunto de abordagens é o fato de que a proteína não é necessariamente ativa durante a interação com a droga, o que pode resultar na identificação de interações falso-positivas e falsas negativas. Os fragmentos proteicos são particularmente vulneráveis a alterações na conformação e falta de atividade e todos os dados obtidos devem ser validados usando proteínas ativas, preferencialmente em sua forma completa. Outra limitação de algumas das plataformas é a capacidade de tela apenas análogos de ATP, limitando seu uso geral.
A maioria dos serviços comercialmente disponíveis para a triagem de TKIs usando abordagens baseadas em enzimas utilizam apenas versões de tipo selvagem da quinase de interesse, e às vezes apenas alguns mutantes selecionados. Sabendo que a resistência à droga é muito comum em pacientes tratados com TKI, é importante ser capaz de medir a resposta medicamentosa em diferentes mutantes, para a seleção do inibidor mais adequado. Devido à natureza da NAPPA, a triagem de mutantes da quinase é simples e pode ser facilmente realizada, e a única ferramenta necessária é a incorporação do mutante da quinase na coleção de cDNA NAPPA, que pode ser feita por mutagenese local específica, por exemplo.
Aplicações futuras
Uma das formas mais comuns de tratamento decorre na terapia com câncer usando inibidores da quinase é a aquisição de mutações no alvo de drogas durante um curso de tratamento. A triagem desses mutantes em relação à sua resposta aos inibidores da quinase é de vital importância para a seleção da segunda/terceira geração de TKIs para alcançar um tratamento personalizado para cada paciente. A aproximação da seleção da droga apresentada aqui, fornece uma plataforma de seleção imparcial em que todo o inibidor da tirosina quinase pode ser testado de encontro a um painel das quinases do tirosina atuais no genoma humano. Uma vez que as proteínas exibidas em matrizes NAPPA são expressas in vitro a partir do cDNA impresso no slide, qualquer variante mutante pode ser facilmente incorporado na coleção cDNA a ser exibido na matriz. A instalação em que os mutantes da quinase podem ser gerados e expressos na disposição, combinada com o poder da elevado-produção da técnica de NAPPA, fornecem um ambiente original para o estudo de mutantes da quinase e de sua resposta às drogas, fazendo NAPPA apropriado para triagem de medicamentos personalizados, um dos objetivos da medicina de precisão.
Divulgações
Os autores não declaram conflitos de interesse.
Agradecimentos
Os autores gostariam de agradecer a todos no laboratório da LaBaer por sua ajuda e críticas durante o desenvolvimento do projeto. Este projeto foi apoiado pela subvenção NIH U01CA117374, U01AI077883 e Virginia G. Piper Foundation.
Materiais
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| 364 well plates (for arraying) | Genetix | x7020 | |
| 800 µL 96-well collection plate | Abgene | AB-0859 | |
| 96-pin device | Boekel | 140500 | |
| Acetic Acid | Millipore-Sigma | 1.00066 | |
| Acetone 99.9% | Millipore Sigma | 650501 | |
| Aluminum seal for 96 well plates | VWR | 76004-236 | |
| Aminosilane (3-aminopropyltriethoxysilane) | Pierce | 80370 | |
| ANTI-FLAG M2 antibody produced in mouse | Millipore Sigma | F3165 | |
| Anti-Flag rabbit Antibody (polyclonal) | Millipore Sigma | F7425 | |
| ATP 10 mM | Cell Signaling | 9804S | |
| β-Glycerophosphate disodium salt hydrate | Millipore-Sigma | G9422 | |
| bacteriological agar | VWR | 97064-336 | |
| Blocking Buffer | ThermoFisher/Pierce | 37535 | |
| Brij 35 | ThermoFisher/Pierce | BP345-500 | |
| BS3 (bis-sulfosuccinimidyl) | ThermoFisher/Pierce | 21580 | |
| BSA (bovine serum albumin) | Millipore Sigma | A2153 | |
| Coverslip 24 x 60 mm | VWR | 48393-106 | |
| Cy3 AffiniPure Donkey Anti-Mouse IgG (H+L) | Jackson ImmunoResearch | 715-165-150 | |
| DeepWell Block, case of 50 | ThermoFisher/AbGene | AB-0661 | |
| DEPC water | Ambion | 9906 | |
| DMSO (Dimethyl Sulfoxide) | Millipore-Sigma | D8418 | |
| DNA-intercalating dye | Invitrogen | P11495 | |
| DNase I | Millipore-Sigma | AMPD1-1KT | |
| DTT | Millipore-Sigma | 43816 | |
| EDTA | Millipore-Sigma | EDS | |
| Ethanol 200 proof | Millipore-Sigma | E7023 | |
| Filter plates | Millipore-Sigma | WHA77002804 | |
| Gas Permeable Seals, box of 50 | ThermoFisher/AbGene | AB-0718 | |
| Glass box | Wheaton | 900201 | |
| Glass slides | VWR | 48300-047 | |
| Glycerol | Millipore-Sigma | G5516 | |
| HCl (Hydrochloric acid) | Millipore-Sigma | H1758 | |
| HEPES Buffer Solution | Millipore-Sigma | 83264 | |
| Human-based IVTT system | Thermo Scientific | 88882 | |
| ImmunoPure Mouse IgG whole molecule | ThermoFisher/Pierce | 31202 | |
| Isopropanol | Millipore-Sigma | I9516 | |
| KCl (Potassium chloride) | Millipore-Sigma | P9333 | |
| KH2PO4(Potassium phosphate monobasic) | Millipore-Sigma | P5655 | |
| Kinase buffer | Cell Signaling | 9802 | |
| KOAc (Potassium acetate) | Millipore-Sigma | P1190 | |
| Lambda Protein Phosphatase | new england biolabs | P0753 | |
| Lifterslips, 24 x 60 mm | ThermoFisher Scientific | 25X60I24789001LS | |
| Metal 30-slide rack with no handles | Wheaton | 900234 | |
| MgCL2 (Magnesium chloride) | Millipore-Sigma | M8266 | |
| Na3VO4 (Sodium orthovanadate) | Millipore-Sigma | S6508 | |
| NaCl (Sodium Chloride) | Millipore-Sigma | S3014 | |
| NaOAc (Sodium acetate) | Millipore-Sigma | S2889 | |
| NaOH (Sodium hydroxide) | Millipore-Sigma | S8045 | |
| NucleoBond Xtra Midi / Maxi | Macherey-Nagel | 740410.10 / 740414.10 | |
| Nucleoprep Anion II | Macherey Nagel | 740503.1 | |
| Phosphoric Acid | Millipore-Sigma | 79617 | |
| Poly-L-Lysine Solution (0.01%) | Millipore-Sigma | A-005-C | |
| Protein Phosphatase (Lambda) | New England Biolabs | P0753 | |
| RNAse | Invitrogen | 12091021 | |
| SDS (Sodium dodecyl sulfate) | Millipore-Sigma | L6026 | |
| SDS (Sodium dodecyl sulfate) | Millipore-Sigma | 05030 | |
| Sealing gasket | Grace Bio-Labs, Inc | 44904 | |
| Silica packets | VWR | 100489-246 | |
| Single well plate | ThermoFisher/Nalge Nunc | 242811 | |
| Sodium acetate (3M, pH 5.5) | Millipore-Sigma | 71196 | |
| TB media (Terrific Broth) | Millipore-Sigma | T0918 | |
| Tris | IBI scientific | IB70144 | |
| Triton X-100 | Millipore-Sigma | T8787 | |
| Tryptone | Millipore-Sigma | T7293 | |
| Tween 20 | Millipore-Sigma | P9416 | |
| Yeast Extract | Millipore-Sigma | Y1625 | |
| Name | Company | Catalog number | Comments |
| Equipments | Maker/model | ||
| Programmable chilling incubator | Torrey Pines IN30 Incubator with Cooling | ||
| Shaker for bacterial growth | ATR Multitron shaker | ||
| Vacuum manifold with liquid waste trap | MultiScreenVacuum Manifold 96 well | ||
| 96 well autopippetor/liquid handler | Genmate or Biomek FX | ||
| Liquid dispenser | Wellmate | ||
| DNA microarrayer | Genetix QArray2 | ||
| Automatic hybridization station | Tecan HS4800 Pro Hybridization Station | ||
| Microarray scanner | Tecan PowerScanner | ||
| Microarray data quantification | Tecan Array-ProAnalyzer 6.3 |
Referências
- Melnikova, I., Golden, J. Targeting protein kinases. Nature Review Drug Discovery. 3 (12), 993-994 (2004).
- Patterson, H., Nibbs, R., McInnes, I., Siebert, S. Protein kinase inhibitors in the treatment of inflammatory and autoimmune diseases. Clinical and Experimental Immunology. 176 (1), 1-10 (2014).
- Wu, P., Nielsen, T. E., Clausen, M. H. FDA-approved small-molecule kinase inhibitors. Trends Pharmacological Sciencies. 36 (7), 422-439 (2015).
- Druker, B. J., et al. Effects of a selective inhibitor of the Abl tyrosine kinase on the growth of Bcr-Abl positive cells. Nature Medicine. 2 (5), 561-566 (1996).
- Heinrich, M. C., et al. Inhibition of c-kit receptor tyrosine kinase activity by STI 571, a selective tyrosine kinase inhibitor. Blood. 96 (3), 925-932 (2000).
- Stagno, F., et al. Imatinib mesylate in chronic myeloid leukemia: frontline treatment and long-term outcomes. Expert Review Anticancer Therapy. 16 (3), 273-278 (2016).
- Ben Ami, E., Demetri, G. D. A safety evaluation of imatinib mesylate in the treatment of gastrointestinal stromal tumor. Expert Opinions in Drug Safety. 15 (4), 571-578 (2016).
- Ramachandran, N., et al. Self-assembling protein microarrays. Science. 305 (5680), 86-90 (2004).
- Festa, F., et al. Robust microarray production of freshly expressed proteins in a human milieu. Proteomics Clinical Applications. 7 (5-6), 372-377 (2013).
- Yazaki, J., et al. Mapping transcription factor interactome networks using HaloTag protein arrays. Proceedings of the National Academy of Sciences of the United States of America. 113 (29), E4238-E4247 (2016).
- Rauf, F., et al. Ibrutinib inhibition of ERBB4 reduces cell growth in a WNT5A-dependent manner. Oncogene. 37 (17), 2237-2250 (2018).
- Anderson, K. S., et al. Protein microarray signature of autoantibody biomarkers for the early detection of breast cancer. Journal of Proteome Research. 10 (1), 85-96 (2011).
- Wang, J., et al. Plasma Autoantibodies Associated with Basal-like Breast Cancers. Cancer Epidemiol Biomarkers Prevention. 24 (9), 1332-1340 (2015).
- Bian, X., et al. Tracking the Antibody Immunome in Type 1 Diabetes Using Protein Arrays. Journal of Proteome Research. 16 (1), 195-203 (2017).
- Song, L., et al. Identification of Antibody Targets for Tuberculosis Serology using High-Density Nucleic Acid Programmable Protein Arrays. Molecular and Cellular Proteomics. 16 (4 suppl 1), S277-S289 (2017).
- Wang, J., et al. Comparative Study of Autoantibody Responses between Lung Adenocarcinoma and Benign Pulmonary Nodules. Journal of Thoracic Oncology. 11 (3), 334-345 (2016).
- Montor, W. R., et al. Genome-wide study of Pseudomonas aeruginosa outer membrane protein immunogenicity using self-assembling protein microarrays. Infection and Immunity. 77 (11), 4877-4886 (2009).
- Tang, Y., Qiu, J., Machner, M., LaBaer, J. Discovering Protein-Protein Interactions Using Nucleic Acid Programmable Protein Arrays. Current Protocols in Cell Biology. 74, 11-15 (2017).
- Yu, X., et al. Copper-catalyzed azide-alkyne cycloaddition (click chemistry)-based detection of global pathogen-host AMPylation on self-assembled human protein microarrays. Molecular and Cellular Proteomics. 13 (11), 3164-3176 (2014).
- Anderson, K. S., et al. Autoantibody signature for the serologic detection of ovarian cancer. Journal of Proteome Research. 14 (1), 578-586 (2015).
- Woyach, J. A., Johnson, A. J., Byrd, J. C. The B-cell receptor signaling pathway as a therapeutic target in CLL. Blood. 120 (6), 1175-1184 (2012).
- Smith, M. R. Ibrutinib in B lymphoid malignancies. Expert Opinion on Pharmacotherapy. 16 (12), 1879-1887 (2015).
- Festa, F., Steel, J., Bian, X., Labaer, J. High-throughput cloning and expression library creation for functional proteomics. Proteomics. 13 (9), 1381-1399 (2013).
- Seiler, C. Y., et al. DNASU plasmid and PSI:Biology-Materials repositories: resources to accelerate biological research. Nucleic Acids Research. 42, D1253-D1260 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados