Method Article
Kinase-Inhibitor-Screening in selbstmontierten humanen Protein-Mikroarrays
In diesem Artikel
Zusammenfassung
Ein detailliertes Protokoll zur Erzeugung von selbstzusammengesetzten menschlichen Proteinmikroarrays zum Screening von Kinase-Inhibitoren wird vorgestellt.
Zusammenfassung
Das Screening von Kinase-Inhibitoren ist entscheidend für ein besseres Verständnis der Eigenschaften eines Arzneimittels und für die Identifizierung potenziell neuer Ziele mit klinischen Implikationen. Es wurden mehrere Methoden berichtet, um ein solches Screening durchzuführen. Jeder hat jedoch seine eigenen Grenzen (z. B. das Screening nur von ATP-Analoga, die Beschränkung auf die Verwendung von gereinigten Kinase-Domänen, erhebliche Kosten im Zusammenhang mit der Prüfung von mehr als ein paar Kinasen gleichzeitig und mangelnde Flexibilität bei der neuartigen Mutationen). Hier wird ein neues Protokoll vorgestellt, das einige dieser Einschränkungen überwindet und für das unvoreingenommene Screening von Kinase-Inhibitoren verwendet werden kann. Eine Stärke dieser Methode ist ihre Fähigkeit, die Aktivität von Kinase-Inhibitoren über mehrere Proteine hinweg zu vergleichen, entweder zwischen verschiedenen Kinasen oder verschiedenen Varianten derselben Kinase. Es werden selbst zusammengesetzte Proteinmikroarrays eingesetzt, die durch die Expression von Proteinkiinasen durch ein humanbasiertes In-vitro-Transkriptions- und -translationssystem (IVTT) erzeugt werden. Die auf dem Mikroarray angezeigten Proteine sind aktiv, was die Messung der Wirkung von Kinase-Inhibitoren ermöglicht. Im folgenden Verfahren werden die Protokollschritte im Detail beschrieben, von der Mikroarraygenerierung und dem Screening bis zur Datenanalyse.
Einleitung
Proteinkiinasen sind für die Phosphorylierung ihrer Targets verantwortlich und können komplexe molekulare Bahnen modulieren, die viele zelluläre Funktionen steuern (d. h. Zellproliferation, Differenzierung, Zelltod und Überleben). Die Deregulierung der Kinase-Aktivität ist mit mehr als 400 Krankheiten verbunden, was Kinase-Hemmer zu einer der wichtigsten Klassen von Medikamenten macht, die für die Behandlung mehrerer Krankheiten, einschließlich Krebs, Herz-Kreislauf- und neurologischen Erkrankungen sowie entzündlicher und Autoimmunerkrankungen1,2,3.
Mit dem Aufkommen der Präzisionsmedizin hat die Identifizierung neuer Therapien, insbesondere Kinase-Inhibitoren, eine große pharmazeutische und klinische Anziehungskraft. Mehrere Ansätze können für die Identifizierung möglicher neuer Paarvon Kinase/Kinase-Inhibitoren verwendet werden, einschließlich der De-Novo-Konstruktion von Kinase-Inhibitoren und der Identifizierung neuer Ziele für bestehende FDA-zugelassene Medikamente. Letzteres ist besonders attraktiv, da der Zeit- und Geldaufwand für die Umsetzung dieser Medikamente in Kliniken aufgrund der Verfügbarkeit früherer klinischer Studiendaten drastisch reduziert wird. Ein kanonisches Beispiel für die Umnutzung eines Kinase-Inhibitors ist Imatinib, das ursprünglich für die Behandlung chronischer myelogenöser Leukämie (CML) durch die Hemmung von BCR-Abl entwickelt wurde, das auch erfolgreich zur Behandlung von c-Kit-Überexsedern eingesetzt werden kann. gastrointestinale Stromaltumoren (GISTs)4,5,6,7.
Das Screening von Kinase-Inhibitoren kann in bindungsbezogenen Assays oder enzymatischen Assays durchgeführt werden. Die erste Klasse von Assays konzentriert sich auf Protein-Arzneimittel-Wechselwirkungen und kann Informationen wie Ligation Sitte und Affinität liefern. Da die Aktivität der Kinase zum Zeitpunkt dieser Tests unbekannt ist, kann eine Reihe von Wechselwirkungen aufgrund von konformen Veränderungen im Protein übersehen oder fälschlicherweise identifiziert werden. Auf der anderen Seite erfordern enzymatische Assays, dass die Proteinkiinasen aktiv sind und wertvolle Informationen über die Wirkung des Inhibitors auf die Enzymaktivität liefern, aber diese Art des Screenings ist in der Regel zeitaufwändiger und teurer. Derzeit sind beide Arten von Assays kommerziell aus verschiedenen Quellen erhältlich. Sie stellen eine zuverlässige Option für das Screening von Kinase-Inhibitoren mit einigen Einschränkungen dar, darunter: I) die meisten Methoden beinhalten das Testen mehrerer Kiinasen einzeln, was das Screening einer großen Gruppe von Proteinen kostspielig machen kann; II) Der zu prüfende Satz von Kiinasen beschränkt sich auf eine Liste vorgewählter, wildartgebundener Kinäasen und mehrerer bekannter mutierter Versionen einiger Kiinasen, was die Prüfung vieler neuer mutierter Isoformen verhindert.
In diesem Zusammenhang sind Protein-Mikroarrays eine leistungsstarke Plattform, die in der Lage ist, einige der Einschränkungen zu überwinden, die durch kommerziell verfügbare Techniken dargestellt werden. Es eignet sich für enzymatische Assays im Hochdurchsatz-Screening mit in voller Länge, aktiven Proteinen jeder Sequenz von Interesse. Die Mikroarrays können durch einen selbstzusammengesetzten Ansatz wie NAPPA (Nukleinsäure programmierbares Proteinarray) erzeugt werden, bei dem Proteine gerade rechtzeitig für die Assays exprimiert werden, was die Wahrscheinlichkeit erhöht, dass die auf dem Array angezeigten Proteine tatsächlich aktiv sind. Die auf NAPPA angezeigten Proteine werden mit humanabgeleiteten Ribosomen und Chaperonproteinen hergestellt, um die Wahrscheinlichkeit natürlicher Faltung und Aktivität zu verbessern.
Die Proteine werden zunächst programmiert, indem cDNAs, die für Gene von Interesse kodiert werden, die mit einem Capture-Tag zusammen mit einem Erfassungsmittel verschmolzen werden, auf die Mikroarray-Oberfläche gedruckt werden. Die Proteine werden dann auf den Mikroarrays mit einem In-vitro-Transkriptions- und -übersetzungssystem (IVTT) produziert, und die frisch exprimierten Proteine werden durch das Erfassungsmittel auf der Mikroarray-Oberfläche immobilisiert. Ausgedrückte NAPPA-Arrays können für die Untersuchung der auf dem Array angezeigten Proteine in einer unvoreingenommenen, hochdurchsatzigen Art und Weise8,9verwendet werden.
Zuvor wurde gezeigt, dass Proteine, die auf NAPPA-Arrays angezeigt werden, richtig gefaltet werden, um mit bekannten Partnern zu interagieren10; Darüber hinaus wurde ihre enzymatische Aktivität erstmals im Jahr 2018 genutzt, als gezeigt wurde, dass Proteinkiinasen auf dem Mikroarray-Autophosphorylat11angezeigt wurden. Bis heute wurde die NAPPA-Methodik für viele verschiedene Anwendungen verwendet, einschließlich der Biomarker-Entdeckung12,13,14,15,16,17, Protein-Protein-Wechselwirkungen10,18, Substrat-Identifikation19und Arzneimittel-Screening11. Seine Flexibilität ist eines der wichtigsten Merkmale der Plattform, die eine Anpassung an jede Anwendung ermöglicht.
Hier wird ein Protokoll zum Screening von Tyrosinkinase-Inhibitoren in selbst zusammengesetzten NAPPA-Arrays vorgestellt. Die Plattform ist für die Anzeige aktiver menschlicher Proteinkinasen und für die Analyse der Proteinkinase-Aktivität mit niedrigem Hintergrund und hohem Dynamikbereich optimiert. Zu den Änderungen, die zur Verwendung von NAPPA für das Screening von Kinase-Inhibitoren implementiert wurden, gehören: I) Veränderungen in der Druckchemie, II) Dephosphorylierung des Proteinmikroarrays vor dem Kinase-Inhibitor-Screening und III) Optimierung des Nachweises von phosphorylierte Proteine auf dem Array. Dieses Protokoll ist das erste seiner Art und liefert einzigartige Informationen über die Kinase-Studie in NAPPA-Mikroarrays.
Protokoll
1. Gemeinsame Puffer und Lösungen, die verwendet werden sollen
- TB-Medium vorbereiten: Terrific Brühe (24 g/L Hefeextrakt; 20 g/L Trypton; 4 ml/L Glycerin; 0,017 M KH2PO4; und 0,072 M K2HPO4). Die Lösungen 0,017 M KH2PO4 und 0,072 M K2HPO4 können als 10x Phosphatpuffer (0,17 M KH2PO4 und 0,72 M K2HPO4)erworben werden.
- Bereiten LB Medium: Luria-Bertani (5 g/L Hefeextrakt; 10 g/L Trypton; und 10 g/L NaCl). Stellen Sie den pH-Wert auf 7,0 mit 5 M NaOH ein.
- 1x TBS vorbereiten: Tris-gepufferte Salin (TBS: 50 mM Tris-Cl, pH = 7,5; 150 mM NaCl).
- Bereiten Sie 1x TBST vor: TBS ergänzt mit 0,1% Tween 20.
2. DNA-Vorbereitung
HINWEIS: Die DNA, die für NAPPA-Arrays verwendet wird, muss sehr rein sein; daher werden kommerzielle DNA-Mini-Preps nicht empfohlen. Derzeit werden zwei Protokolle zur DNA-Vorbereitung verwendet: im Haus High-Throughput Mini-Prep (hier beschrieben) oder kommerzielle Midi- oder Maxi-Prep. Der durchschnittliche Durchsatz des hauseigenen Mini-Prep-Protokolls beträgt 1.500 Proben pro Tag und Person.
- Bakterielles Wachstum für hauseigene Mini-Prep mit hohem Durchsatz
- Bereiten Sie LB/Agar Omni Platte. 30–40 ml LB-Agar (1,5 % bakteriologischer Agar in LB-Medien, ergänzt mit Antibiotikum zur Auswahl positiver Klone) in jede einzelne Brunnenplatte gießen.
- Spot Glycerin Lager auf LB/Agar Platte. Verdünnung des Glycerinbestands in LB-Medien (1:300, in der Regel 2 l in 600 l LB). Schütteln Sie für 10 min. Spot 3 l des verdünnten Vorrats auf die LB/Agar-Platte. Bei 37 °C auf den Kopf stellen, über Nacht.
- Inokulieren Kulturen. Mit dem 96-Pin-Gerät, das in 80% Ethanol und Flamme sterilisiert wurde, impfen Sie die Kultur von der Agarplatte in einem Tiefbrunnenblock mit 1,5 ml pro Brunnen TB-Medium, ergänzt mit Antibiotikum.
- Inkubieren Kulturen. Den Block mit einer gasdurchlässigen Dichtung bedecken und für 22–24 h bei 37 °C, je nach Shaker 300–800 Umdrehungen pro Minute inkubieren.
HINWEIS: Shaker, die auf 800 Rpm eingestellt sind, sind optimal für diese Inkubation. Die Verwendung eines langsameren Geschwindigkeitsstreuers kann zu weniger dichten Kulturen und geringeren DNA-Reinigungserträgen führen. - Pelletkulturen. Spin-Blöcke bei 3.800 x g und 4 °C für 30 min. Überstand entsorgen.
- Inhouse High-Throughput Mini-Prep
HINWEIS: Mehrkanal-Pipettoroder oder automatische Spender können verwendet werden, um die hauseigene High-Throughput-Mini-Vorbereitung durchzuführen. Wenn Sie einen automatischen Spender verwenden, stellen Sie sicher, dass Sie das System vor der Verwendung und zwischen den Lösungen reinigen.- Bereiten Sie alle Lösungen vor, die während der Mini-Vorbereitung verwendet werden:
- Vorbereiten der Lösung 1: TE-Resuspensionspuffer (50 mM Tris, pH = 8,0; 10 mM EDTA, pH = 8,0; 0,1 mg/ml RNAse). Bei 4 °C lagern.
- Vorbereiten von Lösung 2: NaOH/SDS-Lysepuffer (0,2 M NaOH; 1% SDS). Für bessere Ergebnisse sollte eine frisch zubereitete Lösung verwendet werden.
- Vorbereiten von Lösung 3: KOAC-Neutralisationspuffer (2,8 M KOAc). Stellen Sie den pH-Wert der Lösung mit Eisessig auf 5,1 ein. Bei 4 °C lagern.
- Vorbereiten der Lösung N2: Ausgleichspuffer (100 mM Tris; 900 mM KCl; 15% EtOH; 0,15% Triton X-100). Stellen Sie den pH-Wert der Lösung mit Phosphorsäure auf 6,3 ein.
- Vorbereiten der Lösung N3: Bash-Puffer (100 mM Tris; 1,15 M KCl; 15% EtOH). Stellen Sie den pH-Wert der Lösung mit Phosphorsäure auf 6,3 ein.
- Vorbereiten der Lösung N5: Elutionspuffer (100 mM Tris; 1 M KCl; 15% EtOH). Stellen Sie den pH-Wert der Lösung mit Phosphorsäure auf 8,5 ein.
HINWEIS: Die erfolgreiche Kontrolle der DNA-Bindung, -Wasch- und -elution während des Anionenaustauschs ist in hohem Maße von Puffer-KCl-Konzentration und pH-Werten abhängig. Sorgfältige Pufferkomponentenmessungen und pH-Anpassung sind unerlässlich. Kleine Abweichungen von den beschriebenen Messungen können zu signifikanten Ertragsverlusten führen.
- Pellet wieder aufhängen. Fügen Sie 200 l Lösung 1 hinzu und schütteln Sie bei 2.000 Rpross en 5 min bei RT. Für eine erfolgreiche Lyse ist eine vollständige Wiederaufhängung des Pellets erforderlich. Wirbeln Sie den Block bei Bedarf.
- Lyse-Bakterien. Fügen Sie 200 l Lösung 2 hinzu, versiegeln Sie die Platte mit einer Aluminiumdichtung und invertieren Sie 5x. Diesen Schritt sorgfältig vom Anfang der Lösung 2 Ergänzung. Nicht mehr als 5 min.
- Neutralisieren Sie die Lösung. Fügen Sie 200 l Lösung 3 hinzu, versiegeln Sie die Platte mit einer Aluminiumdichtung und invertieren Sie 5x. Die Dichtung kann aufgrund der Lyse-/Neutralisationspuffer locker sein, also vorsichtsgeboten beim Invertieren. Eine partielle Umkehrung, bei der die Lösung niemals die Dichtung berührt, wird empfohlen, um Kreuzkontaminationen zwischen den Proben zu verhindern.
- Klares Lysat. Zentrifugieren Sie die Platten bei 3.800 x g und 4 °C für 30 min.
- Vorbereiten der Anionenaustauschharzschlämme während des Lysat-Pelletzentrifugationsschritts. Mit einer 1 L-Flasche mit dem Anionenaustauschharz füllen, bis es die 300 ml-Marke erreicht, dann Lösung N2 bis 900 ml hinzufügen.
VORSICHT: Dieser Schritt sollte in der Haube durchgeführt werden, um vor Kieselsäure-Inhalation zu schützen. - Anionenaustauschharzplatten vorbereiten. Stapeln Sie die Filterplatten auf einem tiefen Brunnenblock, um als Abfallsammelbehälter zu fungieren. Mischen Sie die Anionenaustauschgülle, bis sie homogen ist, dann in einen Glastrog gießen. Mit weitgebohrten P1000-Spitzen 450 l der Gülle in jeden Brunnen der Filterplatten übertragen.
- Zentrifugengestapelte Platten (Harzplatte/Tiefe Brunnenplatte) bei langsamer Beschleunigung für 5 min bei 130 x g und RT. Entsorgen Sie den Durchfluss.
- Übertragen Sie Lysat-Überstand auf die Harzplatte/Tiefe Brunnenblockstapel. Drehen Sie die gestapelten Platten für 5 min bei 30 x g mit langsamer Anfahrgeschwindigkeit.
- Waschsäule. Fügen Sie jedem Brunnen 400 l lösung N3 (Waschpuffer) hinzu. Übertragen Sie die Harzplatte auf Vakuumkrümmer, um den Waschpuffer zu entfernen. Wiederholen Sie die Waschschritte 3x. Achten Sie beim letzten Waschen darauf, dass alle Brunnen richtig geleert sind. Drehen Sie die Stapelplatten bei 150 x g für 5 min, um einen Restpuffer zu entfernen.
- Elute DNA. Legen Sie die Harzplatte auf eine saubere 800-L-Sammelplatte. Fügen Sie jedem Bohrwert 300 L der Lösung N5 hinzu. Lassen Sie es bei RT für 10 min sitzen, dann drehen Sie die gestapelten Platten für 5 min bei 20 x g mit langsamer Anlaufgeschwindigkeit. Drehen Sie die gestapelten Platten für 1 min bei 233 x g.
- Quantifizieren Sie DNA und speichern Sie Platten bei -20 °C bis zur weiteren Verwendung oder gehen Sie direkt zur DNA-Fällung über.
HINWEIS: Pro Probe sind mindestens 30 g DNA erforderlich. Wenn die DNA-Ausbeute gering ist, wird empfohlen, die DNA-Mini-Vorbereitung zu wiederholen oder alternativ zwei Platten während des Niederschlagsschritts zu kombinieren (Abschnitt 2.3).
- Bereiten Sie alle Lösungen vor, die während der Mini-Vorbereitung verwendet werden:
- DNA-Fällung
- Platten auftauen, Wirbel zur Homogenisierung der DNA-Lösung auftauen und bei 230 x g für 30 s drehen, um alle Lösungen im Boden des Brunnens zu sammeln.
- Fügen Sie 40 l von 3 M NaOAc und 240 l Isopropanol zu jedem Brunnen hinzu. Bedecken Sie die Platte mit einer Aluminiumdichtung und mischen Sie sie, indem Sie 3x invertieren.
- Zentrifugieren Sie die Platten bei 30 min bei 3.800 x g und 25 °C. Entsorgen Sie den Überstand vorsichtig.
ANMERKUNG: Um zwei Platten zu kombinieren, übertragen Sie die DNA von der zweiten Platte in die Pellet von der ersten Platte und wiederholen Sie die Schritte 2.3.2–2.3.3. - Waschen und fällen Sie die DNA. Fügen Sie 400 l 80% Ethanol zu jedem Brunnen hinzu. Dichtungsplatten mit Aluminiumdichtung und Schütteln bei 1.000 U/min für 30 min. Zentrifuge bei 3.800 x g für 30 min bei 25 °C. Entsorgen Sie den Überstand.
- Trocknen Sie die DNA-Pellets. Legen Sie die Teller kopfüber auf Papiertücher und lassen Sie sie 1–2 h trocknen, bis am Boden des Brunnens kein Alkohol vorhanden ist. Versiegeln und Zentrifuge bei 230 x g für 2 min, um alle Pellets nach unten zu bringen.
- Sobald die Platten trocken sind, entweder mit Aluminiumdichtung abdichten und bei -20 °C für den späteren Gebrauch einfrieren oder die DNA weiter aussetzen (Schritt 4.1).
3. Aminosilan-Schlittenbeschichtung
- Legen Sie die Glasgweise in metallene Rack. Überprüfen Sie jede Folie visuell, um sicherzustellen, dass keine Kratzer oder Unvollkommenheiten vorhanden sind.
- Untertauchen von Dias in Beschichtungslösung (2% Aminosilanreagenz in Aceton) für 15 min beim Schaukeln. Die Aminosilan-Lösung kann verwendet werden, um zwei Racks mit jeweils 30 Dias zu beschichten, bevor sie entsorgt werden muss.
- Spülen Sie Schritt. Das Schiebegestell in Acetonwäsche (99% Aceton) untertauchen, hin und her schütteln, dann 5x schnell auf und ab. Neigen Sie zu einer Ecke ab, um abzutropfen, dann tauchen Sie in Ultrapure Wasser auf und ab schnell 5x. Kippen, um abzutropfen, dann auf Servietten legen.
HINWEIS: Acetonwäsche kann zweimal verwendet werden, während das Reinstwasser jedes Mal gewechselt werden muss. - Trockene Schlitten mit Druckluft, blasen auf sie aus allen Winkeln für ca. 3 min, bis alle Wassertröpfchen entfernt wurden. Bewahren Sie die beschichteten Dias bei RT in einem Metallgestell in einer dicht verschlossenen Box auf.
4. Array-Probenvorbereitung
- Das DNA-Pellet aus der hauseigenen Mini-Vorbereitung (Schritt 2.3.6) in 20 l Reinstwasser wieder aufhängen und bei 1.000 Umdrehungen pro Minute für 2 h schütteln. Für Midi/Max-Prep-DNA jede Probe auf eine Endkonzentration von 1,5 g/l verdünnen und 20 l auf eine 800-L-Sammelplatte übertragen.
- Bereiten Sie den Druckmix vor. Für eine 96-Well-Platte 1 ml Druckmischung [237,5 l Reinstwasser; 500 l Polylysin (0,01%); 187,5 l BS3 (Bis-Sulfosuccinimidyl, 50 mg/ml in DMSO) und 75 l polyklonale Antiflag-Antikörper vorbereiten.]
HINWEIS: Die Chemikalien sollten in der angegebenen Reihenfolge hinzugefügt werden, um Niederschläge zu vermeiden. - Fügen Sie jeder Probe 10 l Druckmischung hinzu, versiegeln Sie Platten mit Aluminiumfolie und schütteln Sie bei RT 90 min bei 1.000 Umdrehungen pro Minute. Lagern Sie die Platten über Nacht (ca. 16 h) bei 4 °C.
- Am Drucktag kurz wirbeln und drehen Sie die Platten. Übertragen Sie 28 l jeder Probe auf eine 384-Array-Platte. Diese Übertragung kann mit Automatisierung oder einer Mehrkanalpipette erfolgen. Es ist wichtig, die Position der Proben in der 384-Array-Platte zu verfolgen.
- Drehen Sie die Platte kurz nach unten, um Blasen zu entfernen. Versiegeln Sie die Platten mit Folie.
5. Generierung von NAPPA-Arrays: Mikroarray-Druck
HINWEIS: Alle Druckbedingungen wurden für das in Tabelle der Ausrüstungunden und Materialienaufgeführte Instrument optimiert. Wenn Sie ein anderes Array verwenden, ist möglicherweise eine weitere Optimierung erforderlich.
- Arrayer bereinigen. Vor dem Start alle Abfallbehälter entleeren und die Reservoirs bei Bedarf mit Reinstwasser oder 80% Ethanol auffüllen. Reinigen Sie Die Stifte eins nach dem anderen mit fusselfreien Tüchern und Reinstwasser. Trocknen Sie die Stifte mit fusselfreien Tüchern und legen Sie sie vorsichtig wieder in den Arrayerkopf.
- Arrayer eingerichtet: Druckspezifikationen [maximale Anzahl der Briefmarken pro Tinte: 1; Anzahl der Stempel pro Punkt: 1; Multi-Stempel-Timing: --; Stempelzeit (ms): 0 ms; Farbzeit (ms): 0 ms; Drucktiefeneinstellung: 90 Mikrometer; Anzahl der Touch-offs: 0]. Dann folgen Sie dem Sterilisationsprotokoll: Reinstwasserwäsche für 2.000 ms mit 0 ms Trocknungszeit und 500 ms Wartezeit; wiederholen Sie diese Schritte 6x; gefolgt vom Waschen mit 80% Ethanol für 2.000 ms mit 1.200 ms Trocknungszeit und 500 ms Wartezeit; wiederholen Sie diese Schritte 6x.
- Foliendesign: Richten Sie den Arrayer mit dem gewünschten Arraying-Muster ein. Der Entwurf sollte mehrere Faktoren berücksichtigen [d. h. die Anzahl der Replikate für jedes Beispiel, den Speicherort und die Anzahl der Steuerungsmerkmale, das Arraylayout (ein Block, mehrere identische Blöcke), die Anzahl der zu druckenden Arrays, die Länge der Ausführung usw.].
- Legen Sie die aminosilanbeschichteten Dias (Schritt 3.4) auf das Arrayerdeck. Überprüfen Sie, ob das Vakuum alle Dias sicher an Ort und Stelle hält. Starten Sie den Luftbefeuchter (er sollte auf 60%) eingestellt werden.
- Platzieren Sie die 384 Well Platte auf dem Arrayerdeck. Starten Sie das Programm.
- Beschriften Sie die Mikroarrays. Platzieren Sie beim Drucken Folienetiketten auf der unteren (nicht gedruckten) Seite jeder Folie. Bewahren Sie die Druckreihenfolge der Folien auf dem Deck in numerischer Reihenfolge auf.
- Bewahren Sie die gedruckten Arrays bei RT in einem Metallgestell in einer dicht verschlossenen Box mit einem Kieselsäurepaket auf. In trockener Umgebung gehaltene Dias haben eine Haltbarkeit von bis zu einem Jahr.
- (Optional): Ein zweiter Stapel von 90 Dias kann mit den gleichen Mustern gedruckt werden. Entfernen Sie dazu die 384-Well-Platte vom Arrayer-Deck, sobald der Druck der Platte erfolgt ist. Versiegeln und lagern Sie die Platte bei 4 °C. Nachdem die erste Charge von Arrays vollständig fertig sind, entfernen Sie sie aus dem Deck, platzieren Sie neue Aminosilan-beschichtete Dias und starten Sie einen neuen Lauf. Stellen Sie sicher, dass jede 384 Well Platte bei RT für 30 min vor seiner Verwendung ist. Wenn mehr als vier Replikate pro Probe in einer Reihe von Dias gedruckt werden, wird empfohlen, die 384 Wellplatten in zwei Platten aufzuteilen, um die Probenverdampfung zu verringern, indem die auf dem Arrayerdeck verbrachte Zeit verringert wird.
HINWEIS: Überprüfen Sie alle Reservoirs vor Beginn des zweiten Durchlaufs.
6. Dna-Detektion auf NAPPA-Dias
- Blockieren Sie die Folien. Legen Sie die Dias in eine Pipettenbox und fügen Sie 30 ml Sperrpuffer hinzu. Bei RT für 1 h auf einem Schaukel-Shaker bebrüten.
- Beflecken Sie die Dias. Entsorgen Sie die Blockierlösung und fügen Sie 20 ml Blockierpuffer und 33 l fluoreszierenden DNA-interkalierenden Farbstoff hinzu. 15 min mit Rührung inkubieren. Dann spülen Sie die Dias schnell mit Reinstwasser ab und trocknen sie mit Druckluft. Fahren Sie mit dem Scannen fort (Abschnitt 11).
7. Ausdruck von NAPPA-Folien
- Blockieren Sie die Dias mit Sperrpuffer auf einem Schaukel-Shaker bei RT für 1 h. Verwenden Sie ca. 30 ml in einer Pipettenbox für vier Dias.
- Schlitten mit Reinstwasser spülen und mit gefilterter Druckluft trocknen. Tragen Sie die Dichtung senket auf jeden Schlitten gemäß den Anweisungen des Herstellers auf.
- Fügen Sie IVTT-Mix hinzu. Für jedes Dia werden 150 L IVTT-Mischung benötigt. Verdünnen Sie 82,5 l HeLa-Lysat in 33 l DEPC-Wasser und ergänzen Sie sie mit 16,5 l Zubehörproteinen und 33 uL Reaktionsmix. Fügen Sie ivTT-Mischung aus dem Nicht-Label- oder Nicht-Probe-Ende hinzu. Pipette die Mischung in langsam (es ist akzeptabel, wenn es perlen vorübergehend am Einlass Ende). Massieren Sie die Dichtungsdichtung vorsichtig, so dass sich die IVTT-Mischung ausbreitet und den gesamten Bereich des Arrays abdeckt. Tragen Sie die kleinen runden Hafendichtungen auf beide Ports auf.
- Legen Sie die Dias auf einen Träger und übertragen Sie sie auf den programmierbaren Chilling-Inkubator. Für die Proteinexpression 90 min bei 30 °C inkubieren, gefolgt von 30 min bei 15 °C zur Immobilisierung des Abfrageproteins.
- Waschen und blockieren Sie die Dias. Entfernen Sie die Dichtungsdichtung und tauchen Sie die Dias in eine Pipettenbox mit ca. 30 ml 1x TBST, ergänzt mit 5% Milch zur Proteinanzeige (Abschnitt 8) oder 1x TBST, ergänzt mit 3% Rinderserumalbumin (BSA) für Kinase-Assays oder Arzneimittelscreening (Abschnitt 9). Inkubieren Sie bei RT mit Agitation für 20 min und wiederholen Sie diesen Schritt 2x.
8. Nachweis von Proteinen auf NAPPA-Arrays
- Fügen Sie primären Antikörper hinzu. Entfernen Sie die Dias von der Blockierlösung (Schritt 7.5) und trocknen Sie die Rückseite (nicht bedruckte Seite) vorsichtig mit einem Papiertuch. Legen Sie die Dias auf eine Stütze und wenden Sie 600 l primärer Antikörper (Maus-Anti-Flag) verdünnt 1:200 in 1x TBST + 5% Milch an. 1 h bei RT inkubieren.
- Waschen Sie die Dias mit 1x TBST + 5% Milch auf einem Schaukelschüttler (3x für je 5 min).
- Fügen Sie sekundären Antikörper hinzu. Entfernen Sie die Dias von der Waschlösung und trocknen Sie die Rückseite vorsichtig mit papiertuch. Legen Sie die Dias auf eine Stütze und wenden Sie 600 l Sekundärantikörper (cy3-labbeled Anti-Maus-Antikörper) verdünnt 1:200 in 1x TBST + 5% Milch auf. Schützen Sie die Dias vor Licht und brüten Sie 1 H bei RT.
- Waschen Sie die Dias mit 1x TBST auf einem Schaukel-Shaker (3x für je 5 min). Spülen Sie die Dias schnell mit Reinstwasser und trocknen Sie mit Druckluft. Fahren Sie mit dem Scannen fort (Abschnitt 11).
9. Tyrosin-Kinase-Inhibitor-Screening auf NAPPA-Arrays
HINWEIS: Mehrere Folien können im selben Experiment verarbeitet werden, stellen Sie jedoch sicher, dass in jedem Schritt eine Folie gleichzeitig verarbeitet wird und dass sie zwischen den Schritten nicht trocknen. Fügen Sie alle Lösungen am Nicht-Label- oder Nicht-Specimen-Ende des Dias hinzu.
- Bereiten Sie alle Lösungen vor, die während des Drogenscreenings verwendet werden:
- Bereiten Sie die Phosphatase/DNase-Lösung vor, indem Sie Folgendes kombinieren: 1x Protein-Metallo-Phosphatasen-Puffer (50 mM HEPES, 100 mM NaCl, 2 mM DTT, 0,01% Brij 35 bei pH = 7,5); 1 mM MnCl2; 8.000 Einheiten Lambda-Proteinphosphatase; und 2 Einheiten DNase I. Bereiten Sie 400 l der Lösung für jedes Mikroarray vor. Fügen Sie Phosphatase und DNase kurz vor der Verwendung hinzu.
- Machen Sie die Droge Verdünnung. Die Medikamente werden in DMSO auf eine Endkonzentration von 10 mM rekonstituiert. Um sicherzustellen, dass alle auf dem Array getesteten Arzneimittelkonzentrationen sicherstellen, dass für jede Konzentration das gleiche Volumen an DMSO (ein 10.000-facher Vorrat in DMSO) entsteht und bei -80 °C gehalten wird. Zum Zeitpunkt des Gebrauchs werden die Medikamente 1:100 in Wasser verdünnt.
- Bereiten Sie Die Medikament/Kinase-Lösung durch die Kombination der folgenden: 1x Kinase Puffer (25 mM Tris-HCl von pH = 7.5); 5 mM Beta-Glycerophosphat; 2 mM DTT; 0,1 mM Na3VO4; 10 mM MgCl2; 500 M ATP; und 2 l Desarzneimittel (verdünnt 1:100 in Wasser). Bereiten Sie für jedes Mikroarray 200 l der Lösung vor.
- Führen Sie die Phosphatase- und DNase-Behandlung durch. Entfernen Sie die Dias von der Blockierlösung (Schritt 7.5) und trocknen Sie die Rückseite vorsichtig mit dem Papiertuch. Legen Sie die Dias auf die Stütze und tragen Sie 200 L Phosphatase/DNase-Lösung auf. Legen Sie einen Mikroarray-Abdeckungsslip auf, um Verdunstung zu vermeiden. Bei 30 °C für 45 min im Ofen brüten.
- Phosphatase- und DNase-Behandlung II: Arrays aus dem Ofen entfernen, den Deckelzettel entsorgen, überschüssige Lösung entfernen und 200 L frisch hergestellte Phosphatase- und DNase-Lösung auftragen. Mikroarrays mit Deckelschlupf abdecken und weitere 45 min bei 30 °C im Ofen inkubieren.
- Schlitten mit 1x TBST + 0,2 M NaCl auf einem Schaukelshaker (3x für je 5 min) waschen.
- Führen Sie die medikamentöse Behandlung und Kinase-Reaktion. Entfernen Sie die Dias von der Waschlösung und trocknen Sie die Rückseite vorsichtig mit einem Papiertuch. Legen Sie die Dias auf Unterstützung und wenden Sie 200 L Medikament / Kinase-Lösung. Legen Sie einen Deckelschlupf oben, um Verdunstung zu vermeiden. 1 h bei 30 °C im Ofen brüten.
- Schlitten mit 1x TBST + 0,2 M NaCl auf einem Schaukelshaker (3x für je 5 min) waschen.
- Wiederholen Sie die Schritte 8.1–8.4 mit als primärem Antikörper Antikörper Anti-Phosho-Tyr Antikörper verdünnt 1:100. 1x TBST + 5% Milch in allen Schritten durch 1x TBST + 3% BSA ersetzen.
10. Automatisiertes Hybridisierungsprotokoll
HINWEIS: Alternativ kann eine Hybridisierungsstation verwendet werden, um alle Hybridisierungen und Wärvorgänge auf den NAPPA-Arrays zu automatisieren (Abschnitte 7–9) und das Protokoll wird als Zusatzdatei 1bereitgestellt.
11. Bildaufnahme
HINWEIS: Mikroarray-Bilder sollten mit einer Auflösung von 20 Mikrometern oder höher aufgenommen werden.
- Laden Sie Mikroarrays in das Diahaltermagazin mit den nach oben gerichteten Proteinen. Laden Sie das Magazin in den Microarray-Scanner ein.
- Wählen Sie den grünen Laser mit einem 575/30 nm Emissionsfilter, um das Signal vom cy-3 markierten Sekundärantikörper zu scannen. Wenn ein anderes Fluorophor verwendet wird, wählen Sie den richtigen Laser/Wellenlänge, um das Signal aus dem Fluoreszenzfarbstoff zu erkennen.
- Definieren Sie den Namen für jedes Bild und den Speicherort, an dem sie gespeichert werden sollen.
- (Optional): Für jedes neue Fluorophor wird die Optimierung der Scanbedingungen empfohlen, um den linearen Bereich der Signalintensität zu erkennen. Scannen Sie dazu ein Mikroarray mit einem Bereich von Photomultiplier (PMT) und gewinnen Sie, bis ein klares Bild mit ungesättigtem Signal und niedrigem Hintergrund erhalten ist.
- Scannen Sie alle Microarrays mit den optimierten Einstellungen und denken Sie daran, die Autogain auszuschalten.
HINWEIS: Für die Datenanalyse sollten alle Mikroarrays mit den gleichen Scaneinstellungen gescannt werden. Bei Kinase-Assays, die cy3 als Fluorophor verwenden, werden die Bilder mit einer PMT-Auflösung von 20 %, einer Laserintensität von 25 % und einer Auflösung von 10 Mikrometern mit dem in der Tabelle der Ausrüstungunden und Materialien aufgeführten Scanner gescannt.
12. Datenverarbeitung und -analyse
HINWEIS: Für die Quantifizierung von Mikroarray-Daten mit ähnlichen Funktionen stehen mehrere Softwarepakete zur Verfügung. Das hier beschriebene Verfahren wurde für die in der Tabelle der Ausrüstungunden und Materialienaufgeführte Software entwickelt.
- Laden Sie die zu quantifizierenden TIFF-Dateien, entwerfen Sie das Raster so, dass es dem Mikroarray-Layout entspricht, und passen Sie die Größe der Spots an, um das gesamte Signal mit der minimalen Fläche zu integrieren. Benachbarte Stellen sollten sich nicht überlappen. Überprüfen Sie visuell, wie gut die Software funktioniert, und passen Sie das Raster bei Bedarf manuell an.
- Quantifizieren Sie die Signalintensität des Mikroarrays. Überprüfen Sie die Stellen visuell auf Anomalien (unspezifische Bindung, Staub usw.) und entfernen Sie sie aus der Datenanalyse.
- Korrigieren Sie den Hintergrund lokal mit dem Signal benachbarter Bereiche auf dem Array, in dem kein Fleck vorhanden ist.
- Normalisieren Sie Daten. Um das Signal über verschiedene Arrays hinweg zu vergleichen, muss die Signalintensität jedes Mikroarrays normalisiert werden. Um Ausreißer auszuschließen, normalisieren Sie die Daten mit dem getrimmten Mittelwert 30% des Signals aus der Positivkontrolle (IgG-Spots) der dephosphorylierten Mikroarrays.
HINWEIS: Das Signal des IgG-Spots ändert sich während der Phosphorylierung und Dephosphorylierung der Mikroarrays nicht und ist für die Normalisierung geeignet. - Identifizieren Sie aktive Kiinasen. Berechnen Sie für jedes Feature, das auf dem Mikroarray angezeigt wird, das Verhältnis zwischen der normalisierten Signalintensität in den autophosphorylierten und dephosphorylierten Arrays. Legen Sie einen Schwellenwert von 1,5-facher Änderung für die Identifizierung der aktiven Kinäsen fest und markieren Sie alle anderen Merkmale als nicht in der Lage, sich einer Autophosphorylierung (N/A) zu unterziehen.
- Berechnen Sie die Aktivität jeder kinase, die in Schritt 12.5 identifiziert wird, als Prozentsatz des angepassten Signals (Signalintensität des normalisierten Positivkontrollarrays (DMSO), subtrahiert durch die Signalintensität des normalisierten Negativkontrollarrays (dephosphoryliert).
Ergebnisse
Selbstzusammengesetzte NAPPA-Mikroarrays bieten eine solide Plattform, die für viele verschiedene Anwendungen verwendet werden kann, einschließlich Biomarker-Erkennung, Protein-Protein-Wechselwirkungen, Substrat-Identifikation und Arzneimittelscreening10,11 ,12,13,14,15,16,17,18,19,20.
Die allgemeine Methodik für die Untersuchung der Kinaseaktivität und das Screening von Tyrosin-Kinaseninhibitoren auf NAPPA-Mikroarrays ist schematisch in Abbildung 1dargestellt. Zunächst werden NAPPA-Mikroarrays durch die Immobilisierung von cDNA und Capture-Agent auf die beschichteten Mikroarrays erzeugt. Die cDNAs werden dann als Vorlage für die Transkription und Translation von Proteinen unter Verwendung eines humanbasierten IVTT-Systems verwendet, und die neu synthetisierten Proteine werden durch das Erfassungsmittel9immobilisiert. Die Qualität des gedruckten Mikroarrays kann überwacht werden, indem die DNA-Werte (Bestätigung des konsistenten Drucks) oder des auf dem Array angezeigten Proteins (Bestätigung der Proteinexpression und -erfassung; Abbildung 1). Um das Hintergrundsignal zu verringern und den Dynamikbereich des Experiments zu erhöhen, werden die Mikroarrays mit 1) Lambdaphosphatase behandelt, um Diephosphorylierung aus Ser/Thr/Tyr-Rückständen zu entfernen, dann mit 2) DNase, um die Chemie vor Ort zu vereinfachen und zu verringern Hintergrund (Abbildung 1).
Der nächste Schritt ist die Autophosphorylierungsreaktion, bei der Mikroarrays ohne ATP (negative control array, auch als dephosphorylierte Mikroarrays) mit Kinase-Puffer inkubiert werden und der Kinase-Puffer durch ATP (Positive Control, autophosphorylierte Arrays) oder ATP + DMSO (Fahrzeugsteuerung). Es sollte betont werden, dass in diesem Schritt keine Kinase hinzugefügt wird; Daher wird die intrinsische Aktivität jeder Kinase, die auf dem Mikroarray angezeigt wird, durch Messung ihrer Phosphorylierungswerte mit einem Pan-Anti-Phospho-Tyrosin-Antikörper, gefolgt von einem cy3-labbeled sekundären Antikörper , quantifiziert (Abbildung 1).
Die Qualitätskontrolle der NAPPA-Kinase-Arrays, die ein Panel mit menschlichen Proteinkinasen zeigen, die in Quadruplikat gedruckt sind, ist in Abbildung 2dargestellt. Die Konzentrationen der immobilisierten DNA wurden durch DNA-Färbung gemessen und es zeigte ein gleichmäßiges Signal über das Mikroarray, was darauf hindeutet, dass die Menge der auf dem Array gedruckten DNA einheitlich war. Es ist auch möglich, mehrere Merkmale ohne DNA-Färbung zu beobachten. Diese Merkmale entsprechen einigen Kontrollen, bei denen DNA aus dem Druckmix weggelassen wurde [d.h. leere Flecken (nichts wurde gedruckt), Wasserflecken, gereinigter IgG-Spot (Polylysin, Verlinker und gereinigtes IgG), nur Druckmischung (vollständiger Druckmix: Polylysin plus Verlinker und Anti-Flag-Antikörper, ohne DNA)]. Die auf den NAPPA-Kinase-Mikroarrays angezeigten Proteinmengen wurden nach der IVTT-Reaktion mit Anti-Tag-Antikörpern bewertet.
Für das Kinase-Screening wurde Flag als Tag der Wahl verwendet und der auf dem Mikroarray angezeigte Proteingehalt wurde mit einem Anti-Flag-Antikörper gemessen. Wie gezeigt, zeigte die Mehrheit der Flecken, die cDNA enthalten, erfolgreich nachweisbare Proteinmengen an. Einige der Kontrollpunkte ohne cDNA zeigten auch Signal mit dem Anti-Flag-Antikörper: IgG-Spot (verwendet, um die Aktivität des sekundären Antikörpers zu erkennen) und leere Vektorflecken (cDNA-Codes nur für das Tag) (Abbildung 2). NAPPA-Kinase-Mikroarrays zeigten eine gute Reproduzierbarkeit unter Dias, wobei die Korrelation der Proteindarstellungsniveaus unter verschiedenen Druckchargen höher als 0,88 war (Abbildung 2). Innerhalb desselben Stapels war die Korrelation sogar noch höher, nahe 0,92 (Daten nicht dargestellt).
Als nächstes wurde die Kinase-Autophosphorylierungsaktivität der auf dem Array angezeigten Proteine mit Anti-Phospho-Tyrosin-Antikörpern gemessen (Abbildung 3). Protein, das auf dem Array angezeigt wurde, zeigte einen hohen Gehalt an Proteinphosphorylierung nach Expression(Abbildung 3, links), die durch die intrinsische Kinaseaktivität des auf dem Array angezeigten Proteins oder durch aktive Kinasen im IVTT-Mix verursacht werden kann. Diese Phosphorylierung wurde vollständig mit Lambda-Phosphatase-Behandlung entfernt und diese Mikroarrays wurden für die Kinase-Assays verwendet. Nach der Dephosphorylierung zeigten Autophosphorylierungsreaktionen, die ohne ATP durchgeführt wurden, wie erwartet keine signifikanten Konzentrationen der Phosphorylierung, während Mikroarrays, die in Gegenwart von ATP mit Kinasepuffer inkubiert wurden, eine Proteinphosphorylierung bereits 15 min zeigten ( Abbildung 3). Für das Arzneimittelscreening wurde die Kinaseaktivität nach 60 min Der Autophosphorylierungsreaktion gemessen, um die Anzahl der getesteten Kiinasen zu maximieren.
Der Vergleich zwischen Mikroarrays, bei denen die Phosphorylierungswerte direkt nach der Proteinexpression gemessen wurden(Abbildung 3, links) und nach 60 min Autophosphorylierungsreaktion(Abbildung 3, rechts) zeigte: i) nur phosphorylierte Proteine nach der Expression, was darauf hindeutet, dass sie exogen durch Proteine auf dem IVTT-Mix phosphoryliert werden können, aber nicht autophosphoryliert werden können; ii) Protein, das erst nach der Autophosphorylierungsreaktion phosphoryliert wurde, was darauf hindeutet, dass diese Proteine nach der Proteinexpression nicht aktiv waren und Co-Faktoren im Kinasepuffer erforderten, um aktiv zu sein; oder iii) Protein phosphoryliert auf beiden Arrays, was darauf hindeutet, dass sie in beiden Einstellungen aktiv waren (Abbildung 3).
Als Beispiel für die Ergebnisse, die für das Screening von Tyrosinkinase-Inhibitoren auf NAPPA-Kinase-Arrays erzielt wurden, wurden drei Kinaseninhibitoren mit unterschiedlicher Selektivität über Proteinkinasen hinweg verwendet: Staurosporin, Imatinib und Ibrutinib. Für alle Screenings wurden dephosphorylierte NAPPA-Mikroarrays mit zunehmenden TKI-Konzentrationen (von 100 nM bis 10 uM) während der Autophosphorylierungsreaktion inkubiert. Der erste getestete TKI war Staurosporin, ein globaler Proteinkinase-Inhibitor, der eine starke Kinase-Hemmung auf dem Mikroarray über praktisch alle getesteten Kinasen zeigte11.
Als nächstes wurde Imatinib getestet, ein ABL- und C-Kit-Hemmer zur Behandlung von chronischer myelogener Leukämie und gastrointestinalen Stromaltumoren4,5,6,7. Bei NAPPA-Kinase-Arrays zeigte Imatinib eine signifikante Verringerung der Abl1- und BCR-Abl1-Aktivität, während andere Kinasen weitgehend unberührt blieben (Abbildung 4A). Die Datenquantifizierung für die Kinaseaktivität wurde mit dem dephosphorylierten Array normalisiert und als Prozentsatz des Positivkontrollmikroarrays (nur Fahrzeug) dargestellt. Die Daten für TNK2 (non-relevant kinase), Abl1 und BCR-Abl1 sind in Abbildung 4Bdargestellt. Wie erwartet zeigte Imatinib eine selektive Hemmung gegenüber Abl1 und BCR-ABl1. Die Daten für c-Kit waren aufgrund mangelnder Aktivität auf den Positivkontroll-Arrays nicht schlüssig.
Schließlich wurde Ibrutinib, ein von der FDA zugelassener kovalenter Inhibitor der Bruton-Tyrosinkinase (BTK), getestet. Ibrutinib wird derzeit bei der Behandlung mehrerer blutbedingter Krebsarten mit überaktiver BTK, einschließlich chronischer lymphatischer Leukämie (CLL), Mantelzelllymphom, und Waldenströms Makroglobulinämie21,22. Abbildung 4C, ist repräsentativ für die typischen Ergebnisse, die für das Ibrutinib-Screening erzielt wurden. Die Kinaseaktivität von ABL1 (nicht relevante Kinase) und BTK (kanonisches Ziel) und ERBB4 (potenzielles neues Ziel) ist in Abbildung 4Ddargestellt. Die Daten deuten darauf hin, dass ERBB4 durch Ibrutinib in einer dosisspezifischen Weise gehemmt werden kann. Diese Hemmung wurde in vitro und in zellbasierten Assays11bestätigt, was die Leistungsfähigkeit dieser Plattform demonstrierte.
Zusammengenommen deuten die Daten darauf hin, dass die NAPPA-Kinase-Mikroarray-Plattform für das unvoreingenommene Screening von TK-Inhibitoren verwendet werden könnte. Darüber hinaus ist das Screening schnell und kann leicht angepasst werden, um jede Variation der Protein-Kinasen von Interesse zu umfassen.
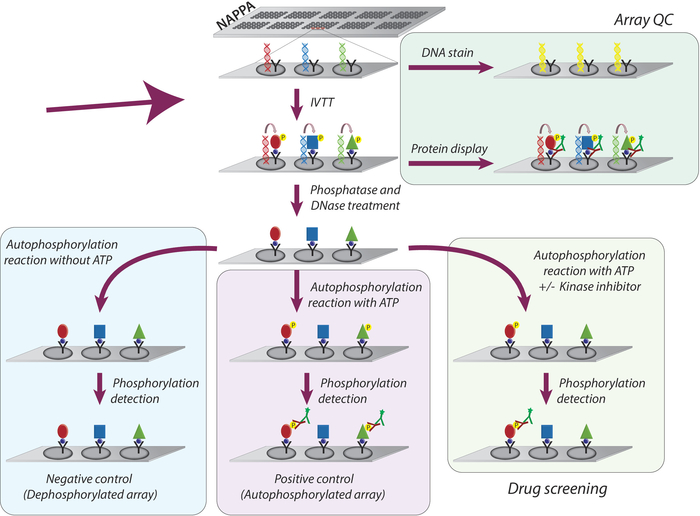
Abbildung 1: Schematische Darstellung der Qualitätskontrolle und Dessiebung von Tyrosinkinase-Inhibitoren in NAPPA-Arrays. NAPPA-Arrays werden mit cDNA-Codierung für das Protein von Interesse gedruckt, das mit einem Tag und einem Capture-Antikörper verschmolzen ist. Während der In-vitro-Transkription und Translationsreaktion (IVTT) werden die synthetisierten Proteine durch den Capture-Antikörper auf der Mikroarray-Oberfläche durch das Tag erfasst. Die Qualitätskontrolle (QC) der Arrays erfolgt durch die Messung der auf dem Dia aufgedruckten DNA-Werte unter Verwendung eines fluoreszierenden DNA-interkalierenden Farbstoffs und der auf dem Array angezeigten Proteinmengen mit tagspezifischen Antikörpern. Für das Kinase-Screening werden die Mikroarrays nach der IVTT-Reaktion mit DNase und Phosphatase behandelt, um die gedruckte DNA und alle Phosphorylierungen zu entfernen, die während der Proteinsynthese aufgetreten sein können. Die dephosphorylierten Arrays sind nun bereit, für den Drogen-Bildschirm verwendet werden. Für jeden Test werden routinemäßig drei Sätze von Kontrollen verwendet: (I) dephosphorylierte Arrays, bei denen die Autophosphorylierungsreaktion ohne ATP durchgeführt wird; (II) autophosphorylierte Mikroarrays, bei denen die Autophosphorylierungsreaktion in Gegenwart von ATP durchgeführt wird; und (III) DMSO-behandeltes Array (Fahrzeug), in dem die Autophosphorylierungsreaktion mit ATP und DMSO durchgeführt wird. Die mit unterschiedlicher Konzentration von Kinase-Inhibitoren behandelten Dias folgen genau dem gleichen Protokoll, das für die DMSO-behandelten Arrays verwendet wird. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
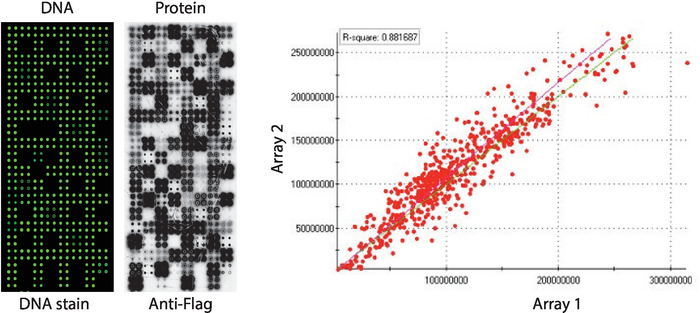
Abbildung 2: Repräsentative Ergebnisse der Qualitätskontrolle für selbstmontierte NAPPA-Kinase-Arrays. Gezeigt werden DNA-Gehalt, gemessen durch einen fluoreszierenden DNA-interkalierenden Farbstoff (links) und Proteingehalte, die auf dem Mikroarray gemessen werden, gemessen durch Anti-Flag-Antikörper (Mitte). Auf der rechten Seite befindet sich ein Korrelationsdiagramm der Proteinspiegel, die auf zwei NAPPA-Kinase-Arrays in separaten Chargen gedruckt sind. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
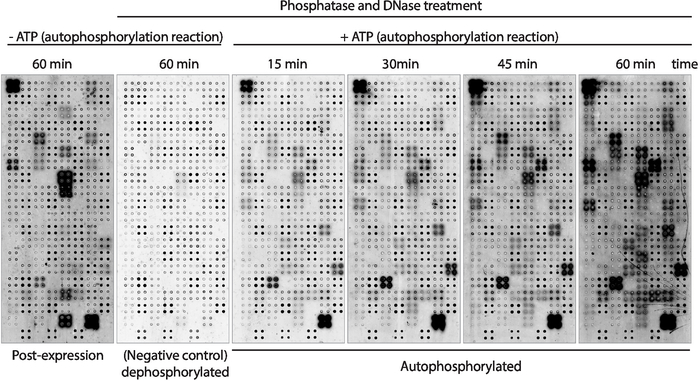
Abbildung 3: Repräsentative Ergebnisse der Kinase-Aktivität in NAPPA-Kinase-Arrays. Mikroarrays, die Proteinkinasen in Quadruplicate zeigen, wurden verwendet, um die Proteinkinase-Aktivität auf dem Array durch die Messung der Proteinphosphorylierung mit Anti-PTyr-Antikörpern zu untersuchen, gefolgt von cy3-markiertem Anti-Maus-Antikörper. Kontrollarrays ohne Phosphatase/DNase-Behandlung und ohne ATP während der Autophosphorylierungsreaktion wurden verwendet, um die Hintergrundphosphorylierung nach Proteinexpression (Postexpression) zu messen. Die übrigen Mikroarrays wurden mit Phosphatase/DNA behandelt, und die Autophosphorylierungsreaktion wurde ohne ATP (dephosphoryliertes Mikroarray, Negativkontrolle) oder mit ATP (autophosphorylierte Mikroarrays) durchgeführt. Bei den autophosphorylierten Mikroarrays wurde die Autophosphorylierungsreaktion für 15 min, 30 min, 45 min oder 60 min durchgeführt, wie gezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
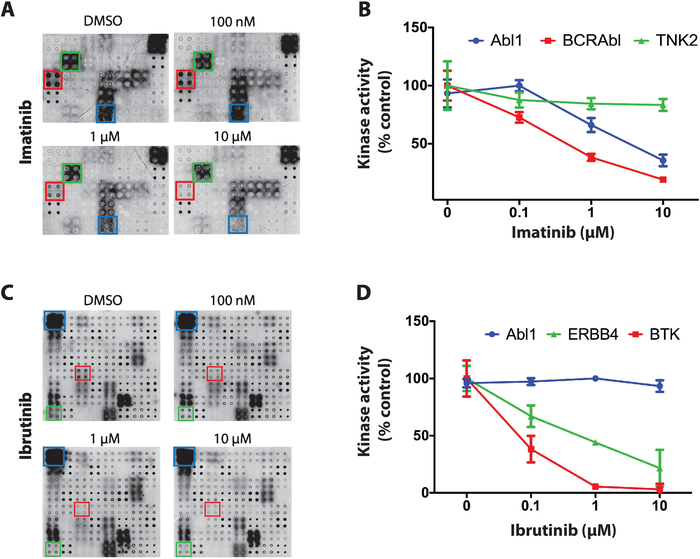
Abbildung 4: Repräsentative Daten vom Tyrosinkinase-Bildschirm auf NAPPA-Kinase-Arrays. (A) Phosphatase/DNase behandelte NAPPA-Kinase-Arrays wurden während der Autophosphorylierungsreaktion in zunehmenden Konzentrationen von Imatinib inkubiert und die Kinaseaktivität mit Anti-Phospho-Tyr-Antikörpern gemessen. (B) Quantifizierung der Kinase-Aktivität, die an NAPPA-Kinase-Arrays beobachtet wurde, die Imatinib ausgesetzt sind. Die Daten wurden gegen das Signal der Negativkontroll-Arrays (dephosphoryliert) normalisiert und als Prozentsatz der positiven Kontrollarrays (Autophosporylierungsreaktion in Gegenwart von DMSO) angezeigt. Ähnliche Daten werden für das Screening von Ibrutinib (C,D) angezeigt. Diese Zahl wurde von Rauf et al.11geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Datei 1. Alternatives Protokoll zum Screening von Tyrosinkinase-Inhibitoren in NAPPA-Arrays mit einer automatisierten Hybridisierungsstation. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Änderungen und Fehlerbehebung
Während der Optimierungsphase der Untersuchung der Kinase-Aktivität auf NAPPA-Arrays war eine der Hauptquellen für Hintergrund und schwacher Dynamikbereich die BSA, die auf dem Druckmix verwendet wurde. BSA lieferte die primären Adern, die für die Vernetzung mit der Aminosilanoberfläche notwendig sind, und die DNA und den Capture-Antikörper an Ort und Stelle ein. BSA ist jedoch stark phosphoryliert, was die Erkennung des Autophosphorylierungssignals auf dem Array über dem Hintergrundrauschen erschwert. Um dieses Problem zu lösen, wurden mehrere Alternativen für BSA im Druckmix getestet, und Polylysin wurde als guter Ersatz identifiziert. Polylysin fehlt jede Phosphorylierung Seisplatz; Daher ist der Hintergrund von nicht ausgedrückten Arrays sehr minimal. Darüber hinaus sind Mikroarrays, die mit Polylysin bedruckt sind, reproduzierbar und weisen gute Proteine auf (Abbildung 2).
Die nächste kritische Änderung, die am Standard-NAPPA-Test durchgeführt wurde, war die Zugabe eines Phosphatase/DNase-Behandlungsschritts. Die Behandlung der Mikroarrays mit Phosphatase ermöglicht die Entfernung jeglicher Phosphorylierung, die im IVTT-Mix während der Proteinsynthese und -erfassung aufgetreten ist (Abbildung 3). Die Quelle dieser Phosphorylierung könnte aus der intrinsischen Autophosphorylierungsaktivität oder aus der Aktivität von Kiinasen stammen, die im IVTT-Mix vorhanden sind. Die Entfernung aller Phosphorylierung nach der Expression ermöglichte eine einfache Identifizierung der Kiinasen, die aktiv sind und einer Autophosphorylierung unterzogen werden können (Abbildung 3).
Kritische Schritte innerhalb des Protokolls
NAPPA ist eine robuste Technologie, aber wie erwartet gibt es mehrere kritische Schritte. Die erste ist der Erwerb hochwertiger DNA in der entsprechenden Konzentration. Die Verwendung von DNA von schlechter Qualität oder in niedrigen Konzentrationen erzeugt Mikroarrays von schlechter Qualität, wobei mehrere Merkmale nicht ausgedrückt und in den entsprechenden Ebenen angezeigt werden, wodurch die Anzahl der auf dem Array analysierten Proteine verringert wird. Der zweite kritische Schritt ist die Expression von Proteinen auf dem Mikroarray. Die Verwendung eines IVTT-Systems, das ein hohes Maß an funktionellem Protein ausdrückt, ist entscheidend für die Untersuchung der Kinase-Aktivität auf dem Array.
Der nächste wichtige Schritt beim TKI-Screening ist der Umgang mit den Mikroarrays. Die Mikroarrays sollten während eines Schritts des Protokolls nicht trocknen, und eine schonende Handhabung wird empfohlen, um Kratzer zu vermeiden, die das Hintergrundsignal erhöhen können. Da die Arrays aus dem gesamten Experiment miteinander verglichen werden, ist es wichtig sicherzustellen, dass jeder Inkubationsschritt gleichmäßig über alle Folien hinweg erfolgt. Beispielsweise sollte die Zeit berücksichtigt werden, die zum Ausführen eines Schritts in einem einzelnen Array erforderlich ist, wenn ein Batch von 20 Arrays verarbeitet wird, um Unterschiede in der Dauer der Inkubation zwischen Arrays zu vermeiden.
Schließlich sind die Konzeption des Experiments und die Einbeziehung von positiven und negativen Kontrollen für die Qualitätskontrolle und Datenanalyse von entscheidender Bedeutung. Der erste Satz von Steuerelementen sind die in jedem Array gedruckten und enthalten Negativkontrollen [d. h. leere Flecken (ohne Material gedruckt), Wasser oder leeren Vektor (nur das Tag ausdrücken)] sowie eine positivgesteuerte (d. h. gereinigte IgG, die vom sekundären Antikörper und ist inert gegenüber Veränderungen der Phosphorylierungsniveaus). Zusammen messen sie die Hintergrundpegel des Mikroarrays, die mögliche Übertragung während des Drucks und die Signalintensität der Detektionsmethode.
Die nächsten Kontrollen sind die Arzneimittel-Screening-Kontrollen und umfassen die dephosphorylierten und autophosphorylierten Mikroarrays (in Gegenwart oder Abwesenheit von DMSO). Wie bereits erwähnt, misst das dephosphorylierte Mikroarray den Phosphorylierungsgrad nach der Phosphatase-Behandlung und damit den Ausgangswert für alle anderen Experimente. Je niedriger die Basisebene ist, desto höher ist der Dynamikbereich der Assays. Die autophosphorylierten Arrays stellen die maximalen Phosphorylierungswerte aller Arrays dar und das Signal sollte stark und klar sein. Es wird für die Datenanalyse verwendet, aber auch als Kontrolle, dass alle Reaktionen erfolgreich auf dem Array durchgeführt wurden.
Einschränkungen der Technik
Ab sofort ist eine der Einschränkungen des hier vorgestellten Arzneimittelscreenings seine Fähigkeit, nur Proteinkiinasen zu überprüfen, die autophosphoryliert werden können. Eine möglichkeit, dies zu überwinden, ist, eine Kinase und ein bekanntes Substrat an der gleichen Stelle zu drucken. Der Co-Druck von DNA für zwei verschiedene Proteine wurde erfolgreich15erfolgreich durchgeführt, was auf die Machbarkeit dieses Ansatzes hindeutet. Darüber hinaus wird das auf dem Array angezeigte Protein möglicherweise nicht richtig gefaltet, was zu einem inaktiven Protein führt. Der Einsatz des humanbasierten Expressionssystems hat die kinase-Aktivität, gemessen auf dem Array, erheblich verbessert; Einige Proteine können jedoch aufgrund ihrer Inaktivität immer noch nicht auf dem Array analysiert werden.
Eine zweite Einschränkung ist die Messung der Phosphorylierung mit einem Pan-Anti-Phospho-Tyr-Antikörper. Trotz seiner Unspezifität bezüglich des Motivs der Phosphorylierungsstelle traten alle gemessenen Phosphorylierungen auf Tyrosinrückständen auf und hinterließen Serinen und Threonine und deren jeweilige Kiinasen. Bis heute wurden mehr als 10 Pan-Phospho-Ser/Thr-Antikörper trotz mehrerer Versuche zur Optimierung der Inkubations- und Waschbedingungen erfolglos getestet. Ein neues Detektionssystem, das unabhängig von Antikörpern ist, kann die beste Option sein, um die Anzahl der Proteinkiinasen zu erweitern, die auf Arzneimittelhemmung untersucht werden können. In diesem Zusammenhang stehen einige Optionen zur Verfügung, darunter Radioaktivität oder chemische Ansätze wie Klickkonjugation. Eine Reihe von Optimierungen sind erforderlich, um das Hintergrundsignal zu minimieren und einen guten Dynamikbereich für die Assays zu bieten.
Die dritte Einschränkung ist die Erfassung von cDNA-Klonen, die auf dem Array gedruckt werden sollen. Die cDNA-Klone können mit jeder Klontechnik generiert werden, einschließlich standortspezifischer Rekombinationssysteme, wie Creator oder Gateway23. Eine weitere Möglichkeit besteht darin, die Klone aus der DNAsu-Bibliothek zu erwerben, die unter gefunden wird, wo mehr als 17.000 cDNAs-Klone, einschließlich des gesamten menschlichen Kinom, für den Bau von NAPPA-Arrays 24 leicht verfügbar sind. .
Die vierte Einschränkung ist, dass nicht jedes Labor mit geeigneten Geräten ausgestattet ist, um ihre eigenen NAPPA-Arrays herzustellen und zu prüfen. Dieses Protokoll bietet alternative Methoden zur Erzeugung der DNA, die auf das Mikroarray gedruckt werden soll, ohne dass Geräte mit hohem Durchsatz benötigt werden, sowie Protokolle, um alle Hybridisierungsschritte manuell auszuführen. Der Zugriff auf einen Arrayer und Einen Microarray-Scanner ist jedoch weiterhin erforderlich. Eine Möglichkeit, dieses Problem zu beheben, ist die Nutzung des NAPPA-Kerndienstes und der NAPPA-Einrichtung, die unter zu finden ist und maßgeschneiderte NAPPA-Mikroarrays zu einem gemeinnützigen akademischen Preis vertreibt. Schließlich sind die auf den Arrays gewonnenen Daten ab jeder Screening-Methodik anfällig für Artefakte (entweder positive oder negative) und sollten daher mit orthogonalen Assays validiert werden.
Bedeutung für bestehende Methoden
Für das Screening von Proteinkiinasen stehen mehrere Plattformen im Handel zur Verfügung. Ein routinemäßig verwendeter Ansatz sind Bindungstests, die mit Proteinfragmenten, Kinase-Domäne, größeren Proteinfragmenten mit der Kinase-Domäne und einigen regulatorischen Regionen und sogar Voll-Längen-Proteinen durchgeführt werden können. Die Proteine werden in der Regel in bakteriellen Systemen aufgrund der Kosten und Einfachheit in der Expression und Reinigung Protokolle ausgedrückt. Die Wechselwirkung zwischen dem Medikament von Interesse und dem Protein wird dann mit einer Art von Berichtstest wie Fluoreszenz oder Vorhandensein von Tags gemessen, zum Beispiel. Die Hauptbeschränkung dieser Reihe von Ansätzen ist die Tatsache, dass das Protein während der Interaktion mit dem Medikament nicht unbedingt aktiv ist, was zur Identifizierung falsch positiver und falsch negativer Wechselwirkungen führen kann. Proteinfragmente sind besonders anfällig für Veränderungen der Konformation und mangelnde Aktivität, und alle erhaltenen Daten sollten mit aktiven Proteinen validiert werden, vorzugsweise in ihrer vollen Form. Eine weitere Einschränkung einiger Plattformen ist die Möglichkeit, nur ATP-Analoga zu überprüfen, wodurch die Gesamtnutzung eingeschränkt wird.
Die meisten kommerziell verfügbaren Dienstleistungen für das Screening von TKIs mit enzymatischen Ansätzen verwenden nur wilde Typversionen der Kinase von Interesse, und manchmal nur ein paar ausgewählte Mutanten. Da sie wissen, dass Arzneimittelresistenz bei Patienten, die mit TKI behandelt werden, sehr häufig ist, ist es wichtig, die Arzneimittelreaktion bei verschiedenen Mutanten messen zu können, um den am besten geeigneten Inhibitor zu ersinnen. Aufgrund der Natur von NAPPA ist das Screening von Kinase-Mutanten einfach und kann leicht durchgeführt werden, und das einzige erforderliche Werkzeug ist die Einbeziehung der Kinase-Mutante in die NAPPA cDNA-Sammlung, die beispielsweise durch ortsspezifische Mutagenese durchgeführt werden kann.
Zukünftige Anwendungen
Eine der häufigsten Behandlungsformen in der Krebstherapie mit Kinase-Inhibitoren ist die Erfassung von Mutationen im Wirkstoffziel während eines Behandlungskurses. Das Screening dieser Mutanten auf ihre Reaktion auf Kinase-Inhibitoren ist von entscheidender Bedeutung für die Auswahl der zweiten/dritten Generation von TKIs, um eine personalisierte Behandlung für jeden Patienten zu erreichen. Der hier vorgestellte Arzneimittel-Screening-Ansatz bietet eine unvoreingenommene Screening-Plattform, auf der jeder Tyrosinkinase-Inhibitor gegen eine Gruppe von Tyrosin-Kiinasen im menschlichen Genom getestet werden kann. Da die auf NAPPA-Arrays angezeigten Proteine in vitro aus der auf dem Dia gedruckten cDNA exprimiert werden, kann jede mutierte Variante problemlos in die cDNA-Sammlung integriert werden, die auf dem Array angezeigt werden soll. Die Anlage, in der die Kinase-Mutanten auf dem Array erzeugt und ausgedrückt werden können, in Kombination mit der hohen Durchsatzleistung der NAPPA-Technik, bietet eine einzigartige Umgebung für die Untersuchung von Kinase-Mutanten und deren Reaktion auf Medikamente, wodurch NAPPA für personalisiertes Arzneimittelscreening, eines der Ziele der Präzisionsmedizin.
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
Die Autoren bedanken sich bei allen im LaBaer-Labor für ihre Hilfe und Kritik bei der Entwicklung des Projekts. Dieses Projekt wurde durch das NIH-Stipendium U01CA117374, U01AI077883 und Virginia G. Piper Foundation unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| 364 well plates (for arraying) | Genetix | x7020 | |
| 800 µL 96-well collection plate | Abgene | AB-0859 | |
| 96-pin device | Boekel | 140500 | |
| Acetic Acid | Millipore-Sigma | 1.00066 | |
| Acetone 99.9% | Millipore Sigma | 650501 | |
| Aluminum seal for 96 well plates | VWR | 76004-236 | |
| Aminosilane (3-aminopropyltriethoxysilane) | Pierce | 80370 | |
| ANTI-FLAG M2 antibody produced in mouse | Millipore Sigma | F3165 | |
| Anti-Flag rabbit Antibody (polyclonal) | Millipore Sigma | F7425 | |
| ATP 10 mM | Cell Signaling | 9804S | |
| β-Glycerophosphate disodium salt hydrate | Millipore-Sigma | G9422 | |
| bacteriological agar | VWR | 97064-336 | |
| Blocking Buffer | ThermoFisher/Pierce | 37535 | |
| Brij 35 | ThermoFisher/Pierce | BP345-500 | |
| BS3 (bis-sulfosuccinimidyl) | ThermoFisher/Pierce | 21580 | |
| BSA (bovine serum albumin) | Millipore Sigma | A2153 | |
| Coverslip 24 x 60 mm | VWR | 48393-106 | |
| Cy3 AffiniPure Donkey Anti-Mouse IgG (H+L) | Jackson ImmunoResearch | 715-165-150 | |
| DeepWell Block, case of 50 | ThermoFisher/AbGene | AB-0661 | |
| DEPC water | Ambion | 9906 | |
| DMSO (Dimethyl Sulfoxide) | Millipore-Sigma | D8418 | |
| DNA-intercalating dye | Invitrogen | P11495 | |
| DNase I | Millipore-Sigma | AMPD1-1KT | |
| DTT | Millipore-Sigma | 43816 | |
| EDTA | Millipore-Sigma | EDS | |
| Ethanol 200 proof | Millipore-Sigma | E7023 | |
| Filter plates | Millipore-Sigma | WHA77002804 | |
| Gas Permeable Seals, box of 50 | ThermoFisher/AbGene | AB-0718 | |
| Glass box | Wheaton | 900201 | |
| Glass slides | VWR | 48300-047 | |
| Glycerol | Millipore-Sigma | G5516 | |
| HCl (Hydrochloric acid) | Millipore-Sigma | H1758 | |
| HEPES Buffer Solution | Millipore-Sigma | 83264 | |
| Human-based IVTT system | Thermo Scientific | 88882 | |
| ImmunoPure Mouse IgG whole molecule | ThermoFisher/Pierce | 31202 | |
| Isopropanol | Millipore-Sigma | I9516 | |
| KCl (Potassium chloride) | Millipore-Sigma | P9333 | |
| KH2PO4(Potassium phosphate monobasic) | Millipore-Sigma | P5655 | |
| Kinase buffer | Cell Signaling | 9802 | |
| KOAc (Potassium acetate) | Millipore-Sigma | P1190 | |
| Lambda Protein Phosphatase | new england biolabs | P0753 | |
| Lifterslips, 24 x 60 mm | ThermoFisher Scientific | 25X60I24789001LS | |
| Metal 30-slide rack with no handles | Wheaton | 900234 | |
| MgCL2 (Magnesium chloride) | Millipore-Sigma | M8266 | |
| Na3VO4 (Sodium orthovanadate) | Millipore-Sigma | S6508 | |
| NaCl (Sodium Chloride) | Millipore-Sigma | S3014 | |
| NaOAc (Sodium acetate) | Millipore-Sigma | S2889 | |
| NaOH (Sodium hydroxide) | Millipore-Sigma | S8045 | |
| NucleoBond Xtra Midi / Maxi | Macherey-Nagel | 740410.10 / 740414.10 | |
| Nucleoprep Anion II | Macherey Nagel | 740503.1 | |
| Phosphoric Acid | Millipore-Sigma | 79617 | |
| Poly-L-Lysine Solution (0.01%) | Millipore-Sigma | A-005-C | |
| Protein Phosphatase (Lambda) | New England Biolabs | P0753 | |
| RNAse | Invitrogen | 12091021 | |
| SDS (Sodium dodecyl sulfate) | Millipore-Sigma | L6026 | |
| SDS (Sodium dodecyl sulfate) | Millipore-Sigma | 05030 | |
| Sealing gasket | Grace Bio-Labs, Inc | 44904 | |
| Silica packets | VWR | 100489-246 | |
| Single well plate | ThermoFisher/Nalge Nunc | 242811 | |
| Sodium acetate (3M, pH 5.5) | Millipore-Sigma | 71196 | |
| TB media (Terrific Broth) | Millipore-Sigma | T0918 | |
| Tris | IBI scientific | IB70144 | |
| Triton X-100 | Millipore-Sigma | T8787 | |
| Tryptone | Millipore-Sigma | T7293 | |
| Tween 20 | Millipore-Sigma | P9416 | |
| Yeast Extract | Millipore-Sigma | Y1625 | |
| Name | Company | Catalog number | Comments |
| Equipments | Maker/model | ||
| Programmable chilling incubator | Torrey Pines IN30 Incubator with Cooling | ||
| Shaker for bacterial growth | ATR Multitron shaker | ||
| Vacuum manifold with liquid waste trap | MultiScreenVacuum Manifold 96 well | ||
| 96 well autopippetor/liquid handler | Genmate or Biomek FX | ||
| Liquid dispenser | Wellmate | ||
| DNA microarrayer | Genetix QArray2 | ||
| Automatic hybridization station | Tecan HS4800 Pro Hybridization Station | ||
| Microarray scanner | Tecan PowerScanner | ||
| Microarray data quantification | Tecan Array-ProAnalyzer 6.3 |
Referenzen
- Melnikova, I., Golden, J. Targeting protein kinases. Nature Review Drug Discovery. 3 (12), 993-994 (2004).
- Patterson, H., Nibbs, R., McInnes, I., Siebert, S. Protein kinase inhibitors in the treatment of inflammatory and autoimmune diseases. Clinical and Experimental Immunology. 176 (1), 1-10 (2014).
- Wu, P., Nielsen, T. E., Clausen, M. H. FDA-approved small-molecule kinase inhibitors. Trends Pharmacological Sciencies. 36 (7), 422-439 (2015).
- Druker, B. J., et al. Effects of a selective inhibitor of the Abl tyrosine kinase on the growth of Bcr-Abl positive cells. Nature Medicine. 2 (5), 561-566 (1996).
- Heinrich, M. C., et al. Inhibition of c-kit receptor tyrosine kinase activity by STI 571, a selective tyrosine kinase inhibitor. Blood. 96 (3), 925-932 (2000).
- Stagno, F., et al. Imatinib mesylate in chronic myeloid leukemia: frontline treatment and long-term outcomes. Expert Review Anticancer Therapy. 16 (3), 273-278 (2016).
- Ben Ami, E., Demetri, G. D. A safety evaluation of imatinib mesylate in the treatment of gastrointestinal stromal tumor. Expert Opinions in Drug Safety. 15 (4), 571-578 (2016).
- Ramachandran, N., et al. Self-assembling protein microarrays. Science. 305 (5680), 86-90 (2004).
- Festa, F., et al. Robust microarray production of freshly expressed proteins in a human milieu. Proteomics Clinical Applications. 7 (5-6), 372-377 (2013).
- Yazaki, J., et al. Mapping transcription factor interactome networks using HaloTag protein arrays. Proceedings of the National Academy of Sciences of the United States of America. 113 (29), E4238-E4247 (2016).
- Rauf, F., et al. Ibrutinib inhibition of ERBB4 reduces cell growth in a WNT5A-dependent manner. Oncogene. 37 (17), 2237-2250 (2018).
- Anderson, K. S., et al. Protein microarray signature of autoantibody biomarkers for the early detection of breast cancer. Journal of Proteome Research. 10 (1), 85-96 (2011).
- Wang, J., et al. Plasma Autoantibodies Associated with Basal-like Breast Cancers. Cancer Epidemiol Biomarkers Prevention. 24 (9), 1332-1340 (2015).
- Bian, X., et al. Tracking the Antibody Immunome in Type 1 Diabetes Using Protein Arrays. Journal of Proteome Research. 16 (1), 195-203 (2017).
- Song, L., et al. Identification of Antibody Targets for Tuberculosis Serology using High-Density Nucleic Acid Programmable Protein Arrays. Molecular and Cellular Proteomics. 16 (4 suppl 1), S277-S289 (2017).
- Wang, J., et al. Comparative Study of Autoantibody Responses between Lung Adenocarcinoma and Benign Pulmonary Nodules. Journal of Thoracic Oncology. 11 (3), 334-345 (2016).
- Montor, W. R., et al. Genome-wide study of Pseudomonas aeruginosa outer membrane protein immunogenicity using self-assembling protein microarrays. Infection and Immunity. 77 (11), 4877-4886 (2009).
- Tang, Y., Qiu, J., Machner, M., LaBaer, J. Discovering Protein-Protein Interactions Using Nucleic Acid Programmable Protein Arrays. Current Protocols in Cell Biology. 74, 11-15 (2017).
- Yu, X., et al. Copper-catalyzed azide-alkyne cycloaddition (click chemistry)-based detection of global pathogen-host AMPylation on self-assembled human protein microarrays. Molecular and Cellular Proteomics. 13 (11), 3164-3176 (2014).
- Anderson, K. S., et al. Autoantibody signature for the serologic detection of ovarian cancer. Journal of Proteome Research. 14 (1), 578-586 (2015).
- Woyach, J. A., Johnson, A. J., Byrd, J. C. The B-cell receptor signaling pathway as a therapeutic target in CLL. Blood. 120 (6), 1175-1184 (2012).
- Smith, M. R. Ibrutinib in B lymphoid malignancies. Expert Opinion on Pharmacotherapy. 16 (12), 1879-1887 (2015).
- Festa, F., Steel, J., Bian, X., Labaer, J. High-throughput cloning and expression library creation for functional proteomics. Proteomics. 13 (9), 1381-1399 (2013).
- Seiler, C. Y., et al. DNASU plasmid and PSI:Biology-Materials repositories: resources to accelerate biological research. Nucleic Acids Research. 42, D1253-D1260 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten