Method Article
Screening dell'inibitore della kinasi in microarray proteici umane auto-assemblati
In questo articolo
Riepilogo
Viene presentato un protocollo dettagliato per la generazione di microarray proteici umani auto-assemblati per lo screening degli inibitori della chinasi.
Abstract
Lo screening degli inibitori della chinasi è fondamentale per comprendere meglio le proprietà di un farmaco e per l'identificazione di potenziali nuovi obiettivi con implicazioni cliniche. Diverse metodologie sono state segnalate per realizzare tale screening. Tuttavia, ciascuno ha i suoi limiti (ad esempio, lo screening di solo analoghi ATP, la restrizione all'uso di domini di chinasi purificati, costi significativi associati al test più di un paio di chinasi alla volta e la mancanza di flessibilità nello screening delle parentesi proteiche con nuove mutazioni). Qui, viene presentato un nuovo protocollo che supera alcuni di questi limiti e può essere utilizzato per lo screening imparziale degli inibitori della chinasi. Un punto di forza di questo metodo è la sua capacità di confrontare l'attività degli inibitori della chinasi su più proteine, tra diverse chinasi o varianti diverse della stessa chinasi. Vengono impiegati microarray proteici autoassemblati generati attraverso l'espressione di chinasi proteiche da un sistema umano di trascrizione e traduzione in vitro (IVTT). Le proteine visualizzate sul microarray sono attive, consentendo la misurazione degli effetti degli inibitori della chinasi. La procedura seguente descrive in dettaglio i passaggi del protocollo, dalla generazione e lo screening di microarray all'analisi dei dati.
Introduzione
Le chinasi proteiche sono responsabili della fosforilazione dei loro bersagli e possono modulare vie molecolari complesse che controllano molte funzioni cellulari (cioè la proliferazione cellulare, la differenziazione, la morte cellulare e la sopravvivenza). La deregolazione dell'attività della chinasi è associata a più di 400 malattie, rendendo gli inibitori della chinasi una delle principali classi di farmaci disponibili per il trattamento di diverse malattie, tra cui il cancro, i disturbi cardiovascolari e neurologici, nonché i disturbi infiammatori e malattie autoimmuni1,2,3.
Con l'avvento della medicina di precisione, l'identificazione di nuove terapie, in particolare gli inibitori della chinasi, ha un grande fascino farmaceutico e clinico. Diversi approcci possono essere utilizzati per l'identificazione di possibili nuove coppie di inibitori della chinasi/ellissi, tra cui la progettazione de novo di inibitori della chinasi e l'identificazione di nuovi bersagli per i farmaci esistenti approvati dalla FDA. Quest'ultimo è particolarmente attraente, dal momento che il tempo e il denaro necessari per implementare questi farmaci nelle cliniche sono drasticamente ridotti a causa della disponibilità di precedenti dati di sperimentazione clinica. Un esempio canonico del riutilizzo di un inibitore della chinasi è l'imatinib, inizialmente progettato per il trattamento della leucemia mieloziana cronica (CML) attraverso l'inibizione di BCR-Abl, che può anche essere utilizzata con successo per il trattamento di tumori stromali gastrointestinali (GIIST)4,5,6,7.
Lo screening degli inibitori della chinasi può essere eseguito in saggi vincolanti o saggi enzimatici. La prima classe di analisi si concentra sulle interazioni proteina-farmaco e può fornire informazioni come il sito di ligazione e l'affinità. Poiché l'attività della chinasi al momento di questi saggi è sconosciuta, una serie di interazioni può essere persa o erroneamente identificata a causa di cambiamenti conformazionali nella proteina. D'altra parte, i saggi a base enzimatica richiedono che le chinasi proteiche siano attive e forniscano informazioni preziose sull'effetto dell'inibitore sull'attività enzimatica, tuttavia, questo tipo di screening è generalmente più dispendioso in termini di tempo e denaro. Attualmente, entrambi i tipi di saggi sono disponibili in commercio da diverse fonti. Essi rappresentano un'opzione affidabile per lo screening degli inibitori della chinasi con alcune limitazioni, tra cui: I) la maggior parte dei metodi comporta la sperimentazione di più chinasi individualmente, che può rendere costoso lo screening di un ampio insieme di proteine; II) l'insieme delle chinasi da testare è limitato a un elenco di chinasi preselezionate e wild-type e diverse versioni mutate ben note di alcune chinasi, ostacolando il test di molte nuove isoforme mutate.
In questo contesto, i microarray proteici sono una potente piattaforma in grado di superare alcune delle limitazioni presentate dalle tecniche disponibili in commercio. È adatto per eseguire saggi a base enzimatica nello screening ad alto rendimento utilizzando proteine attive a lunghezza intera di qualsiasi sequenza di interesse. I microarray possono essere generati da un approccio auto-assemblato come NAPPA (nucleic acid programmable protein array), in cui le proteine sono espresse appena in tempo per i test, aumentando la probabilità che quelli visualizzati sulla matrice siano effettivamente attivi. Le proteine visualizzate su NAPPA sono prodotte utilizzando ribosomi derivati dall'uomo e proteine chaperone al fine di migliorare la probabilità di piegamento naturale e attività.
Le proteine sono inizialmente programmate stampando cDNA codificando per geni di interesse fusi con un tag di cattura, insieme a un agente di cattura, sulla superficie del microarray. Le proteine vengono quindi prodotte sui microarray utilizzando un sistema di trascrizione e traduzione in vitro (IVTT) e le proteine appena espresse vengono immobilizzate sulla superficie del microarray dall'agente di cattura. Gli array NAPPA espressi possono essere utilizzati per lo studio delle proteine visualizzate sull'array in modo imparziale e ad alto contenuto di velocitàeffettiva 8,9.
In precedenza, è stato dimostrato che le proteine visualizzate sugli array NAPPA sono piegate correttamente per interagire con partner noti10; inoltre, la loro attività enzimatica è stata sfruttata per la prima volta nel 2018, quando è stato dimostrato che le chinasi proteiche visualizzate sul microarray autofosforetale11. Ad oggi, la metodologia NAPPA è stata utilizzata per molte applicazioni distinte, tra cui la scoperta di biomarcatori12,13,14,15,16,17, interazioni proteina-proteina10,18, identificazione del substrato19e screening farmacologico11. La sua flessibilità è una delle caratteristiche chiave della piattaforma che consente l'adattamento a ogni applicazione.
Qui, viene presentato un protocollo per lo screening degli inibitori della tirosina della chinasi in array NAPPA auto-assemblati. La piattaforma è ottimizzata per la visualizzazione delle chinasi delle proteine umane attive e per l'analisi dell'attività della chinasi proteica, con basso background e alta gamma dinamica. Tra le modifiche implementate per utilizzare NAPPA per lo screening degli inibitori della chinasi includono: I) cambiamenti nella chimica della stampa, II) de-phophorylation del microarray proteico prima dello screening dell'inibitore della chinasi, e III) ottimizzazione del rilevamento del proteine fosforolate sulla matrice. Questo protocollo è il primo nel suo genere e fornisce informazioni uniche sullo studio della chinasi nei microarray NAPPA.
Protocollo
1. Buffer comuni e soluzioni da utilizzare
- Preparare il TB medio: terrificante brodo (24 g/L lievito; 20 g/L tripptone; 4 mL/L glicerol; 0.017 M KH2PO4; e 0.072 M K2HPO4). Le soluzioni 0.017 M KH2PO4 e 0.072 M K2HPO4 possono essere acquistate come buffer 10x fosfato (0.17 M KH2PO4 e 0.72 M K2HPO4).
- Preparare LB medium: Luria-Bertani (5 g/L estratto di lievito; 10 g/L tryptone; e 10 g/L NaCl). Regolare il pH a 7,0 con 5 M NaOH.
- Preparare 1x TBS: linea salina con buffer di tris (TBS: 50 mM Tris-Cl, pH - 7,5; 150 mM NaCl).
- Preparare 1x TBST: TBS integrato con 0.1% Tween 20.
2. Preparazione del DNA
NOTA: il DNA utilizzato per gli array NAPPA deve essere altamente puro; pertanto, non sono raccomandati mini-prep di DNA commerciale. Attualmente, vengono utilizzati due protocolli per la preparazione del DNA: in casa mini-prep ad alto valore di throughput (descritto qui) o commerciale Midi- o Maxi-prep. La velocità effettiva media del protocollo mini-prep interno è di 1.500 campioni al giorno per persona.
- Crescita batterica per mini-prep ad alto throughput in-house
- Preparare la piastra omni LB/Agar. Versare 30-40 mL di agar LB (1,5 % di agar batteriologico nei supporti LB integrato con antibiotico per la selezione di cloni positivi) in ogni singola piastra del pozzo.
- Avvistare il brodo di glicerolo su piastra LB/agar. Diluire lo stock di glicerolo in supporti LB (1:300, di solito 2 - L in 600 l. Agitare per 10 min. Spot 3 - L del brodo diluito sulla piastra LB/agar. Incubare a 37 gradi centigradi, capovolto, durante la notte.
- Inoculare le culture. Utilizzando il dispositivo a 96 pin che è stato sterilizzato in 80% etanolo e fiamma, inoculare la coltura dalla piastra di agar in un blocco di pozzo profondo con 1,5 mL per pozzetto di mezzo TB integrato con antibiotico.
- Incubano culture. Coprire il blocco con una guarnizione permeabile a gas e incubare per 22-24 h a 37 gradi centigradi, 300-800 rpm a seconda dello shaker.
NOTA: Gli shaker impostati a 800 giri/min sono ottimali per questa incubazione. L'uso di uno shaker di velocità più lento può provocare colture meno dense e minori rese di purificazione del DNA. - Cultura Pellet. Blocco di spin a 3.800 x g e 4 gradi centigradi per 30 min.
- Mini-prep ad alta velocità automatica in-house
NOTA: per eseguire la mini-prep di mini-preconfigurazione ad alta velocità interna è possibile utilizzare pipettors multicanale o distributori automatici. Se si utilizza un dispenser automatico, assicurarsi di pulire il sistema prima dell'uso e tra le soluzioni.- Preparare tutte le soluzioni utilizzate durante la mini-preparazione:
- Preparare la soluzione 1: buffer di sospensione TE (50 mM Tris, pH - 8,0; 10 mM EDTA, pH - 8,0; 0,1 mg/mL di RNAse). Conservare a 4 gradi centigradi.
- Preparare la soluzione 2: buffer di lisi sia NaOH/SDS (0,2 M NaOH; 1% SDS). Per ottenere risultati migliori, si dovrebbe utilizzare una soluzione appena realizzata.
- Preparare la soluzione 3: buffer di neutralizzazione KOAC (2,8 M KOAc). Regolare il pH della soluzione a 5.1 con acido acetico glaciale. Conservare a 4 gradi centigradi.
- Preparare la soluzione N2: equilibratione buffer (100 mM Tris; 900 mM KCl; 15% EtOH; 0.15% Triton X-100). Regolare il pH della soluzione a 6,3 con acido fosforico.
- Preparare la soluzione N3: bash buffer (100 mM Tris; 1.15 M KCl; 15% EtOH). Regolare il pH della soluzione a 6,3 con acido fosforico.
- Preparare la soluzione N5: buffer di eluizione (100 mM Tris; 1 M KCl; 15% EtOH). Regolare il pH della soluzione a 8,5 con acido fosforico.
NOTA: il corretto controllo del legame, del lavaggio e dell'eluzione del DNA durante lo scambio di anioni dipende fortemente dalla concentrazione di CCU e dai valori di pH del buffer. Sono essenziali misurazioni accurate dei componenti tamponati e regolazione del pH. Piccole deviazioni dalle misurazioni descritte possono comportare una significativa perdita di rendimenti.
- Ri-sospendere pellet. Aggiungere 200 l della soluzione 1 e agitare a 2.000 giri/mm per 5 min a RT. È necessaria una ri-sospensione completa del pellet per una lisi di successo. Vorticare il blocco, se necessario.
- Batteri di Lyse. Aggiungere 200 l della soluzione 2, sigillare la piastra con un sigillo in alluminio e invertire 5x. Tempo attentamente questo passaggio dall'inizio della soluzione 2 aggiunta. Non superare i 5 min.
- Neutralizzare la soluzione. Aggiungere 200 l della soluzione 3, sigillare la piastra con un sigillo in alluminio e invertire 5x. Il sigillo può essere allentato a causa dei buffer di lisi/neutralizzazione, quindi prestare attenzione quando si inverte. Si raccomanda un'inversione parziale, in cui la soluzione non tocca mai la guarnizioni, per evitare la contaminazione incrociata tra i campioni.
- Lisato chiaro. Centrifugare le piastre a 3.800 x g e 4 gradi centigradi per 30 min.
- Preparare il liquame di resina di scambio di anion durante la fase di centrifugazione della pelletazione lisata. Utilizzando una bottiglia da 1 L, riempirla con la resina di scambio di anion fino a raggiungere il marchio 300 mL, quindi aggiungere la soluzione N2 fino a 900 mL.
AVVISO: Questo passo dovrebbe essere fatto nel cofano per proteggere contro l'inalazione di silice. - Preparare piastre di resina di scambio di anion. Impilare le piastre di filtro sopra un blocco di pozzi profondi per agire come un recipiente di raccolta dei rifiuti. Mescolare il liquame di scambio di anion fino a quando non è omogeneo, quindi versare in una depressione di vetro. Utilizzando punte P1000 annoiate annoiate, trasferire 450 gradi di liquami in ogni pozzetto delle piastre filtranti.
- Centrifuga piastre impilate (piastra di resina / piastra di pozzo profondo) a lenta accelerazione per 5 min a 130 x g e RT. Scartare il flusso attraverso.
- Trasferire l'unlfato supernatant alla piastra di resina / blocchi di pozzi profondi. Girare le piastre impilate per 5 min a 30 x g con lenta velocità ramp-up.
- Colonna di lavaggio. Aggiungere 400 l della soluzione N3 (lavaggio tampone) ad ogni pozzetto. Trasferire la piastra di resina a vuoto collettore per rimuovere il tampone di lavaggio. Ripetere i passaggi di lavaggio 3x. Nell'ultimo lavaggio, assicurarsi che tutti i pozzi siano svuotati correttamente. Girare le piastre di impilamento a 150 x g per 5 min per rimuovere qualsiasi buffer residuo.
- Dna erida. Collocare la piastra di resina su una piastra di raccolta pulita da 800 luna. Aggiungere 300 l di soluzione N5 a ogni pozzo. Lasciare seduto a RT per 10 min, quindi girare le piastre impilate per 5 min a 20 x g con lenta velocità ramp-up. Girare le piastre impilate per 1 min a 233 x g.
- Quantifica il DNA e conserva le piastre a -20 gradi centigradi fino a nuovo utilizzo o procedi direttamente alla precipitazione del DNA.
NOTA: Sono necessari un minimo di 30 g di DNA per campione. Se la resa del DNA è bassa, si consiglia di ripetere la mini-preparazione del DNA, o in alternativa combinare due piastre durante la fase di precipitazione (sezione 2.3).
- Preparare tutte le soluzioni utilizzate durante la mini-preparazione:
- Precipitazioni del DNA
- Scongelare piastre, vortice per omogeneizzare la soluzione del DNA, e girare a 230 x g per 30 s per raccogliere tutte le soluzioni nella parte inferiore del pozzo.
- Aggiungete 40 l l di 3 M NaOAc e 240 l of isopropanol ad ogni pozzo. Coprire la piastra con un sigillo in alluminio e mescolare invertendo 3x.
- Centrifugare le piastre per 30 min a 3.800 x g e 25 gradi centigradi. Scartate con cura il super-atali.
NOTA: Per combinare due piastre, trasferire il DNA dalla seconda piastra nel pellet dalla prima piastra e ripetere i passaggi 2.3.2–2.3.3. - Lavare e far precipitare il DNA. Aggiungete 400 l dell'80% di etanolo a ogni pozzo. Piastre di sigillare con sigillo in alluminio e agitare a 1.000 giri/mm per 30 min. Centrifughe a 3.800 x g per 30 min a 25 gradi centigradi. Scartare il super-attardato.
- Asciugare i pellet di DNA. Posizionare le piastre a testa in giù con un angolo su carta assorbente per 1-2 h, fino a quando non è presente alcool nella parte inferiore del pozzo. Sigillare e centrifugare a 230 x g per 2 min per portare eventuali pellet verso il basso.
- Una volta che le piastre sono asciutte, sigillano con sigillo di alluminio e congelate a -20 gradi centigradi per un uso successivo o continuate a rimettere in spunto il DNA (passaggio 4.1).
3. Rivestimento scorrevole aminosilane
- Posizionare i vetrini di vetro in cremagliera metallica. Ispezionare visivamente ogni vetrino per assicurarsi che non siano presenti graffi o imperfezioni.
- Sommergiivi in soluzione di rivestimento (2% reagente aminosilane in acetone) per 15 min durante il dondolo. La soluzione di aminosilastra può essere utilizzata per rivestire due rack di 30 vetrini ciascuno prima che debba essere scartato.
- Sciacquare il passo. Immergere il portadiapositive nel lavaggio dell'acetone (99% acetone), scuotere avanti e indietro, poi su e giù rapidamente 5x. Inclinare a un angolo per gocciolare fuori, quindi immergere in acqua Ultrapure su e giù rapidamente 5x. Inclinare per gocciolare fuori, quindi posizionare sui tovaglioli.
NOTA: Il lavaggio degli acetone può essere utilizzato due volte, mentre l'acqua ultrapura deve essere cambiata ogni volta. - Scivoli secchi con aria sotto pressione, soffiando su di essi da tutte le angolazioni per circa 3 min fino a quando tutte le goccioline d'acqua sono state rimosse. Conservare i vetrini rivestiti in una griglia di metallo all'interno di una scatola ben sigillata.
4. Preparazione dell'esempio di matrice
- Risospendere il pellet di DNA dal mini-prep interno (passaggio 2.3.6) in 20 - L di acqua ultrapura e agitare a 1.000 giri/min per 2 h. Per il DNA di preparazione midi/max, diluire ogni campione fino a una concentrazione finale di 1,5 g/l e trasferire 20 l su una piastra di raccolta di 800 .
- Preparare il mix di stampa. Per una piastra da 96 pozzetti, preparate 1 mL di miscela di stampa [237,5 l'l di acqua ultrapura; 500 luna di poli-lisina (0,01%); 187,5 - L di BS3 (bis-sulfosuccinimidyl, 50 mg/mL in DMSO); e 75 l of polyclonal anti-rabbit anti-flaganti)].
NOTA: Le sostanze chimiche devono essere aggiunte nell'ordine specificato per evitare precipitazioni. - Aggiungete 10 l di miscela di stampa ad ogni campione, sigillate le lastre con foglio di alluminio e agitate a 03 00 min a 1.000 giri/m. Conservare le piastre durante la notte a 4 gradi centigradi.
- Il giorno della stampa, vorticare brevemente e girare le lastre. Trasferire 28 l di ogni campione su una piastra di matrice 384. Questo trasferimento può essere fatto utilizzando l'automazione o una pipetta multicanale. È fondamentale tenere traccia della posizione dei campioni nella piastra di matrice 384.
- Ruotare brevemente la piastra per rimuovere eventuali bolle. Sigillare le piastre con un foglio.
5. Generazione di array NAPPA: stampa microarray
NOTA: tutte le condizioni di stampa sono state ottimizzate per lo strumento elencato in Tabella delle attrezzature e dei materiali. Se si utilizza una matrice diversa, potrebbe essere necessaria un'ulteriore ottimizzazione.
- Pulizia dell'array. Prima di iniziare, svuotare tutti i serbatoi di rifiuti e riempire i serbatoi con acqua ultrapura o 80% di etanolo, se necessario. Perni puliti uno per uno con salviette senza lacrime e acqua ultrapura. Asciugare i perni con salviette senza lanucine e rimetterli con cura nella testa dell'arrayer.
- Arrayer impostato: specifiche di stampa [numero massimo di timbri per inchiostro: 1; numero di timbri per punto: 1; tempo di timbro multistampo: --; ora timbro (ms): 0 ms; tempo di inchiostro (ms): 0 ms; regolazione della profondità di stampa: 90 micron; numero di ritocchi: 0]. protocollo di sterilizzazione: lavaggio dell'acqua ultrapuro per 2.000 ms con 0 ms di tempo di essiccazione e 500 ms di tempo di attesa; ripetere questi passaggi 6x; seguita da lavaggio con 80% di etanolo per 2.000 ms con 1.200 ms di tempo di essiccazione e 500 ms di tempo di attesa; ripetere questi passaggi 6x.
- Slide design: impostare l'arrayer con il modello di arraydesiderato. La progettazione deve prendere in considerazione diversi fattori [ad esempio, il numero di repliche per ogni campione, la posizione e il numero delle funzionalità di controllo, il layout della matrice (un blocco, diversi blocchi identici), il numero di matrici da stampare, la lunghezza della conduzione e così via.
- Posizionare i vetrini rivestiti di aminosila (passaggio 3.4) sul ponte dell'arrayer. Verificare se l'aspirapolvere tiene tutti i vetrini saldamente in posizione. Avviare l'umidificatore (dovrebbe essere impostato al 60%).
- Posizionare il 384 ben piastra sul ponte arrayer. Avviare il programma.
- Etichettare i microarray. Al termine della stampa, posizionare le etichette delle diapositive sul lato inferiore (non stampato) di ogni diapositiva. Mantenere l'ordine di stampa delle diapositive sul deck in ordine numerico.
- Conservare gli array stampati in RT in una griglia metallica all'interno di una scatola ben sigillata con un pacchetto di silice. I vetrini conservati in un ambiente asciutto hanno una durata di conservazione fino a un anno.
- (Facoltativo): un secondo lotto di 90 diapositive può essere stampato utilizzando gli stessi campioni. Per farlo, rimuovere la lastra 384 bene dal ponte arrayer non appena la stampa della piastra è fatto. Sigillare e riporre la piastra a 4 gradi centigradi. Dopo che il primo lotto di array è stato completamente fatto, rimuoverli dal ponte, posizionare nuovi vetrini rivestiti di aminosilane e iniziare una nuova corsa. Assicurarsi che ogni piastra 384 bene è a RT per 30 min prima del suo utilizzo. Se più di quattro repliche per campione vengono stampate in un lotto di diapositive, si consiglia di dividere le 384 lastre ben in due piastre per ridurre l'evaporazione del campione diminuendo la quantità di tempo trascorso sul ponte dell'arrayer.
NOTA: Controllare tutti i serbatoi prima dell'inizio della seconda tiratura.
6. Rilevamento del DNA sui vetrini NAPPA
- Bloccare le diapositive. Posizionare i vetrini in una scatola pipetta e aggiungere 30 mL di buffer di blocco. Incubare a RT per 1 h su uno shaker a dondolo.
- Macchia le diapositive. Eliminare la soluzione di blocco e aggiungere 20 mL di buffer di blocco e 33 l di colorante fluorescente del DNA fluorescente. Incubare per 15 min con agitazione. Quindi, sciacquare rapidamente i vetrini con acqua ultrapura e asciugarli con aria sotto pressione. Procedere con la scansione (sezione 11).
7. Espressione delle diapositive NAPPA
- Bloccare le diapositive con buffer di blocco su uno shaker a dondolo a RT per 1 h. Utilizzare circa 30 mL in una scatola di pipetta per quattro vetrini.
- Sciacquare i vetrini con acqua ultrapura e asciugarli con aria compressa filtrata. Applicare la guarnizione di guarnizione a ogni vetrino secondo le istruzioni del produttore.
- Aggiungere il mix IVTT. Ogni scivolo richiederà 150 gradi l di mix IVTT. Diluire 82,5 ll di HeLa lisate in 33 litri di acqua DEPC e integrare con 16,5 litri di proteine accessorie e 33 uL di miscela di reazione. Aggiungere la miscela IVTT dall'estremità non etichetta o non-specimen. Pipette il mix lentamente (è accettabile se perline temporaneamente all'estremità di inserito). Massaggiare delicatamente la guarnizione di guarnizione in modo che il mix IVTT si alpra e copra l'intera area dell'array. Applicare le piccole guarnizioni rotonde a entrambe le porte.
- Posizionare i vetrini su un supporto e trasferirli nell'incubatrice di raffreddamento programmabile. Incubare per 90 min a 30 gradi centigradi per l'espressione delle proteine, seguita da 30 min a 15 gradi per l'immobilizzazione della proteina di query.
- Lavare e bloccare i vetrini. Rimuovere la guarnizione di stufa e immergere i vetrini in una scatola di pipette con circa 30 mL di 1x TBST integrato con il 5% di latte per il display proteico (sezione 8) o 1x TBST integrato con 3% di albumina di siero bovino (BSA) per i saggi di chinasi o lo screening farmacologico (sezione 9). Incubare a RT con agitazione per 20 min e ripetere questo passaggio 2x.
8. Rilevamento di proteine su array NAPPA
- Aggiungere l'anticorpo primario. Rimuovere i vetrini dalla soluzione di blocco (passaggio 7.5) e asciugare delicatamente il lato posteriore (lato non stampato) utilizzando un tovagliolo di carta. Posizionare le diapositive su un supporto e applicare 600 l dell'anticorpo primario (mouse anti-flag) diluito 1:200 in 1x TBST - 5% latte. Incubare per 1 h a RT.
- Lavare i vetrini con 1x TBST - 5% di latte su uno shaker a dondolo (3x per 5 min ciascuno).
- Aggiungere l'anticorpo secondario. Rimuovere i vetrini dalla soluzione di lavaggio e asciugare delicatamente il lato posteriore con un tovagliolo di carta. Posizionare le vetrine su un supporto e applicare 600 l dell'anticorpo secondario (anticorpo antito-tono cy3-labbeled) diluito 1:200 in 1x TBST - 5% latte. Proteggere i vetrini dalla luce e incubare per 1 h a RT.
- Lavare i vetrini con 1x TBST su uno shaker a dondolo (3x per 5 min ciascuno). Sciacquare rapidamente i vetrini con acqua ultrapura e asciugare con aria sotto pressione. Procedere con la scansione (sezione 11).
9. Screening dell'inibitore della chinasi tirosina sugli array NAPPA
NOTA: più diapositive possono essere elaborate nello stesso esperimento, tuttavia, assicurarsi che in ogni passaggio, una diapositiva viene elaborata alla volta e che non si asciugano tra i passaggi. Aggiungere tutte le soluzioni all'estremità non etichetta o non provino del vetrino.
- Preparare tutte le soluzioni utilizzate durante lo screening farmacologico:
- Preparare la soluzione fosfosasi/DNase combinando quanto segue: 1x proteina metallo-phosphatases buffer (50 mM HEPES, 100 mM NaCl, 2 mM DTT, 0.01% Brij 35 a pH - 7,5); 1 mM MnCl2; 8.000 unità di fosfofoculosi proteico lambda; e 2 unità di DNase I. Preparare 400 l della soluzione per ogni microarray. Aggiungere fosforano e DNase appena prima dell'uso.
- Effettuare la diluizione del farmaco. I farmaci vengono ricostituiti nella DMSO ad una concentrazione finale di 10 mM. Per assicurare che tutte le concentrazioni di farmaci testate sull'array, assicurarsi che lo stesso volume di DMSO (uno stock di 10.000x in DMSO) sia creato per ogni concentrazione e mantenuto a -80 gradi centigradi. Al momento dell'uso, i farmaci vengono diluiti 1:100 in acqua.
- Preparare la soluzione farmaco/chinasi combinando quanto segue: 1x ninasi tampone (25 mM Tris-HCl di pH - 7.5); 5 mM beta-glicerofosfato; 2 MM DTT; 0,1 mM Na3VO4; 10 mM MgCl2; 500 bancomat m; e 2 - L di droga (diluito 1:100 in acqua). Preparare 200 l della soluzione per ogni microarray.
- Eseguire il trattamento fosfoculosi e DNase. Rimuovere i vetrini dalla soluzione di blocco (passaggio 7.5) e asciugare delicatamente il retro con un tovagliolo di carta. Posizionare le diapositive sul supporto e applicare 200 -L di fosforasi/DNase soluzione. Posizionare un coperchio microarray per evitare l'evaporazione. Incubare a 30 gradi centigradi per 45 min in forno.
- Trattamento di fosfofofosi e DNase II: rimuovere le matrici dal forno, scartare il coperchio, rimuovere la soluzione in eccesso e applicare 200 l di fosfora e soluzione DNase appena fatto. Coprire i microarray con coverslip e incubare per altri 45 min a 30 gradi centigradi in forno.
- Lavare i vetrini con 1x TBST - 0,2 M NaCl su uno shaker a dondolo (3x per 5 min ciascuno).
- Eseguire il trattamento farmacologico e la reazione della chinasi. Rimuovere i vetrini dalla soluzione di lavaggio e asciugare delicatamente il retro con un tovagliolo di carta. Posizionare i vetrini sul supporto e applicare 200 - L di droga / chinasi soluzione. Posizionare un coperchio sulla parte superiore per evitare l'evaporazione. Incubare per 1 h a 30 gradi centigradi in forno.
- Lavare i vetrini con 1x TBST - 0,2 M NaCl su uno shaker a dondolo (3x per 5 min ciascuno).
- Ripetere i passaggi da 8.1 a 8.4 utilizzando come anticorpo anti-phosho-Tyr primario diluito 1:100. Sostituite 1x TBST e il 5% di latte in tutti i passaggi con 1x TBST - 3% BSA.
10. Protocollo di ibridazione automatizzata
NOTA: In alternativa, una stazione di ibridazione può essere utilizzata per automatizzare tutte le ibridazioni e gli esercizi sugli array NAPPA (sezioni 7–9) e il protocollo viene fornito come File supplementare 1.
11. Acquisizione di immagini
NOTA: le immagini Microarray devono essere acquisite a una risoluzione di 20 micron o superiore.
- Caricare microarray nel caricatore del supporto per diapositive con le proteine rivolte verso l'alto. Caricare il caricatore nello scanner microarray.
- Selezionare il laser verde con un filtro di emissione di 575/30 nm per eseguire la scansione del segnale dall'anticorpo secondario etichettato cy-3. Se si utilizza un fluoroforo diverso, selezionare la lunghezza del laser/onda corretta per rilevare il segnale dal tinrito fluorescente.
- Definire il nome di ogni immagine e la posizione in cui verranno salvate.
- (Facoltativo): per ogni nuovo fluoroforo, si raccomanda l'ottimizzazione delle condizioni di scansione per rilevare l'intervallo lineare dell'intensità del segnale. Per farlo, scansiona un microarray usando una gamma di fotomoltiplicatori (PMT) e guadagna fino a ottenere un'immagine chiara con segnale non saturo e sfondo basso.
- Scansiona tutti i microarray con le impostazioni ottimizzate e ricordati di disattivare il guadagno automatico.
NOTA: per l'analisi dei dati, tutti i microarray devono essere scansionati utilizzando le stesse impostazioni di scansione. Per i saggi chinasi che utilizzano la cia3 come fluoroforo, le immagini vengono scansionate con un PMT del 20%, un'intensità laser del 25% e 10 micron di risoluzione, utilizzando lo scanner elencato nella Tabella delle attrezzature e dei materiali.
12. Trattamento e analisi dei dati
NOTA: sono disponibili diversi pacchetti software per la quantificazione dei dati microarray con funzionalità simili. La procedura qui descritta è stata progettata per il software elencato nella tabella delle attrezzature e dei materiali.
- Caricare i file TIFF da quantificare, progettare la griglia in modo che corrisponda al layout del microarray e regolare le dimensioni dei punti per incorporare l'intero segnale con l'area minima possibile. Le macchie adiacenti non devono sovrapporsi. Controllare visivamente le prestazioni del software e regolare la griglia manualmente, se necessario.
- Quantificare l'intensità del segnale del microarray. Controllare visivamente i punti per eventuali anomalie (rilegatura non specifica, polvere, ecc.) e rimuoverli dall'analisi dei dati.
- Correggere lo sfondo localmente utilizzando il segnale delle aree vicine sull'array in cui non è presente alcun punto.
- Normalizzare i dati. Per confrontare il segnale tra diversi array, l'intensità del segnale di ogni microarray deve essere normalizzata. Per escludere eventuali outlier, normalizzare i dati utilizzando la media tagliata 30% del segnale dal controllo positivo (punti IgG) dei microarray deforhosphollated.
NOTA: Il segnale proveniente dallo spot IgG non cambia durante il fosforo e la dephosphorylation dei microarray ed è adatto per la normalizzazione. - Identificare le chinasi attive. Per ogni funzione visualizzata sul microarray, calcolare il rapporto tra l'intensità del segnale normalizzato negli array autofosforelati e sforinfonati. Impostare una soglia di 1,5 volte di modifica per l'identificazione delle chinasi attive e contrassegnare tutte le altre caratteristiche come in grado di sottoporsi all'autoforforsfororylazione (N/D).
- Calcolare l'attività di ciascuna chinasi identificata al punto 12.5 come percentuale del segnale regolato (intensità del segnale dell'array di controllo positivo normalizzato (DMSO) sottratto dall'intensità del segnale dell'array di controllo negativo normalizzato (dephosphorylated).
Risultati
I microarray NAPPA auto-assemblati forniscono una solida piattaforma che può essere utilizzata per molte applicazioni distinte, tra cui la scoperta di biomarcatori, le interazioni proteina-proteina, l'identificazione del substrato e lo screening farmacologico10,11 ,12,13,14,15,16,17,18,19,20.
La metodologia complessiva adottata per lo studio dell'attività della chinasi e lo screening degli inibitori della chinasi tirosina sui microarray NAPPA è schematicamente rappresentata nella Figura 1. In primo luogo, i microarray NAPPA sono generati dall'immobilizzazione del cDNA e dell'agente di cattura sui microarray rivestiti. I cDNA vengono quindi utilizzati come modello per la trascrizione e la traduzione delle proteine, utilizzando un sistema IVTT basato sull'uomo, e le proteine appena sintetizzate vengono immobilizzate dall'agente di cattura9. La qualità del microarray stampato può essere monitorata misurando i livelli di DNA (confermando una stampa coerente) o di proteina visualizzati sull'array (confermando l'espressione e la cattura delle proteine; Figura 1). Per diminuire il segnale di fondo e aumentare la gamma dinamica dell'esperimento, i microarray sono trattati con 1) fosfosasi lambda per rimuovere il fosforo dai residui di Ser/Thr/Tyr, quindi con 2) DNase per semplificare la chimica sul posto e diminuire sfondo (Figura 1).
Il passo successivo è la reazione dell'autofosfororylazione, in cui i microarray vengono incubati con tampone di chinasi in assenza di ATP (matrice di controllo negativo, definita microarray dephosphorylated), e il buffer di chinasi è integrato con ATP (controllo positivo, come array autofosforolilati) o ATP - DMSO (controllo del veicolo). Va sottolineato che durante questo passaggio non viene aggiunta alcuna chinasi; pertanto, l'attività intrinseca di ogni chinasi visualizzata sul microarray viene quantificata attraverso la misurazione dei suoi livelli di fosforolalazione utilizzando un anticorpo anti-fosforo-tirosina seguito da un anticorpo secondario ci3-labbeled (Figura 1).
Il controllo di qualità degli array NAPPA-kinase che visualizzano un pannello di chinasi proteiche umane stampate in quadruplicato è illustrato nella Figura 2. I livelli di DNA immobilizzato sono stati misurati dalla colorazione del DNA e hanno mostrato un segnale uniforme attraverso il microarray, suggerendo che la quantità di DNA stampato sull'array era uniforme. È anche possibile osservare diverse caratteristiche senza alcuna colorazione del DNA. Queste caratteristiche corrispondono ad alcuni controlli in cui il DNA è stato omesso dal mix di stampa [cioè, macchie vuote (niente è stato stampato), macchie d'acqua, spot IgG purificato (poli-lysina, crosslinker e IgG purificato), solo mix di stampa (mix di stampa completo: poli-lisina più crosslinker e anticorpo anti-bandiera, senza DNA)]. I livelli di proteine visualizzati sui microarray NAPPA-kinase sono stati valutati dopo la reazione IVTT usando anticorpi antitag.
Per lo screening della chinasi, Flag è stato utilizzato come tag di scelta e il livello di proteina visualizzato sul microarray è stato misurato utilizzando un anticorpo anti-bandiera. Come mostrato, la maggior parte dei punti contenenti cDNA ha mostrato con successo livelli rilevabili di proteine. Alcuni dei punti di controllo senza cDNA hanno anche rivelato il segnale con l'anticorpo anti-bandiera: spot IgG (utilizzato per rilevare l'attività dell'anticorpo secondario) e macchie vettoriali vuote (codici cDNA solo per il tag) (Figura 2). I microarray NAPPA-kinase hanno mostrato una buona riproducibilità tra i vetrini, con la correlazione dei livelli di visualizzazione delle proteine tra lotti di stampa distinti superiori a 0,88 (Figura 2). All'interno dello stesso batch la correlazione era ancora più alta, vicina a 0,92 (dati non mostrati).
Successivamente, l'attività di autofosforilazione della chinasi delle proteine visualizzate sull'array è stata misurata utilizzando anticorpi anti-fosforo-tirosina (Figura 3). Le proteine visualizzate sull'array hanno mostrato alti livelli di fosforo lamierelazione delle proteine dopo l'espressione (Figura 3, a sinistra), che possono essere causati dall'attività intrinseca della chinasi della proteina visualizzata sull'array o dalle chinasi attive presenti nel mix IVTT. Questa fosforolalazione è stata completamente rimossa con il trattamento con fosfofasi lambda e questi microarray sono stati utilizzati per i test della chinasi. Dopo la depfororylazione, le reazioni di autofosfororylazione eseguite senza ATP non hanno mostrato livelli significativi di fosfororylazione, come previsto, mentre i microarray incubati con buffer di chinasi in presenza di ATP hanno mostrato la fosfororylazione proteica a partire da 15 min ( Figura 3). Per lo screening farmacologico, l'attività della chinasi è stata misurata dopo 60 min di reazione all'autoforsfororylazione per massimizzare il numero di chinasi testate.
Il confronto tra microarray in cui i livelli di fosforiliato sono stati misurati subito dopo l'espressione proteica (Figura 3, sinistra) e dopo 60 minuti di reazione all'autoforforosforilazione (Figura 3,destra) ha mostrato: i) proteine fosforelate solo dopo l'espressione, suggerendo che possono essere esogenamente fosforilati dalle proteine presenti sul mix IVTT, ma non possono essere autoforsinfolilate; ii) il fosforilato proteico solo dopo la reazione dell'autofosfororylazione, suggerendo che queste proteine non erano attive dopo l'espressione della proteina e richiedevano l'attività di co-fattori presenti nel tampone della chinasi; o iii) fosforalato proteico su entrambi gli array, suggerendo che fossero attivi in entrambe le impostazioni (Figura 3).
Come esempio dei risultati ottenuti per lo screening degli inibitori della tirosina della chinasi sugli array NAPPA-kinase sono stati utilizzati tre inibitori delle chinasi con distinta selettività tra le chinasi proteiche: staurosporina, imatinib e ibrutinib. Per tutti gli screening, microarray NAPPA dephosforrylati sono stati incubati con crescenti concentrazioni di TKI (che vanno da 100 nM a 10 uM) durante la reazione di autofosfororylazione. Il primo TKI testato è stata la staurosporina, un inibitore della chinasi proteica globale, che ha mostrato una potente inibizione della chinasi sul microarray in quasi tutte le chinasi testate11.
Successivamente, è stato testato l'imatinib, un inibitore ABL e c-Kit utilizzato per il trattamento della leucemia mielocericronica cronica e dei tumori stromali gastrointestinali4,5,6,7. Sugli array NAPPA-kinase imatinib ha mostrato una significativa riduzione dell'attività Abl1 e BCR-Abl1, mentre altre chinasi sono rimaste per lo più inalterate (Figura 4A). La quantificazione dei dati per l'attività della chinasi è stata normalizzata rispetto all'array dephosphorylated e rappresentata come percentuale del microarray di controllo positivo (solo veicolo). I dati per TNK2 (chinasi non rilevante), Abl1 e BCR-Abl1 sono illustrati nella figura 4B. Come previsto, imatinib ha mostrato un'inibizione selettiva nei confronti di Abl1 e BCR-ABl1. I dati per c-Kit erano inconcludenti a causa della mancanza di attività sugli array di controllo positivi.
Infine, ibrutinib, un inibitore covalente approvato dalla FDA della chinasi di tirosina di Bruton (BTK), è stato testato. Ibrutinib è attualmente utilizzato nel trattamento di diversi tumori correlati al sangue con BTK iperattivo, tra cui leucemia linfocitica cronica (CLL), linfoma delle cellule del mantello e macroglobulinemia21diWaldenstr'm,22. Figura 4C, è rappresentativo dei risultati tipici ottenuti per lo screening ibrutinib. L'attività della chinasi di ABL1 (chinasi non rilevante) e BTK (bersaglio canonico) ed ERBB4 (potenziale nuovo obiettivo) è illustrata nella figura 4D. I dati suggeriscono che ERBB4 può essere inibito da ibrutinib in modo specifico della dose. Questa inibizione è stata confermata in vitro e nei saggi a base cellulare11, dimostrando la potenza di questa piattaforma.
Nel loro insieme, i dati suggeriscono che la piattaforma di microarray NAPPA-kinase potrebbe essere utilizzata per lo screening imparziale degli inibitori del TK. Inoltre, lo screening è veloce e può essere facilmente personalizzato per includere qualsiasi variazione delle chinasi proteiche di interesse.
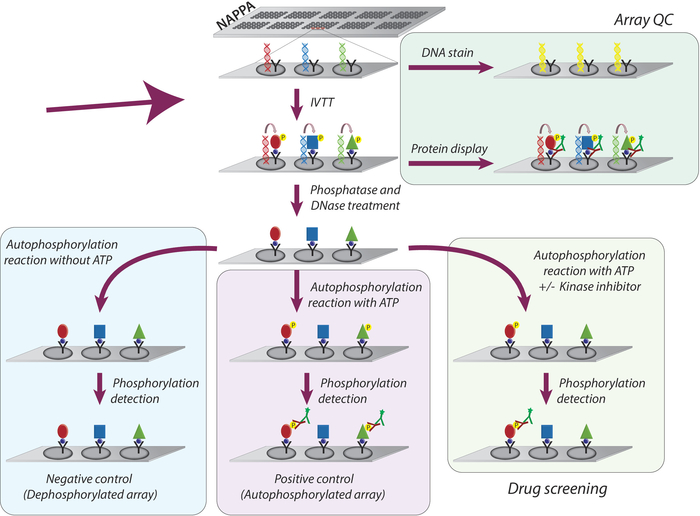
Figura 1: Rappresentazione schematica del controllo di qualità e screening degli inibitori della cinesi nelle matrici NAPPA. Gli array NAPPA sono stampati con codifica cDNA per la proteina di interesse fusa con un tag e un anticorpo di cattura. Durante la trascrizione in vitro e la reazione di traduzione (IVTT) le proteine sintetizzate vengono catturate sulla superficie del microarray attraverso il tag dall'anticorpo di cattura. Il controllo qualità (QC) degli array viene eseguito dalla misurazione dei livelli di DNA stampati sul vetrino, utilizzando un colorante fluorescente di intercalazione del DNA e i livelli di proteine visualizzati sull'array utilizzando anticorpi specifici del tag. Per lo screening della chinasi, i microarray vengono trattati con DNase e fosfosi, dopo la reazione IVTT, per rimuovere il DNA stampato e tutta la fosforolalazione che potrebbe essersi verificata durante la sintesi proteica. Gli array sforphorylated sono ora pronti per essere utilizzati per lo schermo della droga. Per ogni analisi, vengono regolarmente utilizzati tre set di controlli: (I) array dephosphorylated, in cui la reazione di autofosfororylazione viene eseguita senza ATP; (II) microarray autofosforolati, in cui la reazione all'autofosfororylazione viene eseguita in presenza di ATP; e (III) Matrice trattata DMSO (veicolo), in cui la reazione all'autofosfororylazione viene eseguita con ATP e DMSO. I vetrini trattati con una diversa concentrazione di inibitori della chinasi seguono esattamente lo stesso protocollo utilizzato per gli array trattati con DMSO. Fare clic qui per visualizzare una versione più grande di questa figura.
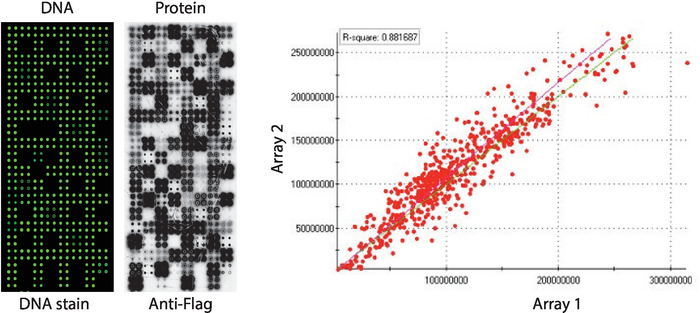
Figura 2: Risultati rappresentativi del controllo qualità per gli array NAPPA-kinase auto-assemblati. Vengono mostrati il contenuto di DNA misurato da un colorante fluorescente che intercalazione del DNA (a sinistra) e i livelli di proteine visualizzati sul microarray misurati dall'anticorpo anti-Bandiera (al centro). Sul lato destro c'è un grafico di correlazione dei livelli di proteine visualizzati su due array NAPPA-kinase stampati in lotti separati. Fare clic qui per visualizzare una versione più grande di questa figura.
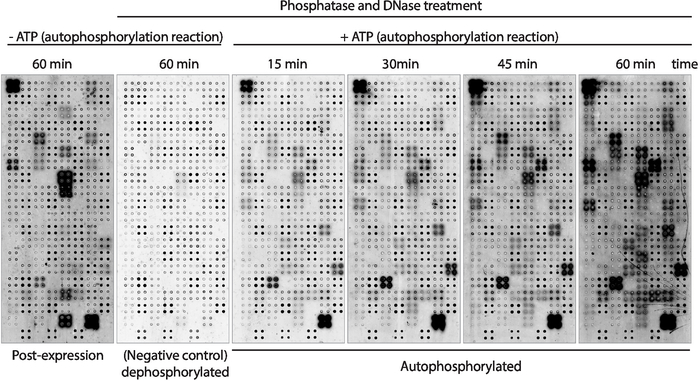
Figura 3: Risultati rappresentativi dell'attività della chinasi negli array NAPPA-kinase. I microarray che mostrano chinasi proteiche in quadruplicato sono stati utilizzati per studiare l'attività della chinasi proteica sull'array attraverso la misurazione della fosforolante delle proteine utilizzando anticorpi anti-pTyr, seguiti da anticorpi antito-topolino con etichetta ci3. Gli array di controllo senza trattamento fosfopobce/DNase e senza ATP durante la reazione all'autoforosforo piano sono stati utilizzati per misurare la fosforilazione di fondo dopo l'espressione proteica (post-espressione). I microarray rimanenti sono stati trattati con fosfoculoe/DNA, e la reazione di autofosfororylazione è stata eseguita senza ATP (microarray dephosphorylated, controllo negativo) o con ATP (microarray autofosforillati). Per i microarray autofosforiliati la reazione dell'autoforsfororylazione è stata eseguita per 15 min, 30 min, 45 min o 60 min, come mostrato. Fare clic qui per visualizzare una versione più grande di questa figura.
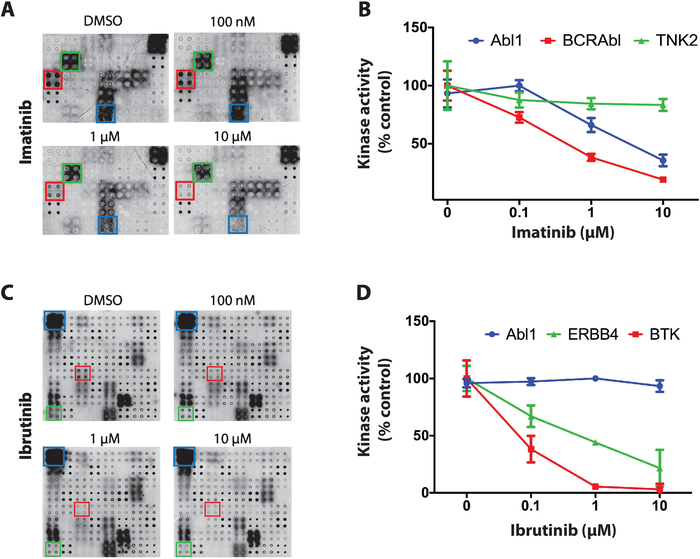
Figura 4: Dati rappresentativi dalla schermata della chinasi della tirosina sugli array NAPPA-kinase. (A) Gli array NAPPA-kinase trattati con fosfofosi sono stati incubati in crescenti concentrazioni di imatinib durante la reazione all'autofosfororylazione e l'attività della chinasi è stata misurata con anti-phospho-anticorpi. (B) Quantificazione dell'attività della chinasi osservata sugli array NAPPA-kinasi esposti all'imatinib. I dati sono stati normalizzati contro il segnale degli array di controllo negativo (dephosphorylated) ed è mostrato come percentuale degli array di controllo positivi (reazione di autofosporylazione eseguita in presenza di DMSO). Dati simili vengono visualizzati per lo screening di ibrutinib (C,D). Questa cifra è stata modificata da Rauf et al.11. Fare clic qui per visualizzare una versione più grande di questa figura.
File supplementare 1. Protocollo alternativo per lo screening degli inibitori della chinasi della tirosina negli array NAPPA utilizzando una stazione di ibridazione automatizzata. Fare clic qui per scaricare questo file.
Discussione
Modifiche e risoluzione dei problemi
Durante la fase di ottimizzazione dello studio dell'attività della chinasi sugli array NAPPA, una delle principali fonti di fondo e bassa gamma dinamica osservata è stata la BSA utilizzata sul mix di stampa. BSA stava fornendo le ammine primarie necessarie per il collegamento incrociato con la superficie dell'ammisornazione e stava intrappolando il DNA e l'anticorpo di cattura sul posto. Tuttavia, BSA è altamente fosforelato, rendendo difficile per il rilevamento del segnale di autoforsfororylation sull'array sopra il rumore di fondo. Per risolvere questo problema, sono state testate diverse alternative per BSA nella miscela di stampa e la poli-lysina è stata identificata come un buon sostituto. La polialisina manca di qualsiasi sito di fosforilio; pertanto, lo sfondo da matrici non espresse è molto minimo. Inoltre, i microarray stampati con polilisina sono riproducibili e mostrano buoni livelli di proteine (Figura 2).
La successiva modifica critica eseguita sul saggio standard NAPPA è stata l'aggiunta di una fase di trattamento Fosfobce/DNase. Il trattamento dei microarray con fosfofasi consente la rimozione di qualsiasi fosforilazione che si è verificato nel mix IVTT durante la sintesi e la cattura delle proteine (Figura 3). La fonte di questa fosforolante potrebbe essere da attività intrinseca di autoforosforelazione o dall'attività delle chinasi presenti nel mix IVTT. La rimozione di tutto il fosforo-espressione ha permesso una facile identificazione delle chinasi che sono attive e possono essere sottoposte all'autoforosfororylation (Figura 3).
Passaggi critici all'interno del protocollo
NAPPA è una tecnologia robusta, ma come previsto, ci sono diversi passaggi critici. Il primo è l'acquisizione di DNA di alta qualità nella concentrazione appropriata. L'utilizzo di DNA di scarsa qualità o a basse concentrazioni genererà microarray di scarsa qualità con diverse caratteristiche non espresse e visualizzate nei livelli appropriati, diminuendo il numero di proteine analizzate sull'array. Il secondo passo critico è l'espressione delle proteine sul microarray. L'uso di un sistema IVTT che esprimerà alti livelli di proteine funzionali è fondamentale per studiare l'attività della chinasi sull'array.
Il prossimo passo critico sullo screening TKI è il modo in cui vengono gestiti i microarray. I microarray non devono asciugarsi durante qualsiasi fase del protocollo, e si raccomanda una manipolazione delicata per evitare graffi che possono aumentare il segnale di fondo. Poiché gli array dell'intero esperimento saranno confrontati tra loro, è importante assicurarsi che ogni fase di incubazione sia uniforme in tutte le diapositive. Ad esempio, il tempo necessario per eseguire un passaggio in una singola matrice deve essere preso in considerazione quando un batch di 20 matrici viene elaborato per evitare differenze nella lunghezza dell'incubazione tra le matrici.
Infine, la progettazione dell'esperimento e l'inclusione di controlli positivi e negativi sono fondamentali per il controllo della qualità e l'analisi dei dati. Il primo set di controlli sono quelli stampati in ogni matrice e includono controlli negativi [cioè punti vuoti (senza materiale stampato), acqua o vettore vuoto (esprimere solo il tag)], nonché un controllo positivo (cioè IgG purificato, che viene rilevato dal anticorpo secondario ed è inerte all'alterazione dei livelli di fosforo). Collettivamente, misurano i livelli di fondo del microarray, possibile riporto durante la stampa e l'intensità del segnale del metodo di rilevamento.
La serie successiva di controlli sono i controlli di screening dei farmaci e includono i microarray dephosphorylated e autofosforelazione (in presenza o assenza di DMSO). Come accennato in precedenza, il microarray dephosphorylated misura il livello di fosforo dopo il trattamento con fosfofore e quindi il livello di base per tutti gli altri esperimenti. Più basso è il livello di base, maggiore è la gamma dinamica dei saggi. Gli array autofosforili presentano i livelli massimi di fosforo di tutti gli array e il segnale deve essere forte e chiaro. Viene utilizzato per l'analisi dei dati, ma anche come controllo che tutte le reazioni sono state eseguite correttamente sulla matrice.
Limitazioni della tecnica
Al momento, una delle limitazioni dello screening farmacologico qui presentato è la sua capacità di vagliare solo le chinasi proteiche che possono essere autofosfore. Un modo possibile per superare questo è quello di stampare una chinasi e substrato noto nello stesso punto. La co-stampa del DNA per due distinte proteine è stata realizzata con successo15, suggerendo la fattibilità di questo approccio. Inoltre, la proteina visualizzata sull'array potrebbe non essere piegata correttamente causando una proteina inattiva. L'uso del sistema di espressione basato sull'uomo ha fatto un miglioramento significativo dell'attività della chinasi misurata sulla matrice; tuttavia, alcune proteine non possono ancora essere analizzate sull'array a causa della sua inattività.
Una seconda limitazione è la misurazione del fosforilo utilizzando un anticorpo pan anti-phospho-tyr. Nonostante la sua non specificità per quanto riguarda il motivo del sito di fosfororylazione, tutte le fosfororylazioni misurate si sono verificate sui residui di tirosina, lasciando dietro di sé serine e treonine e le rispettive chinasi. Ad oggi, più di 10 anticorpi pan fospho-Ser/Thr sono stati testati senza successo, nonostante diversi tentativi di ottimizzare le condizioni di incubazione e lavaggio. Un nuovo sistema di rilevamento indipendente dagli anticorpi può essere l'opzione migliore per espandere il numero di chinasi proteiche che possono essere sottoposte a screening per l'inibizione dei farmaci. In questo contesto, sono disponibili alcune opzioni, tra cui la radioattività o approcci chimici come la coniugazione dei clic. Una serie di ottimizzazioni sono necessarie per ridurre al minimo il segnale di fondo e fornire una buona gamma dinamica per i saggi.
La terza limitazione è l'acquisizione di cloni cDNA da stampare sull'array. I cloni cDNA possono essere generati utilizzando qualsiasi tecnica di clonazione, inclusi sistemi di ricombinazione specifici del sito, ad esempio Creator o Gateway23. Un'altra opzione è quella di acquistare i cloni dalla libreria DNAsu, trovata all'indirizzo , dove più di 17.000 cloni cDNA, tra cui l'intero kino umano, è prontamente disponibile per essere utilizzato per la costruzione di array NAPPA24 .
La quarta limitazione è che non tutti i laboratori sono dotati di attrezzature appropriate per fabbricare e schermare i propri array NAPPA. Questo protocollo fornisce metodi alternativi per generare il DNA da stampare sul microarray, senza la necessità di apparecchiature ad alta velocità, e protocolli per eseguire manualmente tutti i passaggi di ibridazione. Tuttavia, l'accesso a uno scanner arrayer e microarray è ancora necessario. Un'opzione per risolvere questo problema consiste nell'utilizzare il servizio e la struttura di base NAPPA, disponibile all'indirizzo , che distribuisce microarray NAPPA personalizzati a un prezzo accademico senza scopo di lucro. Infine, a partire da qualsiasi metodologia di screening, i dati ottenuti sugli array sono suscettibili di essere artefatti (positivi o negativi) e pertanto devono essere convalidati utilizzando saggi ortogonali.
Significato rispetto ai metodi esistenti
Diverse piattaforme sono disponibili in commercio per lo screening delle chinasi proteiche. Un approccio usato di routine è i saggi vincolanti, che possono essere eseguiti con frammenti proteici, dominio chinasi, frammenti proteici più grandi con il dominio della chinasi e alcune regioni regolatorie, e persino proteine a lunghezza intera. Le proteine sono di solito espresse in sistemi batterici a causa del costo e della semplicità nei protocolli di espressione e purificazione. L'interazione tra il farmaco di interesse e la proteina viene quindi misurata con un qualche tipo di saggio di rapporto come la fluorescenza o la presenza di tag, per esempio. La principale limitazione di questo insieme di approcci è il fatto che la proteina non è necessariamente attiva durante l'interazione con il farmaco, il che può comportare l'identificazione di interazioni false positive e false negative. I frammenti proteici sono particolarmente vulnerabili ai cambiamenti nella conformazione e nella mancanza di attività e tutti i dati ottenuti dovrebbero essere convalidati utilizzando proteine attive, preferibilmente nella loro forma completa. Un'altra limitazione di alcune piattaforme è la capacità di schermare solo gli analoghi ATP, limitandone l'uso complessivo.
La maggior parte dei servizi disponibili in commercio per lo screening dei TCI utilizzando approcci basati enzimatici utilizzano solo versioni di tipo selvaggio della chinasi di interesse, e talvolta solo pochi mutanti selezionati. Sapendo che la resistenza ai farmaci è molto comune nei pazienti trattati con TKI, è importante essere in grado di misurare la risposta dei farmaci in diversi mutanti, per la selezione dell'inibitore più appropriato. A causa della natura di NAPPA, lo screening dei mutanti di chinasi è semplice e può essere facilmente realizzato, e l'unico strumento necessario è l'incorporazione della mutante chinasi nella collezione cDNA NAPPA, che può essere eseguita da mutagenesi site-specific, per esempio.
Applicazioni future
Una delle forme più comuni di trattamento traslano nella terapia del cancro usando inibitori della chinasi è l'acquisizione di mutazioni nel bersaglio farmacologico durante un ciclo di trattamento. Lo screening di questi mutanti per quanto riguarda la loro risposta agli inibitori della chinasi è di vitale importanza per la selezione di seconda/terza generazione di TCI per ottenere un trattamento personalizzato per ogni paziente. L'approccio allo screening farmacologico qui presentato fornisce una piattaforma di screening imparziale in cui qualsiasi inibitore della chinasi tirosina può essere testato contro un pannello di chinasi tirosina presenti nel genoma umano. Poiché le proteine visualizzate sugli array NAPPA sono espresse in vitro dal cDNA stampato sulla diapositiva, qualsiasi variante mutante può essere facilmente incorporata nella collezione cDNA per essere visualizzata sull'array. La struttura in cui i mutanti chinasi possono essere generati ed espressi sulla matrice, combinata con il potere ad alto consumo della tecnica NAPPA, fornisce un ambiente unico per lo studio dei mutanti di chinasi e la loro risposta alle droghe, rendendo NAPPA adatto per screening farmacologico personalizzato, uno degli obiettivi della medicina di precisione.
Divulgazioni
Gli autori non dichiarano conflitti di interesse.
Riconoscimenti
Gli autori desiderano ringraziare tutti nel laboratorio di LaBaer per il loro aiuto e le critiche durante lo sviluppo del progetto. Questo progetto è stato sostenuto dalla sovvenzione NIH U01CA117374, U01AI077883 e Virginia G. Piper Foundation.
Materiali
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| 364 well plates (for arraying) | Genetix | x7020 | |
| 800 µL 96-well collection plate | Abgene | AB-0859 | |
| 96-pin device | Boekel | 140500 | |
| Acetic Acid | Millipore-Sigma | 1.00066 | |
| Acetone 99.9% | Millipore Sigma | 650501 | |
| Aluminum seal for 96 well plates | VWR | 76004-236 | |
| Aminosilane (3-aminopropyltriethoxysilane) | Pierce | 80370 | |
| ANTI-FLAG M2 antibody produced in mouse | Millipore Sigma | F3165 | |
| Anti-Flag rabbit Antibody (polyclonal) | Millipore Sigma | F7425 | |
| ATP 10 mM | Cell Signaling | 9804S | |
| β-Glycerophosphate disodium salt hydrate | Millipore-Sigma | G9422 | |
| bacteriological agar | VWR | 97064-336 | |
| Blocking Buffer | ThermoFisher/Pierce | 37535 | |
| Brij 35 | ThermoFisher/Pierce | BP345-500 | |
| BS3 (bis-sulfosuccinimidyl) | ThermoFisher/Pierce | 21580 | |
| BSA (bovine serum albumin) | Millipore Sigma | A2153 | |
| Coverslip 24 x 60 mm | VWR | 48393-106 | |
| Cy3 AffiniPure Donkey Anti-Mouse IgG (H+L) | Jackson ImmunoResearch | 715-165-150 | |
| DeepWell Block, case of 50 | ThermoFisher/AbGene | AB-0661 | |
| DEPC water | Ambion | 9906 | |
| DMSO (Dimethyl Sulfoxide) | Millipore-Sigma | D8418 | |
| DNA-intercalating dye | Invitrogen | P11495 | |
| DNase I | Millipore-Sigma | AMPD1-1KT | |
| DTT | Millipore-Sigma | 43816 | |
| EDTA | Millipore-Sigma | EDS | |
| Ethanol 200 proof | Millipore-Sigma | E7023 | |
| Filter plates | Millipore-Sigma | WHA77002804 | |
| Gas Permeable Seals, box of 50 | ThermoFisher/AbGene | AB-0718 | |
| Glass box | Wheaton | 900201 | |
| Glass slides | VWR | 48300-047 | |
| Glycerol | Millipore-Sigma | G5516 | |
| HCl (Hydrochloric acid) | Millipore-Sigma | H1758 | |
| HEPES Buffer Solution | Millipore-Sigma | 83264 | |
| Human-based IVTT system | Thermo Scientific | 88882 | |
| ImmunoPure Mouse IgG whole molecule | ThermoFisher/Pierce | 31202 | |
| Isopropanol | Millipore-Sigma | I9516 | |
| KCl (Potassium chloride) | Millipore-Sigma | P9333 | |
| KH2PO4(Potassium phosphate monobasic) | Millipore-Sigma | P5655 | |
| Kinase buffer | Cell Signaling | 9802 | |
| KOAc (Potassium acetate) | Millipore-Sigma | P1190 | |
| Lambda Protein Phosphatase | new england biolabs | P0753 | |
| Lifterslips, 24 x 60 mm | ThermoFisher Scientific | 25X60I24789001LS | |
| Metal 30-slide rack with no handles | Wheaton | 900234 | |
| MgCL2 (Magnesium chloride) | Millipore-Sigma | M8266 | |
| Na3VO4 (Sodium orthovanadate) | Millipore-Sigma | S6508 | |
| NaCl (Sodium Chloride) | Millipore-Sigma | S3014 | |
| NaOAc (Sodium acetate) | Millipore-Sigma | S2889 | |
| NaOH (Sodium hydroxide) | Millipore-Sigma | S8045 | |
| NucleoBond Xtra Midi / Maxi | Macherey-Nagel | 740410.10 / 740414.10 | |
| Nucleoprep Anion II | Macherey Nagel | 740503.1 | |
| Phosphoric Acid | Millipore-Sigma | 79617 | |
| Poly-L-Lysine Solution (0.01%) | Millipore-Sigma | A-005-C | |
| Protein Phosphatase (Lambda) | New England Biolabs | P0753 | |
| RNAse | Invitrogen | 12091021 | |
| SDS (Sodium dodecyl sulfate) | Millipore-Sigma | L6026 | |
| SDS (Sodium dodecyl sulfate) | Millipore-Sigma | 05030 | |
| Sealing gasket | Grace Bio-Labs, Inc | 44904 | |
| Silica packets | VWR | 100489-246 | |
| Single well plate | ThermoFisher/Nalge Nunc | 242811 | |
| Sodium acetate (3M, pH 5.5) | Millipore-Sigma | 71196 | |
| TB media (Terrific Broth) | Millipore-Sigma | T0918 | |
| Tris | IBI scientific | IB70144 | |
| Triton X-100 | Millipore-Sigma | T8787 | |
| Tryptone | Millipore-Sigma | T7293 | |
| Tween 20 | Millipore-Sigma | P9416 | |
| Yeast Extract | Millipore-Sigma | Y1625 | |
| Name | Company | Catalog number | Comments |
| Equipments | Maker/model | ||
| Programmable chilling incubator | Torrey Pines IN30 Incubator with Cooling | ||
| Shaker for bacterial growth | ATR Multitron shaker | ||
| Vacuum manifold with liquid waste trap | MultiScreenVacuum Manifold 96 well | ||
| 96 well autopippetor/liquid handler | Genmate or Biomek FX | ||
| Liquid dispenser | Wellmate | ||
| DNA microarrayer | Genetix QArray2 | ||
| Automatic hybridization station | Tecan HS4800 Pro Hybridization Station | ||
| Microarray scanner | Tecan PowerScanner | ||
| Microarray data quantification | Tecan Array-ProAnalyzer 6.3 |
Riferimenti
- Melnikova, I., Golden, J. Targeting protein kinases. Nature Review Drug Discovery. 3 (12), 993-994 (2004).
- Patterson, H., Nibbs, R., McInnes, I., Siebert, S. Protein kinase inhibitors in the treatment of inflammatory and autoimmune diseases. Clinical and Experimental Immunology. 176 (1), 1-10 (2014).
- Wu, P., Nielsen, T. E., Clausen, M. H. FDA-approved small-molecule kinase inhibitors. Trends Pharmacological Sciencies. 36 (7), 422-439 (2015).
- Druker, B. J., et al. Effects of a selective inhibitor of the Abl tyrosine kinase on the growth of Bcr-Abl positive cells. Nature Medicine. 2 (5), 561-566 (1996).
- Heinrich, M. C., et al. Inhibition of c-kit receptor tyrosine kinase activity by STI 571, a selective tyrosine kinase inhibitor. Blood. 96 (3), 925-932 (2000).
- Stagno, F., et al. Imatinib mesylate in chronic myeloid leukemia: frontline treatment and long-term outcomes. Expert Review Anticancer Therapy. 16 (3), 273-278 (2016).
- Ben Ami, E., Demetri, G. D. A safety evaluation of imatinib mesylate in the treatment of gastrointestinal stromal tumor. Expert Opinions in Drug Safety. 15 (4), 571-578 (2016).
- Ramachandran, N., et al. Self-assembling protein microarrays. Science. 305 (5680), 86-90 (2004).
- Festa, F., et al. Robust microarray production of freshly expressed proteins in a human milieu. Proteomics Clinical Applications. 7 (5-6), 372-377 (2013).
- Yazaki, J., et al. Mapping transcription factor interactome networks using HaloTag protein arrays. Proceedings of the National Academy of Sciences of the United States of America. 113 (29), E4238-E4247 (2016).
- Rauf, F., et al. Ibrutinib inhibition of ERBB4 reduces cell growth in a WNT5A-dependent manner. Oncogene. 37 (17), 2237-2250 (2018).
- Anderson, K. S., et al. Protein microarray signature of autoantibody biomarkers for the early detection of breast cancer. Journal of Proteome Research. 10 (1), 85-96 (2011).
- Wang, J., et al. Plasma Autoantibodies Associated with Basal-like Breast Cancers. Cancer Epidemiol Biomarkers Prevention. 24 (9), 1332-1340 (2015).
- Bian, X., et al. Tracking the Antibody Immunome in Type 1 Diabetes Using Protein Arrays. Journal of Proteome Research. 16 (1), 195-203 (2017).
- Song, L., et al. Identification of Antibody Targets for Tuberculosis Serology using High-Density Nucleic Acid Programmable Protein Arrays. Molecular and Cellular Proteomics. 16 (4 suppl 1), S277-S289 (2017).
- Wang, J., et al. Comparative Study of Autoantibody Responses between Lung Adenocarcinoma and Benign Pulmonary Nodules. Journal of Thoracic Oncology. 11 (3), 334-345 (2016).
- Montor, W. R., et al. Genome-wide study of Pseudomonas aeruginosa outer membrane protein immunogenicity using self-assembling protein microarrays. Infection and Immunity. 77 (11), 4877-4886 (2009).
- Tang, Y., Qiu, J., Machner, M., LaBaer, J. Discovering Protein-Protein Interactions Using Nucleic Acid Programmable Protein Arrays. Current Protocols in Cell Biology. 74, 11-15 (2017).
- Yu, X., et al. Copper-catalyzed azide-alkyne cycloaddition (click chemistry)-based detection of global pathogen-host AMPylation on self-assembled human protein microarrays. Molecular and Cellular Proteomics. 13 (11), 3164-3176 (2014).
- Anderson, K. S., et al. Autoantibody signature for the serologic detection of ovarian cancer. Journal of Proteome Research. 14 (1), 578-586 (2015).
- Woyach, J. A., Johnson, A. J., Byrd, J. C. The B-cell receptor signaling pathway as a therapeutic target in CLL. Blood. 120 (6), 1175-1184 (2012).
- Smith, M. R. Ibrutinib in B lymphoid malignancies. Expert Opinion on Pharmacotherapy. 16 (12), 1879-1887 (2015).
- Festa, F., Steel, J., Bian, X., Labaer, J. High-throughput cloning and expression library creation for functional proteomics. Proteomics. 13 (9), 1381-1399 (2013).
- Seiler, C. Y., et al. DNASU plasmid and PSI:Biology-Materials repositories: resources to accelerate biological research. Nucleic Acids Research. 42, D1253-D1260 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon