Method Article
全染色体、テロメア、遺伝子座を明らかにするためのDNAハロー調製物での蛍光In situハイブリダイゼーション
Erratum Notice
要約
DNAハロー調製物と蛍光in situハイブリダイゼーションを組み合わせることで、核酸骨格とのゲノム相互作用の高分解能解析が可能になります。付着したゲノムは、抽出された残留核内に位置するハイブリダイズした蛍光シグナルをもたらしますが、非付着ゲノムは、残留核を取り囲むDNAのハローにあります。
要約
ゲノムは、その活性を調節し、特定の場所に固定するために、細胞核内のいくつかの構造に関連付けられています。これらの構造は総称して核骨格として知られており、核薄層、核小体、および核体が含まれます。ゲノムとその組織を研究するために、蛍光in situハイブリダイゼーション(FISH)の多くの変異体が存在しますが、これらは分解能によって制限されることが多く、ゲノムと核構造との関連に関する情報が不十分です。DNAハロー法は、高塩濃度と非イオン性界面活性剤を使用して、ゲノム内の付着領域を介して核内の構造に固定されたままのDNAループを生成します。ここでは、ヒストン、脂質、核マトリックスに強固に結合していないDNAなどの可溶性核タンパク質が抽出されます。これにより、内部核構造と抽出耐性タンパク質に密接に関連するDNAを含む残留核の周りに、結合していないDNAのハローが形成されます。これらの拡張されたDNA鎖は、分解能の向上を可能にし、物理的なマッピングを容易にすることができます。FISHと組み合わせることで、この方法には、ゲノムが固定されているすべての構造とのゲノム相互作用を研究するという追加の利点があります。HALO-FISHと呼ばれるこの技術は非常に用途が広く、DNAハローを核酸プローブと組み合わせて、遺伝子座、全染色体、アルファサテライト、テロメア、さらにはRNAを明らかにすることができます。この技術は、正常細胞および癌などの疾患進行における核組織化および機能に関する洞察を提供する。
概要
「核マトリックス」は、1974年にベレズニーとコフィーによって最初に記述されました1。ラットの肝臓核に対して高塩モル濃度の抽出とヌクレアーゼ処理を行った後、彼らはタンパク質性の構造フレームワークを特定しました。DNAハロー手順は、その後この方法から適応され、核マトリックス(NM)およびNM関連タンパク質および染色体のみが存続するように可溶性タンパク質の除去を含む。DNA結合領域はDNAループの基部に位置し、マトリックス結合領域(MAR)または足場付着領域(SAR)と呼ばれ、それぞれ高塩濃度およびイオン性界面活性剤リチウム-3,5-ジヨードサリチル酸リチウム(LIS)による抽出に耐性があります。DNAハローでは、MAR/SARに関連するDNAが残留核内に結合しているのに対し、DNAループはこれらの部位から離れてDNAハローを形成します。現在、ゲノムはラミナ関連ドメイン(LAD)を介して核ラミナに、核小体関連領域(NAD)を介して、そしておそらく特定の核体などの他の核構造を介して固定されていることがわかっています。
DNAハロー法は、クロマチンからヒストンが剥がされ、DNAが引き伸ばされるため、拡張されたDNAとクロマチンがより高い分解能を提供するため、DNA、遺伝子、および染色体領域の物理的マッピングに使用できます2,3,4,5,6。ただし、このアプリケーションにDNAハローを使用する場合は、いくつかの制限があります。例えば、DNAハローの残留核と密接に関連するDNAは、プローブにアクセスできないため、分析や物理的マッピングから除外される可能性があります6。ファイバーフィッシュ2,4,5,7や分子コーミング8などの他の技術も物理マッピングを可能にし、比較的迅速かつ簡単に実行できるという利点があります。どちらも、DNAハローよりも遺伝子のDNAマッピングに優先的に使用されます。これらの方法は、核からの溶媒または塩抽出を使用してクロマチン繊維を抽出しますが、分子コーミングは再現性が高い傾向があります8,9。
核骨格が、DNAの付着部位、クロマチンリモデリング、DNA転写、DNA修復、DNA複製などの重要な核プロセスをサポートする役割を担っているという証拠が増えています11,12。このように、DNAハロー技術は、これらの細胞活動中の核骨格とゲノムの間の相互作用を調べるために開発され、研究で日常的に使用され、報告されてきました。この技術は、疾患の進行に関連するゲノムと核酸骨格の間の相互作用を調査するためにも使用されており、核構造の悪性腫瘍関連の変化が同定されています11。
DNAハロー法は、発生および分化中のゲノムと核酸骨格の関係を調べるためにも使用されています12。多くの研究では、FISH14 と組み合わせた場合、ハロスペルム13またはSpermHalo-FISHとして知られるDNAハロー技術のバリエーションが使用されています。精子クロマチンはプロタミンとして知られるタンパク質にしっかりと結合しており、この技術は精子DNAへのアクセスを改善するために開発されました。ハロスペルマは、精子DNAの完全性を調査し、DNA損傷が存在するかどうかを判断するために使用されています。DNA損傷が少ない精子はDNAハローサイズが大きいと相関しますが、断片化して損傷したDNAのレベルが高い精子は、ハローが小さいか、まったくありませんでした。したがって、ハロスペルマは、胚の質と体外受精13による妊娠の成功の潜在的な予後マーカーとして使用できます。この例は、この技術の潜在的な臨床応用を強調しています。私たちの研究では、HALO-FISHを使用して、早期老化疾患であるハッチンソン-ギルフォード早老症症候群(HGPS)15におけるゲノム挙動の変化と特定の薬物治療の効果を評価しました。
これらおよび他の研究を組み合わせることで、DNAハロー技術が技術の研究と有用性に使用できるプロセス/アプリケーションの幅広さが強調されています。
プロトコル
1.スライドの準備、滅菌、細胞培養
- 500 mLの10%HCl(v / v)を準備し、大きなビーカーに注ぎます。
- 顕微鏡スライドを個別に酸に落とし、2 x gに設定したシェーカーで室温で1時間インキュベートします。
注意: HClは腐食性で刺激性があります。重度の皮膚のやけどや目の損傷、皮膚、目、呼吸器系の刺激を引き起こす可能性があります。ニトリル手袋、目の保護具、白衣など、適切な個人用保護具を着用してください。 - ビーカーから酸をデカントし、水道水で10回、次に脱イオン水でさらに10回スライドします。
- スライドをメタノールで2回すすぎ、炎上滅菌するまでメタノールに保ちます。
注意: メタノールは非常に可燃性の液体であり、飲み込んだり、皮膚に接触したり、吸入したりすると有毒です。.さらに、メタノールは臓器に損傷を与える可能性があり、腐食性および刺激性です。職場の暴露制限を遵守し、ブチルゴム手袋、目の保護具、白衣などの適切な個人用保護具が着用されていることを確認してください。可能であれば、局所排気換気(LEV)ヒューム戸棚で取り扱ってください。 - 金属製のトングまたは長い鉗子を使用して、メタノールを含むビーカーから顕微鏡スライドを取り外します。ブンゼンバーナーで炎上して滅菌し、ブンゼンバーナーの近くにあるスライド用の4つのコンパートメントを含む長方形の細胞培養容器に移します。
注意: 炎上により、使用前に顕微鏡スライドを即座に滅菌できます。ただし、この方法には関連する危険があります。メタノールは可燃性が高いため、スライドを含むビーカーをブンゼンバーナーから離して配置することが重要です。スライドをしっかりと握る長いトングまたは鉗子を使用する必要があります。ビーカー内のメタノールレベルは、使用されるメタノールの量を最小限に抑え、鉗子/トングの端のみがメタノールと接触するように、スライドを覆う必要があります。使用後は、鉗子またはトングからメタノールが蒸発し、これらが冷えていることを確認してから、スライドとメタノールを含むビーカーに戻してください。ビーカーは、メタノールが発火した場合に酸素を飢えさせるために、アルミホイルで覆う必要があります。空気が循環しているクラスII層流フード内で炎が滑ることはありません。 - または、手順1.1〜1.4を実行しますが、メタノールでインキュベートした後にスライドを炎上させるのではなく、糸くずの出ないティッシュにスライドを置いて風乾させます。乾いたら、アルミホイルで包み、滅菌オーブンまたはオートクレーブに入れます。
- 60〜70%のコンフルエントに達するまで、5%CO2 中で37°Cで少なくとも48時間血清を含む適切な培地で細胞を増殖させる。このプロトコルは、ヒト皮膚線維芽細胞(HDF)の早期通過、および古典的なハッチンソン-ギルフォード早老症症候群(HGPS)線維芽細胞(AG06297)および非定型2型HGPS線維芽細胞(AG08466)に対して実施されました。各細胞タイプを採取し、血球計算盤を使用してカウントし、細胞密度を決定します。スライドごとに10 mL培地に1 x 105 細胞を播種します。
注:細胞がコンフルエントになりすぎると、異なる核からのDNAループが収束する可能性があるため、細胞密度は重要です。形質転換細胞はより迅速に増殖する可能性があるため、使用する細胞の種類に応じて播種密度を最適化する必要がありますが、後の継代細胞培養では、目的のコンフルエントに到達するまでに時間がかかる場合があります。 - 静止するために細胞をG0 で停止させる必要がある場合は、スライドごとに1 x 105 細胞(10 mL培地中)を播種し、24時間増殖させます。無血清培地で細胞を2回洗浄し、0.5%の低濃度の血清を含む標準培地(新生ウシ血清、NCS、またはウシ胎児血清、FBS)で7日間インキュベートします。
- DNA Haloアッセイに細胞の増殖状態が必要な場合は、複製中に5-ブロモ-2'-デオキシウリジン(BrdU)をDNAに取り込むことによってS期を通過した細胞を特定します。
- 通常どおり細胞に播種し、24時間増殖させる。培養液を取り出し、BrdUと5-フルオロ-2'-デオキシウリジン(3 μg/μL)を含む培地と交換します。さらに24時間後、培地を取り出し、細胞を培地(10%NCS)で1回洗浄してから、新しい培地(10%NCS)を再供給します。さらに24時間インキュベートしてから、DNAハローアッセイ用のスライドを準備します。
2. プローブの準備
- 染色体全体およびアームペインティングプローブ
- Teleniusらの方法を用いて、縮重オリゴヌクレオチドプライミングポリメラーゼ連鎖反応(DOP-PCR)によってフローソーティングまたはマイクロダイセクションされた染色体の増幅から染色体プローブを作成します16。 表1に示すように、DOP-PCRを使用して、染色体プローブをビオチン-16-dUTPまたはジゴキシゲニン-11-dUTPのいずれかで標識します。増幅プロファイルについてはメーカーの説明書をご確認くださいが、この実験に使用した条件を 表2に示します。
- 標識PCR産物8 μL、Cot-1 DNA7 μL、ニシン精子3 μL、3 M酢酸ナトリウム(pH 5.4)の1/20容量、および2倍量の100%エタノールを一緒に添加して、アームまたは染色体全体プローブを調製します。プローブ溶液を-80°Cで最低30分間インキュベートします。
注:この方法は、単一の染色体プローブ、または目的の染色体ごとに異なる標識(ビオチン-16-UTPとジゴキシゲニン-11-dUTP)が使用されている場合は複数の染色体プローブを作成するために使用できます。
注意: エタノールは非常に可燃性の液体および蒸気であり、深刻な目の炎症を引き起こす可能性があります。熱、高温の表面、発火源から遠ざけてください。職場の暴露制限を遵守し、ブチルゴム手袋、目の保護具、白衣などの適切な個人用保護具が着用されていることを確認してください。可能な場合は、LEVヒューム食器棚でハンドルを取り付けます。 - プローブ溶液を13,700 x g で4°Cで15分間遠心分離し、70%エタノールで洗浄します。遠心分離手順を繰り返し、DNAペレットを乱したり紛失したりしないように注意しながら、上清を廃棄します。DNAペレットを乾燥させます。
- 12 μLのハイブリダイゼーションバッファー(50%ホルムアミド、10%デキストラン硫酸塩、10%20x生理食塩水クエン酸ナトリウム(SSC;3 M NaCl、0.3 Mクエン酸三ナトリウム、pH 7.0)、1%(v/v)ポリオキシエチレンソルビナールモノラウレート(Tween-20))をDNAペレットに加えます。DNAペレットがハイブリダイゼーションバッファーに溶解するまで、37°Cで少なくとも2時間放置します。
注意: ホルムアミドは発がん性および催奇形性であるため、胎児に深刻な害を及ぼす可能性があります。.女性が妊娠している場合、または妊娠が疑われる場合は、ホルムアミドの使用を避ける必要があります。ホルムアミドはLEVヒュームフードで使用する必要があります。職場の暴露制限を遵守し、ブチルゴム手袋、目の保護具、白衣などの適切な個人用保護具が着用されていることを確認してください。
- 細菌人工染色体(BAC)からのDNA単離
- BACクローンのグリセロールストックのごく一部をLuria-Bertani(LB)寒天プレートにストリークします(1%(W / V)NaCl、1%(w / v)トリプトン、0.5%(w / v)酵母エキス、1.5%(w / v)寒天テクニカル、12.5 μg / mL(w / v)クロラムフェニコール)。37°Cで一晩インキュベートします。
- プレートから単一のコロニーを選択し、10 mLのLBブロス(1%(w / v)NaCl、1%(w / v)バクトトリプトン、0.5%(w / v)酵母エキス、12.5 μg / mL(w / v)クロラムフェニコール)を接種します。溶液を振とうインキュベーターに37°Cで一晩放置します。
注意:クロラムフェニコールは癌を引き起こす疑いがあります。取り扱いには注意し、露出を減らしてください。 - 室温で1,700 x g で10分間遠心分離培養します。
- 上清を捨て、300 μLのP1溶液(15 mM Tris(pH 8)、10 mM EDTA、100 μg/mL RNase A)をペレットに加えます。激しくボルテックスし、細胞を2 mLの微量遠心チューブに移します。
- 300 μLのP2溶液(0.2 M NaOH、1%(w/v)ドデシル硫酸ナトリウム(SDS))を細胞に滴下します。閉じた微小遠心チューブを5回反転させ、室温で最大5分間放置します。
注意: 水酸化ナトリウムは腐食性であり、重度の皮膚火傷や目の損傷を引き起こす可能性があります。金属に対して腐食性がある場合があります。取り扱いには注意し、露出を減らしてください。職場の暴露制限を遵守し、ニトリルゴム手袋、目の保護具、白衣などの適切な個人用保護具が着用されていることを確認してください。可能な場合は、LEVヒューム食器棚でハンドルを取り付けます。
注意: ドデシル硫酸ナトリウムは可燃性の固体であり、飲み込むと有害であり、皮膚や呼吸器の炎症を引き起こす可能性があります。.また、深刻な目の損傷を引き起こす可能性があります。ニトリルゴム手袋、目の保護具、白衣など、適切な個人用保護具を着用してください。可能な場合は、LEVヒューム食器棚でハンドルを取り付けます。 - 300 μLのP3(3 M酢酸カリウム)を細胞にゆっくりと加え、穏やかに混合します。微量遠心チューブを氷上に10分間置きます。
- 8,100 x g で4°Cで10分間遠心分離し、上清を800 μLの氷冷イソプロパノールを含むチューブに移します。チューブを数回反転させ、-20°Cで一晩インキュベートします。
注意: イソプロパノールは非常に可燃性の液体および蒸気であり、深刻な目の炎症、眠気、またはめまいを引き起こす可能性があります。.熱、高温の表面、発火源から遠ざけてください。職場の暴露制限を遵守し、ニトリルゴム手袋、目の保護具、白衣などの適切な個人用保護具が着用されていることを確認してください。可能な場合は、LEV)ヒューム食器棚でハンドルを取り付けてください。 - 8,100 x g で4°Cで15分間遠心分離します。 上清を取り除き、別のチューブに移します。氷冷した70%エタノール500μLを加えます。チューブを数回反転させ、8,100 x g で4°Cで5分間遠心分離します。
- 上清を除去し、ペレットを室温で風乾する。ペレットが乾いたら、40 μLのジエチルピロカーボネート処理水(DEPC処理)に再懸濁し、4°Cで一晩放置します。完全に再懸濁したら、5 μLの溶液を除去し、1%アガロースゲルにロードしてDNAの存在を確認します。
- ニック翻訳によるBACの単一遺伝子プローブ調製
- 市販のニック翻訳ラベリングキットを使用してください。または、次のプロトコルを使用します。構成成分と体積については 表3 を参照してください。
- 最後にDNAポリメラーゼIを加えて微量遠心チューブに 表3 の構成成分を一緒に加え、穏やかに混合し、数秒間短時間遠心分離します。溶液を15°Cで2時間インキュベートします。
- フラグメントサイズを確認するには、5 μLの溶液を2%アガロースゲルにロードします。DNAフラグメントサイズの範囲は200〜600 bpである必要があります。DNAフラグメントのサイズが大きい場合は、溶液を15°Cでさらに15分間インキュベートし続け、2%アガロースゲルで製品を実行します。
- 10 mM EDTA、0.1% SDS(0.5 M EDTA2.5 μL、100 μL中のpH 8.0、100 μL中の10%SDS)を添加して、ニック翻訳反応を停止します。溶液を65°Cで5分間加熱します。
- 取り込まれていないヌクレオチドを除去するには、BACプローブをスピンカラムに適用します。市販のスピンカラムを購入することも、シリンジを使用して次のように作成することもできます。
- 30 gのセファデックスG-50を500 mLのカラムバッファー(10 mM Tris-HCl(pH8)、1 mM EDTA、0.1% SDS)に加えます。混合物をオートクレーブする。また、500 mLのカラムバッファー(セファデックスG-50なし)とオートクレーブを作ります。
- 1 mLシリンジの底にオートクレーブ滅菌したグラスウールを加えてスピンカラムを作ります。1 mLシリンジにセファデックスG-50をカラムバッファーで充填します。底に蓋のない微小遠心チューブを備えた15 mLの遠心分離管に1 mLシリンジを入れます。1,600 x g で5分間遠心分離します。
- シリンジを取り外し、下部にある微量遠心チューブを廃棄します。新しい微量遠心チューブを15 mLの遠沈管に戻します。カラムバッファー(Sephadex G-50なし)を1 mLシリンジに加え、15 mL遠沈管に再挿入します。1,600 x g で5分間遠心分離します。この手順をもう一度 2 回繰り返します。
- シリンジを取り外し、新しい清潔なマイクロ遠心チューブを含む15mLの遠心分離チューブに挿入します。プローブをシリンジに塗布し、微量遠心チューブにプローブを収集します。
- DNAプローブを沈殿させるには、5 μLのニシン精子DNA(10 mg / mL)、10 μLの酢酸ナトリウム、および2.25容量の100%エタノールをDNA溶液に加えます。溶液を穏やかに混合し、-80°Cで最低1時間インキュベートします。13,700 x g で4°Cで15分間遠心分離します。
- 上清を廃棄し、ペレットを200 μLの氷冷70%エタノールで4°Cで15分間洗浄します。 上澄み液を取り除き、風乾します。ペレットを20 μLのDECC処理水に室温で数時間、または4°Cで一晩乾燥して再懸濁します。 これでプローブを使用する準備が整い、-20°Cで保存することもできます。
- 各スライドについて、5 μLプローブDNAと5 μLのCot-1 DNAを混合し、Speed Vac真空濃縮器を使用して乾燥させます。ペレットが乾燥したら、12 μLのハイブリダイゼーションミックスに再懸濁します。
3.DNAハローの準備
- スライドと付着した細胞が入っている正方形の培養皿をインキュベーターから取り出します。培地を廃棄し、鉛筆を使用してスライドにラベルを付け、50 mLの氷冷細胞骨格(CSK)バッファー(100 mM NaCl、3 mM MgCl2、0.3 Mスクロース、10 mM 1、4-ピペラジンジエタンスルホン酸(PIPES;pH 7.8)、0.5%(v / v)トリトンX-100を脱イオン水で構成したコプリンジャーに入れます。氷上または4°Cで15分間インキュベートします。
注意: Triton X-100は、皮膚の炎症や深刻な目の損傷を引き起こす可能性があります。ニトリル手袋、ゴーグル、白衣などの適切な個人用保護具を使用して取り扱ってください。 - CSKバッファーを廃棄し、50 mLの1x DNAハローバッファー(DHB;140 mM NaCl、27 mM KCl、110 mM NaHPO4、15 mM KH2PO4;pH7.4)でスライドを3回、すなわちDHBを含むコプリンジャーに浸して取り出します。
- 50 mLの抽出バッファー(2 M NaCl、10 mM PIPES(pH 6.8)、10 mMエチレンジアミン四酢酸(EDTA)、0.1%(w / v)ジギトニン、0.05 mM(v / v)スペルミン、0.125 mM(v / v)スペルミジンを含むCoplinジャーにスライドを移します。室温で4分間インキュベートします。
注意: ジギトニンは、飲み込んだり皮膚に接触したりすると有毒であり、吸入すると致命的です。.ジギトニンがLEVヒューム戸棚で取り扱われていることを確認し、白衣、ニトリル手袋(二重手袋)、安全メガネ、マスクを着用してください。スペルミンとスペルミジンはどちらも重度の皮膚火傷や目の損傷を引き起こす可能性がありますが、EDTAは深刻な目の炎症を引き起こすため、各化学物質の取り扱いには注意してください。
注:粉末を60〜70°Cの温度で水に溶かして、ジギトニンを個別に調製します。 冷却したら、溶解したジギトニンを抽出バッファーに加えます。生物学的活性を維持するために、最後にスペルミン、スペルミジン、ジギトニンを抽出バッファーに加えます。 - スライドを50 mLの10x DHB(1.4 M NaCl、270 mM KCl、1.1 M NaHPO 4、150 mM KH2PO4;PH7.4)、5x、2x、および1x DHBをそれぞれ1分間。
- 10%、30%、70%、95%(v/v)エタノールの50 mLシーケンシャルエタノールシリーズを通してスライド(ストレートインおよびストレートアウト)をディップします。
- 空気乾燥スライドし、2次元蛍光in situハイブリダイゼーション(2D FISH)が行われるまで-80°Cで保存します。
4. 二次元蛍光in situハイブリダイゼーション
- 20x SSCを作る:3 M NaCl、0.3 Mクエン酸三ナトリウム、pH 7.0。このバッファーはオートクレーブ滅菌し、室温で保存し、必要に応じて希釈することができます。
- 70%(v/v)ホルムアミド、2x SSC pH 7.0を作り、水浴中で70°Cに加熱する。
- スライドを、70、90、および100%エタノールの連続した50 mLエタノールシリーズを通して、それぞれ5分間インキュベートします。
- 保温プレート上で風乾スライドし、70°Cのオーブンで5分間焼きます。
- 70%ホルムアミド、2x SSC溶液に70°Cで2分間入れてスライドを変性させます。
メモ: 温度とタイミングは、手順4.5にとって重要です。温度が DNAは変性せず、プローブはハイブリダイズせず、DNAハローフィッシュからシグナルは得られません。 - 変性スライドを50 mLの氷冷70%エタノールに5分間入れ、90%、95%、100%のエタノールシリーズを室温でそれぞれ5分間通過させます。
- 保温プレートで風乾
- 直接標識された全ヒト染色体プローブは、製造元の指示に従って取り扱ってください。これらの実験には、ヒト全染色体塗料1、13、15、17、18を使用しました。また、本実験では、 CCND1 および CTNNA1 遺伝子プローブを用いた。
注:染色体プローブ全体とBAC遺伝子特異的プローブの両方をビオチン-11-dUTPで標識し、シアニン3(Cy3)に結合させたストレプトアビジンによって検出しました。染色体ペインティングプローブ(DOP-PCR)およびニックトランスレーションによって標識されたBAC DNAについては、これらをプロトコルにおいてこの時点からDNAプローブと呼び、以下のように扱う。 - DNAプローブ(全染色体ペイントまたは遺伝子特異的プローブ)を75°Cでホットブロックまたはウォーターバス中で10分間変性させます。
- DNAプローブをホットブロックまたはウォーターバスで37°Cで30分間温めてから、10 μLを適切なスライドにピペッティングします。
注:この手順は、繰り返しの染色体配列をブロックするために重要です。非特異的シグナルを行わない場合は、DNA Halo FISHに産生され得る。 - 21 mm x 21 mmのカバーガラスとゴムセメントを使用したシールを備えたオーバーレイプローブ。
- スライドを加湿ハイブリダイゼーションチャンバーで37°Cで最低18時間インキュベートします。
注:加湿ハイブリダイゼーションチャンバーは、湿らせた組織の数層と、スライドを置くためにカットされた10mLプラスチックピペットから構築された隆起したプラットフォームを含むサンドイッチボックスから作ることができます。これは、光への露出を最小限に抑えるためにアルミホイルで覆われています。 - 鉗子を使用してゴムセメントを慎重に取り除きます。
- スライドを50 mL 50%(v/v)ホルムアミド、2x SSC、pH 7.0溶液でインキュベートし、45°Cに予め加温して5分間3回インキュベートします。
注意: 50%(v / v)ホルムアミド、2x SSC、pH 7.0溶液での最初のインキュベーションで、カバーガラスをスライドから離します。これにより、カバーガラスを「引きずり込む」ことによって引き起こされる可能性のあるDNA Halo調製物の損傷を防ぎます。スライドは、カバーガラスを取り外すのを助けるために鉗子によるグリップを介してバッファー内で攪拌することができます。 - 次に、スライドを60°Cに予熱したが45°Cの水浴に入れた0.1x SSC、pH 7.0溶液の50mLに入れます。5分間インキュベートし、バッファーをさらに2回5分間インキュベーションします。
- 室温で50 mLの4x SSC、pH 7.0溶液を含むCoplinジャーにスライドを入れ、バッファーを3回交換して15分間インキュベートします。
- 100 μLの4%BSA、4x SSC溶液を各スライドに塗布し、パラフィンフィルムをオーバーレイします。室温で10分間インキュベートします。これにより、抗体の非特異的結合が防止されます。
- 標識プローブ(ビオチン-16-dUTP)を検出するには、100 μLの1:200(1% BSA製、4x SSC)ストレプトアビジン-Cy3と室温で1時間インキュベートします。
注:実験前に抗体の希釈とテスト希釈で製造元の指示に従って、良好なシグナルが生成されていることを確認してください - 室温で50 mLの4x SSC(0.5%Tween-20)pH 7.0溶液を含むCoplinジャーにスライドを入れ、バッファーを3回交換して15分間インキュベートします。スライドは、免疫蛍光が必要ない場合は、ステップ4.21に示すようにこの段階で取り付けることができます。
- DNAハローに作られた細胞の増殖状態が必要な場合は、FISHステップの後、組み込みBrdUについてマウントまたは染色する前に抗pKi67抗体で染色する。
- スライドを50 mLの1xリン酸緩衝生理食塩水(PBS)でそれぞれ3回5分間洗浄し、続いてPBS中の4%NCSで室温で1時間ブロッキングします。
- 必要な一次抗体(ウサギ抗ヒトpKi67;マウス抗BrdU)200 μLをスライドに塗布し、パラフィンフィルムのストリップでオーバーレイし、室温で1時間インキュベートします。
- スライドを1x PBSで5分間3回洗浄し、室温で200 μLの蛍光色素結合二次抗体(pKi67:ブタ抗ウサギTRITC;BrdU:ロバアンチマウスCy3)。PBSでさらに3回5分間洗浄します。すべての希釈は、メーカーの推奨希釈率の範囲でPBS中の1%(v / v)NCSを使用して行われます。
- DAPIを含む20 μLのマウンタントにスライドをマウントし、22 mm x 50 mmのカバーガラスでオーバーレイします。
5.テロメアPNAフィッシュ
- テロメアを検出するには、テロメアPNA FISHキット-FITCを使用します。製造元の指示に従って手順を実行してください。手順は、特に明記されていない限り、室温で実行する必要があります。
- スライドをトリス緩衝生理食塩水(TBS、pH 7.5)に2分間浸した後、3.7%ホルムアルデヒド(TBS中、v/v)に正確に2分間入れます。
注意:TBS溶液には、10〜30%のトロメタモールと10〜30%の2-アミノ-2-(ヒドロキシメチル)プロパン-1,3-ジオール塩酸塩が含まれています。.これは深刻な目や皮膚の炎症を引き起こす可能性があるため、保護手袋とゴーグル/顔面保護具を着用してください。換気の良い場所で使用してください。 - コプリンジャーでスライドをTBSで2回ずつ5分間洗浄します。
- スライドを前処理液に10分間浸した後、TBSで2回洗浄し、1回の洗浄で5分間洗浄します。
- 次に、70%、85%、95%(v/v)エタノール50mLからなる氷冷エタノールシリーズを濃度あたり2分間スライドします。その後、スライドを風乾させます。
- 蛍光タグの色の選択に応じて、10 μLのテロメアPNAプローブ/FITC(またはCy3)を各スライドに適用し、カバーガラスでオーバーレイします。80°Cに設定した予熱オーブンで5分間インキュベートし、暗所に約1時間置きます。
注意: テロメアPNAプローブ/ FITCには6〜100%ホルムアミドが含まれているため、深刻な目の炎症を引き起こし、催奇形性であるため、胎児に深刻な害を及ぼす可能性があります。女性が妊娠している場合、または妊娠が疑われる場合は、ホルムアミドの使用を避ける必要があります。ホルムアミドはLEVヒュームフードで使用し、適切な目または顔の保護具を着用する必要があります。. - カバーガラスを取り除くには、スライドを「リンス溶液」に1分間浸してから、「洗浄溶液」に65°Cで5分間入れます。
注意: 洗浄液には、1〜5%のポリオキシエチレンオクチルフェニルエーテルと1〜5%の塩化ナトリウムが含まれています。これは腐食性であり、深刻な目の損傷を引き起こす可能性があります。洗浄液を取り扱うときは、ゴーグルまたは顔面保護具を着用していることを確認してください。 - スライドを50 mLの氷冷エタノールシリーズ(70%、85%、95%(v/v))に通して濃度あたり2分間インキュベートし、風乾します。DAPIを含むマウンタントでドライマウントスライドし、カバーガラスでオーバーレイします。
6.画像のキャプチャと分析
- DNAハローと染色体領域を視覚化するには、落射蛍光顕微鏡(ライカDM4000顕微鏡など)を使用して、HC PL FLUOTAR 100X/1.30オイル対物レンズとDFC365FXカメラを使用して画像をキャプチャします。
- グレースケール画像をキャプチャし、キャプチャされた各チャンネルの色を定義して、画像の疑似カラーリングを有効にします。この実験では、商用ソフトウェア(LAS AFバージョン4.5.0ソフトウェアなど)を使用しました。個々のカラーチャンネルはTIFFとしてエクスポートされました。
- Java画像処理プログラムフィジーImageJを使用して画像を分析します。 [ファイル ]を押して画像をアップロードし 、[開く]をクリックします。
- 個別の画像チャンネルを読み込むか、合成画像を個別のグレースケールチャンネルに分割するには、 Image |カラー |チャンネルを分割します。画像チャンネルを選択し、[ 画像 |調整 し、[ 明るさとコントラスト]を選択します。それに応じて変更し、他のチャネルで繰り返します。
- 核を描いたDAPI染色チャンネルを選択して、残留核のマスクを作成します。[ 画像] |調整 し、[ しきい値] を選択します。しきい値を変更できるダイアログボックスが表示されたら、[ 暗い背景 ]チェックボックスをオンにします。残留核が透明になるまで変更し、[ 適用 ]を押してダイアログボックスを閉じます。
注: これにより、ピクセル強度に基づいてバイナリマスクが作成され、白いピクセルは関心領域を示し、黒いピクセルは背景を示します。プローブチャネルで同じ手順を繰り返します。 - フリーハンド選択を使用して残余核の周辺をアウトライン化し、[ 編集 ]をクリックして [外側をクリア]をクリックします。プローブチャネルを残留核にオーバーレイします。これは、 画像|カラー |チャンネルをマージします。
- ImageJ で計測スケールを設定するには、スケールバー上または 2 つの既知の距離の点間に線を引きます。 [分析 ]に移動し 、[スケールの設定]を押します。ダイアログボックスで距離の長さを追加し、[ OK]をクリックします。距離を計測するには、計測対象のポイント間にラインを描画し、[ 解析 |測定します。これにより、距離値がデータウィンドウに転送されます。
- これは核の中心と一致するので、最も明るいDAPI強度を測定します。この測定から、核中心から最も遠い染色体領域の端(CTE)までの距離を測定します。核中心から核端(NE)までの距離を測定します。
- 結果が CTE/NE 比として表示されることを確認します。ここで、核中心から各最遠端の染色体領域縁までの距離(CTE)を、核中心から各各核縁までの距離(NE)で除算する。これは最低50核で実行する必要があります。これは、棒グラフまたはボックス チャートとして表すことができます。
- テロメアの分析では、データセットごとに最低30個の核を分析します。画像は、Fiji ImageJを使用して分析するか、手動で分析して、残留核内およびDNAハロー内のテロメアの数をカウントできます。BrdUまたはpKi67は、増殖核(BrdU/piK67+)および老化/静止核(BrdU/pKi67-)の分化を可能にした。データは、平均の標準誤差(SEM)に対応する誤差バーを持つ棒グラフで表すことができます。
- スチューデントのt検定(対応なし)を使用して、有意と見なされるp>0.05と結果を統計的に比較します。
結果
このDNAハロー調製法は、若きも若きの細胞内だけでなく、核骨格タンパク質が異常な早期老化疾患に由来する細胞においても、ゲノム挙動の違いを明らかにする試みに役立っています15。 図1 は、残存核の端、残留核内に残っているDNA、および周囲にスプールアウトしてDNAハローを作成している未付着DNAを見ることができるDNAハローの例を示しています。また、残留核の取得方法を示す分析と、NEおよびCTE測定も示しています。細胞がS期にある場合にBrdUなどの標識ヌクレオチドを組み込むか、またはG1細胞において核小体およびヘテロクロマチンの領域を明らかにする診断増殖マーカーanti-pKi67を採用することによって、増殖細胞と非増殖細胞を区別することができる17、18。コンフルエントを達成せずに高血清中で増殖した初代細胞、すなわち増殖マーカーに対して陰性である初代細胞は、老化性であると仮定される。低血清中で増殖した初代細胞、またはコンフルエントになった、すなわち、増殖マーカーに対して陰性である接触阻害細胞は静止しているとみなされ、正しい栄養素および状況が与えられれば増殖性細胞周期に再入することができるであろう。Ki67陽性細胞と陰性細胞を区別することができるようになったことで、増殖、静止、老化のヒト皮膚線維芽細胞の違いを決定することができました。 図2 は、非増殖細胞では起こらないメカニズムであるDNA複製中にBrdUが取り込まれた細胞から作製し、その後抗BrdU抗体で染色した増殖型ヒト皮膚線維芽細胞のDNAハローを示しています。増殖マーカー抗pKi67抗体による染色も 図2に見ることができます。これは堅牢な抗原であり、FISHプロトコルを存続するため、FISH後およびプレマウントのために染色することができます。従って、左側の列にはBrdUおよびanti-pKi67(赤色)に対して増殖細胞が陽性(赤色)であり、右側の列には実際に 図2 の老化細胞が表示されている。緑色の信号は、テロメアPNAフィッシュ/ FITCキットで明らかにされた個々のテロメアです。免疫蛍光法とDNAハローを組み合わせることで、増殖細胞、静止細胞、老化細胞を調査する際に 図2 に示すように、さまざまな細胞状態での分析が可能になります。選択した抗体に応じて、分化、放射線照射によるDNA損傷など、他の条件を調べることができます。
染色体領域は、FISHを使用してDNAハロー内で視覚化することもできます。核からのDNAのスプールを可能にする準備のために、染色体領域の形状は、残留核内のゲノムの固定とその構造に応じて、DNAハローに見られる染色体の量を少量または多くして乱す可能性があります。 図3 は、染色体1、13、17、および18の特定の腕全体の染色体塗装プローブ(赤)で個々の染色体が明らかにされたDNAハローのパネルを明らかにしています。抗pKi67(緑色)は、増殖細胞および同じ培養物内でのその不在を、同じスライド上で、老化細胞を示すために使用されています。CTE / NEとして提示された画像とデータから、小さな遺伝子欠損染色体18は、付着がほとんどなく、残留核から離れたDNAハローにさらにスプールし、他の染色体よりも残留核の中心からかなり離れている染色体であることが非常に明白です。しかし、これは1番染色体にも当てはまります。増殖マーカーanti-pKi67を用いて、老化細胞と、同じ培養物内、同じスライド上で増殖を比較することも可能であり、この分析により、これら2つの非常に異なる細胞状態内の染色体は、残留核構造との付着に関して互いに有意差がないことを明らかにしました。
興味深いことに、遺伝子はまた、残留核内に留まるか、DNA Haloに位置することに関して、増殖細胞と老化細胞の間に統計的に有意な違いを示しています。図4は、赤色の標識BACプローブと緑色の抗Ki67によって描かれた遺伝子座でこれを示しています。DNA Halo調製後、増殖細胞と老化細胞における遺伝子位置の間に有意差はない。しかし、DNAハロー内には、サイクリンD1 CNDD1遺伝子座よりもカテニンα1 CTNNA1遺伝子座が有意に多く存在し、非常に少ない。図5は、テロメアを含むDNAハロー調製物を緑色で示しています。バックグラウンドは、DNAハロー内でテロメア信号を視覚化できるように、意図的に高く残されています。この一連のデータには、静止細胞、すなわち、7日間血清飢餓状態にされた細胞が含まれており、興味深いことに、静止細胞のDNAハロー内には、増殖細胞および老化細胞よりも、付着しておらず、DNAハロー内にあるテロメアが有意に多い。図5aでは、DNAハロー中のテロメアの割合が、特に画像「実験2」について観察することができる。これは、DNAハローにおけるテロメアの平均パーセンテージが静止細胞において約17%である図5bに対応する。老化細胞のすべてのテロメアが、おそらく非常に短いため、見ることができるわけではないといういくつかの証拠があります。
このDNAハローの方法は、罹患細胞の核内のゲノム相互作用の変化を調べることに成功しています15。 図6 は、原発性対照線維芽細胞における染色体結合の違い、および典型的な(ラミンA変異)および非定型のハッチンソン・ギルフォード早老症症候群を有する罹患細胞における、異なるSUN1アイソフォームおよびラミンA変異を発現しないことを示している19。染色体1と13は、対照DNAハローと比較した場合、残留核内の付着に統計的に有意な違いを示しています。 図6b は、染色体領域全体の位置を残留核およびDNA Haloと相関させる。1以下の値は染色体が残留核内にあることを示し、1を超える値は染色体またはDNAハロー内の染色体の一部を示します。
全体として、これは、細胞周期に影響を与えるさまざまな条件(増殖、静止、老化)または早老症や癌細胞株などの疾患細胞内での染色体全体、特定の遺伝子、テロメアのゲノム相互作用を調査する上でのHALO-FISHの有用性を強調しています。実際、これらの状態間の相互作用の違いは、核骨格が核内の重要なプロセスを調節する上で重要な役割を果たしていることを意味します。

図1: HDF抽出核の残留核とDNAハロー、および分析方法の概要を表示。(a)DNAハローアッセイで調製し、DAPIで対比染色したHDF核。明るく染色された残留核は、核骨格に固定されたDNAを示しており、これはDNAのハローを形成する非付着DNAに囲まれています。倍率= x 100;スケールバー10μm。 (b)青色チャネルは、DAPI染色された核と周囲のDNAをキャプチャします。残留核は、ImageJを使用して選択および除去されます。矢印は、核中心から残留核端(NE)までの距離を表しています。(c)赤チャンネルはプローブ信号を示しています。(d)「結果」と表示されている画像は、青チャンネル画像に赤チャンネルを重ね合わせた結果です。これにより、核中心から最も遠い染色体領域縁(CTE)までの距離が可能になります。 この図の拡大版を表示するには、ここをクリックしてください。
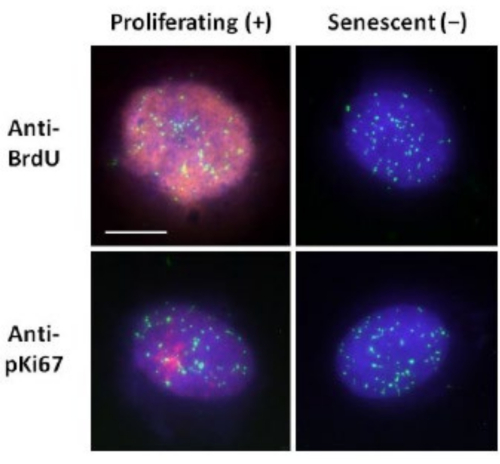
図2: 増殖および老化したHDFに対するテロメアPNA FISHによるDNAハロー調製。DNAハローアッセイに供されたHDF上のテロメアPNA FISH。テロメアシグナルは緑色(FITC)で可視化され、残留DNAとハローDNAはDAPI(青色)を使用して対比染色され、増殖核は赤色の間接免疫蛍光法(TRITC)を介して抗BrdUまたは抗pKi67抗体のいずれかを使用して検出されました。倍率= x 100;スケールバー10μm。 この図の拡大版を表示するには、ここをクリックしてください。
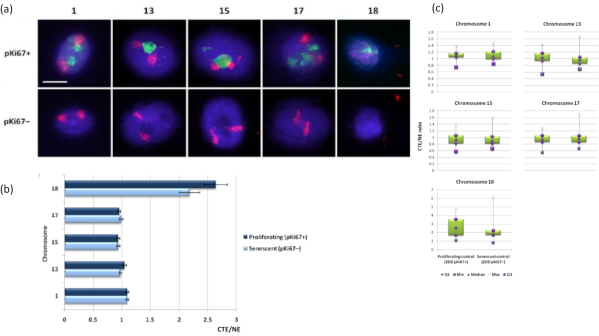
図3: DNAハローアッセイを用いた核酸骨格-染色体相互作用と解析。(a)染色体1、13、15、17および18に特異的なプローブを有する2D-FISHを、DNAハロー調製に供したHDFに対して行った。染色体全体を赤(Cy3)で塗り、核をpKi67で調べて、増殖しているか老化しているかを判断しました。増殖細胞(pKi67+)は緑色(FITC)で描かれたが、老化細胞は染色されず(pKi67-)、すなわち緑色のシグナルは検出されなかった。倍率= x 100;スケールバー10μm。 (b)HALO-FISHを受けた増殖および老化HDFにおける核骨格による染色体固定。測定は、増殖細胞(pKi67+)および老化細胞(pKi67-)における染色体1、13、15、17および18番染色体のそれぞれの核縁(NE)に対する最も遠い染色体領域縁(CTE)の比率を示す。エラーバーはSEM±を表す。 (c)pKi67+およびpKi67-核における特定の染色体のそれぞれの核縁(NE)に対する染色体領域縁(CTE)の修正箱ひげ図表現。Q1 =下の四分位数。最小値 = 記録された最小値。Med = 中央値;最大値 = 記録された最大値。Q3 =上四分位数。 この図の拡大版を表示するには、ここをクリックしてください。
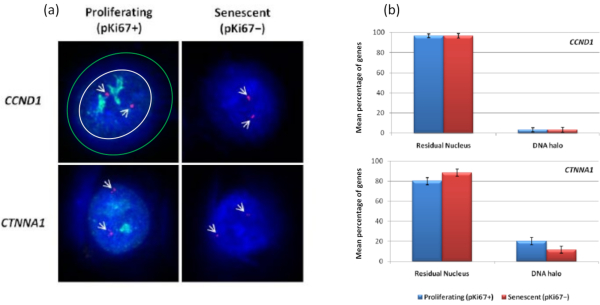
図4: HALO-FISHを用いたHDFにおける遺伝子特異的相互作用。(a)DNAハロー抽出核を遺伝子特異的プローブ(CCND1 および CTNNA1)でプローブし、増殖細胞および老化細胞上のNMへの固定を調べた。遺伝子シグナルは赤色(Cy3)で示され、抗pKi67は増殖細胞を表し、シグナルは緑色(FITC)で視覚化されます。増殖するCCND1画像では、残留核は白い円で囲まれ、白と緑の円の間のスペースはDNAハローを表しています。倍率= x 100;スケールバー10μm。 (b) CCND1 と CTNNA1 の遺伝子特異的シグナルを、残留核とDNAハローの間、および増殖細胞と老化細胞の間で比較します。エラーバー±SEMを表します。 この図の拡大版を表示するには、ここをクリックしてください。
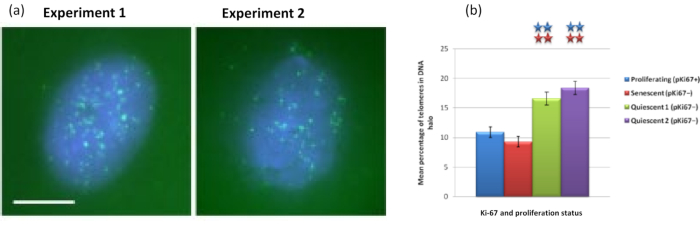
図5: テロメアPNA-FISHでプローブした静止HDFのDNAハローアッセイ。(a)HDFの休止は、低血清培地での7日間の培養によって誘導されました。DNAハローアッセイを実施し、PNA-FISHはFITCシグナル(緑)によるテロメアの可視化を可能にし、残留核と周囲のDNAハローをDAPI(青)で対比染色しました。細胞はまた、核が非増殖であることを確認するために抗pKi67抗体で染色された。これは2つの別々の機会に繰り返されました。倍率= x 100;スケールバー10μm。 (b)増殖、老化および静止HDF細胞におけるDNAハロー内に局在するテロメアの平均割合の比較。エラーバー±SEMを表します。 この図の拡大版を表示するには、ここをクリックしてください。
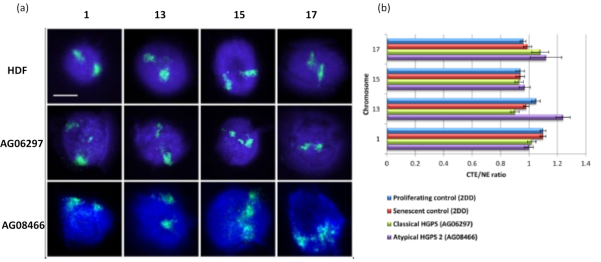
図6:HALO-FISH26を用いたHGPS細胞の核骨格への染色体全固定の検討。(a)コントロールHDF(2DD)、古典HGPS(AG06297)および非定型2型HGPS(AG08466)核は、DNAハロー調製を受け、次に染色体1、13、15および17の全染色体塗料を使用して2D-FISHを受けた。染色体全体は緑色(FITC)で描かれ、DNAはDAPI(青色)で対比染色されました。倍率= x 100;スケールバー10μm。 (b)抽出された核内の染色体の位置は、平均染色体領域縁(CTE)と核縁(NE)の比率を測定することによって決定されました。1を超える比率は、最も遠いCTEがDNAハロー内の対応するNEの外側にあることを示し、1未満の比率は、最も遠いCTEが残留核内のNE内にあることを示します。この図の拡大版を表示するには、ここをクリックしてください。
| 成分 | 容量(μL) |
| 5XDOP-PCRバッファー | 10 |
| dNTPミックス(なしTTP)(2mM) | 5 |
| dTTP(2mM) | 2 |
| ビオチン-16-dUTPまたはジゴキシゲニン-11-dUTP | 10 |
| ドッププライマー(20μM) | 5 |
| タクDNオリメラーゼ(1U/μL) | 1 |
| PCRグレード水用 | 12 |
| テンプレート | 5 |
表1:1x反応のDOP-PCR成分と容量を示す表
| 歩 | サイクル | 温度(摂氏) | 時間 |
| 初期変性 | 1 | 95 | 3 ミン |
| 変性 | 34 | 98 | 20秒 |
| プライマーアニーリング | 62 | 1 ミン | |
| 延長 | 72 | 30秒 | |
| 最終延長 | 1 | 72 | 5 ミン |
| 冷却 | 4 | 持つ |
表2:DOP-PCRサイクル、温度、および時間プロファイルを示す表。
| 構成素 | 容量(μL) |
| 10x NT バッファー (0.5M トリス塩酸塩 pH 8,50 mM MgCl2, 0.5 mg/ml BSA) | 5 |
| 0.1 Mβ-メルカプトエタノール | 5 |
| 10Xヌクレオチドストック(0.5 mM dATP、0.5 mM dCTP、0.5 mM dGTP、0.5 mM dTTP、0.5 mg/mlビオチン-16-dUTP) | 5 |
| ドナーゼ I (1 ng/ml) | 2 |
| DNAポリメラーゼ I | DNA1μgあたり5U |
| DNAテンプレート(1 μg) | 1 |
| DEPC処理水 | 50 μLまで |
表3:1つのプローブのニック変換成分と体積を示す表。
ディスカッション
DNAハロー法は、核酸骨格とゲノムの間の相互作用を分析する際に選択する優れた方法ですが、遵守しなければならないいくつかの重要なステップもあります。最も重要なパラメータの1つは、細胞播種密度の最適化です。細胞がオーバーコンフルエントになると、DNAハローが隣接する細胞と重なり合い、分析を実行できなくなります。CSKおよび抽出バッファーは、生物学的活性を維持するために、調製プロセスの最後にスペルミン、スペルミジン、およびジギトニンを抽出バッファーに追加して、使用日に常に新鮮にする必要があります。Halo-FISHを行う場合、DNAハローの正しい変性温度を使用して、プローブまたはペイントがその後ハイブリダイズできるようにすることが非常に重要です。
電子顕微鏡法は核マトリックスを視覚化するために使用されており、糸状構造が同定されています20。しかし、クロマチンとのマトリックス会合は容易に推測できないため、電子顕微鏡法は限られている。実際、DNA Halo法は、特定の遺伝子、染色体、細胞の状態をすべて調べることができるため、電子顕微鏡と比較してより用途が広いです。さらに、核マトリックスタンパク質のプロテオーム解析が研究されている21,22。この方法は、特に罹患細胞を比較する場合に核マトリックス成分を比較するのに適していますが、標準的なDNA Halo技術で強調されている空間分布と付着は提供されません。
DNA Haloアッセイには限界があります。まず、マトリックスが抽出されるため、これは固定された細胞に対してのみ実行できるため、ライブイメージングは不可能です。DNA Halo法は比較的迅速かつ簡単に実行できますが、細胞培養、プローブ生成、Halo-FISH、および分析をすべて考慮すると、プロセス全体に時間がかかる場合があります。
超解像顕微鏡を使用したDNA HalosおよびHALO-FISHの画像キャプチャは、DNA特異的プローブおよび抗体の分解能を大幅に向上させることができます。さらに、蛍光色素はより簡単にスペクトル分解できるため、1回の実験で多数のDNAプローブを使用して、さらに多くの情報を提供できる可能性があります。染色体コンフォメーションキャプチャ(3C)などの分子生物学的手法の改良は、遺伝子座の相互作用を決定し、細胞内のクロマチン上の空間構成を分析するために使用されてきました。DNAハローアッセイと3Cを組み合わせることができ、M3C23として知られる用語であり、DNAハロー技術の適応性を再び実証しています。
ここで提示される元のデータは、ゲノム挙動調査の可能性とそれらのデータの提示方法を示すためのものです。これらのデータにより、(1)染色体塗装プローブを使用してゲノム付着の有意差を決定できることが実証され、この研究では、18番染色体が分析されたものの中で最も付着していない染色体であることが明らかになりました(図3)。(2)2つの遺伝子座の間に有意差を有する遺伝子座(図4)(3)テロメアは、増殖細胞および老化細胞(図5)と比較して静止細胞においてあまり強く付着しない。不溶性タンパク質であるKi67抗原の存在により、増殖細胞と非増殖細胞を区別することができ、残留核に残るか、ヌクレオチドの取り込みを使用して特定の期間内にS期を通過した細胞を強調表示します(図2)。この技術により、核骨格が損なわれている細胞、すなわちラミノパシー細胞におけるゲノム挙動を分析することもでき、こことBikkul et al., 2018では、対照細胞と比較してゲノムがあまり緊密に付着せず、古典的なHGPS細胞におけるラミンA変異の影響を改善する特定の薬剤で治療すると回復できることを明らかにしました15.しかし、ここでは、ラミンA変異を欠いているが、13番染色体があまり強固に付着していないLINC複合体タンパク質SUN119 の異常な形態を含む非定型HGPS AGO8466細胞の新しいデータを示します(図6)。
HALO-FISHは、核酸骨格とのゲノム相互作用の研究と間接免疫蛍光法を組み合わせて、抽出手順から除去されないタンパク質を分離できるユニークな方法です。核骨格は、特定の癌タイプ19や、診断バイオマーカーとしてのいくつかの核骨格関連タンパク質の重要性など、さまざまな疾患で修飾されることが実証されています24,25。したがって、この技術は、疾患15、24、25、27におけるクロマチン組織化/解体に対する核骨格の影響を調べる上で重要な役割を果たし、ヒト細胞に限定されず、他の動物からの染色体塗装プローブでは、同じDNA-ハロープロトコルを採用することができます28。
開示事項
著者は開示するものは何もありません。
謝辞
染色体アームペインティングプローブの親切な贈り物をしてくれたマイケルビットナー教授に感謝します。LGは、EUが資金提供するEURO-Laminopathiesプロジェクトとブルネル早老症研究基金の支援を受けました。
資料
| Name | Company | Catalog Number | Comments |
| 10X PBS | Thermo Fisher Scientific | 10388739 | Used to create DNA halos |
| 5-bromo-2′-deoxy-uridine | Sigma-Aldrich | B5002-100MG | Labelled nucleotide |
| 5-Fluoro-2′-deoxyuridine | Sigma-Aldrich | F0503-100MG | Labelled nucleotide |
| Agar Technical | Thermo Fisher Scientific | 15562141 | DNA isolation of BAC clones |
| Agarose | Sigma-Aldrich | A939-50G | Check product size of DOP-PCR and nick translation |
| Atypical type 2 HGPS fibroblasts (AG08466) | Coriell Institute | AG08466 | Cell line |
| Bacto tryptone | Thermo Fisher Scientific | 16269751 | DNA isolation of BAC clones |
| Biotin-16-dUTP | Roche Diagnostics | 11093711103 | Labelled nucleotides |
| Chloramphenicol | Sigma-Aldrich | C0378-25G | DNA isolation of BAC clones |
| Classical Hutchinson-Gilford progeria syndrome (HGPS) fibroblasts (AG06297) | Coriell Institute | AG0297 | Cell line |
| Coplin jar | Thermo Fisher Scientific | 12608596 | Holds 5 slides or 8 slides back to back |
| Cot-1 DNA | Thermo Fisher Scientific | 15279011 | Block nonspecific hybridization in HALO FISH |
| DEPC-treated water | Sigma-Aldrich | 693520-1L | DNA isolation of BAC clones |
| Dextran sulphate | Sigma-Aldrich | S4030 | Hybridisation mixture |
| Digitonin | Sigma-Aldrich | D141 | Component of extraction buffer |
| Digoxigenin-11-dUTP | Sigma-Aldrich | 11093088910 | Labelled nucleotides |
| Donkey anti-mouse Cy3 | Jackson Laboratory | 715-165-150 | Secondary antibody |
| EDTA | Sigma-Aldrich | E6758 | Component of extraction buffer |
| Ethanol | Component of extraction buffer | ||
| Ethanol | Sigma-Aldrich | 443611 | Probe precipitation and HALO FISH |
| Fetal bovine system | Thermo Fisher Scientific | 26140079 | Cell culture serum |
| Formamide | Thermo Fisher Scientific | 10523525 | 2D FISH of DNA halos |
| Glass wool | Sigma-Aldrich | 18421 | Spin column |
| Herring sperm | Sigma-Aldrich | D7290 | Probe precipitation |
| HXP™ Lamp (metal halide microscope lamp) | OSRAM | HXP-R120W45C VIS | Image capture of DNA halos |
| Hydrochloric acid | Thermo Fisher Scientific | 10313680 | Cleaning microscope slides |
| Isopropanol | Sigma-Aldrich | I9516-25ML | DNA isolation of BAC clones |
| KAPA HiFi PCR Kit | KAPA Biosystems | KK2103 | PCR Kit |
| Leica DM4000 fluorescent microscope with DFC365 FX camera and LAS AF (Version: 4.5.0) image acquisition software. | Leica Microsystems | Image capture of DNA halos | |
| Luria-Bertani agar | Thermo Fisher Scientific | 13274843 | DNA isolation of BAC clones |
| Magnesium chloride | Sigma-Aldrich | M8266 | Component of CSK buffer |
| Methanol | Thermo Fisher Scientific | 10284580 | Cleaning and sterilizing microscope slides |
| Mouse anti-BrdU antibody | BD Pharmingen | B2531-100UL | BrdU visualisation |
| Newborn calf serum | Thermo Fisher Scientific | 16010159 | Cell culture serum and blocking reagent |
| Nick translation kit | Invitrogen | ||
| PCR grade water | Sigma-Aldrich | 693520-1L | PCR and DNA isolation of BAC clones |
| PCR Primers | Sigma-Aldrich | ||
| PIPES | Sigma-Aldrich | P1851 | Component of CSK and extraction buffers |
| Potassium acetate | Sigma-Aldrich | P1190-100G | DNA isolation of BAC clones |
| QuadriPERM® 4 X 12 | SARSTEDT | 94.6077.307 | Square cell culture dish, polysterene with four compartments. This has hydrophobic surface, is sterile, non-pyrogenic/endotoxin-fee and non-cytotoxic. |
| Rabbit Anti-Ki67 antibody | Sigma-Aldrich | ZRB1007-25UL | Proliferation marker |
| Rnase A | Sigma-Aldrich | R6513 | DNA isolation of BAC clones |
| Rubber cement | Halford's | 101836 | 2D FISH of DNA halos |
| Sephadex G-50 | Sigma-Aldrich | S6022-25G | Spin column |
| Sodium acetate | Sigma-Aldrich | S2889 | Probe precipitation |
| Sodium chloride | Sigma-Aldrich | S5886 | Component of CSK, extraction and SSC buffers |
| Sodium citrate | Sigma-Aldrich | C8532 | Component of SSC buffer |
| Sodium dodecyl sulphate | L3771-100G | DNA isolation of BAC clones | |
| Sodium hydroxide | Sigma-Aldrich | S8045-500G | DNA isolation of BAC clones |
| Spermidine | Sigma-Aldrich | S2626 | Component of extraction buffer |
| Spermine | Sigma-Aldrich | S4264 | Component of extraction buffer |
| Streptavidin-Cy3 | Amersham Life Sciences Ltd, Scientific Laboratory Supplies | pa43001 | Probe antibody |
| Sucrose | Sigma-Aldrich | S0389 | Component of CSK buffer |
| Sucrose | Sigma-Aldrich | S0389 | CSK buffer+A66:D68 |
| SuperFrost™ microscope slides | Thermo Fisher Scientific | 12372098 | Microscope slides: 1 mm thickness, 76 mm length, 26 mm width. Uncoated. |
| Swine anti-rabbit TRITC | Dako | ||
| TELO-PNA FISH KIT | Agilent Dako | K532511-8 | Delineation of telomeres |
| Tris-HCl | Sigma-Aldrich | T3253-100G | Column buffer |
| Triton™ X-100 | Sigma-Aldrich | T9284 | Component of CSK buffer |
| Tryptone | Thermo Fisher Scientific | 10158962 | DNA isolation of BAC clones |
| Tween-20 | Sigma-Aldrich | P9416- 100ML | Detergent |
| Vectashield mountant containing DAPI | Vector Laboratories | H-1200 | 2D FISH of DNA halos |
| Whole human chromosome probes | Calbiochem | 2D FISH of DNA halos | |
| Yeast extract | Thermo Fisher Scientific | 10108202 | DNA isolation of BAC clones |
参考文献
- Berezney, R., Coffey, D. S. Identification of a nuclear protein matrix. Biochemical Biophysical Research Communications. 60 (4), 1410-1417 (1974).
- Haaf, T., Ward, D. C. High resolution ordering of YAC contigs using extended chromatin and chromosomes. Human Molecular Genetics. 3 (4), 629-633 (1994).
- Parra, I., Windle, B. High resolution visual mapping of stretched DNA by fluorescent hybridization. Nature Genetics. 5 (1), 17-21 (1993).
- Senger, G., et al. Released chromatin: linearized DNA for high resolution fluorescence in situ hybridization. Human Molecular Genetics. 3 (8), 1275-1280 (1994).
- Florijn, R. J., et al. High-resolution DNA Fiber-FISH for genomic DNA mapping and colour bar-coding of large genes. Human Molecular Genetics. 4 (5), 831-836 (1995).
- Elcock, L. S., Bridger, J. M. Fluorescence in situ hybridization on DNA halo preparations and extended chromatin fibres. Methods Molecular Biology. 659, 21-31 (2010).
- Heiskanen, M., et al. Visual mapping by fiber-FISH. Genomics. 30 (1), 31-36 (1995).
- Bensimon, A., et al. Alignment and sensitive detection of DNA by a moving interface. Science. 265 (5181), 2096-2098 (1994).
- Michalet, X., et al. Dynamic molecular combing: stretching the whole human genome for high-resolution studies. Science. 277 (5331), 1518-1523 (1997).
- Wilson, R. H., Coverley, D. Relationship between DNA replication and the nuclear matrix. Genes Cells. 18 (1), 17-31 (2013).
- Wilson, R. H. C., Coverley, D. Transformation-induced changes in the DNA-nuclear matrix interface, revealed by high-throughput analysis of DNA halos. Science Reports. 7 (1), 6475 (2017).
- Iarovaia, O. V., Akopov, S. B., Nikolaev, L. G., Sverdlov, E. D., Razin, S. V. Induction of transcription within chromosomal DNA loops flanked by MAR elements causes an association of loop DNA with the nuclear matrix. Nucleic Acids Research. 33 (13), 4157-4163 (2005).
- Tandara, M., et al. Sperm DNA integrity testing: big halo is a good predictor of embryo quality and pregnancy after conventional IVF. Andrology. 2 (5), 678-686 (2014).
- Repping, S., et al. The use of spermHALO-FISH to determine DAZ gene copy number. Mol Human Reproduction. 9 (4), 183-188 (2003).
- Bikkul, M. U., et al. Farnesyltransferase inhibitor and rapamycin correct aberrant genome organisation and decrease DNA damage respectively, in Hutchinson-Gilford progeria syndrome fibroblasts. Biogerontology. 19 (6), 579-602 (2018).
- Telenius, H., et al. Degenerate oligonucleotide-primed PCR: general amplification of target DNA by a single degenerate primer. Genomics. 13 (3), 718-725 (1992).
- Bridger, J. M., et al. Association of pKi-67 with satellite DNA of the human genome in early G1 cells. Chromosome Research. 6, 13-24 (1998).
- Sales Gil, R., Vagnarelli, P. Ki-67: More Hidden behind a 'Classic Proliferation Marker'. Trends in Biochemical Sciences. 43 (10), 747-748 (2018).
- Bikkul, M. U., et al. Telomere elongation through hTERT immortalization leads to chromosome repositioning in control cells and genomic instability in Hutchinson-Gilford progeria syndrome fibroblasts, expressing a novel SUN1 isoform. Genes Chromosomes Cancer. 58 (6), 341-356 (2019).
- Jackson, D. A., Cook, P. R. Visualization of a filamentous nucleoskeleton with a 23 nm axial repeat. EMBO Journal. 7, 3667-3677 (1988).
- Albrethsen, J., et al. Unravelling the nuclear matrix proteome. Journal of Proteomics. 72, 71-81 (2009).
- Mika, S., Rost, B. NMPdb: Database of nuclear matrix proteins. Nucleic Acids Research. 33, 160-163 (2005).
- Gavrilov, A. A., et al. of the nuclear matrix-bound chromatin hubs by a new M3C experimental procedure. Nucleic Acids Research. 38, 8051-8060 (2010).
- Sjakste, N., et al. Role of the nuclear matrix proteins in malignant transformation and cancer diagnosis. Experimental Oncology. 26 (3), 170-178 (2004).
- Leman, E. S., Getzenberg, R. H. Nuclear structure as a source of cancer specific biomarkers. Journal of Cellular Biochemistry. 104 (6), 1988-1993 (2008).
- Volpi, E. V., Bridger, J. M. FISH glossary: an overview of the fluorescence in situ hybridization technique. Biotechniques. 45 (4), 385-386 (2008).
- Bridger, J. M., Foster, H. A. Senescence and the Genome. Human Interphase Chromosomes. , (2021).
- Foster, H. A., Griffin, D. K., Bridger, J. M. Interphase chromosome positioning in in vitro porcine cells and ex vivo porcine tissues. BMC Cell Biology. 13 (1), 30 (2012).
Erratum
Formal Correction: Erratum: Fluorescence In Situ Hybridization on DNA Halo Preparations to Reveal Whole Chromosomes, Telomeres and Gene Loci
Posted by JoVE Editors on 6/05/2023. Citeable Link.
An erratum was issued for: Fluorescence In Situ Hybridization on DNA Halo Preparations to Reveal Whole Chromosomes, Telomeres and Gene Loci. The Authors section was updated from:
Lauren S. Godwin1
Joanna M. Bridger1
Helen A. Foster2
1Laboratory of Nuclear and Genomic Health, Centre for Genome Engineering and Maintenance, Division of Biosciences, Department of Life Sciences, College of Health, Medicine and Life Sciences, Brunel University London
2Biosciences, Department of Clinical, Pharmaceutical and Biological Science, School of Life and Medical Sciences, University of Hertfordshire
to:
Lauren S. Godwin1
Emily Roberts2
Joanna M. Bridger1
Helen A. Foster2
1Laboratory of Nuclear and Genomic Health, Centre for Genome Engineering and Maintenance, Division of Biosciences, Department of Life Sciences, College of Health, Medicine and Life Sciences, Brunel University London
2Biosciences, Department of Clinical, Pharmaceutical and Biological Science, School of Life and Medical Sciences, University of Hertfordshire
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved