Method Article
Tüm Kromozomları, Telomerleri ve Gen Locilerini Ortaya Çıkarmak için DNA Halo Preparatlarında Floresan In Situ Hibridizasyon
Bu Makalede
Özet
DNA halo preparatlarının floresan in situ hibridizasyon ile birleştirilmesi, nükleoiskelet ile genomik etkileşimlerin yüksek çözünürlüklü analizini sağlar. Bağlı genom, artık ekstrakte edilen çekirdeklerin içinde bulunan hibridize floresan sinyallere yol açarken, bağlı olmayan genom, artık çekirdekleri çevreleyen DNA halesindedir.
Özet
Genom, aktivitesini düzenlemek ve belirli yerlere demirlemek için hücre çekirdeği içindeki çeşitli yapılarla ilişkilidir. Bu yapılar toplu olarak nükleoiskelet olarak bilinir ve nükleer lamina, nükleoller ve nükleer cisimleri içerir. Genomu ve organizasyonunu incelemek için birçok floresan in situ hibridizasyon (FISH) varyantı bulunmasına rağmen, bunlar genellikle çözünürlükle sınırlıdır ve genomun nükleer yapılarla ilişkisi hakkında yetersiz bilgi sağlar. DNA halo yöntemi, genom içindeki bağlanma bölgeleri aracılığıyla çekirdeklerdeki yapılara bağlı kalan DNA döngüleri üretmek için yüksek tuz konsantrasyonları ve iyonik olmayan deterjanlar kullanır. Burada, nükleer matrise sıkıca bağlı olmayan histonlar, lipitler ve DNA gibi çözünür nükleer proteinler ekstrakte edilir. Bu, iç nükleer yapılar ve ekstraksiyona dirençli proteinlerle yakından ilişkili DNA'yı içeren artık bir çekirdeği çevreleyen bağlanmamış bir DNA halesinin oluşumuna yol açar. Bu genişletilmiş DNA iplikçikleri artan çözünürlüğü mümkün kılar ve fiziksel haritalamayı kolaylaştırabilir. FISH ile kombinasyon halinde, bu yöntem, genomun demirlendiği tüm yapılarla genomik etkileşimleri incelemenin ek avantajına sahiptir. HALO-FISH olarak adlandırılan bu teknik, DNA halelerinin gen lokuslarını, tüm kromozomları, alfa uydusunu, telomerleri ve hatta RNA'yı ortaya çıkarmak için nükleik asit probları ile birleştirilebildiği çok yönlüdür. Bu teknik, normal hücrelerde ve kanser gibi hastalık ilerlemesinde nükleer organizasyon ve işlev hakkında bir fikir verir.
Giriş
"Nükleer matris" ilk olarak 1974 yılında Berezney ve Coffey tarafından tanımlanmıştır1. Sıçan karaciğer çekirdekleri üzerinde yüksek tuz molariteleri ve nükleaz tedavisi ile ekstraksiyonlar yaptıktan sonra, proteinli bir yapısal çerçeve tanımladılar. DNA halo prosedürü daha sonra bu yöntemden uyarlandı ve çözünür proteinlerin uzaklaştırılmasını içerir, böylece sadece nükleer matris (NM) ve NM ile ilişkili proteinler ve kromozomlar devam eder. DNA bağlanma bölgeleri, DNA döngülerinin tabanında bulunur ve sırasıyla yüksek tuz konsantrasyonları ve iyonik deterjan lityum-3,5-diiyodosalisilat (LIS) ile ekstraksiyona dirençli matrise bağlı bölgeler (MAR'lar) veya iskele bağlanma bölgeleri (SAR'lar) olarak adlandırılır. DNA halelerinde, MAR'lar / SAR'larla ilişkili DNA, artık çekirdek içinde bağlanırken, DNA döngüleri bu bölgelerden uzağa uzanır ve DNA halesini oluşturur. Artık genomun lamina ile ilişkili alanlar (LAD'ler) aracılığıyla nükleer laminaya ve nükleolar ilişkili bölgeler (NAD'ler) ve muhtemelen spesifik nükleer cisimler gibi diğer nükleer yapılar aracılığıyla demirlendiğini biliyoruz.
DNA halo yöntemi, DNA'nın, genlerin ve kromozomal bölgelerin fiziksel haritalanması için kullanılabilir, çünkü genişletilmiş DNA ve kromatin daha büyük bir çözünürlük sağlar, çünkü kromatin histonlardan sıyrılır ve DNA 2,3,4,5,6'ya uzatılır. Bununla birlikte, bu uygulama için DNA halelerini kullanırken bazı sınırlamalar vardır. Örneğin, DNA halelerinin artık çekirdekleriyle sıkı sıkıya ilişkili DNA'ya problar tarafından erişilemez, böylece analiz ve fiziksel haritalama6'dan uzak durulabilir. Fiber-FISH 2,4,5,7 ve moleküler tarama 8 gibi diğer teknikler de fiziksel haritalamayı mümkün kılar ve nispeten hızlı ve gerçekleştirilmesi kolay olma avantajına sahiptir. Her ikisi de tercihen DNA haleleri üzerinden genlerin DNA haritalaması için kullanılır. Bu yöntemler, çekirdekten çözücü veya tuz ekstraksiyonları kullanarak kromatin liflerini çıkarır, ancak moleküler tarama daha iyi tekrarlanabilirliğe sahip olma eğilimindedir 8,9.
Nükleoiskeletin, DNA için bağlanma bölgeleri, kromatin yeniden modellemesi, DNA transkripsiyonu, DNA onarımı ve DNA replikasyonu gibi önemli nükleer süreçleri desteklemede rol oynadığına dair artan kanıtlar vardır11,12. Bu nedenle, DNA halo tekniği, bu hücresel aktiviteler sırasında nükleoiskelet ve genom arasındaki etkileşimleri araştırmak için geliştirilmiştir ve araştırmalarda rutin olarak kullanılmış ve rapor edilmiştir. Bu teknik aynı zamanda genom ve nükleoiskelet arasındaki etkileşimleri, nükleer yapıdaki malignite ile ilişkili değişikliklerin tanımlandığı hastalık ilerlemesi ile ilişkili olarak araştırmak için de kullanılmıştır11.
DNA halo tekniği, gelişim ve farklılaşma sırasında genom ve nükleoiskelet arasındaki ilişkiyi araştırmak için de kullanılmıştır12. Bir dizi çalışma, FISH14 ile birleştiğinde halosperm 13 veya SpermHalo-FISHolarak bilinen DNA halo tekniğinin bir varyasyonunu kullanmıştır. Spermatozoa kromatini, protaminler olarak bilinen proteinlere sıkıca bağlanır ve bu teknik, sperm DNA'sına erişimi iyileştirmek için geliştirilmiştir. Halosperm, spermatozoa DNA'sının bütünlüğünü araştırmak ve DNA hasarının olup olmadığını belirlemek için kullanılmıştır. Daha az DNA hasarına sahip spermatozoa, daha büyük bir DNA halo boyutuyla ilişkilidir, oysa parçalanmış ve hasar görmüş DNA seviyelerinin arttığı spermatozoalarda ya küçük haleler vardı ya da hiç yoktu. Böylece halosperm, IVF13 ile embriyo kalitesinin ve başarılı gebeliğin potansiyel prognostik belirteci olarak kullanılabilir. Bu örnek, bu tekniğin potansiyel klinik uygulamalarını vurgulamaktadır. Çalışmamızda, genom davranışındaki değişiklikleri ve erken yaşlanma hastalığı Hutchinson-Gilford Progeria Sendromu'nda (HGPS) spesifik ilaç tedavilerinin etkisini değerlendirmek için HALO-FISH'i kullandık15.
Bunlar ve diğer çalışmalar birlikte, DNA halo tekniğinin tekniğin incelenmesi ve faydası için kullanılabileceği süreçlerin / uygulamaların genişliğini vurgulamaktadır.
Protokol
1. Slayt hazırlama, sterilizasyon ve hücre kültürü
- 500 mL% 10 HCl (v / v) hazırlayın ve büyük bir beher içine dökün.
- Damla mikroskobu ayrı ayrı aside kayar ve 2 x g'de ayarlanmış bir çalkalayıcıda oda sıcaklığında 1 saat inkübe edin.
DİKKAT: HCl aşındırıcı ve tahriş edicidir. Ciddi cilt yanıklarına ve göz hasarına ve cildin, gözlerin ve solunum sisteminin tahriş olmasına neden olabilir. Nitril eldivenler, göz koruması ve laboratuvar önlüğü dahil olmak üzere uygun kişisel korumanın giyildiğinden emin olun. - Beherden asit alın ve yıkayın, musluk suyunda on kez ve daha sonra deiyonize suda on kez daha kayar.
- Slaytları metanol içinde iki kez durulayın ve alevlendirilerek sterilize edilene kadar metanol içinde tutun.
DİKKAT: Metanol yutulduğunda, ciltle temas ettiğinde veya solunduğunda oldukça yanıcı bir sıvı ve toksiktir. Ek olarak, metanol organlara zarar verebilir, aşındırıcı ve tahriş edicidir. İşyerinde maruz kalma sınırlarına uyun ve bütil-lastik eldivenler, göz koruması ve laboratuvar önlüğü dahil olmak üzere uygun kişisel korumanın giyildiğinden emin olun. Mümkün olduğunda yerel egzoz havalandırma (LEV) duman dolabında tutun. - Metal maşalar veya uzun forseps kullanarak, metanol içeren beherden bir mikroskop slaytını çıkarın. Sterilize etmek için bir Bunsen brülörü üzerinde alev alın ve Bunsen brülörüne yakın bir yerde bulunan slaytlar için dört bölme içeren dikdörtgen bir hücre kültürü kabına aktarın.
DİKKAT: Alevlendirme, kullanımdan önce mikroskop slaytlarının derhal sterilize edilmesini sağlar; Bununla birlikte, bu yöntemin ilişkili tehlikeleri vardır. Metanol oldukça yanıcı olduğundan, slaytları içeren kabın Bunsen brülöründen uzağa yerleştirilmesi önemlidir. Slaytları sıkıca tutan uzun maşa veya forseps kullanılmalıdır. Beherdeki metanol seviyesi, hem kullanılan metanol miktarını en aza indirmek hem de forseps / maşaların sadece uçlarının metanol ile temas halinde olması için slaytları örtmelidir. Her zaman metanolün kullanımdan sonra forseps veya maşadan buharlaştığından ve slaytları ve metanolü içeren beherin içine geri yerleştirmeden önce bunların soğuduğundan emin olun. Beher, metanol alev alırsa oksijeni aç bırakmak için bir parça alüminyum folyo ile kaplanmalıdır. Havanın sirküle edildiği Sınıf II laminer akış davlumbazında asla alev kaymaz. - Alternatif olarak, 1.1 ila 1.4 arasındaki adımları uygulayın, ancak metanol ile inkübe ettikten sonra slaytları yakmak yerine, slaytları tüy bırakmayan doku üzerine hava kuruması için yerleştirin. Kuruduktan sonra, alüminyum folyoya sarın ve bir sterilizatör fırınına veya otoklavına yerleştirin.
- Hücreleri uygun ortamda büyütün, serum ile en az 48 saat boyunca 37 °C'de %5 CO2'de %60-70 akıcılığa ulaşıncaya kadar. Bu protokol, insan dermal fibroblastlarının (HDF'ler) erken geçişi ve klasik Hutchinson-Gilford progeria sendromu (HGPS) fibroblastları (AG06297) ve atipik tip 2 HGPS fibroblastları (AG08466) üzerinde uygulandı. Her hücre tipini hasat edin ve hücre yoğunluğunu belirlemek için bir hemositometre kullanarak sayın. Tohum 1 x 10 slayt başına 10 mL ortamda5 hücre.
NOT: Hücre yoğunluğu önemlidir, çünkü hücreler çok birleşirse farklı çekirdeklerden DNA döngüleri birleşebilir. Tohumlama yoğunluklarının, kullanılan hücre tipine bağlı olarak optimize edilmesi gerekebilir, çünkü dönüştürülmüş hücreler daha hızlı çoğalabilirken, daha sonraki geçiş hücre kültürlerinin istenen akıcılığa ulaşması daha uzun zaman alabilir. - Hücrelerin sessiz hale gelmesi için G0'da tutuklanması gerekiyorsa, slayt başına 1 x 105 hücre (10 mL ortamda) tohumlayın ve 24 saat boyunca büyümeye bırakın. Hücreleri serumsuz ortamla iki kez yıkayın ve 7 gün boyunca% 0.5'te daha düşük bir serum konsantrasyonu (yenidoğan buzağı serumu, NCS; veya fetal sığır serumu, FBS) içeren standart ortamda inkübe edin.
- DNA Halo testi için hücrelerin proliferatif durumu gerekliyse, replikasyon sırasında DNA'ya 5-bromo-2'-deoksi-idrar (BrdU) dahil edilerek S-Fazından geçen hücreleri belirleyin.
- Tohum hücreleri normal olarak ve 24 saat boyunca büyür. Kültür ortamını çıkarın ve BrdU ve 5-floro-2'-deoksiüridin (3 μg / μL) içeren ortam ile değiştirin. 24 saat sonra, ortamı çıkarın, hücreleri bir kez orta (% 10 NCS) ile yıkayın ve ardından taze ortamla (% 10 NCS) yeniden besleyin. Ek bir 24 saat boyunca inkübe edin ve ardından DNA halo testi için slaytları hazırlayın.
2. Prob hazırlama
- Kromozom bütün ve kol boyama probları
- Telenius ve ark.16 tarafından yöntemi kullanarak, dejenere oligonükleotid astarlanmış polimeraz zincir reaksiyonu (DOP-PCR) ile akış sıralanmış veya mikrodisseke kromozomların amplifikasyonundan kromozom probları yapın. Kromozom problarını Tablo 1'de gösterildiği gibi Biotin-16-dUTP veya Digoxigenin-11-dUTP ile etiketlemek için DOP-PCR kullanın. Lütfen amplifikasyon profili için üreticinin talimatlarını kontrol edin, ancak bu deney için kullanılan koşullar Tablo 2'de gösterilmiştir.
- 8 μL etiketli PCR ürünü, 7 μL Cot-1 DNA, 3 μL ringa balığı spermi, 1/20inci hacim 3 M sodyum asetat (pH 5.4) ve 2 hacim% 100 etanol ekleyerek kol veya tüm kromozom probunu hazırlayın. Prob solüsyonunu -80 ° C'de en az 30 dakika inkübe edin.
NOT: Bu yöntem, ilgilenilen her kromozom için farklı etiketler (yani, Biotin-16-UTP ve Digoxigenin-11-dUTP) kullanılıyorsa, tek kromozom probları veya çoklu kromozom probları oluşturmak için kullanılabilir.
DİKKAT: Etanol oldukça yanıcı bir sıvı ve buhardır ve ciddi göz tahrişine neden olabilir. Isıdan, sıcak yüzeylerden ve ateşleme kaynaklarından uzak tutun. İşyerinde maruz kalma sınırlarına uyun ve bütil-lastik eldivenler, göz koruması ve laboratuvar önlüğü dahil olmak üzere uygun kişisel korumanın giyildiğinden emin olun. Mümkün olduğunda bir LEV duman dolabında tutun. - Santrifüj probu çözeltisini 13.700 x g'de 4 °C'de 15 dakika boyunca santrifüj probu çözeltisi ve ardından %70 etanol ile yıkayın. Santrifüjleme prosedürünü tekrarlayın ve DNA peletini rahatsız etmemeye veya kaybetmemeye dikkat ederek süpernatanı atın. DNA peletinin kurumasını bekleyin.
- DNA peletine 12 μL hibridizasyon tamponu (%50 formamid, %10 dekstran sülfat, %10 20x salin sodyum sitrat (SSC; 3 M NaCl, 0,3 M tri-sodyum sitrat; pH 7.0), %1 (v/v) polioksietilen sorbinal monolaurat (Tween-20)) ekleyin. DNA peletinin hibridizasyon tamponunda çözünmesi için 37 ° C'de en az 2 saat bekletin.
DİKKAT: Formamid kanserojen ve teratojeniktir, bu nedenle doğmamış bir çocuğa ciddi zararlar verebilir. Bir kadın hamileyse veya hamile olduğundan şüpheleniyorsa, formamid ile çalışmaktan kaçınmalıdır. Formamid, bir LEV duman davlumbazında kullanılmalıdır. İşyerinde maruz kalma sınırlarına uyun ve bütil-lastik eldivenler, göz koruması ve laboratuvar önlüğü dahil olmak üzere uygun kişisel korumanın giyildiğinden emin olun.
- Bakteriyel Yapay Kromozomlardan (BAC'ler) DNA izolasyonu
- BAC klonundan gliserol stoğunun küçük bir kısmını Luria-Bertani (LB) agar plakasına (% 1 (W / V) NaCl; % 1 (w / v) tripton,% 0.5 (w / v) maya ekstraktı,% 1.5 (w / v) Agar Teknik, 12.5 μg / mL (w / v) kloramfenikol) üzerine çizin. Gece boyunca 37 ° C'de inkübe edin.
- Plakadan tek bir koloni seçin ve 10 mL LB suyu (% 1 (w / v) NaCl,% 1 (w / v) baktotripton,% 0.5 (w / v) maya ekstresi, 12.5 μg / mL (w / v) kloramfenikol) aşılayın). Çözeltiyi gece boyunca 37 ° C'de çalkalayan bir inkübatörde bırakın.
DİKKAT: Kloramfenikolün kansere neden olduğundan şüphelenilmektedir. Dikkatli kullanın ve maruziyeti azaltın. - Oda sıcaklığında 10 dakika boyunca 1.700 x g'de santrifüj kültürü.
- Süpernatantı atın ve peletlere 300 μL P1 çözeltisi (15 mM Tris (pH 8), 10 mM EDTA, 100 μg / mL RNase A) ekleyin. Vorteks kuvvetli bir şekilde ve hücreleri 2 mL'lik bir mikrosantrifüj tüpüne aktarın.
- Hücrelere damla damla 300 μL P2 çözeltisi (0.2 M NaOH,% 1 (w/v) sodyum dodesil sülfat (SDS) ekleyin. Kapalı mikrosantrifüj tüpünü 5 kez ters çevirin ve oda sıcaklığında maksimum 5 dakika bekletin.
DİKKAT: Sodyum hidroksit aşındırıcıdır ve ciddi cilt yanıklarına ve göz hasarına neden olabilir. Metaller için aşındırıcı olabilir. Dikkatli kullanın ve maruziyeti azaltın. İşyerinde maruz kalma sınırlarına uyun ve nitril-lastik eldivenler, göz koruması ve laboratuvar önlüğü dahil olmak üzere uygun kişisel korumanın giyildiğinden emin olun. Mümkün olduğunda bir LEV duman dolabında tutun.
DİKKAT: Sodyum dodesil sülfat yanıcı bir katıdır, yutulduğunda zararlıdır ve cilt ve solunum tahrişine neden olabilir. Ayrıca ciddi göz hasarına neden olabilir. Nitril-lastik eldivenler, göz koruması ve laboratuvar önlüğü dahil olmak üzere uygun kişisel korumanın giyildiğinden emin olun. Mümkün olduğunda bir LEV duman dolabında tutun. - Hücrelere yavaşça 300 μL P3 (3 M potasyum asetat) ekleyin ve yavaşça karıştırın. Mikrosantrifüj tüpünü 10 dakika boyunca buzun üzerine yerleştirin.
- 4 ° C'de 10 dakika boyunca 8.100 x g'de santrifüj yapın ve süpernatantı 800 μL buz gibi soğuk izopropanol içeren bir tüpe aktarın. Tüpü birkaç kez ters çevirin ve gece boyunca -20 ° C'de inkübe edin.
DİKKAT: İzopropanol oldukça yanıcı bir sıvı ve buhardır ve ciddi göz tahrişine, uyuşukluğa veya baş dönmesine neden olabilir. Isıdan, sıcak yüzeylerden ve ateşleme kaynaklarından uzak tutun. İşyerinde maruz kalma sınırlarına uyun ve nitril-lastik eldivenler, göz koruması ve laboratuvar önlüğü dahil olmak üzere uygun kişisel korumanın giyildiğinden emin olun. Mümkünse bir LEV) duman dolabında tutun. - 4 °C'de 15 dakika boyunca 8.100 x g'de santrifüj. Süpernatantı çıkarın ve başka bir tüpe aktarın. 500 μL buz gibi soğuk% 70 etanol ekleyin. Tüpü birkaç kez ters çevirin ve 4 ° C'de 5 dakika boyunca 8.100 x g'de santrifüjleyin.
- Süpernatantı çıkarın ve peleti oda sıcaklığında hava ile kurutun. Pelet kuruduktan sonra, 40 μL dietil pirokarbonatla arıtılmış suda (DEPC ile muamele edilmiş) tekrar askıya alın ve gece boyunca 4 ° C'de bırakın. Tamamen yeniden askıya alındıktan sonra, 5 μL çözeltiyi çıkarın ve DNA'nın varlığını kontrol etmek için% 1'lik bir agaroz jeli üzerine yükleyin.
- Nick translasyonu ile BAC'lerin tek gen probu hazırlanması
- Piyasada satılan takma ad çevirisi etiketleme kitlerini kullanın. Alternatif olarak, aşağıdaki protokolü kullanın. Bileşenler ve ciltler için Tablo 3'e bakınız.
- Tablo 3'teki bileşenleri, DNA Polimeraz I'i ekleyerek bir mikrosantrifüj tüpünde bir araya getirin, yavaşça karıştırın ve birkaç saniye boyunca kısaca santrifüj yapın. Çözeltiyi 2 saat boyunca 15 ° C'de inkübe edin.
- Parça boyutlarını doğrulamak için, çözeltinin 5 μL'sini% 2'lik bir agaroz jeli üzerine yükleyin. DNA fragman boyut aralığı 200-600 bp arasında olmalıdır. DNA fragmanı boyutları daha büyükse, çözeltiyi 15 ° C'de 15 dakika daha inkübe etmeye devam edin ve% 2 agaroz jeli üzerinde ürünleri çalıştırın.
- 10 mM EDTA, %0,1 SDS (2,5 μL 0,5 M EDTA, 100 μL'de pH 8,0 ve 100 μL'de 1 μL %10 SDS) ekleyerek çentik çeviri reaksiyonunu durdurun. Çözeltiyi 65 ° C'de 5 dakika ısıtın.
- Dahil edilmemiş nükleotidleri çıkarmak için BAC probunu bir spin sütununa uygulayın. Ticari spin sütunları satın alınabilir veya aşağıdaki gibi bir şırınga kullanılarak oluşturulabilir:
- 500 mL sütun tamponuna 30 g Sephadex G-50 ekleyin (10 mM Tris-HCl (pH8), 1 mM EDTA, %0,1 SDS). Karışımı otoklavlayın. Ayrıca, 500 mL sütun tamponu (Sephadex G-50 olmadan) ve otoklav yapın.
- 1 mL'lik bir şırınganın dibine otoklavlanmış cam yünü ekleyerek spin sütunları yapın. 1 mL şırıngayı kolon tamponunda Sephadex G-50 ile doldurun. 1 mL şırıngayı, dibinde kapağı olmayan bir mikrosantrifüj tüpüne sahip 15 mL'lik bir santrifüj tüpüne yerleştirin. 5 dakika boyunca 1.600 x g'de santrifüj.
- Şırıngayı çıkarın ve alttaki mikrosantrifüj tüpünü atın. 15 mL santrifüj tüpüne yeni bir mikrosantrifüj tüpü ekleyin. 1 mL şırıngaya kolon tamponu (Sephadex G-50 olmadan) ekleyin ve 15 mL santrifüj tüpüne tekrar takın. 5 dakika boyunca 1.600 x g'de santrifüj. Bu adımı iki kez tekrarlayın.
- Şırıngayı çıkarın ve yeni bir temiz mikrosantrifüj tüpü içeren 15 mL'lik bir santrifüj tüpüne yerleştirin. Şırıngaya prob uygulayın ve mikrosantrifüj tüpünde probu toplayın.
- DNA probunu çökeltmek için DNA çözeltisine 5 μL ringa balığı sperm DNA'sı (10 mg / mL), 10 μL sodyum asetat ve 2.25 hacim% 100 etanol ekleyin. Çözeltiyi yavaşça karıştırın ve en az 1 saat boyunca -80 ° C'de inkübe edin. 4 °C'de 15 dakika boyunca 13.700 x g'de santrifüj.
- Süpernatantı atın ve peleti 4 ° C'de 15 dakika boyunca 200 μL buz gibi soğuk% 70 etanol ile yıkayın. Süpernatantı çıkarın ve havayı kurutun. Kuruduktan sonra, pelet 20 μL DEPC ile arıtılmış suda oda sıcaklığında birkaç saat veya gece boyunca 4 ° C'de tekrar askıya alın. Prob artık kullanıma hazırdır veya -20 °C'de saklanabilir.
- Her slayt için 5 μL prob DNA'sını 5 μL Cot-1 DNA ile karıştırın ve bir Speed Vac vakum yoğunlaştırıcısı kullanarak kurutun. Pelet kuruduktan sonra 12 μL hibridizasyon karışımında tekrar askıya alın.
3. DNA Halo hazırlığı
- Slaytları ve ekli hücreleri içeren kare kültür kabını inkübatörden çıkarın. Ortamı atın, slaytları bir kalem kullanarak etiketleyin ve 50 mL buz gibi soğuk hücre iskeleti (CSK) tamponu içeren bir Coplin kavanozuna yerleştirin: 100 mM NaCl, 3 mM MgCl2, 0,3 M sakaroz, 10 mM 1, 4-piperazinedietansülfonik asit (PIPES; pH 7.8), % 0.5 (v / v) Triton X-100 deiyonize suda oluşur. Buz üzerinde veya 4 ° C'de 15 dakika inkübe edin.
DİKKAT: Triton X-100 cilt tahrişine ve ciddi göz hasarına neden olabilir. Nitril eldivenler, gözlükler ve laboratuvar önlüğü dahil olmak üzere uygun kişisel koruyucu ekipman kullanarak kullanın. - CSK tamponunu atın ve slaytları 50 mL 1x DNA halo tamponunda (DHB; 140 mM NaCl, 27 mM KCl, 110 mM NaHPO 4, 15 mM KH2PO 4; pH7.4) üç kez hızlı bir şekilde durulayın, yani DHB içeren Coplin kavanozuna daldırın ve çıkarın.
- Slaytları 50 mL ekstraksiyon tamponu içeren bir Coplin kavanozuna aktarın: 2 M NaCl, 10 mM BORULAR (pH 6.8), 10 mM etilendiamintetraasetik asit (EDTA), %0.1 (w/v) digitonin, 0.05 mM (v/v) spermin, 0.125 mM (v/v) spermidin. Oda sıcaklığında 4 dakika inkübe edin.
DİKKAT: Digitonin yutulduğunda veya ciltle temas ettiğinde toksiktir ve solunduğunda ölümcüldür. Digitoninin bir LEV duman dolabında kullanıldığından emin olun ve laboratuvar önlüğü, nitril eldiven (çift eldivenli), güvenlik gözlüğü ve maske takın. Hem spermin hem de spermidin ciddi cilt yanıklarına ve göz hasarına neden olabilirken, EDTA ciddi göz tahrişine neden olur, bu nedenle her kimyasalı dikkatli kullanın.
NOT: Tozu suda 60-70 °C sıcaklıkta çözerek digitonini ayrı ayrı hazırlayın. Soğutulduktan sonra ekstraksiyon tamponuna çözünmüş digitonin ekleyin. Biyolojik aktiviteyi korumak için spermin, spermidin ve digitonin en son ekstraksiyon tamponuna ekleyin. - Slaytları ardışık olarak 50 mL 10x DHB (1,4 M NaCl, 270 mM KCl, 1,1 M NaHPO 4, 150 mM KH2PO4; PH7.4), her biri 1 dakika boyunca 5x, 2x ve 1x DHB.
- %10, %30, %70 ve %95 (v/v) etanolden oluşan 50 mL sıralı etanol serisinden daldırma kayar (düz giriş ve düzlük).
- Hava kaydırakları kurutun ve iki boyutlu floresan in situ hibridizasyon (2D FISH) yapılana kadar -80 °C'de saklayın.
4. İki boyutlu floresan in situ hibridizasyon
- 20x SSC yapın: 3 M NaCl, 0.3 M tri-sodyum sitrat, pH 7.0. Bu tampon otoklavlanabilir, oda sıcaklığında saklanabilir ve gerektiğinde seyreltilebilir.
- %70 (v/v) formamid, 2x SSC pH 7.0 yapın ve bir su banyosunda 70 °C'ye ısıtın.
- Slaytları, her biri 5 dakika boyunca, sıralı 50 mL etanol serisi boyunca% 70, 90 ve 100 etanolden inkübe edin.
- Hava ile kuruyan bir ısıtma plakasına kayar ve 70 °C'lik bir fırında 5 dakika pişirin.
- 70 ° C'de 2 dakika boyunca% 70 formamid, 2x SSC çözeltisine yerleştirilerek denatüre kızar.
NOT: Sıcaklık ve zamanlama adım 4.5 için kritik öneme sahiptir. Sıcaklık ise, DNA denatüre olmaz ve problar melezleşmez ve DNA halo FISH'ten sinyal alınamaz. - Denatüre edilmiş slaydı 5 dakika boyunca 50 mL buz gibi soğuk% 70 etanol içine yerleştirin ve her biri 5 dakika boyunca oda sıcaklığında% 90,% 95 ve% 100'lük bir etanol serisinden geçirin.
- Isıtma plakasında kuru hava
- Doğrudan etiketlenmiş toplam insan kromozom problarını üreticinin talimatlarına göre kullanın. Bu deneyler için insan tüm kromozom boyaları 1, 13, 15, 17 ve 18'i kullandık. Ek olarak, bu deneyde, CCND1 ve CTNNA1 gen probları kullanılmıştır.
NOT: Hem tüm kromozom probları hem de BAC genine özgü problar biotin-11-dUTP ile etiketlendi ve Siyanin 3'e (Cy3) konjuge streptavidin ile tespit edildi. Nick translasyonu ile etiketlenmiş (DOP-PCR) ve BAC DNA tarafından yapılan kromozom boyama probları için, bunlar protokolde bu noktadan itibaren DNA probları olarak adlandırılacak ve aşağıdaki gibi ele alınacaktır. - Denatüre DNA probu (tüm kromozom boyası veya gen-spesifik prob) 75 ° C'de bir sıcak blok veya su banyosunda 10 dakika boyunca.
- Sıcak DNA probları, uygun slayta 10 μL pipetlemeden önce bir sıcak blok veya su banyosunda 30 dakika boyunca 37 °C'de çalışır.
NOT: Bu adım, tekrarlayan kromozomal dizileri engellemek için önemlidir. Eğer yapılmazsa, DNA'da spesifik olmayan sinyaller üretilebilir: Halo FISH. - 21 mm x 21 mm kapak kaymalı kaplama probu ve kauçuk çimento kullanarak sızdırmazlık sağlar.
- Slaytları nemlendirilmiş bir hibridizasyon odasında 37 ° C'de en az 18 saat boyunca inkübe edin.
NOT: Nemlendirilmiş hibridizasyon odaları, birkaç kat nemlendirilmiş doku içeren sandviç kutulardan ve slaytları dinlendirmek için kesilmiş 10 mL plastik pipetlerden yapılmış yükseltilmiş bir platformdan yapılabilir. Bu, ışığa maruz kalmayı en aza indirmek için alüminyum folyo ile kaplanmıştır. - Forseps kullanarak kauçuk çimentoyu dikkatlice çıkarın.
- Slaytları, üç adet 5 dakikalık inkübasyon için 45 °C'ye önceden ısıtılmış 50 mL% 50 (v / v) formamid, 2x SSC, pH 7.0 çözeltisinde inkübe edin.
NOT: İlk inkübasyonda kapak kaymasının %50 (v/v) formamid, 2x SSC, pH 7.0 çözeltisi içinde slayttan düşmesine izin verin. Bu, kapağın "sürüklenmesinden" kaynaklanabilecek DNA Halo preparatının zarar görmesini önler. Kızaklar, kapak kaymasının ayrılmasına yardımcı olmak için forseps tarafından kavranarak tamponda çalkalanabilir. - Daha sonra, slaytları 60 ° C'ye önceden ısıtılmış ancak 45 ° C'lik bir su banyosuna yerleştirilmiş 50 mL 0.1x SSC, pH 7.0 çözeltisine yerleştirin. 5 dakika boyunca inkübe edin ve tamponu iki kez daha 5 dakikalık inkübasyonlarla değiştirin.
- Slaytları oda sıcaklığında 50 mL 4x SSC, pH 7.0 çözeltisi içeren bir Coplin kavanozuna yerleştirin ve üç tampon değişimi ile 15 dakika boyunca inkübe edin.
- Her slayta 100 μL% 4 BSA, 4x SSC çözeltisi uygulayın ve bir parça parafin film ile kaplayın. Oda sıcaklığında 10 dakika inkübe edin. Bu, antikorun spesifik olmayan bağlanmasını önler.
- Etiketli probu (biotin-16-dUTP) tespit etmek için, oda sıcaklığında 1 saat boyunca 1:200 (% 1 BSA, 4x SSC'de yapılmış) streptavidin-Cy3'ün 100 μL'si ile inkübe edin.
NOT: İyi bir sinyal üretildiğinden emin olmak için antikorların seyreltilmesi ve deneyden önce seyreltmeyi test ederek üreticinin talimatlarını izleyin - Slaytları oda sıcaklığında 50 mL 4x SSC (% 0.5 Ara-20) pH 7.0 çözeltisi içeren bir Coplin kavanozuna yerleştirin ve üç tampon değişimi ile 15 dakika boyunca inkübe edin. İmmünofloresan gerekli değilse, slaytlar bu aşamada adım 4.21'de gösterildiği gibi monte edilebilir.
- DNA halelerine dönüştürülen hücrelerin proliferatif durumu gerekiyorsa, FISH adımlarından sonra, montajdan önce veya dahil BrdU için boyamadan önce anti-pKi67 antikorları ile boyanır.
- Slaytları 50 mL 1x fosfat tamponlu salin (PBS) içinde her biri 5 dakika boyunca 3 kez yıkayın, ardından oda sıcaklığında 1 saat boyunca PBS'de% 4 NCS ile bloke edin.
- Slayta gerekli birincil antikorun (tavşan anti-insan pKi67; fare anti-BrdU) 200 μL'sini uygulayın, bir parafin film şeridi ile kaplayın ve oda sıcaklığında 1 saat boyunca inkübe edin.
- Slaytları 1x PBS'de 5 dakika boyunca 3 kez yıkayın ve oda sıcaklığında 200 μL florokrom konjuge ikincil antikor (pKi67: domuz anti-tavşan TRITC; BrdU: eşek anti-fare Cy3). PBS ile 3 adet daha 5 dakikalık yıkama yapın. Tüm seyreltmeler, üreticilerin önerdiği seyreltme aralığında PBS'de% 1 (v / v) NCS kullanılarak yapılmalıdır.
- Kızakları DAPI içeren 20 μL montaj maddesine ve 22 mm x 50 mm kapak kaymalı kaplamaya monte edin.
5. Telomer PNA BALIK
- Telomerleri tespit etmek için telomer PNA FISH kiti - FITC; Prosedürü üreticinin talimatlarıyla gerçekleştirin. Prosedür, aksi belirtilmedikçe, oda sıcaklığında yapılmalıdır.
- Slaytları 2 dakika boyunca tris tamponlu saline (TBS, pH 7.5) daldırın ve ardından tam olarak 2 dakika boyunca% 3.7 formaldehit (TBS'de; v / v) içine yerleştirin.
DİKKAT: TBS çözeltisi% 10-30 trometamol ve% 10-30 2-amino-2-(hidroksimetil) propan-1,3-diol hidroklorür içerir. Bu ciddi göz ve cilt tahrişine neden olabilir, bu nedenle koruyucu eldiven ve gözlük / yüz koruyucu kullanın. İyi havalandırılan bir alanda kullanın. - Slaytları bir Coplin kavanozunda her biri 5 dakika boyunca TBS ile iki kez yıkayın.
- Slaytları 10 dakika boyunca ön işlem çözeltisine daldırın ve ardından yıkama başına 5 dakika boyunca TBS ile iki kez yıkayın.
- Daha sonra, slaytları konsantrasyon başına 2 dakika boyunca 50 mL% 70,% 85 ve% 95 (v / v) etanolden oluşan buz gibi soğuk bir etanol serisinden geçirin. Daha sonra slaytların hava kurumasına izin verin.
- Floresan etiket renklendirme seçimine bağlı olarak 10 μL Telomer PNA Probu/FITC (veya Cy3) uygulayın, her slayta ve kaplamayı bir kapak kayması ile örtün. 5 dakika boyunca 80 ° C'de ayarlanmış önceden ısıtılmış bir fırında inkübe edin ve ardından yaklaşık 1 saat boyunca karanlıkta bırakın.
DİKKAT: Telomer PNA Probu / FITC, ciddi göz tahrişine neden olan ve teratojenik olan% 6-100 formamid içerir, bu nedenle doğmamış bir çocuğa ciddi zararlar verebilir. Bir kadın hamileyse veya hamile olduğundan şüpheleniyorsa, formamid ile çalışmaktan kaçınmalıdır. Bir LEV duman başlığında formamid kullanılmalı ve uygun göz veya yüz koruması giyilmelidir. - Kapakları çıkarmak için, slaytları 1 dakika boyunca 'Durulama Çözeltisi'ne daldırın ve ardından 65 ° C'de 5 dakika boyunca 'Yıkama Çözeltisi'ne yerleştirin.
DİKKAT: Yıkama çözeltisi% 1-5 polioksietilen oktil fenil eter ve% 1-5 sodyum klorür içerir. Bu aşındırıcıdır ve ciddi göz hasarına neden olabilir. Yıkama solüsyonunu kullanırken gözlük veya yüz koruyucu takıldığından emin olun. - 50 mL'lik buz gibi soğuk etanol serisinden (%70, %85 ve %95 (v/v)) geçerek konsantrasyon başına 2 dakika boyunca kavanozları inkübe edin ve ardından hava ile kurutun. Bir kez kuruduktan sonra DAPI içeren montaj maddesi ile sürgü ve bir kapak kayması ile bindirme.
6. Görüntü yakalama ve analiz
- DNA halelerini ve kromozom bölgelerini görselleştirmek için bir HC PL FLUOTAR 100X/1.30 yağ hedefi ve DFC365FX kamera kullanarak görüntü yakalayan bir epifloresan mikroskobu (örneğin, Leica DM4000 mikroskobu) kullanın.
- Gri tonlamalı görüntüler yakalayın ve görüntülerin sözde renklendirilmesini sağlamak için yakalanan her kanal için renk tanımlayın. Bu deneyde ticari bir yazılım kullanılmıştır (örneğin, LAS AF sürüm 4.5.0 yazılımı). Tek tek renk kanalları TIFF olarak dışa aktarıldı.
- Java görüntü işleme programı Fiji ImageJ'yi kullanarak görüntüleri analiz edin. Dosya ve Aç'a basarak resim yükleyin.
- Görüntü | Renk | Kanalları Bölme. Bir resim kanalı seçin ve Resim | Ayarlayın ve ardından Parlaklık ve Kontrast'ı seçin. Buna göre değiştirin ve diğer kanallarla tekrarlayın.
- Çekirdeği gösteren DAPI lekeli kanalı seçerek artık çekirdeğin bir maskesini oluşturun. Resme Tıklayın | Ayarlayın ve ardından Eşik'i seçin. Eşiğin değiştirilebileceği bir iletişim kutusu görünecektir, Koyu arka plan kutusunu işaretleyin. Artık çekirdek temizlenene kadar değiştirin ve Uygula'ya basıp iletişim kutusunu kapatın.
NOT: Bu, piksel yoğunluğuna bağlı olarak, ilgilenilen bölgeleri beyaz pikseller ve arka planı gösteren siyah piksellerle ikili bir maske oluşturur. Aynı prosedürü prob kanalında tekrarlayın. - Artık çekirdeğin çevresini ana hatlarıyla belirtmek için serbest seçimi kullanın, ardından Düzenle ve Dışarıyı Temizle'yi tıklayın. Prob kanalını artık çekirdeğe yerleştirin. Bu, Resim| Renk | Kanalları Birleştir.
- ImageJ'de ölçüm ölçeğini ayarlamak için, ölçek çubuğuna veya bilinen iki uzaklığın noktaları arasına bir çizgi çizin. Analiz'e gidin ve Ölçeği Ayarla'ya basın. İletişim kutusunda, mesafe uzunluğunu ekleyin ve Tamam'ı tıklatın. Mesafeleri ölçmek için, ölçülen noktalar arasında bir çizgi çizin ve Analiz Et | Ölçün. Bu, uzaklık değerlerini bir veri penceresine aktarır.
- Çekirdeğin merkezi ile çakıştığı için en parlak DAPI yoğunluğunu ölçün. Bu ölçümden nükleer merkezden en uzak kromozom bölgesi kenarına (CTE) olan mesafe. Nükleer merkezin nükleer kenara olan mesafesini ölçün (NE).
- Sonuçların CTE/NE oranı olarak gösterildiğinden emin olun. Burada nükleer merkezden her bir en uzak kromozom bölgesi kenarına (CTE) olan mesafe, nükleer merkezden her bir nükleer kenara (NE) olan mesafeye bölünür. Bu en az 50 çekirdek üzerinde yapılmalıdır. Bu bir çubuk veya kutu grafik olarak gösterilebilir.
- Telomerlerin analizi için, veri kümesi başına en az 30 çekirdeği analiz edin. Görüntüler, Fiji ImageJ kullanılarak veya artık çekirdek içindeki ve DNA halesi içindeki telomerlerin sayısını saymak için manuel olarak analiz edilebilir. BrdU veya pKi67, proliferasyon yapan (BrdU / piK67+) ve yaşlanan / sessiz (BrdU / pKi67-) çekirdeklerin farklılaşmasını sağladı. Veriler, ortalamanın standart hatasına (SEM) karşılık gelen hata çubuklarına sahip çubuk grafiklerde gösterilebilir.
- Sonuçları istatistiksel olarak anlamlı kabul edilen p> 0.05 ile karşılaştırmak için öğrencinin t-testini (eşleşmemiş) kullanın.
Sonuçlar
Bu DNA halo hazırlama yöntemi, genç ve yaşlı hücrelerdeki genom davranışındaki farklılıkları belirleme çabalarımızda ve aynı zamanda anormal nükleoskelet proteinlerine sahip erken yaşlanma hastalıklarından türetilen hücrelerde bize yardımcı olmuştur15. Şekil 1, artık çekirdeğin kenarını, artık çekirdeğin içinde kalan DNA'yı ve bir DNA halesi oluşturarak çevreye biriken bağlanmamış DNA'yı görmenin mümkün olduğu DNA halelerinin örneklerini göstermektedir. Ayrıca artık çekirdeğin nasıl elde edildiğini gösteren analizi ve NE ve CTE ölçümlerini de göstermektedir. Proliferasyon yapan ve olmayan hücreler arasında, hücreler S-fazındayken BrdU gibi etiketli bir nükleotid ekleyerek veya nükleolleri ortaya çıkaran tanısal proliferasyon belirteci anti-pKi67'yi ve G1 hücrelerinde heterokromatin bölgelerini kullanarak ayırt etmek mümkündür17,18. Yüksek serumda akıcılık sağlanmadan büyüyen, proliferasyon belirteçleri için negatif olan primer hücrelerin yaşlı olduğu varsayılır. Düşük serumda büyüyen veya konfluent hale gelen primer hücreler, yani proliferasyon belirteçleri için negatif olan temas inhibe edilmiş hücreler sessiz kabul edilir ve doğru besinler ve durum göz önüne alındığında proliferatif hücre döngüsüne tekrar girebilirler. Ki67 pozitif ve negatif hücreleri ayırt edebilmek, çoğalan, sessiz ve yaşlanan insan dermal fibroblastları arasındaki farkları belirlememizi sağlamıştır. Şekil 2, DNA replikasyonu sırasında BrdU'nun içine dahil edildiği hücrelerden oluşturulan çoğalan insan dermal fibroblastlarının DNA halelerini, çoğalmayan hücrelerde meydana gelmeyen ve daha sonra anti-BrdU antikoru ile boyanan bir mekanizmayı göstermektedir. Proliferatif belirteç anti-pKi67 antikoru ile boyanma da Şekil 2'de görülmektedir. Bu sağlam bir antijendir ve FISH protokolünden kurtulur ve bu nedenle FISH sonrası ve ön montaj için boyanabilir. Bu nedenle, çoğalan hücreler BrdU için pozitif (kırmızı) ve sol sütunda anti-pKi67 (kırmızı) ve çoğalmayan hücreler, aslında Şekil 2'deki yaşlanan hücreler sağ sütunda gösterilmektedir. Yeşil sinyaller, bir telomer PNA FISH / FITC kiti ile ortaya çıkarılan bireysel telomerlerdir. İmmünofloresansı DNA haleleri ile birleştirmek, çoğalan, sessiz ve yaşlanan hücreleri araştırırken Şekil 2'de gösterildiği gibi farklı hücre durumları sırasında analiz yapılmasını sağlar. Seçilen antikora bağlı olarak, farklılaşma, ışınlama yoluyla DNA hasarı gibi diğer koşullar incelenebilir.
Kromozom bölgeleri, FISH kullanılarak DNA haleleri içinde de görselleştirilebilir. DNA'nın çekirdeklerden dışarı atılmasına izin veren preparat nedeniyle, genomun artık çekirdeğin içindeki ankrajına ve yapılarına bağlı olarak, DNA halesinde bulunan kromozomun daha küçük veya daha büyük miktarları ile kromozom bölgesi şekli bozulabilir. Şekil 3 , kromozom 1, 13, 17 ve 18 için spesifik tam kol kromozomu boyama probları (kırmızı) ile bireysel kromozomların ortaya çıkarıldığı bir DNA haleleri panelini ortaya koymaktadır. Anti-pKi67 (yeşil), çoğalan hücreleri ve aynı kültür içindeki yokluğunu, aynı slayt üzerinde, yaşlanan hücreleri belirtmek için kullanılmıştır. CTE / NE olarak sunulan görüntülerden ve verilerden çok açıktır ki, küçük gen fakir kromozom 18, az sayıda bağlanmaya sahip olan ve artık çekirdeklerden uzakta DNA halesine daha fazla makaralanan ve artık çekirdeklerin merkezinden diğer kromozomlardan önemli ölçüde daha uzakta olan bir kromozomdur. Bununla birlikte, bu kromozom 1 için de geçerlidir. Proliferatif belirteç anti-pKi67'yi kullanarak, aynı kültür içinde ve aynı slaytta yaşlanan hücrelerle çoğalmayı karşılaştırmak da mümkün olmuştur ve bu analiz, bu iki çok farklı hücre durumundaki kromozomların, artık nükleer yapılara bağlanma açısından birbirlerinden önemli ölçüde farklı olmadığını ortaya koymuştur.
İlginçtir ki, genler ayrıca çoğalan ve yaşlanan hücreler arasında, artık bir çekirdek içinde kalma veya DNA Halesinde bulunma açısından istatistiksel olarak anlamlı farklılıklar göstermektedir. Şekil 4 bunu, kırmızı renkte etiketli BAC probları ve yeşil renkte anti-Ki67 ile tanımlanan gen lokusları ile göstermektedir. Bir DNA Halo preparatından sonra, çoğalan hücrelere karşı yaşlanan hücrelerdeki gen konumları arasında anlamlı bir fark yoktur. Bununla birlikte, DNA halesi içinde, çok az sayıda bulunan siklin D1 CNDD1 lokuslarından önemli ölçüde daha fazla katenin alfa 1 CTNNA1 lokusu vardır. Şekil 5, yeşil renkte telomerlerle DNA halo preparatlarını göstermektedir. Arka plan, telomer sinyallerinin DNA halesi içinde görselleştirilmesini sağlamak için kasıtlı olarak yüksek bırakılmıştır. Bu veri kümesine sessiz hücreler, yani 7 gün boyunca serum açlığı çeken hücreler dahil edilmiştir ve ilginç bir şekilde, sessiz hücrelerde DNA halelerinde bağlanmamış ve yerleştirilmiş, çoğalan ve yaşlanan hücrelerden çok daha fazla telomer vardır. Şekil 5a'da, DNA halesindeki telomerlerin oranı, özellikle 'Deney 2' görüntüsü için gözlemlenebilir. Bu, DNA halesindeki telomerlerin ortalama yüzdesinin sessiz hücrelerde yaklaşık% 17 olduğu Şekil 5b'ye karşılık gelir. Yaşlanan hücrelerdeki tüm telomerlerin bazılarının belki de çok kısa olarak görülemeyeceğine dair bazı kanıtlar vardır.
Bu DNA halo yöntemi, hastalıklı hücrelerdeki çekirdeklerdeki genom etkileşimi değişikliklerini araştırmamız için başarılı olmuştur15. Şekil 6 , primer kontrol fibroblastlarında ve tipik (lamin A mutasyonu) ve atipik Hutchinson-Gilford Progeria Sendromu olan hastalıklı hücrelerde kromozom bağlanmasındaki farklılıkları göstermektedir, farklı bir SUN1 izoformu eksprese eder ve lamin A mutasyonuyoktur 19. Kromozom 1 ve 13, kontrol DNA halelerine kıyasla artık çekirdekler içindeki bağlanmalarında istatistiksel olarak anlamlı farklılıklar göstermektedir. Şekil 6b , tüm kromozom bölgesinin konumunu artık çekirdek ve DNA Halosu ile ilişkilendirir. 1 veya daha düşük değerler, kromozomun artık çekirdek içinde bulunduğunu ve 1'in üzerindeki değerlerin DNA Halosu içindeki kromozomları veya kromozom kısımlarını gösterdiğini gösterir.
Genel olarak, bu, HALO-FISH'in tüm kromozomların, spesifik genlerin ve telomerlerin genomik etkileşimlerini, hücre döngüsünü etkileyen çeşitli koşullar altında (proliferasyon, sessizlik ve yaşlanma) veya hastalık hücreleri içinde, örneğin progeria ve kanser hücre hatları içinde araştırmadaki faydasını vurgulamaktadır. Gerçekten de, bu durumlar arasındaki etkileşimlerdeki farklılıklar, nükleoiskeletin çekirdek içindeki anahtar süreçleri düzenlemede önemli bir role sahip olduğunu ima eder.

Şekil 1: Artık çekirdeği ve DNA halesini gösteren HDF ekstrakte edilmiş çekirdek ve analiz yöntemine genel bakış. (a) DNA halo testi ile hazırlanan ve DAPI ile boyanan bir HDF çekirdeği. Parlak lekeli artık çekirdek, nükleoiskelete bağlanmış DNA'yı gösterir ve bu, DNA'nın bir halesini oluşturan bağlanmamış DNA ile çevrilidir. Büyütme = x 100; ölçek çubuğu 10 μm. (b) Mavi kanal, DAPI lekeli çekirdeği ve çevresindeki DNA'yı yakalar. Artık çekirdek ImageJ kullanılarak seçilir ve çıkarılır. Ok, nükleer merkezden artık nükleer kenara (NE) olan mesafeyi göstermektedir. (c) Kırmızı kanal prob sinyalini gösterir. (d) 'Sonuç' olarak gösterilen görüntü, kırmızı kanalın mavi kanal görüntüsünün üzerine bindirilmesinin sonucudur; bu, nükleer merkezden en uzak kromozom bölgesi kenarına (CTE) olan mesafeyi sağlar. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
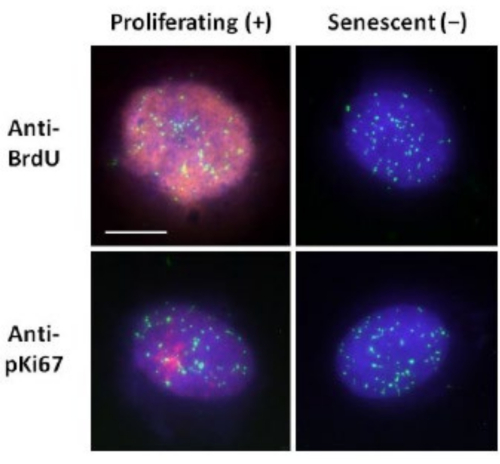
Şekil 2: Proliferasyon ve yaşlanan HDF'lerde telomer PNA FISH ile DNA halo preparasyonu. Telomer PNA FISH DNA halo testine tabi tutulan HDF'lerde. Telomer sinyalleri yeşil (FITC) renkte görselleştirildi, artık ve halo DNA'sı DAPI (mavi) kullanılarak boyandı ve proliferasyon yapan çekirdekler kırmızı (TRITC) dolaylı immünofloresan yoluyla anti-BrdU veya anti-pKi67 antikorları kullanılarak tespit edildi. Büyütme = x 100; ölçek çubuğu 10 μm. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
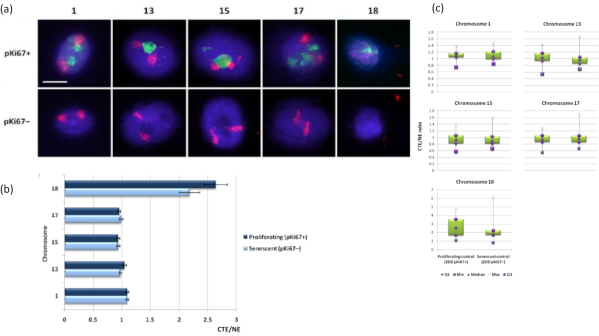
Şekil 3: Nükleoiskelet-kromozom etkileşimleri ve DNA halo testi kullanılarak analiz. (a) DNA halo preparasyonuna tabi tutulan HDF'ler üzerinde 1, 13, 15, 17 ve 18 numaralı kromozomlara özgü problarla 2D-FISH uygulandı. Tüm kromozomlar kırmızıya (Cy3) boyandı ve çekirdekler çoğalan veya yaşlanan olup olmadıklarını belirlemek için pKi67 ile incelendi. Proliferasyon yapan hücreler (pKi67+) yeşil (FITC) ile tanımlanırken, yaşlanan hücreler lekesiz kaldı (pKi67-), yani yeşil sinyal tespit edilmedi. Büyütme = x 100; ölçek çubuğu 10 μm. (b) HALO-FISH geçirmiş olan çoğalan ve yaşlanan HDF'lerde nükleoiskelet tarafından kromozom ankrajı. Ölçümler, çoğalan (pKi67+) ve yaşlanan (pKi67-) hücrelerde 1, 13, 15, 17 ve 18 numaralı kromozomlar için en uzak kromozom bölge kenarının (CTE) ilgili nükleer kenara (NE) oranını göstermektedir. Hata çubukları ± SEM'i temsil eder. (c) Kromozom bölge kenarının (CTE) pKi67+ ve pKi67- çekirdeklerindeki spesifik kromozomların ilgili nükleer kenarına (NE) modifiye kutu grafiği gösterimi. Q1 = alt çeyrek; Min = kaydedilen en düşük değer; Med = medyan; Maksimum = kaydedilen maksimum değer; Q3 = üst çeyrek. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
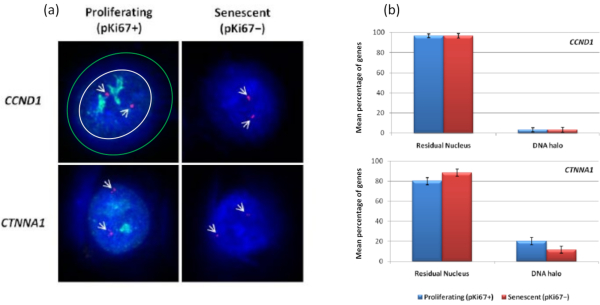
Şekil 4: HALO-FISH kullanan HDF'lerde genlere özgü etkileşimler. (a) DNA halesi ekstrakte edilen çekirdekler, çoğalan ve yaşlanan hücreler üzerinde NM'ye ankrajlarını araştırmak için gen spesifik problarla (CCND1 ve CTNNA1) araştırıldı. Gen sinyalleri kırmızı (Cy3) ile gösterilir ve anti-pKi67 çoğalan hücreleri gösterir ve sinyal yeşil (FITC) ile görselleştirilir. Çoğalan CCND1 görüntüsü için, artık çekirdek beyaz daire içine alınır ve beyaz ve yeşil daire arasındaki boşluk DNA Halesini gösterir. Büyütme = x 100; ölçek çubuğu 10 μm. (b) CCND1 ve CTNNA1 için genlere özgü sinyaller, artık çekirdek ve DNA halesi arasında ve ayrıca çoğalan ve yaşlanan hücreler arasında karşılaştırılır. Hata çubukları SEM±i temsil eder. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
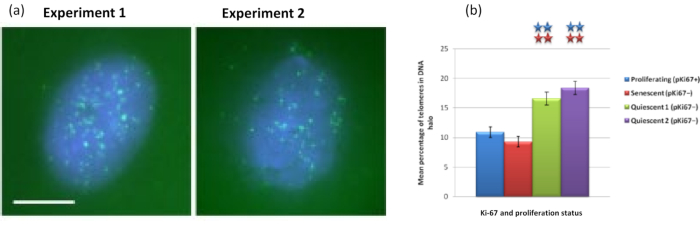
Şekil 5: Telomer PNA-FISH ile araştırılan sessiz HDF'ler üzerinde DNA halo testi. (a) HDF'lerin sessizleşmesi, 7 gün boyunca düşük serum ortamında kültür tarafından indüklendi. DNA halo testi yapıldı ve PNA-FISH, TELOMERLERIN FITC sinyali (yeşil) ile görselleştirilmesini sağladı ve artık çekirdek ve çevresindeki DNA halesi DAPI (mavi) ile boyandı. Hücreler ayrıca çekirdeklerin çoğalmadığından emin olmak için anti-pKi67 antikoru ile boyandı. Bu iki ayrı durumda tekrarlandı. Büyütme = x 100; ölçek çubuğu 10 μm. (b) Proliferasyon, yaşlanan ve sessiz HDF hücrelerinde DNA halesi içinde lokalize olan telomerlerin ortalama yüzdesinin karşılaştırılması. Hata çubukları SEM± temsil eder. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
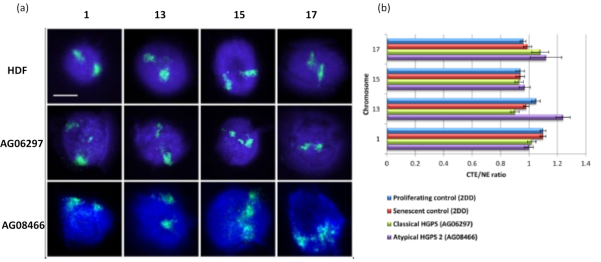
Şekil 6: HGPS hücrelerinde nükleoiskelete tüm kromozom ankrajının HALO-FISH26 kullanılarak incelenmesi. (a) Kontrol HDF (2DD), klasik HGPS (AG06297) ve atipik tip 2 HGPS (AG08466) çekirdeklerine DNA halo hazırlığı ve daha sonra kromozom 1, 13, 15 ve 17 için tüm kromozom boyaları kullanılarak 2D-FISH uygulandı. Tüm kromozomlar yeşil (FITC) ile tasvir edilmiştir ve DNA DAPI (mavi) ile boyanmıştır. Büyütme = x 100; ölçek çubuğu 10 μm. (b) Kromozomların ekstrakte edilen çekirdekler içindeki konumlandırılması, ortalama kromozom bölge kenarının (CTE) nükleer kenara (NE) oranı ölçülerek belirlendi. 1'in üzerindeki bir oran, en uzak CTE'nin DNA halesi içindeki karşılık gelen NE'nin dışında olduğunu gösterirken, 1'in altındaki bir oran, en uzak CTE'nin artık çekirdek içindeki NE içinde bulunduğunu gösterir. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
| Bileşen | Hacim (μL) |
| 5XDOP-PCRtampon | 10 |
| dNTPmix(olmadandTTP)(2mM) | 5 |
| dTTP(2mM) | 2 |
| Biotin-16-dUTPorDigoxigenin-11-dUTP | 10 |
| DOPprimer (20μM) | 5 |
| TaqDNAPolimeraz (1U / μL) | 1 |
| PCRgradewater | 12 |
| Şablon | 5 |
Tablo 1: 1x reaksiyon için DOP-PCR bileşenlerini ve hacimlerini gösteren tablo
| Adım | Döngü | Sıcaklık (derece Santigrat) | Saat |
| İlk Denatürasyon | 1 | 95 | 3 dk |
| Denatürasyon | 34 | 98 | 20 saniye |
| Astar Tavlama | 62 | 1 dk | |
| Uzantı | 72 | 30 saniye | |
| Son Uzatma | 1 | 72 | 5 dk |
| Soğutma | 4 | Tutmak |
Tablo 2: DOP-PCR döngüsünü, sıcaklığını ve zaman profilini gösteren tablo.
| Unsur | Hacim (μL) |
| 10x NT tampon (0,5 M Tris-HCl pH 8,50 mM MgCl2, 0,5 mg/ml BSA) | 5 |
| 0.1 M beta-merkaptoetanol | 5 |
| 10X Nükleotid stoğu (0,5 mM dATP, 0,5 mM dCTP, 0,5 mM dGTP, 0,5 mM dTTP, 0,5 mg/ml biotin-16-dUTP) | 5 |
| Dnase I (1 ng/ml) | 2 |
| DNApolimeraz I | μg DNA başına 5U |
| DNAtemplate (1 μg) | 1 |
| DEPC ile arıtılmış su | 50 μL'ye kadar |
Tablo 3: Bir prob için nick çeviri bileşenlerini ve ciltlerini gösteren tablo.
Tartışmalar
DNA halo yöntemi, nükleoiskelet ve genom arasındaki etkileşimleri analiz ederken mükemmel bir seçim yöntemidir, ancak uyulması gereken bazı kritik adımlar da vardır. En önemli parametrelerden biri, hücre tohumlama yoğunluğunun optimizasyonudur. Hücreler aşırı birleşirse, DNA haleleri komşu hücrelerle üst üste binecek ve bu da analizin yapılmasını imkansız hale getirecektir. CSK ve ekstraksiyon tamponları, biyolojik aktivitelerini korumak için hazırlama işleminin sonunda ekstraksiyon tamponuna spermin, spermidin ve digitonin eklenerek kullanım gününde her zaman taze yapılmalıdır. Halo-FISH yapıyorsanız, probun veya boyanın daha sonra hibritleşmesini sağlamak için DNA halelerinin doğru denatürasyon sıcaklığını kullanmak son derece önemlidir.
Elektron mikroskobu, nükleer matrisi görselleştirmek için kullanılmıştır, filamentli yapılar tanımlanmıştır20. Bununla birlikte, elektron mikroskobu sınırlıdır, çünkü kromatin ile matris ilişkileri kolayca çıkarılamaz. Gerçekten de, DNA Halo yöntemi, elektron mikroskobu ile karşılaştırıldığında daha çok yönlüdür, çünkü spesifik genler, kromozomlar ve hücre durumları incelenebilir. Ayrıca, nükleer matriks proteinlerinin proteomik analizi incelenmektedir21,22. Bu yöntem, özellikle hastalıklı hücreleri karşılaştırırken, nükleer matris bileşenlerini karşılaştırmak için iyidir, ancak standart DNA Halo tekniği tarafından vurgulanan uzamsal dağılımı ve ekleri sağlamaz.
DNA Halo tahlillerinin sınırlamaları vardır. İlk olarak, matris çıkarılırken, bu sadece sabit hücreler üzerinde gerçekleştirilebilir, bu nedenle canlı görüntüleme mümkün değildir. DNA Halo yönteminin gerçekleştirilmesi nispeten hızlı ve kolay olmasına rağmen, hücre kültürü, prob üretimi, Halo-FISH ve analiz dikkate alındığında genel süreç zaman alıcı olabilir.
Süper çözünürlüklü mikroskopi kullanılarak DNA Haloları ve HALO-FISH'in görüntü yakalaması, DNA'ya özgü probların ve antikorların çözünürlüğünü büyük ölçüde artıracaktır. Ek olarak, florokromlar spektral olarak daha kolay çözülebildiğinden, tek bir deneyde bir dizi DNA probu kullanmak mümkün olabilir ve bu da daha fazla bilgi sağlar. Kromozom konformasyon yakalama (3C) gibi moleküler biyoloji tekniklerindeki gelişmeler, gen lokuslarının etkileşimlerini belirlemek ve hücredeki kromatin üzerindeki mekansal organizasyonu analiz etmek için kullanılmıştır. DNA Halo testleri ve 3C, M3C23 olarak bilinen bir terim olan birleştirilebilir ve yine DNA Halo tekniğinin uyarlanabilirliğini gösterir.
Burada sunulan orijinal veriler, genom davranış sorgulaması olanaklarını ve bu verilerin nasıl sunulacağını göstermektir. Bu verilerle, (1) kromozom boyama probları kullanarak genom bağlanmasında önemli farklılıklar belirlemenin mümkün olduğunu gösterdik, bu çalışmada kromozom 18'in analiz edilenlerden en az bağlı kromozom olduğunu ortaya koyduk (Şekil 3); (2) İki gen lokusu arasında önemli farklılıklar gösteren gen lokusları ve (Şekil 4) (3) Telomerler, çoğalan ve yaşlanan hücrelere kıyasla sessiz hücrelere daha az güçlü bir şekilde bağlanırlar (Şekil 5). Proliferasyon yapan ve olmayan hücreler arasında, çözünmez bir protein olan ve bu nedenle artık çekirdeklerle birlikte kalan proliferasyon belirteci Ki67 antijeninin varlığı veya belirli bir zaman periyodu içinde S-fazından geçen hücreleri vurgulamak için nükleotidlerin dahil edilmesi yoluyla ayrım yapabiliyoruz (Şekil 2). Bu teknik aynı zamanda nükleoiskeletlerinde, yani laminopati hücrelerinde tehlikeye giren hücrelerdeki genom davranışını analiz etmemizi sağlamıştır ve burada ve Bikkul ve ark., 2018'de, genomun kontrol hücrelerine kıyasla daha az sıkı bir şekilde bağlanabileceğini ve klasik HGPS hücrelerinde lamin A mutasyonunun etkisini iyileştiren spesifik ilaçlarla tedavi edilirken restore edilebileceğini ortaya koymaktadır15. Bununla birlikte, burada lamin A mutasyonundan yoksun olan, ancak kromozom 13'ün daha az sıkı bir şekilde bağlandığı LINC kompleks proteini SUN119'un alışılmadık bir formunu içeren atipik HGPS AGO8466 hücreleri için yeni veriler gösteriyoruz (Şekil 6).
HALO-FISH, ekstraksiyon prosedüründen çıkarılmayan proteinleri çözmek için dolaylı immünofloresan ile kombinasyon halinde nükleoiskelet ile genomik etkileşimlerin incelenmesini sağlayan benzersiz bir yöntemdir. Nükleoiskeletin bazı kanser tipleri19 gibi çeşitli hastalıklarda modifiye olduğu ve bazı nükleoiskeletle ilişkili proteinlerin tanısal biyobelirteçler olarak öneminin24,25 olduğu gösterilmiştir. Bu nedenle, bu teknik, nükleoiskeletin 15,24,25,27 hastalığında kromatin organizasyonu / düzensizliği üzerindeki etkisini incelemede önemli bir role sahiptir ve insan hücreleri ile sınırlı değildir, diğer hayvanlardan kromozomal boyama probları ile aynı DNA-halo protokolü kullanılabilir 28.
Açıklamalar
Yazarların açıklayacak hiçbir şeyi yok.
Teşekkürler
Prof. Michael Bittner'e kromozom kol boyama problarının nazik hediyesi için teşekkür ederiz. LG, AB tarafından finanse edilen EURO-Laminopatiler projesi ve Brunel Progeria Araştırma Fonu tarafından desteklendi.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 10X PBS | Thermo Fisher Scientific | 10388739 | Used to create DNA halos |
| 5-bromo-2′-deoxy-uridine | Sigma-Aldrich | B5002-100MG | Labelled nucleotide |
| 5-Fluoro-2′-deoxyuridine | Sigma-Aldrich | F0503-100MG | Labelled nucleotide |
| Agar Technical | Thermo Fisher Scientific | 15562141 | DNA isolation of BAC clones |
| Agarose | Sigma-Aldrich | A939-50G | Check product size of DOP-PCR and nick translation |
| Atypical type 2 HGPS fibroblasts (AG08466) | Coriell Institute | AG08466 | Cell line |
| Bacto tryptone | Thermo Fisher Scientific | 16269751 | DNA isolation of BAC clones |
| Biotin-16-dUTP | Roche Diagnostics | 11093711103 | Labelled nucleotides |
| Chloramphenicol | Sigma-Aldrich | C0378-25G | DNA isolation of BAC clones |
| Classical Hutchinson-Gilford progeria syndrome (HGPS) fibroblasts (AG06297) | Coriell Institute | AG0297 | Cell line |
| Coplin jar | Thermo Fisher Scientific | 12608596 | Holds 5 slides or 8 slides back to back |
| Cot-1 DNA | Thermo Fisher Scientific | 15279011 | Block nonspecific hybridization in HALO FISH |
| DEPC-treated water | Sigma-Aldrich | 693520-1L | DNA isolation of BAC clones |
| Dextran sulphate | Sigma-Aldrich | S4030 | Hybridisation mixture |
| Digitonin | Sigma-Aldrich | D141 | Component of extraction buffer |
| Digoxigenin-11-dUTP | Sigma-Aldrich | 11093088910 | Labelled nucleotides |
| Donkey anti-mouse Cy3 | Jackson Laboratory | 715-165-150 | Secondary antibody |
| EDTA | Sigma-Aldrich | E6758 | Component of extraction buffer |
| Ethanol | Component of extraction buffer | ||
| Ethanol | Sigma-Aldrich | 443611 | Probe precipitation and HALO FISH |
| Fetal bovine system | Thermo Fisher Scientific | 26140079 | Cell culture serum |
| Formamide | Thermo Fisher Scientific | 10523525 | 2D FISH of DNA halos |
| Glass wool | Sigma-Aldrich | 18421 | Spin column |
| Herring sperm | Sigma-Aldrich | D7290 | Probe precipitation |
| HXP™ Lamp (metal halide microscope lamp) | OSRAM | HXP-R120W45C VIS | Image capture of DNA halos |
| Hydrochloric acid | Thermo Fisher Scientific | 10313680 | Cleaning microscope slides |
| Isopropanol | Sigma-Aldrich | I9516-25ML | DNA isolation of BAC clones |
| KAPA HiFi PCR Kit | KAPA Biosystems | KK2103 | PCR Kit |
| Leica DM4000 fluorescent microscope with DFC365 FX camera and LAS AF (Version: 4.5.0) image acquisition software. | Leica Microsystems | Image capture of DNA halos | |
| Luria-Bertani agar | Thermo Fisher Scientific | 13274843 | DNA isolation of BAC clones |
| Magnesium chloride | Sigma-Aldrich | M8266 | Component of CSK buffer |
| Methanol | Thermo Fisher Scientific | 10284580 | Cleaning and sterilizing microscope slides |
| Mouse anti-BrdU antibody | BD Pharmingen | B2531-100UL | BrdU visualisation |
| Newborn calf serum | Thermo Fisher Scientific | 16010159 | Cell culture serum and blocking reagent |
| Nick translation kit | Invitrogen | ||
| PCR grade water | Sigma-Aldrich | 693520-1L | PCR and DNA isolation of BAC clones |
| PCR Primers | Sigma-Aldrich | ||
| PIPES | Sigma-Aldrich | P1851 | Component of CSK and extraction buffers |
| Potassium acetate | Sigma-Aldrich | P1190-100G | DNA isolation of BAC clones |
| QuadriPERM® 4 X 12 | SARSTEDT | 94.6077.307 | Square cell culture dish, polysterene with four compartments. This has hydrophobic surface, is sterile, non-pyrogenic/endotoxin-fee and non-cytotoxic. |
| Rabbit Anti-Ki67 antibody | Sigma-Aldrich | ZRB1007-25UL | Proliferation marker |
| Rnase A | Sigma-Aldrich | R6513 | DNA isolation of BAC clones |
| Rubber cement | Halford's | 101836 | 2D FISH of DNA halos |
| Sephadex G-50 | Sigma-Aldrich | S6022-25G | Spin column |
| Sodium acetate | Sigma-Aldrich | S2889 | Probe precipitation |
| Sodium chloride | Sigma-Aldrich | S5886 | Component of CSK, extraction and SSC buffers |
| Sodium citrate | Sigma-Aldrich | C8532 | Component of SSC buffer |
| Sodium dodecyl sulphate | L3771-100G | DNA isolation of BAC clones | |
| Sodium hydroxide | Sigma-Aldrich | S8045-500G | DNA isolation of BAC clones |
| Spermidine | Sigma-Aldrich | S2626 | Component of extraction buffer |
| Spermine | Sigma-Aldrich | S4264 | Component of extraction buffer |
| Streptavidin-Cy3 | Amersham Life Sciences Ltd, Scientific Laboratory Supplies | pa43001 | Probe antibody |
| Sucrose | Sigma-Aldrich | S0389 | Component of CSK buffer |
| Sucrose | Sigma-Aldrich | S0389 | CSK buffer+A66:D68 |
| SuperFrost™ microscope slides | Thermo Fisher Scientific | 12372098 | Microscope slides: 1 mm thickness, 76 mm length, 26 mm width. Uncoated. |
| Swine anti-rabbit TRITC | Dako | ||
| TELO-PNA FISH KIT | Agilent Dako | K532511-8 | Delineation of telomeres |
| Tris-HCl | Sigma-Aldrich | T3253-100G | Column buffer |
| Triton™ X-100 | Sigma-Aldrich | T9284 | Component of CSK buffer |
| Tryptone | Thermo Fisher Scientific | 10158962 | DNA isolation of BAC clones |
| Tween-20 | Sigma-Aldrich | P9416- 100ML | Detergent |
| Vectashield mountant containing DAPI | Vector Laboratories | H-1200 | 2D FISH of DNA halos |
| Whole human chromosome probes | Calbiochem | 2D FISH of DNA halos | |
| Yeast extract | Thermo Fisher Scientific | 10108202 | DNA isolation of BAC clones |
Referanslar
- Berezney, R., Coffey, D. S. Identification of a nuclear protein matrix. Biochemical Biophysical Research Communications. 60 (4), 1410-1417 (1974).
- Haaf, T., Ward, D. C. High resolution ordering of YAC contigs using extended chromatin and chromosomes. Human Molecular Genetics. 3 (4), 629-633 (1994).
- Parra, I., Windle, B. High resolution visual mapping of stretched DNA by fluorescent hybridization. Nature Genetics. 5 (1), 17-21 (1993).
- Senger, G., et al. Released chromatin: linearized DNA for high resolution fluorescence in situ hybridization. Human Molecular Genetics. 3 (8), 1275-1280 (1994).
- Florijn, R. J., et al. High-resolution DNA Fiber-FISH for genomic DNA mapping and colour bar-coding of large genes. Human Molecular Genetics. 4 (5), 831-836 (1995).
- Elcock, L. S., Bridger, J. M. Fluorescence in situ hybridization on DNA halo preparations and extended chromatin fibres. Methods Molecular Biology. 659, 21-31 (2010).
- Heiskanen, M., et al. Visual mapping by fiber-FISH. Genomics. 30 (1), 31-36 (1995).
- Bensimon, A., et al. Alignment and sensitive detection of DNA by a moving interface. Science. 265 (5181), 2096-2098 (1994).
- Michalet, X., et al. Dynamic molecular combing: stretching the whole human genome for high-resolution studies. Science. 277 (5331), 1518-1523 (1997).
- Wilson, R. H., Coverley, D. Relationship between DNA replication and the nuclear matrix. Genes Cells. 18 (1), 17-31 (2013).
- Wilson, R. H. C., Coverley, D. Transformation-induced changes in the DNA-nuclear matrix interface, revealed by high-throughput analysis of DNA halos. Science Reports. 7 (1), 6475 (2017).
- Iarovaia, O. V., Akopov, S. B., Nikolaev, L. G., Sverdlov, E. D., Razin, S. V. Induction of transcription within chromosomal DNA loops flanked by MAR elements causes an association of loop DNA with the nuclear matrix. Nucleic Acids Research. 33 (13), 4157-4163 (2005).
- Tandara, M., et al. Sperm DNA integrity testing: big halo is a good predictor of embryo quality and pregnancy after conventional IVF. Andrology. 2 (5), 678-686 (2014).
- Repping, S., et al. The use of spermHALO-FISH to determine DAZ gene copy number. Mol Human Reproduction. 9 (4), 183-188 (2003).
- Bikkul, M. U., et al. Farnesyltransferase inhibitor and rapamycin correct aberrant genome organisation and decrease DNA damage respectively, in Hutchinson-Gilford progeria syndrome fibroblasts. Biogerontology. 19 (6), 579-602 (2018).
- Telenius, H., et al. Degenerate oligonucleotide-primed PCR: general amplification of target DNA by a single degenerate primer. Genomics. 13 (3), 718-725 (1992).
- Bridger, J. M., et al. Association of pKi-67 with satellite DNA of the human genome in early G1 cells. Chromosome Research. 6, 13-24 (1998).
- Sales Gil, R., Vagnarelli, P. Ki-67: More Hidden behind a 'Classic Proliferation Marker'. Trends in Biochemical Sciences. 43 (10), 747-748 (2018).
- Bikkul, M. U., et al. Telomere elongation through hTERT immortalization leads to chromosome repositioning in control cells and genomic instability in Hutchinson-Gilford progeria syndrome fibroblasts, expressing a novel SUN1 isoform. Genes Chromosomes Cancer. 58 (6), 341-356 (2019).
- Jackson, D. A., Cook, P. R. Visualization of a filamentous nucleoskeleton with a 23 nm axial repeat. EMBO Journal. 7, 3667-3677 (1988).
- Albrethsen, J., et al. Unravelling the nuclear matrix proteome. Journal of Proteomics. 72, 71-81 (2009).
- Mika, S., Rost, B. NMPdb: Database of nuclear matrix proteins. Nucleic Acids Research. 33, 160-163 (2005).
- Gavrilov, A. A., et al. of the nuclear matrix-bound chromatin hubs by a new M3C experimental procedure. Nucleic Acids Research. 38, 8051-8060 (2010).
- Sjakste, N., et al. Role of the nuclear matrix proteins in malignant transformation and cancer diagnosis. Experimental Oncology. 26 (3), 170-178 (2004).
- Leman, E. S., Getzenberg, R. H. Nuclear structure as a source of cancer specific biomarkers. Journal of Cellular Biochemistry. 104 (6), 1988-1993 (2008).
- Volpi, E. V., Bridger, J. M. FISH glossary: an overview of the fluorescence in situ hybridization technique. Biotechniques. 45 (4), 385-386 (2008).
- Bridger, J. M., Foster, H. A. Senescence and the Genome. Human Interphase Chromosomes. , (2021).
- Foster, H. A., Griffin, D. K., Bridger, J. M. Interphase chromosome positioning in in vitro porcine cells and ex vivo porcine tissues. BMC Cell Biology. 13 (1), 30 (2012).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır