Method Article
Hibridização in situ fluorescente em preparações de halo de DNA para revelar cromossomos inteiros, telômeros e loci gênicos
Neste Artigo
Erratum Notice
Resumo
A combinação de preparações de halo de DNA com hibridização in situ fluorescente permite a análise de alta resolução das interações genômicas com o nucleoesqueleto. O genoma anexado leva a sinais fluorescentes hibridizados localizados dentro dos núcleos residuais extraídos, enquanto o genoma não ligado está no halo de DNA ao redor dos núcleos residuais.
Resumo
O genoma está associado a várias estruturas dentro dos núcleos celulares, a fim de regular sua atividade e ancorá-lo em locais específicos. Essas estruturas são coletivamente conhecidas como nucleoesqueleto e incluem a lâmina nuclear, os nucléolos e os corpos nucleares. Embora muitas variantes de hibridação in situ fluorescente (FISH) existam para estudar o genoma e sua organização, estas são frequentemente limitadas pela resolução e fornecem informações insuficientes sobre a associação do genoma com estruturas nucleares. O método do halo de DNA usa altas concentrações de sal e detergentes não iônicos para gerar alças de DNA que permanecem ancoradas a estruturas dentro dos núcleos através de regiões de ligação dentro do genoma. Aqui, proteínas nucleares solúveis, como histonas, lipídios e DNA não fortemente ligados à matriz nuclear, são extraídas. Isso leva à formação de um halo de DNA não anexado ao redor de um núcleo residual que contém DNA intimamente associado a estruturas nucleares internas e proteínas resistentes à extração. Essas cadeias de DNA estendidas permitem maior resolução e podem facilitar o mapeamento físico. Em combinação com FISH, este método tem a vantagem adicional de estudar interações genômicas com todas as estruturas pelas quais o genoma está ancorado. Esta técnica, denominada HALO-FISH, é altamente versátil, pela qual halos de DNA podem ser acoplados a sondas de ácido nucleico para revelar loci gênicos, cromossomos inteiros, satélite alfa, telômeros e até RNA. Esta técnica fornece uma visão sobre a organização nuclear e função em células normais e na progressão de doenças, como no câncer.
Introdução
A "matriz nuclear" foi descrita pela primeira vez por Berezney e Coffey em 19741. Após realizarem extrações com altas molaridades salinas e tratamento com nucleases em núcleos hepáticos de ratos, eles identificaram um arcabouço estrutural proteico. O procedimento do halo de DNA foi posteriormente adaptado deste método e envolve a remoção de proteínas solúveis para que apenas a matriz nuclear (NM) e as proteínas e cromossomos associados à MN persistam. As regiões de ligação ao DNA estão localizadas na base das alças de DNA e são chamadas de regiões ligadas à matriz (MARs) ou regiões de ligação do andaime (SARs), que são resistentes à extração com altas concentrações de sal e detergente iônico lítio-3,5-diiodossalicilato (LIS), respectivamente. Nos halos de DNA, o DNA associado a MARs/SARs está ligado dentro do núcleo residual, enquanto as alças de DNA se estendem para longe desses locais e formam o halo de DNA. Sabemos agora que o genoma está ancorado via domínios associados à lâmina (LADs) à lâmina nuclear e através de regiões nucleolares associadas (NADs) e possivelmente através de outras estruturas nucleares, como corpos nucleares específicos.
O método do halo de DNA pode ser utilizado para mapeamento físico de DNA, genes e regiões cromossômicas, pois o DNA estendido e a cromatina proporcionam maior resolução, pois a cromatina é despida de histonas e o DNA é estirado 2,3,4,5,6. No entanto, existem algumas limitações ao usar halos de DNA para esta aplicação. Por exemplo, o DNA fortemente associado a núcleos residuais de halos de DNA pode ser inacessível às sondas, impedindo-o de análise e mapeamento físico6. Outras técnicas como a fibra-FISH 2,4,5,7 e o pente molecular 8 também possibilitam o mapeamento físico e têm a vantagem de serem relativamente rápidas e fáceis de serem realizadas. Ambos são preferencialmente usados para mapeamento de DNA de genes sobre halos de DNA. Esses métodos extraem fibras de cromatina através do uso de extrações com solvente ou sal do núcleo, entretanto, o penteamento molecular tende a ter melhor reprodutibilidade8,9.
Há evidências crescentes de que o nucleoesqueleto tem um papel no suporte de processos nucleares chave, tais como sítios de ligação para o DNA, remodelamento da cromatina, transcrição do DNA, reparo do DNA e replicação do DNA11,12. Como tal, a técnica do halo de DNA foi desenvolvida para investigar as interações entre o nucleoesqueleto e o genoma durante essas atividades celulares e tem sido rotineiramente usada e relatada em pesquisas. Essa técnica também tem sido utilizada para investigar interações entre o genoma e o nucleoesqueleto em relação à progressão da doença, sendo identificadas alterações na estrutura nuclear associadas àmalignidade11.
A técnica do halo de DNA também tem sido utilizada para investigar a relação entre o genoma e o nucleoesqueleto durante o desenvolvimento ediferenciação12. Vários estudos têm utilizado uma variação da técnica do halo de DNA conhecida como halosperma13 ou SpermHalo-FISH se acoplada à FISH14. A cromatina dos espermatozoides está firmemente ligada a proteínas conhecidas como protaminas e esta técnica foi desenvolvida para melhorar o acesso ao ADN dos espermatozoides. Halosperma tem sido usada para investigar a integridade do DNA dos espermatozoides e determinar se danos ao DNA estão presentes. Os espermatozoides com menos danos ao DNA correlacionam-se a um maior tamanho do halo de DNA, enquanto os espermatozoides com níveis aumentados de DNA fragmentado e danificado tinham halos pequenos ou nenhum. Assim, a halosperma pode ser utilizada como um potencial marcador prognóstico da qualidade embrionária e do sucesso da gestação com FIV13. Este exemplo enfatiza as potenciais aplicações clínicas dessa técnica. Em nosso trabalho utilizamos HALO-FISH para avaliar mudanças no comportamento do genoma e o efeito de tratamentos medicamentosos específicos na doença do envelhecimento precoce Síndrome de Hutchinson-Gilford Progeria (HGPS)15.
Juntos, esses e outros estudos destacam a amplitude de processos/aplicações que a técnica do halo de DNA pode ser utilizada para estudar e a utilidade da técnica.
Protocolo
1. Preparo das lâminas, esterilização e cultura celular
- Preparar 500 mL de HCl 10% (v/v) e despejar em um copo grande.
- Gota microscópio lâminas individualmente no ácido e incubar por 1 h à temperatura ambiente em um agitador regulado a 2 x g.
CUIDADO: HCl é corrosivo e irritante. Pode causar queimaduras graves na pele e danos oculares e irritação da pele, olhos e sistema respiratório. Assegure-se de que a proteção pessoal adequada seja usada, incluindo luvas nitrílicas, proteção ocular e um jaleco de laboratório. - Decantar o ácido do copo e lavar as lâminas dez vezes em água da torneira e depois mais dez vezes em água deionizada.
- Enxágue as lâminas em metanol duas vezes e mantenha em metanol até a esterilização por chamas.
CUIDADO: O metanol é um líquido altamente inflamável e tóxico se ingerido, em contato com a pele ou se inalado. Além disso, o metanol pode causar danos aos órgãos, é corrosivo e irritante. Respeite os limites de exposição no local de trabalho e garanta o uso de proteção pessoal adequada, incluindo luvas de borracha butilada, proteção ocular e jaleco de laboratório. Sempre que possível, manuseie em um armário de fumaça de ventilação de exaustão local (LEV). - Usando pinças de metal ou pinças longas, remova uma lâmina de microscópio do copo contendo metanol. Chama sobre um queimador de Bunsen para esterilizar e transferir para um recipiente retangular de cultura de células contendo quatro compartimentos para lâminas, localizado próximo ao queimador de Bunsen.
CUIDADO: A queima permite a esterilização imediata das lâminas do microscópio antes do uso; no entanto, este método tem riscos associados. Como o metanol é altamente inflamável, é importante que o copo que contém as lâminas seja posicionado longe do queimador de Bunsen. Pinças longas ou pinças devem ser usadas para segurar firmemente as corrediças. O nível de metanol no copo deve apenas cobrir as lâminas, tanto para minimizar a quantidade de metanol utilizada quanto para que apenas as extremidades das pinças/pinças estejam em contato com o metanol. Certifique-se sempre de que o metanol evaporou das pinças ou pinças após o uso e que estas esfriaram antes de colocá-las novamente no copo que contém as lâminas e o metanol. O copo deve ser coberto por um pedaço de papel alumínio para matar a fome de oxigênio caso o metanol pegue fogo. Nunca deslize a chama dentro de uma capela de fluxo laminar Classe II onde o ar está circulando. - Alternativamente, execute as etapas 1.1 a 1.4, mas em vez de inflamar as lâminas após incubar com metanol, coloque as lâminas no tecido sem fiapos para secar ao ar. Depois de seco, envolva em papel alumínio e coloque em um forno esterilizador ou autoclave.
- Cultivar células no meio apropriado com soro por pelo menos 48 h a 37 °C em 5% de CO2 até atingir 60-70% de confluência. Este protocolo foi realizado em uma passagem precoce de fibroblastos dérmicos humanos (HDFs) e em fibroblastos clássicos da síndrome de Hutchinson-Gilford progeria (HGPS) (AG06297) e fibroblastos atípicos do tipo 2 do HGPS (AG08466). Colher cada tipo celular e contar usando um hemocitômetro para determinar a densidade celular. Semear 1 x 105 células em meio de 10 mL por lâmina.
NOTA: A densidade celular é importante, pois as alças de DNA de diferentes núcleos podem convergir se as células se tornarem muito confluentes. As densidades de semeadura podem precisar ser otimizadas dependendo do tipo de célula utilizada, pois as células transformadas podem proliferar mais rapidamente, enquanto culturas de células de passagem posterior podem levar mais tempo para atingir a confluência desejada. - Se as células precisarem ser presas em G0 para se tornarem quiescentes, então semeie 1 x 105 células (em meio de 10 mL) por lâmina e deixe crescer por 24 h. Lavar as células duas vezes com meio livre de soro e incubar em meio padrão que contenha uma concentração menor de soro a 0,5% (soro de bezerro recém-nascido, NCS; ou soro fetal bovino, FBS) por 7 dias.
- Se o status proliferativo das células é necessário para o ensaio de DNA Halo, então determine as células que passaram pela fase S pela incorporação de 5-bromo-2'-desoxi-uridina (BrdU) no DNA durante a replicação.
- Células de sementes normalmente e crescem por 24 h. Retirar o meio de cultura e substituir por meio contendo BrdU e 5-fluoro-2'-desoxiuridina (3 μg/μL). Após mais 24 h, retirar o meio, lavar as células uma vez com meio (10% NCS) e depois realimentar com meio fresco (10% NCS). Incubar por mais 24 h e, em seguida, preparar as lâminas para o ensaio do halo de DNA.
2. Preparação da sonda
- Sondas de pintura cromossômica inteira e de braço
- Realizar sondas cromossômicas a partir da amplificação de cromossomos classificados em fluxo ou microdissecados por reação em cadeia da polimerase com primed de oligonucleotídeos degenerados (DOP-PCR) utilizando o método de Telenius et al.16. Use DOP-PCR para marcar sondas cromossômicas com Biotina-16-dUTP ou Digoxigenina-11-dUTP, conforme mostrado na Tabela 1. Por favor, verifique as instruções do fabricante para o perfil de amplificação, no entanto, as condições utilizadas para este experimento são mostradas na Tabela 2.
- Preparar sonda de braço ou cromossómico inteiro adicionando 8 μL de produto de PCR marcado, 7 μL de DNA Cot-1, 3 μL de esperma de arenque, 1/20º volume de acetato de sódio 3 M (pH 5,4) e 2 volumes de etanol 100%. Incubar a solução de sonda durante um mínimo de 30 minutos a -80 °C.
NOTA: Este método pode ser usado para criar sondas cromossômicas únicas, ou várias sondas cromossômicas se rótulos diferentes (ou seja, Biotina-16-UTP e Digoxigenina-11-dUTP) forem usados para cada cromossomo de interesse.
CUIDADO: O etanol é um líquido e vapor altamente inflamável e pode causar irritação ocular grave. Manter longe do calor, superfícies quentes e fontes de ignição. Respeite os limites de exposição no local de trabalho e garanta o uso de proteção pessoal adequada, incluindo luvas de borracha butilada, proteção ocular e jaleco de laboratório. Sempre que possível, manuseie em um armário de fumaça LEV. - Solução de sonda de centrifugação a 13.700 x g por 15 min a 4 °C e depois lavar com etanol a 70%. Repita o procedimento de centrifugação e descarte o sobrenadante, tomando cuidado para não atrapalhar ou perder a pelota de DNA. Deixe o pellet de DNA secar.
- Adicionar 12 μL de tampão de hibridização (50% formamida, 10% sulfato de dextrana, 10% citrato de sódio salino 20x (SSC; 3 M NaCl, 0,3 M tri-sodium citrate; pH 7,0), 1% (v/v) polioxietileno sorbinal monolaurato (Tween-20)) ao pellet de DNA. Deixar a 37 °C durante, pelo menos, 2 h para que a pastilha de ADN se dissolva no tampão de hibridização.
CUIDADO: A formamida é cancerígena e teratogênica, por isso pode causar sérios danos ao feto. Se uma mulher está grávida ou suspeita que está grávida, eles devem evitar trabalhar com formamida. A formamida deve ser utilizada num exaustor de fumos LEV. Respeite os limites de exposição no local de trabalho e garanta o uso de proteção pessoal adequada, incluindo luvas de borracha butilada, proteção ocular e jaleco de laboratório.
- Isolamento de DNA de Cromossomos Artificiais Bacterianos (CBAs)
- Coloque uma pequena porção do estoque de glicerol do clone BAC em uma placa de ágar Luria-Bertani (LB) (1% (p/v) NaCl; 1% (p/v) triptona, 0,5% (p/v) extrato de levedura, 1,5% (p/v) Agar Technical, 12,5 μg/mL (p/v) cloranfenicol). Incubar durante a noite a 37 °C.
- Selecionar uma única colônia da placa e inocular 10 mL de caldo LB (1% (p/v) de NaCl, 1% (p/v) de bactotriptona, 0,5% (p/v) de extrato de levedura, 12,5 μg/mL (p/v) de cloranfenicol). Deixar a solução numa incubadora de agitação durante a noite a 37 °C.
CUIDADO: O cloranfenicol é suspeito de causar câncer. Manuseie com cuidado e reduza a exposição. - Cultura centrífuga a 1.700 x g por 10 min à temperatura ambiente.
- Eliminar o sobrenadante e adicionar 300 μL de solução P1 (15 mM Tris (pH 8), 10 mM EDTA, 100 μg/mL RNase A) ao pellet. Vórtice vigorosamente e transfira as células para um tubo de microcentrífuga de 2 mL.
- Adicionar 300 μL de solução de P2 (NaOH 0,2 M, dodecil sulfato de sódio (SDS) a 1% (p/v) gota a gota às células. Inverta o tubo de microcentrífuga fechado 5 vezes e deixe à temperatura ambiente por no máximo 5 min.
CUIDADO: O hidróxido de sódio é corrosivo e pode causar queimaduras graves na pele e danos nos olhos. Pode ser corrosivo para metais. Manuseie com cuidado e reduza a exposição. Respeite os limites de exposição no local de trabalho e garanta o uso de proteção individual adequada, incluindo luvas de borracha nitrílica, proteção ocular e jaleco de laboratório. Sempre que possível, manuseie em um armário de fumaça LEV.
CUIDADO: O dodecil sulfato de sódio é um sólido inflamável, prejudicial se ingerido e pode causar irritação cutânea e respiratória. Também pode causar danos oculares graves. Assegure-se de que a proteção pessoal adequada seja usada, incluindo luvas de borracha nitrílica, proteção ocular e um jaleco de laboratório. Sempre que possível, manuseie em um armário de fumaça LEV. - Adicionar 300 μL de P3 (acetato de potássio 3 M) lentamente às células e misturar suavemente. Coloque o tubo da microcentrífuga no gelo por 10 min.
- Centrifugar a 8.100 x g durante 10 min a 4 °C e transferir o sobrenadante para um tubo contendo 800 μL de isopropanol gelado. Inverter o tubo várias vezes e incubar a -20 °C durante a noite.
CUIDADO: O isopropanol é um líquido e vapor altamente inflamável e pode causar irritação ocular grave, sonolência ou tontura. Manter longe do calor, superfícies quentes e fontes de ignição. Respeite os limites de exposição no local de trabalho e garanta o uso de proteção pessoal adequada, incluindo luvas de borracha nitrílica, proteção ocular e jaleco de laboratório. Sempre que possível, manuseie em um armário LEV)fume. - Centrifugar a 8.100 x g por 15 min a 4 °C. Retire o sobrenadante e transfira para outro tubo. Adicionar 500 μL de etanol 70% gelado. Inverter o tubo várias vezes e centrifugar a 8.100 x g por 5 min a 4 °C.
- Retire o sobrenadante e seque o pellet ao ar à temperatura ambiente. Quando o pellet estiver seco, ressuspenda em 40 μL de água tratada com pirocarbonato de dietilo (tratada com DEPC) e deixe a 4 °C durante a noite. Uma vez totalmente ressuspenso, remova 5 μL de solução e carregue em um gel de agarose a 1% para verificar a presença de DNA.
- Preparação de sonda de gene único de CBAs via nick translation
- Use kits de etiquetagem de tradução nick disponíveis comercialmente. Como alternativa, use o seguinte protocolo. Ver quadro 3 para componentes e volumes.
- Adicione os constituintes da Tabela 3 em um tubo de microcentrífuga adicionando a DNA Polimerase I por último, misture suavemente e centrifugue brevemente por alguns segundos. Incubar a solução a 15 °C durante 2 h.
- Para verificar o tamanho dos fragmentos, carregar 5 μL da solução em gel de agarose a 2%. A faixa de tamanho do fragmento de DNA deve estar entre 200-600 pb. Se os tamanhos dos fragmentos de ADN forem maiores, continue a incubar a solução durante mais 15 minutos a 15 °C e execute os produtos em gel de agarose a 2%.
- Pare a reação de tradução de nick adicionando 10 mM EDTA, 0,1% SDS (2,5 μL de 0,5 M EDTA, pH 8,0 em 100 μL e 1 μL de 10% SDS em 100 μL). Aquecer a solução a 65 °C durante 5 min.
- Para remover nucleotídeos não incorporados, aplique a sonda BAC em uma coluna de spin. Colunas de spin comerciais podem ser compradas, ou podem ser criadas usando uma seringa da seguinte maneira:
- Adicionar 30 g de Sephadex G-50 a 500 ml de tampão de coluna (10 mM Tris-HCl (pH8), 1 mM EDTA, 0,1% SDS). Autoclave a mistura. Fazer também 500 mL de tampão de coluna (sem Sephadex G-50) e autoclave.
- Faça colunas de spin adicionando lã de vidro autoclavada ao fundo de uma seringa de 1 mL. Encha a seringa de 1 ml com Sephadex G-50 em tampão de coluna. Coloque 1 mL de seringa em um tubo de centrífuga de 15 mL que tenha um tubo de microcentrífuga sem tampa na parte inferior. Centrifugar a 1.600 x g por 5 min.
- Retire a seringa e descarte o tubo de microcentrífuga na parte inferior. Adicione um tubo de microcentrífuga fresco de volta ao tubo de centrífuga de 15 mL. Adicione tampão de coluna (sem Sephadex G-50) à seringa de 1 ml e volte a introduzir no tubo de centrífuga de 15 ml. Centrifugar a 1.600 x g por 5 min. Repita esta etapa novamente duas vezes.
- Retire a seringa e insira em um tubo de centrífuga de 15 mL que contenha um novo tubo de microcentrífuga limpo. Aplique a sonda na seringa e colete a sonda no tubo da microcentrífuga.
- Para precipitar a sonda de DNA, adicione 5 μL de DNA espermática de arenque (10 mg/mL), 10 μL de acetato de sódio e 2,25 volume de etanol 100% à solução de DNA. Misture suavemente a solução e incube a -80 °C durante um mínimo de 1 h. Centrifugar a 13.700 x g por 15 min a 4 °C.
- Eliminar o sobrenadante e lavar o pellet com 200 μL de etanol a 70 % gelado durante 15 minutos a 4 °C. Retire o sobrenadante e seque ao ar. Uma vez seco, ressuspenda o pellet em 20 μL de água tratada com DEPC à temperatura ambiente por várias horas ou durante a noite a 4 °C. A sonda está agora pronta para ser usada ou pode ser armazenada a -20 °C.
- Para cada lâmina, misturar 5 μL de DNA de sonda com 5 μL de DNA Cot-1 e secar usando um concentrador de vácuo Speed Vac. Uma vez seco o pellet, ressuspenda em 12 μL de mistura de hibridização.
3. Preparação do DNA Halo
- Remova a placa de cultura quadrada que contém as lâminas e as células anexadas da incubadora. Descarte o meio, rotule as lâminas com lápis e coloque em um frasco de Coplin contendo 50 mL de tampão citoesqueleto gelado (CSK): NaCl 100 mM, MgCl2 3 mM, sacarose 0,3 M, 10 mM 1, ácido 4-piperazinediethanesulfônico (PIPES; pH 7,8), Triton X-100 0,5% (v/v) composto em água deionizada. Incubar durante 15 minutos no gelo ou a 4 °C.
CUIDADO: Triton X-100 pode causar irritação da pele e danos oculares graves. Manuseie usando equipamentos de proteção individual apropriados, incluindo luvas nitrílicas, óculos e jaleco de laboratório. - Descarte o tampão CSK e enxágue rapidamente as lâminas em 50 mL de tampão halo de DNA 1x (DHB; NaCl 140 mM, KCl 27 mM, NaHPO4 110 mM, 15 mM KH2PO 4; pH7.4) três vezes, ou seja, mergulhe a lâmina no frasco de Coplin contendo DHB e remova.
- Transfira as lâminas para um frasco Coplin contendo 50 mL de tampão de extração: NaCl 2M, PIPES 10 mM (pH 6,8), ácido etilenodiaminotetracético (EDTA) 10 mM, digitonina a 0,1% (p/v), espermina 0,05 mM (v/v), espermidina 0,125 mM (v/v). Incubar durante 4 minutos à temperatura ambiente.
CUIDADO: A digitonina é tóxica se ingerida ou em contato com a pele e é fatal se inalada. Certifique-se de que a digitonina é manuseada em um armário de fumaça LEV e use um jaleco de laboratório, luvas nitrílicas (luva dupla), óculos de segurança e máscara. Tanto a espermina quanto a espermidina podem causar queimaduras graves na pele e danos nos olhos, enquanto o EDTA causa irritação ocular grave, então manipule cada produto químico com cuidado.
NOTA: Prepare a digitonina separadamente dissolvendo o pó em água a uma temperatura de 60-70 °C. Adicione a digionina dissolvida ao tampão de extração depois de resfriado. Adicione espermina, espermidina e digitonina por último ao tampão de extração para preservar a atividade biológica. - Incubar as lâminas consecutivamente em 50 mL de 10x DHB (NaCl 1,4 M, KCl 270 mM, NaHPO4 1,1 M, KH150mM 2 PO4; PH7,4), 5x, 2x e 1x DHB por 1 min cada.
- Lâminas de imersão (direto e direto) através de uma série sequencial de 50 mL de etanol de etanol de 10%, 30%, 70% e 95% (v/v).
- Lâminas secas ao ar e armazenadas a -80 °C até que a hibridação in situ por fluorescência bidimensional (FISH 2D) seja realizada.
4. Hibridização in situ por fluorescência bidimensional
- Fazer 20x SSC: NaCl 3 M, citrato trissódico 0,3 M, pH 7,0. Esse tampão pode ser autoclavado, armazenado à temperatura ambiente e diluído conforme necessário.
- Fazer formamida a 70% (v/v), 2x SSC pH 7,0 e aquecer a 70 °C em banho-maria.
- Incubar lâminas, por 5 min cada, através de uma série sequencial de 50 mL de etanol de 70, 90 e 100% de etanol.
- Seque ao ar deslize em uma placa de aquecimento e leve ao forno a 70 °C por 5 min.
- Desnaturar as lâminas colocando na solução de formamida a 70%, 2x SSC durante 2 min a 70 °C.
NOTA: A temperatura e o tempo são críticos para o passo 4.5. Se a temperatura for o DNA não desnaturará e as sondas não hibridizarão, e nenhum sinal será obtido a partir do halo de DNA FISH. - Colocar a lâmina desnaturada em 50 mL de etanol gelado a 70% por 5 min e passar por uma série de etanol de 90%, 95% e 100% à temperatura ambiente por 5 min cada.
- Secar ao ar em uma placa de aquecimento
- Manusear diretamente sondas de cromossomos humanos totais rotulados diretamente de acordo com as instruções do fabricante. Para esses experimentos foram utilizadas tintas cromossômicas inteiras humanas 1, 13, 15, 17 e 18. Além disso, neste experimento, foram utilizadas sondas dos genes CCND1 e CTNNA1 .
NOTA: Tanto as sondas cromossômicas inteiras quanto as sondas gene-específicas do BAC foram marcadas com biotina-11-dUTP e detectadas por estreptavidina conjugada à cianina 3 (Cy3). Para as sondas de pintura cromossômica feitas por (DOP-PCR) e DNA BAC marcado por nick translation, estas serão referidas como sondas de DNA a partir deste ponto no protocolo e tratadas da seguinte forma. - Desnaturar a sonda de DNA (tinta cromossômica inteira ou sonda gene-específica) a 75 °C por 10 min em um banho de água ou bloco quente.
- Sondas de ADN quentes a 37 °C durante 30 minutos num bloco quente ou em banho-maria antes de pipetar 10 μL na lâmina apropriada.
NOTA: Esta etapa é importante para bloquear sequências cromossômicas repetitivas. Se não forem realizados, sinais inespecíficos podem ser produzidos no DNA Halo FISH. - Sonda de sobreposição com lamínula de 21 mm x 21 mm e vedação com cimento de borracha.
- Incubar as lâminas durante um mínimo de 18 h a 37 °C numa câmara de hibridização humidificada.
NOTA: As câmaras de hibridização umidificadas podem ser feitas de caixas sanduíche que contêm várias camadas de tecido umedecido e uma plataforma elevada construída a partir de pipetas plásticas cortadas de 10 mL para apoiar as lâminas. Este é coberto em folha de alumínio para minimizar a exposição à luz. - Retire o cimento de borracha cuidadosamente usando pinças.
- Incubar as lâminas em 50 mL de formamida a 50% (v/v), 2x SSC, solução pH 7,0 pré-aquecida a 45 °C por três incubações de 5 min.
OBS: Permitir que a lamínula se afaste da lâmina na primeira incubação em formamida a 50% (v/v), solução de SSC 2x, pH 7,0. Isso evita danos à preparação do Halo de DNA que poderiam ser causados por "arrastar" a tampa para longe. As lâminas podem ser agitadas no tampão através da preensão por pinças para ajudar a desprender a lamínula. - Em seguida, colocar as lâminas em 50 mL de solução de 0,1x SSC, pH 7,0, pré-aquecida a 60 °C, mas colocada em banho-maria a 45 °C. Incubar por 5 min e substituir o tampão mais duas vezes por incubações de 5 min.
- Coloque as lâminas em um frasco de Coplin contendo 50 mL de solução 4x SSC, pH 7,0 à temperatura ambiente e incube por 15 min com três trocas de tampão.
- Aplicar 100 μL de BSA a 4%, solução de SSC 4x em cada lâmina e sobrepor com um pedaço de filme de parafina. Incubar à temperatura ambiente durante 10 min. Isso impede a ligação inespecífica do anticorpo.
- Para detectar a sonda marcada (biotina-16-dUTP), incubar com 100 μL de uma estreptavidina-Cy3 1:200 (fabricada em BSA 1%, 4x SSC) por 1 h à temperatura ambiente.
NOTA: Siga as instruções do fabricante com diluições de anticorpos e diluição de teste antes do experimento para garantir que um bom sinal seja produzido - Coloque as lâminas em um frasco de Coplin contendo 50 mL de solução de SSC 4x (Tween-20 a 0,5%) pH 7,0 à temperatura ambiente e incube por 15 min com três trocas de tampão. As lâminas podem ser montadas nesta fase, como mostrado na etapa 4.21, se a imunofluorescência não for necessária.
- Se o status proliferativo das células transformadas em halos de DNA for necessário, corar com anticorpos anti-pKi67 após as etapas de FISH, antes da montagem ou coloração para BrdU incorporado.
- Lavar as lâminas 3 vezes por 5 min cada em 50 mL de solução salina tamponada com fosfato (PBS) 1x, seguido de bloqueio com NCS a 4% em PBS por 1 h à temperatura ambiente.
- Aplicar 200 μL do anticorpo primário necessário (coelho anti-humano pKi67; camundongo anti-BrdU) na lâmina, sobrepor com uma tira de filme de parafina e incubar à temperatura ambiente por 1 h.
- Lavar as lâminas 3 vezes por 5 min em 1x PBS e incubar à temperatura ambiente por 1 h em 200 μL de anticorpo secundário conjugado com fluorocromo (pKi67: suíno anticoelho TRITC; BrdU: burro anti-camundongo Cy3). Realize mais 3 lavagens de 5 min com PBS. Todas as diluições a serem feitas usando NCS a 1% (v/v) em PBS na faixa de diluições sugeridas pelo fabricante.
- Monte corrediças em 20 μL de montante contendo DAPI e sobreposição com uma lamínula de 22 mm x 50 mm.
5. Telômero PNA FISH
- Para detectar telômeros, utilizar o kit telômero PNA FISH - FITC; Execute o procedimento com as instruções do fabricante. O procedimento deve ser executado à temperatura ambiente, salvo indicação em contrário.
- Imergir as lâminas em solução salina tamponada com tris (TBS, pH 7,5) por 2 min e, em seguida, colocar em formaldeído a 3,7% (em TBS; v/v) por exatamente 2 min.
CUIDADO: A solução de TBS contém 10-30% de trometamol e 10-30% de cloridrato de 2-amino-2-(hidroximetil) propano-1,3-diol. Isso pode causar sérias irritações nos olhos e na pele, por isso use luvas de proteção e óculos de proteção/proteção facial. Use em uma área bem ventilada. - Lave as lâminas em um frasco Coplin duas vezes com TBS por 5 min cada.
- Mergulhe as lâminas na solução de pré-tratamento por 10 min e depois lave duas vezes com TBS por 5 min por lavagem.
- Em seguida, faça as lâminas através de uma série de etanol gelada composta por 50 mL de etanol a 70%, 85% e 95% (v/v) por 2 min por concentração. Depois, deixe as lâminas secarem ao ar.
- Aplique 10 μL de Telomere PNA Probe/FITC (ou Cy3), dependendo da escolha da coloração do tag fluorescente, em cada lâmina e cobertura com uma lamínula. Incubar num forno pré-aquecido regulado a 80 °C durante 5 min e, em seguida, colocar no escuro durante aproximadamente 1 h.
CUIDADO: Telomere PNA Probe/FITC contém 6-100% formamida, que causa irritação ocular grave e é teratogênica, portanto, pode causar sérios danos ao feto. Se uma mulher está grávida ou suspeita que está grávida, eles devem evitar trabalhar com formamida. A formamida deve ser usada em um exaustor de fumaça LEV e a proteção adequada dos olhos ou do rosto deve ser usada. - Para remover as lamínulas, mergulhe as lâminas em 'Solução de Enxágue' por 1 min e, em seguida, coloque na 'Solução de Lavagem' por 5 min a 65 °C.
CUIDADO: A solução de lavagem contém 1-5% de polioxietileno octilfenil éter e 1-5% de cloreto de sódio. Isso é corrosivo e pode causar sérios danos aos olhos. Certifique-se de que os óculos ou a proteção facial são usados ao manusear a solução de lavagem. - Incubar lâminas através de uma série de 50 mL de etanol gelado (70%, 85% e 95% (v/v)) por 2 min por concentração e, em seguida, secar ao ar. Depois de seco, deslize com montador contendo DAPI e sobreponha com uma lamínula.
6. Captura e análise de imagens
- Para visualizar halos de DNA e territórios cromossômicos use um microscópio de epifluorescência (por exemplo, microscópio Leica DM4000) capturando imagens usando uma objetiva de óleo HC PL FLUOTAR 100X/1.30 e uma câmera DFC365FX.
- Capture imagens em escala de cinza e defina a cor para cada canal capturado para permitir a pseudocoloração de imagens. Um software comercial foi usado neste experimento (por exemplo, o software LAS AF versão 4.5.0). Os canais de cores individuais foram exportados como TIFFs.
- Analise imagens usando o programa de processamento de imagens Java Fiji ImageJ. Carregue a imagem pressionando Arquivo e Abrir.
- Carregue canais de imagem separados ou divida uma imagem composta em canais de escala de cinza separados clicando em Imagem | Cor | Canais divididos. Selecione um canal de imagem e clique em Imagem | Ajuste e selecione Brilho e Contraste. Altere de acordo e repita com outros canais.
- Crie uma máscara do núcleo residual selecionando o canal corado por DAPI representando o núcleo. Clique na imagem | Ajuste e selecione Limite. Uma caixa de diálogo aparecerá onde o limite pode ser alterado, marque a caixa Plano de fundo escuro . Altere até que o núcleo residual esteja limpo e pressione Aplicar e feche a caixa de diálogo.
Observação : isso cria uma máscara binária com base na intensidade de pixel, com pixels brancos mostrando regiões de interesse e pixels pretos mostrando plano de fundo. Repita o mesmo procedimento no canal de sonda. - Use a seleção à mão livre para delinear a periferia do núcleo residual e clique em Editar e Limpar Fora. Sobreponha o canal da sonda sobre o núcleo residual. Isso pode ser feito pressionando Image| Cor | Mesclar canais.
- Para definir a escala de medida no ImageJ, desenhe uma linha na barra de escala ou entre os pontos de duas distâncias conhecidas. Vá para Analisar e pressione Definir escala. Na caixa de diálogo, adicione o comprimento da distância e clique em OK. Para medir distâncias, desenhe uma linha entre os pontos que estão sendo medidos e clique em Analisar | Medida. Isso transferirá os valores de distância para uma janela de dados.
- Meça a intensidade DAPI mais brilhante, pois esta coincide com o centro do núcleo. A partir disso, mede-se a distância do centro nuclear até a borda mais distante do território cromossômico (CTE). Meça a distância do centro nuclear à borda nuclear (NE).
- Certifique-se de que os resultados sejam retratados como uma relação CTE/NE. Aqui, a distância do centro nuclear a cada borda de território cromossômico mais distante (CTE) é dividida pela distância do centro nuclear a cada respectiva borda nuclear (NE). Isso deve ser realizado em um mínimo de 50 núcleos. Isso pode ser descrito como um gráfico de barras ou caixas.
- Para análise dos telômeros, analisar um mínimo de 30 núcleos por conjunto de dados. As imagens podem ser analisadas usando Fiji ImageJ ou manualmente para contar o número de telômeros dentro do núcleo residual e dentro do halo de DNA. BrdU ou pKi67 permitiu a diferenciação de núcleos proliferantes (BrdU/piK67+) e senescentes/quiescentes (BrdU/pKi67-). Os dados podem ser representados em gráficos de barras com barras de erro correspondentes ao erro padrão da média (EPM).
- Utilizar o teste t de Student (não pareado) para comparar estatisticamente os resultados com p> 0,05 considerado significativo.
Resultados
Esse método de preparação do halo de DNA tem nos ajudado em nossos esforços para determinar diferenças no comportamento do genoma dentro de células jovens e velhas, mas também em células derivadas de doenças do envelhecimento precoce com proteínas nucleosqueléticas aberrantes15. A Figura 1 mostra exemplos de halos de DNA onde é possível ver a borda de um núcleo residual, o DNA remanescente dentro do núcleo residual e o DNA não anexado que se deslocou para a área circundante criando um halo de DNA. Apresenta também a análise mostrando como o núcleo residual é obtido e as medidas de NE e CTE. É possível diferenciar células em proliferação e não proliferantes incorporando um nucleotídeo marcado, como BrdU, quando as células estão em fase S, ou empregando o marcador diagnóstico de proliferação anti-pKi67, que revela nucléolos, e regiões de heterocromatina nas célulasG117,18. Presume-se que as células primárias crescidas em soro alto sem atingir confluência, que são negativas para os marcadores de proliferação, sejam senescentes. Células primárias crescidas em soro baixo ou que se tornaram confluentes, ou seja, inibidas de contato negativas para os marcadores de proliferação, são consideradas quiescentes e seriam capazes de reentrar no ciclo celular proliferativo dados os nutrientes e a situação correta. A diferenciação entre células Ki67 positivas e negativas nos permitiu determinar diferenças entre fibroblastos dérmicos humanos em proliferação, quiescentes e senescentes. A Figura 2 mostra halos de DNA de fibroblastos dérmicos humanos em proliferação criados a partir de células onde BrdU foi incorporado a eles durante a replicação do DNA, um mecanismo que não ocorre em células não proliferantes, e posteriormente corados com anticorpo anti-BrdU. A coloração com o marcador proliferativo anticorpo anti-pKi67 também é visível na Figura 2. Este é um antígeno robusto e sobrevive ao protocolo de FISH e, portanto, pode ser corado para pós-FISH e pré-montagem. Assim, as células em proliferação são positivas (vermelho) para BrdU e anti-pKi67 (vermelho) na coluna da esquerda e as células não proliferantes, de fato, as células senescentes na Figura 2 são exibidas na coluna da direita. Os sinais verdes são telômeros individuais revelados com um kit telômero PNA FISH/FITC. A combinação de imunofluorescência com halos de DNA permite a análise durante diferentes estados celulares, como mostrado na Figura 2 ao investigar células em proliferação, quiescentes e senescentes. Dependendo do anticorpo escolhido, outras condições podem ser examinadas, como diferenciação, danos ao DNA via irradiação, etc.
Territórios cromossômicos também podem ser visualizados dentro de halos de DNA usando FISH. Devido à preparação que permite o spooling do DNA para fora dos núcleos, a forma do território cromossômico pode ser perturbada, com quantidades menores ou maiores do cromossomo encontradas no halo do DNA, dependendo da ancoragem do genoma dentro do núcleo residual e suas estruturas. A Figura 3 revela um painel de halos de DNA pelo qual cromossomos individuais foram revelados com sondas específicas de pintura cromossômica de braço inteiro (vermelho) para os cromossomos 1, 13, 17 e 18. O anti-pKi67 (verde) tem sido utilizado para marcar células em proliferação e sua ausência dentro de uma mesma cultura, sobre a mesma lâmina, denotando células senescentes. É muito óbvio, pelas imagens e pelos dados apresentados como CTE/NE, que o pequeno cromossomo 18 é um cromossomo que tem poucos anexos e carretéis mais para fora no halo de DNA longe dos núcleos residuais e está significativamente mais longe do centro dos núcleos residuais do que os outros cromossomos. No entanto, isso também é verdade para o cromossomo 1 também. Utilizando o marcador proliferativo anti-pKi67 também foi possível comparar células proliferantes com células senescentes, dentro de uma mesma cultura, e na mesma lâmina, e esta análise revelou que os cromossomos dentro desses dois status celulares muito diferentes não são significativamente diferentes um do outro, no que diz respeito à ligação com as estruturas nucleares residuais.
Curiosamente, os genes também estão mostrando diferenças estatisticamente significativas entre células em proliferação e senescentes com relação a permanecerem dentro de um núcleo residual ou estarem localizadas no DNA Halo. A Figura 4 demonstra isso com loci gênicos delineados por sondas BAC marcadas em vermelho e anti-Ki67 em verde. Não há diferenças significativas entre a localização dos genes nas células em proliferação versus as senescentes, após uma preparação de DNA Halo. No entanto, há significativamente mais loci CTNNA1 de catenina alfa 1 dentro do halo de DNA do que loci CNDD1 de ciclina D1, onde há muito poucos. A Figura 5 mostra as preparações de halo de DNA com telômeros em verde. O fundo é deixado deliberadamente alto para permitir que os sinais teloméricos sejam visualizados dentro do halo de DNA. Neste conjunto de dados, foram incluídas células quiescentes, ou seja, células que passaram fome de soro por 7 dias e, curiosamente, há significativamente mais telômeros desconectados e localizados dentro dos halos de DNA em células quiescentes do que para células em proliferação e senescentes. Na Figura 5a observa-se a proporção de telômeros no halo de DNA, principalmente para a imagem 'Experimento 2'. Isso corresponde à Figura 5b , onde a porcentagem média de telômeros no halo de DNA é de aproximadamente 17% nas células quiescentes. Há algumas evidências de que nem todos os telômeros em células senescentes podem ser vistos como alguns deles talvez muito curtos.
Este método de halo de DNA tem sido bem sucedido para investigarmos alterações na interação genômica dentro de núcleos em célulasdoentes15. A Figura 6 demonstra diferenças na ligação cromossômica em fibroblastos de controle primário e em células doentes com síndrome típica (mutação da lamina A) e síndrome de Hutchinson-Gilford Progeria atípica, expressando uma isoforma SUN1 diferente e nenhuma mutação na lamina A19. Os cromossomos 1 e 13 apresentam diferenças estatisticamente significativas em sua fixação dentro dos núcleos residuais quando comparados aos halos de DNA controle. A Figura 6b correlaciona a posição de todo o território cromossômico com o núcleo residual e o DNA Halo. Valores de 1 ou menos indicam que o cromossomo está localizado dentro do núcleo residual e valores acima de 1 demonstram cromossomos ou porções de cromossomos dentro do DNA Halo.
Em geral, isso destaca a utilidade do HALO-FISH na investigação de interações genômicas de cromossomos inteiros, genes específicos e telômeros sob uma variedade de condições que afetam o ciclo celular (proliferação, quiescência e senescência) ou dentro de células da doença, por exemplo, progéria e linhagens de células cancerosas. De fato, as diferenças nas interações entre esses estados implicam que o nucleoesqueleto tem um papel importante na regulação de processos-chave dentro do núcleo.

Figura 1: Núcleo extraído do HDF exibindo o núcleo residual e o halo de DNA e visão geral do método de análise. a) Um núcleo HDF preparado através de ensaio de halo de ADN e contracorado com DAPI. O núcleo residual brilhantemente corado mostra DNA ancorado ao nucleoesqueleto e este é cercado pelo DNA não aderido que forma um halo de DNA. Ampliação = x 100; barra de escala 10 μm. (b) O canal azul captura o núcleo corado com DAPI e o DNA circundante. O núcleo residual é selecionado e removido usando ImageJ. A seta representa a distância do centro nuclear à borda nuclear residual (NE). (c) O canal vermelho mostra o sinal da sonda. (d) A imagem indicada 'Resultado' é o resultado da sobreposição do canal vermelho à imagem do canal azul; isso permite a distância do centro nuclear até a borda mais distante do território cromossômico (CTE). Clique aqui para ver uma versão maior desta figura.
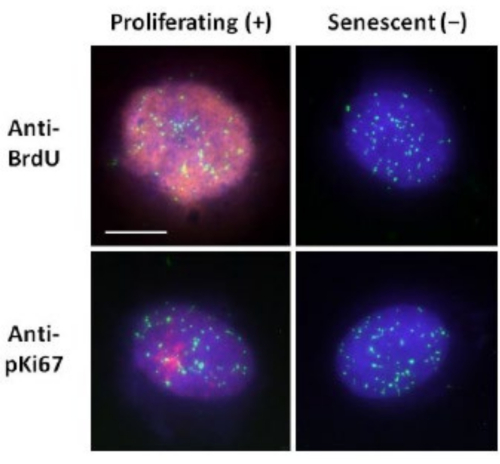
Figura 2: Preparação do halo de DNA com telômero PNA FISH em HDFs em proliferação e senescentes. Telômero PNA FISH em HDFs submetidos ao ensaio de DNA halo. Os sinais teloméricos são visualizados em verde (FITC), o DNA residual e halo foi contracorado usando DAPI (azul) e núcleos em proliferação foram detectados usando anticorpos anti-BrdU ou anti-pKi67 via imunofluorescência indireta em vermelho (TRITC). Ampliação = x 100; barra de escala 10 μm. Clique aqui para ver uma versão maior desta figura.
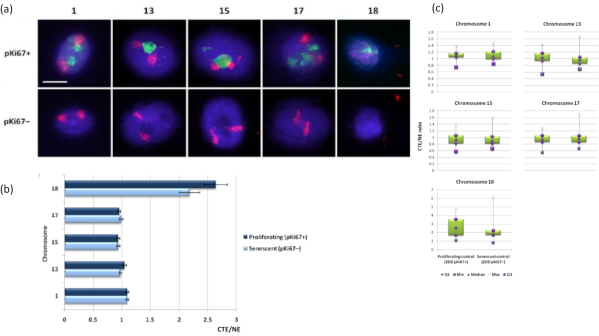
Figura 3: Interações nucleoesqueleto-cromossomo e análise usando ensaio de halo de DNA. (a) FISH 2D com sondas específicas para os cromossomos 1, 13, 15, 17 e 18 foi realizada em HDFs submetidos à preparação de halo de DNA. Cromossomos inteiros foram pintados em vermelho (Cy3) e os núcleos foram sondados com pKi67 para determinar se estavam proliferando ou senescentes. As células em proliferação (pKi67+) foram delineadas em verde (FITC), enquanto as células senescentes permaneceram não coradas (pKi67-), ou seja, nenhum sinal verde foi detectado. Ampliação = x 100; barra de escala 10 μm. (b) Ancoragem cromossômica pelo nucleoesqueleto em HDFs em proliferação e senescentes que sofreram HALO-FISH. As medidas mostram a razão entre a borda do território cromossômico mais distante (CTE) e a respectiva borda nuclear (NE) para os cromossomos 1, 13, 15, 17 e 18 em células em proliferação (pKi67+) e senescentes (pKi67-). ±(c) Representação modificada do box plot da borda do território cromossômico (CTE) para a respectiva borda nuclear (NE) de cromossomos específicos nos núcleos pKi67+ e pKi67-. Q1 = quartil inferior; Min = menor valor registrado; Med = mediana; Max = valor máximo registrado; Q3 = quartil superior. Clique aqui para ver uma versão maior desta figura.
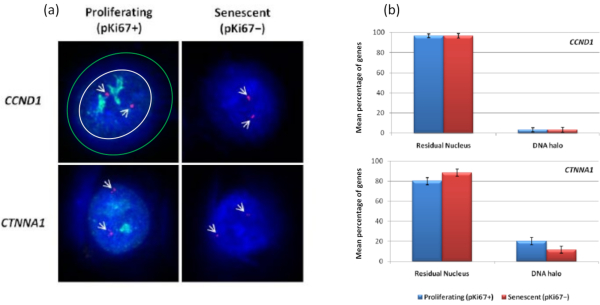
Figura 4: Interações gene-específicas em HDFs usando HALO-FISH. (a) Núcleos extraídos de halo de DNA foram sondados com sondas gênicas específicas (CCND1 e CTNNA1) para investigar sua ancoragem ao MN em células em proliferação e senescentes. Os sinais gênicos são mostrados em vermelho (Cy3) e o anti-pKi67 mostra células em proliferação e o sinal é visualizado em verde (FITC). Para a imagem CCND1 em proliferação, o núcleo residual é fechado dentro do círculo branco, e o espaço entre o círculo branco e verde representa o Halo de DNA. Ampliação = x 100; barra de escala 10 μm. (b) Sinais gene-específicos para CCND1 e CTNNA1 são comparados entre o núcleo residual e o halo de DNA, e também, entre células em proliferação e senescentes. As barras de erro representam ± SEM. Clique aqui para ver uma versão maior desta figura.
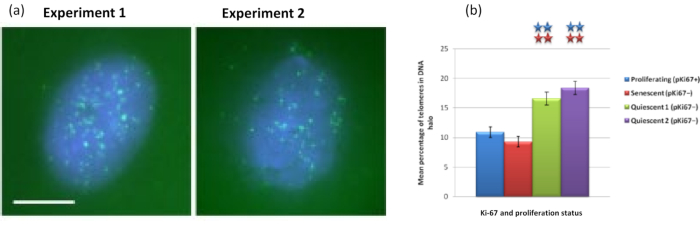
Figura 5: Ensaio de halo de DNA em HDFs quiescentes sondados com telômero PNA-FISH. (a) A quiescência das ICDs foi induzida por cultura em meio de soro baixo por 7 dias. O ensaio do halo de DNA foi realizado, e o PNA-FISH permitiu a visualização dos telômeros pelo sinal FITC (verde) e o núcleo residual e o halo de DNA circundante foram contracorados com DAPI (azul). As células também foram coradas com o anticorpo anti-pKi67 para garantir que os núcleos não proliferassem. Isso se repetiu em duas ocasiões distintas. Ampliação = x 100; barra de escala 10 μm. (b) Comparação da porcentagem média de telômeros localizados dentro do halo de DNA em células HDF proliferantes, senescentes e quiescentes. As barras de erro representam ± SEM. Clique aqui para ver uma versão maior desta figura.
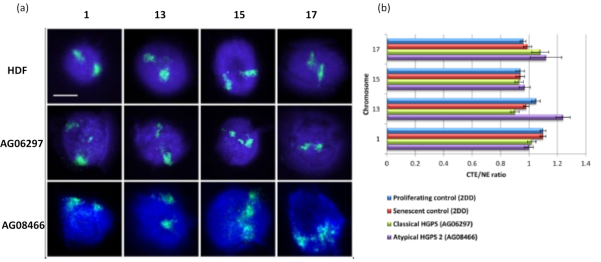
Figura 6: Examinando a ancoragem cromossômica total ao nucleoesqueleto em células HGPS usando HALO-FISH26. (a) Os núcleos controle HDF (2DD), HGPS clássico (AG06297) e HGPS tipo 2 atípico (AG08466) foram submetidos à preparação do halo de DNA e, em seguida, ao 2D-FISH usando tintas cromossômicas inteiras para os cromossomos 1, 13, 15 e 17. Cromossomos inteiros são representados em verde (FITC) e o DNA foi contracorado com DAPI (azul). Ampliação = x 100; barra de escala 10 μm. (b) O posicionamento dos cromossomos dentro dos núcleos extraídos foi determinado medindo-se a razão entre a borda média do território cromossômico (CTE) e a borda nuclear (NE). Uma razão acima de 1 demonstra que a CTE mais distante está fora do NE correspondente dentro do halo de DNA, enquanto uma razão abaixo de 1 significa que a CTE mais distante está dentro do NE dentro do núcleo residual. Clique aqui para ver uma versão maior desta figura.
| Constituintes | Volume (μL) |
| 5XDOP-PCRbuffer | 10 |
| dNTPmix (semdTTP)(2mM) | 5 |
| dTTP (2mM) | 2 |
| Biotina-16-dUTPorDigoxigenina-11-dUTP | 10 |
| DOPprimer (20μM) | 5 |
| TaqDNAPolymerase (1U/μL) | 1 |
| PCRgradewater | 12 |
| Modelo | 5 |
Tabela 1: Tabela mostrando os componentes e volumes do DOP-PCR para uma reação de 1x
| Passo | Ciclos | Temp (grau centígrado) | Hora |
| Desnaturação Inicial | 1 | 95 | 3 minutos |
| Desnaturação | 34 | 98 | Anos 20 |
| Recozimento de Primer | 62 | 1 min | |
| Extensão | 72 | Anos 30 | |
| Extensão Final | 1 | 72 | 5 minutos |
| Arrefecimento | 4 | Segurar |
Tabela 2: Tabela mostrando o ciclo DOP-PCR, temperatura e perfil temporal.
| Constituinte | Volume (μL) |
| Tampão 10x NT (0,5M Tris-HCl pH 8,50 mM MgCl2, 0,5 mg/ml BSA) | 5 |
| 0,1 M de beta-mercaptoetanol | 5 |
| 10X estoque de nucleotídeos (0,5 mM dATP, 0,5 mM dCTP, 0,5 mM dGTP, 0,5 mM dTTP, 0,5 mg/ml biotina-16-dUTP) | 5 |
| Dnase I (1 ng/ml) | 2 |
| DNApolimerase I | 5U por μg de DNA |
| DNAtemplate (1 μg) | 1 |
| Água tratada com DEPC | Até 50 μL |
Tabela 3: Tabela mostrando os componentes e volumes de tradução de nick para uma sonda.
Discussão
O método do halo de DNA é um excelente método de escolha para analisar interações entre o nucleoesqueleto e o genoma, no entanto, existem algumas etapas críticas que também devem ser seguidas. Um dos parâmetros mais importantes é a otimização da densidade de semeadura celular. Se as células se tornarem mais confluentes, os halos de DNA se sobreporão às células vizinhas, impossibilitando a realização da análise. Os tampões CSK e de extracção devem ser sempre renovados no dia da utilização, com espermina, espermidina e digitonina a serem adicionadas ao tampão de extracção no final do processo de preparação para manter a sua actividade biológica. Ao realizar Halo-FISH, é extremamente importante usar a temperatura de desnaturação correta dos halos de DNA para permitir que a sonda ou tinta hibridize posteriormente.
A microscopia eletrônica tem sido utilizada para visualizar a matriz nuclear, sendo identificadas estruturasfilamentosas20. No entanto, a microscopia eletrônica é limitada, pois associações de matriz com cromatina não podem ser facilmente deduzidas. De fato, o método de DNA Halo é mais versátil em comparação com a microscopia eletrônica, pois genes, cromossomos e estados celulares específicos podem ser examinados. Além disso, a análise proteômica de proteínas da matriz nuclear está sendo estudada21,22. Este método é bom para comparar componentes da matriz nuclear, particularmente quando se compara células doentes, no entanto, não fornece a distribuição espacial e os anexos destacados pela técnica padrão de DNA Halo.
Os ensaios de DNA Halo têm limitações. Em primeiro lugar, como a matriz é extraída, isso só pode ser realizado em células fixas, de modo que imagens vivas não são possíveis. Embora o método de Halo de DNA seja relativamente rápido e fácil de executar, o processo geral pode ser demorado quando a cultura de células, a geração de sonda, o Halo-FISH e a análise são levados em consideração.
A captura de imagens de Halos de DNA e HALO-FISH usando microscopia de super-resolução melhoraria muito a resolução de sondas e anticorpos específicos de DNA. Além disso, como os fluorocromos podem ser mais facilmente resolvidos espectralmente, pode ser possível usar várias sondas de DNA em um único experimento, fornecendo ainda mais informações. Melhorias em técnicas de biologia molecular, como a captura de conformação cromossômica (3C), têm sido usadas para determinar interações de loci gênicos e analisar a organização espacial da cromatina na célula. Ensaios de DNA Halo e 3C podem ser combinados, um termo conhecido como M3C23, demonstrando novamente a adaptabilidade da técnica de DNA Halo.
Os dados originais aqui apresentados são para demonstrar as possibilidades de interrogação do comportamento genômico e como apresentar esses dados. Com esses dados demonstramos que é possível determinar diferenças significativas na ligação genômica utilizando (1) sondas de pintura cromossômica, neste estudo revelando que o cromossomo 18 é o menos aderido dentre os analisados (Figura 3); (2) Loci gênicos com diferenças significativas entre dois loci gênicos e (Figura 4) (3) Telômeros, que estão menos fortemente aderidos em células quiescentes em comparação com células em proliferação e senescentes (Figura 5). Podemos diferenciar células em proliferação e não proliferantes através da presença do marcador de proliferação Ki67 antígeno que é uma proteína insolúvel assim permanece com os núcleos residuais ou usando a incorporação de nucleotídeos para destacar células que passaram pela fase S dentro de um período de tempo específico (Figura 2). Essa técnica também nos permitiu analisar o comportamento do genoma em células que estão comprometidas em seus nucleoesqueletos, ou seja, células de laminopatia e aqui e em Bikkul et al., 2018 revelamos que o genoma pode ser menos firmemente ligado quando comparado às células controle e pode ser restaurado quando tratado com drogas específicas que amenizam o efeito da mutação da lamina A em células HGPS clássicas15. No entanto, mostramos novos dados aqui para as células atípicas HGPS AGO8466, sem mutação na lamina A, mas contendo uma forma incomum da proteína do complexo LINC SUN1 19 na qual o cromossomo13 está menos firmemente ligado (Figura 6).
HALO-FISH é um método único por permitir o estudo de interações genômicas com o nucleoesqueleto em combinação com imunofluorescência indireta para resolver proteínas não removidas do procedimento de extração. Tem sido demonstrado que o nucleoesqueleto é modificado em várias doenças, como certos tipos de câncer19 e a importância de algumas proteínas associadas ao nucleoesqueleto como biomarcadoresdiagnósticos 24,25. Assim, essa técnica tem um papel importante no exame do efeito do nucleoesqueleto na organização/desorganização da cromatina na doença 15,24,25,27 e não se restringe às células humanas, com sondas de pintura cromossômica de outros animais, o mesmo protocolo DNA-halo poderia ser empregado 28.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Gostaríamos de agradecer ao Prof. Michael Bittner pelo gentil presente das sondas de pintura de braço cromossômico. A LG foi apoiada pelo projeto EURO-Laminopathies financiado pela UE e pelo Brunel Progeria Research Fund.
Materiais
| Name | Company | Catalog Number | Comments |
| 10X PBS | Thermo Fisher Scientific | 10388739 | Used to create DNA halos |
| 5-bromo-2′-deoxy-uridine | Sigma-Aldrich | B5002-100MG | Labelled nucleotide |
| 5-Fluoro-2′-deoxyuridine | Sigma-Aldrich | F0503-100MG | Labelled nucleotide |
| Agar Technical | Thermo Fisher Scientific | 15562141 | DNA isolation of BAC clones |
| Agarose | Sigma-Aldrich | A939-50G | Check product size of DOP-PCR and nick translation |
| Atypical type 2 HGPS fibroblasts (AG08466) | Coriell Institute | AG08466 | Cell line |
| Bacto tryptone | Thermo Fisher Scientific | 16269751 | DNA isolation of BAC clones |
| Biotin-16-dUTP | Roche Diagnostics | 11093711103 | Labelled nucleotides |
| Chloramphenicol | Sigma-Aldrich | C0378-25G | DNA isolation of BAC clones |
| Classical Hutchinson-Gilford progeria syndrome (HGPS) fibroblasts (AG06297) | Coriell Institute | AG0297 | Cell line |
| Coplin jar | Thermo Fisher Scientific | 12608596 | Holds 5 slides or 8 slides back to back |
| Cot-1 DNA | Thermo Fisher Scientific | 15279011 | Block nonspecific hybridization in HALO FISH |
| DEPC-treated water | Sigma-Aldrich | 693520-1L | DNA isolation of BAC clones |
| Dextran sulphate | Sigma-Aldrich | S4030 | Hybridisation mixture |
| Digitonin | Sigma-Aldrich | D141 | Component of extraction buffer |
| Digoxigenin-11-dUTP | Sigma-Aldrich | 11093088910 | Labelled nucleotides |
| Donkey anti-mouse Cy3 | Jackson Laboratory | 715-165-150 | Secondary antibody |
| EDTA | Sigma-Aldrich | E6758 | Component of extraction buffer |
| Ethanol | Component of extraction buffer | ||
| Ethanol | Sigma-Aldrich | 443611 | Probe precipitation and HALO FISH |
| Fetal bovine system | Thermo Fisher Scientific | 26140079 | Cell culture serum |
| Formamide | Thermo Fisher Scientific | 10523525 | 2D FISH of DNA halos |
| Glass wool | Sigma-Aldrich | 18421 | Spin column |
| Herring sperm | Sigma-Aldrich | D7290 | Probe precipitation |
| HXP™ Lamp (metal halide microscope lamp) | OSRAM | HXP-R120W45C VIS | Image capture of DNA halos |
| Hydrochloric acid | Thermo Fisher Scientific | 10313680 | Cleaning microscope slides |
| Isopropanol | Sigma-Aldrich | I9516-25ML | DNA isolation of BAC clones |
| KAPA HiFi PCR Kit | KAPA Biosystems | KK2103 | PCR Kit |
| Leica DM4000 fluorescent microscope with DFC365 FX camera and LAS AF (Version: 4.5.0) image acquisition software. | Leica Microsystems | Image capture of DNA halos | |
| Luria-Bertani agar | Thermo Fisher Scientific | 13274843 | DNA isolation of BAC clones |
| Magnesium chloride | Sigma-Aldrich | M8266 | Component of CSK buffer |
| Methanol | Thermo Fisher Scientific | 10284580 | Cleaning and sterilizing microscope slides |
| Mouse anti-BrdU antibody | BD Pharmingen | B2531-100UL | BrdU visualisation |
| Newborn calf serum | Thermo Fisher Scientific | 16010159 | Cell culture serum and blocking reagent |
| Nick translation kit | Invitrogen | ||
| PCR grade water | Sigma-Aldrich | 693520-1L | PCR and DNA isolation of BAC clones |
| PCR Primers | Sigma-Aldrich | ||
| PIPES | Sigma-Aldrich | P1851 | Component of CSK and extraction buffers |
| Potassium acetate | Sigma-Aldrich | P1190-100G | DNA isolation of BAC clones |
| QuadriPERM® 4 X 12 | SARSTEDT | 94.6077.307 | Square cell culture dish, polysterene with four compartments. This has hydrophobic surface, is sterile, non-pyrogenic/endotoxin-fee and non-cytotoxic. |
| Rabbit Anti-Ki67 antibody | Sigma-Aldrich | ZRB1007-25UL | Proliferation marker |
| Rnase A | Sigma-Aldrich | R6513 | DNA isolation of BAC clones |
| Rubber cement | Halford's | 101836 | 2D FISH of DNA halos |
| Sephadex G-50 | Sigma-Aldrich | S6022-25G | Spin column |
| Sodium acetate | Sigma-Aldrich | S2889 | Probe precipitation |
| Sodium chloride | Sigma-Aldrich | S5886 | Component of CSK, extraction and SSC buffers |
| Sodium citrate | Sigma-Aldrich | C8532 | Component of SSC buffer |
| Sodium dodecyl sulphate | L3771-100G | DNA isolation of BAC clones | |
| Sodium hydroxide | Sigma-Aldrich | S8045-500G | DNA isolation of BAC clones |
| Spermidine | Sigma-Aldrich | S2626 | Component of extraction buffer |
| Spermine | Sigma-Aldrich | S4264 | Component of extraction buffer |
| Streptavidin-Cy3 | Amersham Life Sciences Ltd, Scientific Laboratory Supplies | pa43001 | Probe antibody |
| Sucrose | Sigma-Aldrich | S0389 | Component of CSK buffer |
| Sucrose | Sigma-Aldrich | S0389 | CSK buffer+A66:D68 |
| SuperFrost™ microscope slides | Thermo Fisher Scientific | 12372098 | Microscope slides: 1 mm thickness, 76 mm length, 26 mm width. Uncoated. |
| Swine anti-rabbit TRITC | Dako | ||
| TELO-PNA FISH KIT | Agilent Dako | K532511-8 | Delineation of telomeres |
| Tris-HCl | Sigma-Aldrich | T3253-100G | Column buffer |
| Triton™ X-100 | Sigma-Aldrich | T9284 | Component of CSK buffer |
| Tryptone | Thermo Fisher Scientific | 10158962 | DNA isolation of BAC clones |
| Tween-20 | Sigma-Aldrich | P9416- 100ML | Detergent |
| Vectashield mountant containing DAPI | Vector Laboratories | H-1200 | 2D FISH of DNA halos |
| Whole human chromosome probes | Calbiochem | 2D FISH of DNA halos | |
| Yeast extract | Thermo Fisher Scientific | 10108202 | DNA isolation of BAC clones |
Referências
- Berezney, R., Coffey, D. S. Identification of a nuclear protein matrix. Biochemical Biophysical Research Communications. 60 (4), 1410-1417 (1974).
- Haaf, T., Ward, D. C. High resolution ordering of YAC contigs using extended chromatin and chromosomes. Human Molecular Genetics. 3 (4), 629-633 (1994).
- Parra, I., Windle, B. High resolution visual mapping of stretched DNA by fluorescent hybridization. Nature Genetics. 5 (1), 17-21 (1993).
- Senger, G., et al. Released chromatin: linearized DNA for high resolution fluorescence in situ hybridization. Human Molecular Genetics. 3 (8), 1275-1280 (1994).
- Florijn, R. J., et al. High-resolution DNA Fiber-FISH for genomic DNA mapping and colour bar-coding of large genes. Human Molecular Genetics. 4 (5), 831-836 (1995).
- Elcock, L. S., Bridger, J. M. Fluorescence in situ hybridization on DNA halo preparations and extended chromatin fibres. Methods Molecular Biology. 659, 21-31 (2010).
- Heiskanen, M., et al. Visual mapping by fiber-FISH. Genomics. 30 (1), 31-36 (1995).
- Bensimon, A., et al. Alignment and sensitive detection of DNA by a moving interface. Science. 265 (5181), 2096-2098 (1994).
- Michalet, X., et al. Dynamic molecular combing: stretching the whole human genome for high-resolution studies. Science. 277 (5331), 1518-1523 (1997).
- Wilson, R. H., Coverley, D. Relationship between DNA replication and the nuclear matrix. Genes Cells. 18 (1), 17-31 (2013).
- Wilson, R. H. C., Coverley, D. Transformation-induced changes in the DNA-nuclear matrix interface, revealed by high-throughput analysis of DNA halos. Science Reports. 7 (1), 6475 (2017).
- Iarovaia, O. V., Akopov, S. B., Nikolaev, L. G., Sverdlov, E. D., Razin, S. V. Induction of transcription within chromosomal DNA loops flanked by MAR elements causes an association of loop DNA with the nuclear matrix. Nucleic Acids Research. 33 (13), 4157-4163 (2005).
- Tandara, M., et al. Sperm DNA integrity testing: big halo is a good predictor of embryo quality and pregnancy after conventional IVF. Andrology. 2 (5), 678-686 (2014).
- Repping, S., et al. The use of spermHALO-FISH to determine DAZ gene copy number. Mol Human Reproduction. 9 (4), 183-188 (2003).
- Bikkul, M. U., et al. Farnesyltransferase inhibitor and rapamycin correct aberrant genome organisation and decrease DNA damage respectively, in Hutchinson-Gilford progeria syndrome fibroblasts. Biogerontology. 19 (6), 579-602 (2018).
- Telenius, H., et al. Degenerate oligonucleotide-primed PCR: general amplification of target DNA by a single degenerate primer. Genomics. 13 (3), 718-725 (1992).
- Bridger, J. M., et al. Association of pKi-67 with satellite DNA of the human genome in early G1 cells. Chromosome Research. 6, 13-24 (1998).
- Sales Gil, R., Vagnarelli, P. Ki-67: More Hidden behind a 'Classic Proliferation Marker'. Trends in Biochemical Sciences. 43 (10), 747-748 (2018).
- Bikkul, M. U., et al. Telomere elongation through hTERT immortalization leads to chromosome repositioning in control cells and genomic instability in Hutchinson-Gilford progeria syndrome fibroblasts, expressing a novel SUN1 isoform. Genes Chromosomes Cancer. 58 (6), 341-356 (2019).
- Jackson, D. A., Cook, P. R. Visualization of a filamentous nucleoskeleton with a 23 nm axial repeat. EMBO Journal. 7, 3667-3677 (1988).
- Albrethsen, J., et al. Unravelling the nuclear matrix proteome. Journal of Proteomics. 72, 71-81 (2009).
- Mika, S., Rost, B. NMPdb: Database of nuclear matrix proteins. Nucleic Acids Research. 33, 160-163 (2005).
- Gavrilov, A. A., et al. of the nuclear matrix-bound chromatin hubs by a new M3C experimental procedure. Nucleic Acids Research. 38, 8051-8060 (2010).
- Sjakste, N., et al. Role of the nuclear matrix proteins in malignant transformation and cancer diagnosis. Experimental Oncology. 26 (3), 170-178 (2004).
- Leman, E. S., Getzenberg, R. H. Nuclear structure as a source of cancer specific biomarkers. Journal of Cellular Biochemistry. 104 (6), 1988-1993 (2008).
- Volpi, E. V., Bridger, J. M. FISH glossary: an overview of the fluorescence in situ hybridization technique. Biotechniques. 45 (4), 385-386 (2008).
- Bridger, J. M., Foster, H. A. Senescence and the Genome. Human Interphase Chromosomes. , (2021).
- Foster, H. A., Griffin, D. K., Bridger, J. M. Interphase chromosome positioning in in vitro porcine cells and ex vivo porcine tissues. BMC Cell Biology. 13 (1), 30 (2012).
Erratum
Formal Correction: Erratum: Fluorescence In Situ Hybridization on DNA Halo Preparations to Reveal Whole Chromosomes, Telomeres and Gene Loci
Posted by JoVE Editors on 6/05/2023. Citeable Link.
An erratum was issued for: Fluorescence In Situ Hybridization on DNA Halo Preparations to Reveal Whole Chromosomes, Telomeres and Gene Loci. The Authors section was updated from:
Lauren S. Godwin1
Joanna M. Bridger1
Helen A. Foster2
1Laboratory of Nuclear and Genomic Health, Centre for Genome Engineering and Maintenance, Division of Biosciences, Department of Life Sciences, College of Health, Medicine and Life Sciences, Brunel University London
2Biosciences, Department of Clinical, Pharmaceutical and Biological Science, School of Life and Medical Sciences, University of Hertfordshire
to:
Lauren S. Godwin1
Emily Roberts2
Joanna M. Bridger1
Helen A. Foster2
1Laboratory of Nuclear and Genomic Health, Centre for Genome Engineering and Maintenance, Division of Biosciences, Department of Life Sciences, College of Health, Medicine and Life Sciences, Brunel University London
2Biosciences, Department of Clinical, Pharmaceutical and Biological Science, School of Life and Medical Sciences, University of Hertfordshire
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados