Method Article
Флуоресцентная гибридизация in situ на препаратах гало ДНК для выявления целых хромосом, теломер и локусов генов
В этой статье
Резюме
Сочетание препаратов ДНК-гало с флуоресцентной гибридизацией in situ позволяет проводить анализ геномных взаимодействий с нуклеоскелетом с высоким разрешением. Присоединенный геном приводит к гибридизованным флуоресцентным сигналам, расположенным внутри остаточных экстрагированных ядер, тогда как неприсоединенный геном находится в ореоле ДНК, окружающем остаточные ядра.
Аннотация
Геном связан с несколькими структурами внутри клеточных ядер, чтобы регулировать его активность и закреплять его в определенных местах. Эти структуры в совокупности известны как нуклеоскелет и включают ядерную пластинку, ядрышки и ядерные тела. Хотя существует множество вариантов флуоресцентной гибридизации in situ (FISH) для изучения генома и его организации, они часто ограничены разрешением и предоставляют недостаточную информацию о связи генома с ядерными структурами. Метод гало ДНК использует высокие концентрации соли и неионогенные детергенты для создания петель ДНК, которые остаются привязанными к структурам внутри ядер через области прикрепления в геноме. Здесь извлекаются растворимые ядерные белки, такие как гистоны, липиды и ДНК, не тесно связанные с ядерным матриксом. Это приводит к образованию ореола неприсоединенной ДНК, окружающей остаточное ядро, которое само содержит ДНК, тесно связанную с внутренними ядерными структурами и устойчивыми к экстракции белками. Эти расширенные нити ДНК обеспечивают повышенное разрешение и могут облегчить физическое картирование. В сочетании с FISH этот метод имеет дополнительное преимущество, заключающееся в изучении геномных взаимодействий со всеми структурами, которыми привязан геном. Этот метод, называемый HALO-FISH, очень универсален, благодаря чему ореолы ДНК могут быть объединены с зондами нуклеиновых кислот для выявления локусов генов, целых хромосом, альфа-сателлитов, теломер и даже РНК. Этот метод дает представление о ядерной организации и функционировании в нормальных клетках и при прогрессировании заболевания, например, при раке.
Введение
«Ядерная матрица» была впервые описана Березни и Коффи в 1974 году1. После выполнения экстракции с высоким содержанием соли и обработки нуклеазами ядер печени крыс они идентифицировали белковый структурный каркас. Процедура гало ДНК была впоследствии адаптирована из этого метода и включает удаление растворимых белков таким образом, чтобы сохранялись только ядерный матрикс (NM) и NM-ассоциированные белки и хромосомы. Области прикрепления ДНК расположены у основания петель ДНК и называются областями, присоединенными к матрице (MAR) или областями прикрепления каркаса (SAR), которые устойчивы к экстракции с высокими концентрациями соли и ионным детергентом литий-3,5-дийодосалицилатом (LIS) соответственно. В ореолах ДНК ДНК, связанная с MAR / SAR, связана в остаточном ядре, тогда как петли ДНК простираются от этих участков и образуют ореол ДНК. Теперь мы знаем, что геном привязан через пластинчатые ассоциированные домены (LAD) к ядерной пластинке и через ядрышкоассоциированные области (NAD) и, возможно, через другие ядерные структуры, такие как специфические ядерные тела.
Метод гало ДНК можно использовать для физического картирования ДНК, генов и хромосомных областей, поскольку расширенная ДНК и хроматин обеспечивают большее разрешение, поскольку хроматин лишен гистонов, а ДНК растягивается 2,3,4,5,6. Однако существуют некоторые ограничения при использовании ореолов ДНК для этого приложения. Например, ДНК, тесно связанная с остаточными ядрами ореолов ДНК, может быть недоступна для зондов, что исключает ее из анализа и физического картирования6. Другие методы, такие как fiber-FISH 2,4,5,7 и молекулярное расчесывание 8, также позволяют проводить физическое картирование и имеют то преимущество, что они относительно быстры и просты в выполнении. Оба предпочтительно используются для картирования ДНК генов, а не ореолов ДНК. Эти методы извлекают волокна хроматина с помощью растворителя или солевой экстракции из ядра, однако молекулярное расчесывание, как правило, имеет лучшую воспроизводимость 8,9.
Появляется все больше доказательств того, что нуклеоскелет играет роль в поддержке ключевых ядерных процессов, таких как сайты прикрепления ДНК, ремоделирование хроматина, транскрипция ДНК, репарация ДНК и репликация ДНК11,12. Таким образом, метод гало ДНК был разработан для исследования взаимодействия между нуклеоскелетом и геномом во время этих клеточных активностей и регулярно использовался и освещался в исследованиях. Этот метод также использовался для исследования взаимодействия между геномом и нуклеоскелетом в связи с прогрессированием заболевания с идентификацией связанных со злокачественными новообразованиями изменений в структуре ядра11.
Метод гало ДНК также использовался для исследования взаимосвязи между геномом и нуклеоскелетом во время развития и дифференцировки12. В ряде исследований использовался вариант метода ореола ДНК, известный как галосперм13 или SpermHalo-FISH, в сочетании с FISH14. Хроматин сперматозоидов тесно связан с белками, известными как протамины, и этот метод был разработан для улучшения доступа к ДНК сперматозоидов. Галосперм использовался для исследования целостности ДНК сперматозоидов и определения наличия повреждений ДНК. Сперматозоиды с меньшим повреждением ДНК коррелируют с большим размером ореола ДНК, тогда как сперматозоиды с повышенным уровнем фрагментированной и поврежденной ДНК имели либо небольшие ореолы, либо вообще не имели их. Таким образом, галосперм может быть использован в качестве потенциального прогностического маркера качества эмбриона и успешной беременности при ЭКО13. Этот пример подчеркивает потенциальное клиническое применение этого метода. В нашей работе мы использовали HALO-FISH для оценки изменений в поведении генома и влияния специфических лекарственных препаратов на болезнь преждевременного старения Синдром прогерии Хатчинсона-Гилфорда (HGPS)15.
Вместе эти и другие исследования подчеркивают широту процессов / приложений, для изучения которых может быть использован метод гало ДНК, и полезность этого метода.
протокол
1. Подготовка предметного стекла, стерилизация и культивирование клеток
- Приготовьте 500 мл 10% HCl (v/v) и перелейте в большой стакан.
- Капельный микроскоп помещают по отдельности в кислоту и инкубируют в течение 1 ч при комнатной температуре на шейкере, установленном на 2 x g.
ВНИМАНИЕ: HCl вызывает коррозию и раздражает. Это может вызвать серьезные ожоги кожи, повреждение глаз и раздражение кожи, глаз и дыхательной системы. Убедитесь, что вы носите соответствующие средства индивидуальной защиты, включая нитриловые перчатки, средства защиты глаз и лабораторный халат. - Сцедить кислоту из стакана и промыть десять раз в водопроводной воде, а затем еще десять раз в деионизированной воде.
- Промойте предметные стекла в метаноле дважды и держите в метаноле до стерилизации пламенем.
ВНИМАНИЕ: Метанол является легковоспламеняющейся жидкостью и токсичен при проглатывании, контакте с кожей или при вдыхании. Кроме того, метанол может вызвать повреждение органов, является разъедающим и раздражающим средством. Соблюдайте пределы воздействия на рабочем месте и убедитесь, что вы носите соответствующие средства индивидуальной защиты, включая перчатки из бутилкаучука, средства защиты глаз и лабораторный халат. По возможности обрабатывайте в вытяжном шкафу местной вытяжной вентиляции (LEV). - С помощью металлических щипцов или длинных щипцов извлеките предметное стекло микроскопа из стакана, содержащего метанол. Пламя над горелкой Бунзена для стерилизации и перенос в прямоугольный сосуд для культивирования клеток, содержащий четыре отсека для слайдов, расположенный рядом с горелкой Бунзена.
ВНИМАНИЕ: Flaming позволяет немедленно стерилизовать предметные стекла микроскопа перед использованием; Однако этот метод сопряжен с опасностями. Поскольку метанол легко воспламеняется, важно, чтобы стакан, содержащий предметные стекла, был расположен вдали от горелки Бунзена. Следует использовать длинные щипцы или щипцы, которые плотно захватывают слайды. Уровень метанола в стакане должен просто покрывать предметные стекла, чтобы свести к минимуму количество используемого метанола и чтобы только концы щипцов / щипцов соприкасались с метанолом. Всегда следите за тем, чтобы метанол испарился из щипцов или щипцов после использования и чтобы они остыли, прежде чем помещать обратно в стакан, содержащий предметные стекла и метанол. Стакан должен быть покрыт куском алюминиевой фольги, чтобы лишить кислорода в случае возгорания метанола. Никогда не пламя скользит по ламинарному колпаку класса II, где происходит циркуляция воздуха. - В качестве альтернативы выполните шаги с 1.1 по 1.4, но вместо того, чтобы обжигать предметные стекла после инкубации с метанолом, поместите предметные стекла на безворсовую ткань для сушки на воздухе. После высыхания заверните в алюминиевую фольгу и поместите в стерилизатор или автоклав.
- Выращивают клетки в соответствующей среде с сывороткой в течение не менее 48 ч при 37 °C в 5% CO2 до тех пор, пока не будет достигнуто 60-70% слияние. Этот протокол был выполнен на раннем прохождении дермальных фибробластов человека (HDF) и на классических фибробластах синдрома прогерии Хатчинсона-Гилфорда (HGPS) (AG06297) и атипичных фибробластах HGPS 2 типа (AG08466). Соберите каждый тип клеток и подсчитайте с помощью гемоцитометра для определения плотности клеток. Посев 1 х 105 клеток в среду 10 мл на предметное стекло.
ПРИМЕЧАНИЕ: Плотность клеток важна, так как петли ДНК из разных ядер могут сходиться, если клетки становятся слишком сливающимися. Плотность посева, возможно, потребуется оптимизировать в зависимости от используемого типа клеток, поскольку трансформированные клетки могут размножаться быстрее, в то время как более поздним культурам клеток может потребоваться больше времени, чтобы достичь желаемого слияния. - Если клетки должны быть задержаны в G0 , чтобы стать спокойными, то высевают 1 x 105 клеток (в среде 10 мл) на предметное стекло и оставляют расти на 24 часа. Дважды промойте клетки средой, не содержащей сыворотки, и инкубируйте в стандартной среде, содержащей более низкую концентрацию сыворотки на 0,5% (сыворотка новорожденного теленка, NCS; или эмбриональная бычья сыворотка, FBS) в течение 7 дней.
- Если для анализа ДНК Halo требуется пролиферативный статус клеток, то определяют клетки, прошедшие через S-фазу, путем включения 5-бром-2'-дезокси-уридина (BrdU) в ДНК во время репликации.
- Семенные клетки в норме и растут в течение 24 ч. Удалите питательную среду и замените средой, содержащей BrdU и 5-фтор-2'-дезоксиуридин (3 мкг/мкл). Еще через 24 часа удалите среду, промойте клетки один раз средой (10% NCS), а затем повторно закормите свежей средой (10% NCS). Инкубируйте еще 24 часа, а затем подготовьте предметные стекла для анализа гало ДНК.
2. Подготовка зонда
- Зонды для рисования хромосом и руки
- Создание хромосомных зондов из амплификации отсортированных потоком или микрорассеченных хромосом методом вырожденной олигонуклеотидной полимеразной цепной реакции (ДОФ-ПЦР) по методу Telenius et al.16. Используйте ДОФ-ПЦР для маркировки хромосомных зондов биотином-16-dUTP или дигоксигенином-11-dUTP, как показано в таблице 1. Пожалуйста, ознакомьтесь с инструкциями производителя по профилю усиления, однако условия, используемые для этого эксперимента, показаны в таблице 2.
- Подготовьте зонд руки или всей хромосомы, сложив 8 мкл меченого продукта ПЦР, 7 мкл ДНК Cot-1, 3 мкл спермы сельди, 1/20объема 3 М ацетата натрия (рН 5,4) и 2 объема 100% этанола. Инкубируйте зондовый раствор не менее 30 минут при -80 °C.
ПРИМЕЧАНИЕ: Этот метод может быть использован для создания зондов с одной хромосомой или зондов с несколькими хромосомами, если для каждой интересующей хромосомы используются разные метки (например, биотин-16-UTP и дигоксигенин-11-dUTP).
ВНИМАНИЕ: Этанол является легковоспламеняющейся жидкостью и паром и может вызвать серьезное раздражение глаз. Хранить вдали от источников тепла, горячих поверхностей и источников воспламенения. Соблюдайте пределы воздействия на рабочем месте и убедитесь, что вы носите соответствующие средства индивидуальной защиты, включая перчатки из бутилкаучука, средства защиты глаз и лабораторный халат. Там, где это возможно, работайте в вытяжном шкафу LEV. - Раствор центрифужного зонда при 13 700 x g в течение 15 мин при 4 °C, а затем промыть 70% этанолом. Повторите процедуру центрифугирования и выбросьте надосадочную жидкость, стараясь не нарушить и не потерять гранулу ДНК. Дайте грануле ДНК высохнуть.
- Добавьте 12 мкл гибридизационного буфера (50% формамида, 10% сульфата декстрана, 10% 20-кратного солевого цитрата натрия (SSC; 3 M NaCl, 0,3 М три-цитрата натрия; pH 7,0), 1% (об./об.) полиоксиэтиленсорбинального монолаурата (Tween-20)) к грануле ДНК. Оставьте при 37 °C не менее чем на 2 часа, чтобы гранула ДНК растворилась в буфере гибридизации.
ВНИМАНИЕ: Формамид является канцерогенным и тератогенным, поэтому может нанести серьезный вред будущему ребенку. Если женщина беременна или подозревает, что она беременна, им следует избегать работы с формамидом. Формамид следует использовать в вытяжном шкафу LEV. Соблюдайте пределы воздействия на рабочем месте и убедитесь, что вы носите соответствующие средства индивидуальной защиты, включая перчатки из бутилкаучука, средства защиты глаз и лабораторный халат.
- Выделение ДНК из бактериальных искусственных хромосом (BAC)
- Нанесите небольшую часть запаса глицерина из клона BAC на агаровую пластину Лурия-Бертани (LB) (1% (мас./об.) NaCl; 1% (мас./об.) триптон, 0,5% (мас./об.) дрожжевой экстракт, 1,5% (мас./об.) агар технический, 12,5 мкг/мл (мас./об.) хлорамфеникола). Инкубировать в течение ночи при температуре 37 °C.
- Выберите одну колонию из пластины и инокулируйте 10 мл бульона LB (1% (мас./об.) NaCl, 1% (мас./об.) бактотриптон, 0,5% (мас./об.) дрожжевого экстракта, 12,5 мкг/мл (мас./об.) хлорамфеникола). Оставьте раствор в встряхивающем инкубаторе на ночь при температуре 37 °C.
ВНИМАНИЕ: Хлорамфеникол подозревается в том, что он вызывает рак. Обращайтесь с осторожностью и уменьшите воздействие. - Центрифужную культуру при 1 700 х г в течение 10 мин при комнатной температуре.
- Выбросьте надосадочную жидкость и добавьте в гранулу 300 мкл раствора P1 (15 мМ Tris (pH 8), 10 мМ ЭДТА, 100 мкг/мл РНКазы А). Энергично вихревите и переносите клетки в микроцентрифужную пробирку объемом 2 мл.
- Добавьте в клетки 300 мкл раствора P2 (0,2 М NaOH, 1% (мас./об.) додецилсульфата натрия (SDS) по каплям. Переверните закрытую микроцентрифужную пробирку 5 раз и оставьте при комнатной температуре максимум на 5 минут.
ВНИМАНИЕ: Гидроксид натрия вызывает коррозию и может вызвать серьезные ожоги кожи и повреждение глаз. Он может вызывать коррозию металлов. Обращайтесь с осторожностью и уменьшите воздействие. Соблюдайте ограничения воздействия на рабочем месте и обеспечьте надлежащую индивидуальную защиту, включая перчатки из нитрилового каучука, средства защиты глаз и лабораторный халат. Там, где это возможно, работайте в вытяжном шкафу LEV.
ВНИМАНИЕ: Додецилсульфат натрия является легковоспламеняющимся твердым веществом, вредным при проглатывании и может вызвать раздражение кожи и дыхательных путей. Это также может привести к серьезному повреждению глаз. Убедитесь, что вы носите соответствующие средства индивидуальной защиты, включая перчатки из нитрилового каучука, средства защиты глаз и лабораторный халат. Там, где это возможно, работайте в вытяжном шкафу LEV. - Медленно добавьте 300 мкл P3 (3 М ацетата калия) в клетки и аккуратно перемешайте. Поместите микроцентрифужную пробирку на лед на 10 минут.
- Центрифугу при 8,100 x g в течение 10 мин при 4 °C и перенесите надосадочную жидкость в пробирку, содержащую 800 мкл ледяного изопропанола. Переверните пробирку несколько раз и выдержите при температуре -20 °C в течение ночи.
ВНИМАНИЕ: Изопропанол является легковоспламеняющейся жидкостью и паром и может вызвать серьезное раздражение глаз, сонливость или головокружение. Хранить вдали от источников тепла, горячих поверхностей и источников воспламенения. Соблюдайте ограничения воздействия на рабочем месте и обеспечьте надлежащую индивидуальную защиту, включая перчатки из нитрилового каучука, средства защиты глаз и лабораторный халат. Там, где это возможно, ручка в вытяжном шкафу LEV). - Центрифуга при 8 100 x g в течение 15 мин при 4 °C. Удалите надосадочную жидкость и перенесите в другую трубку. Добавьте 500 мкл ледяного 70% этанола. Переверните пробирку несколько раз и центрифугу при 8 100 x g в течение 5 мин при 4 °C.
- Удалите надосадочную жидкость и высушите гранулы на воздухе при комнатной температуре. После высыхания гранул повторно суспендировать в 40 мкл воды, обработанной диэтилпирокарбонатом (обработанной DEPC), и оставить при температуре 4 °C на ночь. После полной ресуспендирования удалите 5 мкл раствора и нанесите 1% агарозный гель, чтобы проверить наличие ДНК.
- Подготовка БАК с помощью зонда одного гена с помощью трансляции ника
- Используйте имеющиеся в продаже наборы для маркировки переводов ников. Кроме того, используйте следующий протокол. Компоненты и объемы см. в таблице 3 .
- Добавьте компоненты из таблицы 3 вместе в микроцентрифужную пробирку с добавлением ДНК-полимеразы I, осторожно перемешайте и ненадолго центрифугируйте в течение нескольких секунд. Инкубировать раствор при 15 °C в течение 2 ч.
- Чтобы проверить размеры фрагментов, загрузите 5 мкл раствора в 2% агарозный гель. Диапазон размеров фрагмента ДНК должен составлять от 200 до 600.н. Если размеры фрагментов ДНК больше, то продолжают инкубировать раствор еще 15 мин при 15 °C и запускают продукты на 2% агарозном геле.
- Остановите реакцию трансляции ника, добавив 10 мМ ЭДТА, 0,1% SDS (2,5 мкл 0,5 М ЭДТА, pH 8,0 в 100 мкл и 1 мкл 10% SDS в 100 мкл). Нагрейте раствор при 65 °C в течение 5 мин.
- Чтобы удалить неинкорпорированные нуклеотиды, приложите датчик BAC к спиновой колонке. Коммерческие спиновые колонки можно приобрести, или их можно создать с помощью шприца следующим образом:
- Добавьте 30 г Sephadex G-50 к 500 мл буфера колонки (10 мМ Tris-HCl (pH8), 1 мМ ЭДТА, 0,1% SDS). Автоклавная смесь. Кроме того, сделайте 500 мл буфера колонки (без Sephadex G-50) и автоклава.
- Сделайте спиновые колонны, добавив автоклавную стекловату на дно шприца объемом 1 мл. Наполните шприц объемом 1 мл препаратом Sephadex G-50 в буферной колонке. Поместите шприц объемом 1 мл в центрифужную пробирку объемом 15 мл с микроцентрифужной пробиркой без крышки внизу. Центрифуга в дозе 1 600 x g в течение 5 мин.
- Извлеките шприц и выбросьте микроцентрифужную пробирку внизу. Добавьте свежую микроцентрифужную пробирку обратно в центрифужную пробирку объемом 15 мл. Добавьте буфер колонки (без Sephadex G-50) в шприц объемом 1 мл и снова вставьте обратно в центрифужную пробирку объемом 15 мл. Центрифуга в дозе 1 600 x g в течение 5 мин. Повторите этот шаг еще раз дважды.
- Извлеките шприц и вставьте его в центрифужную пробирку объемом 15 мл, содержащую новую чистую микроцентрифужную пробирку. Приложите зонд к шприцу и соберите зонд в микроцентрифужную пробирку.
- Для осаждения ДНК-зонда добавляют в раствор ДНК 5 мкл ДНК сперматозоидов сельди (10 мг/мл), 10 мкл ацетата натрия и 2,25 объема 100% этанола. Аккуратно перемешайте раствор и инкубируйте при температуре -80 °C не менее 1 часа. Центрифуга при 13 700 x g в течение 15 мин при 4 °C.
- Выбросьте надосадочную жидкость и промойте гранулу 200 мкл ледяного 70% этанола в течение 15 минут при 4 ° C. Удалите надосадочную жидкость и высушите на воздухе. После высыхания повторно суспендируйте гранулы в 20 мкл воды, обработанной DEPC, при комнатной температуре в течение нескольких часов или на ночь при 4 ° C. Теперь зонд готов к использованию или может храниться при температуре -20 °C.
- Для каждого предметного стекла смешайте 5 мкл зондовой ДНК с 5 мкл ДНК Cot-1 и высушите с помощью вакуумного концентратора Speed Vac. После высыхания гранулы повторно суспендируют в 12 мкл гибридизационной смеси.
3. Подготовка ДНК Halo
- Извлеките из инкубатора квадратную культуральную чашку, содержащую предметные стекла и прикрепленные клетки. Выбросьте среду, пометьте предметные стекла карандашом и поместите в банку Coplin, содержащую 50 мл буфера ледяного цитоскелета (CSK): 100 мМ NaCl, 3 мМ MgCl2, 0,3 М сахарозы, 10 мМ 1, 4-пиперазиндиетическую анестезульфоновую кислоту (PIPES; pH 7,8),0,5% (об. / об.) Triton X-100, приготовленную в деионизированной воде. Инкубировать 15 минут на льду или при температуре 4 °C.
ВНИМАНИЕ: Triton X-100 может вызвать раздражение кожи и серьезное повреждение глаз. Обращайтесь, используя соответствующие средства индивидуальной защиты, включая нитриловые перчатки, защитные очки и лабораторный халат. - Выбросьте буфер CSK и быстро промойте предметные стекла в 50 мл 1x гало-буфера ДНК (DHB; 140 мМ NaCl, 27 мМ KCl, 110 мМ NaHPO 4, 15 мМ KH2PO 4; pH7,4) три раза, т.е. окуните слайд в банку Coplin, содержащую DHB, и удалите.
- Перенесите предметные стекла в банку Coplin, содержащую 50 мл экстракционного буфера: 2 М NaCl, 10 мМ ТРУБОК (рН 6,8), 10 мМ этилендиаминтетрауксусной кислоты (ЭДТА), 0,1% (мас./об.) дигитонина, 0,05 мМ (об./об.) спермина, 0,125 мМ (об./об.) спермидина. Выдерживать 4 мин при комнатной температуре.
ВНИМАНИЕ: Дигитонин токсичен при проглатывании или контакте с кожей и смертелен при вдыхании. Убедитесь, что дигитонин обрабатывается в вытяжном шкафу LEV, и наденьте лабораторный халат, нитриловые перчатки (с двойной перчаткой), защитные очки и маску. И спермин, и спермидин могут вызвать серьезные ожоги кожи и повреждение глаз, в то время как ЭДТА вызывает серьезное раздражение глаз, поэтому обращайтесь с каждым химическим веществом с осторожностью.
ПРИМЕЧАНИЕ: Приготовьте дигитонин отдельно, растворив порошок в воде при температуре 60-70 °C. Добавьте растворенный дигитонин в экстракционный буфер после охлаждения. Добавьте спермин, спермидин и дигитонин в последний состав экстракционного буфера, чтобы сохранить биологическую активность. - Инкубируйте предметные стекла последовательно в 50 мл 10x DHB (1,4 М NaCl, 270 мМ KCl, 1,1 М NaHPO 4, 150 мМ KH2PO4; PH7.4), 5x, 2x и 1x DHB в течение 1 мин каждый.
- Погружение скользит (прямо внутрь и прямо) через серию последовательного этанола объемом 50 мл, содержащую 10%, 30%, 70% и 95% (об. / об.) этанола.
- Сушите предметные стекла на воздухе и храните при температуре -80 °C до тех пор, пока не будет выполнена двумерная флуоресцентная гибридизация in situ (2D FISH).
4. Двумерная флуоресцентная гибридизация in situ
- Сделайте 20x SSC: 3 M NaCl, 0,3 М три-цитрат натрия, pH 7,0. Этот буфер можно автоклавировать, хранить при комнатной температуре и разбавлять по мере необходимости.
- Изготовьте формамид 70% (об./об.), 2x SSC pH 7,0 и нагрейте до 70 °C на водяной бане.
- Инкубируйте предметные стекла в течение 5 минут каждый через последовательную серию этанола объемом 50 мл 70, 90 и 100% этанола.
- Сушить на воздухе на подогревающей тарелке и выпекать в духовке при температуре 70 °C в течение 5 минут.
- Денатурируют слайды, помещая в 70% формамид, 2x раствор SSC на 2 мин при 70 °C.
ПРИМЕЧАНИЕ: Температура и время имеют решающее значение для шага 4.5. При температуре ДНК не будет денатурировать и зонды не будут гибридизоваться, и никакого сигнала от ДНК ореола FISH не будет получено. - Поместите денатурированное предметное стекло в 50 мл ледяного 70% этанола на 5 минут и пропустите через серию этанола 90%, 95% и 100% при комнатной температуре в течение 5 минут каждый.
- Сушка на воздухе на согревающей плите
- Обращайтесь с зондами общей хромосомы человека с прямой меткой в соответствии с инструкциями производителя. Для этих экспериментов мы использовали краски для цельных хромосом человека 1, 13, 15, 17 и 18. Кроме того, в этом эксперименте использовались зонды генов CCND1 и CTNNA1 .
ПРИМЕЧАНИЕ: Как целые хромосомные зонды, так и ген-специфические зонды BAC были помечены биотином-11-dUTP и обнаружены стрептавидином, конъюгированным с цианином 3 (Cy3). Для зондов для рисования хромосом, изготовленных с помощью (DOP-PCR) и ДНК BAC, помеченных ретрансляцией ника, с этого момента они будут называться зондами ДНК в протоколе и обрабатываться следующим образом. - Денатурировать ДНК-зонд (целую хромосомную краску или геноспецифический зонд) при 75 °C в течение 10 мин в горячем блоке или на водяной бане.
- Нагрейте ДНК-зонды при 37 °C в течение 30 минут в горячем блоке или на водяной бане перед пипеткой 10 мкл на соответствующем предметном стекле.
ПРИМЕЧАНИЕ: Этот шаг важен для блокировки повторяющихся хромосомных последовательностей. Если не выполнять, в ДНК Halo FISH могут продуцироваться неспецифические сигналы. - Наложите зонд на покровное стекло размером 21 мм x 21 мм и уплотните его резиновым цементом.
- Инкубируйте предметные стекла не менее 18 часов при температуре 37 °C в увлажненной гибридизационной камере.
ПРИМЕЧАНИЕ: Увлажненные гибридизационные камеры могут быть изготовлены из сэндвич-боксов, которые содержат несколько слоев увлажненной ткани, и приподнятой платформы, построенной из разрезанных пластиковых пипеток объемом 10 мл для размещения предметных стекол. Он покрыт алюминиевой фольгой, чтобы свести к минимуму воздействие света. - Аккуратно удалите резиновый цемент с помощью щипцов.
- Инкубируйте предметные стекла в 50 мл 50% (об./об.) формамида, 2x SSC, раствор pH 7,0, предварительно подогретый до 45 °C в течение трех 5-минутных инкубаций.
ПРИМЕЧАНИЕ: Дайте покровному стеклу отпасть от предметного стекла при первой инкубации в 50% (об./об.) формамиде, 2x SSC, раствор pH 7,0. Это предотвращает повреждение препарата DNA Halo, которое может быть вызвано «перетаскиванием» покровного стекла. Предметные стекла можно перемешивать в буфере с помощью захвата щипцами, чтобы помочь отсоединить покровное стекло. - Затем поместите предметные стекла в 50 мл 0,1x SSC, раствора pH 7,0, предварительно нагретого до 60 ° C, но помещенного на водяную баню 45 ° C. Инкубируйте в течение 5 минут и замените буфер еще два раза на 5-минутные инкубации.
- Поместите предметные стекла в банку Coplin, содержащую 50 мл 4x SSC, раствор pH 7,0 при комнатной температуре, и инкубируйте в течение 15 минут с тремя сменами буфера.
- Нанесите 100 мкл 4% раствора BSA, 4x SSC на каждое предметное стекло и наложите кусочек парафиновой пленки. Инкубировать при комнатной температуре 10 мин. Это предотвращает неспецифическое связывание антитела.
- Для обнаружения меченого зонда (биотин-16-dUTP) инкубируют со 100 мкл 1:200 (изготовленного в 1% BSA, 4x SSC) стрептавидина-Cy3 в течение 1 ч при комнатной температуре.
ПРИМЕЧАНИЕ: Следуйте инструкциям производителя с разбавлениями антител и тестовым разведением перед экспериментом, чтобы убедиться, что получен хороший сигнал - Поместите предметные стекла в банку Coplin, содержащую 50 мл 4x SSC (0,5% Tween-20) раствора pH 7,0 при комнатной температуре, и инкубируйте в течение 15 минут с тремя сменами буфера. На этом этапе можно установить слайды, как показано на шаге 4.21, если иммунофлюоресценция не требуется.
- Если требуется пролиферативный статус клеток, превращенных в ореолы ДНК, окрашивают антителами против pKi67 после этапов FISH, перед установкой или окрашиванием для инкорпорированного BrdU.
- Промыть предметными стеклами 3 раза по 5 мин каждое в 50 мл 1x фосфатного буферного физиологического раствора (PBS) с последующей блокировкой 4% NCS в PBS в течение 1 ч при комнатной температуре.
- Нанесите на предметное стекло 200 мкл необходимого первичного антитела (кролик античеловеческий pKi67; мышиный анти-BrdU) на предметное стекло, наложите полоску парафиновой пленки и выдержите при комнатной температуре в течение 1 ч.
- Промыть предметные стекла 3 раза в течение 5 мин в 1x PBS и инкубировать при комнатной температуре в течение 1 ч в 200 мкл вторичного антитела, конъюгированного с флуорохромом (pKi67: TRITC против свиней против кроликов; BrdU: ослиная антимышь Cy3). Выполните еще 3 5-минутных стирки с PBS. Все разбавления должны производиться с использованием 1% (об./об.) NCS в PBS в диапазоне рекомендуемых производителями разведений.
- Монтируйте слайды в 20 мкл крепления, содержащего DAPI, и накладывайте покровное стекло размером 22 мм x 50 мм.
5. Теломеры ПНА РЫБЫ
- Для обнаружения теломер используйте набор теломер PNA FISH - FITC; Выполняйте процедуру в соответствии с инструкциями производителя. Процедуру следует проводить при комнатной температуре, если не указано иное.
- Погрузите предметные стекла в солевой раствор с трис-буфером (TBS, pH 7,5) на 2 мин, а затем поместите в 3,7% формальдегид (в TBS; v/v) ровно на 2 мин.
ВНИМАНИЕ: Раствор TBS содержит 10-30% трометамола и 10-30% 2-амино-2- (гидроксиметил) пропан-1,3-диола гидрохлорида. Это может вызвать серьезное раздражение глаз и кожи, поэтому надевайте защитные перчатки и очки/средства защиты лица. Использовать в хорошо проветриваемом помещении. - Вымойте горки в банке Coplin дважды с TBS по 5 минут каждая.
- Погрузите предметные стекла в раствор предварительной обработки на 10 минут, а затем дважды промойте TBS в течение 5 минут на стирку.
- Затем проведите слайды через серию ледяного этанола, содержащую 50 мл 70%, 85% и 95% (об. / об.) этанола в течение 2 мин на концентрацию. После этого дайте горкам высохнуть на воздухе.
- Нанесите 10 мкл Telomere PNA Probe/FITC (или Cy3) в зависимости от выбранного цвета флуоресцентной метки на каждое предметное стекло и накройте покровным стеклом. Инкубировать в предварительно разогретой духовке при температуре 80 °C в течение 5 минут, а затем поставить в темноту примерно на 1 час.
ВНИМАНИЕ: Telomere PNA Probe / FITC содержит 6-100% формамида, который вызывает серьезное раздражение глаз и является тератогенным, поэтому может нанести серьезный вред будущему ребенку. Если женщина беременна или подозревает, что она беременна, им следует избегать работы с формамидом. Формамид следует использовать в вытяжном шкафу LEV и носить соответствующие средства защиты глаз или лица. - Чтобы удалить покровные стекла, погрузите предметные стекла в «Раствор для полоскания» на 1 минуту, а затем поместите в «Раствор для стирки» на 5 минут при 65 ° C.
ВНИМАНИЕ: Промывочный раствор содержит 1-5% полиоксиэтиленоктилфенилового эфира и 1-5% хлорида натрия. Это вызывает коррозию и может привести к серьезному повреждению глаз. Убедитесь, что при работе с моющим раствором надеты защитные очки или средства защиты лица. - Инкубация проходит через 50 мл ледяного этанола (70%, 85% и 95% (об./об.)) в течение 2 мин на концентрацию, а затем сушит на воздухе. После сухого монтажа сдвиньте с помощью крепления, содержащего DAPI, и наложите покровное стекло.
6. Захват и анализ изображений
- Для визуализации ореолов ДНК и хромосомных территорий используйте эпифлуоресцентный микроскоп (например, микроскоп Leica DM4000), захватывающий изображения с помощью масляного объектива HC PL FLUOTAR 100X/1.30 и камеры DFC365FX.
- Захват изображений в оттенках серого и определение цвета для каждого захваченного канала, чтобы включить псевдораскрашивание изображений. В этом эксперименте использовалось коммерческое программное обеспечение (например, программное обеспечение LAS AF версии 4.5.0). Отдельные цветовые каналы были экспортированы в формате TIFF.
- Анализируйте изображения с помощью программы обработки изображений Java Fiji ImageJ. Загрузите изображение, нажав «Файл» и «Открыть».
- Загрузите отдельные каналы изображения или разделите составное изображение на отдельные каналы в оттенках серого, нажав «Изображение» | Цвет | Разделение каналов. Выберите канал изображения и нажмите Изображение | Отрегулируйте , а затем выберите «Яркость и контрастность». Измените соответствующим образом и повторите с другими каналами.
- Создайте маску остаточного ядра, выбрав окрашенный DAPI канал, изображающий ядро. Нажмите на изображение | Отрегулируйте , а затем выберите Порог. Появится диалоговое окно, в котором порог можно изменить, установите флажок «Темный фон ». Изменяйте до тех пор, пока остаточное ядро не станет чистым, нажмите «Применить » и закройте диалоговое окно.
ПРИМЕЧАНИЕ: Это создает двоичную маску на основе интенсивности пикселей, где белые пиксели показывают области интереса, а черные пиксели показывают фон. Повторите ту же процедуру на канале зонда. - Используйте выделение от руки, чтобы обвести периферию остаточного ядра, затем нажмите « Изменить» и «Очистить снаружи». Наложите зондовый канал на остаточное ядро. Это можно сделать, нажав Image| Цвет | Слияние каналов.
- Чтобы установить масштаб измерения в ImageJ, нарисуйте линию на масштабной линейке или между точками двух известных расстояний. Перейдите в раздел « Анализ» и нажмите «Установить масштаб». В диалоговом окне добавьте длину расстояния и нажмите кнопку ОК. Чтобы измерить расстояния, проведите линию между измеряемыми точками и нажмите «Анализировать» | Измерьте. Это передаст значения расстояния в окно данных.
- Измерьте самую яркую интенсивность DAPI, так как она совпадает с центром ядра. От этого измеряют расстояние от ядерного центра до края самой дальней территории хромосомы (CTE). Измерьте расстояние от ядерного центра до ядерной кромки (NE).
- Убедитесь, что результаты представлены в виде соотношения CTE/NE. Здесь расстояние от ядерного центра до края каждой самой дальней территории хромосомы (CTE) делится на расстояние от ядерного центра до каждого соответствующего ядерного края (NE). Это должно быть выполнено минимум на 50 ядрах. Это может быть изображено в виде гистограммы или прямоугольной диаграммы.
- Для анализа теломер проанализируйте минимум 30 ядер на набор данных. Изображения могут быть проанализированы с помощью Fiji ImageJ или вручную для подсчета количества теломер в остаточном ядре и в ореоле ДНК. BrdU или pKi67 позволили дифференцировать пролиферирующие (BrdU/piK67+) и стареющие/покоящиеся (BrdU/pKi67-) ядра. Данные могут быть отображены в виде гистограмм с полосами погрешности, соответствующими стандартной погрешности среднего значения (SEM).
- Используйте t-критерий Стьюдента (непарный) для статистического сравнения результатов с p> 0,05 считается значимым.
Результаты
Этот метод получения гало ДНК помог нам в наших усилиях определить различия в поведении генома в молодых и старых клетках, а также в клетках, полученных в результате преждевременных заболеваний старения с аберрантными нуклеоскелетными белками15. На рисунке 1 показаны примеры ореолов ДНК, где можно увидеть край остаточного ядра, ДНК, оставшуюся внутри остаточного ядра, и неприсоединенную ДНК, которая наматывалась в окружающую область, создавая ореол ДНК. На нем также изображен анализ, показывающий, как получается остаточное ядро, а также измерения NE и CTE. Можно дифференцировать пролиферирующие и непролиферирующие клетки, либо включив меченый нуклеотид, такой как BrdU, когда клетки находятся в S-фазе, либо используя диагностический маркер пролиферации анти-pKi67, который выявляет ядрышки и участки гетерохроматина в клетках G117,18. Предполагается, что первичные клетки, выращенные в сыворотке с высоким содержанием сыворотки без достижения слияния, которые отрицательны для маркеров пролиферации, являются стареющими. Первичные клетки, выращенные в сыворотке с низким содержанием сыворотки или ставшие сливающимися, т.е. ингибированные контактом, которые отрицательны для маркеров пролиферации, считаются покоящимися и смогут вернуться в пролиферативный клеточный цикл при условии правильных питательных веществ и ситуации. Возможность дифференцировать положительные и отрицательные клетки Ki67 позволила нам определить различия между пролиферирующими, покоящимися и стареющими дермальными фибробластами человека. На рисунке 2 показаны ореолы ДНК пролиферирующих дермальных фибробластов человека, созданных из клеток, где BrdU был включен в них во время репликации ДНК, механизм, который не происходит в непролиферирующих клетках, и впоследствии окрашен антителом против BrdU. Окрашивание пролиферативным маркером антителом анти-pKi67 также видно на рисунке 2. Это надежный антиген, который выживает в протоколе FISH и поэтому может быть окрашен для пост-FISH и предварительного монтажа. Таким образом, пролиферирующие клетки положительны (красные) для BrdU и анти-pKi67 (красные) в левой колонке, а непролиферирующие клетки, действительно стареющие клетки на рисунке 2, отображаются в правой колонке. Зеленые сигналы - это отдельные теломеры, выявленные с помощью набора теломер PNA FISH/FITC. Сочетание иммунофлуоресценции с ореолами ДНК позволяет проводить анализ в различных состояниях клеток, как показано на рисунке 2 при исследовании пролиферирующих, покоящихся и стареющих клеток. В зависимости от выбранного антитела могут быть исследованы другие условия, такие как дифференцировка, повреждение ДНК при облучении и т. Д.
Хромосомные территории также могут быть визуализированы в ореолах ДНК с помощью FISH. Из-за препарата, позволяющего сматывать ДНК из ядер, форма территории хромосомы может быть нарушена, при этом в ореоле ДНК обнаруживается меньшее или большее количество хромосомы, в зависимости от закрепления генома внутри остаточного ядра и его структур. На рисунке 3 показана панель ореолов ДНК, на которой отдельные хромосомы были выявлены с помощью специфических зондов для окрашивания хромосом всего плеча (красный) для хромосом 1, 13, 17 и 18. Anti-pKi67 (зеленый) использовался для обозначения пролиферирующих клеток и их отсутствия в одной культуре на одном и том же предметном стекле, обозначая стареющие клетки. Из изображений и данных, представленных в виде CTE/NE, совершенно очевидно, что маленькая бедная генами хромосома 18 представляет собой хромосому, которая имеет мало прикреплений и уходит дальше в ореол ДНК вдали от остаточных ядер и находится значительно дальше от центра остаточных ядер, чем другие хромосомы. Однако это верно и для хромосомы 1. Используя пролиферативный маркер анти-pKi67, также можно было сравнить пролиферирующие со стареющими клетками в одной культуре и на одном и том же предметном стекле, и этот анализ показал, что хромосомы в этих двух очень разных клеточных статусах существенно не отличаются друг от друга в отношении прикрепления к остаточным ядерным структурам.
Интересно, что гены также демонстрируют статистически значимые различия между пролиферирующими и стареющими клетками в отношении того, чтобы оставаться в остаточном ядре или располагаться в ореоле ДНК. На рисунке 4 показано это с локусами генов, очерченными мечеными зондами BAC красного цвета и анти-Ki67 зеленым цветом. Нет существенных различий между расположением генов в пролиферирующих и стареющих клетках после подготовки ДНК Halo. Однако в ореоле ДНК значительно больше локусов катенина альфа 1 CTNNA1 , чем локусов циклина D1 CNDD1 , где их очень мало. На рисунке 5 зеленым цветом показаны препараты гало ДНК с теломерами. Фон намеренно оставляют высоким, чтобы сигналы теломер можно было визуализировать в ореоле ДНК. В этот набор данных были включены спокойные клетки, т. е. клетки, которые голодали в сыворотке в течение 7 дней, и, что интересно, в неприкрепленных и расположенных в ореолах ДНК в покоящихся клетках значительно больше теломер, чем для пролиферирующих и стареющих клеток. На рисунке 5а можно наблюдать долю теломер в ореоле ДНК, особенно для изображения «Эксперимент 2». Это соответствует рисунку 5b , где средний процент теломер в ореоле ДНК составляет примерно 17% в покоящихся клетках. Есть некоторые свидетельства того, что не все теломеры в стареющих клетках можно рассматривать как некоторые из них, возможно, очень короткие.
Этот метод гало ДНК был успешным для нас для исследования изменений взаимодействия генома в ядрах больных клеток15. На рисунке 6 показаны различия в прикреплении хромосом в фибробластах первичного контроля и в пораженных клетках с типичным (мутация ламина А) и атипичным синдромом прогерии Хатчинсона-Гилфорда, экспрессирующим другую изоформу SUN1 и не имеющую мутации ламина А19. Хромосомы 1 и 13 демонстрируют статистически значимые различия в их прикреплении в остаточных ядрах по сравнению с контрольными ореолами ДНК. Рисунок 6b коррелирует положение всей территории хромосомы с остаточным ядром и гало ДНК. Значения 1 или менее указывают на то, что хромосома расположена в остаточном ядре, а значения более 1 демонстрируют хромосомы или части хромосом в ореоле ДНК.
В целом, это подчеркивает полезность HALO-FISH в исследовании геномных взаимодействий целых хромосом, специфических генов и теломер при различных условиях, которые влияют на клеточный цикл (пролиферация, покой и старение) или в болезнетворных клетках, например, прогерия и линии раковых клеток. Действительно, различия во взаимодействиях между этими состояниями подразумевают, что нуклеоскелет играет важную роль в регуляции ключевых процессов в ядре.

Рисунок 1: Извлеченное ядро HDF с отображением остаточного ядра и ореола ДНК и обзор метода анализа. (a) Ядро HDF, полученное с помощью анализа гало ДНК и окрашенное DAPI. Ярко окрашенное остаточное ядро показывает ДНК, прикрепленную к нуклеоскелету, и она окружена неприсоединенной ДНК, которая образует ореол ДНК. Увеличение = х 100; масштабная линейка 10 мкм. (b) Синий канал захватывает ядро, окрашенное DAPI, и окружающую ДНК. Остаточное ядро выбирается и удаляется с помощью ImageJ. Стрелкой показано расстояние от ядерного центра до остаточного ядерного края (NE). c) Красный канал показывает сигнал пробника. (d) изображение, обозначенное как «Результат», является результатом наложения красного канала на изображение синего канала; это позволяет определить расстояние от ядерного центра до края самой дальней территории хромосомы (CTE). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
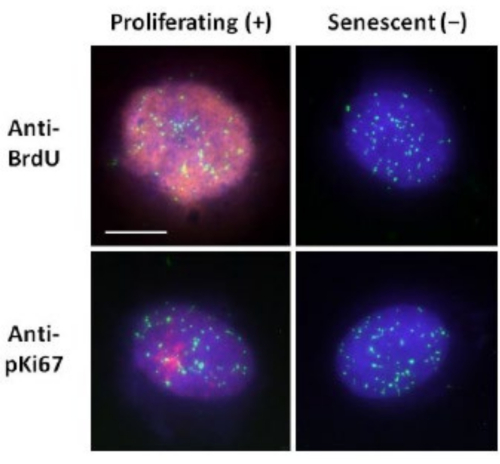
Рисунок 2: Получение гало ДНК с теломерами PNA FISH на пролиферирующих и стареющих HDF. Теломеры PNA FISH на HDF, подвергнутые анализу гало ДНК. Сигналы теломер визуализируются зеленым цветом (FITC), остаточная и гало-ДНК была окрашена с помощью DAPI (синий), а пролиферирующие ядра были обнаружены с использованием антител против BrdU или против pKi67 с помощью непрямой иммунофлюоресценции красного цвета (TRITC). Увеличение = х 100; масштабная линейка 10 мкм. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
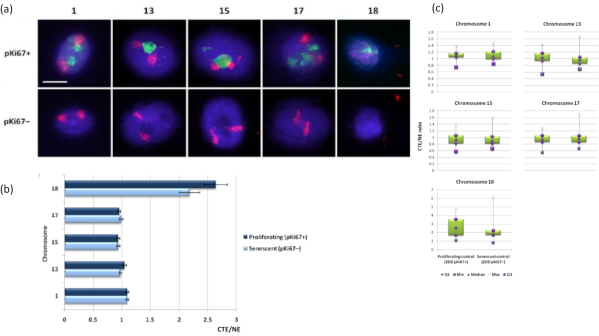
Рисунок 3: Взаимодействие нуклеоскелета и хромосомы и анализ с использованием анализа гало ДНК. (а) 2D-FISH с зондами, специфичными для хромосом 1, 13, 15, 17 и 18, проводили на HDF, подвергнутых препарату ДНК-ореола. Целые хромосомы были окрашены в красный цвет (Cy3), а ядра были исследованы pKi67, чтобы определить, размножаются ли они или стареют. Пролиферирующие клетки (pKi67+) были очерчены зеленым цветом (FITC), тогда как стареющие клетки оставались неокрашенными (pKi67-), т.е. зеленый сигнал не обнаружен. Увеличение = х 100; масштабная линейка 10 мкм. (b) Закрепление хромосом нуклеоскелетом в пролиферирующих и стареющих HDF, подвергшихся HALO-FISH. Измерения показывают отношение самого дальнего края территории хромосомы (CTE) к соответствующему ядерному краю (NE) для хромосом 1, 13, 15, 17 и 18 в пролиферирующих (pKi67+) и стареющих (pKi67-) клетках. Полосы погрешностей представляют ± SEM. (c) Модифицированная коробочная диаграмма представления края территории хромосомы (CTE) до соответствующего ядерного края (NE) конкретных хромосом в ядрах pKi67+ и pKi67-. Q1 = нижний квартиль; Min = наименьшее зарегистрированное значение; Med = медиана; Max = максимальное зарегистрированное значение; Q3 = верхний квартиль. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
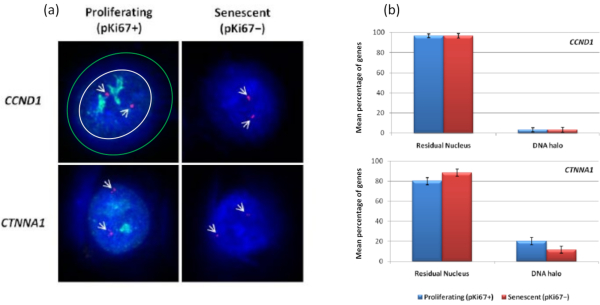
Рисунок 4: Геноспецифические взаимодействия в HDF с использованием HALO-FISH. (a) ядра, извлеченные из ореола ДНК, были исследованы с помощью геноспецифических зондов (CCND1 и CTNNA1) для изучения их привязки к NM на пролиферирующих и стареющих клетках. Сигналы генов показаны красным цветом (Cy3), а анти-pKi67 изображает пролиферирующие клетки, а сигнал визуализируется зеленым цветом (FITC). Для изображения пролиферирующего CCND1 остаточное ядро заключено в белый круг, а пространство между белым и зеленым кругом изображает ореол ДНК. Увеличение = х 100; масштабная линейка 10 мкм. (b) Геноспецифические сигналы для CCND1 и CTNNA1 сравниваются между остаточным ядром и ореолом ДНК, а также между пролиферирующими и стареющими клетками. Полосы погрешностей представляют ± SEM. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
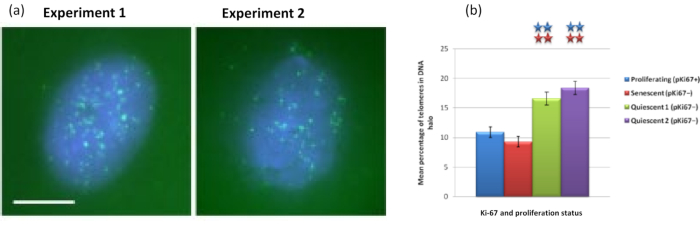
Рисунок 5: Анализ ореола ДНК на покоящихся HDF, исследуемых теломерами PNA-FISH. (a) Покой HDF индуцировали посевом в низкой сывороточной среде в течение 7 дней. Был проведен анализ ореола ДНК, и PNA-FISH позволила визуализировать теломеры сигналом FITC (зеленый), а остаточное ядро и окружающий ореол ДНК были окрашены DAPI (синий). Клетки также окрашивали антителами против pKi67, чтобы убедиться, что ядра не размножаются. Это повторилось в двух отдельных случаях. Увеличение = х 100; масштабная линейка 10 мкм. (b) Сравнение среднего процента теломер, локализованных в ореоле ДНК в пролиферирующих, стареющих и покоящихся клетках HDF. Полосы погрешностей представляют ± SEM. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
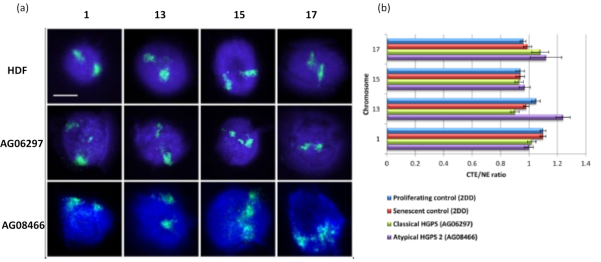
Рисунок 6: Исследование крепления всей хромосомы к нуклеоскелету в клетках HGPS с использованием HALO-FISH26. (a) Контрольные ядра HDF (2DD), классического HGPS (AG06297) и атипичного типа 2 HGPS (AG08466) подверглись подготовке ДНК-гало, а затем 2D-FISH с использованием целых хромосомных красок для хромосом 1, 13, 15 и 17. Целые хромосомы изображены зеленым цветом (FITC), а ДНК была окрашена DAPI (синий). Увеличение = х 100; масштабная линейка 10 мкм. (b) Расположение хромосом в экстрагированных ядрах определяли путем измерения отношения среднего края территории хромосомы (CTE) к ядерному краю (NE). Отношение выше 1 показывает, что самый дальний CTE находится за пределами соответствующего NE в ореоле ДНК, в то время как отношение ниже 1 означает, что самый дальний CTE находится в NE в остаточном ядре. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
| Составляющих | Объем (мкл) |
| 5XDOP-PCRбуфер | 10 |
| dNTPmix (без dTTP) (2 мМ) | 5 |
| dTTP (2 мМ) | 2 |
| Биотин-16-dUTPorDigoxigenin-11-dUTP | 10 |
| DOPprimer (20 мкМ) | 5 |
| TaqDNAPolymeраза (1 Ед / мкл) | 1 |
| PCRgradewater | 12 |
| Шаблон | 5 |
Таблица 1: Таблица, показывающая компоненты и объемы ДОФ-ПЦР для 1-кратной реакции
| Шаг | Циклов | Температура (градус по Цельсию) | Время |
| Первичная денатурация | 1 | 95 | 3 мин |
| Денатурация | 34 | 98 | 20 с |
| Отжиг грунтовки | 62 | 1 мин | |
| Расширение | 72 | 30 с | |
| Окончательное расширение | 1 | 72 | 5 мин |
| Охлаждение | 4 | Держать |
Таблица 2: Таблица, показывающая цикл ДОФ-ПЦР, температурный и временной профиль.
| Составляющая | Объем (мкл) |
| 10x буфер NT (0,5 М Tris-HCl, рН, 8,50 мМ MgCl2, 0,5 мг/мл BSA) | 5 |
| 0,1 М бета-меркаптоэтанол | 5 |
| 10-кратный запас нуклеотидов (0,5 мМ dATP, 0,5 мМ dCTP, 0,5 мМ dGTP, 0,5 мМ dTTP, 0,5 мг/мл биотин-16-dUTP) | 5 |
| Dnase I (1 нг/мл) | 2 |
| ДНКаполимераза I | 5U на мкг ДНК |
| Матрица ДНК (1 мкг) | 1 |
| Вода, очищенная DEPC | До 50 мкл |
Таблица 3: Таблица, показывающая компоненты и объемы перевода ника для одного зонда.
Обсуждение
Метод ореола ДНК является отличным методом выбора при анализе взаимодействия между нуклеоскелетом и геномом, однако есть некоторые важные шаги, которые также необходимо соблюдать. Одним из важнейших параметров является оптимизация плотности засева клеток. Если клетки станут более сливающимися, то ореолы ДНК будут перекрываться с соседними клетками, что сделает невозможным выполнение анализа. ЦСК и экстракционные буферы всегда должны быть свежими в день использования, при этом спермин, спермидин и дигитонин добавляются в экстракционный буфер в конце процесса приготовления для поддержания их биологической активности. При выполнении Halo-FISH чрезвычайно важно использовать правильную температуру денатурации ореолов ДНК, чтобы зонд или краска впоследствии гибридизировались.
Электронная микроскопия была использована для визуализации ядерной матрицы, при этом нитевидные структуры были идентифицированы20. Однако электронная микроскопия ограничена, так как матричные ассоциации с хроматином не могут быть легко выведены. Действительно, метод DNA Halo более универсален по сравнению с электронной микроскопией, поскольку можно исследовать определенные гены, хромосомы и состояния клеток. Кроме того, изучается протеомный анализ белков ядерного матрикса21,22. Этот метод хорош для сравнения компонентов ядерного матрикса, особенно при сравнении больных клеток, однако он не обеспечивает пространственного распределения и прикреплений, выделенных стандартным методом ДНК Halo.
Анализы ДНК Halo имеют ограничения. Во-первых, поскольку матрица извлекается, это может быть выполнено только на фиксированных клетках, поэтому визуализация в реальном времени невозможна. Несмотря на то, что метод DNA Halo относительно быстр и прост в исполнении, общий процесс может занять много времени, если принять во внимание культуру клеток, генерацию зондов, Halo-FISH и анализ.
Захват изображений ДНК Halos и HALO-FISH с использованием микроскопии сверхвысокого разрешения значительно улучшит разрешение ДНК-специфических зондов и антител. Кроме того, поскольку флуорохромы могут быть более легко спектрально разрешены, можно использовать несколько зондов ДНК в одном эксперименте, предоставляя еще больше информации. Усовершенствования в методах молекулярной биологии, такие как захват конформации хромосом (3C), были использованы для определения взаимодействий локусов генов и анализа пространственной организации хроматина в клетке. Анализы ДНК Halo и 3C могут быть объединены, термин, известный как M3C23, что еще раз демонстрирует адаптивность метода DNA Halo.
Исходные данные, представленные здесь, предназначены для демонстрации возможностей опроса поведения генома и того, как представить эти данные. С помощью этих данных мы продемонстрировали, что можно определить значительные различия в прикреплении генома с помощью (1) зондов для рисования хромосом, в этом исследовании выяснилось, что хромосома 18 является наименее прикрепленной хромосомой из проанализированных (рис. 3); (2) Генные локусы со значительными различиями между двумя генными локусами и (рис. 4) (3) теломеры, которые менее сильно прикреплены к покоящимся клеткам по сравнению с пролиферирующими и стареющими клетками (рис. 5). Мы можем различать пролиферирующие и непролиферирующие клетки по присутствию маркера пролиферации антигена Ki67, который является нерастворимым белком, поэтому остается с остаточными ядрами, или используя включение нуклеотидов для выделения клеток, которые прошли через S-фазу в течение определенного периода времени (рис. 2). Этот метод также позволил нам проанализировать поведение генома в клетках, которые скомпрометированы в своих нуклеоскелетах, то есть клетках ламинопатии, и здесь и в Bikkul et al., 2018 мы показываем, что геном может быть менее плотно прикреплен по сравнению с контрольными клетками и может быть восстановлен при лечении конкретными препаратами, которые улучшают эффект мутации ламина А в классических клетках HGPS15. Тем не менее, мы показываем здесь новые данные для атипичных клеток HGPS AGO8466, лишенных мутации ламина А, но содержащих необычную форму белка комплекса LINC SUN1 19, в котором хромосома13 менее плотно прикреплена (рис. 6).
HALO-FISH является уникальным методом, позволяющим изучать геномные взаимодействия с нуклеоскелетом в сочетании с непрямой иммунофлюоресценцией для разрешения белков, не удаленных из процедуры экстракции. Было продемонстрировано, что нуклеоскелет модифицируется при различных заболеваниях, таких как некоторые типы рака19, и важность некоторых белков, связанных с нуклеоскелетом, в качестве диагностических биомаркеров24,25. Таким образом, этот метод играет важную роль в изучении влияния нуклеоскелета на организацию/дезорганизацию хроматина при заболевании 15,24,25,27 и не ограничивается клетками человека, с зондами хромосомного рисования от других животных может быть использован тот же протокол ДНК-гало 28.
Раскрытие информации
Авторам раскрывать нечего.
Благодарности
Мы хотели бы поблагодарить профессора Майкла Биттнера за любезный подарок в виде зондов для рисования хромосом. LG была поддержана финансируемым ЕС проектом EURO-Laminopathies и Исследовательским фондом Брунеля Прогерии.
Материалы
| Name | Company | Catalog Number | Comments |
| 10X PBS | Thermo Fisher Scientific | 10388739 | Used to create DNA halos |
| 5-bromo-2′-deoxy-uridine | Sigma-Aldrich | B5002-100MG | Labelled nucleotide |
| 5-Fluoro-2′-deoxyuridine | Sigma-Aldrich | F0503-100MG | Labelled nucleotide |
| Agar Technical | Thermo Fisher Scientific | 15562141 | DNA isolation of BAC clones |
| Agarose | Sigma-Aldrich | A939-50G | Check product size of DOP-PCR and nick translation |
| Atypical type 2 HGPS fibroblasts (AG08466) | Coriell Institute | AG08466 | Cell line |
| Bacto tryptone | Thermo Fisher Scientific | 16269751 | DNA isolation of BAC clones |
| Biotin-16-dUTP | Roche Diagnostics | 11093711103 | Labelled nucleotides |
| Chloramphenicol | Sigma-Aldrich | C0378-25G | DNA isolation of BAC clones |
| Classical Hutchinson-Gilford progeria syndrome (HGPS) fibroblasts (AG06297) | Coriell Institute | AG0297 | Cell line |
| Coplin jar | Thermo Fisher Scientific | 12608596 | Holds 5 slides or 8 slides back to back |
| Cot-1 DNA | Thermo Fisher Scientific | 15279011 | Block nonspecific hybridization in HALO FISH |
| DEPC-treated water | Sigma-Aldrich | 693520-1L | DNA isolation of BAC clones |
| Dextran sulphate | Sigma-Aldrich | S4030 | Hybridisation mixture |
| Digitonin | Sigma-Aldrich | D141 | Component of extraction buffer |
| Digoxigenin-11-dUTP | Sigma-Aldrich | 11093088910 | Labelled nucleotides |
| Donkey anti-mouse Cy3 | Jackson Laboratory | 715-165-150 | Secondary antibody |
| EDTA | Sigma-Aldrich | E6758 | Component of extraction buffer |
| Ethanol | Component of extraction buffer | ||
| Ethanol | Sigma-Aldrich | 443611 | Probe precipitation and HALO FISH |
| Fetal bovine system | Thermo Fisher Scientific | 26140079 | Cell culture serum |
| Formamide | Thermo Fisher Scientific | 10523525 | 2D FISH of DNA halos |
| Glass wool | Sigma-Aldrich | 18421 | Spin column |
| Herring sperm | Sigma-Aldrich | D7290 | Probe precipitation |
| HXP™ Lamp (metal halide microscope lamp) | OSRAM | HXP-R120W45C VIS | Image capture of DNA halos |
| Hydrochloric acid | Thermo Fisher Scientific | 10313680 | Cleaning microscope slides |
| Isopropanol | Sigma-Aldrich | I9516-25ML | DNA isolation of BAC clones |
| KAPA HiFi PCR Kit | KAPA Biosystems | KK2103 | PCR Kit |
| Leica DM4000 fluorescent microscope with DFC365 FX camera and LAS AF (Version: 4.5.0) image acquisition software. | Leica Microsystems | Image capture of DNA halos | |
| Luria-Bertani agar | Thermo Fisher Scientific | 13274843 | DNA isolation of BAC clones |
| Magnesium chloride | Sigma-Aldrich | M8266 | Component of CSK buffer |
| Methanol | Thermo Fisher Scientific | 10284580 | Cleaning and sterilizing microscope slides |
| Mouse anti-BrdU antibody | BD Pharmingen | B2531-100UL | BrdU visualisation |
| Newborn calf serum | Thermo Fisher Scientific | 16010159 | Cell culture serum and blocking reagent |
| Nick translation kit | Invitrogen | ||
| PCR grade water | Sigma-Aldrich | 693520-1L | PCR and DNA isolation of BAC clones |
| PCR Primers | Sigma-Aldrich | ||
| PIPES | Sigma-Aldrich | P1851 | Component of CSK and extraction buffers |
| Potassium acetate | Sigma-Aldrich | P1190-100G | DNA isolation of BAC clones |
| QuadriPERM® 4 X 12 | SARSTEDT | 94.6077.307 | Square cell culture dish, polysterene with four compartments. This has hydrophobic surface, is sterile, non-pyrogenic/endotoxin-fee and non-cytotoxic. |
| Rabbit Anti-Ki67 antibody | Sigma-Aldrich | ZRB1007-25UL | Proliferation marker |
| Rnase A | Sigma-Aldrich | R6513 | DNA isolation of BAC clones |
| Rubber cement | Halford's | 101836 | 2D FISH of DNA halos |
| Sephadex G-50 | Sigma-Aldrich | S6022-25G | Spin column |
| Sodium acetate | Sigma-Aldrich | S2889 | Probe precipitation |
| Sodium chloride | Sigma-Aldrich | S5886 | Component of CSK, extraction and SSC buffers |
| Sodium citrate | Sigma-Aldrich | C8532 | Component of SSC buffer |
| Sodium dodecyl sulphate | L3771-100G | DNA isolation of BAC clones | |
| Sodium hydroxide | Sigma-Aldrich | S8045-500G | DNA isolation of BAC clones |
| Spermidine | Sigma-Aldrich | S2626 | Component of extraction buffer |
| Spermine | Sigma-Aldrich | S4264 | Component of extraction buffer |
| Streptavidin-Cy3 | Amersham Life Sciences Ltd, Scientific Laboratory Supplies | pa43001 | Probe antibody |
| Sucrose | Sigma-Aldrich | S0389 | Component of CSK buffer |
| Sucrose | Sigma-Aldrich | S0389 | CSK buffer+A66:D68 |
| SuperFrost™ microscope slides | Thermo Fisher Scientific | 12372098 | Microscope slides: 1 mm thickness, 76 mm length, 26 mm width. Uncoated. |
| Swine anti-rabbit TRITC | Dako | ||
| TELO-PNA FISH KIT | Agilent Dako | K532511-8 | Delineation of telomeres |
| Tris-HCl | Sigma-Aldrich | T3253-100G | Column buffer |
| Triton™ X-100 | Sigma-Aldrich | T9284 | Component of CSK buffer |
| Tryptone | Thermo Fisher Scientific | 10158962 | DNA isolation of BAC clones |
| Tween-20 | Sigma-Aldrich | P9416- 100ML | Detergent |
| Vectashield mountant containing DAPI | Vector Laboratories | H-1200 | 2D FISH of DNA halos |
| Whole human chromosome probes | Calbiochem | 2D FISH of DNA halos | |
| Yeast extract | Thermo Fisher Scientific | 10108202 | DNA isolation of BAC clones |
Ссылки
- Berezney, R., Coffey, D. S. Identification of a nuclear protein matrix. Biochemical Biophysical Research Communications. 60 (4), 1410-1417 (1974).
- Haaf, T., Ward, D. C. High resolution ordering of YAC contigs using extended chromatin and chromosomes. Human Molecular Genetics. 3 (4), 629-633 (1994).
- Parra, I., Windle, B. High resolution visual mapping of stretched DNA by fluorescent hybridization. Nature Genetics. 5 (1), 17-21 (1993).
- Senger, G., et al. Released chromatin: linearized DNA for high resolution fluorescence in situ hybridization. Human Molecular Genetics. 3 (8), 1275-1280 (1994).
- Florijn, R. J., et al. High-resolution DNA Fiber-FISH for genomic DNA mapping and colour bar-coding of large genes. Human Molecular Genetics. 4 (5), 831-836 (1995).
- Elcock, L. S., Bridger, J. M. Fluorescence in situ hybridization on DNA halo preparations and extended chromatin fibres. Methods Molecular Biology. 659, 21-31 (2010).
- Heiskanen, M., et al. Visual mapping by fiber-FISH. Genomics. 30 (1), 31-36 (1995).
- Bensimon, A., et al. Alignment and sensitive detection of DNA by a moving interface. Science. 265 (5181), 2096-2098 (1994).
- Michalet, X., et al. Dynamic molecular combing: stretching the whole human genome for high-resolution studies. Science. 277 (5331), 1518-1523 (1997).
- Wilson, R. H., Coverley, D. Relationship between DNA replication and the nuclear matrix. Genes Cells. 18 (1), 17-31 (2013).
- Wilson, R. H. C., Coverley, D. Transformation-induced changes in the DNA-nuclear matrix interface, revealed by high-throughput analysis of DNA halos. Science Reports. 7 (1), 6475 (2017).
- Iarovaia, O. V., Akopov, S. B., Nikolaev, L. G., Sverdlov, E. D., Razin, S. V. Induction of transcription within chromosomal DNA loops flanked by MAR elements causes an association of loop DNA with the nuclear matrix. Nucleic Acids Research. 33 (13), 4157-4163 (2005).
- Tandara, M., et al. Sperm DNA integrity testing: big halo is a good predictor of embryo quality and pregnancy after conventional IVF. Andrology. 2 (5), 678-686 (2014).
- Repping, S., et al. The use of spermHALO-FISH to determine DAZ gene copy number. Mol Human Reproduction. 9 (4), 183-188 (2003).
- Bikkul, M. U., et al. Farnesyltransferase inhibitor and rapamycin correct aberrant genome organisation and decrease DNA damage respectively, in Hutchinson-Gilford progeria syndrome fibroblasts. Biogerontology. 19 (6), 579-602 (2018).
- Telenius, H., et al. Degenerate oligonucleotide-primed PCR: general amplification of target DNA by a single degenerate primer. Genomics. 13 (3), 718-725 (1992).
- Bridger, J. M., et al. Association of pKi-67 with satellite DNA of the human genome in early G1 cells. Chromosome Research. 6, 13-24 (1998).
- Sales Gil, R., Vagnarelli, P. Ki-67: More Hidden behind a 'Classic Proliferation Marker'. Trends in Biochemical Sciences. 43 (10), 747-748 (2018).
- Bikkul, M. U., et al. Telomere elongation through hTERT immortalization leads to chromosome repositioning in control cells and genomic instability in Hutchinson-Gilford progeria syndrome fibroblasts, expressing a novel SUN1 isoform. Genes Chromosomes Cancer. 58 (6), 341-356 (2019).
- Jackson, D. A., Cook, P. R. Visualization of a filamentous nucleoskeleton with a 23 nm axial repeat. EMBO Journal. 7, 3667-3677 (1988).
- Albrethsen, J., et al. Unravelling the nuclear matrix proteome. Journal of Proteomics. 72, 71-81 (2009).
- Mika, S., Rost, B. NMPdb: Database of nuclear matrix proteins. Nucleic Acids Research. 33, 160-163 (2005).
- Gavrilov, A. A., et al. of the nuclear matrix-bound chromatin hubs by a new M3C experimental procedure. Nucleic Acids Research. 38, 8051-8060 (2010).
- Sjakste, N., et al. Role of the nuclear matrix proteins in malignant transformation and cancer diagnosis. Experimental Oncology. 26 (3), 170-178 (2004).
- Leman, E. S., Getzenberg, R. H. Nuclear structure as a source of cancer specific biomarkers. Journal of Cellular Biochemistry. 104 (6), 1988-1993 (2008).
- Volpi, E. V., Bridger, J. M. FISH glossary: an overview of the fluorescence in situ hybridization technique. Biotechniques. 45 (4), 385-386 (2008).
- Bridger, J. M., Foster, H. A. Senescence and the Genome. Human Interphase Chromosomes. , (2021).
- Foster, H. A., Griffin, D. K., Bridger, J. M. Interphase chromosome positioning in in vitro porcine cells and ex vivo porcine tissues. BMC Cell Biology. 13 (1), 30 (2012).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены