Method Article
Ibridazione fluorescenza in situ su preparati di alone di DNA per rivelare interi cromosomi, telomeri e loci genici
In questo articolo
Erratum Notice
Riepilogo
La combinazione di preparazioni di alone di DNA con ibridazione fluorescenza in situ consente un'analisi ad alta risoluzione delle interazioni genomiche con il nucleoscheletro. Il genoma attaccato porta a segnali fluorescenti ibridati situati all'interno dei nuclei residui estratti, mentre il genoma non attaccato è nell'alone di DNA che circonda i nuclei residui.
Abstract
Il genoma è associato a diverse strutture all'interno dei nuclei cellulari, al fine di regolarne l'attività e ancorarla in posizioni specifiche. Queste strutture sono conosciute collettivamente come nucleoscheletro e includono la lamina nucleare, i nucleoli e i corpi nucleari. Sebbene esistano molte varianti dell'ibridazione fluorescenza in situ (FISH) per studiare il genoma e la sua organizzazione, queste sono spesso limitate dalla risoluzione e forniscono informazioni insufficienti sull'associazione del genoma con le strutture nucleari. Il metodo dell'alone di DNA utilizza alte concentrazioni di sale e detergenti non ionici per generare anelli di DNA che rimangono ancorati alle strutture all'interno dei nuclei attraverso regioni di attaccamento all'interno del genoma. Qui vengono estratte proteine nucleari solubili, come istoni, lipidi e DNA non strettamente legati alla matrice nucleare. Ciò porta alla formazione di un alone di DNA non attaccato che circonda un nucleo residuo che a sua volta contiene DNA strettamente associato a strutture nucleari interne e proteine resistenti all'estrazione. Questi filamenti di DNA estesi consentono una maggiore risoluzione e possono facilitare la mappatura fisica. In combinazione con FISH, questo metodo ha l'ulteriore vantaggio di studiare le interazioni genomiche con tutte le strutture a cui è ancorato il genoma. Questa tecnica, denominata HALO-FISH, è altamente versatile per cui gli aloni di DNA possono essere accoppiati con sonde di acido nucleico per rivelare loci genici, cromosomi interi, satelliti alfa, telomeri e persino RNA. Questa tecnica fornisce una visione dell'organizzazione e della funzione nucleare nelle cellule normali e nella progressione della malattia come con il cancro.
Introduzione
La "matrice nucleare" fu descritta per la prima volta da Berezney e Coffey nel 19741. Dopo aver eseguito estrazioni con elevate molarità saline e trattamento con nucleasi su nuclei di fegato di ratto, hanno identificato una struttura strutturale proteica. La procedura dell'alone di DNA è stata successivamente adattata da questo metodo e comporta la rimozione di proteine solubili in modo che persistano solo la matrice nucleare (NM) e le proteine e i cromosomi associati alla NM. Le regioni di attaccamento del DNA si trovano alla base dei circuiti di DNA e sono chiamate regioni attaccate alla matrice (MAR) o regioni di attacco dell'impalcatura (SAR), che sono resistenti all'estrazione con alte concentrazioni di sale e detergente ionico litio-3,5-diiodosalicilato (LIS) rispettivamente. Negli aloni di DNA, il DNA associato a MAR/SAR è legato all'interno del nucleo residuo mentre le anse di DNA si estendono lontano da questi siti e formano l'alone di DNA. Ora sappiamo che il genoma è ancorato attraverso domini associati alla lamina (LAD) alla lamina nucleare e attraverso regioni nucleolari associate (NAD) e possibilmente attraverso altre strutture nucleari come specifici corpi nucleari.
Il metodo dell'alone di DNA può essere utilizzato per la mappatura fisica del DNA, dei geni e delle regioni cromosomiche poiché il DNA esteso e la cromatina forniscono una risoluzione maggiore perché la cromatina viene spogliata degli istoni e il DNA viene allungato 2,3,4,5,6. Tuttavia, ci sono alcune limitazioni quando si utilizzano aloni di DNA per questa applicazione. Ad esempio, il DNA strettamente associato ai nuclei residui degli aloni di DNA può essere inaccessibile alle sonde, precludendogli così l'analisi e la mappatura fisica6. Altre tecniche come il fiber-FISH 2,4,5,7 e la pettinatura molecolare 8 consentono anche la mappatura fisica e hanno il vantaggio di essere relativamente veloci e facili da eseguire. Entrambi sono utilizzati preferenzialmente per la mappatura del DNA dei geni rispetto agli aloni di DNA. Questi metodi estraggono le fibre della cromatina attraverso l'uso di estrazioni di solventi o sali dal nucleo, tuttavia, la pettinatura molecolare tende ad avere una migliore riproducibilità 8,9.
Vi è una crescente evidenza che il nucleoscheletro ha un ruolo nel supportare processi nucleari chiave, come i siti di attaccamento per il DNA, il rimodellamento della cromatina, la trascrizione del DNA, la riparazione del DNA e la replicazione del DNA11,12. Come tale, la tecnica dell'alone di DNA è stata sviluppata per studiare le interazioni tra il nucleoscheletro e il genoma durante queste attività cellulari ed è stata regolarmente utilizzata e riportata nella ricerca. Questa tecnica è stata utilizzata anche per studiare le interazioni tra il genoma e il nucleoscheletro in relazione alla progressione della malattia con cambiamenti associati alla malignità nella struttura nucleare identificati11.
La tecnica dell'alone di DNA è stata utilizzata anche per studiare la relazione tra il genoma e il nucleoscheletro durante lo sviluppo e la differenziazione12. Numerosi studi hanno utilizzato una variazione della tecnica dell'alone di DNA nota come alosperma13 o SpermHalo-FISH se accoppiato con FISH14. La cromatina degli spermatozoi è strettamente legata alle proteine note come protamine e questa tecnica è stata sviluppata per migliorare l'accesso al DNA degli spermatozoi. Halosperm è stato utilizzato per studiare l'integrità del DNA degli spermatozoi e determinare se il danno al DNA è presente. Gli spermatozoi con meno danni al DNA sono correlati a una dimensione dell'alone di DNA più grande, mentre gli spermatozoi con livelli aumentati di DNA frammentato e danneggiato avevano aloni piccoli o nessuno. Pertanto, l'alosperma può essere utilizzato come potenziale marcatore prognostico della qualità embrionale e della gravidanza di successo con la fecondazione in vitro13. Questo esempio sottolinea le potenziali applicazioni cliniche di questa tecnica. Nel nostro lavoro abbiamo utilizzato HALO-FISH per valutare i cambiamenti nel comportamento del genoma e l'effetto di specifici trattamenti farmacologici nella malattia da invecchiamento precoce Sindrome di Hutchinson-Gilford Progeria (HGPS)15.
Insieme, questi e altri studi, evidenziano l'ampiezza dei processi / applicazioni che la tecnica dell'alone di DNA può essere utilizzata per studiare e l'utilità della tecnica.
Protocollo
1. Preparazione dei vetrini, sterilizzazione e coltura cellulare
- Preparare 500 mL di HCl al 10% (v/v) e versare in un becher grande.
- Il microscopio a goccia scorre singolarmente nell'acido e incubare per 1 ora a temperatura ambiente su uno shaker impostato a 2 x g.
ATTENZIONE: HCl è corrosivo e irritante. Può causare gravi ustioni cutanee e danni agli occhi e irritazione della pelle, degli occhi e del sistema respiratorio. Assicurarsi che venga indossata un'adeguata protezione personale, compresi guanti in nitrile, protezione per gli occhi e un camice da laboratorio. - Decantare l'acido dal becher e lavare i vetrini dieci volte in acqua di rubinetto e poi altre dieci volte in acqua deionizzata.
- Risciacquare i vetrini in metanolo due volte e conservare in metanolo fino alla sterilizzazione mediante fiamma.
ATTENZIONE: Il metanolo è un liquido altamente infiammabile e tossico se ingerito, a contatto con la pelle o se inalato. Inoltre, il metanolo può causare danni agli organi, è corrosivo e irritante. Rispettare i limiti di esposizione sul posto di lavoro e assicurarsi che venga indossata un'adeguata protezione personale, compresi guanti di gomma butilica, protezione per gli occhi e un camice da laboratorio. Ove possibile, maneggiare in una cappa aspirante locale di ventilazione (LEV). - Utilizzando pinze metalliche o pinze lunghe, rimuovere un vetrino da microscopio dal becher contenente metanolo. Fiamma su un bruciatore Bunsen per sterilizzare e trasferire in un recipiente rettangolare per la coltura cellulare contenente quattro scomparti per vetrini, situato vicino al bruciatore Bunsen.
ATTENZIONE: La fiammatura consente la sterilizzazione immediata dei vetrini del microscopio prima dell'uso; Tuttavia, questo metodo ha rischi associati. Poiché il metanolo è altamente infiammabile, è importante che il becher contenente i vetrini sia posizionato lontano dal bruciatore Bunsen. Dovrebbero essere usate lunghe pinze o pinze che afferrino saldamente le diapositive. Il livello di metanolo nel becher dovrebbe coprire solo i vetrini, sia per ridurre al minimo la quantità di metanolo utilizzato sia in modo che solo le estremità della pinza / pinza siano in contatto con il metanolo. Assicurarsi sempre che il metanolo sia evaporato dalla pinza o dalle pinze dopo l'uso e che questi si siano raffreddati prima di riporlo nel becher contenente i vetrini e il metanolo. Il becher dovrebbe essere coperto da un pezzo di foglio di alluminio per far morire di fame l'ossigeno nel caso in cui il metanolo prenda fuoco. Non scorrere mai la fiamma all'interno di una cappa a flusso laminare di Classe II dove l'aria viene fatta circolare. - In alternativa, eseguire i passaggi da 1.1 a 1.4, ma piuttosto che bruciare i vetrini dopo l'incubazione con metanolo, posizionare i vetrini sul tessuto privo di lanugine per asciugare all'aria. Una volta asciutto, avvolgere in un foglio di alluminio e mettere in forno sterilizzatore o autoclave.
- Far crescere le cellule nel terreno appropriato con siero per almeno 48 ore a 37 °C in 5% di CO2 fino a raggiungere il 60-70% di confluenza. Questo protocollo è stato eseguito su un passaggio precoce di fibroblasti dermici umani (HDF) e su fibroblasti classici della sindrome di progeria di Hutchinson-Gilford (HGPS) (AG06297) e fibroblasti HGPS di tipo 2 atipici (AG08466). Raccogliere ogni tipo di cellula e contare utilizzando un emocitometro per determinare la densità cellulare. Seme 1 x 105 celle in 10 ml di terreno per vetrino.
NOTA: La densità cellulare è importante in quanto i circuiti di DNA provenienti da nuclei diversi possono convergere se le cellule diventano troppo confluenti. Potrebbe essere necessario ottimizzare le densità di semina a seconda del tipo di cellula utilizzata, poiché le cellule trasformate possono proliferare più rapidamente, mentre le colture cellulari di passaggio successivo possono richiedere più tempo per raggiungere la confluenza desiderata. - Se le cellule devono essere arrestate in G0 per diventare quiescenti, seminare 1 x 105 cellule (in 10 ml di mezzo) per vetrino e lasciarle crescere per 24 ore. Lavare le cellule due volte con terreno privo di siero e incubare in terreno standard che contenga una concentrazione inferiore di siero allo 0,5% (siero di vitello neonato, NCS; o siero fetale bovino, FBS) per 7 giorni.
- Se lo stato proliferativo delle cellule è richiesto per il test dell'alone del DNA, determinare le cellule che sono passate attraverso la fase S mediante l'incorporazione di 5-bromo-2'-deossi-uridina (BrdU) nel DNA durante la replicazione.
- Cellule da seme come al solito e crescono per 24 ore. Rimuovere il terreno di coltura e sostituirlo con terreno contenente BrdU e 5-fluoro-2'-deossiuridina (3 μg/μL). Dopo altre 24 ore, rimuovere il substrato, lavare le celle una volta con mezzo (10% NCS) e quindi ri-alimentare con terreno fresco (10% NCS). Incubare per altre 24 ore e quindi preparare i vetrini per il test dell'alone del DNA.
2. Preparazione della sonda
- Sonde per pittura cromosomica e braccio
- Realizzare sonde cromosomiche dall'amplificazione di cromosomi a flusso ordinati o microdissezionati mediante reazione a catena della polimerasi degenerata da oligonucleotidi (DOP-PCR) utilizzando il metodo di Telenius et al.16. Utilizzare la DOP-PCR per marcare le sonde cromosomiche con Biotina-16-dUTP o Digossigenina-11-dUTP come mostrato nella Tabella 1. Si prega di controllare le istruzioni del produttore per il profilo di amplificazione, tuttavia, le condizioni utilizzate per questo esperimento sono mostrate nella Tabella 2.
- Preparare una sonda a braccio o a un intero cromosoma sommando 8 μL di prodotto PCR marcato, 7 μL di DNA Cot-1, 3 μL di sperma di aringa, 1/20° volume di acetato di sodio 3 M (pH 5,4) e 2 volumi di etanolo al 100%. Incubare la soluzione della sonda per almeno 30 minuti a -80 °C.
NOTA: Questo metodo può essere utilizzato per creare sonde cromosomiche singole o sonde cromosomiche multiple se vengono utilizzate etichette diverse (ad esempio, Biotina-16-UTP e Digossigenina-11-dUTP) per ciascun cromosoma di interesse.
ATTENZIONE: L'etanolo è un liquido e vapore altamente infiammabile e può causare gravi irritazioni agli occhi. Tenere lontano da fonti di calore, superfici calde e fonti di accensione. Rispettare i limiti di esposizione sul posto di lavoro e assicurarsi che venga indossata un'adeguata protezione personale, compresi guanti di gomma butilica, protezione per gli occhi e un camice da laboratorio. Ove possibile, maneggiare in una cappa aspirante LEV. - Soluzione di sonda da centrifuga a 13.700 x g per 15 minuti a 4 °C e quindi lavare con etanolo al 70%. Ripetere la procedura di centrifugazione e scartare il surnatante, facendo attenzione a non disturbare o perdere il pellet di DNA. Lasciare asciugare il pellet di DNA.
- Aggiungere 12 μL di tampone di ibridazione (50% formammide, 10% destrano solfato, 10% 20x citrato salino di sodio (SSC; 3 M NaCl, 0,3 M citrato trisodico; pH 7,0), 1% (v/v) poliossietilene sorbinale monolaurato (Tween-20)) al pellet di DNA. Lasciare agire a 37 °C per almeno 2 ore affinché il pellet di DNA si dissolva nel tampone di ibridazione.
ATTENZIONE: La formammide è cancerogena e teratogena, quindi può causare gravi danni al feto. Se una donna è incinta o sospetta di essere incinta, dovrebbe evitare di lavorare con la formammide. La formammide deve essere utilizzata in una cappa aspirante LEV. Rispettare i limiti di esposizione sul posto di lavoro e assicurarsi che venga indossata un'adeguata protezione personale, compresi guanti di gomma butilica, protezione per gli occhi e un camice da laboratorio.
- Isolamento del DNA da cromosomi artificiali batterici (BAC)
- Strisciare una piccola porzione dello stock di glicerolo dal clone BAC su una piastra di agar Luria-Bertani (LB) (1% (W/V) NaCl; 1% (p/v) triptone, 0,5% (p/v) estratto di lievito, 1,5% (p/v) Agar Technical, 12,5 μg/mL (p/v) cloramfenicolo). Incubare per una notte a 37 °C.
- Selezionare una singola colonia dalla piastra e inoculare 10 ml di brodo LB (1% (p/v) NaCl, 1% (p/v) bactotriptone, 0,5% (p/v) estratto di lievito, 12,5 μg/mL (p/v) cloramfenicolo). Lasciare la soluzione in un incubatore agitatore per una notte a 37 °C.
ATTENZIONE: Il cloramfenicolo è sospettato di causare il cancro. Maneggiare con cura e ridurre l'esposizione. - Coltura in centrifuga a 1.700 x g per 10 minuti a temperatura ambiente.
- Scartare il surnatante e aggiungere 300 μL di soluzione P1 (15 mM Tris (pH 8), 10 mM EDTA, 100 μg/mL RNasi A) al pellet. Vortice vigorosamente e trasferire le cellule in una provetta da microcentrifuga da 2 ml.
- Aggiungere 300 μL di soluzione P2 (0,2 M NaOH, 1% (p/v) di sodio dodecilsolfato (SDS) goccia per goccia nelle cellule. Capovolgere il tubo della microcentrifuga chiuso 5 volte e lasciare a temperatura ambiente per un massimo di 5 minuti.
ATTENZIONE: L'idrossido di sodio è corrosivo e può causare gravi ustioni cutanee e lesioni agli occhi. Può essere corrosivo per i metalli. Maneggiare con cura e ridurre l'esposizione. Rispettare i limiti di esposizione sul posto di lavoro e assicurarsi che venga indossata un'adeguata protezione personale, compresi guanti in gomma nitrilica, protezione per gli occhi e un camice da laboratorio. Ove possibile, maneggiare in una cappa aspirante LEV.
ATTENZIONE: Il sodio dodecilsolfato è un solido infiammabile, dannoso se ingerito e può causare irritazione cutanea e respiratoria. Può anche causare gravi danni agli occhi. Assicurarsi che venga indossata un'adeguata protezione personale, compresi guanti in gomma nitrilica, protezione per gli occhi e un camice da laboratorio. Ove possibile, maneggiare in una cappa aspirante LEV. - Aggiungere lentamente 300 μL di P3 (acetato di potassio 3 M) alle cellule e mescolare delicatamente. Posizionare il tubo della microcentrifuga sul ghiaccio per 10 minuti.
- Centrifugare a 8.100 x g per 10 minuti a 4 °C e trasferire il surnatante in una provetta contenente 800 μL di isopropanolo ghiacciato. Capovolgere il tubo più volte e incubare a -20 °C durante la notte.
ATTENZIONE: L'isopropanolo è un liquido e vapore altamente infiammabile e può causare grave irritazione agli occhi, sonnolenza o vertigini. Tenere lontano da fonti di calore, superfici calde e fonti di accensione. Rispettare i limiti di esposizione sul posto di lavoro e assicurarsi che venga indossata un'adeguata protezione personale, compresi guanti in gomma nitrilica, protezione per gli occhi e un camice da laboratorio. Se possibile, maneggiare in una cappa aspirante. - Centrifugare a 8.100 x g per 15 minuti a 4 °C. Rimuovere il surnatante e trasferirlo in un altro tubo. Aggiungere 500 μL di etanolo ghiacciato al 70%. Invertire più volte il tubo e centrifugare a 8.100 x g per 5 minuti a 4 °C.
- Rimuovere il surnatante e asciugare all'aria il pellet a temperatura ambiente. Una volta che il pellet è asciutto, risospendere in 40 μL di acqua trattata con dietil pirocarbonato (trattata con DEPC) e lasciare a 4 °C per una notte. Una volta completamente risospeso, rimuovere 5 μL di soluzione e caricarli su un gel di agarosio all'1% per verificare la presenza di DNA.
- Preparazione di BAC con sonda a singolo gene tramite traduzione nick
- Utilizza kit di etichettatura per la traduzione dei nick disponibili in commercio. In alternativa, utilizzare il seguente protocollo. Cfr. tabella 3 per i componenti e i volumi.
- Aggiungere i costituenti della Tabella 3 insieme in una provetta da microcentrifuga aggiungendo la DNA Polimerasi I per ultimo, mescolare delicatamente e centrifugare brevemente per alcuni secondi. Incubare la soluzione a 15 °C per 2 ore.
- Per verificare le dimensioni dei frammenti, caricare 5 μL della soluzione su un gel di agarosio al 2%. L'intervallo di dimensioni del frammento di DNA dovrebbe essere compreso tra 200-600 bp. Se le dimensioni dei frammenti di DNA sono maggiori, continuare a incubare la soluzione per altri 15 minuti a 15 °C ed eseguire i prodotti su gel di agarosio al 2%.
- Interrompere la reazione di traduzione del nick aggiungendo 10 mM EDTA, 0,1% SDS (2,5 μL di 0,5 M EDTA, pH 8,0 in 100 μL e 1 μL di 10% SDS in 100 μL). Riscaldare la soluzione a 65 °C per 5 min.
- Per rimuovere i nucleotidi non incorporati, applicare la sonda BAC a una colonna di spin. Le colonne di spin commerciali possono essere acquistate oppure possono essere create utilizzando una siringa come segue:
- Aggiungere 30 g di Sephadex G-50 a 500 mL di tampone a colonna (10 mM Tris-HCl (pH8), 1 mM EDTA, 0,1% SDS). Autoclavare la miscela. Inoltre, fare 500 mL di buffer di colonna (senza Sephadex G-50) e autoclave.
- Crea colonne di rotazione aggiungendo lana di vetro autoclavata sul fondo di una siringa da 1 ml. Riempire la siringa da 1 mL con Sephadex G-50 in tampone a colonna. Introdurre 1 mL di siringa in una provetta da centrifuga da 15 mL dotata di una provetta per microcentrifuga senza coperchio sul fondo. Centrifugare a 1.600 x g per 5 min.
- Rimuovere la siringa ed eliminare il tubo della microcentrifuga sul fondo. Aggiungere nuovamente un tubo di microcentrifuga fresco nel tubo da centrifuga da 15 ml. Aggiungere tampone a colonna (senza Sephadex G-50) alla siringa da 1 mL e reinserirla nuovamente nella provetta da centrifuga da 15 ml. Centrifugare a 1.600 x g per 5 min. Ripetere nuovamente questo passaggio due volte.
- Rimuovere la siringa e inserirla in una provetta da centrifuga da 15 mL contenente una nuova provetta da microcentrifuga pulita. Applicare la sonda alla siringa e raccogliere la sonda nella provetta della microcentrifuga.
- Per precipitare la sonda del DNA aggiungere 5 μL di DNA spermatico di aringa (10 mg/ml), 10 μL di acetato di sodio e 2,25 volume di etanolo al 100% alla soluzione di DNA. Mescolare delicatamente la soluzione e incubare a -80 °C per almeno 1 h. Centrifugare a 13.700 x g per 15 minuti a 4 °C.
- Eliminare il surnatante e lavare il pellet con 200 μL di etanolo ghiacciato al 70 % per 15 minuti a 4 °C. Rimuovere il surnatante e asciugare all'aria. Una volta asciutto, risospendere il pellet in 20 μL di acqua trattata con DEPC a temperatura ambiente per diverse ore o per una notte a 4 °C. La sonda è ora pronta per essere utilizzata o può essere conservata a -20 °C.
- Per ogni vetrino mescolare 5 μL di DNA sonda con 5 μL di DNA Cot-1 e asciugare utilizzando un concentratore di vuoto Speed Vac. Una volta che il pellet si è asciugato risospendere in 12 μL di miscela di ibridazione.
3. Preparazione dell'alone di DNA
- Rimuovere il piatto di coltura quadrato contenente i vetrini e le cellule attaccate dall'incubatore. Scartare il mezzo, etichettare i vetrini con una matita e metterli in un barattolo Coplin contenente 50 mL di tampone citoscheletro ghiacciato (CSK): 100 mM NaCl, 3 mM MgCl2, 0,3 M saccarosio, 10 mM 1, acido 4-piperazinediethanesulfonic (PIPES; pH 7,8), 0,5% (v / v) Triton X-100 costituito in acqua deionizzata. Incubare per 15 minuti su ghiaccio o a 4 °C.
ATTENZIONE: Triton X-100 può causare irritazione cutanea e gravi danni agli occhi. Maneggiare utilizzando dispositivi di protezione individuale appropriati tra cui guanti in nitrile, occhiali e camice da laboratorio. - Eliminare il tampone CSK e sciacquare rapidamente i vetrini in 50 mL di 1x tampone alone DNA (DHB; 140 mM NaCl, 27 mM KCl, 110 mM NaHPO 4, 15 mM KH2PO 4; pH7.4) tre volte, cioè immergere il vetrino nel barattolo Coplin contenente DHB e rimuovere.
- Trasferire i vetrini in un barattolo Coplin contenente 50 mL di tampone di estrazione: 2 M NaCl, 10 mM PIPES (pH 6,8), 10 mM di acido etilendiamminotetraacetico (EDTA), 0,1% (p/v) digitonina, 0,05 mM (v/v) di spermina, 0,125 mM (v/v) di spermidina. Incubare per 4 minuti a temperatura ambiente.
ATTENZIONE: La digitonina è tossica se ingerita o a contatto con la pelle ed è fatale se inalata. Assicurarsi che la digitonina sia maneggiata in una cappa aspirante LEV e indossare un camice da laboratorio, guanti in nitrile (doppio guanto), occhiali di sicurezza e maschera. Sia la spermina che la spermidina possono causare gravi ustioni cutanee e danni agli occhi, mentre l'EDTA provoca gravi irritazioni agli occhi, quindi maneggiare ogni sostanza chimica con cura.
NOTA: Preparare la digitonina separatamente sciogliendo la polvere in acqua ad una temperatura di 60-70 °C. Aggiungere la digitonina disciolta al tampone di estrazione una volta raffreddato. Aggiungere spermina, spermidina e digitonina per ultime al tampone di estrazione per preservare l'attività biologica. - Incubare i vetrini consecutivamente in 50 mL di 10x DHB (1,4 M NaCl, 270 mM KCl, 1,1 M NaHPO 4, 150 mM KH2PO4; PH7.4), 5x, 2x e 1x DHB per 1 min ciascuno.
- Immergere i vetrini (dritti dentro e fuori) attraverso una serie sequenziale di etanolo da 50 ml di etanolo al 10%, 30%, 70% e 95% (v/v).
- Asciugare all'aria i vetrini e conservare a -80 °C fino a quando non viene eseguita l'ibridazione bidimensionale in situ a fluorescenza (2D FISH).
4. Ibridazione bidimensionale a fluorescenza in situ
- Fare 20x SSC: 3 M NaCl, 0,3 M citrato trisodico, pH 7,0. Questo tampone può essere autoclavato, conservato a temperatura ambiente e diluito secondo necessità.
- Produrre formammide al 70% (v/v), 2x SSC pH 7,0 e portare a 70 °C a bagnomaria.
- Incubare i vetrini, per 5 minuti ciascuno, attraverso una serie sequenziale di etanolo da 50 ml di etanolo da 70, 90 e 100%.
- Asciugare all'aria su una piastra riscaldante e cuocere in forno a 70 °C per 5 minuti.
- Denaturare i vetrini ponendo nella soluzione 2x SSC al 70% di formammide per 2 minuti a 70 °C.
NOTA: la temperatura e la temporizzazione sono fondamentali per il passaggio 4.5. Se la temperatura è il DNA non si denatura e le sonde non si ibridano, e nessun segnale sarà ottenuto dall'alone di DNA FISH. - Posizionare il vetrino denaturato in 50 ml di etanolo ghiacciato al 70% per 5 minuti e assumere una serie di etanolo del 90%, 95% e 100% a temperatura ambiente per 5 minuti ciascuno.
- Asciugare all'aria su una piastra riscaldante
- Maneggiare sonde cromosomiche umane totali etichettate direttamente secondo le istruzioni del produttore. Per questi esperimenti abbiamo usato vernici cromosomiche umane 1, 13, 15, 17 e 18. Inoltre, in questo esperimento, sono state utilizzate sonde genetiche CCND1 e CTNNA1 .
NOTA: Sia le sonde cromosomiche intere che le sonde specifiche del gene BAC sono state marcate con biotina-11-dUTP e rilevate da streptavidina coniugata a cianina 3 (Cy3). Per le sonde di pittura cromosomica realizzate da (DOP-PCR) e BAC DNA marcato mediante traduzione nick, queste saranno indicate come sonde DNA da questo punto in avanti nel protocollo e trattate come segue. - Sonda di DNA denaturare (vernice cromosomica intera o sonda gene-specifica) a 75 °C per 10 minuti in un blocco caldo o a bagnomaria.
- Sonde di DNA calde a 37 °C per 30 minuti in un blocco caldo o a bagnomaria prima di pipettare 10 μL sul vetrino appropriato.
NOTA: questo passaggio è importante per bloccare le sequenze cromosomiche ripetitive. Se non eseguiti, possono essere prodotti segnali non specifici nel DNA Halo FISH. - Sonda di sovrapposizione con coprislip da 21 mm x 21 mm e sigillatura con cemento gommato.
- Incubare vetrini per un minimo di 18 h a 37 °C in camera di ibridazione umidificata.
NOTA: Le camere di ibridazione umidificate possono essere realizzate con scatole sandwich che contengono diversi strati di tessuto inumidito e una piattaforma rialzata costruita con pipette di plastica tagliate da 10 ml per appoggiare i vetrini. Questo è coperto in un foglio di alluminio per ridurre al minimo l'esposizione alla luce. - Rimuovere accuratamente il cemento di gomma usando una pinza.
- Vetrini da incubare in 50 mL di formammide al 50% (v/v), 2x SSC, soluzione pH 7,0 preriscaldata a 45 °C per tre incubazioni di 5 minuti.
NOTA: Lasciare cadere il vetrino di copertura dalla vetrina nella prima incubazione in una soluzione di formammide al 50% (v/v), 2x SSC, pH 7,0. Ciò previene danni alla preparazione dell'alone di DNA che potrebbero essere causati dal "trascinamento" del foglietto di copertura. Le guide possono essere agitate nel tampone tramite presa con pinze per aiutare a staccare la copertura. - Quindi, posizionare i vetrini in 50 ml di soluzione 0,1x SSC, pH 7,0 preriscaldata a 60 °C ma posta a bagnomaria a 45 °C. Incubare per 5 minuti e sostituire il buffer altre due volte con incubazioni di 5 minuti.
- Introdurre i vetrini in un barattolo Coplin contenente 50 ml di soluzione 4x SSC, pH 7,0 a temperatura ambiente e incubare per 15 minuti con tre cambi di tampone.
- Applicare 100 μL di soluzione BSA al 4%, 4x SSC su ciascun vetrino e sovrapporre con un pezzo di pellicola di paraffina. Incubare a temperatura ambiente per 10 min. Ciò impedisce il legame non specifico dell'anticorpo.
- Per rilevare la sonda marcata (biotina-16-dUTP), incubare con 100 μL di streptavidina-Cy3 1:200 (prodotta in BSA all'1%, 4x SSC) per 1 ora a temperatura ambiente.
NOTA: Seguire le istruzioni del produttore con le diluizioni degli anticorpi e la diluizione del test prima dell'esperimento per assicurarsi che venga prodotto un buon segnale - Introdurre i vetrini in un barattolo Coplin contenente 50 ml di soluzione 4x SSC (0,5% Tween-20) pH 7,0 a temperatura ambiente e incubare per 15 minuti con tre cambi di tampone. I vetrini possono essere montati in questa fase, come mostrato al punto 4.21, se l'immunofluorescenza non è richiesta.
- Se è richiesto lo stato proliferativo delle cellule trasformate in aloni di DNA, colorare con anticorpi anti-pKi67 dopo le fasi FISH, prima del montaggio o colorare per BrdU incorporato.
- Lavare i vetrini 3 volte per 5 minuti ciascuno in 50 ml di soluzione salina tamponata con fosfato 1x (PBS), quindi bloccare con NCS al 4% in PBS per 1 ora a temperatura ambiente.
- Applicare 200 μL dell'anticorpo primario necessario (coniglio anti-umano pKi67; topo anti-BrdU) sul vetrino, sovrapporre con una striscia di pellicola di paraffina e incubare a temperatura ambiente per 1 ora.
- Lavare i vetrini 3 volte per 5 minuti in 1x PBS e incubare a temperatura ambiente per 1 ora in 200 μL di anticorpo secondario coniugato con fluorocromo (pKi67: suino anti-coniglio TRITC; BrdU: asino anti-topo Cy3). Eseguire altri 3 lavaggi di 5 minuti con PBS. Tutte le diluizioni devono essere effettuate utilizzando NCS all'1% (v/v) in PBS nella gamma di diluizioni suggerite dal produttore.
- Montare vetrini in 20 μL di montante contenente DAPI e sovrapporre con un coprivetrino da 22 x 50 mm.
5. PESCE PNA DEI TELOMERI
- Per rilevare i telomeri, utilizzare il kit PNA FISH per telomeri - FITC; Eseguire la procedura con le istruzioni del produttore. La procedura deve essere eseguita a temperatura ambiente, salvo diversa indicazione.
- Immergere i vetrini in soluzione salina tris-tamponata (TBS, pH 7,5) per 2 minuti e quindi posizionare in formaldeide al 3,7% (in TBS; v / v) per esattamente 2 minuti.
ATTENZIONE: La soluzione TBS contiene il 10-30% di trometamolo e il 10-30% di 2-ammino-2-(idrossimetil) propano-1,3-diolo cloridrato. Ciò può causare gravi irritazioni agli occhi e alla pelle, quindi indossare guanti protettivi e occhiali protettivi / protezioni per il viso. Utilizzare in un'area ben ventilata. - Lavare i vetrini in un barattolo Coplin due volte con TBS per 5 minuti ciascuno.
- Immergere i vetrini in soluzione di pretrattamento per 10 minuti e quindi lavare due volte con TBS per 5 minuti per lavaggio.
- Quindi, prendere le diapositive attraverso una serie di etanolo ghiacciato comprendente 50 ml di etanolo al 70%, 85% e 95% (v / v) per 2 minuti per concentrazione. Successivamente lasciare asciugare i vetrini all'aria.
- Applicare 10 μL di Telomere PNA Probe/FITC (o Cy3) a seconda della scelta della colorazione fluorescente del tag, su ogni vetrino e sovrapposizione di coperchio con un vetrino. Incubare in forno preriscaldato impostato a 80 °C per 5 minuti e poi porre al buio per circa 1 ora.
ATTENZIONE: Telomere PNA Probe/FITC contiene 6-100% formammide, che provoca grave irritazione oculare ed è teratogeno, quindi può causare gravi danni al feto. Se una donna è incinta o sospetta di essere incinta, dovrebbe evitare di lavorare con la formammide. La formammide deve essere utilizzata in una cappa aspirante LEV e deve essere indossata un'adeguata protezione per gli occhi o il viso. - Per rimuovere i vetrini, immergere i vetrini in "Soluzione di risciacquo" per 1 minuto e quindi posizionare nella "Soluzione di lavaggio" per 5 minuti a 65 °C.
ATTENZIONE: La soluzione di lavaggio contiene 1-5% di poliossietilene ottilfenil etere e 1-5% di cloruro di sodio. Questo è corrosivo e può causare gravi danni agli occhi. Assicurarsi che gli occhiali o le protezioni per il viso siano indossati durante la manipolazione della soluzione di lavaggio. - L'incubazione scorre attraverso una serie di etanolo ghiacciato da 50 ml (70%, 85% e 95% (v/v)) per 2 minuti per concentrazione e quindi asciuga all'aria. Una volta a secco, scorrere con un montante contenente DAPI e sovrapporre con un coprivetrino.
6. Acquisizione e analisi delle immagini
- Per visualizzare aloni di DNA e territori cromosomici utilizzare un microscopio a epifluorescenza (ad esempio, microscopio Leica DM4000) che cattura immagini utilizzando un obiettivo olio HC PL FLUOTAR 100X/1.30 e una fotocamera DFC365FX.
- Acquisisci immagini in scala di grigi e definisci il colore per ogni canale acquisito per consentire la pseudocolorazione delle immagini. In questo esperimento è stato utilizzato un software commerciale (ad esempio, software LAS AF versione 4.5.0). I singoli canali di colore sono stati esportati come TIFF.
- Analizzare le immagini utilizzando il programma di elaborazione delle immagini Java Fiji ImageJ. Carica l'immagine premendo File e Apri.
- Caricare canali immagine separati o dividere un'immagine composita in canali in scala di grigi separati facendo clic su Immagine | Colore | Dividere i canali. Seleziona un canale immagine e fai clic su Immagine | Regola, quindi seleziona Luminosità e contrasto. Modificare di conseguenza e ripetere con altri canali.
- Create una maschera del nucleo residuo selezionando il canale colorato DAPI raffigurante il nucleo. Clicca sull'immagine | Regolare , quindi selezionare Soglia. Apparirà una finestra di dialogo in cui la soglia può essere modificata, seleziona la casella Sfondo scuro . Modificare fino a quando il nucleo residuo non è chiaro e premere Applica e chiudere la finestra di dialogo.
NOTA: in questo modo viene creata una maschera binaria basata sull'intensità dei pixel, con pixel bianchi che mostrano le aree di interesse e pixel neri che mostrano lo sfondo. Ripetere la stessa procedura sul canale della sonda. - Utilizzate la selezione a mano libera per delineare la periferia del nucleo residuo, quindi fate clic su Modifica (Edit ) e Cancella all'esterno (Clear Outside). Sovrapporre il canale della sonda al nucleo residuo. Questo può essere fatto premendo Image| Colore | Unisci canali.
- Per impostare la scala di misurazione in ImageJ, tracciare una linea sulla barra della scala o tra i punti di due distanze note. Vai su Analizza e premi Imposta scala. Nella finestra di dialogo aggiungete la lunghezza della distanza e fate clic su OK. Per misurare le distanze, tracciare una linea tra i punti misurati e fare clic su Analizza | Misura. In questo modo i valori di distanza verranno trasferiti in una finestra dati.
- Misurare l'intensità DAPI più brillante in quanto questa coincide con il centro del nucleo. Da questa misura la distanza dal centro nucleare al margine del territorio cromosomico più lontano (CTE). Misurare la distanza del centro nucleare dal bordo nucleare (NE).
- Assicurarsi che i risultati siano rappresentati come un rapporto CTE/NE. Qui la distanza dal centro nucleare a ciascun margine del territorio cromosomico più lontano (CTE) è divisa per la distanza dal centro nucleare a ciascun rispettivo bordo nucleare (NE). Questo dovrebbe essere eseguito su un minimo di 50 nuclei. Questo può essere rappresentato come un grafico a barre o a caselle.
- Per l'analisi dei telomeri, analizzare un minimo di 30 nuclei per set di dati. Le immagini possono essere analizzate utilizzando Fiji ImageJ o manualmente per contare il numero di telomeri all'interno del nucleo residuo e all'interno dell'alone di DNA. BrdU o pKi67 hanno permesso la differenziazione dei nuclei proliferanti (BrdU/piK67+) e senescenti/quiescenti (BrdU/pKi67-). I dati possono essere rappresentati in grafici a barre con barre di errore corrispondenti all'errore standard della media (SEM).
- Utilizzare il test t dello studente (non accoppiato) per confrontare statisticamente i risultati con p> 0,05 considerati significativi.
Risultati
Questo metodo di preparazione dell'alone di DNA ci ha aiutato nei nostri sforzi per determinare le differenze nel comportamento del genoma all'interno di cellule giovani e vecchie, ma anche in cellule derivate da malattie dell'invecchiamento precoce con proteine nucleoscheletriche aberranti15. La Figura 1 mostra esempi di aloni di DNA in cui è possibile vedere il bordo di un nucleo residuo, il DNA che rimane all'interno del nucleo residuo e il DNA non attaccato che si è infilato nell'area circostante creando un alone di DNA. Descrive anche l'analisi che mostra come viene ottenuto il nucleo residuo e le misurazioni di NE e CTE. È possibile distinguere tra cellule proliferanti e non proliferanti incorporando un nucleotide marcato come BrdU quando le cellule sono in fase S o impiegando il marcatore di proliferazione diagnostica anti-pKi67, che rivela nucleoli, e regioni di eterocromatina nelle cellule G117,18. Si presume che le cellule primarie cresciute in alto siero senza raggiungere la confluenza, che sono negative per i marcatori di proliferazione, siano senescenti. Le cellule primarie cresciute in siero basso o sono diventate confluenti, cioè inibite dal contatto che sono negative per i marcatori di proliferazione, sono considerate quiescenti e sarebbero in grado di rientrare nel ciclo cellulare proliferativo dati i nutrienti e la situazione corretti. Essere in grado di distinguere tra cellule Ki67 positive e negative ci ha permesso di determinare le differenze tra fibroblasti dermici umani proliferanti, quiescenti e senescenti. La Figura 2 mostra aloni di DNA di fibroblasti dermici umani proliferanti creati da cellule in cui BrdU è stato incorporato in essi durante la replicazione del DNA, un meccanismo che non si verifica nelle cellule non proliferanti, e successivamente colorato con anticorpi anti-BrdU. La colorazione con l'anticorpo marcatore proliferativo anti-pKi67 è visibile anche nella Figura 2. Questo è un antigene robusto e sopravvive al protocollo FISH e quindi può essere colorato per post-FISH e pre-montaggio. Pertanto, le cellule proliferanti sono positive (rosse) per BrdU e anti-pKi67 (rosso) nella colonna di sinistra e le cellule non proliferanti, anzi le cellule senescenti nella Figura 2 sono visualizzate nella colonna di destra. I segnali verdi sono singoli telomeri rivelati con un kit PNA FISH/FITC dei telomeri. La combinazione di immunofluorescenza con aloni di DNA consente l'analisi durante diversi stati cellulari, come mostrato nella Figura 2 quando si studiano le cellule proliferanti, quiescenti e senescenti. A seconda dell'anticorpo scelto possono essere esaminate altre condizioni, come la differenziazione, il danno al DNA attraverso l'irradiazione, ecc.
I territori cromosomici possono anche essere visualizzati all'interno di aloni di DNA utilizzando FISH. A causa della preparazione che consente lo spooling del DNA fuori dai nuclei, la forma del territorio cromosomico può essere disturbata, con quantità più o meno grandi del cromosoma trovate nell'alone di DNA, a seconda dell'ancoraggio del genoma all'interno del nucleo residuo e delle sue strutture. La Figura 3 rivela un pannello di aloni di DNA in cui sono stati rivelati singoli cromosomi con specifiche sonde di pittura cromosomica dell'intero braccio (rosso) per i cromosomi 1, 13, 17 e 18. Anti-pKi67 (verde) è stato usato per marcare le cellule proliferanti e la sua assenza all'interno della stessa coltura, sullo stesso vetrino, denotando cellule senescenti. È molto evidente dalle immagini e dai dati presentati come CTE / NE che il piccolo cromosoma 18 povero di geni è un cromosoma che ha pochi attaccamenti e bobina più lontano nell'alone di DNA lontano dai nuclei residui ed è significativamente più lontano dal centro dei nuclei residui rispetto agli altri cromosomi. Tuttavia, questo vale anche per il cromosoma 1. Utilizzando il marcatore proliferativo anti-pKi67 è stato anche possibile confrontare la proliferazione con cellule senescenti, all'interno della stessa coltura e sullo stesso vetrino, e questa analisi ha rivelato che i cromosomi all'interno di questi due stati cellulari molto diversi non sono significativamente diversi l'uno dall'altro, per quanto riguarda l'attaccamento con le strutture nucleari residue.
È interessante notare che i geni mostrano anche differenze statisticamente significative tra cellule proliferanti e senescenti rispetto al rimanere all'interno di un nucleo residuo o essere situati nell'alone del DNA. La Figura 4 lo dimostra con loci genici delineati da sonde BAC marcate in rosso e anti-Ki67 in verde. Non ci sono differenze significative tra le posizioni dei geni nelle cellule proliferanti rispetto a quelle senescenti, dopo una preparazione dell'alone di DNA. Tuttavia, ci sono significativamente più loci di catenina alfa 1 CTNNA1 all'interno dell'alone del DNA rispetto ai loci della ciclina D1 CNDD1 , dove ce ne sono pochissimi. La Figura 5 mostra i preparati di alone di DNA con telomeri in verde. Lo sfondo è lasciato volutamente alto per consentire la visualizzazione dei segnali dei telomeri all'interno dell'alone di DNA. In questo insieme di dati sono state incluse cellule quiescenti, cioè cellule che sono state affamate di siero per 7 giorni e curiosamente ci sono significativamente più telomeri non attaccati e situati all'interno degli aloni di DNA nelle cellule quiescenti che per le cellule proliferanti e senescenti. Nella figura 5a si può osservare la proporzione di telomeri nell'alone di DNA, in particolare per l'immagine "Esperimento 2". Ciò corrisponde alla Figura 5b dove la percentuale media di telomeri nell'alone di DNA è di circa il 17% nelle cellule quiescenti. Ci sono alcune prove che non tutti i telomeri nelle cellule senescenti possono essere visti come alcuni di loro forse molto corti.
Questo metodo di alone di DNA ha avuto successo per noi per studiare le alterazioni dell'interazione del genoma all'interno dei nuclei nelle cellule malate15. La Figura 6 mostra le differenze nell'attaccamento cromosomico nei fibroblasti di controllo primario e nelle cellule malate con sindrome di progeria tipica (lamina A) e atipica, che esprime una diversa isoforma SUN1 e nessuna mutazione della lamina A19. I cromosomi 1 e 13 mostrano differenze statisticamente significative nel loro attaccamento all'interno dei nuclei residui rispetto agli aloni di DNA di controllo. La figura 6b correla la posizione dell'intero territorio cromosomico al nucleo residuo e all'alone di DNA. Valori pari o inferiori a 1 indicano che il cromosoma si trova all'interno del nucleo residuo e valori superiori a 1 dimostrano cromosomi o porzioni di cromosomi all'interno dell'alone di DNA.
Nel complesso, ciò evidenzia l'utilità di HALO-FISH nello studio delle interazioni genomiche di interi cromosomi, geni specifici e telomeri in una varietà di condizioni che influenzano il ciclo cellulare (proliferazione, quiescenza e senescenza) o all'interno di cellule patologiche, ad esempio linee cellulari di progeria e cancro. In effetti, le differenze nelle interazioni tra questi stati implicano che il nucleoscheletro ha un ruolo importante nella regolazione dei processi chiave all'interno del nucleo.

Figura 1: Nucleo estratto HDF che mostra il nucleo residuo e l'alone di DNA e panoramica del metodo di analisi. (a) Un nucleo HDF preparato mediante test dell'alone di DNA e controcolorato con DAPI. Il nucleo residuo brillantemente colorato mostra DNA ancorato al nucleoscheletro e questo è circondato dal DNA non attaccato che forma un alone di DNA. Ingrandimento = x 100; barra di scala 10 μm. (b) Il canale blu cattura il nucleo colorato con DAPI e il DNA circostante. Il nucleo residuo viene selezionato e rimosso utilizzando ImageJ. La freccia rappresenta la distanza dal centro nucleare al bordo nucleare residuo (NE). (c) Il canale rosso mostra il segnale della sonda. (d) L'immagine indicata con "Risultato" è il risultato della sovrapposizione del canale rosso all'immagine del canale blu; ciò consente la distanza dal centro nucleare al margine del territorio cromosomico più lontano (CTE). Fare clic qui per visualizzare una versione ingrandita di questa figura.
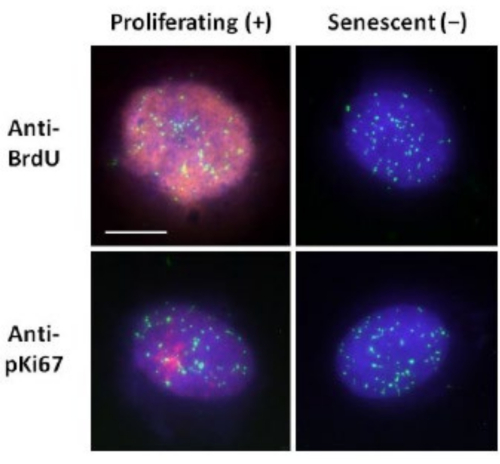
Figura 2: Preparazione dell'alone di DNA con telomeri PNA FISH su HDF proliferanti e senescenti. Telomere PNA FISH su HDF sottoposti ad analisi dell'alone di DNA. I segnali dei telomeri sono visualizzati in verde (FITC), il DNA residuo e dell'alone è stato controcolorato utilizzando DAPI (blu) e i nuclei proliferanti sono stati rilevati utilizzando anticorpi anti-BrdU o anti-pKi67 tramite immunofluorescenza indiretta in rosso (TRITC). Ingrandimento = x 100; Barra di scala 10 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
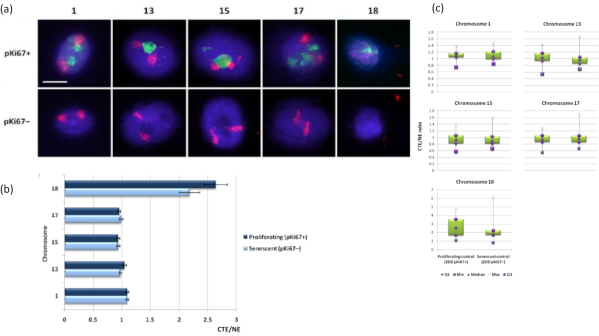
Figura 3: Interazioni nucleoscheletro-cromosoma e analisi mediante saggio dell'alone di DNA. (a) 2D-FISH con sonde specifiche per i cromosomi 1, 13, 15, 17 e 18 è stato eseguito su HDF sottoposti a preparazione di alone di DNA. Interi cromosomi sono stati dipinti in rosso (Cy3) e i nuclei sono stati sondati con pKi67 per determinare se fossero proliferanti o senescenti. Le cellule proliferanti (pKi67+) sono state delineate in verde (FITC), mentre le cellule senescenti sono rimaste non colorate (pKi67-) cioè nessun segnale verde rilevato. Ingrandimento = x 100; barra di scala 10 μm. (b) Ancoraggio cromosomico da parte del nucleoscheletro in HDF proliferanti e senescenti che avevano subito HALO-FISH. Le misurazioni mostrano il rapporto tra il margine del territorio cromosomico più lontano (CTE) e il rispettivo bordo nucleare (NE) per i cromosomi 1, 13, 15, 17 e 18 nelle cellule proliferanti (pKi67+) e senescenti (pKi67-). Le barre di errore rappresentano ± SEM. (c) Rappresentazione modificata del box plot del bordo del territorio cromosomico (CTE) al rispettivo bordo nucleare (NE) di cromosomi specifici nei nuclei di pKi67+ e pKi67-. Q1 = quartile inferiore; Min = valore più basso registrato; Med = mediana; Max = valore massimo registrato; Q3 = quartile superiore. Fare clic qui per visualizzare una versione ingrandita di questa figura.
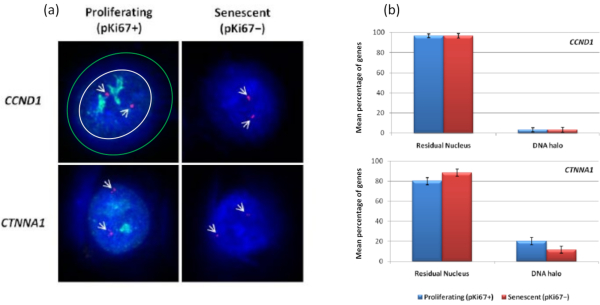
Figura 4: Interazioni gene-specifiche nelle HDF usando HALO-FISH. (a) I nuclei estratti dall'alone di DNA sono stati sondati con sonde gene-specifiche (CCND1 e CTNNA1) per studiare il loro ancoraggio al NM su cellule proliferanti e senescenti. I segnali del gene sono mostrati in rosso (Cy3) e anti-pKi67 raffigura le cellule proliferanti e il segnale viene visualizzato in verde (FITC). Per l'immagine proliferante di CCND1, il nucleo residuo è racchiuso all'interno del cerchio bianco e lo spazio tra il cerchio bianco e verde raffigura l'alone di DNA. Ingrandimento = x 100; Barra di scala 10 μm. (b) I segnali gene-specifici per CCND1 e CTNNA1 sono confrontati tra il nucleo residuo e l'alone di DNA, e anche tra cellule proliferanti e senescenti. Le barre di errore rappresentano ± SEM. Fare clic qui per visualizzare una versione ingrandita di questa figura.
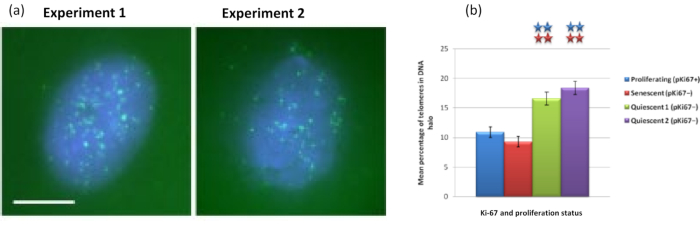
Figura 5: Saggio dell'alone di DNA su HDF quiescenti sondati con telomero PNA-FISH. (a) La quiescenza delle HDF è stata indotta mediante coltura in terreno sierico basso per 7 giorni. È stato eseguito il test dell'alone di DNA e PNA-FISH ha permesso la visualizzazione dei telomeri tramite segnale FITC (verde) e il nucleo residuo e l'alone di DNA circostante sono stati controcolorati con DAPI (blu). Le cellule sono state anche colorate con anticorpi anti-pKi67 per garantire che i nuclei non proliferassero. Ciò è stato ripetuto in due diverse occasioni. Ingrandimento = x 100; Barra di scala 10 μm. (b) Confronto della percentuale media di telomeri localizzati all'interno dell'alone di DNA in cellule HDF proliferanti, senescenti e quiescenti. Le barre di errore rappresentano ± SEM. Fare clic qui per visualizzare una versione ingrandita di questa figura.
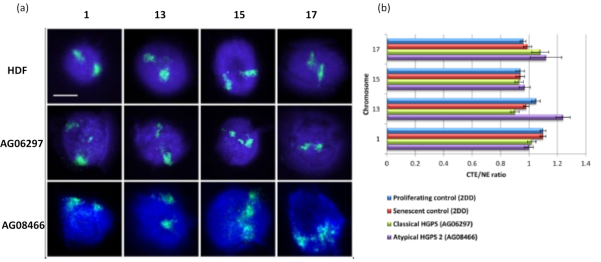
Figura 6: Esame dell'ancoraggio dell'intero cromosoma al nucleoscheletro nelle cellule HGPS utilizzando HALO-FISH26. (a) I nuclei di controllo HDF (2DD), HGPS classico (AG06297) e HGPS atipico di tipo 2 (AG08466) sono stati sottoposti a preparazione di alone di DNA e quindi 2D-FISH utilizzando vernici cromosomiche intere per i cromosomi 1, 13, 15 e 17. I cromosomi interi sono raffigurati in verde (FITC) e il DNA è stato controcolorato con DAPI (blu). Ingrandimento = x 100; Barra di scala 10 μm. (b) Il posizionamento dei cromosomi all'interno dei nuclei estratti è stato determinato misurando il rapporto tra il margine medio del territorio cromosomico (CTE) e il bordo nucleare (NE). Un rapporto superiore a 1 dimostra che il CTE più lontano si trova al di fuori del corrispondente NE all'interno dell'alone di DNA, mentre un rapporto inferiore a 1 significa che il CTE più lontano si trova all'interno del NE all'interno del nucleo residuo. Fare clic qui per visualizzare una versione ingrandita di questa figura.
| Costituenti | Volume (μL) |
| 5XDOP-PCRbuffer | 10 |
| dNTPmix (senzaTTP) (2mM) | 5 |
| dTTP (2mM) | 2 |
| Biotina-16-dUTPorDigoxigenina-11-dUTP | 10 |
| DOPprimer(20μM) | 5 |
| TaqDNAPolymerase (1U/μL) | 1 |
| PCRgradewater | 12 |
| Sagoma | 5 |
Tabella 1: Tabella che mostra i componenti e i volumi della DOP-PCR per una reazione 1x
| Passo | Cicli | Temp (grado centigrado) | Ore |
| Denaturazione iniziale | 1 | 95 | 3 minuti |
| Denaturazione | 34 | 98 | 20 secondi |
| Ricottura del primer | 62 | 1 min | |
| Estensione | 72 | 30 secondi | |
| Estensione finale | 1 | 72 | 5 minuti |
| Raffreddamento | 4 | Tenere |
Tabella 2: Tabella che mostra il ciclo DOP-PCR, la temperatura e il profilo temporale.
| Costituente | Volume (μL) |
| Tampone NT 10x (0,5M Tris-HCl pH 8,50 mM MgCl2, 0,5 mg/ml BSA) | 5 |
| 0,1 M beta-mercaptoetanolo | 5 |
| 10X Stock nucleotidico (0,5 mM dATP, 0,5 mM dCTP, 0,5 mM dGTP, 0,5 mM dTTP, 0,5 mg/ml biotina-16-dUTP) | 5 |
| Dnase I (1 ng/ml) | 2 |
| DNApolimerasi I | 5U per μg di DNA |
| DNAtemplate (1 μg) | 1 |
| Acqua trattata con DEPC | Fino a 50 μL |
Tabella 3: Tabella che mostra i componenti e i volumi di traduzione nick per una sonda.
Discussione
Il metodo dell'alone di DNA è un metodo eccellente di scelta quando si analizzano le interazioni tra il nucleoscheletro e il genoma, tuttavia, ci sono anche alcuni passaggi critici che devono essere rispettati. Uno dei parametri più importanti è l'ottimizzazione della densità di semina cellulare. Se le cellule diventano troppo confluenti, gli aloni di DNA si sovrapporranno alle cellule vicine rendendo impossibile eseguire l'analisi. Il CSK e i tamponi di estrazione devono essere sempre fatti freschi il giorno dell'uso con spermina, spermidina e digitonina aggiunte al tampone di estrazione alla fine del processo di preparazione per mantenere la loro attività biologica. Se si esegue Halo-FISH è estremamente importante utilizzare la corretta temperatura di denaturazione degli aloni di DNA per consentire alla sonda o alla vernice di ibridarsi successivamente.
La microscopia elettronica è stata utilizzata per visualizzare la matrice nucleare, con strutture filamentose identificate20. Tuttavia, la microscopia elettronica è limitata in quanto le associazioni della matrice con la cromatina non possono essere facilmente dedotte. In effetti, il metodo dell'alone del DNA è più versatile rispetto alla microscopia elettronica poiché è possibile esaminare geni, cromosomi e stati cellulari specifici. Inoltre, l'analisi proteomica delle proteine della matrice nucleare è in fase di studio21,22. Questo metodo è utile per confrontare i componenti della matrice nucleare, in particolare quando si confrontano le cellule malate, tuttavia, non fornisce la distribuzione spaziale e gli attaccamenti evidenziati dalla tecnica standard dell'alone del DNA.
I test dell'alone del DNA hanno dei limiti. In primo luogo, poiché la matrice viene estratta, questo può essere eseguito solo su cellule fisse, quindi l'imaging dal vivo non è possibile. Sebbene il metodo DNA Halo sia relativamente veloce e facile da eseguire, il processo complessivo può richiedere molto tempo quando si tiene conto della coltura cellulare, della generazione di sonde, dell'Halo-FISH e dell'analisi.
L'acquisizione di immagini di aloni di DNA e HALO-FISH utilizzando la microscopia a super-risoluzione migliorerebbe notevolmente la risoluzione delle sonde e degli anticorpi specifici del DNA. Inoltre, poiché i fluorocromi possono essere risolti spettralmente più facilmente, potrebbe essere possibile utilizzare un certo numero di sonde di DNA in un singolo esperimento, fornendo ancora più informazioni. Miglioramenti nelle tecniche di biologia molecolare come la cattura della conformazione cromosomica (3C) sono stati utilizzati per determinare le interazioni dei loci genici e analizzare l'organizzazione spaziale della cromatina nella cellula. I saggi di DNA Halo e 3C possono essere combinati, un termine noto come M3C23, dimostrando ancora una volta l'adattabilità della tecnica DNA Halo.
I dati originali presentati qui servono a dimostrare le possibilità di interrogare il comportamento del genoma e come presentare tali dati. Con questi dati abbiamo dimostrato che è possibile determinare differenze significative nell'attaccamento del genoma utilizzando (1) sonde di pittura cromosomica, in questo studio rivelando che il cromosoma 18 è il cromosoma meno attaccato tra quelli analizzati (Figura 3); (2) Loci genici con differenze significative tra due loci genici e (Figura 4) (3) Telomeri, che sono meno fortemente attaccati nelle cellule quiescenti rispetto alle cellule proliferanti e senescenti (Figura 5). Siamo in grado di distinguere tra cellule proliferanti e non proliferanti attraverso la presenza dell'antigene marcatore di proliferazione Ki67 che è una proteina insolubile quindi rimane con i nuclei residui o utilizzando l'incorporazione di nucleotidi per evidenziare le cellule che hanno attraversato la fase S entro un periodo di tempo specifico (Figura 2). Questa tecnica ci ha anche permesso di analizzare il comportamento del genoma in cellule che sono compromesse nei loro nucleoscheletri, cioè cellule di lamininopatia e qui e in Bikkul et al., 2018 riveliamo che il genoma può essere meno strettamente attaccato rispetto alle cellule di controllo e può essere ripristinato quando trattato con farmaci specifici che migliorano l'effetto della mutazione della lamina A nelle cellule HGPS classiche15. Tuttavia, mostriamo qui nuovi dati per le cellule HGPS atipiche AGO8466, prive di una mutazione della lamina A ma contenenti una forma insolita della proteina del complesso LINC SUN119 in cui il cromosoma 13 è meno strettamente attaccato (Figura 6).
HALO-FISH è un metodo unico che consente lo studio delle interazioni genomiche con il nucleoscheletro in combinazione con l'immunofluorescenza indiretta per risolvere le proteine non rimosse dalla procedura di estrazione. È stato dimostrato che il nucleoscheletro è modificato in varie malattie come alcuni tipi di cancro19 e l'importanza di alcune proteine associate al nucleoscheletro come biomarcatori diagnostici24,25. Pertanto, questa tecnica ha un ruolo importante nell'esaminare l'effetto del nucleoscheletro sull'organizzazione/disorganizzazione della cromatina nella malattia 15,24,25,27 e non è limitata alle cellule umane, con sonde di pittura cromosomica di altri animali, lo stesso protocollo DNA-alone potrebbe essere impiegato 28.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Vorremmo ringraziare il Prof. Michael Bittner per il gentile dono delle sonde per la pittura del braccio cromosomico. LG è stato sostenuto dal progetto EURO-Laminopathies, finanziato dall'UE, e dal Brunel Progeria Research Fund.
Materiali
| Name | Company | Catalog Number | Comments |
| 10X PBS | Thermo Fisher Scientific | 10388739 | Used to create DNA halos |
| 5-bromo-2′-deoxy-uridine | Sigma-Aldrich | B5002-100MG | Labelled nucleotide |
| 5-Fluoro-2′-deoxyuridine | Sigma-Aldrich | F0503-100MG | Labelled nucleotide |
| Agar Technical | Thermo Fisher Scientific | 15562141 | DNA isolation of BAC clones |
| Agarose | Sigma-Aldrich | A939-50G | Check product size of DOP-PCR and nick translation |
| Atypical type 2 HGPS fibroblasts (AG08466) | Coriell Institute | AG08466 | Cell line |
| Bacto tryptone | Thermo Fisher Scientific | 16269751 | DNA isolation of BAC clones |
| Biotin-16-dUTP | Roche Diagnostics | 11093711103 | Labelled nucleotides |
| Chloramphenicol | Sigma-Aldrich | C0378-25G | DNA isolation of BAC clones |
| Classical Hutchinson-Gilford progeria syndrome (HGPS) fibroblasts (AG06297) | Coriell Institute | AG0297 | Cell line |
| Coplin jar | Thermo Fisher Scientific | 12608596 | Holds 5 slides or 8 slides back to back |
| Cot-1 DNA | Thermo Fisher Scientific | 15279011 | Block nonspecific hybridization in HALO FISH |
| DEPC-treated water | Sigma-Aldrich | 693520-1L | DNA isolation of BAC clones |
| Dextran sulphate | Sigma-Aldrich | S4030 | Hybridisation mixture |
| Digitonin | Sigma-Aldrich | D141 | Component of extraction buffer |
| Digoxigenin-11-dUTP | Sigma-Aldrich | 11093088910 | Labelled nucleotides |
| Donkey anti-mouse Cy3 | Jackson Laboratory | 715-165-150 | Secondary antibody |
| EDTA | Sigma-Aldrich | E6758 | Component of extraction buffer |
| Ethanol | Component of extraction buffer | ||
| Ethanol | Sigma-Aldrich | 443611 | Probe precipitation and HALO FISH |
| Fetal bovine system | Thermo Fisher Scientific | 26140079 | Cell culture serum |
| Formamide | Thermo Fisher Scientific | 10523525 | 2D FISH of DNA halos |
| Glass wool | Sigma-Aldrich | 18421 | Spin column |
| Herring sperm | Sigma-Aldrich | D7290 | Probe precipitation |
| HXP™ Lamp (metal halide microscope lamp) | OSRAM | HXP-R120W45C VIS | Image capture of DNA halos |
| Hydrochloric acid | Thermo Fisher Scientific | 10313680 | Cleaning microscope slides |
| Isopropanol | Sigma-Aldrich | I9516-25ML | DNA isolation of BAC clones |
| KAPA HiFi PCR Kit | KAPA Biosystems | KK2103 | PCR Kit |
| Leica DM4000 fluorescent microscope with DFC365 FX camera and LAS AF (Version: 4.5.0) image acquisition software. | Leica Microsystems | Image capture of DNA halos | |
| Luria-Bertani agar | Thermo Fisher Scientific | 13274843 | DNA isolation of BAC clones |
| Magnesium chloride | Sigma-Aldrich | M8266 | Component of CSK buffer |
| Methanol | Thermo Fisher Scientific | 10284580 | Cleaning and sterilizing microscope slides |
| Mouse anti-BrdU antibody | BD Pharmingen | B2531-100UL | BrdU visualisation |
| Newborn calf serum | Thermo Fisher Scientific | 16010159 | Cell culture serum and blocking reagent |
| Nick translation kit | Invitrogen | ||
| PCR grade water | Sigma-Aldrich | 693520-1L | PCR and DNA isolation of BAC clones |
| PCR Primers | Sigma-Aldrich | ||
| PIPES | Sigma-Aldrich | P1851 | Component of CSK and extraction buffers |
| Potassium acetate | Sigma-Aldrich | P1190-100G | DNA isolation of BAC clones |
| QuadriPERM® 4 X 12 | SARSTEDT | 94.6077.307 | Square cell culture dish, polysterene with four compartments. This has hydrophobic surface, is sterile, non-pyrogenic/endotoxin-fee and non-cytotoxic. |
| Rabbit Anti-Ki67 antibody | Sigma-Aldrich | ZRB1007-25UL | Proliferation marker |
| Rnase A | Sigma-Aldrich | R6513 | DNA isolation of BAC clones |
| Rubber cement | Halford's | 101836 | 2D FISH of DNA halos |
| Sephadex G-50 | Sigma-Aldrich | S6022-25G | Spin column |
| Sodium acetate | Sigma-Aldrich | S2889 | Probe precipitation |
| Sodium chloride | Sigma-Aldrich | S5886 | Component of CSK, extraction and SSC buffers |
| Sodium citrate | Sigma-Aldrich | C8532 | Component of SSC buffer |
| Sodium dodecyl sulphate | L3771-100G | DNA isolation of BAC clones | |
| Sodium hydroxide | Sigma-Aldrich | S8045-500G | DNA isolation of BAC clones |
| Spermidine | Sigma-Aldrich | S2626 | Component of extraction buffer |
| Spermine | Sigma-Aldrich | S4264 | Component of extraction buffer |
| Streptavidin-Cy3 | Amersham Life Sciences Ltd, Scientific Laboratory Supplies | pa43001 | Probe antibody |
| Sucrose | Sigma-Aldrich | S0389 | Component of CSK buffer |
| Sucrose | Sigma-Aldrich | S0389 | CSK buffer+A66:D68 |
| SuperFrost™ microscope slides | Thermo Fisher Scientific | 12372098 | Microscope slides: 1 mm thickness, 76 mm length, 26 mm width. Uncoated. |
| Swine anti-rabbit TRITC | Dako | ||
| TELO-PNA FISH KIT | Agilent Dako | K532511-8 | Delineation of telomeres |
| Tris-HCl | Sigma-Aldrich | T3253-100G | Column buffer |
| Triton™ X-100 | Sigma-Aldrich | T9284 | Component of CSK buffer |
| Tryptone | Thermo Fisher Scientific | 10158962 | DNA isolation of BAC clones |
| Tween-20 | Sigma-Aldrich | P9416- 100ML | Detergent |
| Vectashield mountant containing DAPI | Vector Laboratories | H-1200 | 2D FISH of DNA halos |
| Whole human chromosome probes | Calbiochem | 2D FISH of DNA halos | |
| Yeast extract | Thermo Fisher Scientific | 10108202 | DNA isolation of BAC clones |
Riferimenti
- Berezney, R., Coffey, D. S. Identification of a nuclear protein matrix. Biochemical Biophysical Research Communications. 60 (4), 1410-1417 (1974).
- Haaf, T., Ward, D. C. High resolution ordering of YAC contigs using extended chromatin and chromosomes. Human Molecular Genetics. 3 (4), 629-633 (1994).
- Parra, I., Windle, B. High resolution visual mapping of stretched DNA by fluorescent hybridization. Nature Genetics. 5 (1), 17-21 (1993).
- Senger, G., et al. Released chromatin: linearized DNA for high resolution fluorescence in situ hybridization. Human Molecular Genetics. 3 (8), 1275-1280 (1994).
- Florijn, R. J., et al. High-resolution DNA Fiber-FISH for genomic DNA mapping and colour bar-coding of large genes. Human Molecular Genetics. 4 (5), 831-836 (1995).
- Elcock, L. S., Bridger, J. M. Fluorescence in situ hybridization on DNA halo preparations and extended chromatin fibres. Methods Molecular Biology. 659, 21-31 (2010).
- Heiskanen, M., et al. Visual mapping by fiber-FISH. Genomics. 30 (1), 31-36 (1995).
- Bensimon, A., et al. Alignment and sensitive detection of DNA by a moving interface. Science. 265 (5181), 2096-2098 (1994).
- Michalet, X., et al. Dynamic molecular combing: stretching the whole human genome for high-resolution studies. Science. 277 (5331), 1518-1523 (1997).
- Wilson, R. H., Coverley, D. Relationship between DNA replication and the nuclear matrix. Genes Cells. 18 (1), 17-31 (2013).
- Wilson, R. H. C., Coverley, D. Transformation-induced changes in the DNA-nuclear matrix interface, revealed by high-throughput analysis of DNA halos. Science Reports. 7 (1), 6475 (2017).
- Iarovaia, O. V., Akopov, S. B., Nikolaev, L. G., Sverdlov, E. D., Razin, S. V. Induction of transcription within chromosomal DNA loops flanked by MAR elements causes an association of loop DNA with the nuclear matrix. Nucleic Acids Research. 33 (13), 4157-4163 (2005).
- Tandara, M., et al. Sperm DNA integrity testing: big halo is a good predictor of embryo quality and pregnancy after conventional IVF. Andrology. 2 (5), 678-686 (2014).
- Repping, S., et al. The use of spermHALO-FISH to determine DAZ gene copy number. Mol Human Reproduction. 9 (4), 183-188 (2003).
- Bikkul, M. U., et al. Farnesyltransferase inhibitor and rapamycin correct aberrant genome organisation and decrease DNA damage respectively, in Hutchinson-Gilford progeria syndrome fibroblasts. Biogerontology. 19 (6), 579-602 (2018).
- Telenius, H., et al. Degenerate oligonucleotide-primed PCR: general amplification of target DNA by a single degenerate primer. Genomics. 13 (3), 718-725 (1992).
- Bridger, J. M., et al. Association of pKi-67 with satellite DNA of the human genome in early G1 cells. Chromosome Research. 6, 13-24 (1998).
- Sales Gil, R., Vagnarelli, P. Ki-67: More Hidden behind a 'Classic Proliferation Marker'. Trends in Biochemical Sciences. 43 (10), 747-748 (2018).
- Bikkul, M. U., et al. Telomere elongation through hTERT immortalization leads to chromosome repositioning in control cells and genomic instability in Hutchinson-Gilford progeria syndrome fibroblasts, expressing a novel SUN1 isoform. Genes Chromosomes Cancer. 58 (6), 341-356 (2019).
- Jackson, D. A., Cook, P. R. Visualization of a filamentous nucleoskeleton with a 23 nm axial repeat. EMBO Journal. 7, 3667-3677 (1988).
- Albrethsen, J., et al. Unravelling the nuclear matrix proteome. Journal of Proteomics. 72, 71-81 (2009).
- Mika, S., Rost, B. NMPdb: Database of nuclear matrix proteins. Nucleic Acids Research. 33, 160-163 (2005).
- Gavrilov, A. A., et al. of the nuclear matrix-bound chromatin hubs by a new M3C experimental procedure. Nucleic Acids Research. 38, 8051-8060 (2010).
- Sjakste, N., et al. Role of the nuclear matrix proteins in malignant transformation and cancer diagnosis. Experimental Oncology. 26 (3), 170-178 (2004).
- Leman, E. S., Getzenberg, R. H. Nuclear structure as a source of cancer specific biomarkers. Journal of Cellular Biochemistry. 104 (6), 1988-1993 (2008).
- Volpi, E. V., Bridger, J. M. FISH glossary: an overview of the fluorescence in situ hybridization technique. Biotechniques. 45 (4), 385-386 (2008).
- Bridger, J. M., Foster, H. A. Senescence and the Genome. Human Interphase Chromosomes. , (2021).
- Foster, H. A., Griffin, D. K., Bridger, J. M. Interphase chromosome positioning in in vitro porcine cells and ex vivo porcine tissues. BMC Cell Biology. 13 (1), 30 (2012).
Erratum
Formal Correction: Erratum: Fluorescence In Situ Hybridization on DNA Halo Preparations to Reveal Whole Chromosomes, Telomeres and Gene Loci
Posted by JoVE Editors on 6/05/2023. Citeable Link.
An erratum was issued for: Fluorescence In Situ Hybridization on DNA Halo Preparations to Reveal Whole Chromosomes, Telomeres and Gene Loci. The Authors section was updated from:
Lauren S. Godwin1
Joanna M. Bridger1
Helen A. Foster2
1Laboratory of Nuclear and Genomic Health, Centre for Genome Engineering and Maintenance, Division of Biosciences, Department of Life Sciences, College of Health, Medicine and Life Sciences, Brunel University London
2Biosciences, Department of Clinical, Pharmaceutical and Biological Science, School of Life and Medical Sciences, University of Hertfordshire
to:
Lauren S. Godwin1
Emily Roberts2
Joanna M. Bridger1
Helen A. Foster2
1Laboratory of Nuclear and Genomic Health, Centre for Genome Engineering and Maintenance, Division of Biosciences, Department of Life Sciences, College of Health, Medicine and Life Sciences, Brunel University London
2Biosciences, Department of Clinical, Pharmaceutical and Biological Science, School of Life and Medical Sciences, University of Hertfordshire
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon