Method Article
전체 염색체, 텔로미어 및 유전자좌를 밝히기 위한 DNA Halo Preparations의 형광 In Situ 혼성화
Erratum Notice
요약
DNA 후광 제제와 형광 in situ 하이브리드화를 결합하면 뉴클레오스켈레톤과의 게놈 상호작용에 대한 고분해능 분석이 가능합니다. 부착된 게놈은 추출된 잔류 핵 내에 위치한 혼성화된 형광 신호를 유도하는 반면, 부착되지 않은 게놈은 잔류 핵을 둘러싸고 있는 DNA의 후광에 있습니다.
초록
게놈은 활동을 조절하고 특정 위치에 고정하기 위해 세포핵 내부의 여러 구조와 관련이 있습니다. 이러한 구조는 총칭하여 핵골격으로 알려져 있으며 핵층, 핵소체 및 핵체를 포함합니다. 게놈과 그 조직을 연구하기 위해 형광 FISH(Fluorescence in situ hybridization)의 많은 변이체가 존재하지만, 이들은 종종 분해능에 의해 제한되고 게놈과 핵 구조의 연관성에 대한 정보가 충분하지 않습니다. DNA 헤일로 방법은 높은 염분 농도와 비이온성 세제를 사용하여 게놈 내의 부착 영역을 통해 핵 내 구조에 고정된 상태로 유지되는 DNA 루프를 생성합니다. 여기에서 히스톤, 지질 및 핵 매트릭스에 단단히 결합되지 않은 DNA와 같은 가용성 핵 단백질이 추출됩니다. 이것은 내부 핵 구조 및 추출 저항성 단백질과 밀접하게 관련된 DNA를 포함하는 잔류 핵을 둘러싼 부착되지 않은 DNA의 후광을 형성합니다. 이러한 확장된 DNA 가닥은 분해능을 높이고 물리적 매핑을 용이하게 할 수 있습니다. FISH와 함께 이 방법은 게놈이 고정되어 있는 모든 구조와의 게놈 상호 작용을 연구하는 추가 이점이 있습니다. HALO-FISH라고 하는 이 기술은 DNA 후광을 핵산 프로브와 결합하여 유전자좌, 전체 염색체, 알파 위성, 텔로미어 및 RNA까지 밝혀낼 수 있는 매우 다재다능합니다. 이 기술은 정상 세포와 암과 같은 질병 진행에서 핵 조직과 기능에 대한 통찰력을 제공합니다.
서문
"핵 매트릭스"는 19741에서 Berezney와 Coffey에 의해 처음 기술되었습니다. 쥐의 간 핵에 대해 높은 염분 몰 농도와 뉴클레아제 처리로 추출을 수행한 후, 그들은 단백질성 구조 프레임워크를 확인했습니다. DNA 후광 절차는 이후에 이 방법에서 채택되었으며 가용성 단백질을 제거하여 핵 매트릭스(NM) 및 NM 관련 단백질 및 염색체만 지속되도록 합니다. DNA 부착 영역은 DNA 루프의 기저부에 위치하며 매트릭스 부착 영역(MAR) 또는 스캐폴드 부착 영역(SAR)이라고 하며, 각각 고염 농도 및 이온 세제 리튬-3,5-디요오도살리실레이트(LIS)를 사용한 추출에 내성이 있습니다. DNA 후광에서 MAR/SAR과 관련된 DNA는 잔류 핵 내에 결합되어 있는 반면 DNA 루프는 이러한 부위에서 멀리 확장되어 DNA 후광을 형성합니다. 우리는 이제 게놈이 라미나 관련 도메인(LAD)을 통해 핵층과 핵층 관련 영역(NAD)을 통해 그리고 아마도 특정 핵체와 같은 다른 핵 구조를 통해 고정된다는 것을 알고 있습니다.
DNA 후광 방법은 염색질에서 히스톤이 제거되고 DNA가 2,3,4,5,6으로 늘어나기 때문에 확장된 DNA와 염색질이 더 큰 분해능을 제공하기 때문에 DNA, 유전자 및 염색체 영역의 물리적 매핑에 사용할 수 있습니다. 그러나 이 응용 프로그램에 DNA 후광을 사용할 때 몇 가지 제한 사항이 있습니다. 예를 들어, DNA 후광의 잔류 핵과 밀접하게 관련된 DNA는 프로브에 접근할 수 없어 분석 및 물리적 매핑에서 제외될 수 있다6. fiber-FISH 2,4,5,7 및 분자 빗질(molecular combining)8과 같은 다른 기술들 또한 물리적 맵핑을 가능하게 하고, 비교적 빠르고 쉽게 수행할 수 있다는 장점을 갖는다. 둘 다 DNA 후광에 대한 유전자의 DNA 매핑에 우선적으로 사용됩니다. 이러한 방법은 핵에서 용매 또는 염 추출을 사용하여 염색질 섬유를 추출하지만 분자 빗질은 더 나은 재현성을 갖는 경향이 있습니다 8,9.
뉴클레오스켈레톤이 DNA의 부착 부위, 염색질 리모델링, DNA 전사, DNA 복구 및 DNA 복제와 같은 주요 핵 과정을 지원하는 역할을 한다는 증거가 증가하고 있습니다11,12. 따라서 DNA 후광 기술은 이러한 세포 활동 동안 뉴클레오스켈레톤과 게놈 사이의 상호 작용을 조사하기 위해 개발되었으며 연구에서 일상적으로 사용 및 보고되었습니다. 이 기술은 또한 질병 진행과 관련하여 게놈과 뉴클레오스켈레톤 사이의 상호 작용을 조사하는 데 사용되었으며 핵 구조의 악성 종양 관련 변화가 확인되었습니다11.
DNA 후광 기술은 또한 발달과 분화 과정에서 게놈과 뉴클레오스켈레톤 사이의 관계를 조사하는 데 사용되었다12. 많은 연구에서 FISH14와 결합된 경우 halosperm13 또는 SpermHalo-FISH로 알려진 DNA 후광 기술의 변형을 사용했습니다. 정자 염색질은 프로타민으로 알려진 단백질에 밀접하게 결합되어 있으며 이 기술은 정자 DNA에 대한 접근성을 향상시키기 위해 개발되었습니다. 연씨식물은 정자 DNA의 무결성을 조사하고 DNA 손상이 있는지 확인하는 데 사용되었습니다. DNA 손상이 적은 정자는 더 큰 DNA 후광 크기와 관련이 있는 반면, 파편화되고 손상된 DNA 수준이 증가한 정자는 후광이 작거나 전혀 없었습니다. 따라서 염씨식물은 IVF를 통한 배아의 질과 성공적인 임신의 잠재적인 예후 마커로 사용될 수 있습니다13. 이 예는 이 기술의 잠재적인 임상 적용을 강조합니다. 우리의 연구에서 우리는 HALO-FISH를 사용하여 게놈 행동의 변화와 조기 노화 질환 인 허친슨-길 포드 (Hutchinson-Gilford) Progeria 증후군 (HGPS)에서 특정 약물 치료의 효과를 평가했습니다 15.
이러한 연구와 다른 연구를 함께 사용하면 DNA 후광 기술을 연구하는 데 사용할 수 있는 프로세스/응용 프로그램의 폭과 기술의 유용성을 강조합니다.
프로토콜
1. 슬라이드 준비, 멸균 및 세포 배양
- 500mL의 10% HCl(v/v)을 준비하고 큰 비커에 붓습니다.
- 현미경 슬라이드를 산에 개별적으로 떨어뜨리고 2 x g로 설정된 셰이커에서 실온에서 1시간 동안 배양합니다.
주의 : HCl은 부식성이며 자극성입니다. 심한 피부 화상과 눈 손상 및 피부, 눈 및 호흡기 자극을 유발할 수 있습니다. 니트릴 장갑, 보안경 및 실험실 코트를 포함하여 적절한 개인 보호구를 착용하십시오. - 비커에서 산을 내리고 슬라이드를 수돗물에서 10 번 세척 한 다음 탈 이온수에서 10 번 더 세척합니다.
- 슬라이드를 메탄올로 두 번 헹구고 화염에 의해 살균 될 때까지 메탄올에 보관하십시오.
주의: 메탄올은 인화성이 높은 액체이며 삼키거나 피부에 접촉하거나 흡입하면 유독합니다. 또한, 메탄올은 장기에 손상을 줄 수 있고, 부식성 및 자극성이다. 작업장 노출 제한을 준수하고 부틸 고무 장갑, 보안경 및 실험실 코트를 포함한 적절한 개인 보호 장비를 착용하십시오. 가능하면 국소 배기 환기(LEV) 흄 찬장에서 다루십시오. - 금속 집게 또는 긴 집게를 사용하여 메탄올이 들어 있는 비커에서 현미경 슬라이드를 제거합니다. 분젠 버너 위에 불을 붙여 살균하고 분젠 버너 근처에 위치한 슬라이드 용 구획 4 개가 들어있는 직사각형 세포 배양 용기로 옮깁니다.
주의 : Flaming은 사용하기 전에 현미경 슬라이드를 즉시 멸균할 수 있습니다. 그러나 이 방법에는 관련 위험이 있습니다. 메탄올은 가연성이 높기 때문에 슬라이드가 들어 있는 비커를 분젠 버너에서 멀리 떨어뜨리는 것이 중요합니다. 슬라이드를 단단히 잡는 긴 집게나 집게를 사용해야 합니다. 비커의 메탄올 수준은 사용되는 메탄올의 양을 최소화하고 집게/집게의 끝만 메탄올과 접촉하도록 슬라이드를 덮어야 합니다. 사용 후 집게나 집게에서 메탄올이 증발하고 슬라이드와 메탄올이 들어 있는 비커에 다시 넣기 전에 식었는지 항상 확인하십시오. 비커는 메탄올에 불이 붙을 경우 산소를 굶주리게 하기 위해 알루미늄 호일로 덮어야 합니다. 공기가 순환되는 Class II 층류 후드 내에서 화염이 미끄러지지 않도록 하십시오. - 또는 1.1-1.4단계를 수행하되 메탄올로 배양한 후 슬라이드를 태우지 말고 보푸라기가 없는 조직에 슬라이드를 놓아 자연 건조시킵니다. 건조되면 알루미늄 호일로 싸서 살균기 오븐이나 오토클레이브에 넣습니다.
- 60-70% 컨플루언스에 도달할 때까지 5%CO2 중에서 37°C에서 적어도 48시간 동안 혈청과 함께 적절한 배지에서 세포를 성장시킨다. 이 프로토콜은 인간 진피 섬유 아세포 (HDF)와 고전적인 허친슨-길 포드 프로 게 리아 증후군 (HGPS) 섬유 아세포 (AG06297) 및 비정형 유형 2 HGPS 섬유 아세포 (AG08466)의 초기 계대에서 수행되었습니다. 각 세포 유형을 수확하고 혈구계를 사용하여 계수하여 세포 밀도를 결정합니다. 슬라이드당 10mL 배지에 1 x 105 개의 세포를 시드합니다.
참고: 세포가 너무 합류하면 다른 핵의 DNA 루프가 수렴될 수 있으므로 세포 밀도가 중요합니다. 형질전환된 세포가 더 빨리 증식할 수 있으므로 사용되는 세포 유형에 따라 파종 밀도를 최적화해야 할 수 있으며, 이후 계대 세포 배양은 원하는 합류도에 도달하는 데 더 오랜 시간이 걸릴 수 있습니다. - 세포가 정지 상태가 되기 위해 G0 에서 정지되어야 하는 경우 슬라이드당 1 x 105 세포(10mL 배지 중)를 시딩하고 24시간 동안 성장하도록 둡니다. 무혈청 배지로 세포를 2회 세척하고 0.5%의 낮은 농도의 혈청(신생아 송아지 혈청, NCS 또는 소 태아 혈청, FBS)이 포함된 표준 배지에서 7일 동안 배양합니다.
- DNA Halo 분석에 세포의 증식 상태가 필요한 경우 복제 중에 5-bromo-2'-deoxy-uridine(BrdU)을 DNA에 통합하여 S-Phase를 통과한 세포를 결정합니다.
- 종자 세포는 정상으로 24 시간 동안 자랍니다. 배양 배지를 제거하고 BrdU 및 5-플루오로-2'-데옥시유리딘(3μg/μL)을 함유한 배지로 교체합니다. 추가 24시간 후, 배지를 제거하고, 배지(10% NCS)로 세포를 한 번 세척한 다음, 새로운 배지(10% NCS)로 다시 공급한다. 추가로 24시간 동안 배양한 다음 DNA 후광 분석을 위한 슬라이드를 준비합니다.
2. 프로브 준비
- 염색체 전체 및 팔 페인팅 프로브
- Telenius et al.16의 방법을 사용하여 퇴화 올리고뉴클레오티드 프라이밍 중합효소 연쇄 반응(DOP-PCR)에 의해 유동 분류 또는 미세 해부된 염색체의 증폭에서 염색체 프로브를 만듭니다. DOP-PCR을 사용하여 표 1과 같이 염색체 프로브에 Biotin-16-dUTP 또는 Digoxigenin-11-dUTP를 표시합니다. 증폭 프로파일에 대한 제조업체의 지침을 확인하시기 바랍니다, 그러나, 이 실험에 사용된 조건은 표 2에 나타내었다.
- 표지된 PCR 산물 8μL, Cot-1 DNA 7μL, 청어 정자 3μL, 3M 아세트산나트륨(pH 5.4) 1/20 부피 및 100% 에탄올2부피를 함께 추가하여 팔 또는 전체 염색체 프로브를 준비합니다. 프로브 용액을 -80°C에서 최소 30분 동안 배양합니다.
참고: 이 방법은 단일 염색체 프로브를 만드는 데 사용할 수 있으며, 관심 있는 각 염색체에 대해 서로 다른 레이블(예: 비오틴-16-UTP 및 디곡시제닌-11-dUTP)을 사용하는 경우 다중 염색체 프로브를 만들 수 있습니다.
주의 : 에탄올은 인화성이 높은 액체 및 증기이며 심각한 눈 자극을 유발할 수 있습니다. 열, 뜨거운 표면 및 발화원으로부터 멀리하십시오. 작업장 노출 제한을 준수하고 부틸 고무 장갑, 보안경 및 실험실 코트를 포함한 적절한 개인 보호 장비를 착용하십시오. 가능하면 LEV 흄 찬장에서 다루십시오. - 프로브 용액을 13,700 x g 에서 4°C에서 15분 동안 원심분리한 다음 70% 에탄올로 세척한다. 원심분리 절차를 반복하고 DNA 펠릿을 방해하거나 잃지 않도록 주의하면서 상층액을 버립니다. DNA 펠릿을 건조시키십시오.
- 12μL의 하이브리드화 완충액(50% 포름아미드, 10% 덱스트란 설페이트, 10% 20x 식염수 시트르산나트륨(SSC; 3M NaCl, 0.3M 트리소듐시트레이트, pH 7.0), 1%(v/v) 폴리옥시에틸렌 소르비날 모노라우레이트(Tween-20))를 DNA 펠릿에 추가합니다. DNA 펠릿이 혼성화 완충액 내로 용해될 때까지 적어도 2시간 동안 37°C에서 방치한다.
주의: 포름아미드는 발암성 및 기형유발성이므로 태아에게 심각한 해를 끼칠 수 있습니다. 여성이 임신 중이거나 임신이 의심되는 경우 포름 아미드 사용을 피해야합니다. 포름 아미드는 LEV 흄 후드에 사용해야합니다. 작업장 노출 제한을 준수하고 부틸 고무 장갑, 보안경 및 실험실 코트를 포함한 적절한 개인 보호 장비를 착용하십시오.
- 박테리아 인공 염색체(BAC)에서 DNA 분리
- BAC 클론의 글리세롤 스톡의 작은 부분을 Luria-Bertani(LB) 한천 플레이트(1%(W/V) NaCl, 1%(w/v) 트립톤, 0.5%(w/v) 효모 추출물, 1.5%(w/v) 한천 기술, 12.5μg/mL(w/v) 클로람페니콜). 37°C에서 하룻밤 동안 배양한다.
- 플레이트에서 단일 콜로니를 선택하고 LB 브로스 10mL(1%(w/v) NaCl, 1%(w/v) 박토트립톤, 0.5%(w/v) 효모 추출물, 12.5μg/mL(w/v) 클로람페니콜)를 접종합니다. 용액을 37°C에서 밤새 진탕 인큐베이터에 방치한다.
주의: 클로람페니콜은 암을 유발하는 것으로 의심됩니다. 조심해서 다루고 노출을 줄이십시오. - 실온에서 1,700 x g 에서 10분 동안 원심분리하여 배양합니다.
- 상청액을 버리고 300 μL의 P1 용액 (15 mM Tris (pH 8), 10 mM EDTA, 100 μg / mL RNase A)을 펠렛에 첨가한다. 격렬하게 소용돌이치고 세포를 2mL 미세원심분리기 튜브로 옮깁니다.
- 300 μL의 P2 용액(0.2 M NaOH, 1%(w/v) 소듐 도데실 설페이트(SDS))을 세포에 적가합니다. 닫힌 미세원심분리기 튜브를 5회 뒤집고 실온에서 최대 5분 동안 그대로 둡니다.
주의 : 수산화나트륨은 부식성이며 심한 피부 화상과 눈 손상을 유발할 수 있습니다. 금속을 부식시킬 수 있습니다. 조심해서 다루고 노출을 줄이십시오. 작업장 노출 제한을 준수하고 니트릴 고무 장갑, 보안경 및 실험실 코트를 포함한 적절한 개인 보호 장비를 착용하십시오. 가능하면 LEV 흄 찬장에서 다루십시오.
주의 : 도데실 황산나트륨은 가연성 고체로 삼키면 유해하며 피부와 호흡기 자극을 유발할 수 있습니다. 또한 심각한 눈 손상을 일으킬 수 있습니다. 니트릴 고무 장갑, 보안경 및 실험실 코트를 포함하여 적절한 개인 보호구를 착용하십시오. 가능하면 LEV 흄 찬장에서 다루십시오. - 300 μL의 P3 (3 M potassium acetate)를 세포에 천천히 넣고 부드럽게 혼합합니다. 미세 원심분리기 튜브를 얼음 위에 10분 동안 놓습니다.
- 4°C에서 10분 동안 8,100 x g 에서 원심분리하고 상층액을 800μL의 빙냉 이소프로판올이 들어 있는 튜브로 옮깁니다. 튜브를 여러 번 뒤집고 -20°C에서 밤새 배양합니다.
주의: 이소프로판올은 가연성이 높은 액체 및 증기이며 심각한 눈 자극, 졸음 또는 현기증을 유발할 수 있습니다. 열, 뜨거운 표면 및 발화원으로부터 멀리하십시오. 작업장 노출 제한을 준수하고 니트릴 고무 장갑, 보안경 및 실험실 코트를 포함하여 적절한 개인 보호 장비를 착용하십시오. 가능하면 LEV) 흄 찬장에 보관하십시오. - 4°C에서 15분 동안 8,100 x g 로 원심분리합니다. 상청액을 제거하고 다른 튜브로 옮깁니다. 얼음처럼 차가운 70% 에탄올 500μL를 넣습니다. 튜브를 여러 번 뒤집고 4°C에서 5분 동안 8,100 x g 로 원심분리합니다.
- 상층액을 제거하고 펠릿을 실온에서 자연 건조시킵니다. 펠릿이 건조되면 40 μL의 디에틸 피로카보네이트 처리수(DEPC 처리)에 다시 현탁하고 4°C에서 밤새 방치합니다. 완전히 재현탁되면 5μL의 용액을 제거하고 1% 아가로스 겔에 로드하여 DNA의 존재 여부를 확인합니다.
- 닉 번역을 통한 BAC의 단일 유전자 프로브 준비
- 시중에서 판매되는 닉 번역 라벨링 키트를 사용하십시오. 또는 다음 프로토콜을 사용합니다. 성분과 부피에 대해서는 표 3 을 참조하십시오.
- 마지막으로 DNA Polymerase I을 첨가하는 미세 원심분리 튜브에 표 3 의 성분을 함께 넣고 부드럽게 혼합하고 몇 초 동안 짧게 원심분리합니다. 용액을 15°C에서 2시간 동안 인큐베이션합니다.
- 절편 크기를 확인하려면 5μL의 용액을 2% 아가로스 겔에 로드합니다. DNA 단편 크기 범위는 200-600bp 사이여야 합니다. DNA 단편 크기가 더 큰 경우, 15°C에서 추가로 15분 동안 용액을 계속 인큐베이션하고 생성물을 2% 아가로스 겔 상에서 실행한다.
- 10 mM EDTA, 0.1% SDS (100 μL 중 100 μL 중 0.5 M EDTA, pH 8.0 중 2.5 μL 및 100 μL 중 10% SDS 중 1 μL)를 첨가하여 닉 번역 반응을 중지시켰다. 용액을 65°C에서 5분 동안 가열합니다.
- 통합되지 않은 뉴클레오티드를 제거하려면 BAC 프로브를 스핀 컬럼에 적용합니다. 상업용 스핀 컬럼을 구입하거나 다음과 같이 주사기를 사용하여 만들 수 있습니다.
- 30g의 Sephadex G-50을 500mL의 컬럼 완충액(10mM Tris-HCl(pH8), 1mM EDTA, 0.1% SDS)에 추가합니다. 혼합물을 오토클레이브합니다. 또한 500mL의 컬럼 버퍼(Sephadex G-50 제외)와 오토클레이브를 만듭니다.
- 1mL 주사기 바닥에 오토클레이브 유리솜을 추가하여 스핀 컬럼을 만듭니다. 1mL 주사기를 컬럼 버퍼에 Sephadex G-50으로 채웁니다. 바닥에 뚜껑이 없는 미세 원심분리기 튜브가 있는 15mL 원심분리기 튜브에 1mL 주사기를 넣습니다. 1,600 x g 에서 5분 동안 원심분리기.
- 주사기를 제거하고 하단에 있는 미세 원심분리기 튜브를 버립니다. 새 미세 원심분리기 튜브를 15mL 원심분리기 튜브에 다시 추가합니다. 컬럼 버퍼(Sephadex G-50 제외)를 1mL 주사기에 넣고 15mL 원심분리기 튜브에 다시 삽입합니다. 1,600 x g 에서 5분 동안 원심분리기. 이 단계를 다시 두 번 반복합니다.
- 주사기를 제거하고 깨끗한 새 미세 원심분리기 튜브가 들어 있는 15mL 원심분리기 튜브에 삽입합니다. 주사기에 프로브를 적용하고 미세 원심 분리기 튜브에 프로브를 수집합니다.
- DNA 프로브를 침전시키기 위해 5 μL의 청어 정자 DNA(10 mg/mL), 10 μL의 아세트산 나트륨 및 2.25 부피의 100% 에탄올을 DNA 용액에 첨가합니다. 용액을 부드럽게 혼합하고 -80°C에서 최소 1시간 동안 배양합니다. 4°C에서 15분 동안 13,700 x g 로 원심분리합니다.
- 상층액을 버리고 4°C에서 15분 동안 얼음처럼 차가운 70% 에탄올 200μL로 펠릿을 세척합니다. 상층액을 제거하고 자연 건조하십시오. 일단 건조되면 펠렛을 실온에서 20μL의 DEPC 처리수에 몇 시간 동안 또는 4°C에서 밤새 재현탁합니다. 이제 프로브를 사용할 준비가 되었거나 -20°C에서 보관할 수 있습니다.
- 각 슬라이드에 대해 5 μL 프로브 DNA와 5 μL Cot-1 DNA를 혼합하고 Speed Vac 진공 농축기를 사용하여 건조시킵니다. 일단 펠렛이 건조되면 12 μL의 혼성화 혼합물에 재현탁한다.
3. DNA 헤일로 준비
- 슬라이드와 부착된 세포가 들어 있는 정사각형 배양 접시를 인큐베이터에서 꺼냅니다. 배지를 버리고 연필을 사용하여 슬라이드에 라벨을 붙이고 50mL의 얼음-차가운 세포골격(CSK) 완충액이 들어 있는 코플린 병에 넣습니다: 100mM NaCl, 3mMMgCl2, 0.3M 수크로오스, 10mM 1, 4-피페라진디에탄술폰산(PIPES; pH 7.8), 0.5%(v/v) 트리톤 X-100을 탈이온수로 구성하였다. 얼음 또는 4°C에서 15분 동안 배양합니다.
주의 : Triton X-100은 피부 자극과 심각한 눈 손상을 일으킬 수 있습니다. 니트릴 장갑, 고글 및 실험실 코트를 포함한 적절한 개인 보호 장비를 사용하여 다루십시오. - CSK 완충액을 버리고, 슬라이드를 50 mL의 1x DNA 할로 완충액(DHB; 140 mM NaCl, 27 mM KCl, 110 mM NaHPO4, 15 mM KH2PO4; pH7.4)으로 빠르게 헹구고, 즉, 슬라이드를 DHB가 들어 있는 코플린 병에 담그고 제거한다.
- 50mL의 추출 완충액이 들어 있는 코플린 병으로 슬라이드를 옮깁니다: 2M NaCl, 10mM PIPES(pH 6.8), 10mM 에틸렌디아민테트라아세트산(EDTA), 0.1%(w/v) 디지토닌, 0.05mM(v/v) 스페르민, 0.125mM(v/v) 스페르미딘. 실온에서 4분 동안 배양합니다.
주의: 디지토닌은 삼키거나 피부에 닿으면 독성이 있으며 흡입하면 치명적입니다. 디지토닌은 LEV 흄 찬장에서 취급되는지 확인하고 실험실 코트, 니트릴 장갑(이중 장갑), 보안경 및 마스크를 착용하십시오. 스페르민과 스페르미딘은 모두 심각한 피부 화상과 눈 손상을 유발할 수 있으며 EDTA는 심각한 눈 자극을 유발하므로 각 화학 물질을 주의해서 다루십시오.
참고: 분말을 60-70°C의 온도에서 물에 용해시켜 디지토닌을 별도로 준비합니다. 냉각된 후 용해된 디지토닌을 추출 버퍼에 추가합니다. 생물학적 활성을 보존하기 위해 추출 완충액에 스페르민, 스페르미딘 및 디지토닌을 마지막에 첨가합니다. - 슬라이드를 50 mL의 10x DHB (1.4 M NaCl, 270 mM KCl, 1.1 M NaHPO4, 150 mM KH2PO4; PH7.4), 5x, 2x 및 1x DHB를 각각 1분 동안 투여합니다.
- 10%, 30%, 70% 및 95%(v/v) 에탄올의 50mL 순차 에탄올 시리즈를 통해 딥 슬라이드(스트레이트 인 및 스트레이트 아웃).
- 슬라이드를 공기 건조하고, 2차원 형광 계내 하이브리드화(2D FISH)가 수행될 때까지 -80°C에서 보관한다.
4. 2차원 형광 in situ hybridization
- 20x SSC 만들기: 3M NaCl, 0.3M 구연산 삼나트륨, pH 7.0. 이 완충액은 고압 증기 멸균, 실온에서 보관 및 필요에 따라 희석 할 수 있습니다.
- 70%(v/v) 포름아미드, 2x SSC pH 7.0을 만들고 수조에서 70°C로 가열합니다.
- 슬라이드를 각각 5분 동안 70, 90 및 100% 에탄올의 순차적 50mL 에탄올 시리즈를 통해 배양합니다.
- 보온 접시에서 슬라이드를 자연 건조하고 70°C 오븐에서 5분 동안 굽습니다.
- 70% 포름아미드, 2x SSC 용액에 넣고 70°C에서 2분 동안 슬라이드를 변성시킨다.
알림: 온도와 타이밍은 4.5단계에서 매우 중요합니다. 온도가 같으면 DNA가 변성되지 않고 프로브가 혼성화되지 않으며 DNA halo FISH에서 신호를 얻지 못합니다. - 변성 슬라이드를 얼음처럼 차가운 70% 에탄올 50mL에 5분 동안 넣고 실온에서 각각 5분 동안 90%, 95% 및 100%의 에탄올 시리즈를 통과시킵니다.
- 보온 접시에서 자연 건조
- 제조업체의 지침에 따라 직접 라벨링된 전체 인간 염색체 프로브를 취급합니다. 이 실험을 위해 우리는 인간 전체 염색체 페인트 1, 13, 15, 17 및 18을 사용했습니다. 또한, 본 실험에서는 CCND1 및 CTNNA1 유전자 프로브를 사용하였다.
참고: 전체 염색체 프로브와 BAC 유전자 특이적 프로브는 모두 비오틴-11-dUTP로 표지되었고 시아닌 3(Cy3)에 접합된 스트렙타비딘에 의해 검출되었습니다. 염색체 페인팅 프로브(DOP-PCR) 및 BAC DNA 표지닉 번역에 의해, 이들은 프로토콜에서 이 시점부터 DNA 프로브로서 지칭될 것이고 다음과 같이 취급될 것이다. - DNA 프로브(전체 염색체 페인트 또는 유전자 특이적 프로브)를 75°C에서 고온 블록 또는 수조에서 10분 동안 변성시킨다.
- DNA 프로브를 37°C에서 핫 블록 또는 수조에서 30분 동안 따뜻하게 한 후 10μL를 적절한 슬라이드에 피펫팅합니다.
참고: 이 단계는 반복적인 염색체 서열을 차단하는 데 중요합니다. 비특이적 신호가 수행되지 않을 경우 HALO-FISH DNA에서 생성될 수 있다. - 21mm x 21mm 커버슬립이 있는 오버레이 프로브와 고무 시멘트를 사용한 밀봉.
- 가습된 하이브리드화 챔버에서 37°C에서 최소 18시간 동안 슬라이드를 배양합니다.
참고: 가습 하이브리드화 챔버는 여러 층의 축축한 조직이 포함된 샌드위치 상자와 슬라이드를 놓을 수 있도록 절단된 10mL 플라스틱 피펫으로 구성된 융기된 플랫폼으로 만들 수 있습니다. 이것은 빛에 대한 노출을 최소화하기 위해 알루미늄 호일로 덮여 있습니다. - 집게를 사용하여 고무 시멘트를 조심스럽게 제거하십시오.
- 슬라이드를 50mL 50%(v/v) 포름아미드, 2x SSC, pH 7.0 용액에서 45°C로 예열하여 3회 5분 동안 배양합니다.
알림: 50%(v/v) 포름아미드, 2x SSC, pH 7.0 용액의 첫 번째 배양에서 커버슬립이 슬라이드에서 떨어지도록 합니다. 이렇게 하면 커버슬립을 '끌기' 때문에 발생할 수 있는 DNA Halo 준비의 손상을 방지할 수 있습니다. 슬라이드는 커버슬립을 분리하는 데 도움이 되도록 집게로 잡아 버퍼에서 교반할 수 있습니다. - 다음에, 슬라이드를 60°C로 예열된 0.1x SSC, pH 7.0 용액 50 mL에 넣되 45°C 수조에 넣는다. 5분 동안 인큐베이션하고 버퍼를 5분 인큐베이션으로 두 번 더 교체합니다.
- 실온에서 50mL의 4x SSC, pH 7.0 용액이 들어 있는 Coplin 병에 슬라이드를 넣고 완충액을 세 번 교체하면서 15분 동안 배양합니다.
- 각 슬라이드에 4% BSA, 4x SSC 용액 100μL를 바르고 파라핀 필름으로 오버레이합니다. 실온에서 10분 동안 배양합니다. 이는 항체의 비특이적 결합을 방지한다.
- 표지된 프로브(비오틴-16-dUTP)를 검출하기 위해 실온에서 1시간 동안 1:200(1% BSA, 4x SSC로 제조) 스트렙타비딘-Cy3 100μL와 함께 배양합니다.
알림: 좋은 신호가 생성되는지 확인하기 위해 실험 전에 항체 희석 및 테스트 희석과 함께 제조업체의 지침을 따르십시오 - 실온에서 4x SSC(0.5% Tween-20) pH 7.0 용액 50mL가 들어 있는 Coplin 병에 슬라이드를 넣고 완충액을 세 번 교체하면서 15분 동안 배양합니다. 슬라이드는 면역형광이 필요하지 않은 경우 단계 4.21에 표시된 대로 이 단계에서 장착될 수 있습니다.
- DNA 후광으로 만들어진 세포의 증식 상태가 필요한 경우, FISH 단계 후, 장착 전에 항-pKi67 항체로 염색하거나 혼입된 BrdU에 대해 염색합니다.
- 슬라이드를 50mL의 1x 인산염 완충 식염수(PBS)에서 각각 5분 동안 3회 세척하고, 실온에서 1시간 동안 PBS 중 4% NCS로 블로킹하였다.
- 필요한 1차 항체(토끼 항-인간 pKi67; 마우스 항-BrdU) 200μL를 슬라이드에 적용하고 파라핀 필름 스트립으로 오버레이한 다음 실온에서 1시간 동안 배양합니다.
- 슬라이드를 1x PBS에서 5분 동안 3회 세척하고 200 μL의 형광색소 접합 2차 항체(pKi67: swine anti-rabbit TRITC; BrdU: 당나귀 안티-마우스 Cy3). PBS로 5분 더 3회 더 세척합니다. 모든 희석은 제조업체가 제안한 희석 범위에서 PBS의 1%(v/v) NCS를 사용하여 만듭니다.
- DAPI가 포함된 20μL의 마운팅제에 슬라이드를 장착하고 22mm x 50mm 커버슬립을 오버레이합니다.
5. 텔로미어 PNA 물고기
- 텔로미어를 검출하려면 텔로미어 PNA FISH 키트 - FITC를 사용하십시오. 제조업체의 지침에 따라 절차를 수행하십시오. 절차는 달리 명시되지 않는 한 실온에서 실행되어야 합니다.
- 슬라이드를 트리스 완충 식염수(TBS, pH 7.5)에 2분 동안 담근 다음 3.7% 포름알데히드(TBS, v/v)에 정확히 2분 동안 넣습니다.
주의 : TBS 용액은 10-30 % 트로 메타 몰과 10-30 % 2- 아미노 -2- (히드 록시 메틸) 프로판 -1,3- 디올 하이드로 클로라이드를 함유한다. 심각한 눈과 피부에 자극을 줄 수 있으므로 보호 장갑과 고글/안면 보호구를 착용하십시오. 환기가 잘 되는 곳에서 사용하십시오. - 코플린 병에 슬라이드를 TBS로 각각 5분 동안 두 번 세척합니다.
- 슬라이드를 전처리 용액에 10분 동안 담근 다음 세척당 5분 동안 TBS로 두 번 세척합니다.
- 다음으로, 농도당 2분 동안 70%, 85% 및 95%(v/v) 에탄올 50mL로 구성된 얼음처럼 차가운 에탄올 시리즈를 통해 슬라이드를 가져옵니다. 그런 다음 슬라이드를 자연 건조시킵니다.
- 선택한 형광 태그 색상에 따라 10μL의 Telomere PNA Probe/FITC(또는 Cy3)를 커버슬립이 있는 각 슬라이드 및 커버 오버레이에 적용합니다. 80°C로 예열된 오븐에서 5분 동안 배양한 다음 약 1시간 동안 어두운 곳에 두십시오.
주의: 텔로미어 PNA 프로브/FITC는 6-100% 포름아미드를 함유하고 있어 심각한 눈 자극을 유발하고 기형을 유발하므로 태아에게 심각한 해를 끼칠 수 있습니다. 여성이 임신 중이거나 임신이 의심되는 경우 포름 아미드 사용을 피해야합니다. 포름아미드는 LEV 흄 후드에 사용해야 하며 적절한 눈 또는 얼굴 보호구를 착용해야 합니다. - 커버슬립을 제거하려면 슬라이드를 '헹굼 용액'에 1분 동안 담근 다음 5°C에서 65분 동안 '세척 용액'에 넣습니다.
주의 : 세척 용액에는 1-5 % 폴리 옥시 에틸렌 옥틸 페닐 에테르와 1-5 % 염화나트륨이 포함되어 있습니다. 이것은 부식성이며 심각한 눈 손상을 일으킬 수 있습니다. 세척액을 취급할 때는 고글이나 안면 보호구를 착용해야 합니다. - 50mL의 얼음처럼 차가운 에탄올 시리즈(70%, 85% 및 95%(v/v))를 통해 농도당 2분 동안 슬라이드를 배양한 다음 자연 건조합니다. 일단 건식 마운트는 DAPI가 포함된 마운팅제로 슬라이드하고 커버슬립으로 오버레이합니다.
6. 이미지 캡처 및 분석
- DNA 후광과 염색체 영역을 시각화하려면 HC PL FLUOTAR 100X/1.30 오일 대물렌즈와 DFC365FX 카메라를 사용하여 이미지를 캡처하는 에피형광 현미경(예: Leica DM4000 현미경)을 사용합니다.
- 그레이 스케일 이미지를 캡처하고 캡처된 각 채널의 색상을 정의하여 이미지의 유사 색상을 사용할 수 있습니다. 상용 소프트웨어(예: LAS AF 버전 4.5.0 소프트웨어)가 이 실험에 사용되었습니다. 개별 색상 채널은 TIFF로 내보내졌습니다.
- Java 이미지 처리 프로그램인 Fiji ImageJ를 사용하여 이미지를 분석합니다. 파일 및 열기를 눌러 이미지를 업로드합니다.
- 이미지 | 색상 | 채널 분할. 이미지 채널을 선택하고 이미지 | 조정한 다음 Brightness & Contrast(밝기 및 대비)를 선택합니다. 그에 따라 변경하고 다른 채널로 반복하십시오.
- 핵을 묘사하는 DAPI 염색 채널을 선택하여 잔류 핵의 마스크를 만듭니다. 이미지 | 조정 한 다음 임계값을 선택합니다. 임계값을 변경할 수 있는 대화 상자가 나타나면 어두운 배경 상자를 선택합니다. 잔여 핵이 깨끗해질 때까지 변경하고 적용 을 누르고 대화 상자를 닫습니다.
참고: 이렇게 하면 픽셀 강도에 따라 이진 마스크가 생성되며 흰색 픽셀은 관심 영역을 나타내고 검은색 픽셀은 배경을 표시합니다. 프로브 채널에서 동일한 절차를 반복합니다. - 자유형 선택을 사용하여 잔여 핵의 주변을 윤곽선으로 표시한 다음 편집 및 외부 지우기(Edit and Clear Outside)를 클릭합니다. 프로브 채널을 잔류 핵에 오버레이합니다. 이 작업은 Image| 색상 | 채널을 병합합니다.
- ImageJ에서 측정 배율을 설정하려면 배율 막대 위에 또는 알려진 두 거리의 점 사이에 선을 그립니다. Analyze( 분석 )로 이동하여 Set Scale(배율 설정)을 누릅니다. 대화 상자에서 거리 길이를 추가하고 확인을 클릭합니다. 거리를 측정하려면 측정 중인 지점 사이에 선을 긋고 분석 | 측정. 이렇게 하면 거리 값이 데이터 창으로 전송됩니다.
- 핵의 중심과 일치하는 가장 밝은 DAPI 강도를 측정합니다. 이 측정에서 핵 중심에서 가장 먼 염색체 영역 가장자리(CTE)까지의 거리를 측정합니다. 핵 중심에서 핵 가장자리(NE)까지의 거리를 측정합니다.
- 결과가 CTE/NE 비율로 표시되는지 확인합니다. 여기서 핵 중심에서 각 가장 먼 염색체 영역 가장자리(CTE)까지의 거리는 핵 중심에서 각 핵 가장자리(NE)까지의 거리로 나뉩니다. 이것은 최소 50 개의 핵에서 수행되어야합니다. 이것은 막대 또는 상자 차트로 표시될 수 있습니다.
- 텔로미어 분석을 위해 데이터 세트당 최소 30개의 핵을 분석합니다. Fiji ImageJ를 사용하여 이미지를 분석하거나 수동으로 분석하여 잔여 핵 및 DNA 후광 내의 텔로미어 수를 계산할 수 있습니다. BrdU 또는 pKi67은 증식(BrdU/piK67+) 및 노화/정지(BrdU/pKi67-) 핵의 분화를 가능하게 했습니다. 데이터는 평균의 표준 오차(SEM)에 대응하는 오차 막대를 갖는 막대 차트로 묘사될 수 있다.
- 스튜던트 t-검정(쌍을 이루지 않음)을 사용하여 유의한 것으로 간주되는 p> 0.05와 결과를 통계적으로 비교합니다.
결과
이 DNA 후광 준비 방법은 젊은 세포와 노인 세포 내에서 게놈 행동의 차이를 확인하려는 노력뿐만 아니라 비정상적인 핵골격계 단백질을 가진 조기 노화 질환에서 파생된 세포에서도 차이를 결정하는 데 도움이 되었습니다15. 그림 1은 잔류 핵의 가장자리, 잔류 핵 내에 남아 있는 DNA, 주변 영역으로 스풀링되어 DNA 후광을 생성하는 부착되지 않은 DNA를 볼 수 있는 DNA 후광의 예를 보여줍니다. 또한 잔류 핵을 얻는 방법과 NE 및 CTE 측정을 보여주는 분석을 보여줍니다. 세포가 S-단계에 있을 때 BrdU와 같은 표지된 뉴클레오티드를 통합하거나 G1 세포에서 핵소체 및 헤테로크로마틴 영역을 나타내는 진단 증식 마커 anti-pKi67을 사용하여 증식 세포와 비증식 세포를 구별할 수 있습니다17,18. 증식 마커에 대해 음성인 confluency를 달성하지 않고 높은 혈청에서 성장한 일차 세포는 노화된 것으로 간주됩니다. 저혈청에서 성장하거나 합류하게 된 일차 세포, 즉 증식 마커에 대해 음성인 접촉 억제는 정지 상태로 간주되며 올바른 영양소와 상황이 주어지면 증식 세포 주기에 다시 들어갈 수 있습니다. Ki67 양성 세포와 음성 세포를 구별할 수 있게 됨으로써 증식, 정지 및 노화 인간 진피 섬유아세포의 차이를 결정할 수 있었습니다. 그림 2는 비증식 세포에서는 발생하지 않는 메커니즘인 DNA 복제 중에 BrdU가 혼입된 세포에서 생성된 증식하는 인간 진피 섬유아세포의 DNA 후광을 표시한 후 항-BrdU 항체로 염색됩니다. 증식 마커인 항-pKi67 항체를 사용한 염색도 도 2에서 볼 수 있습니다. 이것은 강력한 항원이며 FISH 프로토콜에서 살아남으므로 FISH 후 및 사전 장착을 위해 염색할 수 있습니다. 따라서, 증식 세포는 BrdU에 대해 양성(적색)이고 pKi67(적색)은 좌측 열에 항-pKi67(적색)이며, 비증식 세포는 도 2의 우측 열에 실제로 노화 세포가 표시된다. 녹색 신호는 텔로미어 PNA FISH/FITC 키트로 밝혀진 개별 텔로미어입니다. 면역형광과 DNA 후광을 결합하면 증식 세포, 정지 세포 및 노화 세포를 조사할 때 그림 2와 같이 다양한 세포 상태에서 분석할 수 있습니다. 선택한 항체에 따라 분화, 방사선 조사를 통한 DNA 손상 등과 같은 다른 조건을 검사할 수 있습니다.
염색체 영역은 FISH를 사용하여 DNA 후광 내에서 시각화할 수도 있습니다. DNA가 핵 밖으로 스풀링될 수 있도록 하기 때문에 잔류 핵 내부의 게놈 고정 및 그 구조에 따라 DNA 후광에서 발견되는 염색체의 양이 적거나 많기 때문에 염색체 영역 모양이 방해받을 수 있습니다. 그림 3 은 염색체 1, 13, 17 및 18에 대한 특정 전체 팔 염색체 페인팅 프로브(빨간색)와 함께 개별 염색체가 드러난 DNA 후광 패널을 보여줍니다. 항-pKi67(녹색)은 노화 세포를 나타내는 동일한 슬라이드에서 동일한 배양 내에서 증식하는 세포와 그 부재를 표시하는 데 사용되었습니다. CTE/NE로 제시된 이미지와 데이터에서 작은 유전자 결핍 염색체 18은 부착물이 거의 없고 잔류 핵에서 멀리 떨어진 DNA 후광으로 더 멀리 스풀링되고 다른 염색체보다 잔류 핵의 중심에서 훨씬 더 멀리 떨어져 있는 염색체임이 매우 분명합니다. 그러나 이것은 1번 염색체에도 해당됩니다. 증식 마커인 anti-pKi67을 사용하여 동일한 배양 및 동일한 슬라이드에서 노화 세포와 증식하는 것을 비교할 수 있었으며, 이 분석은 이 두 가지 매우 다른 세포 상태 내의 염색체가 잔류 핵 구조와의 부착과 관련하여 서로 크게 다르지 않다는 것을 보여주었습니다.
흥미롭게도, 유전자는 또한 잔류 핵 내에 남아 있거나 DNA 헤일로에 위치하는 것과 관련하여 증식 세포와 노화 세포 사이에 통계적으로 유의미한 차이를 보여주고 있습니다. 그림 4는 빨간색의 표지된 BAC 프로브와 녹색의 항-Ki67로 묘사된 유전자좌로 이를 보여줍니다. DNA Halo 준비 후 증식 세포와 노화 세포의 유전자 위치 사이에는 유의미한 차이가 없습니다. 그러나 DNA 후광 내에는 사이클린 D1 CNDD1 유전자좌보다 훨씬 더 많은 카테닌 알파 1 CTNNA1 유전자좌가 있습니다. 그림 5는 텔로미어가 녹색으로 표시된 DNA 후광 준비를 보여줍니다. 배경은 DNA 후광 내에서 텔로미어 신호를 시각화할 수 있도록 의도적으로 높게 둡니다. 이 데이터 세트에는 정지 세포, 즉 7일 동안 혈청이 굶주린 세포가 포함되었으며 흥미롭게도 증식 및 노화 세포보다 정지 세포의 DNA 후광 내에 부착되지 않고 위치하는 텔로미어가 훨씬 더 많습니다. 도 5a에서 DNA 후광에서 텔로미어의 비율이 관찰될 수 있는데, 특히 이미지 '실험 2'에 대해 관찰할 수 있다. 이는 DNA 후광에서 텔로미어의 평균 백분율이 정지 세포에서 약 17%인 그림 5b와 일치합니다. 노화 세포의 모든 텔로미어가 그 중 일부가 매우 짧기 때문에 볼 수 있는 것은 아니라는 증거가 있습니다.
이 DNA 후광 방법은 병든 세포의 핵 내에서 게놈 상호 작용 변화를 조사하는 데 성공적이었습니다15. 그림 6 은 1 차 대조 섬유 아세포와 전형적인 (라민 A 돌연변이) 및 비정형 허친슨-길 포드 프로 게 리아 증후군이있는 병든 세포에서 염색체 부착의 차이를 보여 주며, 다른 SUN1 이소형과 라민 A 돌연변이가 없습니다19. 염색체 1과 13은 대조군 DNA 후광과 비교할 때 잔류 핵 내에서 부착에 통계적으로 유의미한 차이를 보여줍니다. 그림 6b 는 전체 염색체 영역의 위치를 잔류 핵 및 DNA 헤일로와 연관시킵니다. 1 이하의 값은 염색체가 잔류 핵 내에 위치함을 나타내고 1 이상의 값은 DNA 헤일로 내의 염색체 또는 염색체의 일부를 나타냅니다.
전반적으로, 이것은 세포주기 (증식, 정지 및 노화) 또는 질병 세포 (예 : 프로 게 리아 및 암 세포주)에 영향을 미치는 다양한 조건 하에서 전체 염색체, 특정 유전자 및 텔로미어의 게놈 상호 작용을 조사하는 데 HALO-FISH의 유용성을 강조합니다. 실제로, 이러한 상태 간의 상호 작용의 차이는 핵골격이 핵 내의 주요 과정을 조절하는 데 중요한 역할을 한다는 것을 의미합니다.

그림 1: 잔류 핵과 DNA 후광을 표시하는 HDF 추출 핵 및 분석 방법 개요. (a) DNA 후광 분석을 통해 준비되고 DAPI로 대조염색된 HDF 핵. 밝게 염색된 잔류 핵은 핵골격에 고정된 DNA를 보여주며, 이것은 DNA의 후광을 형성하는 부착되지 않은 DNA로 둘러싸여 있습니다. 배율 = x 100; 스케일 바 10 μm. (b) 파란색 채널은 DAPI로 염색된 핵과 주변 DNA를 포착합니다. 잔류 핵은 ImageJ를 사용하여 선택 및 제거됩니다. 화살표는 핵 중심에서 잔류 핵 가장자리(NE)까지의 거리를 나타냅니다. (c) 빨간색 채널은 프로브 신호를 나타냅니다. (d) '결과'로 표시된 이미지는 파란색 채널 이미지에 빨간색 채널을 겹쳐 놓은 결과입니다. 이것은 핵 중심에서 가장 먼 염색체 영역 가장자리 (CTE)까지의 거리를 허용합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
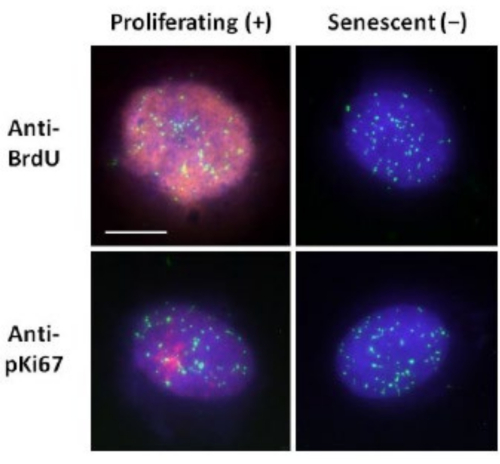
그림 2: 증식 및 노화 HDF에 대한 텔로미어 PNA FISH를 사용한 DNA 후광 준비. DNA 후광 분석을 거친 HDF의 텔로미어 PNA FISH. 텔로미어 신호는 녹색(FITC)으로 시각화하고, 잔류 및 후광 DNA는 DAPI(파란색)를 사용하여 대조염색하고, 증식 핵은 적색의 간접 면역형광(TRITC)을 통해 항-BrdU 또는 항-pKi67 항체를 사용하여 검출했습니다. 배율 = x 100; 스케일 바 10 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
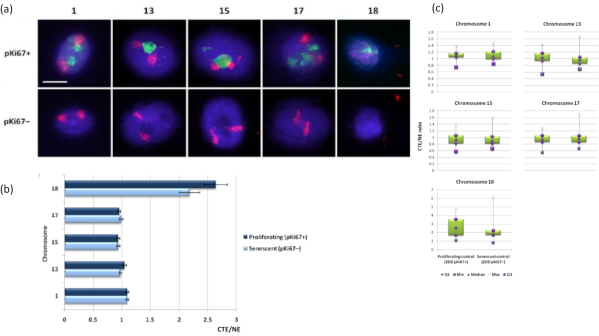
그림 3: DNA 후광 분석을 사용한 핵골격-염색체 상호 작용 및 분석. (a) 염색체 1, 13, 15, 17 및 18에 특이적인 프로브를 사용하여 DNA 후광 준비를 거친 HDF에서 2D-FISH를 수행했습니다. 전체 염색체를 빨간색(Cy3)으로 칠하고 핵을 pKi67로 조사하여 증식 또는 노화 여부를 확인했습니다. 증식 세포(pKi67+)는 녹색(FITC)으로 묘사된 반면, 노화 세포는 염색되지 않은 상태(pKi67-), 즉 녹색 신호가 감지되지 않았습니다. 배율 = x 100; 스케일 바 10 μm. (b) HALO-FISH를 겪은 증식 및 노화 HDF에서 뉴클레오스켈레톤에 의한 염색체 고정. 측정은 증식(pKi67+) 및 노화(pKi67-) 세포에서 염색체 1, 13, 15, 17 및 18에 대한 각 핵 가장자리(NE)에 대한 가장 먼 염색체 영역 가장자리(CTE)의 비율을 보여줍니다. 오차 막대는 SEM± 나타냅니다. (c) pKi67+ 및 pKi67- 핵에서 특정 염색체의 각 핵 가장자리(NE)에 대한 염색체 영역 가장자리(CTE)의 수정된 상자 그림 표현. Q1 = 하위 사분위수; 최소 = 기록된 최저값; Med = 중앙값; Max = 기록된 최대값; Q3 = 상위 사분위수. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
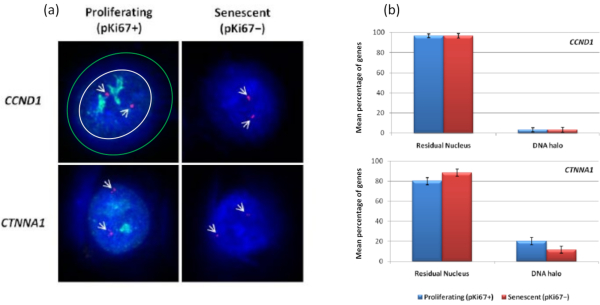
그림 4: HALO-FISH를 사용한 HDF의 유전자 특이적 상호 작용. (a) DNA 후광 추출 핵을 유전자 특이적 프로브(CCND1 및 CTNNA1)로 조사하여 증식 및 노화 세포에 대한 NM에 대한 고정을 조사했습니다. 유전자 신호는 빨간색(Cy3)으로 표시되고 항-pKi67은 증식하는 세포를 나타내고 신호는 녹색(FITC)으로 시각화됩니다. 증식하는 CCND1 이미지의 경우 잔류 핵이 흰색 원 안에 둘러싸여 있고 흰색과 녹색 원 사이의 공간은 DNA 헤일로를 나타냅니다. 배율 = x 100; 스케일 바 10 μm. (b) CCND1 및 CTNNA1에 대한 유전자 특이적 신호는 잔류 핵과 DNA 후광 사이, 그리고 증식 세포와 노화 세포 사이에서 비교됩니다. 오차 막대± SEM을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
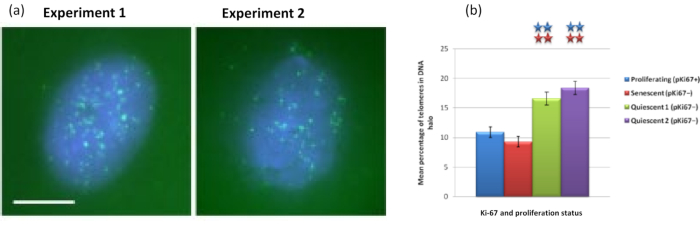
그림 5: 텔로미어 PNA-FISH로 프로브된 정지 HDF의 DNA 후광 분석. (a) HDFs의 정지는 7일 동안 저혈청 배지에서 배양함으로써 유도되었다. DNA 후광 분석을 수행하고, FITC 신호(녹색)에 의한 텔로미어의 PNA-FISH를 시각화하고, 잔류 핵 및 주변 DNA 후광을 DAPI(파란색)로 대조염색하였다. 세포는 또한 핵이 증식하지 않도록 항-pKi67 항체로 염색되었습니다. 이것은 두 차례에 걸쳐 반복되었습니다. 배율 = x 100; 스케일 바 10 μm. (b) 증식, 노화 및 정지 HDF 세포에서 DNA 후광 내에 국한된 텔로미어의 평균 백분율 비교. 오차 막대± SEM을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
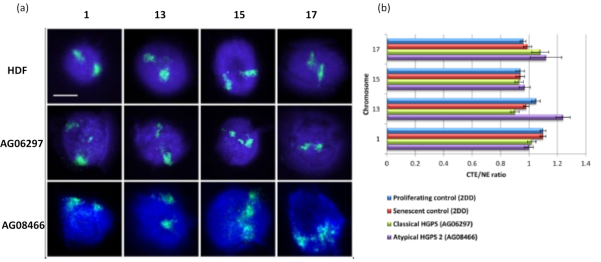
그림 6 : HALO-FISH26을 사용하여 HGPS 세포에서 뉴클레오스 골격에 대한 전체 염색체 앵커리지 검사. (a) 대조군 HDF (2DD), 고전적 HGPS (AG06297) 및 비정형 유형 2 HGPS (AG08466) 핵은 DNA 후광 준비를 거친 다음 염색체 1, 13, 15 및 17에 대한 전체 염색체 페인트를 사용하여 2D-FISH를 거쳤습니다. 전체 염색체는 녹색(FITC)으로 표시되고 DNA는 DAPI(파란색)로 대조염색되었습니다. 배율 = x 100; 스케일 바 10μm. (b) 추출된 핵 내에서 염색체의 위치는 핵 가장자리(NE)에 대한 평균 염색체 영역 가장자리(CTE)의 비율을 측정하여 결정되었습니다. 1보다 큰 비율은 가장 먼 CTE가 DNA 후광 내의 해당 NE 외부에 있음을 나타내고, 1보다 작은 비율은 가장 먼 CTE가 잔류 핵 내의 NE 내에 있음을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 성분 | 부피(μL) |
| 5XDOP-PCR 버퍼 | 10 |
| dNTPmix(제외dTTP)(2mM) | 5 |
| 디티피(2mM) | 2 |
| 비오틴-16-dUTPor디곡시제닌-11-dUTP | 10 |
| DOP프라이머(20μM) | 5 |
| TaqDNAPolymerase (1U/μL) | 1 |
| PCR그레이드워터 | 12 |
| 템플렛 | 5 |
표 1: 1x 반응에 대한 DOP-PCR 성분 및 부피를 나타내는 표
| 걸음 | 사이클 | 온도 (섭씨도) | 시간 |
| 초기 변성 | 1 | 95 | 3 분 |
| 변성 | 34 | 98 | 20초 |
| 프라이머 어닐링 | 62 | 1 분 | |
| 확장 | 72 | 30초 | |
| 최종 연장 | 1 | 72 | 5 분 |
| 냉각 | 4 | 들다 |
표 2: DOP-PCR 주기, 온도 및 시간 프로파일을 나타내는 표.
| 성분 | 부피(μL) |
| 10x NT 완충액(0.5M Tris-HCl pH 8,50 mM MgCl2, 0.5 mg/ml BSA) | 5 |
| 0.1 M 베타-메르캅토에탄올 | 5 |
| 10X 뉴클레오티드 스톡(0.5 mM dATP, 0.5 mM dCTP, 0.5 mM dGTP, 0.5 mM dTTP, 0.5 mg/ml 비오틴-16-dUTP) | 5 |
| 드나제 I(1ng/ml) | 2 |
| DNA중합효소 I | DNA μg당 5U |
| DNAtemplate (1 μg) | 1 |
| DEPC 처리수 | 최대 50 μL |
표 3: 하나의 프로브에 대한 닉 변환 성분 및 부피를 보여주는 표.
토론
DNA 후광 방법은 뉴클레오스켈레톤과 게놈 간의 상호 작용을 분석할 때 선택하는 훌륭한 방법이지만 준수해야 하는 몇 가지 중요한 단계도 있습니다. 가장 중요한 매개 변수 중 하나는 세포 파종 밀도의 최적화입니다. 세포가 과도하게 융합되면 DNA 후광이 이웃 세포와 겹쳐 분석을 수행할 수 없게 됩니다. CSK 및 추출 완충액은 생물학적 활성을 유지하기 위해 준비 과정이 끝날 때 추출 완충액에 스페르민, 스페르미딘 및 디지토닌을 첨가하여 사용 당일에 항상 신선하게 만들어야 합니다. Halo-FISH를 수행하는 경우 DNA 후광의 정확한 변성 온도를 사용하여 프로브 또는 페인트가 이후에 혼성화될 수 있도록 하는 것이 매우 중요합니다.
전자 현미경은 필라멘트 구조가 확인된 핵 매트릭스를 시각화하는 데 사용되어 왔다20. 그러나 전자 현미경은 염색질과의 매트릭스 연관성을 쉽게 추론할 수 없기 때문에 제한적입니다. 실제로 DNA Halo 방법은 특정 유전자, 염색체 및 세포 상태를 모두 검사할 수 있기 때문에 전자 현미경에 비해 더 다재다능합니다. 또한, 핵 매트릭스 단백질의 단백질체 분석이 연구되고 있다21,22. 이 방법은 특히 병든 세포를 비교할 때 핵 매트릭스 구성 요소를 비교하는 데 유용하지만 표준 DNA Halo 기술로 강조된 공간 분포 및 부착을 제공하지 않습니다.
DNA Halo 분석에는 한계가 있습니다. 첫째, 매트릭스가 추출될 때 고정된 세포에서만 수행할 수 있으므로 라이브 이미징이 불가능합니다. DNA Halo 방법은 비교적 빠르고 쉽게 수행할 수 있지만 세포 배양, 프로브 생성, Halo-FISH 및 분석을 모두 고려할 때 전체 프로세스에 시간이 많이 소요될 수 있습니다.
초해상도 현미경을 사용한 DNA Halos 및 HALO-FISH의 이미지 캡처는 DNA 특이적 프로브 및 항체의 해상도를 크게 향상시킬 것입니다. 또한, 형광색소는 스펙트럼으로 더 쉽게 분리될 수 있기 때문에 단일 실험에서 많은 DNA 프로브를 사용하여 더 많은 정보를 제공할 수 있습니다. 염색체 형태 포획(3C)과 같은 분자 생물학 기술의 개선은 유전자좌의 상호 작용을 결정하고 세포의 염색질에 대한 공간 조직을 분석하는 데 사용되었습니다. DNA 헤일로 분석과 3C를 결합할 수 있으며, 이는 M3C23으로 알려진 용어로, DNA 헤일로 기술의 적응성을 다시 보여줍니다.
여기에 제시된 원본 데이터는 게놈 행동 조사의 가능성과 이러한 데이터를 제시하는 방법을 보여주기 위한 것입니다. 이러한 데이터를 통해 우리는 (1) 염색체 페인팅 프로브를 사용하여 게놈 부착의 유의미한 차이를 결정할 수 있음을 입증했으며, 이 연구에서 18번 염색체가 분석된 염색체 중 가장 적게 부착된 염색체임을 밝혔습니다(그림 3). (2) 두 유전자좌 사이에 상당한 차이가 있는 유전자좌(그림 4) (3) 증식 및 노화 세포에 비해 정지 세포에서 덜 강하게 부착되는 텔로미어(그림 5). 우리는 불용성 단백질인 증식 마커 Ki67 항원의 존재를 통해 증식 세포와 비증식 세포를 구별할 수 있으며, 이는 잔류 핵과 함께 남아 있거나 특정 기간 내에 S상을 통과한 세포를 강조하기 위해 뉴클레오티드의 결합을 사용합니다(그림 2). 이 기술을 통해 우리는 핵골격, 즉 laminopathy 세포에서 손상된 세포의 게놈 거동을 분석 할 수 있었으며 Bikkul et al., 2018 우리는 게놈이 대조군 세포와 비교할 때 덜 밀접하게 부착 될 수 있으며 고전적인 HGPS 세포에서 lamin A 돌연변이의 효과를 개선하는 특정 약물로 치료할 때 회복 될 수 있음을 밝혔습니다15. 그러나, 우리는 라민 A 돌연변이가 없지만 염색체 13이 덜 밀접하게 부착되어있는 LINC 복합체 단백질 SUN19 1의 특이한 형태를 포함하는 비정형 HGPS AGO8466 세포에 대한 새로운 데이터를 보여준다 (그림 6).
HALO-FISH는 추출 절차에서 제거되지 않은 단백질을 분해하기 위해 간접 면역형광과 함께 뉴클레오스켈레톤과의 게놈 상호작용 연구를 가능하게 하는 독특한 방법입니다. 뉴클레오스켈레톤은 특정 암 유형19과 같은 다양한 질병에서 변형되고 진단 바이오마커로서 일부 뉴클레오스켈레톤 관련 단백질의 중요성이 입증되었습니다24,25. 따라서, 이 기술은 질병 15,24,25,27에서 염색질 조직/해체에 대한 뉴클레오스켈레톤의 효과를 조사 하는 데 중요한 역할을 하며, 인간 세포에 국한되지 않고, 다른 동물의 염색체 페인팅 프로브를 사용하여 동일한 DNA-후광 프로토콜을 사용할 수 있다 28.
공개
저자는 공개 할 것이 없습니다.
감사의 말
염색체 팔 페인팅 프로브의 친절한 선물에 대해 Michael Bittner 교수에게 감사드립니다. LG는 EU가 자금을 지원하는 EURO-Laminopathies 프로젝트와 Brunel Progeria Research Fund의 지원을 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| 10X PBS | Thermo Fisher Scientific | 10388739 | Used to create DNA halos |
| 5-bromo-2′-deoxy-uridine | Sigma-Aldrich | B5002-100MG | Labelled nucleotide |
| 5-Fluoro-2′-deoxyuridine | Sigma-Aldrich | F0503-100MG | Labelled nucleotide |
| Agar Technical | Thermo Fisher Scientific | 15562141 | DNA isolation of BAC clones |
| Agarose | Sigma-Aldrich | A939-50G | Check product size of DOP-PCR and nick translation |
| Atypical type 2 HGPS fibroblasts (AG08466) | Coriell Institute | AG08466 | Cell line |
| Bacto tryptone | Thermo Fisher Scientific | 16269751 | DNA isolation of BAC clones |
| Biotin-16-dUTP | Roche Diagnostics | 11093711103 | Labelled nucleotides |
| Chloramphenicol | Sigma-Aldrich | C0378-25G | DNA isolation of BAC clones |
| Classical Hutchinson-Gilford progeria syndrome (HGPS) fibroblasts (AG06297) | Coriell Institute | AG0297 | Cell line |
| Coplin jar | Thermo Fisher Scientific | 12608596 | Holds 5 slides or 8 slides back to back |
| Cot-1 DNA | Thermo Fisher Scientific | 15279011 | Block nonspecific hybridization in HALO FISH |
| DEPC-treated water | Sigma-Aldrich | 693520-1L | DNA isolation of BAC clones |
| Dextran sulphate | Sigma-Aldrich | S4030 | Hybridisation mixture |
| Digitonin | Sigma-Aldrich | D141 | Component of extraction buffer |
| Digoxigenin-11-dUTP | Sigma-Aldrich | 11093088910 | Labelled nucleotides |
| Donkey anti-mouse Cy3 | Jackson Laboratory | 715-165-150 | Secondary antibody |
| EDTA | Sigma-Aldrich | E6758 | Component of extraction buffer |
| Ethanol | Component of extraction buffer | ||
| Ethanol | Sigma-Aldrich | 443611 | Probe precipitation and HALO FISH |
| Fetal bovine system | Thermo Fisher Scientific | 26140079 | Cell culture serum |
| Formamide | Thermo Fisher Scientific | 10523525 | 2D FISH of DNA halos |
| Glass wool | Sigma-Aldrich | 18421 | Spin column |
| Herring sperm | Sigma-Aldrich | D7290 | Probe precipitation |
| HXP™ Lamp (metal halide microscope lamp) | OSRAM | HXP-R120W45C VIS | Image capture of DNA halos |
| Hydrochloric acid | Thermo Fisher Scientific | 10313680 | Cleaning microscope slides |
| Isopropanol | Sigma-Aldrich | I9516-25ML | DNA isolation of BAC clones |
| KAPA HiFi PCR Kit | KAPA Biosystems | KK2103 | PCR Kit |
| Leica DM4000 fluorescent microscope with DFC365 FX camera and LAS AF (Version: 4.5.0) image acquisition software. | Leica Microsystems | Image capture of DNA halos | |
| Luria-Bertani agar | Thermo Fisher Scientific | 13274843 | DNA isolation of BAC clones |
| Magnesium chloride | Sigma-Aldrich | M8266 | Component of CSK buffer |
| Methanol | Thermo Fisher Scientific | 10284580 | Cleaning and sterilizing microscope slides |
| Mouse anti-BrdU antibody | BD Pharmingen | B2531-100UL | BrdU visualisation |
| Newborn calf serum | Thermo Fisher Scientific | 16010159 | Cell culture serum and blocking reagent |
| Nick translation kit | Invitrogen | ||
| PCR grade water | Sigma-Aldrich | 693520-1L | PCR and DNA isolation of BAC clones |
| PCR Primers | Sigma-Aldrich | ||
| PIPES | Sigma-Aldrich | P1851 | Component of CSK and extraction buffers |
| Potassium acetate | Sigma-Aldrich | P1190-100G | DNA isolation of BAC clones |
| QuadriPERM® 4 X 12 | SARSTEDT | 94.6077.307 | Square cell culture dish, polysterene with four compartments. This has hydrophobic surface, is sterile, non-pyrogenic/endotoxin-fee and non-cytotoxic. |
| Rabbit Anti-Ki67 antibody | Sigma-Aldrich | ZRB1007-25UL | Proliferation marker |
| Rnase A | Sigma-Aldrich | R6513 | DNA isolation of BAC clones |
| Rubber cement | Halford's | 101836 | 2D FISH of DNA halos |
| Sephadex G-50 | Sigma-Aldrich | S6022-25G | Spin column |
| Sodium acetate | Sigma-Aldrich | S2889 | Probe precipitation |
| Sodium chloride | Sigma-Aldrich | S5886 | Component of CSK, extraction and SSC buffers |
| Sodium citrate | Sigma-Aldrich | C8532 | Component of SSC buffer |
| Sodium dodecyl sulphate | L3771-100G | DNA isolation of BAC clones | |
| Sodium hydroxide | Sigma-Aldrich | S8045-500G | DNA isolation of BAC clones |
| Spermidine | Sigma-Aldrich | S2626 | Component of extraction buffer |
| Spermine | Sigma-Aldrich | S4264 | Component of extraction buffer |
| Streptavidin-Cy3 | Amersham Life Sciences Ltd, Scientific Laboratory Supplies | pa43001 | Probe antibody |
| Sucrose | Sigma-Aldrich | S0389 | Component of CSK buffer |
| Sucrose | Sigma-Aldrich | S0389 | CSK buffer+A66:D68 |
| SuperFrost™ microscope slides | Thermo Fisher Scientific | 12372098 | Microscope slides: 1 mm thickness, 76 mm length, 26 mm width. Uncoated. |
| Swine anti-rabbit TRITC | Dako | ||
| TELO-PNA FISH KIT | Agilent Dako | K532511-8 | Delineation of telomeres |
| Tris-HCl | Sigma-Aldrich | T3253-100G | Column buffer |
| Triton™ X-100 | Sigma-Aldrich | T9284 | Component of CSK buffer |
| Tryptone | Thermo Fisher Scientific | 10158962 | DNA isolation of BAC clones |
| Tween-20 | Sigma-Aldrich | P9416- 100ML | Detergent |
| Vectashield mountant containing DAPI | Vector Laboratories | H-1200 | 2D FISH of DNA halos |
| Whole human chromosome probes | Calbiochem | 2D FISH of DNA halos | |
| Yeast extract | Thermo Fisher Scientific | 10108202 | DNA isolation of BAC clones |
참고문헌
- Berezney, R., Coffey, D. S. Identification of a nuclear protein matrix. Biochemical Biophysical Research Communications. 60 (4), 1410-1417 (1974).
- Haaf, T., Ward, D. C. High resolution ordering of YAC contigs using extended chromatin and chromosomes. Human Molecular Genetics. 3 (4), 629-633 (1994).
- Parra, I., Windle, B. High resolution visual mapping of stretched DNA by fluorescent hybridization. Nature Genetics. 5 (1), 17-21 (1993).
- Senger, G., et al. Released chromatin: linearized DNA for high resolution fluorescence in situ hybridization. Human Molecular Genetics. 3 (8), 1275-1280 (1994).
- Florijn, R. J., et al. High-resolution DNA Fiber-FISH for genomic DNA mapping and colour bar-coding of large genes. Human Molecular Genetics. 4 (5), 831-836 (1995).
- Elcock, L. S., Bridger, J. M. Fluorescence in situ hybridization on DNA halo preparations and extended chromatin fibres. Methods Molecular Biology. 659, 21-31 (2010).
- Heiskanen, M., et al. Visual mapping by fiber-FISH. Genomics. 30 (1), 31-36 (1995).
- Bensimon, A., et al. Alignment and sensitive detection of DNA by a moving interface. Science. 265 (5181), 2096-2098 (1994).
- Michalet, X., et al. Dynamic molecular combing: stretching the whole human genome for high-resolution studies. Science. 277 (5331), 1518-1523 (1997).
- Wilson, R. H., Coverley, D. Relationship between DNA replication and the nuclear matrix. Genes Cells. 18 (1), 17-31 (2013).
- Wilson, R. H. C., Coverley, D. Transformation-induced changes in the DNA-nuclear matrix interface, revealed by high-throughput analysis of DNA halos. Science Reports. 7 (1), 6475 (2017).
- Iarovaia, O. V., Akopov, S. B., Nikolaev, L. G., Sverdlov, E. D., Razin, S. V. Induction of transcription within chromosomal DNA loops flanked by MAR elements causes an association of loop DNA with the nuclear matrix. Nucleic Acids Research. 33 (13), 4157-4163 (2005).
- Tandara, M., et al. Sperm DNA integrity testing: big halo is a good predictor of embryo quality and pregnancy after conventional IVF. Andrology. 2 (5), 678-686 (2014).
- Repping, S., et al. The use of spermHALO-FISH to determine DAZ gene copy number. Mol Human Reproduction. 9 (4), 183-188 (2003).
- Bikkul, M. U., et al. Farnesyltransferase inhibitor and rapamycin correct aberrant genome organisation and decrease DNA damage respectively, in Hutchinson-Gilford progeria syndrome fibroblasts. Biogerontology. 19 (6), 579-602 (2018).
- Telenius, H., et al. Degenerate oligonucleotide-primed PCR: general amplification of target DNA by a single degenerate primer. Genomics. 13 (3), 718-725 (1992).
- Bridger, J. M., et al. Association of pKi-67 with satellite DNA of the human genome in early G1 cells. Chromosome Research. 6, 13-24 (1998).
- Sales Gil, R., Vagnarelli, P. Ki-67: More Hidden behind a 'Classic Proliferation Marker'. Trends in Biochemical Sciences. 43 (10), 747-748 (2018).
- Bikkul, M. U., et al. Telomere elongation through hTERT immortalization leads to chromosome repositioning in control cells and genomic instability in Hutchinson-Gilford progeria syndrome fibroblasts, expressing a novel SUN1 isoform. Genes Chromosomes Cancer. 58 (6), 341-356 (2019).
- Jackson, D. A., Cook, P. R. Visualization of a filamentous nucleoskeleton with a 23 nm axial repeat. EMBO Journal. 7, 3667-3677 (1988).
- Albrethsen, J., et al. Unravelling the nuclear matrix proteome. Journal of Proteomics. 72, 71-81 (2009).
- Mika, S., Rost, B. NMPdb: Database of nuclear matrix proteins. Nucleic Acids Research. 33, 160-163 (2005).
- Gavrilov, A. A., et al. of the nuclear matrix-bound chromatin hubs by a new M3C experimental procedure. Nucleic Acids Research. 38, 8051-8060 (2010).
- Sjakste, N., et al. Role of the nuclear matrix proteins in malignant transformation and cancer diagnosis. Experimental Oncology. 26 (3), 170-178 (2004).
- Leman, E. S., Getzenberg, R. H. Nuclear structure as a source of cancer specific biomarkers. Journal of Cellular Biochemistry. 104 (6), 1988-1993 (2008).
- Volpi, E. V., Bridger, J. M. FISH glossary: an overview of the fluorescence in situ hybridization technique. Biotechniques. 45 (4), 385-386 (2008).
- Bridger, J. M., Foster, H. A. Senescence and the Genome. Human Interphase Chromosomes. , (2021).
- Foster, H. A., Griffin, D. K., Bridger, J. M. Interphase chromosome positioning in in vitro porcine cells and ex vivo porcine tissues. BMC Cell Biology. 13 (1), 30 (2012).
Erratum
Formal Correction: Erratum: Fluorescence In Situ Hybridization on DNA Halo Preparations to Reveal Whole Chromosomes, Telomeres and Gene Loci
Posted by JoVE Editors on 6/05/2023. Citeable Link.
An erratum was issued for: Fluorescence In Situ Hybridization on DNA Halo Preparations to Reveal Whole Chromosomes, Telomeres and Gene Loci. The Authors section was updated from:
Lauren S. Godwin1
Joanna M. Bridger1
Helen A. Foster2
1Laboratory of Nuclear and Genomic Health, Centre for Genome Engineering and Maintenance, Division of Biosciences, Department of Life Sciences, College of Health, Medicine and Life Sciences, Brunel University London
2Biosciences, Department of Clinical, Pharmaceutical and Biological Science, School of Life and Medical Sciences, University of Hertfordshire
to:
Lauren S. Godwin1
Emily Roberts2
Joanna M. Bridger1
Helen A. Foster2
1Laboratory of Nuclear and Genomic Health, Centre for Genome Engineering and Maintenance, Division of Biosciences, Department of Life Sciences, College of Health, Medicine and Life Sciences, Brunel University London
2Biosciences, Department of Clinical, Pharmaceutical and Biological Science, School of Life and Medical Sciences, University of Hertfordshire
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유