このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
足骨造茎および前駆細胞株における短いヘアピンRNA媒介遺伝子ノックダウンによる樹状細胞の発達に関する研究
要約
ここでは、shRNAのレンチウイルス伝達を用いて樹状細胞(DC)の開発に関与する潜在的な転写因子をスクリーニングするためのプロトコルを提供し、 インビトロ DC分化のための安定したノックダウン細胞株を得る。
要約
樹状細胞(DC)は、自然免疫応答と適応免疫応答を結ぶ重要な抗原提示細胞です。DC は異種であり、従来の DC (cDC) とプラズマサイトイド DC (pDC) に分割できます。cDCは、ナイーブT細胞に抗原を提示し、活性化することを専門としています。一方、pDCはウイルス感染中にI型インターフェロン(IFN-I)を大量に産生する可能性がある。DC の仕様は、骨髄 (BM) の DC 前駆物質の初期段階で発生し、転写因子 (TF) のネットワークによって定義されます。たとえば、cdc は ID2 を高度に表現し、pDC は E2-2 を高度に表現します。DC のサブセットが増えるため、DC 開発を制御する特定の TF を理解することに関心が高まっています。ここでは、短いヘアピンRNA(shRNA)を含むレンチウイルスを不死化造血幹細胞および前駆細胞(iHSPCs)ラインに送達することにより、 インビトロで DC分化に不可欠なTFをスクリーニングする方法を確立します。選択および インビトロ 分化後、安定したノックダウン細胞株のcDCおよびpDC電位は、フローサイトメトリーによって分析される。このアプローチは、 インビトロの前駆物質からDC運命を支配する可能性のある遺伝子を同定するためのプラットフォームを提供します。
概要
DC は、自然免疫と適応性イミュニティ1の主要なレギュレータです。DC は、主に 2 つの機能的に異なる集団、すなわち pDC と cDC に分類されます。さらに、cdc は 2 つのサブセット、すなわち、タイプ I とタイプ II cDC または cDC1s と cDC2s をそれぞれ 2 から構成します。pDCは、マウス3,4におけるCD11cのBST2、Siglec-H、および中間レベルを発現し、炎症およびウイルス感染時に大量のIFN-Iを分泌することができる細胞である5。また、その堅牢なIFN-I産生能により、全身性エリテマトース(SLE)6を含む自己免疫疾患の進行に重要な役割を果たしていると考えられています。cDC1sは、マウス7におけるXCR1、CD8a、CLEC9AおよびCD103の表面発現によって定義され、抗原クロスプレゼンテーションを通じて細胞傷害性CD8+T細胞(CTLs)の活性化および分極化に特化し、細胞内病原体および癌に応答してI型免疫を始める8,9。一方、cDC2sは、ヒトとマウスの両方でCD11bおよびCD172α(Sirpαとも呼ばれる)を発現し、CD4+T細胞を活性化し、アレルゲンおよび寄生虫10に対するII型免疫応答を促進し、ならびに細胞外細菌および微生物叢認識に続くIII型免疫を調節することができる11,12。
DCの多様化は、造血幹細胞および前駆細胞(HSPCs)から取得したTF群によって決定される。対照的に、DNA結合2(ID2)の阻害剤はcDCの仕様を駆動し、Eタンパク質活性を遮断してpDCの発達を阻害する15。さらに、cDC1sの開発にはIRF8とBATF3が必要ですが、cDC2sの分化はIRF416に大きく依存しています。最近の研究では、pDC17とccdcの異質性とその転写規則18を探っています。DC ネットワークは複雑であるため、DC の開発と機能を制御する他の TF を識別するプラットフォームを確立する必要があります。
ここでは、BM細胞(Hoxb8-FL細胞とも呼ばれる)19においてホックスb8のエストロゲン制御核転座を発現して生成したiHSPCを用いた。iHSPCsは増殖し、βエストラジオールおよびFlt3リガンド(FL)の存在下で未分化段階にとどまる一方、βエストラジオール19の離脱時にFLの存在下で異なるDCタイプに分化し始める。この特徴を踏まえて、前駆細胞で目的の遺伝子を倒し、続いてpDCやCCの インビトロ 分化に及ぼす影響を調べることができる。したがって、この方法は、DCの開発と機能を調節する遺伝子を発見するための強力なツールです。
プロトコル
レンチウイルスの取扱いは、国立台湾大学医学部環境衛生学科の規制に従って行われます。
1. 不死化造血幹細胞および前駆細胞株(iHSPCs)の調製
- 100 ng/mL FL および 1 μM βエストラジオールを含む完全な RPMI 1640 培地で iHSPC 細胞株を維持します。
- 3日ごとに1:10の比率で細胞を通過させる。
注:10%のウシ胎児血清(FBS)、5 x 10-5 M βメルカプトエタノール、および10 μg/mLのゲンタマイシンを補うことによって完全なRPMI 1640培地を作ります。組換えマウスFLも市販されている。
2. レンチウイルス導入
- 100 ng/mL FL、1 μM βエストラジオール、8 μg/mL ポリブレンを含む完全な培地の1 mLの12ウェルプレートで1 x 105 細胞/ウェルの密度でiHSPCsをプレートします。
注:ポリブレンの濃度は細胞の種類に依存し、通常は4〜8 μg/mLの範囲です。 - 100の感染の多重度(MOI)で各ウェルにshRNAを運ぶレンチウイルスを加える。
注意:レンチウイルスベクターは、pLKO.1-PLKO.1-POマイシン選択マーカーを有する(図1)。 LacZ、 Tcf4、 および Id2 に対する shRNA のターゲット シーケンスは、それぞれ 、材料表に記載されています。MOIは、感染時に細胞当たりのビリオンの数によって定義されます。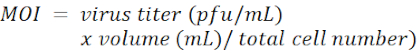
- プレートを37°Cで90分間1,100 x g で回転させます。
注:スピン感染は、スイングバケットローターを使用して行われます。 - インキュベーターで37°Cで一晩感染した細胞を含むプレートをインキュベートします。
注:細胞がポリブレンに敏感な場合は、スピン感染後にポリブレンなしで完全な培地で細胞をリフレッシュしてください。 - 感染後100ng/mL FLおよび1μM βエストラジオール24時間を含む完全な培地で細胞をリフレッシュする。
- 6 μg/mLのピューロマイシンを培地に加え、さらに24時間後に感染細胞を選択します。
注:トランスデューシングレンチウイルスベクターは、通常、ピューロマイシン耐性遺伝子を含む遺伝子を発現するために48時間かかります。ピューロマイシンの濃度が細胞株ごとに最適化されていることを確認してください。 - 100 ng/mL FL、1 μM βエストラジオール、6 μg/mL ピューロマイシンを含む選択培地を3日ごとにリフレッシュし、少なくとも1週間は細胞を維持して安定してトランスデューシングされたiHSPC細胞を拡張します。
注:プロマイシンの選択は、通常48時間後に有効になり、選択期間は、細胞の種類に依存します。
3. 逆転写とリアルタイムPCR(RT-PCR)によるノックダウン効率の測定
- 1 x 107 shLacZ、shTcf4、およびshId2 安定したノックダウンiHSPC細胞から、市販のRNA抽出試薬を用いてRNAを抽出し、水性層からイソプロパノールでRNAを沈殿させ、続いて75%エタノールでRNA沈殿物を洗浄した。
- RNA(~5 μg)をDEPC処理H2Oの5 μLで溶解し、濃度を1 μg/μLに調整します。
- RNAを1〜3μgとし、DEPC処理H2Oと混合して最終体積17.4 μLに、1単位/μL RNaseフリーDNase Iを1 μL加え、37°Cで20分間インキュベートします。
注:このステップは、RNAサンプル中のゲノムDNAを消化することです。 - RNAサンプルに1μLの20mM EDTAを加え、65°Cで10分間インキュベートしてDNase Iを不活性化し、RNAサンプルを4°Cに直ちに入れます。
- 1μLのオリゴ(dT)プライマー(45μM)、5x 1st ストランドバッファの6μL、dNTP(2 mM)の3 μL、RNase阻害剤(50単位/μL)の0.6 μL、逆転写酵素1μL(200単位/μL)を含む反応ミックスを11.6μL加えます。
- 70°Cで10分間加熱して反応を停止し、30 μLのH2Oで反応ミックスを希釈します。
- 希釈したRT反応ミックスを2μLをDNAテンプレートとしてとり、PCRはTcf4またはId2に対するプライマーを使用して増幅します(サーモサイクラー条件については 表1 を参照)。
注: プライマー シーケンスは 、マテリアル テーブルに含まれています。
4. 安定したノックダウン iHSPC 細胞株の インビトロ 分化
- FLの100 ng/mLおよび1 μMのβエストラジオールを含む完全な媒体の LacZ (shLacZ)、 Tcf4 (shTcf4)、または Id2 (shId2)の安定した単一のノックダウンを維持する。
- shLacZ、shTcf4、およびshId2未分化iHSpcsを15 mLチューブに集め、遠心分離機を500 x gで5分間回収し、細胞をペレット化します。
- 上清を捨て、10mLのPBSを加え、細胞を洗浄します。この手順を 2 回繰り返します。
- iHSPC細胞を2 x 105 細胞/mLの密度で12ウェルプレートに再播種し、100 ng/mL FLのみを含む完全な培地の1mLで12ウェルプレートに播種します。
- 3日後に100 ng/mL FLを含む新鮮な完全培地を1mL追加します。
- 2日後にフローサイトメトリーによって分化細胞(shLacZ、shTcf4、およびshId2 iHSPCs)を分析します。
5. 分化されたDCのフローサイトメトリック解析
- プレート内で細胞を上下に2~3回ピペット化し、室温で500 x g で5分間遠心分離機を使用して細胞を1.5 mLチューブに集めます(
メモ: インビトロ で差別化されたDCはプレートにわずかに付着します。穏やかなピペットはプレートからDCを回復するのに役立ちます。 - 上清を捨て、50 μLのFACSバッファで細胞を再中断します。次に、抗CD16/32ハイブリドーマ上清を50μL加え、氷上で5〜10分間インキュベートします。
注:Fcブロッキング抗体は、いくつかの骨髄細胞およびB細胞上で発現するFc受容体に対する抗体の非特異的結合を防止し、様々なベンダーを通じて市販されています。 - 蛍光色素共役抗体(各抗体に0.04 μg)を直接細胞に加え、暗闇の中で氷上で15分間インキュベートします。使用される抗体は、APC/cy7抗マウスCD11c、FITC抗マウスCD11b、およびPE抗マウスB220です。
注: pDC は CD11c+CD11b-B220+ として定義され、cdc は CD11c+ CD11b+ B220- として定義されます。 - 1 mLのFACSバッファーと遠心分離機を用いて、RTで500 x g で5分間洗浄します。
- 細胞を100 μLのFACSバッファに再懸濁し、フローサイトメトリーで分析します。
結果
レンチウイルスベクターpLKO.1-Poの地図を示す(図1)。 iHSPではLacZ(非標的対照)、Tcf4、およびId2に対してレンチウイルス発現shRNAを送達した後、RT-qPCRによって確認されたノックダウン効率は、shLacZ iHSPCsと比較してshTcf4 iHSPCsでTcf4の発現が低下したことを明らかにした(図2A)。一方、shId2 iHSPCsでは、shLacZ
ディスカッション
レンチウイルスベースのshRNAベクターは、ウイルスによる細胞への導入による遺伝子サイレンシングに使用されることが多く、宿主ゲノムへの安定的な統合が可能です。しかし、様々な細胞種における様々な導入効率を考慮する必要があり、この問題を克服するために多くのアプローチがとられている。
ポリブレンは、細胞膜上の電荷を中和し、それによって細胞への?...
開示事項
著者らは開示するものは何もない。
謝辞
Tz-Ling Chen博士の技術サポートに感謝しています。私たちは、shRNAレンチウイルス(http://rnai.genmed.sinica.edu.tw)を提供してくれた国立RNAiコア施設(台湾のアカデミア・シニカ)に感謝します。この研究は、台湾の科学技術省(MOST 108-2320-B-002-037-MY3およびMOST 109-2320-B-002-054-MY3)によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| APC/Cy7 anti-mouse CD11c Antibody | Biolegend | 117324 | (Clone: N418) |
| FITC anti-mouse/human CD11b Antibody | Biolegend | 101206 | (Clone: M1/70) |
| PE anti-mouse/human B220 Antibody | Biolegend | 103208 | (Clone: RA3-6B2) |
| Cell culture | |||
| 1.5 mL Micro tube | ExtraGene | TUBE-170-C | |
| 12-well tissue culture-treated plate | Falcon | 353043 | |
| Fetal bovine serum (FBS) | Corning | 35-010-CV | |
| RPMI 1640 medium | gibco | 11875-085 | |
| Reagent | |||
| β-estradiol | Sigma-Aldrich | E2758-250MG | |
| β-mercaptoethanol (β-ME) | Sigma-Aldrich | M6250 | |
| FACS buffer | home-made | Formula: 1xPBS+0.5 %FBS+0.1%NaN3 | |
| Flt3 ligand (FL) | home-made | ||
| Polybrene | Sigma-Aldrich | TR-1003-G | |
| Puromycin | Invivogen | ant-pr-1 | |
| TRIsure | BIOLINE | BIO-38032 | |
| shRNA (Taregt sequence/clone ID) | Company | ||
| shId2 (GCTTATGTCGAATGATAGCAA/TRCN0000054390) | The RNAi Consortium (TRC) | ||
| shLacZ (CGCGATCGTAATCACCCGAGT/TRCN0000072224) | The RNAi Consortium (TRC) | ||
| shTcf4 (GCTGAGTGATTTACTGGATTT/TRCN0000012094) | The RNAi Consortium (TRC) | ||
参考文献
- Steinman, R. M., Witmer, M. D. Lymphoid dendritic cells are potent stimulators of the primary mixed leukocyte reaction in mice. Proceedings of the National Academy of Sciences. 75 (10), 5132-5136 (1978).
- Guilliams, M., et al. Dendritic cells, monocytes and macrophages: a unified nomenclature based on ontogeny. Nature Reviews Immunology. 14 (8), 571-578 (2014).
- Blasius, A. L., Cella, M., Maldonado, J., Takai, T., Colonna, M. Siglec-H is an IPC-specific receptor that modulates type I IFN secretion through DAP12. Blood. 107 (6), 2474-2476 (2006).
- Swiecki, M., Colonna, M. Unraveling the functions of plasmacytoid dendritic cells during viral infections, autoimmunity, and tolerance. Immunology Reviews. 234 (1), 142-162 (2010).
- Liu, Y. -. J. IPC: Professional Type 1 interferon-producing cells and plasmacytoid dendritic cell precursors. Annual Review of Immunology. 23 (1), 275-306 (2005).
- Panda, S. K., Kolbeck, R., Sanjuan, M. A. Plasmacytoid dendritic cells in autoimmunity. Current Opinion in Immunology. 44, 20-25 (2017).
- Durai, V., Murphy, K. M. Functions of murine dendritic cells. Immunity. 45 (4), 719-736 (2016).
- Mashayekhi, M., et al. CD8α(+) dendritic cells are the critical source of interleukin-12 that controls acute infection by Toxoplasma gondii tachyzoites. Immunity. 35 (2), 249-259 (2011).
- Anderson, D. A., Dutertre, C. -. A., Ginhoux, F., Murphy, K. M. Genetic models of human and mouse dendritic cell development and function. Nature Reviews Immunology. 21 (2), 101-115 (2021).
- Plantinga, M., et al. Conventional and monocyte-derived CD11b(+) dendritic cells initiate and maintain T helper 2 cell-mediated immunity to house dust mite allergen. Immunity. 38 (2), 322-335 (2013).
- Persson, E. K., et al. IRF4 transcription-factor-dependent CD103(+)CD11b(+) dendritic cells drive mucosal T helper 17 cell differentiation. Immunity. 38 (5), 958-969 (2013).
- Kumamoto, Y., et al. CD301b+ dermal dendritic cells drive T helper 2 cell-mediated immunity. Immunity. 39 (4), 733-743 (2013).
- Cisse, B., et al. Transcription factor E2-2 is an essential and specific regulator of plasmacytoid dendritic cell development. Cell. 135 (1), 37-48 (2008).
- Grajkowska, L. T., et al. Isoform-specific expression and feedback regulation of E protein TCF4 control dendritic cell lineage specification. Immunity. 46 (1), 65-77 (2017).
- Jackson, J. T., et al. Id2 expression delineates differential checkpoints in the genetic program of CD8α+ and CD103+ dendritic cell lineages. The EMBO Journal. 30 (13), 2690-2704 (2011).
- Suzuki, S., et al. Critical roles of interferon regulatory factor 4 in CD11bhighCD8alpha- dendritic cell development. Proceedings of the National Academy of Science U. S. A. 101 (24), 8981-8986 (2004).
- Rodrigues, P. F., et al. Distinct progenitor lineages contribute to the heterogeneity of plasmacytoid dendritic cells. Nature Immunology. 19 (7), 711-722 (2018).
- Brown, C. C., et al. Transcriptional basis of mouse and human dendritic cell heterogeneity. Cell. 179 (4), 846-863 (2019).
- Redecke, V., et al. Hematopoietic progenitor cell lines with myeloid and lymphoid potential. Nature Methods. 10 (8), 795-803 (2013).
- Abe, A., Miyanohara, A., Friedmann, T. Polybrene increases the efficiency of gene transfer by lipofection. Gene Therapy. 5 (5), 708-711 (1998).
- Sorgi, F. L., Bhattacharya, S., Huang, L. Protamine sulfate enhances lipid-mediated gene transfer. Gene Therapy. 4 (9), 961-968 (1997).
- Kirkling, M. E., et al. Notch signaling facilitates in vitro generation of cross-presenting classical dendritic cells. Cell Reports. 23 (12), 3658-3672 (2018).
- Pearce, E. J., Everts, B. Dendritic cell metabolism. Nature Reviews. Immunology. 15 (1), 18-29 (2015).
- Wculek, S. K., Khouili, S. C., Priego, E., Heras-Murillo, I., Sancho, D. Metabolic Control of Dendritic Cell Functions: Digesting Information. Frontiers in Immunology. 10 (775), (2019).
- Saxton, R. A., Sabatini, D. M. mTOR signaling in growth, metabolism, and disease. Cell. 168 (6), 960-976 (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved