Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Untersuchung der dendritischen Zellentwicklung durch kurze Haarnadel RNA-vermittelte Gen-Knockdown in einem hämatopoetischen Stamm und Vorläuferzelllinie In vitro
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll für das Screening potenzieller Transkriptionsfaktoren zur Verfügung, die an der Entwicklung dendritischer Zellen (DC) beteiligt sind, unter Verwendung der lentiviralen Transduktion von shRNA, um stabile Knockdown-Zelllinien für die In-vitro-DC-Differenzierung zu erhalten.
Zusammenfassung
Dendritische Zellen (DCs) sind wichtige Antigen-präsentierende Zellen, die angeborene und adaptive Immunantworten verbinden. DCs sind heterogen und können in konventionelle DCs (cDCs) und plasmazytoide DCs (pDCs) unterteilt werden. cDCs ist darauf spezialisiert, naiven T-Zellen Antigene zu präsentieren und zu aktivieren. Auf der anderen Seite können pDCs während einer Virusinfektion große Mengen an Typ-I-Interferonen (IFN-I) produzieren. Die Spezifizierung von DCs erfolgt in einem frühen Stadium von DC-Vorläufern im Knochenmark (BM) und wird durch ein Netzwerk von Transkriptionsfaktoren (TFs) definiert. Zum Beispiel drücken CDCs ID2 hoch aus, während pDCs E2-2 stark ausdrücken. Da immer mehr Teilmengen von DCs identifiziert werden, besteht ein wachsendes Interesse daran, spezifische TFs zu verstehen, die die DC-Entwicklung steuern. Hier etablieren wir eine Methode zum Screening von TFs, die für die DCs-Differenzierung in vitro entscheidend sind, indem lentivirus-tragende kurzhaarnadelige RNA (shRNA) in eine immortalisierte hämatopoetische Stamm- und Vorläuferzelllinie (iHSPCs) abgegeben wird. Nach der Selektion und In-vitro-Differenzierung werden cDC- und pDC-Potential der stabilen Knockdown-Zelllinien mittels Durchflusszytometrie analysiert. Dieser Ansatz bietet eine Plattform, um Gene zu identifizieren, die möglicherweise das DC-Schicksal von Vorläufern in vitro bestimmen.
Einleitung
DCs sind wichtige Regulatoren der angeborenen und adaptiven Immunität1. DCs werden hauptsächlich in zwei funktional unterschiedliche Populationen eingeteilt, nämlich pDCs und cDCs. Darüber hinaus umfassen cDCs zwei Untergruppen, nämlich Typ I und Typ II cDCs oder cDC1s bzw. cDC2s2s. pDCs, die BST2, Siglec-H und mittlere CD11c-Spiegel in Mäusen exprimieren3,4, sind die Zellen, die während Entzündungen und Virusinfektionen große Mengen an IFN-I sezernieren können5. Aufgrund ihrer robusten IFN-I-produzierenden Fähigkeit stehen sie auch im Verdacht, eine Schlüsselrolle beim Fortschreiten von Autoimmunerkrankungen, einschließlich des systemischen Lupus erythematodes (SLE)6, zu spielen. cDC1s, definiert durch die Oberflächenexpression von XCR1, CD8a, CLEC9A und CD103 in Mäusen7, sind auf die Aktivierung und Polarisation von zytotoxischen CD8+ T-Zellen (CTLs) durch die Antigen-Kreuzpräsentation spezialisiert und initiieren dadurch eine Typ-I-Immunität als Reaktion auf intrazelluläre Erreger und Krebs8,9. Auf der anderen Seite können cDC2s, die CD11b und CD172α (auch bekannt als Sirpα) sowohl bei Menschen als auch bei Mäusen exprimieren, CD4+ T-Zellen aktivieren und die Typ-II-Immunantwort gegen Allergene und Parasiten10 fördern sowie die Typ-III-Immunität nach extrazellulärer Bakterien und Mikrobiota-Erkennung modulieren11,12.
Die Diversifikation von DCs wird durch eine Gruppe von TFs aus hämatopoetischen Stamm- und Vorläuferzellen (HSPCs) im BM bestimmt. E2-2 (kodiert von Tcf4) ist ein Hauptregulator für die Differenzierung und Funktion von pDCs13,14. Im Gegensatz dazu treibt der Inhibitor der DNA-Bindung 2 (ID2) die cDC-Spezifikation voran und hemmt die pDC-Entwicklung durch Blockierung der E-Proteinaktivität15. Darüber hinaus erfordert die Entwicklung von cDC1 IRF8 und BATF3, während die Differenzierung von cDC2s stark von IRF416 abhängt. Neuere Arbeiten haben die Heterogenität von pDCs17 und cDCs und deren transkriptioneller Regulation untersucht18. Aufgrund der Komplexität des DC-Netzwerks besteht ein unerfüllter Bedarf, eine Plattform einzurichten, um andere TFs zu identifizieren, die die Entwicklung und Funktionalität von DCs steuern.
Hier verwendeten wir einen iHSPC, der durch Exprimieren von Östrogen-regulierter Kerntranslokation von Hoxb8 in BM-Zellen (auch hoxb8-FL-Zellen genannt) erzeugt wurde19. iHSPCs können sich in Gegenwart von β-Östradiol und Flt3-Liganden (FL) vermehren und in einem undifferenzierten Stadium verbleiben, während sie sich in Gegenwart von FL nach Abzug von β-Östradiol19 in verschiedene DC-Typen zu differenzieren beginnen. Basierend auf dieser Eigenschaft können wir Interessante Gene im Vorläuferstadium niederschlagen, gefolgt von der Untersuchung der Wirkung auf die In-vitro-Differenzierung von pDCs und cDCs. Daher ist diese Methode ein leistungsfähiges Werkzeug, um die Gene zu entdecken, die die Entwicklung und Funktion von DCs regulieren.
Protokoll
Die Handhabung des Lentivirus erfolgt gemäß den Vorschriften der Abteilung für Umweltgesundheit und -sicherheit des National Taiwan University College of Medicine.
1. Herstellung von immortalisierten hämatopoetischen Stamm- und Vorläuferzelllinien (iHSPCs)
- Halten Sie die iHSPC-Zelllinie in einem vollständigen RPMI 1640-Medium mit 100 ng/ml FL und 1 μM β-Östradiol.
- Passieren Sie die Zellen in einem Verhältnis von 1:10 alle 3 Tage.
HINWEIS: Machen Sie das vollständige RPMI 1640-Medium, indem Sie es mit 10% fetalem Rinderserum (FBS), 5 x 10-5 M β-Mercaptoethanol und 10 μg / ml Gentamicin ergänzen. Rekombinantes murines FL ist auch im Handel erhältlich.
2. Lentivirale Transduktion
- Platten-iHSPCs mit einer Dichte von 1 x 105 Zellen/Well in 12-Well-Platten in 1 ml komplettem Medium, das 100 ng/ml FL, 1 μM β-Östradiol und 8 μg/ml Polybrenn enthält.
HINWEIS: Die Konzentration von Polybren hängt von den Zelltypen ab und liegt in der Regel im Bereich von 4-8 μg/ml. - Fügen Sie shRNA-tragendes Lentivirus in jede Vertiefung bei einer Vielzahl von Infektionen (MOI) von 100 hinzu.
HINWEIS: Der lentivirale Vektor ist pLKO.1-Puro mit einem Puromycin-Selektionsmarker (Abbildung 1). Die Zielsequenzen von shRNAs gegen LacZ, Tcf4 bzw. Id2 sind in der Materialtabelle aufgeführt. Der MOI wird durch die Anzahl der Virionen definiert, die während der Infektion pro Zelle hinzugefügt werden.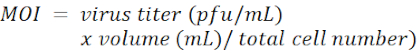
- Die Platten bei 1.100 x g für 90 min bei 37 °C drehen.
HINWEIS: Die Spin-Infektion wird mit einem Schwenklöffelrotor durchgeführt. - Inkubieren Sie die Platte mit infizierten Zellen über Nacht bei 37 °C in einem Inkubator.
HINWEIS: Wenn die Zellen empfindlich auf Polybren reagieren, erfrischen Sie die Zellen nach der Spininfektion mit dem vollständigen Medium ohne Polybrenn. - Aktualisieren Sie die Zellen mit einem vollständigen Medium, das 100 ng / ml FL und 1 μM β-Östradiol 24 h nach der Infektion enthält.
- 6 μg/ml Puromycin in das Medium geben, um die infizierten Zellen nach weiteren 24 h auszuwählen.
HINWEIS: Transduzierter lentiviraler Vektor benötigt normalerweise 48 h für die Exprimierung von Genen, einschließlich Puromycin-resistentem Gen. Stellen Sie sicher, dass die Konzentration von Puromycin für jede Zelllinie optimiert ist. - Aktualisieren Sie das Selektionsmedium mit 100 ng/ml FL, 1 μM β-Östradiol und 6 μg/ml Puromycin alle 3 Tage und halten Sie die Zellen mindestens eine Woche lang aufrecht, um die stabil transduzierten iHSPC-Zellen zu erweitern.
HINWEIS: Die Puromycin-Selektion tritt in der Regel 48 h später in Kraft, und der Zeitraum der Selektion hängt von den Zelltypen ab.
3. Messung der Knockdown-Effizienz durch Reverse Transcription und Real-Time PCR (RT-PCR)
- Extrahieren Sie die Gesamt-RNA aus 1 x 107 shLacZ-, shTcf4- und shId2-stabilen Knockdown-iHSPC-Zellen unter Verwendung eines kommerziellen RNA-Extraktionsreagenzes und scheiden Sie RNA aus der wässrigen Schicht mit Isopropanol aus, gefolgt von einem Waschen der RNA-Niederschlagung mit 75% Ethanol.
- Lösen Sie die RNA (~ 5 μg) mit 5 μL DEPC-behandeltem H2O auf und stellen Sie die Konzentration auf 1 μg/μL ein.
- 1-3 μg RNA nehmen, mit DEPC-behandeltem H2O auf ein Endvolumen von 17,4 μL mischen, 1 μL 1 Einheit/μL RNase-freie DNase I hinzufügen und 20 min bei 37 °C inkubieren.
HINWEIS: Dieser Schritt besteht darin, die genomische DNA in den RNA-Proben zu verdauen. - 1 μL 20 mM EDTA zu den RNA-Proben geben, bei 65 °C für 10 min inkubieren, um DNase I zu inaktivieren, und die RNA-Proben sofort bei 4 °C platzieren.
- Den RNA-Proben werden 11,6 μL der Reaktionsmischung mit 1 μL Oligo (dT)-Primern (45 μM), 6 μL 5x 1st-Strangpuffer, 3 μL dNTP (2 mM), 0,6 μL RNase-Inhibitor (50 Unit/μL) und 1 μL Reversease (200 Unit/μL) zugegeben und bei 40 °C für 1 h inkubiert.
- Stoppen Sie die Reaktion durch Erhitzen bei 70 °C für 10 min und verdünnen Sie die Reaktionsmischung mit 30 μL H2O.
- Man nimmt 2 μL der verdünnten RT-Reaktionsmischung als DNA-Vorlage und verstärkt sie mit Primern gegen Tcf4 oder Id2 (siehe Tabelle 1 für Thermocycler-Bedingungen).
HINWEIS: Die Primersequenzen sind in der Materialtabelle enthalten.
4. In vitro Differenzierung der stabilen Knockdown-iHSPC-Zelllinien
- Halten Sie den stabilen einzelnen Knockdown von LacZ (shLacZ), Tcf4 (shTcf4) oder Id2 (shId2) in iHSPCs in einem vollständigen Medium aufrecht, das 100 ng/ml FL und 1 μM β-Östradiol enthält.
- Sammeln Sie shLacZ, shTcf4 und shId2 undifferenzierte iHSPCs in einem 15-ml-Röhrchen und zentrifugieren Sie für 5 min bei 500 x g , um die Zellen zu pelletieren.
- Verwerfen Sie den Überstand und fügen Sie 10 ml PBS hinzu, um die Zellen zu waschen. Wiederholen Sie diesen Schritt zweimal.
- Resuspendieren und säen Sie die iHSPC-Zellen mit einer Dichte von 2 x 105 Zellen / ml in eine 12-Well-Platte in 1 ml vollständigem Medium, das nur 100 ng / ml FL enthält.
- Fügen Sie drei Tage später 1 ml frisches komplettes Medium mit 100 ng / ml FL hinzu.
- Analysieren Sie die differenzierten Zellen (shLacZ, shTcf4 und shId2 iHSPCs) zwei Tage später mittels Durchflusszytometrie.
5. Durchflusszytometrische Analyse der differenzierten DCs
- Sammeln Sie die Zellen in 1,5 ml Röhrchen, indem Sie die Zellen 2-3 Mal in der Platte auf und ab pipettieren und für 5 min bei 500 x g bei Raumtemperatur (RT) zentrifugieren.
HINWEIS: In vitro ätzen sich differenzierte DCs leicht an die Platten. Sanftes Pipettieren hilft, DCs von den Platten zurückzugewinnen. - Verwerfen Sie den Überstand und resuspenieren Sie die Zellen in 50 μL FACS-Puffer. Als nächstes fügen Sie 50 μL Anti-CD16/32-Hybridom-Überstand hinzu und inkubieren Sie für 5-10 min auf Eis.
HINWEIS: Der Fc-blockierende Antikörper verhindert die unspezifische Bindung von Antikörpern an Fc-Rezeptoren, die auf einigen myeloischen Zellen und B-Zellen exprimieren, und ist kommerziell über verschiedene Anbieter erhältlich. - Fluoreszierende Farbstoff-konjugierte Antikörper (0,04 μg für jeden Antikörper) direkt in die Zellen geben und 15 min auf Eis im Dunkeln inkubieren. Die verwendeten Antikörper sind APC/cy7 Anti-Maus CD11c, FITC Anti-Maus CD11b und PE Anti-Maus B220.
HINWEIS: pDCs sind definiert als CD11c+CD11b-B220+, und cDCs sind definiert als CD11c+CD11b+B220-. - Zellen mit 1 ml FACS-Puffer waschen und 5 min bei 500 x g RT zentrifugieren.
- Resuspendieren Sie die Zellen in 100 μL FACS-Puffer und analysieren Sie sie durch Durchflusszytometrie.
Ergebnisse
Die Karte des lentiviralen Vektors pLKO.1-Puro ist dargestellt (Abbildung 1). Nach der Verabreichung von Lentivirus- exprimierender shRNA gegen LacZ (eine Nicht-Targeting-Kontrolle), Tcf4 und Id2 in iHSPCs zeigte die durch RT-qPCR bestätigte Knockdown-Effizienz, dass die Expression von Tcf4 in shTcf4-iHSPCs im Vergleich zu shLacZ-iHSPCs reduziert war (Abbildung 2A). Auf der anderen Seite wurde die vermindert...
Diskussion
Lentivirus-basierte shRNA-Vektoren werden häufig für das Gen-Silencing durch virale Transduktion in Zellen verwendet und ermöglichen eine stabile Integration in das Wirtsgenom. Es müssen jedoch verschiedene Transduktionseffizienzen in verschiedenen Zelltypen berücksichtigt werden, und es wurden eine Reihe von Ansätzen verfolgt, um dieses Problem zu lösen.
Polybren ist ein polykationisches Polymer, das die Ladungen auf der Zellmembran neutralisieren kann, wodurch die Bindung des Virions...
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Wir sind dankbar für die technische Unterstützung von Dr. Tz-Ling Chen. Wir danken der National RNAi Core Facility (Academia Sinica, Taiwan) für die Bereitstellung von shRNA Lentivirus (http://rnai.genmed.sinica.edu.tw). Diese Arbeit wurde vom Ministerium für Wissenschaft und Technologie, Taiwan (MOST 108-2320-B-002-037-MY3 und MOST 109-2320-B-002-054-MY3) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| APC/Cy7 anti-mouse CD11c Antibody | Biolegend | 117324 | (Clone: N418) |
| FITC anti-mouse/human CD11b Antibody | Biolegend | 101206 | (Clone: M1/70) |
| PE anti-mouse/human B220 Antibody | Biolegend | 103208 | (Clone: RA3-6B2) |
| Cell culture | |||
| 1.5 mL Micro tube | ExtraGene | TUBE-170-C | |
| 12-well tissue culture-treated plate | Falcon | 353043 | |
| Fetal bovine serum (FBS) | Corning | 35-010-CV | |
| RPMI 1640 medium | gibco | 11875-085 | |
| Reagent | |||
| β-estradiol | Sigma-Aldrich | E2758-250MG | |
| β-mercaptoethanol (β-ME) | Sigma-Aldrich | M6250 | |
| FACS buffer | home-made | Formula: 1xPBS+0.5 %FBS+0.1%NaN3 | |
| Flt3 ligand (FL) | home-made | ||
| Polybrene | Sigma-Aldrich | TR-1003-G | |
| Puromycin | Invivogen | ant-pr-1 | |
| TRIsure | BIOLINE | BIO-38032 | |
| shRNA (Taregt sequence/clone ID) | Company | ||
| shId2 (GCTTATGTCGAATGATAGCAA/TRCN0000054390) | The RNAi Consortium (TRC) | ||
| shLacZ (CGCGATCGTAATCACCCGAGT/TRCN0000072224) | The RNAi Consortium (TRC) | ||
| shTcf4 (GCTGAGTGATTTACTGGATTT/TRCN0000012094) | The RNAi Consortium (TRC) |
Referenzen
- Steinman, R. M., Witmer, M. D. Lymphoid dendritic cells are potent stimulators of the primary mixed leukocyte reaction in mice. Proceedings of the National Academy of Sciences. 75 (10), 5132-5136 (1978).
- Guilliams, M., et al. Dendritic cells, monocytes and macrophages: a unified nomenclature based on ontogeny. Nature Reviews Immunology. 14 (8), 571-578 (2014).
- Blasius, A. L., Cella, M., Maldonado, J., Takai, T., Colonna, M. Siglec-H is an IPC-specific receptor that modulates type I IFN secretion through DAP12. Blood. 107 (6), 2474-2476 (2006).
- Swiecki, M., Colonna, M. Unraveling the functions of plasmacytoid dendritic cells during viral infections, autoimmunity, and tolerance. Immunology Reviews. 234 (1), 142-162 (2010).
- Liu, Y. -. J. IPC: Professional Type 1 interferon-producing cells and plasmacytoid dendritic cell precursors. Annual Review of Immunology. 23 (1), 275-306 (2005).
- Panda, S. K., Kolbeck, R., Sanjuan, M. A. Plasmacytoid dendritic cells in autoimmunity. Current Opinion in Immunology. 44, 20-25 (2017).
- Durai, V., Murphy, K. M. Functions of murine dendritic cells. Immunity. 45 (4), 719-736 (2016).
- Mashayekhi, M., et al. CD8α(+) dendritic cells are the critical source of interleukin-12 that controls acute infection by Toxoplasma gondii tachyzoites. Immunity. 35 (2), 249-259 (2011).
- Anderson, D. A., Dutertre, C. -. A., Ginhoux, F., Murphy, K. M. Genetic models of human and mouse dendritic cell development and function. Nature Reviews Immunology. 21 (2), 101-115 (2021).
- Plantinga, M., et al. Conventional and monocyte-derived CD11b(+) dendritic cells initiate and maintain T helper 2 cell-mediated immunity to house dust mite allergen. Immunity. 38 (2), 322-335 (2013).
- Persson, E. K., et al. IRF4 transcription-factor-dependent CD103(+)CD11b(+) dendritic cells drive mucosal T helper 17 cell differentiation. Immunity. 38 (5), 958-969 (2013).
- Kumamoto, Y., et al. CD301b+ dermal dendritic cells drive T helper 2 cell-mediated immunity. Immunity. 39 (4), 733-743 (2013).
- Cisse, B., et al. Transcription factor E2-2 is an essential and specific regulator of plasmacytoid dendritic cell development. Cell. 135 (1), 37-48 (2008).
- Grajkowska, L. T., et al. Isoform-specific expression and feedback regulation of E protein TCF4 control dendritic cell lineage specification. Immunity. 46 (1), 65-77 (2017).
- Jackson, J. T., et al. Id2 expression delineates differential checkpoints in the genetic program of CD8α+ and CD103+ dendritic cell lineages. The EMBO Journal. 30 (13), 2690-2704 (2011).
- Suzuki, S., et al. Critical roles of interferon regulatory factor 4 in CD11bhighCD8alpha- dendritic cell development. Proceedings of the National Academy of Science U. S. A. 101 (24), 8981-8986 (2004).
- Rodrigues, P. F., et al. Distinct progenitor lineages contribute to the heterogeneity of plasmacytoid dendritic cells. Nature Immunology. 19 (7), 711-722 (2018).
- Brown, C. C., et al. Transcriptional basis of mouse and human dendritic cell heterogeneity. Cell. 179 (4), 846-863 (2019).
- Redecke, V., et al. Hematopoietic progenitor cell lines with myeloid and lymphoid potential. Nature Methods. 10 (8), 795-803 (2013).
- Abe, A., Miyanohara, A., Friedmann, T. Polybrene increases the efficiency of gene transfer by lipofection. Gene Therapy. 5 (5), 708-711 (1998).
- Sorgi, F. L., Bhattacharya, S., Huang, L. Protamine sulfate enhances lipid-mediated gene transfer. Gene Therapy. 4 (9), 961-968 (1997).
- Kirkling, M. E., et al. Notch signaling facilitates in vitro generation of cross-presenting classical dendritic cells. Cell Reports. 23 (12), 3658-3672 (2018).
- Pearce, E. J., Everts, B. Dendritic cell metabolism. Nature Reviews. Immunology. 15 (1), 18-29 (2015).
- Wculek, S. K., Khouili, S. C., Priego, E., Heras-Murillo, I., Sancho, D. Metabolic Control of Dendritic Cell Functions: Digesting Information. Frontiers in Immunology. 10 (775), (2019).
- Saxton, R. A., Sabatini, D. M. mTOR signaling in growth, metabolism, and disease. Cell. 168 (6), 960-976 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten