Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Étude du développement de cellules dendritiques par knockdown de gènes médiés par l’ARN en épingle à cheveux court dans une lignée cellulaire hématopoïétique et progénitrice in vitro
Dans cet article
Résumé
Nous fournissons ici un protocole pour le dépistage des facteurs de transcription potentiels impliqués dans le développement de cellules dendritiques (DC) en utilisant la transduction lentivirale de l’ARNh pour obtenir des lignées cellulaires knockdown stables pour la différenciation DC in vitro .
Résumé
Les cellules dendritiques (DC) sont d’importantes cellules présentatrices d’antigènes qui relient les réponses immunitaires innées et adaptatives. Les DC sont hétérogènes et peuvent être divisés en DC conventionnels (cDC) et en DC plasmocytoïdes (pDC). cDCs se spécialise dans la présentation d’antigènes et l’activation de lymphocytes T naïfs. D’autre part, les pDC peuvent produire de grandes quantités d’interférons de type I (IFN-I) lors d’une infection virale. La spécification des DC se produit à un stade précoce des progéniteurs DC dans la moelle osseuse (BM) et est définie par un réseau de facteurs de transcription (TF). Par exemple, les cDC expriment fortement ID2, tandis que les pDC expriment fortement E2-2. Étant donné que de plus en plus de sous-ensembles de DC sont identifiés, il existe un intérêt croissant pour la compréhension de TF spécifiques contrôlant le développement de DC. Ici, nous établissons une méthode pour dépister les TF critiques pour la différenciation des DC in vitro en délivrant un lentivirus porteur d’ARN en épingle à cheveux court (shRNA) dans une lignée de cellules souches et progénitrices hématopoïétiques immortalisées (iHSPC). Après la sélection et la différenciation in vitro , le potentiel cDC et pDC des lignées cellulaires stables est analysé par cytométrie en flux. Cette approche fournit une plate-forme pour identifier les gènes potentiellement régissant le destin des DC des progéniteurs in vitro.
Introduction
Les DC sont des régulateurs clés de l’immunité innée et adaptative1. Les PAYS sont principalement classés en deux populations fonctionnellement distinctes, à savoir les PDC et les CDC. En outre, les CDC comprennent deux sous-ensembles, à savoir les CDC de type I et de type II ou cDC1 et cDC2, respectivement2. Les pDC, exprimant BST2, Siglec-H et des niveaux intermédiaires de CD11c chez la souris3,4, sont les cellules qui peuvent sécréter de grandes quantités d’IFN-I lors de l’inflammation et de l’infection virale5. En raison de leur capacité robuste à produire des IFN-I, ils sont également soupçonnés de jouer un rôle clé dans la progression des maladies auto-immunes, y compris le lupus érythémateux disséminé (LED)6. Les cDC1, définis par l’expression de surface de XCR1, CD8a, CLEC9A et CD103 chez la souris7, sont spécialisés dans l’activation et la polarisation des lymphocytes T CD8+ cytotoxiques (CTL) par la présentation croisée de l’antigène, initiant ainsi une immunité de type I en réponse aux agents pathogènes intracellulaires et au cancer8,9. D’autre part, les cDC2, exprimant CD11b et CD172α (également connus sous le nom de Sirpα) chez l’homme et la souris, peuvent activer les lymphocytes T CD4+ et favoriser la réponse immunitaire de type II contre les allergènes et les parasites10, ainsi que moduler l’immunité de type III après la reconnaissance des bactéries extracellulaires et du microbiote11,12.
La diversification des DC est déterminée par un groupe de TF à partir de cellules souches et progénitrices hématopoïétiques (HSPC) dans le BM. E2-2 (codé par Tcf4) est un régulateur maître pour la différenciation et la fonction des pDC13,14. En revanche, l’inhibiteur de la liaison à l’ADN 2 (ID2) détermine la spécification du cDC et inhibe le développement du pDC en bloquant l’activité de la protéine E15. De plus, le développement des cDC1 nécessite IRF8 et BATF3, tandis que la différenciation des cDC2 dépend fortement de l’IRF416. Des travaux récents ont exploré l’hétérogénéité des pDC17 et des cDC et leur régulation transcriptionnelle18. En raison de la complexité du réseau CC, il existe un besoin non satisfait d’établir une plate-forme pour identifier d’autres TF contrôlant le développement et la fonctionnalité des DC.
Ici, nous avons utilisé un iHSPC qui a été généré en exprimant la translocation nucléaire régulée par les œstrogènes de Hoxb8 dans les cellules BM (également appelées cellules Hoxb8-FL)19. Les iHSPC peuvent proliférer et rester dans un stade indifférencié en présence de β-estradiol et de ligand Flt3 (FL), alors qu’ils commencent à se différencier en différents types DC en présence de FL lors du retrait du β-estradiol19. Sur la base de cette caractéristique, nous pouvons détruire les gènes d’intérêt au stade progéniteur, puis examiner l’effet sur la différenciation in vitro des pDC et des cDC. Par conséquent, cette méthode est un outil puissant pour découvrir les gènes qui régulent le développement et la fonction des DC.
Protocole
La manipulation du lentivirus est effectuée conformément à la réglementation du Département de la santé et de la sécurité environnementales du Collège de médecine de l’Université nationale de Taiwan.
1. Préparation de lignées cellulaires hématopoïétiques et progénitrices immortalisées (iHSPC)
- Maintenir la lignée cellulaire iHSPC dans un milieu RPMI 1640 complet contenant 100 ng/mL FL et 1 μM β-estradiol.
- Passage des cellules à un rapport de 1:10 tous les 3 jours.
REMARQUE: Rendre le milieu RPMI 1640 complet en complétant avec 10% de sérum bovin fœtal (FBS), 5 x 10-5 M β-mercaptoéthanol et 10 μg / mL de gentamicine. Recombinant murin FL est également disponible dans le commerce.
2. Transduction lentivirale
- Plaques iHSPC à une densité de 1 x 105 cellules/puits dans des plaques à 12 puits dans 1 mL de milieu complet contenant 100 ng/mL FL, 1 μM de β-estradiol et 8 μg/mL de polybrene.
REMARQUE: La concentration de polybrene dépend des types de cellules et est généralement comprise entre 4 et 8 μg / mL. - Ajouter le lentivirus porteur d’ARNh dans chaque puits à une multiplicité d’infection (MOI) de 100.
REMARQUE: Le vecteur lentiviral est pLKO.1-Puro avec un marqueur de sélection de puromycine (Figure 1). Les séquences cibles des shRNA contre LacZ, Tcf4 et Id2, respectivement, sont répertoriées dans le tableau des matériaux. Le MOI est défini par le nombre de virions qui sont ajoutés par cellule pendant l’infection.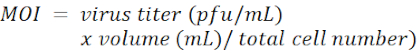
- Faire tourner les plaques à 1 100 x g pendant 90 min à 37 °C.
REMARQUE: L’infection par rotation est effectuée à l’aide d’un rotor à godet oscillant. - Incuber la plaque contenant les cellules infectées pendant la nuit à 37 °C dans un incubateur.
REMARQUE: Si les cellules sont sensibles au polybrène, rafraîchissez les cellules avec le milieu complet sans polybrène après l’infection par spin. - Rafraîchir les cellules avec un milieu complet contenant 100 ng/mL FL et 1 μM β-estradiol 24 h après l’infection.
- Ajouter 6 μg/mL de puromycine au milieu pour sélectionner les cellules infectées après 24 h supplémentaires.
REMARQUE: Le vecteur lentiviral transducté prend généralement 48 heures pour exprimer les gènes, y compris le gène résistant à la puromycine. S’assurer que la concentration de puromycine est optimisée pour chaque lignée cellulaire. - Actualiser le milieu de sélection contenant 100 ng/mL FL, 1 μM de β-estradiol et 6 μg/mL de puromycine tous les 3 jours et maintenir les cellules pendant au moins une semaine pour dilater les cellules iHSPC transduites de manière stable.
REMARQUE: La sélection de la puromycine prend généralement effet 48 heures plus tard, et la période de sélection dépend des types de cellules.
3. Mesure de l’efficacité du knockdown par transcription inverse et PCR en temps réel (RT-PCR)
- Extraire l’ARN total de 1 x 107 shLacZ, shTcf4 et shId2 stable knockdown iHSPC cellules à l’aide d’un réactif d’extraction d’ARN commercial et précipiter l’ARN de la couche aqueuse avec de l’isopropanol, puis laver le précipité d’ARN avec de l’éthanol à 75%.
- Dissoudre l’ARN (~ 5 μg) avec 5 μL de H2O traité par DEPC et ajuster la concentration à 1 μg/μL.
- Prélever 1 à 3 μg d’ARN, mélanger avec du H2O traité par DEPC jusqu’à un volume final de 17,4 μL, ajouter 1 μL de DNase I sans RNase 1 unité/μL et incuber pendant 20 min à 37 °C.
REMARQUE: Cette étape consiste à digérer l’ADN génomique dans les échantillons d’ARN. - Ajouter 1 μL d’EDTA de 20 mM aux échantillons d’ARN, incuber à 65 °C pendant 10 min pour inactiver la DNase I, et mettre immédiatement les échantillons d’ARN à 4 °C.
- Ajouter 11,6 μL du mélange réactionnel contenant 1 μL d’amorces oligo(dT) (45 μM), 6 μL de 5x tampon 1er brin, 3 μL de dNTP (2 mM), 0,6 μL d’inhibiteur de la RNase (50 Unité/μL) et 1 μL de transcriptase inverse (200 Unité/μL) aux échantillons d’ARN et incuber à 40 °C pendant 1 h.
- Arrêter la réaction en chauffant à 70 °C pendant 10 min et diluer le mélange réactionnel avec 30 μL de H2O.
- Prendre 2 μL du mélange de réaction RT dilué comme modèle d’ADN et la PCR l’amplifier à l’aide d’amorces contre Tcf4 ou Id2 (voir le tableau 1 pour les conditions du thermocycleur).
REMARQUE : Les séquences d’amorce sont incluses dans la table des matériaux.
4. Différenciation in vitro des lignées cellulaires stables knockdown iHSPC
- Maintenir l’élimination unique stable de LacZ (shLacZ), Tcf4 (shTcf4) ou Id2 (shId2) dans des iHSPC dans un milieu complet contenant 100 ng/mL de FL et 1 μM de β-estradiol.
- Collectez les iHSPC non différenciés shLacZ, shTcf4 et shId2 dans un tube de 15 mL et centrifugez pendant 5 min à 500 x g pour granuler les cellules.
- Jeter le surnageant et ajouter 10 mL de PBS pour laver les cellules. Répétez cette étape deux fois.
- Remettre en suspension et ensemencer les cellules iHSPC à une densité de 2 x 105 cellules/mL dans une plaque de 12 puits dans 1 mL de milieu complet contenant 100 ng/mL FL seulement.
- Ajouter 1 mL de milieu frais complet contenant 100 ng/mL FL trois jours plus tard.
- Analysez les cellules différenciées (shLacZ, shTcf4 et shId2 iHSPC) par cytométrie en flux deux jours plus tard.
5. Analyse cytométrique en flux des DC différenciés
- Recueillir les cellules dans des tubes de 1,5 mL en pipetant les cellules de haut en bas 2-3 fois dans la plaque, et centrifuger pendant 5 min à 500 x g à température ambiante (RT).
REMARQUE: Les DC différenciés in vitro se fixeront légèrement aux plaques. Un pipetage doux aidera à récupérer les DC des plaques. - Jeter le surnageant et remettre les cellules en suspension dans 50 μL de tampon FACS. Ensuite, ajoutez 50 μL de surnageant d’hybridome anti-CD16/32 et incubez pendant 5 à 10 minutes sur de la glace.
REMARQUE: L’anticorps bloquant Fc empêche la liaison non spécifique des anticorps aux récepteurs Fc exprimés sur certaines cellules myéloïdes et cellules B et est disponible dans le commerce auprès de divers fournisseurs. - Ajouter des anticorps conjugués à des colorants fluorescents (0,04 μg pour chaque anticorps) directement dans les cellules et incuber pendant 15 min sur de la glace dans l’obscurité. Les anticorps utilisés sont APC/cy7 anti-souris CD11c, FITC anti-souris CD11b et PE anti-souris B220.
REMARQUE : les pDC sont définis comme CD11c+CD11b-B220+, et les cDC sont définis comme CD11c+CD11b+B220-. - Laver les cellules avec 1 mL de tampon FACS et centrifuger pendant 5 min à 500 x g à RT.
- Remettre en suspension les cellules dans 100 μL de tampon FACS et les analyser par cytométrie en flux.
Résultats
La carte du vecteur lentiviral pLKO.1-Puro est montrée (Figure 1). Après l’administration de lentivirus exprimant l’ARNh contre LacZ (un contrôle non ciblé), Tcf4 et Id2 dans les iHSPC, l’efficacité de knockdown confirmée par RT-qPCR a révélé que l’expression de Tcf4 était réduite dans les iHSPC shTcf4, par rapport aux iHSPC shLacZ (Figure 2A). D’autre part, la diminution de l’expressio...
Discussion
Les vecteurs shRNA à base de lentivirus sont souvent utilisés pour le silençage génique par transduction virale dans les cellules et permettent une intégration stable dans le génome de l’hôte. Cependant, diverses efficacités de transduction dans différents types de cellules doivent être prises en compte, et un certain nombre d’approches ont été adoptées pour surmonter ce problème.
Le polybrène est un polymère polycationique qui peut neutraliser les charges sur la membrane ...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous sommes reconnaissants du soutien technique du Dr Tz-Ling Chen. Nous remercions le National RNAi Core Facility (Academia Sinica, Taïwan) d’avoir fourni le lentivirus shRNA (http://rnai.genmed.sinica.edu.tw). Ce travail a été soutenu par le ministère de la Science et de la Technologie de Taïwan (MOST 108-2320-B-002-037-MY3 et MOST 109-2320-B-002-054-MY3).
matériels
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| APC/Cy7 anti-mouse CD11c Antibody | Biolegend | 117324 | (Clone: N418) |
| FITC anti-mouse/human CD11b Antibody | Biolegend | 101206 | (Clone: M1/70) |
| PE anti-mouse/human B220 Antibody | Biolegend | 103208 | (Clone: RA3-6B2) |
| Cell culture | |||
| 1.5 mL Micro tube | ExtraGene | TUBE-170-C | |
| 12-well tissue culture-treated plate | Falcon | 353043 | |
| Fetal bovine serum (FBS) | Corning | 35-010-CV | |
| RPMI 1640 medium | gibco | 11875-085 | |
| Reagent | |||
| β-estradiol | Sigma-Aldrich | E2758-250MG | |
| β-mercaptoethanol (β-ME) | Sigma-Aldrich | M6250 | |
| FACS buffer | home-made | Formula: 1xPBS+0.5 %FBS+0.1%NaN3 | |
| Flt3 ligand (FL) | home-made | ||
| Polybrene | Sigma-Aldrich | TR-1003-G | |
| Puromycin | Invivogen | ant-pr-1 | |
| TRIsure | BIOLINE | BIO-38032 | |
| shRNA (Taregt sequence/clone ID) | Company | ||
| shId2 (GCTTATGTCGAATGATAGCAA/TRCN0000054390) | The RNAi Consortium (TRC) | ||
| shLacZ (CGCGATCGTAATCACCCGAGT/TRCN0000072224) | The RNAi Consortium (TRC) | ||
| shTcf4 (GCTGAGTGATTTACTGGATTT/TRCN0000012094) | The RNAi Consortium (TRC) | ||
Références
- Steinman, R. M., Witmer, M. D. Lymphoid dendritic cells are potent stimulators of the primary mixed leukocyte reaction in mice. Proceedings of the National Academy of Sciences. 75 (10), 5132-5136 (1978).
- Guilliams, M., et al. Dendritic cells, monocytes and macrophages: a unified nomenclature based on ontogeny. Nature Reviews Immunology. 14 (8), 571-578 (2014).
- Blasius, A. L., Cella, M., Maldonado, J., Takai, T., Colonna, M. Siglec-H is an IPC-specific receptor that modulates type I IFN secretion through DAP12. Blood. 107 (6), 2474-2476 (2006).
- Swiecki, M., Colonna, M. Unraveling the functions of plasmacytoid dendritic cells during viral infections, autoimmunity, and tolerance. Immunology Reviews. 234 (1), 142-162 (2010).
- Liu, Y. -. J. IPC: Professional Type 1 interferon-producing cells and plasmacytoid dendritic cell precursors. Annual Review of Immunology. 23 (1), 275-306 (2005).
- Panda, S. K., Kolbeck, R., Sanjuan, M. A. Plasmacytoid dendritic cells in autoimmunity. Current Opinion in Immunology. 44, 20-25 (2017).
- Durai, V., Murphy, K. M. Functions of murine dendritic cells. Immunity. 45 (4), 719-736 (2016).
- Mashayekhi, M., et al. CD8α(+) dendritic cells are the critical source of interleukin-12 that controls acute infection by Toxoplasma gondii tachyzoites. Immunity. 35 (2), 249-259 (2011).
- Anderson, D. A., Dutertre, C. -. A., Ginhoux, F., Murphy, K. M. Genetic models of human and mouse dendritic cell development and function. Nature Reviews Immunology. 21 (2), 101-115 (2021).
- Plantinga, M., et al. Conventional and monocyte-derived CD11b(+) dendritic cells initiate and maintain T helper 2 cell-mediated immunity to house dust mite allergen. Immunity. 38 (2), 322-335 (2013).
- Persson, E. K., et al. IRF4 transcription-factor-dependent CD103(+)CD11b(+) dendritic cells drive mucosal T helper 17 cell differentiation. Immunity. 38 (5), 958-969 (2013).
- Kumamoto, Y., et al. CD301b+ dermal dendritic cells drive T helper 2 cell-mediated immunity. Immunity. 39 (4), 733-743 (2013).
- Cisse, B., et al. Transcription factor E2-2 is an essential and specific regulator of plasmacytoid dendritic cell development. Cell. 135 (1), 37-48 (2008).
- Grajkowska, L. T., et al. Isoform-specific expression and feedback regulation of E protein TCF4 control dendritic cell lineage specification. Immunity. 46 (1), 65-77 (2017).
- Jackson, J. T., et al. Id2 expression delineates differential checkpoints in the genetic program of CD8α+ and CD103+ dendritic cell lineages. The EMBO Journal. 30 (13), 2690-2704 (2011).
- Suzuki, S., et al. Critical roles of interferon regulatory factor 4 in CD11bhighCD8alpha- dendritic cell development. Proceedings of the National Academy of Science U. S. A. 101 (24), 8981-8986 (2004).
- Rodrigues, P. F., et al. Distinct progenitor lineages contribute to the heterogeneity of plasmacytoid dendritic cells. Nature Immunology. 19 (7), 711-722 (2018).
- Brown, C. C., et al. Transcriptional basis of mouse and human dendritic cell heterogeneity. Cell. 179 (4), 846-863 (2019).
- Redecke, V., et al. Hematopoietic progenitor cell lines with myeloid and lymphoid potential. Nature Methods. 10 (8), 795-803 (2013).
- Abe, A., Miyanohara, A., Friedmann, T. Polybrene increases the efficiency of gene transfer by lipofection. Gene Therapy. 5 (5), 708-711 (1998).
- Sorgi, F. L., Bhattacharya, S., Huang, L. Protamine sulfate enhances lipid-mediated gene transfer. Gene Therapy. 4 (9), 961-968 (1997).
- Kirkling, M. E., et al. Notch signaling facilitates in vitro generation of cross-presenting classical dendritic cells. Cell Reports. 23 (12), 3658-3672 (2018).
- Pearce, E. J., Everts, B. Dendritic cell metabolism. Nature Reviews. Immunology. 15 (1), 18-29 (2015).
- Wculek, S. K., Khouili, S. C., Priego, E., Heras-Murillo, I., Sancho, D. Metabolic Control of Dendritic Cell Functions: Digesting Information. Frontiers in Immunology. 10 (775), (2019).
- Saxton, R. A., Sabatini, D. M. mTOR signaling in growth, metabolism, and disease. Cell. 168 (6), 960-976 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon