È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Studio dello sviluppo delle cellule dendritiche mediante knockdown genico mediato da RNA a forcina corta in una linea cellulare ematopoietica e progenitrice in vitro
In questo articolo
Riepilogo
Qui forniamo un protocollo per lo screening di potenziali fattori di trascrizione coinvolti nello sviluppo di cellule dendritiche (DC) utilizzando la trasduzione lentivirale di shRNA per ottenere linee cellulari di knockdown stabili per la differenziazione DC in vitro .
Abstract
Le cellule dendritiche (DC) sono importanti cellule che presentano l'antigene che collegano le risposte immunitarie innate e adattative. I DC sono eterogenei e possono essere suddivisi in DC convenzionali (cDC) e DC plasmacitoidi (pDC). cDCs è specializzata nella presentazione di antigeni e attivare cellule T naïve. D'altra parte, i pDC possono produrre grandi quantità di interferoni di tipo I (IFN-I) durante l'infezione virale. La specifica delle DC si verifica in una fase iniziale dei progenitori DC nel midollo osseo (BM) ed è definita da una rete di fattori di trascrizione (TF). Ad esempio, i cDC esprimono altamente ID2, mentre i pDC esprimono altamente E2-2. Poiché vengono identificati sempre più sottoinsiemi di DC, c'è un crescente interesse per la comprensione di specifici TF che controllano lo sviluppo DC. Qui, stabiliamo un metodo per lo screening dei TF critici per la differenziazione delle DC in vitro consegnando lentivirus che trasporta RNA a forcina corta (shRNA) in una linea immortalizzata di cellule staminali e progenitrici ematopoietiche (iHSPC). Dopo la selezione e la differenziazione in vitro , il potenziale cDC e pDC delle linee cellulari stabili di knockdown viene analizzato mediante citometria a flusso. Questo approccio fornisce una piattaforma per identificare i geni che potenzialmente governano i destini della DC dai progenitori in vitro.
Introduzione
I DC sono regolatori chiave dell'immunità innata e adattativa1. I DC sono principalmente classificati in due popolazioni funzionalmente distinte, vale a dire pDC e cDC. Inoltre, i cDC comprendono due sottoinsiemi, vale a dire, rispettivamente, cDC di tipo I e di tipo II o cDC1 e cDC22. Le pDC, che esprimono BST2, Siglec-H e livelli intermedi di CD11c nei topi3,4, sono le cellule che possono secernere grandi quantità di IFN-I durante l'infiammazione e l'infezione virale5. Grazie alla loro robusta capacità di produrre IFN-I, si sospetta anche che svolgano un ruolo chiave nella progressione delle malattie autoimmuni, tra cui il lupus eritematoso sistemico (LES)6. I cDC1, definiti dall'espressione superficiale di XCR1, CD8a, CLEC9A e CD103 nei topi7, sono specializzati nell'attivazione e polarizzazione delle cellule T CD8+ citotossiche (CTL) attraverso la presentazione incrociata dell'antigene, avviando così l'immunità di tipo I in risposta a patogeni intracellulari e cancro8,9. D'altra parte, i cDC2, che esprimono CD11b e CD172α (noto anche come Sirpα) sia nell'uomo che nei topi, possono attivare le cellule T CD4 + e promuovere la risposta immunitaria di tipo II contro allergeni e parassiti10, oltre a modulare l'immunità di tipo III a seguito di batteri extracellulari e riconoscimento del microbiota11,12.
La diversificazione delle DC è determinata da un gruppo di TF da cellule staminali e progenitrici ematopoietiche (HSPC) nel BM. E2-2 (codificato da Tcf4) è un regolatore principale per la differenziazione e la funzione dei pDC13,14. Al contrario, l'inibitore del legame del DNA 2 (ID2) guida la specifica cDC e inibisce lo sviluppo di pDC bloccando l'attività della proteina E15. Inoltre, lo sviluppo di cDC1 richiede IRF8 e BATF3, mentre la differenziazione di cDC2 dipende fortemente da IRF416. Lavori recenti hanno esplorato l'eterogeneità dei pDC17 e dei cDC e la loro regolazione trascrizionale18. A causa della complessità della rete DC, vi è una necessità insoddisfatta di stabilire una piattaforma per identificare altri TF che controllano lo sviluppo e la funzionalità dei DC.
Qui, abbiamo usato un iHSPC che è stato generato esprimendo la traslocazione nucleare regolata dagli estrogeni di Hoxb8 nelle cellule BM (note anche come cellule Hoxb8-FL)19. Le iHSPC possono proliferare e rimanere in uno stadio indifferenziato in presenza di β-estradiolo e ligando Flt3 (FL), mentre iniziano a differenziarsi in diversi tipi di DC in presenza di FL al momento del ritiro di β-estradiolo19. Sulla base di questa caratteristica, possiamo abbattere i geni di interesse nella fase progenitrice, seguita dall'esame dell'effetto sulla differenziazione in vitro di pDC e cDC. Pertanto, questo metodo è un potente strumento per scoprire i geni che regolano lo sviluppo e la funzione dei DC.
Protocollo
La gestione del lentivirus viene eseguita secondo il regolamento del Dipartimento di salute e sicurezza ambientale del National Taiwan University College of Medicine.
1. Preparazione di linee cellulari staminali e progenitrici ematopoietiche immortalizzate (iHSPC)
- Mantenere la linea cellulare iHSPC in un mezzo RPMI 1640 completo contenente 100 ng/mL FL e 1 μM β-estradiolo.
- Passare le cellule ad un rapporto di 1:10 ogni 3 giorni.
NOTA: Rendere completo RPMI 1640 medio integrando con 10% di siero bovino fetale (FBS), 5 x 10-5 M β-mercaptoetanolo e 10 μg / mL di gentamicina. FL murino ricombinante è anche disponibile in commercio.
2. Trasduzione lentivirale
- Piastra iHSPC ad una densità di 1 x 105 celle/pozzetto in piastre a 12 pozzetti in 1 mL di mezzo completo contenente 100 ng/mL FL, 1 μM β-estradiolo e 8 μg/mL di polibrene.
NOTA: La concentrazione di polybrene dipende dai tipi di cellule ed è solitamente compresa tra 4-8 μg/mL. - Aggiungere il lentivirus portatore di shRNA in ogni pozzetto ad una molteplicità di infezione (MOI) di 100.
NOTA: Il vettore lentivirale è pLKO.1-Puro con un marcatore di selezione della puromicina (Figura 1). Le sequenze target di shRNA contro LacZ, Tcf4 e Id2, rispettivamente, sono elencate nella Tabella dei materiali. Il MOI è definito dal numero di virioni che vengono aggiunti per cellula durante l'infezione.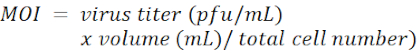
- Ruotare le piastre a 1.100 x g per 90 minuti a 37 °C.
NOTA: l'infezione da spin viene effettuata utilizzando un rotore a benna oscillante. - Incubare la piastra contenente cellule infette per una notte a 37 °C in un'incubatrice.
NOTA: se le cellule sono sensibili al polibrene, aggiornare le cellule con il mezzo completo senza polibrene dopo l'infezione da spin. - Aggiornare le cellule con mezzo completo contenente 100 ng/mL FL e 1 μM β-estradiolo 24 ore dopo l'infezione.
- Aggiungere 6 μg/mL di puromicina al mezzo per selezionare le cellule infette dopo ulteriori 24 ore.
NOTA: Il vettore lentivirale trasdotto di solito richiede 48 ore per esprimere i geni, incluso il gene resistente alla puromicina. Assicurarsi che la concentrazione di puromicina sia ottimizzata per ogni linea cellulare. - Aggiornare il mezzo di selezione contenente 100 ng / mL FL, 1 μM β-estradiolo e 6 μg / mL puromicina ogni 3 giorni e mantenere le cellule per almeno una settimana per espandere le cellule iHSPC trasdotte stabilmente.
NOTA: La selezione di puromicina di solito ha effetto 48 ore dopo e il periodo di selezione dipende dai tipi di cellule.
3. Misurazione dell'efficienza di abbattimento mediante trascrizione inversa e REAL-TIME PCR (RT-PCR)
- Estrarre l'RNA totale da 1 x 107 shLacZ, shTcf4 e shId2 cellule iHSPC stabili di knockdown utilizzando il reagente di estrazione dell'RNA commerciale e precipitare l'RNA dallo strato acquoso con isopropanolo, seguito dal lavaggio del precipitato di RNA con etanolo al 75%.
- Sciogliere l'RNA (~ 5 μg) con 5 μL di H2O trattato con DEPC e regolare la concentrazione a 1 μg/μL.
- Prendere 1-3 μg di RNA, mescolare con H2O trattato con DEPC per un volume finale di 17,4 μL, aggiungere 1 μL di 1 unità/μL di DNasi I priva di RNasi e incubare per 20 minuti a 37 °C.
NOTA: Questo passaggio consiste nel digerire il DNA genomico nei campioni di RNA. - Aggiungere 1 μL di 20 mM EDTA ai campioni di RNA, incubare a 65 °C per 10 minuti per inattivare la DNasi I e mettere immediatamente i campioni di RNA a 4 °C.
- Aggiungere 11,6 μL della miscela di reazione contenente 1 μL di primer oligo (dT) (45 μM), 6 μL di 5x tampone del 1° filamento, 3 μL di dNTP (2 mM), 0,6 μL di inibitore della RNasi (50 Unità/μL) e 1 μL di trascrittasi inversa (200 Unità/μL) ai campioni di RNA e incubare a 40 °C per 1 ora.
- Arrestare la reazione riscaldando a 70 °C per 10 minuti e diluire la miscela di reazione con 30 μL di H2O.
- Prendi 2 μL della miscela di reazione RT diluita come modello di DNA e la PCR la amplifica usando primer contro Tcf4 o Id2 (vedi Tabella 1 per le condizioni del termociclatore).
NOTA: le sequenze di primer sono incluse nella Tabella dei materiali.
4. Differenziazione in vitro delle linee cellulari iHSPC a knockdown stabile
- Mantenere stabile il singolo knockdown di LacZ (shLacZ), Tcf4 (shTcf4) o Id2 (shId2) in iHSPC in mezzo completo contenente 100 ng/ mL di FL e 1 μM β-estradiolo.
- Raccogliere gli iHSPC indifferenziati shLacZ, shTcf4 e shId2 in un tubo da 15 mL e centrifugare per 5 minuti a 500 x g per pellettare le celle.
- Scartare il surnatante e aggiungere 10 ml di PBS per lavare le cellule. Ripetere questo passaggio due volte.
- Risospesciare e seminare le cellule iHSPC ad una densità di 2 x 105 cellule/mL in una piastra a 12 pozzetti in 1 mL di mezzo completo contenente solo 100 ng/mL FL.
- Aggiungere 1 mL di mezzo fresco completo contenente 100 ng/mL FL tre giorni dopo.
- Analizzare le cellule differenziate (shLacZ, shTcf4 e shId2 iHSPC) mediante citometria a flusso due giorni dopo.
5. Analisi citometrica a flusso delle DC differenziate
- Raccogliere le cellule in tubi da 1,5 ml tubando le celle su e giù 2-3 volte nella piastra e centrifugare per 5 minuti a 500 x g a temperatura ambiente (RT).
NOTA: i DC differenziati in vitro si attaccheranno leggermente alle piastre. Il pipettaggio delicato aiuterà a recuperare i DC dalle piastre. - Scartare il surnatante e risospese le cellule in 50 μL di tampone FACS. Quindi, aggiungere 50 μL di surnatante ibridoma anti-CD16/32 e incubare per 5-10 minuti sul ghiaccio.
NOTA: L'anticorpo bloccante Fc impedisce il legame non specifico degli anticorpi ai recettori Fc che si esprimono su alcune cellule mieloidi e cellule B ed è disponibile in commercio attraverso vari fornitori. - Aggiungere anticorpi coniugati con colorante fluorescente (0,04 μg per ciascun anticorpo) direttamente alle cellule e incubare per 15 minuti sul ghiaccio al buio. Gli anticorpi utilizzati sono APC/cy7 anti-mouse CD11c, FITC anti-mouse CD11b e PE anti-mouse B220.
NOTA: i pDC sono definiti come CD11c+CD11b-B220+ e i cDC sono definiti come CD11c+CD11b+B220-. - Lavare le celle con 1 mL di tampone FACS e centrifugare per 5 min a 500 x g a RT.
- Risospendare le cellule in 100 μL di tampone FACS e analizzarle mediante citometria a flusso.
Risultati
Viene mostrata la mappa del vettore lentivirale pLKO.1-Puro (Figura 1). Dopo la somministrazione di lentivirus che esprimono shRNA contro LacZ (un controllo non mirato), Tcf4 e Id2 in iHSPC, l'efficienza di knockdown confermata da RT-qPCR ha rivelato che l'espressione di Tcf4 era ridotta nelle iHSPC shTcf4, rispetto alle iHSPC shLacZ (Figura 2A). D'altra parte, la diminuzione dell'espressione di Id2 ...
Discussione
I vettori shRNA basati su lentivirus sono spesso utilizzati per il silenziamento genico mediante trasduzione virale nelle cellule e consentono un'integrazione stabile nel genoma dell'ospite. Tuttavia, è necessario considerare varie efficienza di trasduzione in diversi tipi di cellule e sono stati adottati numerosi approcci per superare questo problema.
Polybrene è un polimero policanico in grado di neutralizzare le cariche sulla membrana cellulare, migliorando così il legame del virione al...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Siamo grati per il supporto tecnico del Dr. Tz-Ling Chen. Ringraziamo la National RNAi Core Facility (Academia Sinica, Taiwan) per aver fornito shRNA lentivirus (http://rnai.genmed.sinica.edu.tw). Questo lavoro è stato sostenuto dal Ministero della Scienza e della Tecnologia, Taiwan (MOST 108-2320-B-002-037-MY3 e MOST 109-2320-B-002-054-MY3).
Materiali
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| APC/Cy7 anti-mouse CD11c Antibody | Biolegend | 117324 | (Clone: N418) |
| FITC anti-mouse/human CD11b Antibody | Biolegend | 101206 | (Clone: M1/70) |
| PE anti-mouse/human B220 Antibody | Biolegend | 103208 | (Clone: RA3-6B2) |
| Cell culture | |||
| 1.5 mL Micro tube | ExtraGene | TUBE-170-C | |
| 12-well tissue culture-treated plate | Falcon | 353043 | |
| Fetal bovine serum (FBS) | Corning | 35-010-CV | |
| RPMI 1640 medium | gibco | 11875-085 | |
| Reagent | |||
| β-estradiol | Sigma-Aldrich | E2758-250MG | |
| β-mercaptoethanol (β-ME) | Sigma-Aldrich | M6250 | |
| FACS buffer | home-made | Formula: 1xPBS+0.5 %FBS+0.1%NaN3 | |
| Flt3 ligand (FL) | home-made | ||
| Polybrene | Sigma-Aldrich | TR-1003-G | |
| Puromycin | Invivogen | ant-pr-1 | |
| TRIsure | BIOLINE | BIO-38032 | |
| shRNA (Taregt sequence/clone ID) | Company | ||
| shId2 (GCTTATGTCGAATGATAGCAA/TRCN0000054390) | The RNAi Consortium (TRC) | ||
| shLacZ (CGCGATCGTAATCACCCGAGT/TRCN0000072224) | The RNAi Consortium (TRC) | ||
| shTcf4 (GCTGAGTGATTTACTGGATTT/TRCN0000012094) | The RNAi Consortium (TRC) |
Riferimenti
- Steinman, R. M., Witmer, M. D. Lymphoid dendritic cells are potent stimulators of the primary mixed leukocyte reaction in mice. Proceedings of the National Academy of Sciences. 75 (10), 5132-5136 (1978).
- Guilliams, M., et al. Dendritic cells, monocytes and macrophages: a unified nomenclature based on ontogeny. Nature Reviews Immunology. 14 (8), 571-578 (2014).
- Blasius, A. L., Cella, M., Maldonado, J., Takai, T., Colonna, M. Siglec-H is an IPC-specific receptor that modulates type I IFN secretion through DAP12. Blood. 107 (6), 2474-2476 (2006).
- Swiecki, M., Colonna, M. Unraveling the functions of plasmacytoid dendritic cells during viral infections, autoimmunity, and tolerance. Immunology Reviews. 234 (1), 142-162 (2010).
- Liu, Y. -. J. IPC: Professional Type 1 interferon-producing cells and plasmacytoid dendritic cell precursors. Annual Review of Immunology. 23 (1), 275-306 (2005).
- Panda, S. K., Kolbeck, R., Sanjuan, M. A. Plasmacytoid dendritic cells in autoimmunity. Current Opinion in Immunology. 44, 20-25 (2017).
- Durai, V., Murphy, K. M. Functions of murine dendritic cells. Immunity. 45 (4), 719-736 (2016).
- Mashayekhi, M., et al. CD8α(+) dendritic cells are the critical source of interleukin-12 that controls acute infection by Toxoplasma gondii tachyzoites. Immunity. 35 (2), 249-259 (2011).
- Anderson, D. A., Dutertre, C. -. A., Ginhoux, F., Murphy, K. M. Genetic models of human and mouse dendritic cell development and function. Nature Reviews Immunology. 21 (2), 101-115 (2021).
- Plantinga, M., et al. Conventional and monocyte-derived CD11b(+) dendritic cells initiate and maintain T helper 2 cell-mediated immunity to house dust mite allergen. Immunity. 38 (2), 322-335 (2013).
- Persson, E. K., et al. IRF4 transcription-factor-dependent CD103(+)CD11b(+) dendritic cells drive mucosal T helper 17 cell differentiation. Immunity. 38 (5), 958-969 (2013).
- Kumamoto, Y., et al. CD301b+ dermal dendritic cells drive T helper 2 cell-mediated immunity. Immunity. 39 (4), 733-743 (2013).
- Cisse, B., et al. Transcription factor E2-2 is an essential and specific regulator of plasmacytoid dendritic cell development. Cell. 135 (1), 37-48 (2008).
- Grajkowska, L. T., et al. Isoform-specific expression and feedback regulation of E protein TCF4 control dendritic cell lineage specification. Immunity. 46 (1), 65-77 (2017).
- Jackson, J. T., et al. Id2 expression delineates differential checkpoints in the genetic program of CD8α+ and CD103+ dendritic cell lineages. The EMBO Journal. 30 (13), 2690-2704 (2011).
- Suzuki, S., et al. Critical roles of interferon regulatory factor 4 in CD11bhighCD8alpha- dendritic cell development. Proceedings of the National Academy of Science U. S. A. 101 (24), 8981-8986 (2004).
- Rodrigues, P. F., et al. Distinct progenitor lineages contribute to the heterogeneity of plasmacytoid dendritic cells. Nature Immunology. 19 (7), 711-722 (2018).
- Brown, C. C., et al. Transcriptional basis of mouse and human dendritic cell heterogeneity. Cell. 179 (4), 846-863 (2019).
- Redecke, V., et al. Hematopoietic progenitor cell lines with myeloid and lymphoid potential. Nature Methods. 10 (8), 795-803 (2013).
- Abe, A., Miyanohara, A., Friedmann, T. Polybrene increases the efficiency of gene transfer by lipofection. Gene Therapy. 5 (5), 708-711 (1998).
- Sorgi, F. L., Bhattacharya, S., Huang, L. Protamine sulfate enhances lipid-mediated gene transfer. Gene Therapy. 4 (9), 961-968 (1997).
- Kirkling, M. E., et al. Notch signaling facilitates in vitro generation of cross-presenting classical dendritic cells. Cell Reports. 23 (12), 3658-3672 (2018).
- Pearce, E. J., Everts, B. Dendritic cell metabolism. Nature Reviews. Immunology. 15 (1), 18-29 (2015).
- Wculek, S. K., Khouili, S. C., Priego, E., Heras-Murillo, I., Sancho, D. Metabolic Control of Dendritic Cell Functions: Digesting Information. Frontiers in Immunology. 10 (775), (2019).
- Saxton, R. A., Sabatini, D. M. mTOR signaling in growth, metabolism, and disease. Cell. 168 (6), 960-976 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon