このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
逆転写ループを介した等温増幅によるSARS-CoV-2ウイルスの検出
要約
ここでは、逆転写ループ媒介等温増幅(RT-LAMP)によりヒトサンプル中のSARS-CoV-2ウイルスを検出する方法を標準化し、実装するための完全なプロトコルを提供します。この方法は60分以内に完了し、低コストで安価な機器を使用して、あらゆる検査室やポイントオブケアに適応できます。
要約
重症急性呼吸器症候群コロナウイルス2(SARS-CoV-2)ウイルスは、人間の健康に劇的な影響を与えています。感染によって多くの人が亡くなるため、現代社会にとって脅威であり続けています。この疾患は、ゴールドスタンダードのリアルタイムポリメラーゼ連鎖反応(RT-PCR)などの血清学的および分子学的検査を使用して診断されます。後者には、特殊なインフラストラクチャ、高価な機器、および訓練を受けた人員が必要になるため、いくつかの欠点があります。ここでは、ヒトサンプルの逆転写ループ媒介等温増幅法(RT-LAMP)を使用してSARS-CoV-2ウイルスを検出するために必要な手順を概説したプロトコルを示します。このプロトコールには、 プライマーin silicoの設計、試薬の調製、増幅、および可視化の指示が含まれています。標準化されると、この方法は簡単に実装でき、60分以内に低コストで安価な機器を使用して、あらゆるラボやポイントオブケアに適応させることができます。さまざまな病原体の検出に適応できます。したがって、現場や保健センターでタイムリーな疫学監視を実施するために使用できる可能性があります。
概要
重症急性呼吸器症候群コロナウイルス2(SARS-CoV-2)は、コロナウイルス病2019(COVID-19)を引き起こします。世界保健機関(WHO)は、2020年1月30日に国際的に懸念される公衆衛生上の緊急事態を宣言し、2020年3月11日にパンデミックを宣言しました。パンデミックにより、この記事が書かれた時点で7億6,000万人以上の症例と687万人の死亡者が出ています1。
このウイルスの影響により、感染症の検出と制御を改善するために、より優れた、より正確で、より速く、より広く利用可能な監視ツールの必要性が浮き彫りになりました2,3。パンデミック時には、SARS-CoV-2の診断検査は核酸、抗体、タンパク質の検出が基本でしたが、核酸のRT-PCR検出はゴールドスタンダードです4。ただし、RT-PCRにはいくつかの制限があります。これには、分子生物学の訓練を受けた特殊な機器、インフラストラクチャ、および人員が必要であり、その適用は専門の研究所に限定されています。さらに、それは時間がかかり(4〜6時間)、標本を実験室に輸送する時間を含まない、これには5日かかることがあります。これらの制約により、効率的なサンプル処理や、緊急時対応計画や疫学管理に必要な情報の取得が妨げられます。
逆転写ループ媒介等温増幅法(RT-LAMP)は、RT-PCRに比べていくつかの利点があり、特にリソースに制約のある環境で、将来のポイントオブケア診断検査(POCT)を設計するための魅力的な戦略となっています6。まず、DNAやRNAなど、標的配列の6〜8つの領域を認識する4〜6つのプライマーを使用するため、非常に特異的です7,8。次に、一定の温度で動作するため、増幅を生成するためにリアルタイムのサーマルサイクラーなどの高度な機器を必要とせず、高度な訓練を受けた人員を操作する必要もありません。第三に、反応時間が非常に短く(~60分)、あまり専門化されていない試薬が使用されるため、費用対効果の高いツールになります6。上記とCOVID-19のパンデミックによって引き起こされた健康上の緊急事態を考えると、この技術は、迅速、安価、かつ簡単に実施できる代替診断方法と見なすことができる9。
この記事では、サーモサイクラーとウォーターバスを使用した比色法によりSARS-CoV-2を検出するためのRT-LAMPを標準化および実装するためのプロトコルについて説明します(図1)。重要なポイント、その制限、およびそれらを進めるための代替案について説明します。
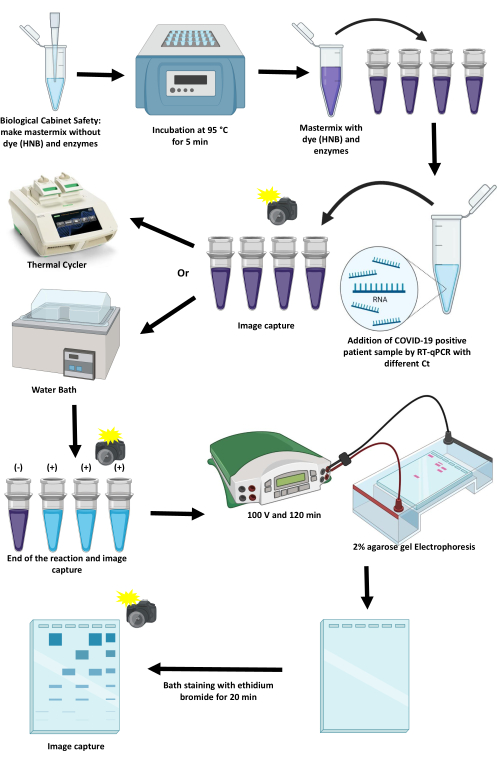
図1:RT-LAMP技術を使用してSARS-CoV-2を増幅するためのプロトコルのスキーム。 この 図の拡大版を表示するには、ここをクリックしてください。
プロトコル
使用されたサンプルは、Fundación Valle del Lili大学病院の臨床検査室から提供され、RT-qPCR技術を使用してCOVID-19の検査で陽性となった患者の精製されたRNAに対応しました。すべての患者が研究にインフォームドコンセントを提供し、この研究は、Fundación Valle del Lili大学病院のヒト研究の生命倫理委員会によって承認されました。
1. RT-LAMPプライマーの設計と調製
注:LAMPプライマーは、New England BioLabs(NEB)LAMP、Primer Explorer、LAMPアッセイ多用途分析(LAVA)など、さまざまなプラットフォームで使用できます。ただし、このプロトコルでは、NEB LAMPツールが使用されました。プライマー設計は、NextStrain database10から取得した SARS-CoV-2 ゲノムを使用して行うことができます。 表1 に、このプロトコールで使用されるプライマーセットを示します。
- LAMPのプライマー設計
- ウイルスのゲノム配列を取得します。
- 配列アラインメントを実行して、コンセンサス配列を取得します。
- NEB LAMPプライマー設計ツールプラットフォーム11に移動し、クイックガイドの指示に従います。このツールは、プライマーエクスプローラーV5と同じ結果を生成しますが、出力ははるかにユーザーフレンドリーです。プライマーエクスプローラーのユーザーマニュアルをプライマーデザインのガイドとして使用してください。
- プライマーセットの熱力学的評価
- ツールPrimer-Dimer12 を使用して、取得したプライマーの熱力学分析を実行します。
- プライマー配列をツールに入れます。次に、 オプション「マルチプレックス解析およびダイマー構造レポート」を選択します。
- ΔGが-5以上のプライマーセットを選択します。
- 設計されたプライマーの特異性評価
- BLAST13 のヌクレオチドコレクション(nt/nt)データベースを使用して、各プライマーを解析します。
- 最初のBLAST解析を実行するには、Refseq_rnaデータベースを選択し、Orthocoronavirinae亜科に属する属のグループで検索をフィルタリングします。それらは、アルファコロナウイルス(taxid:693996)、ガンマコロナウイルス(taxid:694013)、およびデルタコロナウイルス(taxid:1159901)です。さらに、H1N1亜型 (taxid:114727)、インフルエンザA型ウイルス (taxid:11320)、インフルエンザB型ウイルス (taxid:11520) として共循環している他のウイルスに対して配列を評価します。
- 2回目のBLAST解析を行うには、ベータコロナウイルスGenBankを選択し、コロナウイルス科(taxid:11118)とSARS(taxid:694009)で検索をフィルタリングします。これらのグループには、コウモリ、ベータコロナウイルス(taxid:694002)で見つかったゲノムを含む、特定されたすべてのSARSコロナウイルスゲノムの配列が含まれています。
- このプロトコルでは、プライマーが標的ゲノムであるSARS-CoV-2以外のゲノムと一致していないことを確認してください。
- プライマー調製
- 凍結乾燥プライマーを含むバイアルを微量遠心分離機(10,000 x g、室温[RT]で1分間)で回転させ、チューブ開封時の損失を防ぎます。
- 凍結乾燥粉末を0.1%ジエチルピロカーボネート(DEPC)水またはヌクレアーゼフリー水に再水和し、最終濃度100μM(表2)まで再水和し、ピペッティングで上下させて十分に溶解します。次に、マイクロ遠心分離機で最高速度(10,000 x g、室温で1分)で回転し、チューブの底にあるすべてのプライマー溶液を収集します。
- 表2に示すように、フォワードインナープライマー(FIP)、バックワードインナープライマー(BIP)、フォワードアウタープライマー(F3)、バックワードアウタープライマー(B3)、ループバックワード(LB)、ループフォワード(LF)プライマーを使用して、バイオセーフティキャビネットの下に10xプライマーミックスを調製します。損失を防ぐには、マイクロ遠心分離機で急速スピン(10,000 x g、RTで1分)を行う前に、プライマー溶液をピペットでピペットまたは穏やかにボルテックスします。
- 10倍プライマーミックスは、長期保存のために-20°Cで保存してください。ただし、凍結融解サイクルが多すぎるのを避けるために、複数のサンプルに関係なく、最大5回の実験に十分な量を準備してください。
注:プライマーミックスの量を少なくする必要がある場合は、新しい容量を計算して値を調整します(表2)。また、RT-LAMP反応にはループプライマーが不要なため、RdRpセットとRdRp/HelセットにはLFプライマーは含まれていません。そのため、LFプライマーの容量をヌクレアーゼフリー水または0.1%DEPC水に交換してください。
2. RT-LAMP反応
- 製造元の指示に従って層流キャビネットの電源を入れ、空気の流れが安定するまで少なくとも3分間待ちます。
- 空気の流れが安定したら、無菌技術を使用してキャビネットの内面を清掃し、消毒します。これを達成するには、次の消毒剤をこの順序で使用します:1000 ppm第四級アンモニウム(塩化ベンザルコニウム)、2%次亜塩素酸、3%過酸化水素、および70%エタノール。
注:この場合、無菌技術では、消毒剤を塗布し、事前に洗浄した表面を越えずにキャビン内から外側にナプキンで除去する必要があります。 - 手順2.2の消毒剤を使用して、キャビンに入る材料を同じ順序で清掃します。
注:マイクロピペット、フィルターチップボックス、1.5 mLおよび0.6 mLチューブ付きフラスコ、0.2 mL PCRチューブ、ラック、および400 mLビーカーをキャビネットに持ち込む必要があります。 - ナプキンとニトリル手袋を機内にお持ちください。その後、キャビネットの電源を切り、紫外線(UV)に15分間さらします。
注意: 長時間の放射線被曝による組織やDNAの損傷を避けるために、手順2.4で設定した時間が経過するまで紫外線を避けてください。
注意 プロトコルを開始する前に 図2 に示す組み立てを実行し、手順2.4を完了した後にウォーターバスを開始します。金属製の容器をほぼ縁まで飲料水で満たし、鉄製の実験室用加熱プレートの温度を90°Cに設定することが重要です。これにより、システム内の温度が~66.3°Cになり、水銀温度計で監視されます。 - 照射期間が終了したら、キャビネットを再起動し、手順1.1の推奨事項に従ってください。
- 試薬(表3、表4、および 表5)を氷で満たされたクーラーまたは小さなポリスチレン冷蔵庫に入れます。70%エタノールで洗浄した後、容器をキャビネットに入れます。
- 0.6 mLの微量遠心チューブに、増幅する遺伝子のLAMP混合物(RdRp、N-A、およびRdRp/Hel)を調製し、次の成分のみを追加します:10x Buffer、MgSO4、dNTPs、1x Primers Mix、ヌクレアーゼフリー水または0.1% DEPC水。ピペッティングでよく混ぜて均質化します。
注意: キャビネット内での取り扱いや動作が不適切なため、試薬が汚染されるリスクが高くなります。この問題を軽減するには、次のルールに従う必要があります:(i)滅菌チップとフィルターチップを使用します。(ii)試薬ごとに1つのチップを使用します。(iii)層流を乱さないようにゆっくりと慎重に移動します。(iv)秩序を保ち、最小限の材料を使用します。(v)異なる手袋を使用して混合物を調製し、遺伝物質を添加する。
注:温度変化がそれらを変性させ、ポリメラーゼ活性を変化させる可能性があるため、すべての試薬、特に酵素を氷上に保管してください。 - キャップを閉めた状態で0.6 mLチューブを加熱ブロックに入れ、95°Cで5分間インキュベートします。
注:LAMPミックス調製を開始する前に、キャビネットの外側にある1.5〜2.0 mLチューブの加熱ブロックを少なくとも30分間オンにし、水銀またはアルコール温度計で温度(95°C)を監視します。 - インキュベーションが完了したら、チューブを氷入りポリスチレンクーラーに5分間入れます。
- チューブを層流キャビネットに戻し、酵素のDNAポリメラーゼ(Bst 3.0)、逆転写酵素、およびハイフィデリティDNAポリメラーゼ(表3、表4、および 表5)を添加してLAMPミックス調製を完了します。比色検出を使用する場合は、染料ヒドロキシナフトールブルー(HNB)を追加します。
- これらの試薬を添加した後、LAMP試薬をピペッティングして非常によく混合し、酵素と色素を可溶化します。
- 各PCRチューブに22.0μLの混合物を充填し、泡を作らないように注意してください。次に、3.0 μL の 0.1% DEPC 水またはヌクレアーゼフリー水をネガティブコントロールまたはチューブノーテンプレートコントロール (NTC) に添加し、残りのチューブ (遺伝物質) を添加用に置いておきます。
注:サンプルが追加されるまで、PCRチューブを氷で満たされたクーラーに入れておき、 Bst 3.0酵素の活性化や反応の早期開始を避けてください。 - キャビネットからすべての材料を取り出し、70%エタノールを使用して表面を清掃します。その後、製造元の指示に従って電源を切ります。
- 別の領域で、サンプル3 μLを各PCRチューブに加え、完全に均質化します。これを行うには、20μLのマイクロピペットとフィルターチップを使用します。
注意:遺伝物質の添加に使用されるマイクロピペットは、この目的にのみ使用する必要があり、混合物の調製には使用できません。このようにして、試薬の汚染が回避されます。さらに、RNA分解の可能性を減らすために、RNAサンプルは常に氷の上に置いてください。サンプルの追加には、使い捨てガウン、キャップ、N95マスク、レギンス、ラボ用ゴーグル、ニトリル手袋などの個人用保護具(PPE)を使用してください。 - 比色反応を行う前に、高品質のカメラでPCRチューブの写真を撮ります。HNBの開始色は紫です。
- 次のシステムまたは機器で反応を実行します:(i)サーマルサイクラーおよび(ii)ウォーターバス。
- サーマルサイクラー:チューブを反応ブロックに堆積し、サーモプロファイル( 表6を参照)を装置にセットアップします。
- ウォーターバス:チューブを円形の容器に入れ、チューブが出ないようによく調整します。その後、容器を表2にリストされている温度のウォーターバス(図6A、B)に入れます。
- ウォーターバスの場合、チューブがシステム内に入ったら、タイマーを60分間開始します(表6)。
- 反応時間後にサーマルサイクラーまたはウォーターバスからチューブを取り外し、電気泳動運転の場合は4°Cで、使用時までは-20°Cで保管します。
- 比色反応を行った場合は、高品質のカメラを使用してPCRチューブの写真を撮ります。HNBの最終的な色はスカイブルーです。
3. アガロースゲル中の増幅産物の解析
注:これらのステップは、標準化ステップ中の性能に対する比色反応または制御に対する追加のチェックとして推奨されます。これは、この技術がこれらのテストを行うラボに大きな汚染リスクをもたらす可能性があるためです。
- エッジラバーが壁に接触するようにベッドを電気泳動チャンバー内に置き、アガロース(内部チャンバー)を追加するための密閉スペースを作成します(図3A、B)。
- ステップ3.1を完了した後、500 mLビーカーで必要な量のアガロースを秤量し、2%ゲルを得ます。その後、必要な量の0.5倍トリス酢酸EDTA(TAE)バッファーとマイクロ波を1〜2分間追加します。
注:アガロースは、オーブンから取り出したときに半透明で塊がないときに完全に溶けます。これが確認されないと、ゲル化が不十分な領域が残り、電気泳動の実行と増幅生成物の視覚化が変わる可能性があります。 - ビーカーをオーブンから取り出し、ステップ3.1で作成した内部チャンバーにアガロースを注ぎます(図3C)。その後、気泡がないことを確認し、気泡がある場合はマイクロピペットチップを使用して気泡を取り除きます。
- コームを配置してウェルを形成し、アガロースを室温(RT)で約30分間ゲル化します。
- この時間の後、0.5x TAEバッファーを5 mL追加して、コームとゲルを含むベッドの取り外しを容易にします。次に、ウェルがアノードにくるようにゲルを配置します(図3D)。
- 電気泳動チャンバーに0.5x TAEバッファーをメーカー指定の容量まで充填し、電極がバッファーに接触していることを確認します。
- ゲルの最初のウェルに3 μLの分子量マーカーを加え、9 μLのNTCと各サンプルを次のウェルに加えます。これらは、7 μL の増幅生成物と 3 μL のローディングバッファーを組み合わせて作成します。次に、この混合物の9μLをゲルのウェルにロードします。
- 電気泳動チャンバーを蓋で覆い、ケーブルをカラーパターンで電源ポートに接続します。電源を次のパラメータに設定します:100 Vおよび一定 amp120分間。
- 電気泳動ランが完了したら、ゲルを染色液(臭化エチジウム)の入った容器に入れ、30分間インキュベートします。
- インキュベーション後、ゲルを染色液から取り出し、ジップロックバッグに入れます。これにより、アンプリコンの可視化に使用される機器の汚染を防ぐことができます。
- トランスイルミネーターやAmersham Imager 600のようなイメージャーでゲルを可視化します。
結果
プロトコールの実施は、上記のプロトコールに従って各標的遺伝子のプライマーのセットを設計することから始まります。2020年6月、NextStrainデータベースから5,000のSARS-CoV-2ゲノムが取得され、コロンビアのゲノムの代表性は10%でした。これらの配列は、プライマー設計プロセスで使用されたコンセンサス配列を得るためにアラインメントされました。 表1 に、プライマーRdRp/Helと...
ディスカッション
RT-LAMPは、分子診断を実行するための補完的な方法論と見なされていますが、プロトコルを標準化して実装する際に考慮しなければならないいくつかの制限と重要なステップもあります。
SARS-CoV-2の検出のためのLAMP標準化では、マスターミックスの次のパラメーターと成分が評価されました:(a)プライマーのアライメントの濃度と温度。(b)酵素の濃度。(c)マグネシウム濃度?...
開示事項
ナタリア・カンピージョ・ペドロサは、BioDx: Diagnóstico y Soluciones Biotecnológicas S.A.S.のCEOです。残りの著者は、利益相反を宣言しません。
謝辞
この研究は、コロンビアのSistema General de Regalías(助成金番号BPIN 2020000100092)とUniversidad Icesi - Convocatoria Interna(助成金番号CA0413119)によって資金提供されました。MFVTは、ロスアンデス大学の助教授基金からも資金提供を受けました。資金提供機関は、原稿の設計、活動の実行、データ収集、データ分析、および準備には参加していません。SARS-CoV-2サンプルからのウイルスRNAを提供してくださったUniversity Hospital Fundación Valle del Liliと、原稿へのコメントを提供してくださったAlvaro Barrera-Ocampo博士に感謝します。
資料
| Name | Company | Catalog Number | Comments |
| 1 kb DNA Ladder | SOLIS BIODYNE | 07-12-00050 | Store at -20 °C |
| 50x TAE Electrophoresis Buffer | ThermoScientific | B49 | Store at roome temperature |
| Accuris High Fidelity Polymerase | ACCURIS LIFE SCIENCE REAGENTS | PR1000-HF-200 | It can be used in case Q5 High-Fidelity DNA polymerase cannot be purchased. For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| Agarose | PanReacAppliChem | A8963,0100 | N/A |
| Bst 3.0 DNA Polymerase 8000 IU/mL | New England BioLabs | M0374S/M0374L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| Deoxynucleotide (dNTP) Solution Set | New England BioLabs | N0446S | Store at -20 °C |
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220-25G | Handle it with caution under an extraction cabinet |
| GeneRuler 100 bp Plus DNA Ladder, ready-to-use | ThermoScientific | SM0322 | Store at -20 °C |
| Hydroxy naphthol blue disodium salt | Santa Cruz Biotechnology | sc-215156B | N/A |
| Q5 High-Fidelity DNA polymerase 2000 IU/mL | New England BioLabs | M0491S/M0491L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| WarmStart RTx Reverse Transcriptase 15000 IU/mL | New England BioLabs | M0380S/M0380L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
参考文献
- World Health Organization. . Who coronavirus (COVID-19) dashboard (no date). , (2023).
- Ibrahim, N. K. Epidemiologic surveillance for controlling Covid-19 pandemic: types, challenges and implications. Journal of Infection and Public Health. 13 (11), 1630-1638 (2020).
- Rojas-Gallardo, D. M., et al. COVID-19 in Latin America: Contrasting phylodynamic inference with epidemiological surveillance. (Molecular epidemiology of COVID-19 in Latin America). medRxiv. , (2020).
- Liu, R., et al. Positive rate of RT-PCR detection of SARS-CoV-2 infection in 4880 cases from one hospital in Wuhan, China, from Jan to Feb 2020. Clinica Chimica Acta. 505, 172-175 (2020).
- Kevadiya, B. D., et al. Diagnostics for SARS-CoV-2 infections. Nature Materials. 20 (5), 593-605 (2021).
- Tomita, N., Mori, Y., Kanda, H., Notomi, T. Loop-mediated isothermal amplification (LAMP) of gene sequences and simple visual detection of products. Nature Protocols. 3 (5), 877-882 (2008).
- Li, Y., Fan, P., Zhou, S., Zhang, L. Loop-mediated isothermal amplification (LAMP): A novel rapid detection platform for pathogens. Microbial Pathogenesis. 107, 54-61 (2017).
- Notomi, T., Mori, Y., Tomita, N., Kanda, H. Loop-mediated isothermal amplification (LAMP): principle, features, and future prospects. Journal of Microbiology. 53 (1), 1-5 (2015).
- Augustine, R., et al. Loop-mediated isothermal amplification (LAMP): A rapid, sensitive, specific, and cost-effective point-of-care test for coronaviruses in the context of COVID-19 pandemic. Biology (Basel). 9 (8), 182 (2020).
- . Nextstrain Available from: https://nextstrain.org/ (2023)
- . Neb Lamp, NEB LAMP Available from: https://lamp.neb.com/ (2023)
- . Blast: Basic local alignment search tool (no date) Available from: https://blast.ncbi.nlm.nih.gov/ (2023)
- Zhang, Y., et al. Rapid molecular detection of SARS-CoV-2 (COVID-19) virus RNA using colorimetric LAMP. medRxiv. , (2020).
- Lu, R., et al. Development of a novel reverse transcription loop-mediated isothermal amplification method for rapid detection of SARS-CoV-2. Virologica Sinica. 35 (3), 344-347 (2020).
- Najafov, A., Hoxhaj, G. . PCR Guru. , (2017).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. Biotechniques. 69 (3), 178-185 (2020).
- Ramírez-Chavarría, R. G., et al. Automatic analysis of isothermal amplification via impedance time-constant-domain spectroscopy: A SARS-CoV-2 case study. Chemosensors. 11 (4), 230 (2023).
- Haque, M. F. U., et al. A novel RdRp-based colorimetric RT-LAMP assay for rapid and sensitive detection of SARS-CoV-2 in clinical and sewage samples from Pakistan. Virus Research. 302, 198484 (2021).
- Donia, A., et al. Integration of RT-LAMP and microfluidic technology for detection of SARS-CoV-2 in wastewater as an advanced point-of-care platform. Food and Environmental Virology. 14, 364-373 (2022).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved