É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Detecção do vírus SARS-CoV-2 por amplificação isotérmica mediada por loop de transcrição reversa
Neste Artigo
Resumo
Aqui, fornecemos um protocolo completo para padronizar e implementar o método de detecção do vírus SARS-CoV-2 em amostras humanas por amplificação isotérmica mediada por loop de transcrição reversa (RT-LAMP). Este método, feito em 60 min, pode ser adaptado a qualquer laboratório ou ponto de atendimento a baixo custo e usando equipamentos baratos.
Resumo
O vírus coronavírus 2 da síndrome respiratória aguda grave (SARS-CoV-2) afetou drasticamente a saúde humana. Continua a ser uma ameaça para a sociedade moderna porque muitas pessoas morrem como resultado de uma infecção. A doença é diagnosticada por meio de testes sorológicos e moleculares, como a reação em cadeia da polimerase em tempo real padrão-ouro (RT-PCR). Este último tem várias desvantagens porque requer infraestrutura especializada, equipamentos caros e pessoal treinado. Aqui, apresentamos um protocolo descrevendo as etapas necessárias para detectar o vírus SARS-CoV-2 usando amplificação isotérmica mediada por loop de transcrição reversa (RT-LAMP) em amostras humanas. O protocolo inclui instruções para projetar primers in silico, preparar reagentes, amplificação e visualização. Uma vez padronizado, esse método pode ser facilmente implementado e adaptado a qualquer laboratório ou ponto de atendimento em 60 minutos a baixo custo e usando equipamentos baratos. É adaptável à detecção de diferentes patógenos. Assim, pode potencialmente ser usado no campo e nos centros de saúde para realizar vigilância epidemiológica oportuna.
Introdução
O coronavírus 2 da síndrome respiratória aguda grave (SARS-CoV-2) causa a doença do coronavírus 2019 (COVID-19). A Organização Mundial da Saúde declarou uma emergência de saúde pública de interesse internacional em 30 de janeiro de 2020 e uma pandemia em 11 de março de 2020. A pandemia resultou em mais de 760 milhões de casos e 6,87 milhões de mortes na data em que este artigo foi escrito1.
O impacto desse vírus destacou a necessidade de ferramentas de vigilância melhores, mais precisas, mais rápidas e mais amplamente disponíveis para melhorar a detecção e o controle de doenças infecciosas 2,3. Durante a pandemia, os testes diagnósticos de SARS-CoV-2 foram baseados na detecção de ácido nucleico, anticorpos e proteínas, mas a detecção de ácido nucleico por RT-PCR é o padrão-ouro4. No entanto, a RT-PCR tem algumas limitações; Requer equipamentos especializados, infraestrutura e pessoal treinado em biologia molecular, limitando sua aplicação a laboratórios especializados. Além disso, é demorado (4-6 h), não incluindo o tempo de transporte das amostras para o laboratório, que pode levardias 5. Essas restrições impedem o processamento eficiente da amostra e a obtenção das informações necessárias para o planejamento de contingência e gerenciamento epidemiológico.
A amplificação isotérmica mediada por loop de transcrição reversa (RT-LAMP) tem várias vantagens sobre o RT-PCR, tornando-se uma estratégia atraente para projetar futuros testes diagnósticos no local de atendimento (POCT), particularmente em ambientes com recursos limitados6. Primeiro, é muito específico porque usa entre quatro e seis primers que reconhecem de seis a oito áreas na sequência alvo, seja DNA ou RNA 7,8. Em segundo lugar, por operar a uma temperatura constante, não requer equipamentos sofisticados, como termocicladores em tempo real, para gerar a amplificação, nem requer pessoal altamente treinado para operá-la. Em terceiro lugar, o tempo de reação é muito curto (~ 60 min) e reagentes não muito especializados são empregados, o que o torna uma ferramenta econômica6. Diante do exposto e da emergência sanitária causada pela pandemia de COVID-19, essa técnica pode ser vista como um método diagnóstico alternativo, rápido, barato e simples de implementar em qualquer laboratório de pesquisa9.
O protocolo de padronização e implementação de um RT-LAMP para detecção de SARS-CoV-2 por métodos colorimétricos usando um termociclador e um banho-maria é descrito neste artigo (Figura 1). Discutem-se os pontos críticos, suas limitações e alternativas para avançá-los.
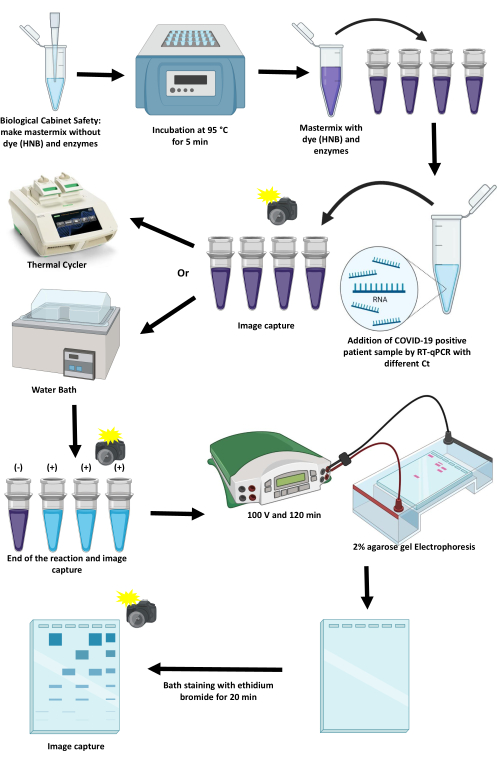
Figura 1: Esquema do protocolo de amplificação do SARS-CoV-2 usando a técnica RT-LAMP. Clique aqui para ver uma versão maior desta figura.
Protocolo
As amostras utilizadas foram fornecidas pelo laboratório clínico do Hospital Universitário Fundación Valle del Lili e corresponderam ao RNA purificado de pacientes que testaram positivo para COVID-19 usando a técnica de RT-qPCR. Todos os pacientes assinaram o termo de consentimento livre e esclarecido para a pesquisa, e este estudo foi aprovado pelo comitê de bioética para estudos em seres humanos do Hospital Universitário Fundación Valle del Lili.
1. Projeto e preparação do primer RT-LAMP
NOTA: Os primers LAMP podem ser usados com uma variedade de plataformas, incluindo LAMP da New England BioLabs (NEB), Primer Explorer e análise versátil de ensaio LAMP (LAVA). No entanto, para este protocolo, foi utilizada a ferramenta NEB LAMP. O design do primer pode ser feito usando genomas SARS-CoV-2 obtidos do banco de dados NextStrain10. A Tabela 1 mostra o conjunto de primers utilizado neste protocolo.
- Design de primer para LAMP
- Obtenha sequências do genoma viral.
- Execute alinhamentos de sequência para obter a sequência de consenso.
- Navegue até a plataforma de ferramentas de design de primer NEB LAMP11 e siga as instruções no guia rápido. Esta ferramenta produz os mesmos resultados que o primer explorer V5, mas é muito mais fácil de usar em sua saída. Use os manuais do usuário do primer explorer como um guia para o design do primer.
- Avaliação termodinâmica do conjunto de primers
- Use a ferramenta Primer-Dimer12 para realizar uma análise termodinâmica nos primers obtidos.
- Coloque as sequências de primer na ferramenta. Em seguida, selecione a opção Análise Multiplex e Relatório de Estrutura de Dímero.
- Selecione os conjuntos de primers que tenham ΔG não inferior a -5.
- Avaliação da especificidade dos primers projetados
- Use o banco de dados de coleta de nucleotídeos (nt/nt) no BLAST13 para analisar cada primer.
- Para realizar a primeira análise BLAST, selecione a Base de Dados Refseq_rna e filtre a busca com o grupo de gêneros que pertencem à subfamília Orthocoronavirinae. Eles são Alphacoronavirus (taxid:693996), Gammacoronavirus (taxid:694013) e Deltacoronavirus (taxid:1159901). Além disso, avalie a sequência contra outros vírus que estão co-circulando como subtipo H1N1 (taxid:114727), vírus Influenza A (taxid:11320) e vírus Influenza B (taxid:11520).
- Para realizar a segunda análise BLAST, selecione o Betacoronavirus GenBank e filtre a pesquisa com Coronaviridae (taxid:11118) e SARS (taxid:694009). Esses grupos contêm sequências de todos os genomas identificados do SARS Coronavirus, incluindo genomas encontrados em morcegos, Betacoronavirus (taxid:694002).
- Para este protocolo, certifique-se de que os primers não estejam alinhados com genomas diferentes do genoma alvo, SARS-CoV-2.
- Preparação de primer
- Gire os frascos contendo os primers iofilizados com uma microcentrífuga (10.000 x g, 1 min à temperatura ambiente [RT]) para evitar perdas durante a abertura do tubo.
- Reidrate o pó iofilizado em água com 0,1% de dietilpirocarbonato (DEPC) ou água livre de nuclease até uma concentração final de 100 μM (Tabela 2) e dissolva completamente pipetando para cima e para baixo. Em seguida, gire na velocidade máxima (10.000 x g, 1 min em RT) em uma microcentrífuga para coletar todas as soluções de primer no fundo do tubo.
- Prepare a mistura de primer 10x em um gabinete de biossegurança com o primer interno dianteiro (FIP), primer interno reverso (BIP), primer externo dianteiro (F3), primer externo reverso (B3), loop backward (LB) e loop forward (LF), conforme relatado na Tabela 2. Para evitar perdas, pipete ou vórtice suavemente a solução de primer antes de realizar uma centrifugação rápida (10.000 x g, 1 min em RT) com uma microcentrífuga.
- Armazene a mistura de primer 10x a -20 °C para armazenamento de longo prazo; no entanto, prepare-se o suficiente para um máximo de cinco experimentos, independentemente de várias amostras, para evitar muitos ciclos de congelamento e descongelamento.
NOTA Se for necessário um volume menor da mistura de primer, ajuste os valores calculando os novos volumes (Tabela 2). Além disso, os conjuntos RdRp e RdRp/Hel não incluem o primer LF porque os primers de alça não são necessários para reações RT-LAMP. Como resultado, substitua o volume do primer LF por água livre de nuclease ou água DEPC a 0,1%.
2. Reação RT-LAMP
- Ligue o gabinete de fluxo laminar de acordo com as instruções do fabricante e aguarde pelo menos 3 minutos para que o fluxo de ar se estabilize.
- Quando o fluxo de ar estiver estável, limpe e higienize as superfícies internas do gabinete usando uma técnica asséptica. Para isso, use os seguintes desinfetantes nesta ordem: 1000 ppm de amônio quaternário (cloreto de benzalcônio), 2% de hipoclorito, 3% de peróxido de hidrogênio e 70% de etanol.
NOTA: Neste caso, a técnica asséptica consiste em aplicar o desinfetante e removê-lo com guardanapos de dentro da cabine para o exterior sem passar por cima de superfícies previamente limpas. - Usando os desinfetantes da etapa 2.2, limpe os materiais que entrarão na cabine na mesma ordem.
NOTA: Micropipetas, caixas de pontas de filtro, frascos com tubos de 1.5 mL e 0.6 mL, tubos de PCR de 0.2 mL, racks e um copo de 400 mL devem ser trazidos para o gabinete. - Traga alguns guardanapos e luvas de nitrilo para a cabine. Depois disso, desligue o gabinete e exponha-o à luz ultravioleta (UV) por 15 min.
CUIDADO: Para evitar danos aos tecidos e ao DNA devido à exposição prolongada à radiação, evite a luz ultravioleta até que o tempo definido na etapa 2.4 expire.
NOTA Execute a montagem mostrada na Figura 2 antes de iniciar o protocolo e inicie o banho-maria após concluir a etapa 2.4. É crucial encher o recipiente de metal quase até a borda com água potável e ajustar a temperatura da placa de aquecimento do laboratório de ferro para 90 °C, pois isso resultará em uma temperatura de ~66,3 °C no sistema, que é monitorada com o termômetro de mercúrio. - Após o término do período de irradiação, reinicie o gabinete e siga as recomendações da etapa 1.1.
- Coloque os reagentes (Tabela 3, Tabela 4 e Tabela 5) em um refrigerador cheio de gelo ou em uma pequena geladeira de poliestireno. Coloque o recipiente dentro do gabinete após limpá-lo com etanol 70%.
- Em um tubo de microcentrífuga de 0,6 mL, prepare a mistura LAMP do gene a ser amplificado (RdRp, N-A e RdRp/Hel), adicionando apenas os seguintes componentes: 10x Tampão, MgSO4, dNTPs, 1x mistura de primers e água livre de nuclease ou 0,1% de água DEPC; misture bem pipetando para homogeneizar.
CUIDADO: Devido ao manuseio e comportamento inadequados dentro do gabinete, há um alto risco de contaminação do reagente. As seguintes regras devem ser seguidas para mitigar esse problema: (i) usar pontas estéreis e filtrantes; ii) utilizar uma ponta para cada reagente; iii) Deslocar-se lenta e cuidadosamente para evitar perturbar o fluxo laminar; (iv) manter a ordem e usar o menor número de materiais; e (v) usar luvas diferentes para preparar a mistura e adicionar o material genético.
NOTA: Mantenha todos os reagentes, especialmente enzimas, no gelo, pois as mudanças de temperatura podem desnaturá-los e alterar a atividade da polimerase. - Coloque o(s) tubo(s) de 0,6 ml com a tampa fechada num bloco de aquecimento e incube a 95 °C durante 5 min.
NOTA: Ligue o bloco de aquecimento para tubos de 1.5-2.0 mL localizados fora do gabinete por pelo menos 30 min antes de iniciar o LAMP preparação da mistura e monitore a temperatura (95 °C) com um termômetro de mercúrio ou álcool. - Quando a incubação estiver concluída, coloque os tubos em um refrigerador de poliestireno cheio de gelo por 5 min.
- Retorne os tubos ao gabinete de fluxo laminar e conclua a preparação da mistura LAMP adicionando as enzimas DNA polimerase (Bst 3.0), transcriptase reversa e DNA polimerase de alta fidelidade (Tabela 3, Tabela 4 e Tabela 5). No caso de usar a detecção colorimétrica, adicione o corante azul de hidroxinaftol (HNB).
- Depois de adicionar esses reagentes, misture muito bem os reagentes LAMP, pipetando-os para solubilizar as enzimas e o corante.
- Encha cada tubo de PCR com 22,0 μL da mistura, tomando cuidado para não criar bolhas. Em seguida, adicione 3,0 μL de água a 0,1% de DEPC ou água livre de nuclease ao controle negativo ou ao controle de tubo sem modelo (NTC) e reserve o (s) tubo (s) restante (s) para a adição (material genético).
NOTA: Mantenha os tubos de PCR em um refrigerador cheio de gelo até que a amostra seja adicionada para evitar a ativação da enzima Bst 3.0 e iniciar a reação prematuramente. - Remova todos os materiais do gabinete e use etanol 70% para limpar as superfícies. Em seguida, desligue-o seguindo as instruções do fabricante.
- Em uma área separada, adicione 3 μL da amostra a cada tubo de PCR e homogeneize-o completamente. Use uma micropipeta de 20 μL e pontas de filtro para fazer isso.
CUIDADO: A micropipeta usada para adicionar o material genético deve ser usada exclusivamente para esse fim e não pode ser usada para preparar a mistura. Desta forma, evita-se a contaminação dos reagentes. Além disso, mantenha amostras de RNA no gelo o tempo todo para reduzir a possibilidade de degradação do RNA. Use o seguinte equipamento de proteção individual (EPI) para a adição da amostra: avental descartável, boné, máscara N95, leggings, óculos de laboratório e luvas de nitrilo. - Antes de realizar a reação colorimétrica, tire fotos dos tubos de PCR com uma câmera de alta qualidade. A cor inicial com HNB é violeta.
- Realizar a reação no seguinte sistema ou equipamento: (i) termociclador e (ii) banho-maria.
- Termociclador: Deposite os tubos no bloco de reação e configure o termoperfil (ver Tabela 6) no equipamento.
- Banho-maria: Deposite os tubos em recipientes circulares e ajuste-os muito bem para evitar que saiam. Em seguida, coloque os recipientes no banho-maria (Figura 2A, B) na temperatura listada na Tabela 6.
- No caso do banho-maria, uma vez que os tubos estejam dentro do sistema, inicie o cronômetro por 60 min (Tabela 6).
- Remova os tubos do termociclador ou banho-maria após o tempo de reação e armazene-os a 4 ° C para a corrida eletroforética ou a -20 ° C até o uso.
- Se uma reação colorimétrica foi realizada, tire fotos dos tubos de PCR usando uma câmera de alta qualidade. A cor final com HNB é azul celeste.
3. Análise de produtos de amplificação em gel de agarose
NOTA: Essas etapas são sugeridas como verificações adicionais para a reação colorimétrica ou controle de desempenho durante a etapa de padronização. Isso ocorre porque a técnica pode apresentar um enorme risco de contaminação para o laboratório que faz esses testes.
- Coloque o leito dentro da câmara de eletroforese de forma que as borrachas da borda toquem as paredes, criando um espaço vedado para a adição de agarose (câmara interna) (Figura 3A, B).
- Depois de concluir a etapa 3.1, pesar a quantidade necessária de agarose em um béquer de 500 mL para obter um gel a 2%. Depois disso, adicione o volume necessário de tampão EDTA de trisacetato (TAE) 0,5x e leve ao microondas por 1-2 min.
NOTA: A agarose é completamente derretida quando é translúcida e sem grumos quando removida do forno. Se isso não for confirmado, regiões mal gelificadas podem permanecer, fazendo com que a corrida eletroforética e a visualização dos produtos de amplificação sejam alteradas. - Retirar o copo da estufa e deitar a agarose na câmara interna criada no passo 3.1 (figura 3C). Em seguida, verifique se não há bolhas e, se houver, remova-as usando uma ponta de micropipeta.
- Disponha o pente para formar os poços e deixe a agarose gelificar por cerca de 30 min em temperatura ambiente (RT).
- Após este tempo, adicione 5 mL de tampão TAE 0,5x para facilitar a remoção dos favos e do leito contendo o gel. Em seguida, posicione o gel de forma que os poços fiquem no ânodo (Figura 3D).
- Encha a câmara de eletroforese com tampão TAE 0.5x até a capacidade especificada pelo fabricante, garantindo que os eletrodos estejam em contato com o tampão.
- Adicione 3 μL de marcador de peso molecular ao primeiro poço do gel e adicione 9 μL de NTC e cada amostra aos seguintes poços. Faça-os combinando 7 μL do produto de amplificação com 3 μL de tampão de carga; em seguida, carregue 9 μL dessa mistura nos poços do gel.
- Cubra a câmara de eletroforese com a tampa e conecte os cabos às portas de alimentação no padrão de cores. Defina a fonte de alimentação para os seguintes parâmetros: 100 V e amperagem constante por 120 min.
- Após a conclusão da corrida eletroforética, coloque o gel no recipiente com a solução de coloração (brometo de etídio) e incube por 30 min.
- Após a incubação, remova o gel da solução de coloração e coloque-o em um saco zip-lock. Isso evita a contaminação do equipamento que será usado para visualizar os amplicons.
- Visualize o gel em um transiluminador ou gerador de imagens como o Amersham Imager 600.
Resultados
A implementação do protocolo começa com o desenho do conjunto de primers para cada gene alvo seguindo o protocolo descrito acima. Em junho de 2020, 5.000 genomas de SARS-CoV-2 foram obtidos do banco de dados NextStrain, com 10% de representatividade dos genomas colombianos. Essas sequências foram alinhadas para obter a sequência de consenso que foi usada no processo de design do primer. A Tabela 1 mostra o conjunto de primers escolhido para os primers RdRp/Hel e RdRp. O conjunto de primers para ampl...
Discussão
Embora o RT-LAMP seja considerado uma metodologia complementar para a realização de diagnósticos moleculares, ele também apresenta algumas limitações e etapas críticas que devem ser consideradas quando o protocolo é padronizado e implementado.
A padronização LAMP para a detecção de SARS-CoV-2 avaliou os seguintes parâmetros e componentes no master mix: (a) concentração e temperatura de alinhamento dos primers; b) Concentração das enzimas; c) Concentração de magnésio; d) Tem...
Divulgações
Natalia Campillo-Pedroza é CEO da empresa BioDx: Diagnóstico y Soluciones Biotecnológicas S.A.S. O restante dos autores declara não haver conflito de interesses.
Agradecimentos
Este trabalho foi financiado pelo Sistema General de Regalías da Colômbia, número de processo BPIN 2020000100092, e Universidad Icesi - Convocatoria Interna, número de processo CA0413119. O MFVT também foi financiado pelos Fundos de Professor Assistente da Universidad de los Andes. As entidades financiadoras não participaram da concepção, execução das atividades, coleta de dados e análise e preparação dos dados do manuscrito. Agradecemos ao Hospital Universitário Fundación Valle del Lili pelo RNA viral das amostras de Sars-CoV-2 e ao Dr. Alvaro Barrera-Ocampo pelos comentários sobre o manuscrito.
Materiais
| Name | Company | Catalog Number | Comments |
| 1 kb DNA Ladder | SOLIS BIODYNE | 07-12-00050 | Store at -20 °C |
| 50x TAE Electrophoresis Buffer | ThermoScientific | B49 | Store at roome temperature |
| Accuris High Fidelity Polymerase | ACCURIS LIFE SCIENCE REAGENTS | PR1000-HF-200 | It can be used in case Q5 High-Fidelity DNA polymerase cannot be purchased. For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| Agarose | PanReacAppliChem | A8963,0100 | N/A |
| Bst 3.0 DNA Polymerase 8000 IU/mL | New England BioLabs | M0374S/M0374L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| Deoxynucleotide (dNTP) Solution Set | New England BioLabs | N0446S | Store at -20 °C |
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220-25G | Handle it with caution under an extraction cabinet |
| GeneRuler 100 bp Plus DNA Ladder, ready-to-use | ThermoScientific | SM0322 | Store at -20 °C |
| Hydroxy naphthol blue disodium salt | Santa Cruz Biotechnology | sc-215156B | N/A |
| Q5 High-Fidelity DNA polymerase 2000 IU/mL | New England BioLabs | M0491S/M0491L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| WarmStart RTx Reverse Transcriptase 15000 IU/mL | New England BioLabs | M0380S/M0380L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
Referências
- World Health Organization. . Who coronavirus (COVID-19) dashboard (no date). , (2023).
- Ibrahim, N. K. Epidemiologic surveillance for controlling Covid-19 pandemic: types, challenges and implications. Journal of Infection and Public Health. 13 (11), 1630-1638 (2020).
- Rojas-Gallardo, D. M., et al. COVID-19 in Latin America: Contrasting phylodynamic inference with epidemiological surveillance. (Molecular epidemiology of COVID-19 in Latin America). medRxiv. , (2020).
- Liu, R., et al. Positive rate of RT-PCR detection of SARS-CoV-2 infection in 4880 cases from one hospital in Wuhan, China, from Jan to Feb 2020. Clinica Chimica Acta. 505, 172-175 (2020).
- Kevadiya, B. D., et al. Diagnostics for SARS-CoV-2 infections. Nature Materials. 20 (5), 593-605 (2021).
- Tomita, N., Mori, Y., Kanda, H., Notomi, T. Loop-mediated isothermal amplification (LAMP) of gene sequences and simple visual detection of products. Nature Protocols. 3 (5), 877-882 (2008).
- Li, Y., Fan, P., Zhou, S., Zhang, L. Loop-mediated isothermal amplification (LAMP): A novel rapid detection platform for pathogens. Microbial Pathogenesis. 107, 54-61 (2017).
- Notomi, T., Mori, Y., Tomita, N., Kanda, H. Loop-mediated isothermal amplification (LAMP): principle, features, and future prospects. Journal of Microbiology. 53 (1), 1-5 (2015).
- Augustine, R., et al. Loop-mediated isothermal amplification (LAMP): A rapid, sensitive, specific, and cost-effective point-of-care test for coronaviruses in the context of COVID-19 pandemic. Biology (Basel). 9 (8), 182 (2020).
- . Nextstrain Available from: https://nextstrain.org/ (2023)
- . Neb Lamp, NEB LAMP Available from: https://lamp.neb.com/ (2023)
- . Blast: Basic local alignment search tool (no date) Available from: https://blast.ncbi.nlm.nih.gov/ (2023)
- Zhang, Y., et al. Rapid molecular detection of SARS-CoV-2 (COVID-19) virus RNA using colorimetric LAMP. medRxiv. , (2020).
- Lu, R., et al. Development of a novel reverse transcription loop-mediated isothermal amplification method for rapid detection of SARS-CoV-2. Virologica Sinica. 35 (3), 344-347 (2020).
- Najafov, A., Hoxhaj, G. . PCR Guru. , (2017).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. Biotechniques. 69 (3), 178-185 (2020).
- Ramírez-Chavarría, R. G., et al. Automatic analysis of isothermal amplification via impedance time-constant-domain spectroscopy: A SARS-CoV-2 case study. Chemosensors. 11 (4), 230 (2023).
- Haque, M. F. U., et al. A novel RdRp-based colorimetric RT-LAMP assay for rapid and sensitive detection of SARS-CoV-2 in clinical and sewage samples from Pakistan. Virus Research. 302, 198484 (2021).
- Donia, A., et al. Integration of RT-LAMP and microfluidic technology for detection of SARS-CoV-2 in wastewater as an advanced point-of-care platform. Food and Environmental Virology. 14, 364-373 (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados