Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Detección del virus SARS-CoV-2 mediante amplificación isotérmica mediada por bucle de transcripción inversa
En este artículo
Resumen
Aquí proporcionamos un protocolo completo para estandarizar e implementar el método de detección del virus SARS-CoV-2 en muestras humanas mediante amplificación isotérmica mediada por bucle de transcripción inversa (RT-LAMP). Este método, realizado en 60 minutos, podría adaptarse a cualquier laboratorio o punto de atención a un bajo costo y utilizando equipos económicos.
Resumen
El virus del coronavirus 2 del síndrome respiratorio agudo grave (SARS-CoV-2) ha tenido un impacto drástico en la salud humana. Sigue siendo una amenaza para la sociedad moderna porque muchas personas mueren como resultado de la infección. La enfermedad se diagnostica mediante pruebas serológicas y moleculares, como la reacción en cadena de la polimerasa en tiempo real (RT-PCR). Este último tiene varias desventajas porque requiere infraestructura especializada, equipos costosos y personal capacitado. En este artículo, presentamos un protocolo que describe los pasos necesarios para detectar el virus SARS-CoV-2 mediante la amplificación isotérmica mediada por bucle de transcripción inversa (RT-LAMP) en muestras humanas. El protocolo incluye instrucciones para el diseño de cebadores in silico, la preparación de reactivos, la amplificación y la visualización. Una vez estandarizado, este método puede implementarse fácilmente y adaptarse a cualquier laboratorio o punto de atención en 60 minutos a un bajo costo y utilizando equipos económicos. Es adaptable para detectar diferentes patógenos. Por lo tanto, potencialmente puede ser utilizado en el campo y en los centros de salud para realizar una vigilancia epidemiológica oportuna.
Introducción
El coronavirus 2 del síndrome respiratorio agudo grave (SARS-CoV-2) causa la enfermedad por coronavirus 2019 (COVID-19). La Organización Mundial de la Salud declaró una emergencia de salud pública de importancia internacional el 30 de enero de 2020 y una pandemia el 11 de marzo de 2020. La pandemia provocó más de 760 millones de casos y 6,87 millones de muertes hasta la fecha de redacción de este artículo1.
El impacto de este virus ha puesto de manifiesto la necesidad de contar con herramientas de vigilancia mejores, más precisas, más rápidas y más ampliamente disponibles para mejorar la detección y el control de las enfermedades infecciosas 2,3. Durante la pandemia, las pruebas diagnósticas del SARS-CoV-2 se basaron en la detección de ácido nucleico, anticuerpos y proteínas, pero la detección por RT-PCR de ácido nucleico es el estándar de oro4. Sin embargo, la RT-PCR tiene algunas limitaciones; Requiere equipos especializados, infraestructura y personal capacitado en biología molecular, limitando su aplicación a laboratorios especializados. Además, requiere mucho tiempo (4-6 h), sin incluir el tiempo de transporte de las muestras al laboratorio, que puede llevardías 5. Estas limitaciones impiden el procesamiento eficiente de las muestras y la obtención de la información necesaria para la planificación de contingencias y la gestión epidemiológica.
La amplificación isotérmica mediada por bucle de transcripción inversa (RT-LAMP) tiene varias ventajas sobre la RT-PCR, lo que la convierte en una estrategia atractiva para el diseño de futuras pruebas diagnósticas en el punto de atención (POCT), especialmente en entornos con recursos limitados6. En primer lugar, es muy específico porque utiliza entre cuatro y seis cebadores que reconocen de seis a ocho áreas de la secuencia diana, ya sea ADN o ARN 7,8. En segundo lugar, debido a que funciona a una temperatura constante, no requiere equipos sofisticados como termocicladores en tiempo real para generar la amplificación, ni requiere personal altamente capacitado para operarlo. En tercer lugar, el tiempo de reacción es muy corto (~60 min) y se emplean reactivos poco especializados, lo que lo convierte en una herramienta rentable6. Teniendo en cuenta lo anterior y la emergencia sanitaria provocada por la pandemia de COVID-19, esta técnica puede ser vista como un método diagnóstico alternativo, rápido, económico y sencillo de implementar en cualquier laboratorio de investigación9.
En este artículo se describe el protocolo para estandarizar e implementar un RT-LAMP para detectar el SARS-CoV-2 por métodos colorimétricos utilizando un termociclador y un baño de agua (Figura 1). Se discuten los puntos críticos, sus limitaciones y las alternativas para avanzarlos.
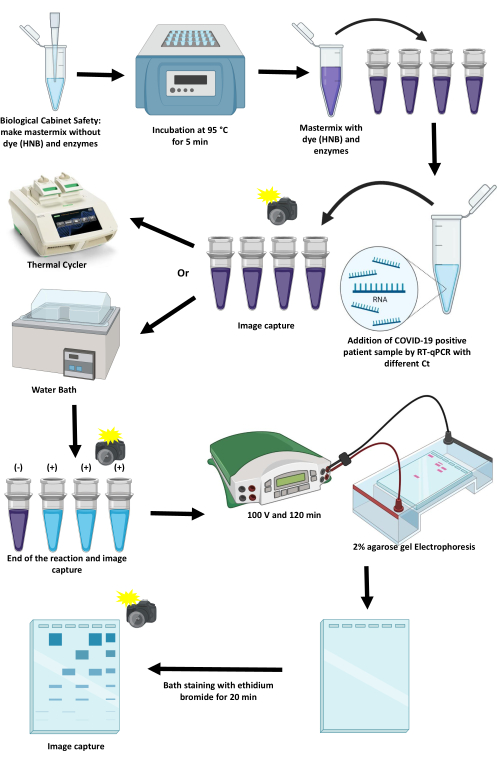
Figura 1: Esquema del protocolo de amplificación del SARS-CoV-2 mediante la técnica RT-LAMP. Haga clic aquí para ver una versión ampliada de esta figura.
Protocolo
Las muestras utilizadas fueron proporcionadas por el laboratorio clínico del Hospital Universitario Fundación Valle del Lili y correspondieron al ARN purificado de pacientes que dieron positivo a COVID-19 mediante la técnica RT-qPCR. Todos los pacientes dieron su consentimiento informado para la investigación, y este estudio fue aprobado por el comité de bioética para estudios en humanos del hospital universitario Fundación Valle del Lili.
1. Diseño y preparación de la imprimación RT-LAMP
NOTA: Los cebadores LAMP se pueden utilizar con una variedad de plataformas, incluidas LAMP de New England BioLabs (NEB), Primer Explorer y análisis versátil de ensayo LAMP (LAVA). Sin embargo, para este protocolo, se utilizó la herramienta NEB LAMP. El diseño del cebador se puede realizar utilizando genomas del SARS-CoV-2 obtenidos de la base de datos NextStrain10. En la Tabla 1 se muestra el conjunto de cebadores utilizados en este protocolo.
- Diseño de imprimación para LAMP
- Obtener secuencias del genoma viral.
- Realice alineaciones de secuencia para obtener la secuencia de consenso.
- Navegue hasta la plataforma11 de la herramienta de diseño de cebadores NEB LAMP y siga las instrucciones de la guía rápida. Esta herramienta produce los mismos resultados que el explorador de cebador V5, pero es mucho más fácil de usar en su salida. Utilice los manuales de usuario del explorador de imprimaciones como guía para el diseño de imprimaciones.
- Evaluación termodinámica del conjunto de cebadores
- Utilice la herramienta Imprimación-Dímero12 para realizar un análisis termodinámico de los cebadores obtenidos.
- Coloque las secuencias de cebadores en la herramienta. A continuación, seleccione la opción Análisis múltiplex e informe de estructura de dímeros.
- Seleccione los juegos de cebadores que tengan un ΔG no inferior a -5.
- Evaluación de la especificidad de los cebadores diseñados
- Utilice la base de datos de colección de nucleótidos (nt/nt) de BLAST13 para analizar cada cebador.
- Para realizar el primer análisis BLAST, seleccione la Base de Datos Refseq_rna y filtre la búsqueda con el grupo de géneros que pertenecen a la subfamilia Orthocoronavirinae. Se trata de Alphacoronavirus (taxid:693996), Gammacoronavirus (taxid:694013) y Deltacoronavirus (taxid:1159901). Además, evalúe la secuencia frente a otros virus que circulan simultáneamente como el subtipo H1N1 (taxid:114727), el virus de la influenza A (taxid:11320) y el virus de la influenza B (taxid:11520).
- Para realizar el segundo análisis BLAST, seleccione el GenBank de Betacoronavirus y filtre la búsqueda con Coronaviridae (taxid:11118) y SARS (taxid:694009). Estos grupos contienen secuencias de todos los genomas identificados del coronavirus SARS, incluidos los genomas encontrados en murciélagos, Betacoronavirus (taxid:694002).
- Para este protocolo, asegúrese de que los cebadores no se alineen con genomas que no sean el genoma objetivo, el SARS-CoV-2.
- Preparación de la imprimación
- Centrifugar los viales que contienen los cebadores iyofilizados con una microcentrífuga (10.000 x g, 1 min a temperatura ambiente [RT]) para evitar pérdidas durante la apertura del tubo.
- Rehidratar el polvo iyofilizado en agua de pirocarbonato de dietilo (DEPC) al 0,1% o agua libre de nucleasas hasta una concentración final de 100 μM (Tabla 2) y disolver completamente pipeteando hacia arriba y hacia abajo. A continuación, centrifugar a máxima velocidad (10.000 x g, 1 min en RT) en una microcentrífuga para recoger todas las soluciones de cebador en el fondo del tubo.
- Prepare la mezcla de imprimación 10x debajo de una cabina de bioseguridad con la imprimación interna directa (FIP), la imprimación interna trasera (BIP), la imprimación exterior delantera (F3), la imprimación exterior trasera (B3), el ciclo hacia atrás (LB) y la imprimación de bucle hacia adelante (LF), como se informa en la Tabla 2. Para evitar pérdidas, pipetee o agite suavemente la solución del cebador antes de realizar un centrifugado rápido (10.000 x g, 1 min en RT) con una microcentrífuga.
- Almacene la mezcla de cebador 10x a -20 °C para un almacenamiento a largo plazo; Sin embargo, prepárese lo suficiente para un máximo de cinco experimentos, independientemente de varias muestras, para evitar demasiados ciclos de congelación y descongelación.
NOTA Si se necesita un volumen menor de la mezcla de cebadores, ajuste los valores calculando los nuevos volúmenes (Tabla 2). Además, los conjuntos RdRp y RdRp/Hel no incluyen el cebador LF porque los cebadores de bucle no son necesarios para las reacciones RT-LAMP. Como resultado, reemplace el volumen de cebador LF con agua libre de nucleasas o agua DEPC al 0,1%.
2. Reacción RT-LAMP
- Encienda la cabina de flujo laminar de acuerdo con las instrucciones del fabricante y espere al menos 3 minutos para que el flujo de aire se estabilice.
- Una vez que el flujo de aire sea estable, limpie y desinfecte las superficies internas del gabinete utilizando una técnica aséptica. Para lograr esto, use los siguientes desinfectantes en este orden: 1000 ppm de amonio cuaternario (cloruro de benzalconio), 2% de hipoclorito, 3% de peróxido de hidrógeno y 70% de etanol.
NOTA: En este caso, la técnica aséptica consiste en aplicar el desinfectante y retirarlo con servilletas desde el interior de la cabina hacia el exterior sin pasar por superficies previamente limpias. - Utilizando los desinfectantes del paso 2.2, limpie los materiales que entrarán en la cabina en el mismo orden.
NOTA: Se deben introducir en el armario micropipetas, cajas de puntas de filtro, matraces con tubos de 1,5 mL y 0,6 mL, tubos de PCR de 0,2 mL, gradillas y un vaso de precipitados de 400 mL. - Traiga algunas servilletas y guantes de nitrilo a la cabina. Después de eso, apague el gabinete y expóngalo a la luz ultravioleta (UV) durante 15 minutos.
PRECAUCIÓN: Para evitar daños en los tejidos y el ADN por la exposición prolongada a la radiación, evite la luz ultravioleta hasta que expire el tiempo establecido en el paso 2.4.
NOTA Realice el montaje que se muestra en la Figura 2 antes de comenzar el protocolo y comience el baño de agua después de completar el paso 2.4. Es crucial llenar el recipiente metálico casi hasta el borde con agua potable y ajustar la temperatura de la placa calefactora de laboratorio de hierro a 90 °C, ya que esto dará como resultado una temperatura de ~66,3 °C en el sistema, que se controla con el termómetro de mercurio. - Una vez finalizado el período de irradiación, reinicie el armario y siga las recomendaciones del paso 1.1.
- Coloque los reactivos (Tabla 3, Tabla 4 y Tabla 5) en una hielera llena de hielo o en un pequeño refrigerador de poliestireno. Coloque el recipiente dentro del gabinete después de limpiarlo con etanol al 70%.
- En un tubo de microcentrífuga de 0,6 mL, prepare la mezcla LAMP del gen a amplificar (RdRp, N-A y RdRp/Hel), añadiendo solo los siguientes componentes: 10x Tampón, MgSO4, dNTPs, 1x mezcla de cebadores y agua libre de nucleasas o 0,1% de agua DEPC; Mezclar bien pipeteando para homogeneizar.
PRECAUCIÓN: Debido a la manipulación y el comportamiento inadecuados dentro del armario, existe un alto riesgo de contaminación del reactivo. Se deben seguir las siguientes reglas para mitigar este problema: (i) utilizar puntas estériles y de filtro; ii) utilizar una punta para cada reactivo; (iii) moverse lenta y cuidadosamente para evitar interrumpir el flujo laminar; (iv) mantener el orden y utilizar la menor cantidad de materiales; y (v) utilizar diferentes guantes para preparar la mezcla y añadir el material genético.
NOTA: Mantenga todos los reactivos, especialmente las enzimas, en hielo porque los cambios de temperatura pueden desnaturalizarlos y alterar la actividad de la polimerasa. - Coloque el tubo o los tubos de 0,6 ml con el tapón cerrado en un bloque calefactor e incube a 95 °C durante 5 min.
NOTA: Encienda el bloque calefactor para tubos de 1,5-2,0 mL ubicados fuera del gabinete durante al menos 30 minutos antes de comenzar la preparación de la mezcla LAMP y controle la temperatura (95 °C) con un termómetro de mercurio o alcohol. - Cuando se complete la incubación, coloque los tubos en un enfriador de poliestireno lleno de hielo durante 5 minutos.
- Regrese los tubos a la cabina de flujo laminar y complete la preparación de la mezcla LAMP agregando las enzimas ADN polimerasa (Bst 3.0), transcriptasa inversa y ADN polimerasa de alta fidelidad (Tabla 3, Tabla 4 y Tabla 5). En el caso de utilizar detección colorimétrica, añadir el colorante azul de hidroxinaftol (HNB).
- Después de agregar estos reactivos, mezcle muy bien los reactivos LAMP pipeteándolos para solubilizar las enzimas y el colorante.
- Llene cada tubo de PCR con 22,0 μL de la mezcla, teniendo cuidado de no crear burbujas. A continuación, añada 3,0 μL de agua con DEPC al 0,1% o agua libre de nucleasas al control negativo o al tubo sin control de plantilla (NTC) y reserve los tubos restantes para la adición (material genético).
NOTA: Mantenga los tubos de PCR en un enfriador lleno de hielo hasta que se agregue la muestra para evitar activar la enzima Bst 3.0 y comenzar la reacción prematuramente. - Retire todos los materiales del gabinete y use etanol al 70% para limpiar las superficies. A continuación, apáguelo siguiendo las instrucciones del fabricante.
- En un área separada, agregue 3 μL de la muestra a cada tubo de PCR y homogeneícelo completamente. Para ello, utilice una micropipeta de 20 μL y puntas de filtro.
ATENCIÓN: La micropipeta utilizada para añadir el material genético debe utilizarse exclusivamente para este fin y no se puede utilizar para preparar la mezcla. De esta manera, se evita la contaminación de los reactivos. Además, mantenga las muestras de ARN en el hielo en todo momento para reducir la posibilidad de degradación del ARN. Utilice el siguiente equipo de protección personal (EPP) para la adición de la muestra: bata desechable, gorro, mascarilla N95, mallas, gafas de laboratorio y guantes de nitrilo. - Antes de realizar la reacción colorimétrica, tome fotografías de los tubos de PCR con una cámara de alta calidad. El color inicial de HNB es el violeta.
- Realizar la reacción en el siguiente sistema o equipo: (i) termociclador y (ii) baño maría.
- Termociclador: Depositar los tubos en el bloque de reacción y colocar el termoperfil (ver Tabla 6) en el equipo.
- Baño María: Deposita los tubos en recipientes circulares y ajústalos muy bien para evitar que se salgan. Después de eso, coloque los recipientes en el baño de agua (Figura 2A, B) a la temperatura indicada en la Tabla 6.
- En el caso del baño maría, una vez que los tubos están dentro del sistema, inicie el temporizador durante 60 min (Tabla 6).
- Retire los tubos del termociclador o baño de agua después del tiempo de reacción y guárdelos a 4 °C para el funcionamiento electroforético o a -20 °C hasta su uso.
- Si se realizó una reacción colorimétrica, tome fotografías de los tubos de PCR con una cámara de alta calidad. El color final con HNB es azul cielo.
3. Análisis de productos de amplificación en gel de agarosa
NOTA: Estos pasos se sugieren como comprobaciones adicionales a la reacción colorimétrica o control del rendimiento durante la etapa de estandarización. Esto se debe a que la técnica podría presentar un gran riesgo de contaminación para el laboratorio que realiza estas pruebas.
- Coloque el lecho dentro de la cámara de electroforesis de modo que las gomas de los bordes toquen las paredes, creando un espacio sellado para la adición de agarosa (cámara interna) (Figura 3A, B).
- Después de completar el paso 3.1, pese la cantidad necesaria de agarosa en un vaso de precipitados de 500 ml para obtener un gel al 2%. Después de eso, agregue el volumen requerido de tampón de trisacetato EDTA (TAE) 0.5x y cocine en el microondas durante 1-2 minutos.
NOTA: La agarosa se derrite completamente cuando está translúcida y sin grumos cuando se retira del horno. Si esto no se confirma, pueden quedar regiones mal gelificadas, lo que hace que se altere el funcionamiento electroforético y la visualización de los productos de amplificación. - Saca el vaso del horno y vierte la agarosa en la cámara interna creada en el paso 3.1 (Figura 3C). Posteriormente, comprueba que no haya burbujas, y si las hay, retíralas con la punta de una micropipeta.
- Coloque el peine para formar los pocillos y deje que la agarosa se gelifique durante unos 30 minutos a temperatura ambiente (RT).
- Pasado este tiempo, añadir 5 mL de tampón TAE 0,5x para facilitar la retirada de los peines y del lecho que contiene el gel. A continuación, coloque el gel de tal manera que los pocillos queden en el ánodo (Figura 3D).
- Llene la cámara de electroforesis con tampón TAE 0,5x hasta la capacidad especificada por el fabricante, asegurándose de que los electrodos estén en contacto con el tampón.
- Agregue 3 μL de marcador de peso molecular al primer pocillo del gel y agregue 9 μL de NTC y cada muestra a los pocillos siguientes. Hágalos combinando 7 μL del producto de amplificación con 3 μL de tampón de carga; luego cargue 9 μL de esta mezcla en los pocillos del gel.
- Cubra la cámara de electroforesis con la tapa y conecte los cables a los puertos de la fuente de alimentación en el patrón de color. Ajuste la fuente de alimentación a los siguientes parámetros: 100 V y amperaje constante durante 120 min.
- Una vez finalizada la ejecución electroforética, coloque el gel en el recipiente con la solución de tinción (bromuro de etidio) e incube durante 30 minutos.
- Después de la incubación, retire el gel de la solución de tinción y colóquelo en una bolsa con cierre hermético. Esto evita la contaminación del equipo que se utilizará para visualizar los amplicones.
- Visualice el gel en un transiluminador o generador de imágenes como el Amersham Imager 600.
Resultados
La implementación del protocolo comienza con el diseño del conjunto de cebadores para cada gen diana siguiendo el protocolo descrito anteriormente. En junio de 2020 se obtuvieron 5.000 genomas de SARS-CoV-2 de la base de datos NextStrain, con una representatividad del 10% de los genomas colombianos. Estas secuencias se alinearon para obtener la secuencia de consenso que se utilizó en el proceso de diseño del cebador. En la Tabla 1 se muestra el conjunto de cebadores elegido para los cebadores RdRp/He...
Discusión
Aunque el RT-LAMP se considera una metodología complementaria para realizar diagnósticos moleculares, también tiene algunas limitaciones y pasos críticos que deben tenerse en cuenta cuando se estandariza e implementa el protocolo.
La estandarización de LAMP para la detección de SARS-CoV-2 evaluó los siguientes parámetros y componentes en la mezcla maestra: (a) concentración y temperatura de alineación de los cebadores; b) concentración de las enzimas; c) concentración de magnesio; ...
Divulgaciones
Natalia Campillo-Pedroza es CEO de la empresa BioDx: Diagnóstico y Soluciones Biotecnológicas S.A.S. El resto de los autores declaran no tener conflicto de intereses.
Agradecimientos
Este trabajo fue financiado por el Sistema General de Regalías de Colombia, beca número BPIN 2020000100092, y Universidad Icesi - Convocatoria Interna, beca número CA0413119. El MFVT también fue financiado por los Fondos de Cátedra Asistente de la Universidad de los Andes. Las entidades financiadoras no participaron en el diseño, la ejecución de las actividades, la recopilación de datos, el análisis de datos y la preparación del manuscrito. Agradecemos al Hospital Universitario Fundación Valle del Lili por el ARN viral de las muestras de Sars-CoV-2 y al Dr. Álvaro Barrera-Ocampo por los comentarios al manuscrito.
Materiales
| Name | Company | Catalog Number | Comments |
| 1 kb DNA Ladder | SOLIS BIODYNE | 07-12-00050 | Store at -20 °C |
| 50x TAE Electrophoresis Buffer | ThermoScientific | B49 | Store at roome temperature |
| Accuris High Fidelity Polymerase | ACCURIS LIFE SCIENCE REAGENTS | PR1000-HF-200 | It can be used in case Q5 High-Fidelity DNA polymerase cannot be purchased. For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| Agarose | PanReacAppliChem | A8963,0100 | N/A |
| Bst 3.0 DNA Polymerase 8000 IU/mL | New England BioLabs | M0374S/M0374L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| Deoxynucleotide (dNTP) Solution Set | New England BioLabs | N0446S | Store at -20 °C |
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220-25G | Handle it with caution under an extraction cabinet |
| GeneRuler 100 bp Plus DNA Ladder, ready-to-use | ThermoScientific | SM0322 | Store at -20 °C |
| Hydroxy naphthol blue disodium salt | Santa Cruz Biotechnology | sc-215156B | N/A |
| Q5 High-Fidelity DNA polymerase 2000 IU/mL | New England BioLabs | M0491S/M0491L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| WarmStart RTx Reverse Transcriptase 15000 IU/mL | New England BioLabs | M0380S/M0380L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
Referencias
- World Health Organization. . Who coronavirus (COVID-19) dashboard (no date). , (2023).
- Ibrahim, N. K. Epidemiologic surveillance for controlling Covid-19 pandemic: types, challenges and implications. Journal of Infection and Public Health. 13 (11), 1630-1638 (2020).
- Rojas-Gallardo, D. M., et al. COVID-19 in Latin America: Contrasting phylodynamic inference with epidemiological surveillance. (Molecular epidemiology of COVID-19 in Latin America). medRxiv. , (2020).
- Liu, R., et al. Positive rate of RT-PCR detection of SARS-CoV-2 infection in 4880 cases from one hospital in Wuhan, China, from Jan to Feb 2020. Clinica Chimica Acta. 505, 172-175 (2020).
- Kevadiya, B. D., et al. Diagnostics for SARS-CoV-2 infections. Nature Materials. 20 (5), 593-605 (2021).
- Tomita, N., Mori, Y., Kanda, H., Notomi, T. Loop-mediated isothermal amplification (LAMP) of gene sequences and simple visual detection of products. Nature Protocols. 3 (5), 877-882 (2008).
- Li, Y., Fan, P., Zhou, S., Zhang, L. Loop-mediated isothermal amplification (LAMP): A novel rapid detection platform for pathogens. Microbial Pathogenesis. 107, 54-61 (2017).
- Notomi, T., Mori, Y., Tomita, N., Kanda, H. Loop-mediated isothermal amplification (LAMP): principle, features, and future prospects. Journal of Microbiology. 53 (1), 1-5 (2015).
- Augustine, R., et al. Loop-mediated isothermal amplification (LAMP): A rapid, sensitive, specific, and cost-effective point-of-care test for coronaviruses in the context of COVID-19 pandemic. Biology (Basel). 9 (8), 182 (2020).
- . Nextstrain Available from: https://nextstrain.org/ (2023)
- . Neb Lamp, NEB LAMP Available from: https://lamp.neb.com/ (2023)
- . Blast: Basic local alignment search tool (no date) Available from: https://blast.ncbi.nlm.nih.gov/ (2023)
- Zhang, Y., et al. Rapid molecular detection of SARS-CoV-2 (COVID-19) virus RNA using colorimetric LAMP. medRxiv. , (2020).
- Lu, R., et al. Development of a novel reverse transcription loop-mediated isothermal amplification method for rapid detection of SARS-CoV-2. Virologica Sinica. 35 (3), 344-347 (2020).
- Najafov, A., Hoxhaj, G. . PCR Guru. , (2017).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. Biotechniques. 69 (3), 178-185 (2020).
- Ramírez-Chavarría, R. G., et al. Automatic analysis of isothermal amplification via impedance time-constant-domain spectroscopy: A SARS-CoV-2 case study. Chemosensors. 11 (4), 230 (2023).
- Haque, M. F. U., et al. A novel RdRp-based colorimetric RT-LAMP assay for rapid and sensitive detection of SARS-CoV-2 in clinical and sewage samples from Pakistan. Virus Research. 302, 198484 (2021).
- Donia, A., et al. Integration of RT-LAMP and microfluidic technology for detection of SARS-CoV-2 in wastewater as an advanced point-of-care platform. Food and Environmental Virology. 14, 364-373 (2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados