Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Nachweis des SARS-CoV-2-Virus durch reverse Transkriptionsschleifen-vermittelte isotherme Amplifikation
In diesem Artikel
Zusammenfassung
Hier stellen wir ein vollständiges Protokoll zur Standardisierung und Implementierung der Methode zum Nachweis des SARS-CoV-2-Virus in menschlichen Proben durch reverse Transkriptionsschleifen-vermittelte isotherme Amplifikation (RT-LAMP) zur Verfügung. Diese Methode, die innerhalb von 60 Minuten durchgeführt wird, kann kostengünstig und mit kostengünstigen Geräten an jedes Labor oder jeden Point-of-Care angepasst werden.
Zusammenfassung
Das Virus des schweren akuten respiratorischen Syndroms Coronavirus 2 (SARS-CoV-2) hat dramatische Auswirkungen auf die menschliche Gesundheit. Es ist nach wie vor eine Bedrohung für die moderne Gesellschaft, da viele Menschen an den Folgen einer Infektion sterben. Die Diagnose der Krankheit erfolgt mit serologischen und molekularen Tests, wie z. B. dem Goldstandard der Echtzeit-Polymerase-Kettenreaktion (RT-PCR). Letzteres hat mehrere Nachteile, da es eine spezielle Infrastruktur, teure Ausrüstung und geschultes Personal erfordert. Im Folgenden stellen wir ein Protokoll vor, das die Schritte beschreibt, die zum Nachweis des SARS-CoV-2-Virus mittels reverser Transkriptionsschleifen-vermittelter isothermer Amplifikation (RT-LAMP) in menschlichen Proben erforderlich sind. Das Protokoll enthält Anweisungen für das Design von Primern in silico, die Vorbereitung von Reagenzien, die Amplifikation und die Visualisierung. Einmal standardisiert, kann diese Methode innerhalb von 60 Minuten zu geringen Kosten und mit kostengünstigen Geräten einfach implementiert und an jedes Labor oder jeden Point-of-Care angepasst werden. Es ist anpassungsfähig, um verschiedene Krankheitserreger zu erkennen. Daher kann es potenziell vor Ort und in Gesundheitszentren eingesetzt werden, um eine rechtzeitige epidemiologische Überwachung durchzuführen.
Einleitung
Das schwere akute respiratorische Syndrom Coronavirus 2 (SARS-CoV-2) verursacht die Coronavirus-Krankheit 2019 (COVID-19). Die Weltgesundheitsorganisation erklärte am 30. Januar 2020 eine gesundheitliche Notlage von internationaler Tragweite und am 11. März 2020 eine Pandemie (Pandemie). Die Pandemie führte zum Zeitpunkt der Erstellung dieses Artikels zu über 760 Millionen Fällen und 6,87 Millionen Todesfällen1.
Die Auswirkungen dieses Virus haben den Bedarf an besseren, genaueren, schnelleren und breiter verfügbaren Überwachungsinstrumenten zur Verbesserung der Erkennung und Kontrolle von Infektionskrankheiten deutlich gemacht 2,3. Während der Pandemie basierten die diagnostischen Tests von SARS-CoV-2 auf dem Nachweis von Nukleinsäuren, Antikörpern und Proteinen, aber der RT-PCR-Nachweis von Nukleinsäuren ist der Goldstandard4. Die RT-PCR hat jedoch einige Einschränkungen; Es erfordert spezielle Geräte, Infrastrukturen und in Molekularbiologie geschultes Personal, was seine Anwendung auf spezialisierte Labore beschränkt. Darüber hinaus ist es zeitaufwändig (4-6 Stunden), ohne die Zeit für den Transport der Proben zum Labor, der Tage dauern kann5. Diese Einschränkungen verhindern eine effiziente Probenverarbeitung und die Gewinnung der für die Notfallplanung und das epidemiologische Management erforderlichen Informationen.
Die reverse-transkriptionsschleifen-vermittelte isotherme Amplifikation (RT-LAMP) hat gegenüber der RT-PCR mehrere Vorteile und ist damit eine attraktive Strategie für die Entwicklung zukünftiger Point-of-Care-Diagnosetests (POCT), insbesondere in ressourcenbeschränkten Umgebungen6. Erstens ist es sehr spezifisch, da es zwischen vier und sechs Primer verwendet, die sechs bis acht Bereiche in der Zielsequenz erkennen, sei es DNA oder RNA 7,8. Zweitens benötigt es aufgrund des Betriebs bei konstanter Temperatur weder hochentwickelte Geräte wie Echtzeit-Thermocycler zur Erzeugung der Verstärkung noch hochqualifiziertes Personal, um es zu bedienen. Drittens ist die Reaktionszeit sehr kurz (~60 min), und es werden Reagenzien verwendet, die nicht sehr spezialisiert sind, was es zu einem kostengünstigen Werkzeug macht6. In Anbetracht des Vorstehenden und des durch die COVID-19-Pandemie verursachten gesundheitlichen Notfalls kann diese Technik als alternative Diagnosemethode angesehen werden, die schnell, kostengünstig und einfach in jedem Forschungslabor umzusetzen ist9.
In diesem Artikel wird das Protokoll zur Standardisierung und Implementierung einer RT-LAMP zum Nachweis von SARS-CoV-2 durch kolorimetrische Verfahren unter Verwendung eines Thermocyclers und eines Wasserbades beschrieben (Abbildung 1). Kritische Punkte, ihre Grenzen und Alternativen, um sie voranzutreiben, werden diskutiert.
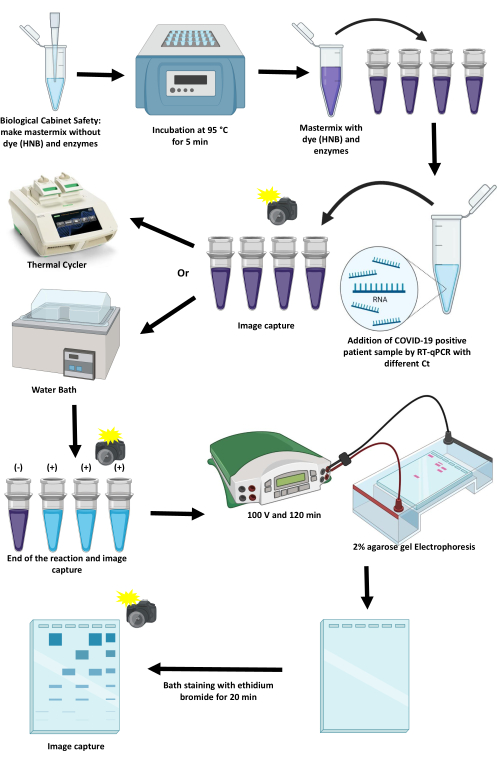
Abbildung 1: Schema des Protokolls zur Amplifikation von SARS-CoV-2 mit der RT-LAMP-Technik. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
Die verwendeten Proben wurden vom klinischen Labor des Universitätskrankenhauses Fundación Valle del Lili zur Verfügung gestellt und entsprachen der gereinigten RNA von Patienten, die mit der RT-qPCR-Technik positiv auf COVID-19 getestet wurden. Alle Patienten gaben eine Einverständniserklärung für die Forschung ab, und diese Studie wurde von der bioethischen Kommission für Humanstudien des Universitätskrankenhauses Fundación Valle del Lili genehmigt.
1. Design und Vorbereitung der RT-LAMP-Grundierung
HINWEIS: LAMP-Primer können mit einer Vielzahl von Plattformen verwendet werden, einschließlich New England BioLabs (NEB) LAMP, Primer Explorer und LAMP Assay Versatile Analysis (LAVA). Für dieses Protokoll wurde jedoch das NEB LAMP-Tool verwendet. Das Primer-Design kann unter Verwendung von SARS-CoV-2-Genomen erfolgen, die aus der NextStrain-Datenbank10 gewonnen wurden. Tabelle 1 zeigt das in diesem Protokoll verwendete Primer-Set.
- Primer-Design für LAMP
- Erhalten Sie virale Genomsequenzen.
- Führen Sie Sequenzausrichtungen durch, um die Konsenssequenz zu erhalten.
- Navigieren Sie zur NEB LAMP Primer Design Tool Platform11 und folgen Sie den Anweisungen in der Kurzanleitung. Dieses Tool liefert die gleichen Ergebnisse wie Primer Explorer V5, ist aber viel benutzerfreundlicher in der Ausgabe. Verwenden Sie die Benutzerhandbücher des Primer-Explorers als Leitfaden für die Primer-Gestaltung.
- Thermodynamische Bewertung des Primer-Satzes
- Verwenden Sie das Werkzeug Primer-Dimer12 , um eine thermodynamische Analyse der erhaltenen Primer durchzuführen.
- Legen Sie die Primersequenzen in das Werkzeug. Wählen Sie dann die Option Multiplex-Analyse und Dimer-Struktur-Bericht.
- Wählen Sie die Grundierungssätze aus, deren ΔG nicht kleiner als -5 ist.
- Spezifitätsbewertung der entworfenen Primer
- Verwenden Sie die Datenbank Nukleotidsammlung (nt/nt) in BLAST13 , um die einzelnen Primer zu analysieren.
- Um die erste BLAST-Analyse durchzuführen, wählen Sie die Datenbank Refseq_rna aus und filtern Sie die Suche nach der Gruppe der Gattungen, die zur Unterfamilie Orthocoronavirinae gehören. Es handelt sich um Alphacoronavirus (taxid:693996), Gammacoronavirus (taxid:694013) und Deltacoronavirus (taxid:1159901). Bewerten Sie außerdem die Sequenz mit anderen Viren, die als H1N1-Subtyp (taxid:114727), Influenza-A-Virus (taxid:11320) und Influenza-B-Virus (taxid:11520) zirkulieren.
- Um die zweite BLAST-Analyse durchzuführen, wählen Sie die Betacoronavirus-Genbank aus und filtern Sie die Suche nach Coronaviridae (taxid:11118) und SARS (taxid:694009). Diese Gruppen enthalten Sequenzen aller identifizierten SARS-Coronavirus-Genome, einschließlich der Genome, die in Fledermäusen, Betacoronavirus (taxid:694002) gefunden wurden.
- Stellen Sie für dieses Protokoll sicher, dass die Primer nicht mit anderen Genomen als dem Zielgenom SARS-CoV-2 übereinstimmen.
- Vorbereitung der Grundierung
- Drehen Sie die Fläschchen mit den iyophilisierten Primern mit einer Mikrozentrifuge (10.000 x g, 1 min bei Raumtemperatur [RT]), um Verluste beim Öffnen des Röhrchens zu vermeiden.
- Rehydrieren Sie das Iyophilisierte Pulver in 0,1 %igem Diethylpyrocarbonat (DEPC)-Wasser oder nukleasefreiem Wasser auf eine Endkonzentration von 100 μM (Tabelle 2) und lösen Sie es durch Auf- und Abpipettieren gründlich auf. Schleudern Sie dann mit maximaler Geschwindigkeit (10.000 x g, 1 min bei RT) in einer Mikrozentrifuge, um alle Primerlösungen am Boden des Röhrchens zu sammeln.
- Bereiten Sie die 10x-Primermischung unter einer Biosicherheitswerkbank mit dem Forward Inner Primer (FIP), dem Backward Inner Primer (BIP), dem Forward Outer Primer (F3), dem Backward Outer Primer (B3), dem Loop Backward (LB) und dem Loop Forward (LF) Primer vor, wie in Tabelle 2 angegeben. Um Verluste zu vermeiden, pipettieren Sie die Primerlösung oder wirbeln Sie sie vorsichtig ein, bevor Sie eine schnelle Drehung (10.000 x g, 1 min bei RT) mit einer Mikrozentrifuge durchführen.
- Lagern Sie die 10x Primermischung bei -20 °C für die Langzeitlagerung; Bereiten Sie jedoch genug für maximal fünf Experimente vor, unabhängig von mehreren Proben, um zu viele Frost-Tau-Zyklen zu vermeiden.
HINWEIS Wenn ein kleineres Volumen der Primermischung benötigt wird, passen Sie die Werte an, indem Sie die neuen Volumina berechnen (Tabelle 2). Darüber hinaus enthalten die RdRp- und RdRp/Hel-Sets den LF-Primer nicht, da für RT-LAMP-Reaktionen keine Schleifenprimer erforderlich sind. Ersetzen Sie daher das Volumen des LF-Primers durch nukleasefreies Wasser oder 0,1 % DEPC-Wasser.
2. RT-LAMP-Reaktion
- Schalten Sie den Laminar-Flow-Schrank gemäß den Anweisungen des Herstellers ein und warten Sie mindestens 3 Minuten, bis sich der Luftstrom stabilisiert hat.
- Sobald der Luftstrom stabil ist, reinigen und desinfizieren Sie die Innenflächen des Schranks mit einer aseptischen Technik. Um dies zu erreichen, verwenden Sie die folgenden Desinfektionsmittel in dieser Reihenfolge: 1000 ppm quartäres Ammonium (Benzalkoniumchlorid), 2 % Hypochlorit, 3 % Wasserstoffperoxid und 70 % Ethanol.
HINWEIS: In diesem Fall besteht die aseptische Technik darin, das Desinfektionsmittel aufzutragen und mit Servietten von innen nach außen zu entfernen, ohne über die zuvor gereinigten Oberflächen zu gehen. - Reinigen Sie mit den Desinfektionsmitteln aus Schritt 2.2 die Materialien, die in die Kabine gelangen, in der gleichen Reihenfolge.
HINWEIS: Mikropipetten, Filterspitzenboxen, Kolben mit 1,5 mL und 0,6 mL Röhrchen, 0,2 mL PCR Röhrchen, Racks und ein 400 mL Becherglas müssen in den Schrank gebracht werden. - Bringen Sie Servietten und Nitrilhandschuhe in die Kabine mit. Schalten Sie danach den Schrank aus und setzen Sie ihn 15 Minuten lang ultraviolettem (UV) Licht aus.
ACHTUNG: Um Gewebe- und DNA-Schäden durch längere Strahlenbelastung zu vermeiden, vermeiden Sie UV-Licht bis zum Ablauf der in Schritt 2.4 eingestellten Zeit.
HINWEIS Führen Sie die in Abbildung 2 gezeigte Montage durch, bevor Sie mit dem Protokoll beginnen, und beginnen Sie mit dem Wasserbad, nachdem Sie Schritt 2.4 abgeschlossen haben. Entscheidend ist, den Metallbehälter fast bis zum Rand mit Trinkwasser zu füllen und die Temperatur der eisernen Laborheizplatte auf 90 °C einzustellen, da dies zu einer Temperatur von ~66,3 °C im System führt, die mit dem Quecksilberthermometer überwacht wird. - Starten Sie nach Ablauf der Bestrahlungszeit den Schrank neu und befolgen Sie die Empfehlungen in Schritt 1.1.
- Legen Sie die Reagenzien (Tabelle 3, Tabelle 4 und Tabelle 5) in einen mit Eis gefüllten Kühler oder einen kleinen Styroporkühlschrank. Stellen Sie den Behälter in den Schrank, nachdem Sie ihn mit 70% Ethanol gereinigt haben.
- Bereiten Sie in einem 0,6-ml-Mikrozentrifugenröhrchen den LAMP-Mix des zu amplifizierenden Gens vor (RdRp, N-A und RdRp/Hel) und fügen Sie nur die folgenden Komponenten hinzu: 10x Puffer, MgSO4, dNTPs, 1x Primer-Mix und nukleasefreies Wasser oder 0,1 % DEPC-Wasser; Durch Pipettieren gut mischen, um zu homogenisieren.
VORSICHT: Aufgrund unsachgemäßer Handhabung und unsachgemäßen Verhaltens im Schrank besteht ein hohes Risiko einer Kontamination des Reagenzes. Die folgenden Regeln müssen befolgt werden, um dieses Problem zu entschärfen: (i) Verwenden Sie sterile und Filterspitzen; ii) für jedes Reagenz eine Spitze verwenden; iii) sich langsam und vorsichtig bewegen, um eine Störung der laminaren Strömung zu vermeiden; (iv) Ordnung zu halten und die wenigsten Materialien zu verwenden; und (v) verwenden Sie unterschiedliche Handschuhe, um die Mischung vorzubereiten und das genetische Material hinzuzufügen.
HINWEIS: Bewahren Sie alle Reagenzien, insbesondere Enzyme, auf Eis auf, da Temperaturänderungen sie denaturieren und die Polymeraseaktivität verändern können. - Das/die 0,6-ml-Röhrchen(e) mit geschlossener Kappe in einen Heizblock legen und 5 Minuten lang bei 95 °C inkubieren.
HINWEIS: Schalten Sie den Heizblock für 1,5-2,0-ml-Röhrchen, die sich außerhalb des Schranks befinden, mindestens 30 Minuten lang ein, bevor Sie mit der Zubereitung der LAMP-Mischung beginnen, und überwachen Sie die Temperatur (95 °C) mit einem Quecksilber- oder Alkoholthermometer. - Wenn die Inkubation abgeschlossen ist, legen Sie die Röhrchen für 5 Minuten in einen mit Eis gefüllten Styroporkühler.
- Setzen Sie die Röhrchen wieder in den Laminar-Flow-Schrank ein und vervollständigen Sie die Vorbereitung des LAMP-Mixes, indem Sie die Enzyme DNA-Polymerase (Bst 3.0), Reverse Transkriptase und High-Fidelity-DNA-Polymerase hinzufügen (Tabelle 3, Tabelle 4 und Tabelle 5). Bei Verwendung der kolorimetrischen Detektion ist der Farbstoff Hydroxinaphtholblau (HNB) hinzuzufügen.
- Nachdem Sie diese Reagenzien hinzugefügt haben, mischen Sie die LAMP-Reagenzien sehr gut, indem Sie sie pipettieren, um die Enzyme und den Farbstoff zu lösen.
- Füllen Sie jedes PCR-Röhrchen mit 22,0 μl der Mischung und achten Sie darauf, keine Blasen zu bilden. Geben Sie dann 3,0 μl 0,1 % DEPC-Wasser oder nukleasefreies Wasser in die Negativkontrolle oder das Röhrchen ohne Template-Kontrolle (NTC) und legen Sie das/die restliche(n) Röhrchen(n) für die Zugabe (genetisches Material) beiseite.
HINWEIS : Bewahren Sie die PCR-Röhrchen bis zur Zugabe der Probe in einem mit Eis gefüllten Kühler auf, um eine Aktivierung des Bst 3.0-Enzyms und einen vorzeitigen Start der Reaktion zu vermeiden. - Entfernen Sie alle Materialien aus dem Schrank und verwenden Sie 70% Ethanol, um die Oberflächen zu reinigen. Schalten Sie es dann gemäß den Anweisungen des Herstellers aus.
- Geben Sie in einem separaten Bereich 3 μl der Probe in jedes PCR-Röhrchen und homogenisieren Sie sie gründlich. Verwenden Sie dazu eine 20-μl-Mikropipette und Filterspitzen.
ACHTUNG: Die Mikropipette, die zur Zugabe des genetischen Materials verwendet wird, darf ausschließlich zu diesem Zweck verwendet werden und darf nicht zur Zubereitung der Mischung verwendet werden. Auf diese Weise wird eine Kontamination der Reagenzien vermieden. Bewahren Sie außerdem immer RNA-Proben auf dem Eis auf, um die Möglichkeit eines RNA-Abbaus zu verringern. Verwenden Sie für die Probenzugabe die folgende persönliche Schutzausrüstung (PSA): Einwegkittel, Mütze, N95-Maske, Leggings, Laborbrille und Nitrilhandschuhe. - Bevor Sie die kolorimetrische Reaktion durchführen, machen Sie Fotos von den PCR-Röhrchen mit einer hochwertigen Kamera. Die Ausgangsfarbe bei HNB ist Violett.
- Die Reaktion wird in folgendem System oder Gerät durchgeführt: i) Thermocycler und ii) Wasserbad.
- Thermocycler: Legen Sie die Rohre in den Reaktionsblock ab und richten Sie das Thermoprofil (siehe Tabelle 6) an der Anlage ein.
- Wasserbad: Legen Sie die Schläuche in kreisförmige Behälter und stellen Sie sie sehr gut ein, um ein Herauskommen zu verhindern. Stellen Sie danach die Behälter bei der in Tabelle 6 angegebenen Temperatur in das Wasserbad (Abbildung 2A, B).
- Im Falle des Wasserbades starten Sie den Timer für 60 Minuten, sobald sich die Schläuche im System befinden (Tabelle 6).
- Nehmen Sie die Schläuche nach der Reaktionszeit aus dem Thermocycler oder Wasserbad und lagern Sie sie bei 4 °C für den Elektrophorelauf oder bei -20 °C bis zur Verwendung.
- Wenn eine kolorimetrische Reaktion durchgeführt wurde, machen Sie Fotos der PCR-Röhrchen mit einer hochwertigen Kamera. Die endgültige Farbe bei HNB ist Himmelblau.
3. Analyse von Amplifikationsprodukten in Agarosegel
HINWEIS: Diese Schritte werden als zusätzliche Überprüfung der kolorimetrischen Reaktion oder als Leistungskontrolle während des Standardisierungsschritts empfohlen. Dies liegt daran, dass die Technik ein enormes Kontaminationsrisiko für das Labor darstellen könnte, das diese Tests durchführt.
- Platzieren Sie das Bett so in der Elektrophoresekammer, dass die Kantengummis die Wände berühren, wodurch ein geschlossener Raum für die Zugabe von Agarose (innere Kammer) entsteht (Abbildung 3A, B).
- Nach Abschluss von Schritt 3.1 wird die erforderliche Menge Agarose in einem 500-ml-Becherglas gewogen, um ein 2%iges Gel zu erhalten. Danach das erforderliche Volumen von 0,5x Tris-Acetat EDTA (TAE) Puffer hinzufügen und 1-2 Minuten in der Mikrowelle erhitzen.
HINWEIS: Die Agarose ist vollständig geschmolzen, wenn sie glasig und klumpenfrei ist, wenn sie aus dem Ofen genommen wird. Wenn dies nicht bestätigt wird, können schlecht gelierte Bereiche zurückbleiben, die dazu führen, dass der elektrophoretische Lauf und die Visualisierung der Amplifikationsprodukte verändert werden. - Nehmen Sie den Becher aus dem Ofen und gießen Sie die Agarose in die in Schritt 3.1 entstandene innere Kammer (Abbildung 3C). Überprüfen Sie anschließend, ob keine Blasen vorhanden sind, und entfernen Sie diese gegebenenfalls mit einer Mikropipettenspitze.
- Ordnen Sie den Kamm so an, dass er die Vertiefungen bildet, und lassen Sie die Agarose etwa 30 Minuten bei Raumtemperatur (RT) gelieren.
- Nach dieser Zeit fügen Sie 5 ml 0,5x TAE-Puffer hinzu, um die Entnahme der Waben und des Bettes mit dem Gel zu erleichtern. Positionieren Sie dann das Gel so, dass sich die Vertiefungen in der Anode befinden (Abbildung 3D).
- Füllen Sie die Elektrophoresekammer mit 0,5x TAE-Puffer bis zu der vom Hersteller angegebenen Kapazität und stellen Sie sicher, dass die Elektroden in Kontakt mit dem Puffer sind.
- Geben Sie 3 μl Molekulargewichtsmarker in die erste Vertiefung des Gels und fügen Sie 9 μl NTC und jede Probe in die folgenden Vertiefungen hinzu. Kombinieren Sie dazu 7 μl des Amplifikationsprodukts mit 3 μl Ladepuffer. Laden Sie dann 9 μl dieser Mischung in die Vertiefungen des Gels.
- Decken Sie die Elektrophoresekammer mit dem Deckel ab und verbinden Sie die Kabel mit den Stromversorgungsanschlüssen im Farbmuster. Stellen Sie die Stromquelle auf folgende Parameter ein: 100 V und konstante Stromstärke für 120 min.
- Nachdem der elektrophoretische Lauf abgeschlossen ist, geben Sie das Gel in den Behälter mit der Färbelösung (Ethidiumbromid) und inkubieren Sie es 30 Minuten lang.
- Entfernen Sie nach der Inkubation das Gel aus der Färbelösung und geben Sie es in einen Zip-Lock-Beutel. Dadurch wird eine Kontamination der Geräte vermieden, die zur Visualisierung der Amplikons verwendet werden.
- Visualisieren Sie das Gel auf einem Transilluminator oder Imager wie dem Amersham Imager 600.
Ergebnisse
Die Implementierung des Protokolls beginnt mit der Entwicklung des Primersatzes für jedes Zielgen gemäß dem oben beschriebenen Protokoll. Im Juni 2020 wurden 5.000 SARS-CoV-2-Genome aus der NextStrain-Datenbank gewonnen, mit einer Repräsentativität von 10 % für kolumbianische Genome. Diese Sequenzen wurden ausgerichtet, um die Konsensussequenz zu erhalten, die im Primer-Designprozess verwendet wurde. Tabelle 1 zeigt die Primer-Sets, die für die Primer RdRp/Hel und RdRp ausgewählt wurden. Das Prim...
Diskussion
Obwohl die RT-LAMP als ergänzende Methodik für die Durchführung molekularer Diagnostik angesehen wird, weist sie auch einige Einschränkungen und kritische Schritte auf, die bei der Standardisierung und Implementierung des Protokolls berücksichtigt werden müssen.
Die LAMP-Standardisierung für den Nachweis von SARS-CoV-2 bewertete die folgenden Parameter und Komponenten im Mastermix: (a) Konzentration und Temperatur der Ausrichtung der Primer; b) Konzentration der Enzyme; c) Magnesiumkonz...
Offenlegungen
Natalia Campillo-Pedroza ist CEO des Unternehmens BioDx: Diagnóstico y Soluciones Biotecnológicas S.A.S. Die übrigen Autoren erklären, dass kein Interessenkonflikt besteht.
Danksagungen
Diese Arbeit wurde vom Sistema General de Regalías aus Kolumbien, Fördernummer BPIN 2020000100092, und der Universidad Icesi - Convocatoria Interna, Fördernummer CA0413119, finanziert. MFVT wurde auch aus den Assistant Professorship Funds der Universidad de los Andes finanziert. Die Fördereinrichtungen waren nicht an der Konzeption, Durchführung der Aktivitäten, der Datenerhebung, der Datenanalyse und der Vorbereitung des Manuskripts beteiligt. Wir danken dem Universitätskrankenhaus Fundación Valle del Lili für die virale RNA aus Sars-CoV-2-Proben und Dr. Alvaro Barrera-Ocampo für die Kommentare zum Manuskript.
Materialien
| Name | Company | Catalog Number | Comments |
| 1 kb DNA Ladder | SOLIS BIODYNE | 07-12-00050 | Store at -20 °C |
| 50x TAE Electrophoresis Buffer | ThermoScientific | B49 | Store at roome temperature |
| Accuris High Fidelity Polymerase | ACCURIS LIFE SCIENCE REAGENTS | PR1000-HF-200 | It can be used in case Q5 High-Fidelity DNA polymerase cannot be purchased. For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| Agarose | PanReacAppliChem | A8963,0100 | N/A |
| Bst 3.0 DNA Polymerase 8000 IU/mL | New England BioLabs | M0374S/M0374L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| Deoxynucleotide (dNTP) Solution Set | New England BioLabs | N0446S | Store at -20 °C |
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220-25G | Handle it with caution under an extraction cabinet |
| GeneRuler 100 bp Plus DNA Ladder, ready-to-use | ThermoScientific | SM0322 | Store at -20 °C |
| Hydroxy naphthol blue disodium salt | Santa Cruz Biotechnology | sc-215156B | N/A |
| Q5 High-Fidelity DNA polymerase 2000 IU/mL | New England BioLabs | M0491S/M0491L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| WarmStart RTx Reverse Transcriptase 15000 IU/mL | New England BioLabs | M0380S/M0380L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
Referenzen
- World Health Organization. . Who coronavirus (COVID-19) dashboard (no date). , (2023).
- Ibrahim, N. K. Epidemiologic surveillance for controlling Covid-19 pandemic: types, challenges and implications. Journal of Infection and Public Health. 13 (11), 1630-1638 (2020).
- Rojas-Gallardo, D. M., et al. COVID-19 in Latin America: Contrasting phylodynamic inference with epidemiological surveillance. (Molecular epidemiology of COVID-19 in Latin America). medRxiv. , (2020).
- Liu, R., et al. Positive rate of RT-PCR detection of SARS-CoV-2 infection in 4880 cases from one hospital in Wuhan, China, from Jan to Feb 2020. Clinica Chimica Acta. 505, 172-175 (2020).
- Kevadiya, B. D., et al. Diagnostics for SARS-CoV-2 infections. Nature Materials. 20 (5), 593-605 (2021).
- Tomita, N., Mori, Y., Kanda, H., Notomi, T. Loop-mediated isothermal amplification (LAMP) of gene sequences and simple visual detection of products. Nature Protocols. 3 (5), 877-882 (2008).
- Li, Y., Fan, P., Zhou, S., Zhang, L. Loop-mediated isothermal amplification (LAMP): A novel rapid detection platform for pathogens. Microbial Pathogenesis. 107, 54-61 (2017).
- Notomi, T., Mori, Y., Tomita, N., Kanda, H. Loop-mediated isothermal amplification (LAMP): principle, features, and future prospects. Journal of Microbiology. 53 (1), 1-5 (2015).
- Augustine, R., et al. Loop-mediated isothermal amplification (LAMP): A rapid, sensitive, specific, and cost-effective point-of-care test for coronaviruses in the context of COVID-19 pandemic. Biology (Basel). 9 (8), 182 (2020).
- . Nextstrain Available from: https://nextstrain.org/ (2023)
- . Neb Lamp, NEB LAMP Available from: https://lamp.neb.com/ (2023)
- . Blast: Basic local alignment search tool (no date) Available from: https://blast.ncbi.nlm.nih.gov/ (2023)
- Zhang, Y., et al. Rapid molecular detection of SARS-CoV-2 (COVID-19) virus RNA using colorimetric LAMP. medRxiv. , (2020).
- Lu, R., et al. Development of a novel reverse transcription loop-mediated isothermal amplification method for rapid detection of SARS-CoV-2. Virologica Sinica. 35 (3), 344-347 (2020).
- Najafov, A., Hoxhaj, G. . PCR Guru. , (2017).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. Biotechniques. 69 (3), 178-185 (2020).
- Ramírez-Chavarría, R. G., et al. Automatic analysis of isothermal amplification via impedance time-constant-domain spectroscopy: A SARS-CoV-2 case study. Chemosensors. 11 (4), 230 (2023).
- Haque, M. F. U., et al. A novel RdRp-based colorimetric RT-LAMP assay for rapid and sensitive detection of SARS-CoV-2 in clinical and sewage samples from Pakistan. Virus Research. 302, 198484 (2021).
- Donia, A., et al. Integration of RT-LAMP and microfluidic technology for detection of SARS-CoV-2 in wastewater as an advanced point-of-care platform. Food and Environmental Virology. 14, 364-373 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten