Method Article
粒状ハイドロゲル生体材料内および粒状ハイドロゲル生体材料内への三次元細胞移動の定量化
要約
ここでは、粒状ハイドロゲルの界面内および界面への3D細胞遊走を定量的に評価するためのプロトコールを紹介します。
要約
粒状ハイドロゲルスキャフォールドは、再生医療において大きな可能性を秘めており、細胞送達の担体として、または組織統合のインターフェースとして機能します。この記事では、粒状ハイドロゲル内および粒状ハイドロゲルへの細胞移動を定量化するための2つの新しいアプローチを紹介し、これらのスキャフォールドの明確なアプリケーションに焦点を当てます。まず、組織を顆粒状ハイドロゲルに成長させて統合する細胞単層界面アッセイを紹介します。次に、ハイドロゲルマトリックス内の細胞の動きを追跡するように設計されたスフェロイドベースのアッセイについて説明し、特に細胞送達を含むアプリケーションに適しています。どちらの方法も、細胞遊走の正確で制御された測定を可能にし、粒状ハイドロゲルスキャフォールドを利用する研究者に包括的なツールキットを提供します。これらの方法の動機は、特定のアプリケーションに合わせて、スキャフォールド内の細胞移動をオーダーメイドで制御する必要性に起因しています。これらの定量技術を最適化および標準化することで、研究者は粒状ハイドロゲルの特性を反復的に改良し、さまざまな再生医療の状況での有効性を確保できます。この堅牢な定量ツールセットは、粒状ハイドロゲルスキャフォールドを強化する新たな機会を提供し、細胞導入と組織統合アプリケーションの両方での使用を促進します。
概要
治療用途向けのバイオマテリアルは、組織統合を研究するために、より複雑で関連性の高い細胞環境モデルへとますます進化しています。生体材料スキャフォールドは、細胞増殖のための三次元(3D)構造を提供し、目的の組織を模倣することを目的としています1,2。三次元細胞培養モデルには、天然マトリックスと合成スキャフォールドが含まれており、ハプトタクティックまたは走化性の手がかり3,4を介して細胞にさらなる複雑さを提供します。従来のハイドロゲル足場は、バルクで架橋され、ナノ多孔質メッシュを生じ、低分子5,6の拡散を可能にするが、修復が必要な組織領域への細胞スケールの移動には分解が必要である7。粒状ハイドロゲルは、生体適合性、不規則な形状に適合する能力、および多くの場合、その注入性8,9により、臨床応用の可能性が高い生体材料のサブセットです。それらのビルディングブロックの性質は、組織浸潤および血管新生を強化するための細胞スケールの多孔性の利点、ならびに細胞挙動10,11,12の不均一な手がかりの追加を可能にするモジュール性を提供する。3Dスキャフォールド内の細胞応答と動きを理解することは、組織統合のための生体材料を使用するすべてのアプリケーションにおいて、生理学的関連性を得るために不可欠です。
しかし、組織の内部成長を3次元で研究することは、定量的な精度で達成するのは難しいことが証明されています。3D環境の複雑さが増すには、細胞の挙動に関する洞察だけでなく、材料条件の最適化も提供できる細胞遊走のin vitroモデルが必要です。以前に発表された3D顆粒足場細胞の移動に関する報告では、局所播種を使用して細胞の挙動を調査し、多孔質構造および細胞形態への浸潤を報告し、他のスフェロイド発芽14,15を報告し、成長長および発芽数を測定している。局所播種移動の長さは、重力の影響を不均一に受ける可能性があり、顕微鏡法の限界により、結果を縦断的にすることはできません。スフェロイド発芽法は、最大投影による2次元定量化に限定されており、制御された侵入のメカニズムを捉えることができません。どちらの方法もXY平面で測定されるため、3D細胞の動きと足場の浸潤を完全に再現するために必要なニュアンスが不足しています。
このプロトコルは、多孔質3D粒状ヒドロゲル足場へのin vitro浸潤など、細胞遊走を定量化するための2つのアプローチ、特に微多孔性アニール粒子(MAP)足場16,17,18,19を使用する方法を説明する。以下の方法の目的は、粒状ゲルの移動の方向性を制御することにより、粒状ゲル中の細胞挙動を3次元解析のために研究することです。最初の単層ベースの上昇移動アッセイ(MAMA)アプローチは、均一な細胞-材料相互作用を示し、細胞が粒状ハイドロゲルとインターフェースする初期環境を表し、足場に浸潤する前に個々の行動を分離するためのプラットフォームとして機能する内因性細胞統合の簡略化されたモデルです。2つ目は、並行層型外向きスフェロイド移動アッセイ(PLOSMA)法と呼ばれ、複雑な足場環境に完全に囲まれた状態での細胞の動きを調査し、送達後の細胞の動きと、細胞が顆粒状ゲルに完全に入った後の動きをモデル化する3D細胞スフェロイド移動アッセイです。
どちらの方法も3D画像解析によって定量化でき、細胞の動きの促進または制限が設計基準内にある再生医療および組織工学アプリケーションの縦断的時点を使用した材料と細胞の相互作用の研究と最適化に適用できます。さらに、これらの方法は、均一なマルチウェルアッセイ調製のためのプレート遠心分離を利用します。
プロトコル
本試験で使用した試薬および機器の詳細は、 資料表に記載されています。
1.粒状ヒドロゲル調製
注:このプロトコール全体で使用される MAP 粒子は、45.88 mg/mL PEG-MAL(10 kDa)、0.82 mg/mL RGD、8.06 mg/mL MethMal19、および 5.62 mg/mL MMP-2 分解性架橋剤を含む 3.2 wt% w/v ゲルです。ゲルの機械的剛性は、真皮の剛性17と一致するように15〜20kPaである。
- 粒状のハイドロゲル粒子を生成し、通常どおり細胞培養の準備をします。
注:このプロトコルは、Roosaら18によって製造が詳述されている微多孔性アニール粒子ゲルの無菌調製について説明しています。 - 顆粒状ヒドロゲル粒子を in vitro で使用するために、70%イソプロピルアルコールを3回洗浄した後、滅菌1x PBSを3回洗浄して滅菌します。
- LAP粉末を超純水に溶解し、溶液を0.22μmの滅菌フィルターに通すことにより、培地溶液中の滅菌0.2 mMリチウムフェニル-2,4,6-トリメチルベンゾイルホスフィン酸(LAP)を調製します。この溶液を1:1 v / v/vで加え、実験を行うのに必要な量のゲルを加えます。
- 0.2 mM LAP、ゲル、および培地溶液を37°Cでチューブローテーター上で20 rpmで少なくとも30分間インキュベートしてから、マイクロ粒子全体にLAPを拡散させます。
- 30分が経過したら、粒子懸濁液を18,000 x g で25°Cで5分間遠心分離します。 上清を吸引します。
注:MAP粒子の全体的な乾燥度は、化学的性質、親水性、および粒子サイズの違いにより異なる場合があります。一貫性を保つために、説明されているように粒子懸濁液を遠心分離し、容積式ピペットを使用してチューブ内で粒子を混合し、遠心分離ステップを繰り返すことがベストプラクティスです。
2. Monolayer-based Ascending Migration Assay (MAMA)法:細胞培養とイメージング
注:細胞はFITCチャンネル(488nm)を使用してイメージングしました。細胞に使用した色素は、492 nmで励起され、517 nmで発光しました。10倍の倍率では、4倍の倍率よりもディテールが増加しますが、どちらでも使用できます。
- ヒト真皮線維芽細胞(HDF)は、製造元のプロトコルに従って解凍します。所望の通過が達成されるまで、必要に応じて通過する。一般に、初代細胞はP5を通じて遺伝的および表現型の構成を維持します。
- 96ウェルプレートまたは懸濁液のウェルあたりの推奨培地量を使用して、96ウェルプレートまたは所望のプレートサイズで120,000細胞/cm2 を少なくともn = 6でプレート化し、各添加前に穏やかに吸引します。 図1Aに示すように、細胞を一晩接着させると、翌日には約80%のコンフルエントが得られます。
注:一般的に、センターカラムウェルとローウェルは、最適なゲルの広がりに最適です。著者らは、96ウェルプレート(行B-Gおよび列5-8)に対して最大24ウェルを使用します。 - 上記のようにゲル条件を調製し、LAPでインキュベートしながら、吸引器またはピペットで培地を細胞のウェルプレートから取り出します。プレートの底面を乱さないように注意してください。
- 図1Bに示すメーカーの指示に従って、細胞追跡色素をウェルに添加します。ウェルから細胞追跡色素を吸引する前に、ステップ1で説明したようにゲルが完全に調製されていることを確認してください。
- 各ゲル条件の20 μLを、プレートの底部に触れずにポジティブディスプレイスメント式ピペットでウェルに加えます。
注:最良のアッセイ条件は、ゲルをウェルの中心に直接ピペットで移動するときに発生します。 - 25°Cのプレートスピニングローター遠心分離機アタッチメントを使用して、100 x gで15秒間、加速と減速を8で遠心してゲルを平坦化します。プレートを180°反転させ、図1Cに示すように、ウェル底全体にゲルが均一に分布するように、100 x gで再度15秒間回転させます。
- 集束光(365 nm、34.4 mW/cm2)をサンプルに30秒間印加して足場をアニールし、すべての足場が形成された後に細胞の各ウェルに200 μLの培地を添加することにより、ゲルを上から無菌的に光架橋します(図1D)。細胞を37°Cで30分間インキュベートし、イメージング前に顆粒足場に付着させます。
- 図1Eに要約した移動挙動を捕捉するために、共焦点顕微鏡を使用して細胞を画像化します。細胞が少なくとも 80% コンフルエントであるプレートの領域の最も低い焦点を見つけ、それを Z スタックの下端として設定します。
- 細胞の蛍光シグナルの最高点を見つけ、それをZスタックの上端として設定します。最高の解像度を得るには、5 μm以下のステップサイズを使用してください。
- 細胞単層および非移動細胞の高さを表すために、少なくとも3つのウェルを画像化します。t = 0 の時点は、 図 2A の上面図と側面図の両方から示されています。イメージング終了後、一晩インキュベートします。
注:これらの細胞は4倍対物レンズでイメージングされ、曝露はすべてのウェルで一貫していました。 - t = 24時間で、細胞は、 図2B の上面図および側面図から見られるように、顆粒足場を通って上昇し始めます。同じパラメータを使用して、t = 0 hに対して実行したイメージング手順を繰り返します。ステージの以前の高さを参照として使用するか、まだ移行されていないセルを見つけて、それを Z スタックの下端として設定します。
- すべてのウェルに対して手順を繰り返し、分析を容易にするために各ウェルが個別の画像として保存されるようにします。
注:タイムポイントは、実験上の制約および細胞追跡蛍光の方法により、より長いタイムポイントでイメージングされる場合があります。 - 足場は、追加の指標のために固定して染色することができます。所望の終了時点で、ピペットでウェルから培地を吸引し、廃棄します。各ウェルを200 μLの滅菌PBSで5分間ずつ2回、穏やかに洗浄します。200 μLの4%パラホルムアルデヒド(PFA)を20分間加え、吸引して廃棄します。ウェルはすぐに染色するか、1x PBSで4°Cで最大1週間保存できます。
3. Monolayer-based Ascending Migration Assay (MAMA)法:3D画像解析
- バッチ変換の場合は、IMARIS画像変換ソフトウェアを開きます。顕微鏡画像を変換ソフトウェアにドラッグアンドドロップし、インポートするソフトウェアArena内のフォルダーを選択します。 [すべて開始] を押します。変換された画像は、アリーナに.imsファイルとして表示されます。
注:ボクセルサイズが画像メタデータに含まれていない場合は、個々の共焦点イメージング仕様を参照して値を見つけてください。あるいは、ボクセルサイズは、イメージング中に使用されるステップサイズとして近似することもできます。 - デスクトップの IMARIS Arenaアイコン をダブルクリックし、Arenaから画像を選択して、ソフトウェアを開きます。
- 画像は、上部のツールバーアイコンパネルにある「3Dビュー」分析タブに自動的にロードされます。メインツールバーの 「Image Proc 」タブをクリックします。
- サイドパネルの左上にある「Channel 1」のドロップダウンメニューをクリックし、「Background Subtraction」を選択します。パネルの下部にある[OK]を押して、「3Dビュー」に戻ります。t = 0 h と t = 24 h の代表的な画像をそれぞれ図 2A,B に示します。
- サイドパネルメニューのすぐ上にある小さなツールバーで、丸みを帯びた青い形状のアイコン「 新しいサーフェスを追加」をクリックして、「サーフェス1」という名前の編集可能なオブジェクトタブを作成します。
- 作成パラメータアルゴリズム設定のインターフェースがメニューパネルの下部に開きます。インターフェースの下部にある 青い矢印ボタンをクリックして 、すべての反復に使用するパラメータを手動で生成します。正しいソースチャンネルが選択されていることを確認し、「スムーズ」ボックスにチェックを入れます。
- 表面の詳細を 0.7 μm に設定し、[ Background Subtraction (Local Contrast)] を選択します。[オブジェクトに収まる最大の球の直径]ボックスに平均セル長を入力します。終了したら、下部にある同じ 青い矢印 を押します。
注:この値は、ツールバーの[スライス]タブを使用して推定し、平均セルの幅を測定できます。
- 表面の詳細を 0.7 μm に設定し、[ Background Subtraction (Local Contrast)] を選択します。[オブジェクトに収まる最大の球の直径]ボックスに平均セル長を入力します。終了したら、下部にある同じ 青い矢印 を押します。
- しきい値処理では、最も明るいセルのみがセグメント化される強度ヒストグラムを決定します。スライサーを使用して、画像スタック内を上下に移動し、可能な限り正確であることを確認します。
- [Split Touching Objects (Region Growing)]で [有効] を選択し、[シード ポイントの直径]を以前使用したのと同じ直径に設定します。[Intensity Based] しきい値が選択されていることを確認し、 青い右矢印ボタンをクリックします。
- 次の 2 つのステップ、フィルター シード ポイントとフィルター サーフェスは、生成されたサーフェスが正確であることを確実にするために、いくつかの測定によって調整できます。ただし、ベースライン分析では、追加のフィルタリングは必要ありません。変更が不要な場合は、 青い矢印 ボタンをクリックしてください。
注:最後のステップでは、目的の出力に応じて表面をさらに分類できます。編集を行った後、 緑色の矢印ボタンをクリックして サーフェスの作成を終了します。 - バッチ分析のために作成パラメータを保存するには、ワンドアイコン「作成」をクリックします。[Batch の Store Parameters (Store Parameters for Batch)] をクリックして名前を付け、[Ok] をクリックします。t = 0 と t = 24 h の代表的な処理済み画像をそれぞれ図 2C,D に示します。
注:選択した「サーフェス」内のすべてのオブジェクトは、表面積、体積、強度、作成された他のサーフェスからの距離など、画像変換ソフトウェアによって与えられるさまざまなプロパティに基づいてセットに分類できます。 - サーフェス プロパティは、[ 統計 ] タブを選択すると表示されます。すべてのセルの高さを収集するには、[ 詳細 ] タブをクリックし、連続するドロップダウン メニューから [特定の値 ] と [位置 Z ] を選択します。1 つの [保存 ] アイコンをクリックして、すべての Z 位置と行った分類を .xls ファイルに保存します。すべての画像に対して繰り返します。
- 代表画像から非移動細胞のz位置の中央値を求め、各試験条件からその数値より下のすべてのz位置をよく減算します。
注:移行値は、非移行セルの高さを超える各テクニカルレプリケート条件の中央値の平均として報告されます。これらは、 図 3A に示すように高さの中央値として報告することも、 図 3B に示すように、t = 0 と比較した目的の時点の移動高さの倍率変化として報告することもできます。
4. Parallel Layered Outward Spheroid Migration Assay(PLOSMA)法:3Dスフェロイドの細胞培養と吊り下げ液滴培養
注:このプロトコルは、Nandiらによって執筆されたプロトコルから適応された細胞培養および吊り下げ培養について説明しています.14。
- メーカーのプロトコルに従ってHDFを解凍します。目的の通過が達成されるまで、必要に応じて通過します。
- 無菌細胞培養フードでは、底部に10 mLのPBSを加え、蓋を裏返して外部が細胞培養フードの上に置かれるようにして、ペトリ皿を調製します。
- 適切な量の細胞(細胞数から決定)を微量遠心チューブに加えます。培地で希釈した細胞追跡色素を添加します。温めた培地で総容量を1mLにします。
注:スフェロイドはスフェロイドあたり約8000個の細胞である必要がありますが、細胞の種類によって変化する可能性があります。 - 細胞溶液を37°Cで45分間インキュベートします。 メーカーが推奨する速度に従って細胞をスピンダウンし、上清を吸引します。
- 培地中の細胞を1:100メチルセルロースに再懸濁します。
- 細胞/培地溶液の液滴20μLをペトリ皿の蓋にピペットで移します。
- 自信を持って、すばやく、慎重に蓋を反転させ、PBSを含むシャーレの下半分の上に置きます。
- 液滴を少なくとも24時間インキュベートします。
注:スフェロイド形成は明視野顕微鏡でモニターできます。
5. Parallel Layered Outward Spheroid Migration Assay (PLOSMA) 法: 粒状ハイドロゲル上に細胞スフェロイドを播種する
注: 次のプロセスを 図 4A にまとめます。
- 図 4A で概説した PLOSMA メソッドを設定するには、ポジティブディスプレイスメント式ピペットを使用して 15 μL のゲルを透明な 96 ウェルプレートのウェルに無菌で添加します。
- プレートスピニングローター遠心分離機アタッチメントを使用して、1000 x g で10秒間遠心してゲルを平坦化します。プレートを180°反転させ、再度1000 x g で10秒間回転させ、ウェル底全体にゲルが均一に分布するようにします。
- 均一な平坦性が得られたら、集光光(365 nm、33.4 mW/cm2)をサンプルに30秒間印加して足場をアニールすることにより、ゲルを上から無菌的にフォトクロスリンクします。
- 吊り下げられた液滴のペトリ皿を無菌組織培養フードに無菌的に移動させ、蓋を反転させます。
- 20 μLのピペットを使用して、スフェロイドがピペットチップに入るまで液滴をゆっくりと取り込みます。ウェルの中央にある足場に液滴を排出します。
- すべてのウェルについて、前の手順を繰り返します。各ウェルにスフェロイドがあることを確認するには、明視野顕微鏡または蛍光顕微鏡で確認します。
- ウェルプレートを37°Cで2時間インキュベートし、スフェロイドが足場に付着できるようにします。
- 各回転楕円体の上に追加の15 μLのゲルをピペットで移します。ゲルが均一に分布するように、プレートを300 x g で各方向に15秒間遠心分離します。
- ゲルの最上層をUV(365 nm)光を用いて33.4 mW/cm2で30秒間アニールします。各足場の上に培地をピペットで固定し、ウェルの総容量を200 μLにします。
注:この時点で足場は非常に乾燥しているため、回転楕円体が外れないように、ウェルの側面にメディアをドロップダウンで追加します。
6. Parallel Layered Outward Spheroid Migration Assay(PLOSMA)法:スフェロイドの共焦点イメージング
メモ: イメージングのしやすさは、イメージングシステムによって異なります。スフェロイドをウェル内の低露光時間で配置します。細胞はFITCチャネル(488 nm)を使用してイメージングしました。細胞に使用した色素は、492 nmで励起され、517 nmで発光しました。10倍の倍率で、4倍の倍率よりもディテールが増します。
- 細胞にまだ焦点が合っている最下段レベル (z 高さ) を見つけます。これを Z スタックの下限として設定します。
- 細胞にまだ焦点が合っている最高ステージレベル (z 高さ) を見つけます。これを Z スタックの上限として設定します。
注:最良のイメージング結果には、細胞スケールの分解能を維持するために5μm以下のステップサイズが含まれます。共焦点顕微鏡システムによっては、イメージング速度と解像度の間にトレードオフがある場合があります。 - t = 0 および 24 h で記述されているすべてのスフェロイドを画像化します。48時間の時点も、実験上の制約に応じて画像化できます。t = 0 と t = 24 の代表的な最大射影画像を 図 4B に示します。
7. Parallel Layered Outward Spheroid Migration Assay(PLOSMA)法:3D画像解析
- 手順3.1から3.4で概説されているように、画像を分析ソフトウェアにインポートします。左パネルの右上にある「Channel 1」 のドロップダウンメニュー をクリックし、「 Background Subtraction」を選択します。パネルの下部にある [OK] を押します。
- 3Dビューに戻ったら、ディスプレイ調整ポップアップウィンドウで すべてのチャンネルを自動調整 を押し、必要に応じて修正します。
- サイドメニューのすぐ上にある小さなツールバーで、図5Aに示す「Add new Reference Frame」アイコンと3つの直交する矢印をクリックして、「Reference Frame 1」という新しいタブを追加します。
- 図 5B に示すように、3 つの平面すべてで回転楕円体の中心に原点を移動します。
- 3つの直交する矢印と同じツールバーで、オレンジ色の球のアイコンをクリックし、新しいスポットを追加して「スポット1」というタブを作成します。 青い矢印ボタンを押します。
- [スポット検出] で、[推定 XY 直径] をセルの推定直径に設定します。青い矢印ボタンを押します。
注:HDFの場合、この数は15.0μmです。 - しきい値処理では、最も明るい部分のみを囲むように強度ヒストグラムを調整します。 スライサーを使用して、画像スタック内を上下に移動し、可能な限り正確であることを確認します。 青い次の矢印を押します。
- 緑色の [実行 ]ボタンを押して、解析を終了します。
- [スライサーでレンダリング] のチェックを外すか、セットアップ パネルの右側にある黄色の四角いアイコンをクリックします。
- [統計]タブをクリックします。最初のドロップダウンメニューで、[特定の値]を選択します。2番目のドロップダウンメニューで、[Origin Reference Frameからの距離]を選択します。選択したサーフェスのすべての値が表示されます(図 5Cを参照)。1 つの保存アイコンをクリックすると、.xls ファイルがダウンロードされます。
- 画像と解析に加えた変更を保存するには、メインツールバーの 「保存 」アイコンを押します。 図6A は、24時間後に画像化されたスフェロイドの3Dレンダリングを表し、 図6B は、拡散セルをマーキングするIMARISスポット関数を表し、原点参照フレームからの距離に応じて色分けされています。
- エクスポートしたデータを t = 0 の画像に正規化し、各回転楕円体の移動距離と Z 高さの平均を計算して、各サンプルの 1 つの値を取得します。 図7A、B は、それぞれ各出力の代表的なグラフを示しています。
結果
このプロトコルは、2つの新しい粒状足場移動アッセイに必要なステップを詳述することを目的としています。MAMA法は、組織界面での細胞浸潤を評価するために利用できます。粒状ハイドロゲルは、バルクハイドロゲルよりも複雑なシステムであるため、移動のための処理が本質的に複雑である9,20。図 1 で概説した段階的なプロセスを理解することが重要です。各ステップは次のステップに基づいて構築され、このプロトコルで最適化されています。120,000細胞/cm2の密度でHDFを播種すると、一晩で少なくとも80%のコンフルエントが得られ(図1A)、これらの非蛍光細胞は、イメージングの可能性を最大化するために、実験当日に細胞追跡色素でタグ付けするのが最適です(図1B)。このプロトコールは、細胞の生存率を維持するためにHDF継代で使用される低い遠心力と一致します。1回の遠心分離ステップで生じる角度のため、プレートを180°反転させてゲルがシフトし、ウェルの底面を完全に覆うようにする必要があります(図1C)。アニーリング後30分間インキュベーター内で細胞を回復させると(図1D)、細胞の生存率が維持され、最適な遊走が得られます(図1E)。96ウェルプレートの広い領域を4倍対物レンズでイメージングし、0〜24時間の時点から一致させることができます(図2A、B)細胞の挙動を広く評価できます。結果のZスタック画像を解析ソフトウェアで処理すると、使いやすいインターフェースで複数の大規模データセットの高度な解析が可能になります。このプロトコルは、図 2B、C の代表的な画像で視覚化された各時点におけるセルの高さまたは位置 Z のデータセットを作成する手順をまとめたものです。処理されたデータの分析は、図3Aに見られ、中央値の高さの平均と各時点のそれらの標準偏差を使用して視覚化され、t = 0時間での非移動セルからの折り畳み変化高さが図3Bに示されています。この方法の基礎となるデータは通常、非正規分布であるため、中央値は比較のためのより堅牢な尺度であり、したがってデータの要約に使用されます。
同様に、PLOSMA法は、3D顆粒状ハイドロゲル足場内の送達細胞の運動性を評価するために利用することができます。 図4A は、PLOSMA法の手順を概説しており、ウェルの中央にスフェロイドを播種することが特に重要です。スフェロイドを視野の中央に配置することをお勧めしますが、顕微鏡の能力によります。 図4B は、FITCチャネル(488nm)で10倍の倍率で撮影された、t = 0時間およびt = 24時間のスフェロイドの広がりの代表的な画像を示しています。ソフトウェアでは、原点参照フレームを作成し、各zスタックに合わせて調整できます(図5A、B)。ソフトウェアは、その原点参照フレームからの半径方向の距離を追跡し、目的のデータセットとしてエクスポートできます(図5C)。 図6A はt = 24h回転楕円体の3Dレンダリングの代表的な画像を示し、 図6B はソフトウェアのスポット機能を示しています。処理されたデータの例を 図 7 に示します。 図 7A は、中心から移動した平均距離を Day 0 の距離に正規化したものを表しています。 図7B は、PLOSMA法が研究しようとしている方向であるため、z平面だけで移動した距離を分離しています。
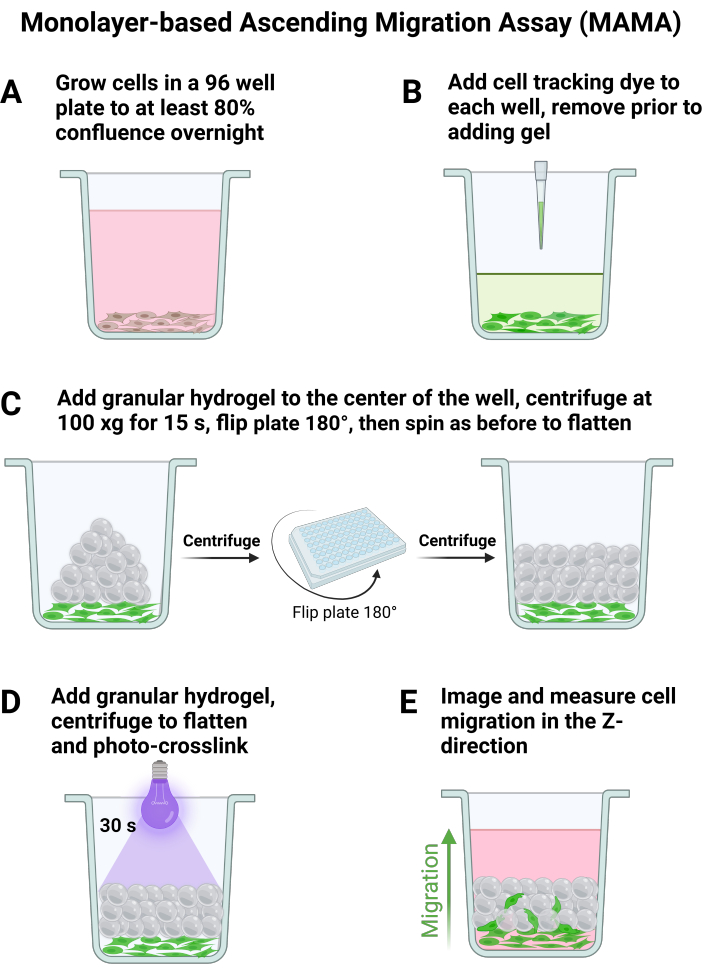
図1:単層ベースのAscending Migration Assay細胞培養とイメージング。 MAMAの細胞およびゲルプロセッシングにおける主要なステップの概略図。(A)細胞を一晩でコンフルエントするまで増殖させ、(B)細胞追跡色素を顆粒状ゲルを添加する直前に添加します。足場は、(C)プレート遠心分離によって組み立てられ、(D)光架橋で安定化されます。複数の時点でのイメージングにより、(E)上向きの細胞移動を視覚化できます。この図の拡大版を表示するには、ここをクリックしてください。
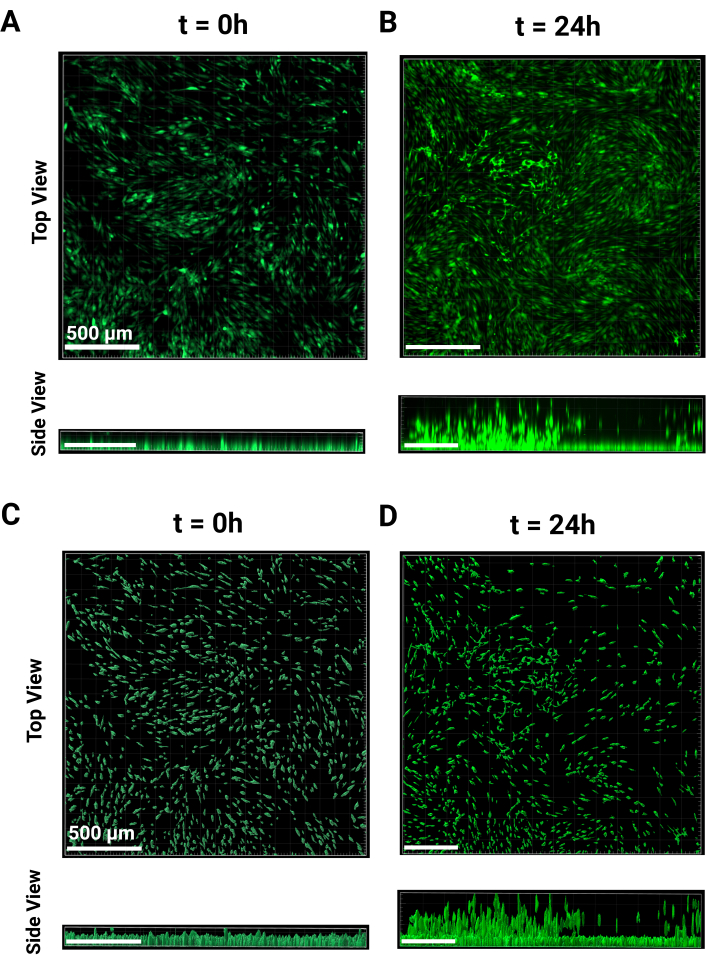
図2:MAMA画像処理。 画像処理の代表的な画像です。FITCチャンネルの(A)t = 0および(B)t = 24hの生の共焦点画像を4倍倍で上面図と側面図で比較。(B)処理されたセル位置Zの高さを(C)t = 0および(D)t = 24時間で上面図と側面図で比較。24 時間の処理には、移動しない Z 高さの中央値の減算が含まれます。スケールバー = 500 μm。略語:MAMA = MonolayerベースのAscending Migration Assay。 この図の拡大版を表示するには、ここをクリックしてください。
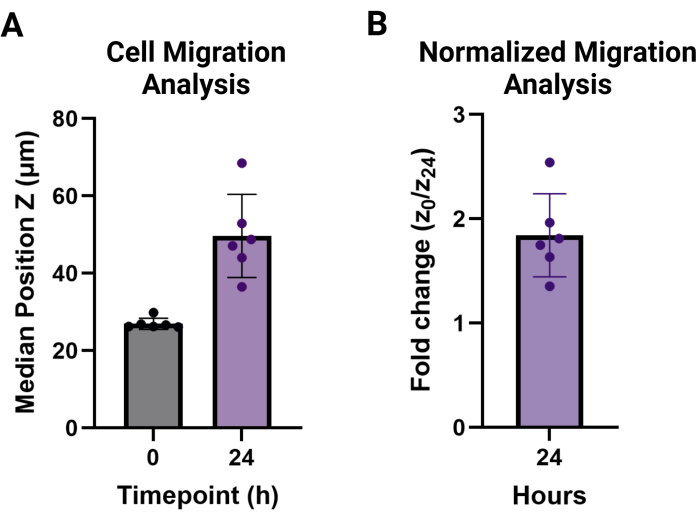
図3:MAMA細胞遊走出力解析。 (A)各反復における細胞高さの中央値Zおよび標準偏差(n = 6)t=0(27.0μm±1.4μm)およびt=24時間(46.6μm±10.8μm)における。(B)24時間での細胞の移動を0時間に正規化し、倍率変化(1.8 ± 0.4)として報告しました。 この図の拡大版を表示するには、ここをクリックしてください。
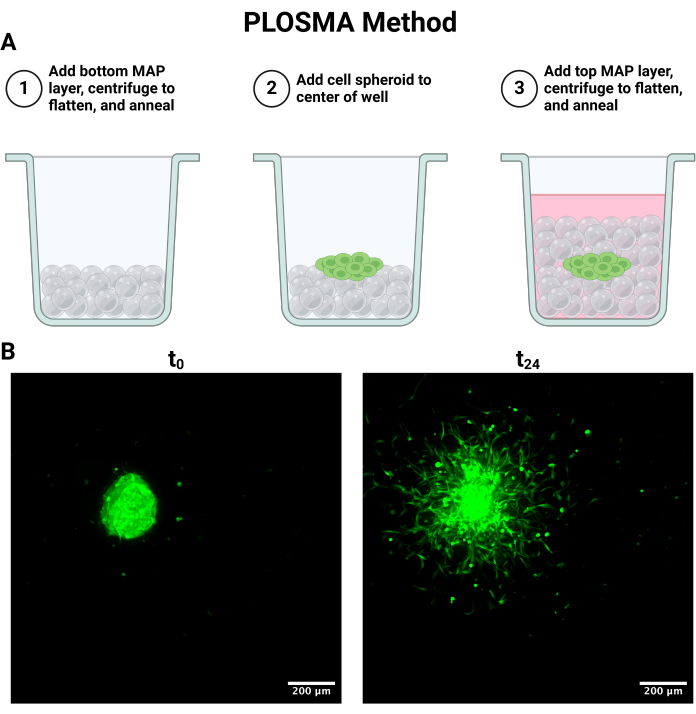
図4:Parallel Layer Outward Spheroid Migration Assay(PLOSMA)細胞培養とイメージング(A)スキャフォールドレイヤリングの手順を説明する概略図。(B)0時間および24時間で撮影された回転楕円体の最大強度投影。画像は、FITCチャネル(488 nm)の共焦点蛍光顕微鏡法で10倍の倍率で撮影しました。スケールバー = 200 μm。略語:PLOSMA = Parallel Layer Outward Spheroid Migration Assay。この図の拡大版を表示するには、ここをクリックしてください。
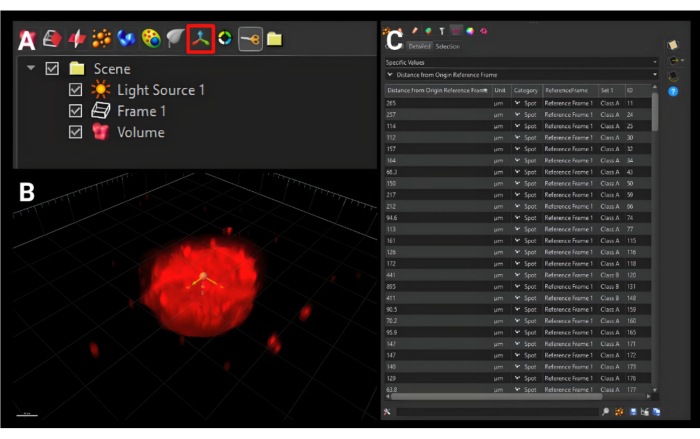
図5:新しい原点参照フレームの作成。 (A)赤いボックスで囲まれた新しい原点参照フレームボタン。(B) 新しい原点は、3次元すべてで回転楕円体の中心に設定されます。(C)表示される出力メトリックは、原点参照フレームからのセル表面の距離であり、セルが中心からどれだけ移動したかを示します。スケールバー = 120 μm. この図の拡大版を表示するには、ここをクリックしてください。
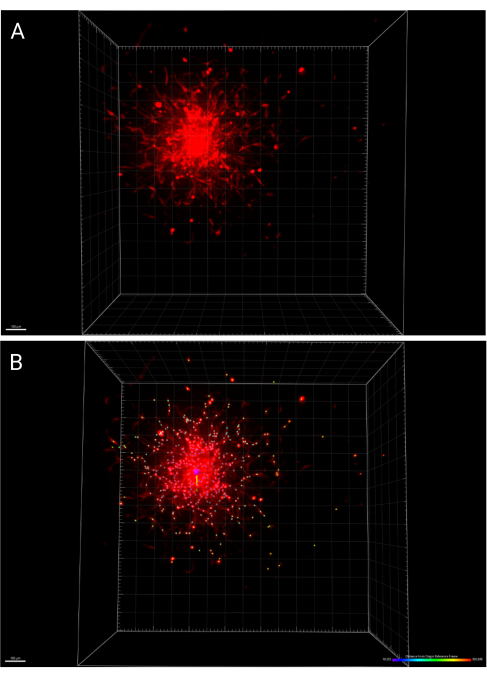
図6:24時間後の埋め込み回転楕円体の3Dレンダリング(A)3D空間で処理された回転楕円体。(B)IMARISの原点参照系機能を用いて同一スフェロイドの中心を決定し、原点からの距離により細胞の広がりを色分けしています。この図の拡大版を表示するには、ここをクリックしてください。
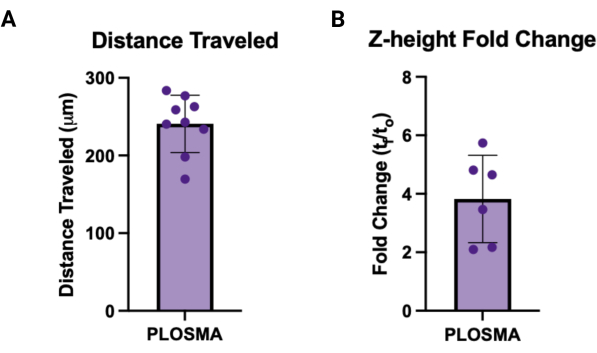
図7:PLOSMAの出力例 (A) 移動距離をμmで示すPLOSMAの結果の例。平均移動距離は240.8μm±36.87μmであった。平均フォールドチェンジは3.82±1.495でした。この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
このプロトコルでは、創傷治癒と組織統合のための細胞遊走を3Dで特徴付けるための2つのin vitroモデルについて説明します。最初のモデルである単分子膜ベースの移動アッセイは、適切に付着したコンフルエントな細胞に依存しています。このプロトコールは、線維芽細胞タイプで開発され、1,20,000細胞/ cm2の播種密度で最適化されました。この密度により、細胞はウェルプレートの底部全体で少なくとも80%のコンフルエントまで一晩で増殖することができます。この手順により、少なくとも 24 時間以内に z 方向への移動が保証されます。ゲル層を添加した際のコンフルエント度が低すぎると、細胞が組織培養プラスチック全体およびゲル内に広がり続ける可能性があり、その結果、最適化中に観察された不均一で遅い移動パターンが生じる可能性があります。不均一な移動高さは、密度の低い細胞の領域では、80%のコンフルエントでも観察される場合があります。Well replicatesは、これらの細胞の挙動のノイズを低減します。過度にコンフルエントな細胞は、遠心分離期間中に細胞が浮き上がり、細胞死を引き起こす可能性があります。このばらつきは、一貫した数の細胞で播種し、適切なデータ比較を可能にするために一貫した画像領域をキャプチャすることで対処されます。著者の知る限り、プレート遠心分離はゲルの平坦化については発表されていませんが、遠心分離は細胞の継代と生体物質の取り扱いに一般的に使用されています21,22。継代速度に合わせて速度を調整することで、細胞の生存率を維持し、さらに最適な細胞プロセッシングを行うことができます。
この方法の主な課題は、イメージングの解像度と深度を最大化しながら、イメージング時間を最小限に抑えて最適な分析を確保することです。グリーンセルトラッキング色素は、5 μm以下のステップサイズで1000 msまでの露光時間で96ウェルをイメージングするのに十分な明るさです。曝露時間を短縮すると、細胞がインキュベーション条件にない時間が短縮されますが、分解能も低下します。これらのパラメータは個々の顕微鏡ごとに最適化する必要がありますが、1つの研究内ですべての画像が同じ設定でキャプチャされるようにすることで、ばらつきが減少します。
MAMAの解析における重要な注意点は、移動細胞のみが統計的検定の対象となるようにするために、単層の高さ以下の細胞を排除する必要があることです。したがって、リピートウェルの中央値は、フィルタリング後の細胞位置の非ガウス分布の性質により報告されます。グループ間の比較はヒストグラムで視覚化でき、中央値はノンパラメトリック検定で統計的に分析できます。
これらの課題にもかかわらず、単分子膜ベースの上方移動法は、最も単純な場合、多孔質足場の3D細胞浸潤のための再現性のあるアッセイです。細胞遊走のメカニズム効果を研究するには、パラメーターが研究対象の細胞タイプに適合していることを確認してください。これには、ゲル内または培地中に走化性成分またはハプトタクティック成分を添加することが含まれる場合があります。ヒト経皮線維芽細胞完全培地には遊走性ケモカインが含まれますが、より特異的な手がかりを使用する他の細胞タイプでは、それに応じてアッセイを適応させる必要があります。このアッセイは、複数のタイプの変数をテストするのに適しています。ただし、これらの範囲は、このプロトコルではカバーされていません。MAMAは、 生体内でバルク組織から注入された多孔質ハイドロゲルへの細胞移動に類似した生理学的に適切な環境を提供します。
PLOSMA法では、スフェロイドをスキャフォールドの中心に配置することが、イメージングを成功させ、3次元で意味のある細胞移動を行うために重要です。ゲルの中心にスフェロイドが正確に播種されるかどうかは、ユーザーによって異なります。この目的のために、ユーザーの非利き手でピペットをバレルに固定すると、センタリングが容易になり、播種位置の有効性は明視野顕微鏡または蛍光顕微鏡を使用して確認できます。中心から外れたスフェロイドは、同じ足場または新しい足場で新しい回転楕円体を使用して2回目の試行を行うことで修正できます。このため、著者らは、必要以上に多くのスフェロイドを作成し、必要以上に多くのMAPゲルを調製することを推奨しています。
第2層の遠心分離ステップでは、スフェロイドが(1)ゲルで均一に覆われ、(2)ゲル内で上向きおよび下向きに均一に広がることができることが保証されます。これは、送達された細胞を研究するために重要です。遠心分離はまた、回転楕円体をウェルの中心から端に向かって移動させることがあり、このプロトコルは、遠心分離ステップと各層(15μL)に使用されるゲルの量を均一に分布させることによりこの現象を制限しますが、その動きを完全に排除するわけではありません。スフェロイドの動きを減らすために必要な正確な遠心分離速度とタイミングは、遠心分離機のモデルに応じて調整する必要がある場合があります。ただし、このプロトコルで説明されている仕様は、個々の最適化のベンチマークとして使用できます。別のアプローチは、スフェロイドを2時間のインキュベーション時間で足場に付着させてから、ゲルの第2層を添加することです。スフェロイドの動きは、両方の戦略が実施されている場合に特によく緩和されます。最後に、多段階の遠心分離プロセスのため、この方法は強度の低い細胞株には適していない可能性があります。
PLOSMA法でのスフェロイドのめっきのロジスティクスとは別に、画像取得には制限があります。スフェロイドは4倍または10倍の倍率でイメージングできますが、最良の結果を得るには、少なくとも10倍の倍率を使用し、zスタックのステップサイズを2〜5μmに縮小します。倍率は、研究全体を通じて一貫している必要があります。イメージング時間は分解能が高くなると増加するため、各ウェルプレート内のサンプル数(プレートあたり4〜8ウェル)を制限して、インキュベーターの外での時間を最小限に抑えます。ライブイメージングのセットアップは、追跡を改善し、より深い洞察を提供することもできます。
粒状ハイドロゲルは、固有の体積、多孔性、機械的強度、場合によっては生物活性など、独自のトポロジーと設計パラメータを持っているため、これらの側面に関連する細胞の挙動を可能な限り忠実に研究する必要があります。PLOSMA法は、送達後または細胞が顆粒ゲルに完全に入った後の細胞の動きをモデル化するように設計されています。細胞は粒状ハイドロゲル形状に固有の細孔を通って移動することを余儀なくされるため、PLOSMA法は細胞の挙動に影響を与えるものとして多孔性を効果的に分離します。このアッセイの潜在的な用途は、特に創傷治癒空間23における顆粒足場内での細胞送達および顆粒足場内での組織統合である。
どちらのプロトコルも、組織の修復およびリモデリングにおける線維芽細胞の移動の役割により、初代ヒト真皮線維芽細胞を用いて開発されました4,24、ただし、接着細胞の遊走挙動は、多孔質足場の変化に応答して測定することができます - 成長因子の添加およびゲルの表面/バルク組成を含む。これらの変更により、顕著な結果を得るためには、これらのアッセイの調整が必要になる場合があります。さらに最適化が必要なパラメータには、細胞播種密度、実験時間、および/または解析パイプラインが含まれます。IMARISは、細胞移動解析に利用される強力なイメージング解析ツールであり、ここで概説されている以上の機能を備えており、選択した「表面」内のすべてのオブジェクトを、表面積、体積、強度、他の作成された表面からの距離などのさまざまな特性に基づいてセットに分類するなど、ここで概説されている機能を備えています。さらなる分析方法を決定するための多くのオンラインリソースがあります。
ここで概説する2つの方法は、生理学的な方法で顆粒状物質への組織導入の初期状態に対処するだけでなく、材料内に完全に埋め込まれたときのその後の細胞応答にも対処します。すべての遊走アッセイと同様に、存在する細胞は移動と並行して増殖することができますが、記載されているアッセイのデザインは増殖を妨げないため、分析に過度の影響を与えません。どちらの方法も、PFA固定を使用して細胞骨格、コラーゲン沈着、増殖などの指標を検出する縦断的イメージングに加えて、エンドポイント染色にも対応しています。概説された方法の使用は、以前の方法1,6,14,15,25,26,27とは対照的に、細胞浸潤を測定可能なパラメータとして利用する3D細胞移動のより正確な時空間表現に向かっています。
開示事項
著者には、開示すべき利益相反はありません。
謝辞
この研究の資金は、米国国立衛生研究所の高優先度短期プロジェクト賞(1R56DK126020-01)とKurtin Trustからの慈善寄付を通じて部分的に支援されました。J.T.は、全米科学財団大学院研究フェローシップから資金提供を受けました。BioRender.com で作成された図の概略図。
資料
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 647 Phalloidin | ThermoFisher | A22287 | |
| Bovine Serum Albumin | VWR International | 332 | |
| CellTracker Green CMFDS Dye, 1 mg | ThermoFisher | C2925 | 20 x 50 ug units, 492/517 nm |
| Centrifuge | ThermoFisher | 75016085 | ST Plus Series |
| Clear 96 well plate | MilliPore Sigma | CLS3997-50EA | |
| Dimethyl Sulfoxide | Fisher Scientific | MT-25950CQC | 250 mL |
| Fibroblast Basal Medium | ATCC | PCS-201-030 | 480 mL, phenol-red-free |
| Fibroblast Growth Kit - Low Serum | ATCC | PCS-201-041 | 7.5 mM L-glut,5 ng/mL rh FGF basic, 5 ug/mL rh Insulin, 1 ug/mL Hydrocortisone, 50 ug/mL Ascorbic acid, 2% FBS |
| FIJI (ImageJ) | NIH | Public access download | |
| Human Dermal Fibroblasts | ATCC | PCS-201-012 | Adult human dermal fibroblasts |
| ImageXpress Micro Confocal | Molecular Devices | Spinning Disc confocal microscope with 4x, 10x magnifications | |
| IMARIS | Oxford Instruments | 3/4D Imaged Visualizaiton and Analysis Software, Proprietary | |
| Incubator | ThermoFisher | Finnpipette F2 Variable volume Pipettes | HeraCell Vios 160i CO2 Incubator, 165L |
| M-20 Microplate Swinging Bucket Rotor | ThermoFisher | 75003624 | |
| Methylcellulose | Fisher Scientific | 9004-67-5 | Lab grade, powder form |
| Microcentrifuge tube | Fisherbrand | 05-408-129 | 1.5 mL microcentrifuge tubes |
| Paraformaldehyde (4%) | Alfa Aesar | AAJ19943K2 | For fixing |
| Petri dish | Corning | 08-757-100A | Bacteriological Petri Dishes with Lid 35 x 10 mm |
| Pipettes | ThermoFisher | 4642080 | Finnpipette F2 Variable volume Pipettes |
| Sterile PBS | Gibco | 10010-023 | |
| Triton-X | Fisher Scientific | 327371000 |
参考文献
- Jerka, D., et al. Unraveling endothelial cell migration: Insights into fundamental forces, inflammation, biomaterial applications, and tissue regeneration strategies. ACS Appl Bio Mater. 7 (4), 2054-2069 (2024).
- Edmondson, R., Broglie, J. J., Adcock, A. F., Yang, L. Three-dimensional cell culture systems and their applications in drug discovery and cell-based biosensors. Assay Drug Dev Technol. 12 (4), 207-218 (2014).
- Yamada, K. M., Sixt, M. Mechanisms of 3D cell migration. Nat Rev Mol Cell Biol. 20 (12), 738-752 (2019).
- Grinnell, F. Fibroblast biology in three-dimensional collagen matrices. Trends Cell Biol. 13 (5), 264-269 (2003).
- Kloxin, A. M., Kloxin, C. J., Bowman, C. N., Anseth, K. S. Mechanical properties of cellularly responsive hydrogels and their experimental determination. Adv Mater. 22 (31), 3484-3494 (2010).
- Solbu, A. A., et al. Assessing cell migration in hydrogels: An overview of relevant materials and methods. Mater Today Bio. 18, 100537 (2023).
- Madl, C. M., Katz, L. M., Heilshorn, S. C. Tuning bulk hydrogel degradation by simultaneous control of proteolytic cleavage kinetics and hydrogel network architecture. ACS Macro Lett. 7 (11), 1302-1307 (2018).
- Daly, A. C., Riley, L., Segura, T., Burdick, J. A. Hydrogel microparticles for biomedical applications. Nat Rev Mater. 5 (1), 20-43 (2020).
- Qazi, T. H., Burdick, J. A. Granular hydrogels for endogenous tissue repair. Biomater Biosyst. 1, 100008 (2021).
- Tanner, G. I., Schiltz, L., Narra, N., Figueiredo, M. L., Qazi, T. H. Granular hydrogels improve myogenic invasion and repair after volumetric muscle loss. Adv Healthc Mater. 25, e2303576 (2024).
- Sussman, E. M., Halpin, M. C., Muster, J., Moon, R. T., Ratner, B. D. Porous implants modulate healing and induce shifts in local macrophage polarization in the foreign body reaction. Ann Biomed Eng. 42 (7), 1508-1516 (2014).
- Roosa, C. A., et al. Conjugation of IL-33 to microporous annealed particle scaffolds enhances type 2-like immune responses in vitro and in vivo. Adv Healthc Mater. 13 (25), 2400249 (2024).
- Jaberi, A., et al. Engineering microgel packing to tailor the physical and biological properties of gelatin methacryloyl granular hydrogel scaffolds. Adv Healthc Mater. 13 (25), 2402489 (2024).
- Nandi, S., Brown, A. C. Characterizing cell migration within three-dimensional in vitro wound environments. J Vis Exp. 126, e56099 (2017).
- Qazi, T. H., et al. Anisotropic rod-shaped particles influence injectable granular hydrogel properties and cell invasion. Adv Mater. 34 (12), 2109194 (2022).
- Griffin, D. R., Weaver, W. M., Scumpia, P. O., Di Carlo, D., Segura, T. Accelerated wound healing by injectable microporous gel scaffolds assembled from annealed building blocks. Nat Mater. 14 (7), 737-744 (2015).
- Pruett, L. J., Jenkins, C. H., Singh, N. S., Catallo, K. J., Griffin, D. R. Heparin microislands in microporous annealed particle scaffolds for accelerated diabetic wound healing. Adv Funct Mater. 31 (35), 2104337 (2021).
- Roosa, C., et al. Microfluidic synthesis of microgel building blocks for microporous annealed particle scaffold. J Vis Exp. (184), e64119 (2022).
- Pfaff, B. N., et al. Selective and improved photoannealing of microporous annealed particle (MAP) scaffolds. ACS Biomater Sci Eng. 7 (2), 422-427 (2021).
- Riley, L., Schirmer, L., Segura, T. Granular hydrogels: Emergent properties of jammed hydrogel microparticles and their applications in tissue repair and regeneration. Curr Opin Biotechnol. 60, 1-8 (2019).
- Jo, C. H., Roh, Y. H., Kim, J. E., Shin, S., Yoon, K. S. Optimizing platelet-rich plasma gel formation by varying time and gravitational forces during centrifugation. J Oral Implantol. 39 (5), 525-532 (2013).
- Mironov, V., et al. Fabrication of tubular tissue constructs by centrifugal casting of cells suspended in an in situ. crosslinkable hyaluronan-gelatin hydrogel. Biomaterials. 26 (36), 7628-7635 (2005).
- Vu, L. T., Jain, G., Veres, B. D., Rajagopalan, P. Cell migration on planar and three-dimensional matrices: A hydrogel-based perspective. Tissue Eng Part B Rev. 21 (1), 67-74 (2015).
- Cialdai, F., Risaliti, C., Monici, M. Role of fibroblasts in wound healing and tissue remodeling on Earth and in space. Front Bioeng Biotechnol. 10, 958381 (2022).
- Qazi, T. H., Muir, V. G., Burdick, J. A. Methods to characterize granular hydrogel rheological properties, porosity, and cell invasion. ACS Biomater Sci Eng. 8 (4), 1427-1442 (2022).
- Shaik, R., et al. Cardiac matrix-derived granular hydrogel enhances cell function in 3D culture. ACS Appl Mater Interfaces. 16 (43), 58346-58356 (2024).
- Puiggalí-Jou, A., Asadikorayem, M., Maniura-Weber, K., Zenobi-Wong, M. Growth factor-loaded sulfated microislands in granular hydrogels promote hMSCs migration and chondrogenic differentiation. Acta Biomater. 166, 69-84 (2023).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved