Method Article
Quantifizierung der dreidimensionalen Zellmigration innerhalb und in granuläre Hydrogel-Biomaterialien
In diesem Artikel
Zusammenfassung
Ein Protokoll zur quantitativen Bewertung der 3D-Zellmigration innerhalb und in die Grenzfläche von granulären Hydrogelen wird hier vorgestellt.
Zusammenfassung
Granuläre Hydrogel-Gerüste bergen ein erhebliches Potenzial in der regenerativen Medizin, da sie entweder als Träger für die Zellabgabe oder als Schnittstellen für die Gewebeintegration fungieren. In diesem Artikel werden zwei neuartige Ansätze zur Quantifizierung der Zellmigration innerhalb und in granulare Hydrogele vorgestellt und die unterschiedlichen Anwendungen dieser Gerüste hervorgehoben. Zunächst wird ein Zell-Monolayer-Interface-Assay vorgestellt, der das Gewebewachstum in granulare Hydrogele zu Integrationszwecken simuliert. Zweitens wird ein sphäroidbasierter Assay beschrieben, der entwickelt wurde, um die Zellbewegung innerhalb der Hydrogelmatrix zu verfolgen, der speziell für Anwendungen mit Zellabgabe geeignet ist. Beide Methoden ermöglichen präzise und kontrollierte Messungen der Zellmigration und bieten Forschern, die granulare Hydrogel-Gerüste verwenden, ein umfassendes Toolkit. Die Motivation für diese Methoden ergibt sich aus der Notwendigkeit einer maßgeschneiderten Kontrolle der Zellmigration innerhalb des Gerüsts, um sie an spezifische Anwendungen anzupassen. Durch die Optimierung und Standardisierung dieser Quantifizierungstechniken können Forscher die Eigenschaften von granularen Hydrogelen iterativ verfeinern und so ihre Wirksamkeit in verschiedenen Kontexten der regenerativen Medizin sicherstellen. Dieses robuste Set an quantitativen Werkzeugen bietet neue Möglichkeiten zur Verbesserung von granularen Hydrogel-Gerüsten und fördert deren Einsatz sowohl bei der Zellverabreichung als auch bei der Gewebeintegration.
Einleitung
Biomaterialien für therapeutische Anwendungen entwickeln sich zunehmend hin zu komplexeren und relevanteren Modellen von Zellumgebungen, um die Gewebeintegration zu untersuchen. Biomaterial-Gerüste bieten eine dreidimensionale (3D) Struktur für das Zellwachstum und zielen darauf ab, ein gewünschtes Gewebe nachzuahmen 1,2. Dreidimensionale Zellkulturmodelle umfassen natürliche Matrizen und synthetische Gerüste, die den Zellen über haptotaktische oder chemotaktische Signale weitere Komplexität verleihen 3,4. Herkömmliche Hydrogel-Gerüste sind in großen Mengen vernetzt, wodurch ein nanoporöses Netz entsteht, das die Diffusion kleiner Moleküle ermöglicht 5,6, aber für die Migration im Zellmaßstab in einen reparaturbedürftigen Gewebebereich einen Abbau erfordert7. Granuläre Hydrogele sind eine Untergruppe von Biomaterialien, die aufgrund ihrer Biokompatibilität, ihrer Fähigkeit, sich an unregelmäßige Formen anzupassen, und in vielen Fällen ihrer Injektionsfähigkeit ein hohes Potenzial für die klinische Umsetzung aufweisen 8,9. Ihre Bausteinnatur bietet den Vorteil der Porosität auf zellischer Ebene, um die Gewebeinfiltration und Angiogenese zu verbessern, sowie die Modularität, die die Hinzufügung heterogener Signale für das Zellverhalten ermöglicht 10,11,12. Das Verständnis der Zellreaktion und -bewegung innerhalb eines 3D-Gerüsts ist entscheidend für die physiologische Relevanz in allen Anwendungen, bei denen Biomaterialien für die Gewebeintegration verwendet werden.
Die dreidimensionale Untersuchung des Einwachsens von Gewebe hat sich jedoch als schwierig erwiesen, dies mit quantitativer Genauigkeit zu erreichen. Die erweiterte Komplexität einer 3D-Umgebung erfordert in vitro Modelle der Zellmigration, die nicht nur Einblicke in das Zellverhalten, sondern auch die Optimierung des Materialzustands liefern können. Zuvor veröffentlichte Berichte über die Migration von 3D-granularen Gerüstzellen haben die topische Aussaat verwendet, um das Zellverhalten zu untersuchen, die Infiltration in die poröse Struktur und die Zellmorphologie13 und andere sphäroide Keimungen 14,15 zu berichten und die Länge des Auswuchses und die Anzahl der Sprossen zu messen. Die Migrationslängen der topischen Aussaat können durch die Schwerkraft ungleichmäßig beeinflusst werden, und aufgrund der Einschränkungen der Mikroskopie können die Ergebnisse nicht longitudinal sein. Die Methode der Sphäroidskeimung war bisher auf die 2D-Quantifizierung mittels maximaler Projektion beschränkt, die den Mechanismus der kontrollierten Invasion nicht erfassen kann. Beide Methoden werden in einer xy-Ebene gemessen, der die notwendige Nuance fehlt, um die 3D-Zellbewegung und die Gerüstinfiltration vollständig zu rekapitulieren.
Dieses Protokoll beschreibt zwei Ansätze zur Quantifizierung der Zellmigration, wie z. B. die Infiltration in die porösen 3D-körnigen Hydrogel-Gerüste, insbesondere unter Verwendung der Gerüste 16,17,18,19 mit mikroporösen geglühten Partikeln(MAP). Der Zweck der folgenden Methoden besteht darin, das Zellverhalten in granularen Gelen zu untersuchen, indem die Richtungsabhängigkeit ihrer Migration für die dreidimensionale Analyse kontrolliert wird. Der erste, auf Monolayern basierende Ansatz des aufsteigenden Migrationsassays (MAMA) ist ein vereinfachtes Modell der endogenen Zellintegration, das einheitliche Zell-Material-Wechselwirkungen veranschaulicht und als Plattform dient, um die anfängliche Umgebung darzustellen, in der Zellen mit granularen Hydrogelen interagieren, sowie um individuelles Verhalten vor der Infiltration des Gerüsts zu isolieren. Die zweite, die sogenannte PLOSMA-Methode (Parallel Layered Outward Spheroid Migration Assay), ist ein 3D-Zell-Sphäroidmigrationsassay, der die Zellbewegung untersucht, wenn sie vollständig von einer komplexen Gerüstumgebung umgeben ist, und die Zellbewegung nach der Verabreichung sowie die Bewegung nach dem vollständigen Eintritt der Zellen in ein granulares Gel modelliert.
Beide Methoden sind durch 3D-Bildanalyse quantifizierbar und können zur Untersuchung und Optimierung von Material-Zell-Wechselwirkungen unter Verwendung von Längszeitpunkten für regenerative Medizin und Tissue Engineering-Anwendungen angewendet werden, bei denen die Förderung oder Einschränkung der Zellbewegung innerhalb der Designkriterien liegt. Darüber hinaus nutzen diese Methoden die Vorteile der Plattenzentrifugation für eine einheitliche Multi-Well-Assay-Vorbereitung.
Protokoll
Die Einzelheiten zu den Reagenzien und der in der Studie verwendeten Ausrüstung sind in der Materialtabelle aufgeführt.
1. Zubereitung des körnigen Hydrogels
HINWEIS: MAP-Partikel, die in diesem Protokoll verwendet werden, bestehen aus 3,2 Gew.-% w/v Gel mit 45,88 mg/mL PEG-MAL (10 kDa), 0,82 mg/ml RGD, 8,06 mg/ml MethMal19 und 5,62 mg/ml MMP-2 abbaubarem Vernetzer. Die mechanische Steifigkeit des Gels beträgt 15-20 kPa und entspricht der dermalen Steifigkeit17.
- Erzeugen Sie körnige Hydrogelpartikel und bereiten Sie die Zellkultur wie gewohnt vor.
HINWEIS: Dieses Protokoll beschreibt die sterile Herstellung von mikroporösem geglühtem Partikelgel, dessen Herstellung von Roosa et al.18 detailliert beschrieben wird. - Bereiten Sie die körnigen Hydrogelpartikel für die In-vitro-Anwendung vor, indem Sie sie mit drei Waschgängen mit 70%igem Isopropylalkohol sterilisieren, gefolgt von drei Waschgängen mit sterilem 1x PBS.
- Bereiten Sie ein steriles 0,2 mM Lithiumphenyl-2,4,6-trimethylbenzoylphosphinat (LAP) in einer Medienlösung vor, indem Sie LAP-Pulver in Reinstwasser auflösen und die Lösung durch einen 0,22 μm Sterilfilter leiten. Fügen Sie diese Lösung 1:1 v/v mit der Menge an Gel hinzu, die für die Durchführung des Experiments erforderlich ist.
- Inkubieren Sie das 0,2 mM LAP, das Gel und die Medienlösung bei 37 °C auf einem Röhrchenrotator bei 20 U/min für mindestens 30 Minuten, bevor Sie fortfahren, um eine Diffusion von LAP durch die Mikropartikel zu ermöglichen.
- Nach Ablauf von 30 min wird die Partikelsuspension bei 18.000 x g 5 min bei 25 °C zentrifugiert. Aspirieren Sie den Überstand.
HINWEIS: Die Gesamttrockenheit von MAP-Partikeln kann aufgrund von Unterschieden in der Chemie, Hydrophilie und Partikelgröße variieren. Aus Gründen der Konsistenz empfiehlt es sich, die Partikelsuspension wie beschrieben zu zentrifugieren, die Partikel im Röhrchen mit einer Verdrängerpipette zu mischen und dann den Zentrifugationsschritt zu wiederholen.
2. Monolayer-based Ascending Migration Assay (MAMA) Methode: Zellkultur und Bildgebung
HINWEIS: Die Zellen wurden mit dem FITC-Kanal (488 nm) abgebildet. Der für die Zellen verwendete Farbstoff hatte eine Anregung bei 492 nm und eine Emission bei 517 nm. Die 10-fache Vergrößerung bietet mehr Details als die 4-fache Vergrößerung, aber beide können verwendet werden.
- Tauen Sie humane dermale Fibroblasten (HDFs) gemäß dem Protokoll des Herstellers auf. Durchgang nach Bedarf, bis der gewünschte Durchgang erreicht ist; Im Allgemeinen behalten Primärzellen ihre genetische und phänotypische Zusammensetzung durch P5 bei.
- Platte 120.000 Zellen/cm³ 2 für mindestens n = 6 in einer 96-Well-Platte oder der gewünschten Plattengröße unter Verwendung des empfohlenen Medienvolumens pro Vertiefung für die Suspension, wobei vor jeder Zugabe vorsichtig aspiriert wird. Lassen Sie die Zellen über Nacht anheften, wie in Abbildung 1A gezeigt, was am nächsten Tag zu einer Konfluenz von etwa 80 % führt.
HINWEIS: Im Allgemeinen eignen sich die Vertiefungen in der mittleren Säule und Reihe am besten für eine optimale Gelverteilung. Die Autoren verwenden bis zu 24 Wells für eine 96-Well-Platte (Zeilen B-G und Spalten 5-8). - Bereiten Sie die Gelbedingungen wie oben beschrieben vor und entfernen Sie während der Inkubation in LAP das Medium mit einem Aspirator oder einer Pipette aus der Well-Platte der Zellen. Achten Sie darauf, den Boden der Platte nicht zu stören.
- Geben Sie den Zellverfolgungsfarbstoff gemäß den Anweisungen des Herstellers in die Vertiefungen, wie in Abbildung 1B dargestellt. Stellen Sie sicher, dass das Gel vollständig wie in Schritt 1 beschrieben vorbereitet ist, bevor Sie den Zellverfolgungsfarbstoff aus den Vertiefungen aspirieren.
- Geben Sie 20 μl jeder Gelbedingung mit einer Verdrängerpipette in die Vertiefungen, ohne den Boden der Platte zu berühren.
HINWEIS: Die besten Assay-Bedingungen treten auf, wenn das Gel direkt in die Mitte der Vertiefung pipettiert wird. - Mit einem Plattenspinnrotor-Zentrifugenaufsatz bei 25 °C wird 15 s lang bei 100 x g mit einer Beschleunigung und Verzögerung von 8 geschleudert, um das Gel zu glätten. Drehen Sie die Platte um 180° und drehen Sie sie erneut 15 s lang mit 100 x g , um eine gleichmäßige Gelverteilung über den Well-Boden zu gewährleisten, wie in Abbildung 1C zu sehen ist.
- Aseptisch vernetzen Sie das Gel von oben (Abbildung 1D), indem Sie 30 s lang fokussiertes Licht (365 nm, 34,4 mW/cm 2) auf die Probe aufbringen, um das Gerüst zu glühen, und fügen Sie 200 μl Medium zu jeder Vertiefung der Zellen hinzu, nachdem alle Gerüste gebildet wurden. Lassen Sie die Zellen 30 Minuten lang bei 37 °C inkubieren, damit sie sich vor der Bildgebung an das körnige Gerüst anheften können.
- Um das in Abbildung 1E zusammengefasste Migrationsverhalten zu erfassen, bilden Sie Zellen mit einem konfokalen Mikroskop ab. Ermitteln Sie den niedrigsten Fokuspunkt für einen Bereich der Platte, in dem die Zellen zu mindestens 80 % zusammenfließen, und legen Sie ihn als unteren Rand des Z-Stapels fest.
- Suchen Sie den höchsten Punkt des Fluoreszenzsignals der Zelle und legen Sie ihn als obere Kante des Z-Stapels fest. Verwenden Sie eine Schrittweite von 5 μm oder kleiner, um die beste Auflösung zu erzielen.
- Stellen Sie mindestens drei Vertiefungen dar, um die Zellmonoschicht und die nicht migrierenden Zellhöhen darzustellen. Der Zeitpunkt t = 0 wird sowohl in der Draufsicht als auch in der Seitenansicht in Abbildung 2A dargestellt. Nach Abschluss der Bildgebung über Nacht inkubieren.
HINWEIS: Diese Zellen wurden mit einem 4-fach-Objektiv abgebildet, und die Belichtung wurde für alle Vertiefungen konstant gehalten. - Bei t = 24 h beginnen die Zellen durch das körnige Gerüst aufzusteigen, wie in Abbildung 2B aus der Draufsicht und der Seitenansicht zu sehen ist. Wiederholen Sie die Bildgebungsschritte, wie sie für t = 0 h mit denselben Parametern durchgeführt wurden. Verwenden Sie die vorherige Höhe der Bühne als Referenz, oder suchen Sie nach Zellen, die noch nicht migriert wurden, und legen Sie diese als untere Kante des Z-Stapels fest.
- Wiederholen Sie die Schritte für alle Wells und stellen Sie sicher, dass jedes Well als separates Bild gespeichert wird, um die Analyse zu vereinfachen.
HINWEIS: Zeitpunkte können je nach experimentellen Einschränkungen und Methode der Zellverfolgungsfluoreszenz für längere Zeitpunkte abgebildet werden. - Gerüste können für zusätzliche Metriken fixiert und gebeizt werden. Saugen Sie das Medium zum gewünschten Endzeitpunkt mit einer Pipette aus den Vertiefungen an und entsorgen Sie es. Waschen Sie jede Vertiefung vorsichtig mit 200 μl sterilem PBS zweimal für jeweils 5 Minuten. 20 μl 4 % Paraformaldehyd (PFA) für 20 min zugeben, dann aspirieren und verwerfen. Die Vertiefungen können sofort gefärbt oder bei 4 °C in 1x PBS bis zu einer Woche gelagert werden.
3. Monolayer-based Ascending Migration Assay (MAMA) Methode: 3D-Bildanalyse
- Öffnen Sie die Software, indem Sie auf das Desktop-Symbol der IMARIS Arena doppelklicken und ein Bild aus der Arena auswählen.
- Das Bild wird automatisch in die Analyseregisterkarte "3D-Ansicht" geladen, die in der Symbolleiste oben zu sehen ist. Klicken Sie auf die Registerkarte Image Proc in der Hauptsymbolleiste.
- Klicken Sie oben links im Seitenbereich auf das Dropdown-Menü für Kanal 1 und wählen Sie Hintergrundsubtraktion. Drücken Sie OK am unteren Rand des Bedienfelds, um zur "3D-Ansicht" zurückzukehren. Repräsentative Bilder für t = 0 h und t = 24 h sind in Abbildung 2A,B zu sehen.
- Klicken Sie in der kleinen Symbolleiste direkt über dem Seitenbereichsmenü auf das Symbol mit den abgerundeten blauen Formen, Neue Oberflächen hinzufügen, um eine Registerkarte für bearbeitbare Objekte mit dem Namen "Oberflächen 1" zu erstellen.
- Eine Schnittstelle für die Einstellungen des Erstellungsparameters und des Algorithmus öffnet sich am unteren Rand des Menübereichs. Generieren Sie manuell die Parameter, die für alle Replikate verwendet werden sollen, indem Sie auf die blaue Pfeilschaltfläche am unteren Rand der Benutzeroberfläche klicken. Stellen Sie sicher, dass der richtige Quellkanal ausgewählt ist, und aktivieren Sie das Kontrollkästchen "Glatt".
- Stellen Sie die Oberflächendetails auf 0,7 μm ein, und wählen Sie Hintergrundsubtraktion (Lokaler Kontrast). Geben Sie die durchschnittliche Zellenlänge in das Feld "Durchmesser der größten Kugel, die in das Objekt passt" ein. Drücken Sie den gleichen blauen Pfeil unten, wenn Sie fertig sind.
HINWEIS: Dieser Wert kann über die Registerkarte "Segment" in der Symbolleiste und durch Messen der Breite der durchschnittlichen Zellen geschätzt werden.
- Stellen Sie die Oberflächendetails auf 0,7 μm ein, und wählen Sie Hintergrundsubtraktion (Lokaler Kontrast). Geben Sie die durchschnittliche Zellenlänge in das Feld "Durchmesser der größten Kugel, die in das Objekt passt" ein. Drücken Sie den gleichen blauen Pfeil unten, wenn Sie fertig sind.
- Bestimmen Sie für die Schwellenwertbestimmung das Intensitätshistogramm, in dem nur die hellsten Zellen segmentiert sind. Bewegen Sie sich mit dem Slicer im Bildstapel auf und ab, um sicherzustellen, dass er so genau wie möglich ist.
- Wählen Sie Aktivieren für "Berührende Objekte teilen (Bereich wachsen)" und stellen Sie den Durchmesser der Kernpunkte auf den gleichen Durchmesser wie zuvor ein. Stellen Sie sicher, dass der Schwellenwert "Intensitätsbasiert" ausgewählt ist, und klicken Sie auf die blaue Schaltfläche mit dem Pfeil nach rechts.
- Die nächsten beiden Schritte, Filter-Seed-Punkte und Filteroberflächen, können durch verschiedene Messungen angepasst werden, um sicherzustellen, dass die generierten Oberflächen genau sind. Für die Baseline-Analyse ist jedoch keine zusätzliche Filterung erforderlich. Wenn keine Änderung erforderlich ist, klicken Sie auf die blaue Pfeilschaltfläche.
HINWEIS: Der letzte Schritt ermöglicht eine weitere Klassifizierung der Oberfläche in Abhängigkeit von der gewünschten Leistung. Nachdem Sie alle Änderungen vorgenommen haben, klicken Sie auf die grüne Pfeilschaltfläche , um die Erstellung der Flächen abzuschließen. - Um die Erstellungsparameter für die Stapelanalyse zu speichern, klicken Sie auf das Zauberstabsymbol Erstellung. Klicken Sie auf Store Parameters for Batch und benennen Sie es, dann klicken Sie auf Ok. Repräsentativ verarbeitete Bilder für t = 0 und t = 24 h sind in den Abbildungen 2C,D dargestellt.
HINWEIS: Alle Objekte innerhalb einer ausgewählten "Oberfläche" können basierend auf verschiedenen Eigenschaften, die von der Bildkonvertierungssoftware vorgegeben werden, wie z. B. Oberfläche, Volumen, Intensität und Entfernung zu anderen erstellten Oberflächen, in Sätze eingeteilt werden. - Oberflächeneigenschaften finden Sie, indem Sie auf der Registerkarte Statistik klicken. Um alle Zellenhöhen zu erfassen, klicken Sie auf die Registerkarte Detailliert und wählen Sie Spezifische Werte und Position Z aus den aufeinanderfolgenden Dropdown-Menüs aus. Klicken Sie auf das einzelne Speichern-Symbol , um alle Z-Positionen und alle vorgenommenen Klassifizierungen in einer .xls Datei zu speichern. Wiederholen Sie den Vorgang für alle Bilder.
- Ermitteln Sie die mittlere Z-Position von nicht migrierenden Zellen aus dem repräsentativen Bild, und subtrahieren Sie alle Z-Positionen unterhalb dieser Zahl von jedem Testbedingungs-Well.
HINWEIS: Die Migrationswerte werden als Mittelwert der Mediane für jede technische Replikationsbedingung oberhalb der nicht migrierenden Zellenhöhe angegeben. Sie können als mediane Höhe angegeben werden, wie in Abbildung 3A zu sehen, oder als Faltenänderung der Migrationshöhe für den gewünschten Zeitpunkt im Vergleich zu t = 0, wie in Abbildung 3B zu sehen.
4. PLOSMA-Methode (Parallel Layered Outward Spheroid Migration Assay): Zellkultur und hängende Tröpfchenkultur für 3D-Sphäroide
HINWEIS: Dieses Protokoll beschreibt Zellkulturen und hängende Tropfenkulturen, die an das von Nandi et al.14 verfasste Protokoll angepasst sind.
- HDFs gemäß dem Protokoll des Herstellers auftauen. Durchgang nach Bedarf, bis der gewünschte Durchgang erreicht ist.
- Bereiten Sie in einer aseptischen Zellkulturhaube eine Petrischale vor, indem Sie 10 ml PBS auf den Boden geben und den Deckel so umklappen, dass die Außenseite auf der Zellkulturhaube aufliegt.
- Geben Sie das entsprechende Zellvolumen (bestimmt anhand der Zellzahl) in ein Mikrozentrifugenröhrchen. Geben Sie einen in Medium verdünnten Zellverfolgungsfarbstoff hinzu. Bringen Sie das Gesamtvolumen mit erwärmtem Medium auf 1 ml.
HINWEIS: Sphäroide sollten etwa 8000 Zellen pro Sphäroid betragen, können sich jedoch je nach Zelltyp ändern. - Inkubieren Sie die Zelllösung 45 Minuten lang bei 37 °C. Schleudern Sie die Zellen entsprechend der vom Hersteller empfohlenen Geschwindigkeit herunter und saugen Sie den Überstand an.
- Resuspendieren Sie Zellen in 1:100 Methylcellulose in Medien.
- Pipettieren Sie 20 μl Tröpfchen der Zell-/Medienlösung auf den Deckel der Petrischale.
- Drehen Sie den Deckel sicher, schnell und vorsichtig um und legen Sie ihn auf die untere Hälfte der Petrischale, die PBS enthält.
- Inkubieren Sie die Tröpfchen mindestens 24 Stunden lang.
HINWEIS: Die Sphäroidbildung kann durch Hellfeldmikroskopie überwacht werden.
5. PLOSMA-Methode (Parallel Layered Outward Spheroid Migration Assay): Aussaat von Zellsphäroiden auf granuläres Hydrogel
HINWEIS: Der folgende Prozess ist in Abbildung 4A zusammengefasst.
- Um die in Abbildung 4A beschriebene PLOSMA-Methode einzurichten, geben Sie aseptisch 15 μl Gel mit einer Direktverdrängerpipette in die Vertiefungen in einer durchsichtigen 96-Well-Platte.
- Mit einem Plattenspinnrotor-Zentrifugenaufsatz 10 s lang bei 1000 x g drehen, um das Gel zu glätten. Drehen Sie die Platte um 180° und drehen Sie sie erneut 10 s lang mit 1000 x g , um eine gleichmäßige Gelverteilung auf dem Vertiefungsboden zu gewährleisten.
- Sobald eine gleichmäßige Planlage erreicht ist, wird das Gel von oben aseptisch photovernetzt, indem 30 s lang fokussiertes Licht (365 nm, 33,4 mW/cm2) auf die Probe angewendet wird, um das Gerüst zu glühen.
- Schieben Sie die Petrischale mit den hängenden Tröpfchen aseptisch in die aseptische Gewebekulturhaube und drehen Sie den Deckel um.
- Nehmen Sie mit einer 20-μl-Pipette langsam ein Tröpfchen auf, bis das Sphäroid in die Pipettenspitze gelangt. Werfen Sie den Tropfen auf das Gerüst in der Mitte der Vertiefung.
- Wiederholen Sie die vorherigen Schritte für alle Wells. Stellen Sie sicher, dass jede Vertiefung ein Sphäroid enthält, indem Sie dies entweder mit Hellfeld- oder Fluoreszenzmikroskopie bestätigen.
- Inkubieren Sie die Well-Platte 2 Stunden lang bei 37 °C, damit die Sphäroide am Gerüst befestigt werden können.
- Pipettieren Sie weitere 15 μl Gel auf jedes Sphäroide. Um eine gleichmäßige Gelverteilung zu gewährleisten, zentrifugieren Sie die Platte bei 300 x g für 15 s in jede Richtung.
- Die oberste Gelschicht wird 30 s lang mit UV-Licht (365 nm) bei 33,4 mW/cm2 geglüht. Pipettieren Sie das Medium auf jedes Gerüst, um das Gesamtvolumen der Vertiefung auf 200 μl zu bringen.
HINWEIS: Gerüste sind zu diesem Zeitpunkt sehr trocken, fügen Sie also tropfenweise Medien an der Seite des Bohrlochs hinzu, um ein Ablösen des Sphäroids zu vermeiden.
6. PLOSMA-Methode (Parallel Layered Outward Spheroid Migration Assay): Konfokale Bildgebung von Sphäroiden
HINWEIS: Wie einfach die Bildgebung ist, hängt vom Bildgebungssystem ab. Platzieren Sie das Sphäroid bei geringer Belichtungszeit in der Vertiefung. Die Zellen wurden mit dem FITC-Kanal (488 nm) abgebildet. Der für die Zellen verwendete Farbstoff hatte eine Anregung bei 492 nm und eine Emission bei 517 nm. Die 10-fache Vergrößerung bietet mehr Details als bei 4-facher Vergrößerung.
- Ermitteln Sie die unterste Bühnenebene (z-Höhe), auf der Zellen noch scharf sind. Legen Sie dies als untere Grenze des Z-Stacks fest.
- Ermitteln Sie die höchste Stufenebene (z-Höhe), auf der Zellen noch scharf sind. Legen Sie dies als Obergrenze des Z-Stapels fest.
HINWEIS: Die besten Bildgebungsergebnisse umfassen eine Schrittweite von weniger als oder gleich 5 μm, um die Auflösung im Zellmaßstab beizubehalten. Je nach konfokalem Mikroskopsystem kann es zu Kompromissen zwischen Bildgebungsgeschwindigkeit und Auflösung kommen. - Bilden Sie alle Sphäroide wie beschrieben bei t = 0 und 24 h ab. Abhängig von experimentellen Einschränkungen kann auch ein 48-Stunden-Zeitpunkt abgebildet werden. Repräsentative maximale Projektionsbilder von t = 0 und t = 24 sind in Abbildung 4B zu sehen.
7. PLOSMA-Methode (Parallel Layered Outward Spheroid Migration Assay): 3D-Bildanalyse
- Importieren Sie Bilder in die Analysesoftware, wie in den Schritten 3.1 bis 3.4 beschrieben. Klicken Sie in der oberen rechten Ecke des linken Bereichs auf das Dropdown-Menü für Kanal 1 und wählen Sie Hintergrundsubtraktion. Drücken Sie unten im Bedienfeld auf OK .
- Sobald Sie wieder in der 3D-Ansicht sind, drücken Sie im Popup-Fenster für die Anzeigeanpassung auf Alle Kanäle automatisch anpassen und korrigieren Sie nach Bedarf.
- Klicken Sie in der kleineren Symbolleiste direkt über dem Seitenmenü auf das Symbol "Neuen Referenzrahmen hinzufügen ", das in Abbildung 5A mit drei orthogonalen Pfeilen gezeigt wird, um eine neue Registerkarte mit dem Namen "Referenzrahmen 1" hinzuzufügen.
- Verschieben Sie den Ursprung in allen drei Ebenen in die Mitte des Sphäroids, wie in Abbildung 5B dargestellt.
- Klicken Sie in der gleichen Symbolleiste wie die drei orthogonalen Pfeile auf das Symbol mit den orangefarbenen Kugeln und fügen Sie neue Spots hinzu, um eine Registerkarte mit dem Namen "Spots 1" zu erstellen. Drücken Sie die blaue Pfeiltaste.
- Legen Sie unter Spot-Erkennung den geschätzten XY-Durchmesser auf den geschätzten Durchmesser der Zellen fest. Drücken Sie die blaue Pfeiltaste.
HINWEIS: Für HDFs beträgt diese Zahl 15,0 μm. - Passen Sie für die Festlegung des Schwellenwerts das Intensitätshistogramm so an, dass nur die hellsten Teile eingekreist werden. Bewegen Sie sich mit dem Slicer im Bildstapel auf und ab, um sicherzustellen, dass er so genau wie möglich ist. Klicken Sie auf den blauen Pfeil Weiter.
- Klicken Sie auf die grüne Schaltfläche Ausführen , um die Analyse abzuschließen.
- Deaktivieren Sie Auf Slicer rendern oder klicken Sie auf das gelbe quadratische Symbol auf der rechten Seite des Setup-Fensters.
- Klicken Sie auf die Registerkarte Statistiken . Wählen Sie im ersten Dropdown-Menü die Option Spezifische Werte aus. Wählen Sie im zweiten Dropdown-Menü die Option Abstand vom Ursprungsreferenzrahmen aus. Es werden alle Werte für die ausgewählten Flächen angezeigt, wie in Abbildung 5C dargestellt. Klicken Sie auf das Symbol für das einzelne Speichern , um eine .xls Datei herunterzuladen.
- Speichern Sie die am Bild und den Analysen vorgenommenen Änderungen, indem Sie auf das Symbol Speichern in der Hauptsymbolleiste klicken. Abbildung 6A zeigt ein 3D-Rendering eines Sphäroids, das nach 24 Stunden abgebildet wurde, während Abbildung 6B die IMARIS-Spots-Funktion darstellt, die ausgebreitete Zellen markiert, farbcodiert entsprechend der Entfernung vom Ursprungsreferenzsystem.
- Normalisieren Sie die exportierten Daten auf die t = 0-Bilder und berechnen Sie den Mittelwert der zurückgelegten Mobilfunkstrecke und der z-Höhe für jedes Sphäroid, um einen einzelnen Wert für jede Probe zu erhalten. Abbildung 7A,B zeigt repräsentative Diagramme für jeden Ausgang.
Ergebnisse
Dieses Protokoll zielt darauf ab, die notwendigen Schritte für zwei neuartige granulare Gerüstmigrationsassays zu beschreiben. Die MAMA-Methode kann verwendet werden, um die zelluläre Infiltration an einer Gewebegrenzfläche zu bewerten. Granulierte Hydrogele sind ein komplexeres System als Bulk-Hydrogele und daher von Natur aus komplexer zu verarbeiten für die Migration 9,20. Es ist wichtig, den in Abbildung 1 beschriebenen schrittweisen Prozess zu verstehen. Jeder Schritt baut auf dem nächsten auf und wurde in diesem Protokoll optimiert. Das Aussäen von HDFs bei einer Dichte von 120.000 Zellen/cm2 führt über Nacht zu einer Konfluenz von mindestens 80 % (Abbildung 1A), und diese nicht fluoreszierenden Zellen werden am besten am Tag des Experiments mit einem Zellverfolgungsfarbstoff markiert, um das Bildgebungspotenzial zu maximieren (Abbildung 1B). Dieses Protokoll entspricht der geringeren Zentrifugalkraft, die bei der HDF-Passage verwendet wird, um die Lebensfähigkeit der Zellen zu erhalten. Aufgrund des Winkels, der für einen einzigen Zentrifugationsschritt erzeugt wird, ist es notwendig, die Platte um 180° zu drehen, um sicherzustellen, dass sich das Gel verschiebt und die Bodenfläche der Wells vollständig bedeckt (Abbildung 1C). Wenn sich die Zellen nach dem Glühen 30 Minuten lang im Inkubator erholen können (Abbildung 1D), bleibt die Lebensfähigkeit der Zellen erhalten und führt zu einer optimalen Migration (Abbildung 1E). Ein großer Bereich einer 96-Well-Platte kann mit einem 4-fach-Objektiv abgebildet und von einem Zeitpunkt von 0 bis 24 Uhr angepasst werden (Abbildung 2A,B), um das Zellverhalten umfassend zu beurteilen. Die Verarbeitung der resultierenden Z-Stack-Bilder in der Analysesoftware bietet erweiterte Analysen für mehrere große Datensätze in einer benutzerfreundlichen Oberfläche. Dieses Protokoll fasst die Schritte zum Erstellen von Datensätzen für die Zellenhöhe oder Position Z zu jedem Zeitpunkt zusammen, der mit den repräsentativen Bildern in Abbildung 2B, C visualisiert wird. Die Analyse der verarbeiteten Daten ist in Abbildung 3A dargestellt, visualisiert mit dem Mittelwert der Medianhöhen und ihrer Standardabweichung für jeden Zeitpunkt, und die Faltenänderungshöhe von nicht migrierenden Zellen bei t = 0 h ist in Abbildung 3B dargestellt. Die dieser Methode zugrunde liegenden Daten sind in der Regel nicht normalverteilt, so dass Mediane robustere Vergleichsmaße sind und daher zur Zusammenfassung der Daten verwendet werden.
Ebenso kann die PLOSMA-Methode verwendet werden, um die Motilität von abgegebenen Zellen innerhalb eines 3D-körnigen Hydrogel-Gerüsts zu bewerten. Abbildung 4A zeigt die Schritte zur PLOSMA-Methode, und es ist besonders wichtig, das Sphäroid in der Mitte des Wells zu säen. Es wird empfohlen, das Sphäroid im Sichtfeld zu zentrieren, hängt aber von den Fähigkeiten des Mikroskops ab. Abbildung 4B zeigt repräsentative Bilder der Sphäroidausbreitung für t = 0 h und t = 24 h, aufgenommen bei 10-facher Vergrößerung im FITC-Kanal (488 nm). In der Software kann ein Ursprungsreferenzrahmen erstellt und an jeden Z-Stapel angepasst werden (Abbildung 5A, B). Die Software kann den radialen Abstand von diesem Ursprungsreferenzrahmen verfolgen und als gewünschten Datensatz exportieren (Abbildung 5C). Abbildung 6A zeigt ein repräsentatives Bild des 3D-Renderings des Sphäroids t = 24 h, während Abbildung 6B die Spots-Funktion der Software zeigt. Ein Beispiel für die verarbeiteten Daten ist in Abbildung 7 dargestellt. Abbildung 7A zeigt die durchschnittliche Entfernung, die vom Zentrum zurückgelegt wurde, normiert auf die Entfernungen von Tag 0. Abbildung 7B isoliert die zurückgelegten Entfernungen nur in der Z-Ebene, da dies die Richtung ist, die die PLOSMA-Methode untersuchen soll.
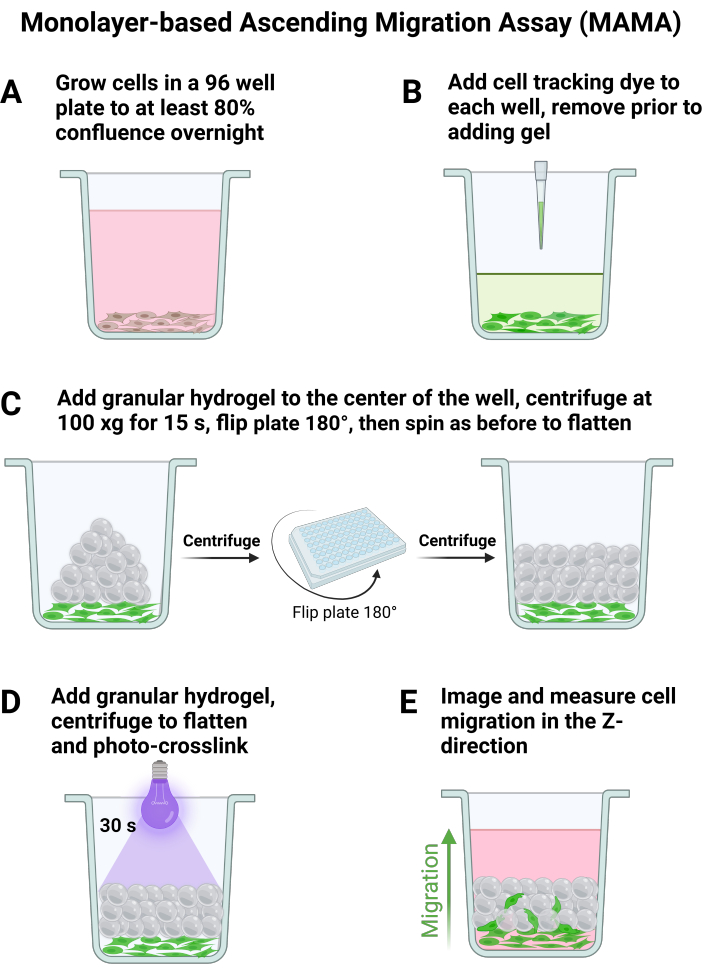
Abbildung 1: Monolayer-basierter Ascending Migration Assay, Zellkultur und Bildgebung. Schematische Darstellung der wichtigsten Schritte in der Zell- und Gelverarbeitung für MAMA. (A) Die Zellen werden über Nacht zur Konfluenz gezüchtet, und (B) der Zellverfolgungsfarbstoff wird kurz vor der Zugabe des körnigen Gels hinzugefügt. Das Gerüst wird mittels (C)-Plattenzentrifugation zusammengebaut und mit (D)-Fotovernetzung stabilisiert. Die Bildgebung zu mehreren Zeitpunkten ermöglicht (E) die Visualisierung der Zellmigration nach oben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
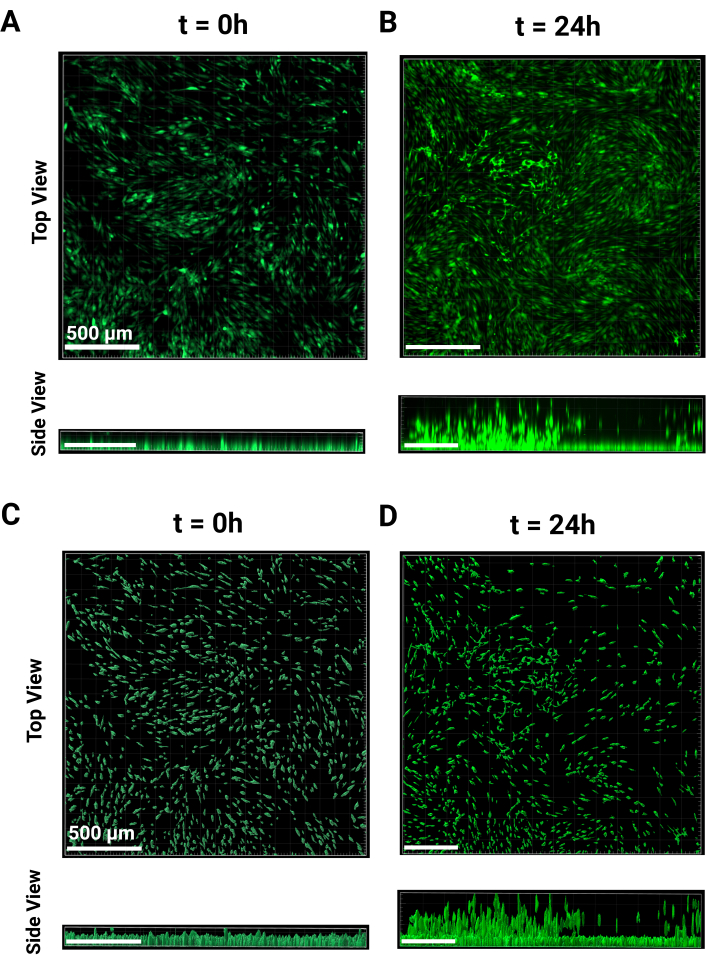
Abbildung 2: MAMA-Bildverarbeitung. Repräsentative Bilder der Bildverarbeitung. Vergleich von konfokalen Rohbildern in der Draufsicht und Seitenansicht bei (A) t = 0 und (B) t = 24 h im FITC-Kanal bei 4-facher Vergrößerung. (B) Vergleich der Draufsicht und Seitenansicht der prozessierten Zellenposition Z Höhen bei (C) t = 0 und (D) t = 24 h. Die Verarbeitung für 24 h umfasst die Subtraktion von medianen, nicht migrierenden Z-Höhen. Maßstabsleisten = 500 μm. Abkürzungen: MAMA = Monolayer-based Ascending Migration Assay. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
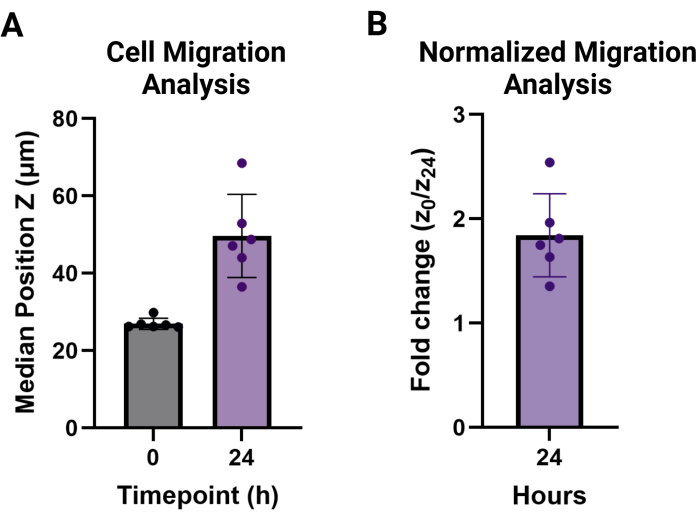
Abbildung 3: MAMA-Zellmigrations-Output-Analyse. (A) Mediane Position Z und Standardabweichung der Zellhöhen in jedem Replikat (n = 6) zu den Zeitpunkten t = 0 (27,0 μm ± 1,4 μm) und t = 24 h (46,6 μm ± 10,8 μm). (B) Migration der Zellen nach 24 h normalisiert auf 0 h und berichtet als Faltenänderung (1,8 ± 0,4). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
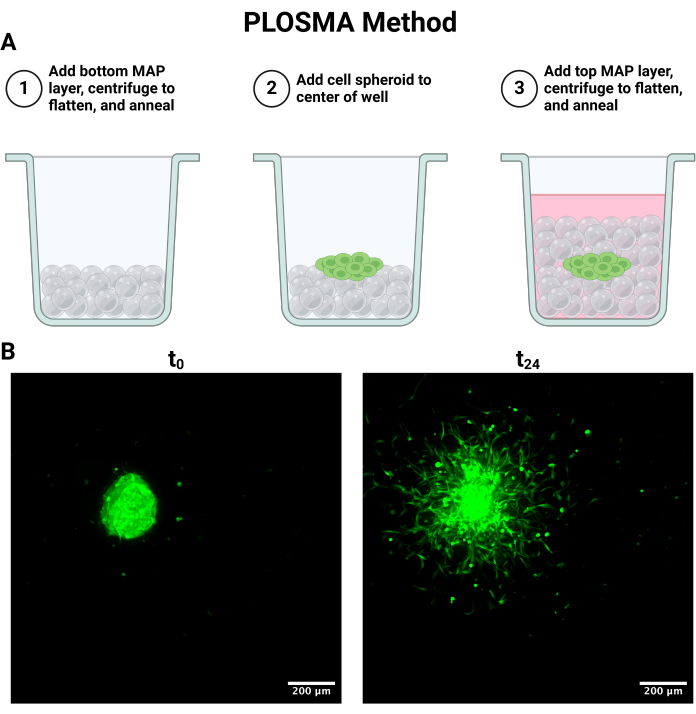
Abbildung 4: Zellkultur und Bildgebung des Parallel Layer Outward Spheroid Migration Assay (PLOSMA). (A) Schematische Darstellung der Schritte der Gerüstschichtierung. (B) Projektionen der maximalen Intensität des Sphäroids, die bei 0 und 24 h aufgenommen wurden. Die Bilder wurden mittels konfokaler Fluoreszenzmikroskopie im FITC-Kanal (488 nm) bei 10-facher Vergrößerung aufgenommen. Maßstabsleisten = 200 μm. Abkürzungen: PLOSMA = Parallel Layer Outward Spheroid Migration Assay. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
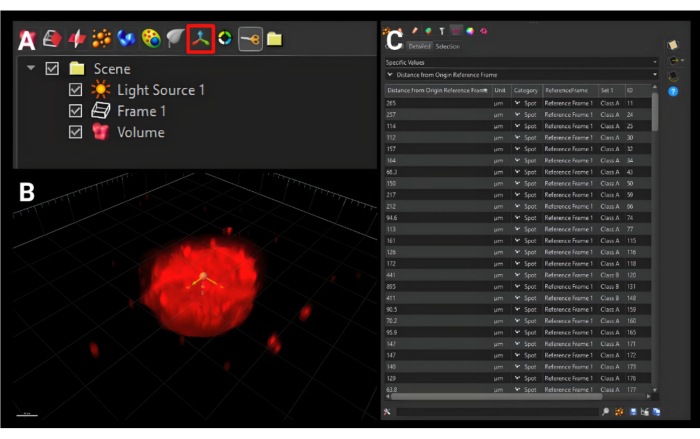
Abbildung 5: Erstellen eines neuen Ursprungsreferenzrahmens. (A) Die Schaltfläche für den neuen Ursprungsreferenzrahmen, die mit einem roten Feld umrandet ist. (B) Der neue Ursprung wird so eingestellt, dass er sich in allen drei Dimensionen in der Mitte des Sphäroids befindet. (C) Bei den angezeigten Ausgabemetriken handelt es sich um die Abstände der Zellenoberflächen vom Ursprungsbezugssystem, das beschreibt, wie weit sich die Zellen vom Mittelpunkt entfernt haben. Maßstabsleiste = 120 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
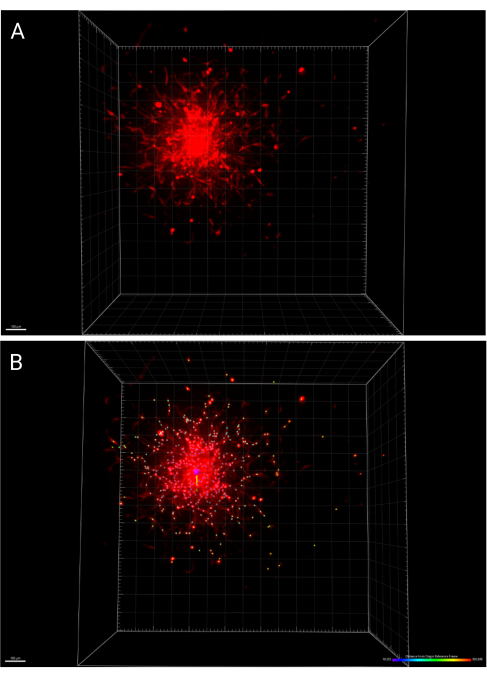
Abbildung 6: 3D-Renderings des eingebetteten Sphäroids nach 24 Stunden. (A) Verarbeitetes Sphäroid im 3D-Raum. (B) Das Zentrum desselben Sphäroids wurde mit der Herkunftsbezugsrahmenfunktion in IMARIS bestimmt, und die Zellausbreitung wird durch die Entfernung vom Ursprung farbcodiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
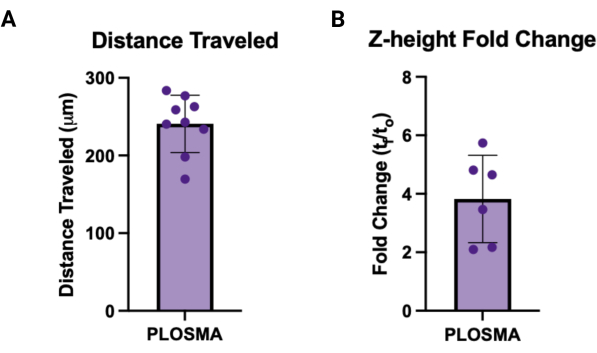
Abbildung 7: Beispielausgaben für PLOSMA. (A) Beispiel für PLOSMA-Ergebnisse, die die zurückgelegte Strecke in μm anzeigen. Die durchschnittlich zurückgelegte Strecke betrug 240,8 μm ± 36,87 μm. (B) Faltenänderung der Z-Höhe (tf/t 0) des Sphäroideskeimens. Die durchschnittliche Fold-Veränderung betrug 3,82 ± 1,495. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Dieses Protokoll beschreibt zwei in vitro Modelle zur Charakterisierung der Zellmigration in 3D für die Wundheilung und Gewebeintegration. Das erste Modell, der Monolayer-basierte Migrationsassay, stützt sich auf richtig angeheftete und konfluente Zellen. Dieses Protokoll wurde mit einem Fibroblasten-Zelltyp entwickelt und bei einer Aussaatdichte von 1.20.000 Zellen/cm2 optimiert. Diese Dichte ermöglicht es den Zellen, über Nacht auf mindestens 80 % Konfluenz gleichmäßig über den Boden der Well-Platte zu wachsen. Dieser Schritt stellt sicher, dass die Migration in z-Richtung innerhalb von mindestens 24 h erfolgt. Wenn die Konfluenz bei der Zugabe der Gelschicht zu gering ist, können sich die Zellen weiterhin über den Gewebekulturkunststoff sowie in das Gel ausbreiten, was zu einem ungleichmäßigen, verlangsamten Migrationsmuster führt, das während der Optimierung beobachtet wurde. Ungleichmäßige Migrationshöhen können in Bereichen mit weniger dichten Zellen immer noch beobachtet werden, selbst bei einer Konfluenz von 80 %. Well-Replikate reduzieren das Rauschen dieser Zellverhaltensweisen. Zu konfluente Zellen können während der Zentrifugationsphase zum Anheben von Zellen und möglicherweise zum Zelltod führen. Diese Variabilität wird durch das Seeding bei einer konsistenten Anzahl von Zellen und durch die Erfassung eines konsistenten Bildbereichs berücksichtigt, um angemessene Datenvergleiche zu ermöglichen. Nach Kenntnis des Autors wurde die Plattenzentrifugation für die Gelabflachung nicht veröffentlicht, aber die Zentrifugation wird häufig für die Zellpassage und den Umgang mit Biomaterie verwendet21,22. Durch die Anpassung der Geschwindigkeit an die Übergabegeschwindigkeit bleibt die Lebensfähigkeit der Zellen für eine weitere optimale Zellverarbeitung erhalten.
Die größte Herausforderung bei dieser Methode besteht darin, die Bildauflösung und -tiefe zu maximieren und gleichzeitig die Bildgebungszeit zu minimieren, um die beste Analyse zu gewährleisten. Der grüne Zellverfolgungsfarbstoff ist hell genug, um ein 96-Well-Modell mit einer Schrittweite von 5 μm oder weniger und einer Belichtungszeit von bis zu 1000 ms abzubilden. Durch die Verkürzung der Belichtungszeit wird die Zeit, in der sich die Zellen nicht in Inkubationsbedingungen befinden, verringert sich jedoch auch die Auflösung. Diese Parameter müssen individuell mikroskopisch optimiert werden, aber die Variabilität wird reduziert, indem sichergestellt wird, dass alle Bilder mit den gleichen Einstellungen innerhalb einer Studie aufgenommen werden.
Ein wichtiger Hinweis für die Analyse von MAMAs ist, dass die Zellen auf oder unter der Monolagenhöhe eliminiert werden müssen, um sicherzustellen, dass nur migrierende Zellen für statistische Tests berücksichtigt werden. Dementsprechend werden die Mediane der Replikatvertiefungen aufgrund der nicht-Gaußschen Verteilungsnatur der Zellpositionen nach der Filterung angegeben. Der Vergleich zwischen den Gruppen kann mit einem Histogramm visualisiert werden, und die Mediane können mit einem nicht-parametrischen Test statistisch analysiert werden.
Trotz dieser Herausforderungen ist die Monolayer-basierte Aufwärtsmigrationsmethode in ihrer einfachsten Form ein reproduzierbarer Assay für die 3D-Zellinfiltration von porösen Gerüsten. Um die mechanistischen Auswirkungen der Zellmigration zu untersuchen, stellen Sie sicher, dass die Parameter zum untersuchten Zelltyp passen. Dies kann die Zugabe von chemotaktischen oder haptotaktischen Komponenten innerhalb des Gels oder in den Medien umfassen. Zu den vollständigen Medien für humane dermale Fibroblasten gehören migratorische Chemokine, aber andere Zelltypen, die spezifischere Signale verwenden, erfordern eine entsprechende Anpassung des Assays. Dieser Assay eignet sich zum Testen mehrerer Arten von Variablen. Der Anwendungsbereich dieser Bestimmungen wird jedoch in diesem Protokoll nicht behandelt. Das MAMA bietet eine physiologisch relevante Umgebung, die der Zellbewegung aus dem Gewebe in ein injiziertes poröses Hydrogel in vivo entspricht.
Für die PLOSMA-Methode ist die Platzierung der Sphäroide in der Mitte des Gerüsts entscheidend für eine erfolgreiche Bildgebung und eine aussagekräftige Zellmigration in drei Dimensionen. Die genaue Aussaat des Sphäroids in der Mitte des Gels ist abhängig vom Anwender. Zu diesem Zweck hilft das Stabilisieren der Pipette am Zylinder mit der nicht dominanten Hand des Benutzers bei der Zentrierung, und die Wirksamkeit der Aussaatposition kann mit Hilfe der Hellfeld- oder Fluoreszenzmikroskopie bestätigt werden. Ein außermittiger Sphäroid kann durch einen zweiten Versuch mit einem neuen Sphäroid behoben werden, entweder auf demselben Gerüst oder auf einem neuen Gerüst. Aus diesem Grund empfehlen die Autoren, mehr Sphäroide als nötig herzustellen und mehr MAP-Gel als nötig herzustellen.
Der zweite Schichtzentrifugationsschritt stellt sicher, dass das Sphäroid (1) gleichmäßig vom Gel bedeckt ist und (2) sich gleichmäßig nach oben und unten in das Gel verteilen kann, was für die Untersuchung der abgegebenen Zellen von entscheidender Bedeutung ist. Die Zentrifugation kann auch dazu führen, dass sich das Sphäroid von der Mitte zu den Rändern der Vertiefung bewegt, und obwohl dieses Protokoll dieses Phänomen einschränkt, indem es die Zentrifugationsschritte und das Volumen des für jede Schicht verwendeten Gels (15 μl) für eine gleichmäßige Verteilung optimiert, eliminiert es seine Bewegung nicht vollständig. Die genaue Zentrifugationsgeschwindigkeit und der Zeitpunkt, die erforderlich sind, um die Bewegung des Sphäroids zu reduzieren, müssen möglicherweise entsprechend dem Modell der Zentrifuge angepasst werden. Die in diesem Protokoll beschriebene Spezifikation kann jedoch als Benchmark für die individuelle Optimierung herangezogen werden. Ein anderer Ansatz besteht darin, den Sphäroiden eine Inkubationszeit von 2 Stunden zu geben, um sich am Gerüst zu befestigen, bevor die zweite Schicht Gel hinzugefügt wird. Die Bewegung der Sphäroide wird besonders gut abgemildert, wenn beide Strategien implementiert werden. Aufgrund des mehrstufigen Zentrifugationsprozesses ist diese Methode möglicherweise nicht für weniger widerstandsfähige Zelllinien geeignet.
Abgesehen von der Logistik der Beschichtung der Sphäroide bei der PLOSMA-Methode gibt es Einschränkungen bei der Bildaufnahme. Das Sphäroid kann mit 4- oder 10-facher Vergrößerung abgebildet werden, aber für beste Ergebnisse verwenden Sie mindestens eine 10-fache Vergrößerung und reduzieren Sie die Schrittweite der z-Stacks auf 2-5 μm. Die Vergrößerung sollte während der gesamten Studie konsistent sein. Die Bildgebungszeit nimmt mit höherer Auflösung zu, daher begrenzen Sie die Anzahl der Proben in jeder Well-Platte (4-8 Wells pro Platte), um die Zeit außerhalb des Inkubators zu minimieren. Ein Live-Imaging-Setup könnte auch die Nachverfolgung verbessern und bessere Einblicke liefern.
Da granulare Hydrogele eine einzigartige Topologie und Designparameter aufweisen, die das inhärente Volumen, die Porosität, die mechanische Festigkeit und in einigen Fällen die Bioaktivität umfassen, ist es notwendig, das Zellverhalten in Bezug auf diese Aspekte so genau wie möglich zu untersuchen. Die PLOSMA-Methode wurde entwickelt, um die Zellbewegung nach der Verabreichung oder nachdem die Zellen vollständig in ein körniges Gel eingedrungen sind, zu modellieren. Da die Zellen gezwungen sind, durch die Poren zu wandern, die der körnigen Hydrogelgeometrie innewohnen, isoliert die PLOSMA-Methode effektiv die Porosität als Einfluss auf das Zellverhalten. Mögliche Anwendungen für diesen Assay sind die Zellverabreichung in situ und die Gewebeintegration innerhalb eines körnigen Gerüsts, insbesondere im Wundheilungsraum23.
Beide Protokolle wurden aufgrund der Rolle der Fibroblastenmigration bei der Gewebereparatur und dem Umbau mit primären humanen dermalen Fibroblasten entwickelt 4,24, jedoch kann das Migrationsverhalten aller adhärenten Zellen als Reaktion auf eine Veränderung des porösen Gerüsts gemessen werden - einschließlich der Zugabe von Wachstumsfaktoren und der Oberflächen-/Volumenzusammensetzung des Gels. Diese Änderungen können eine Anpassung dieser Assays erfordern, um nennenswerte Ergebnisse zu erzielen. Zu den Parametern, die weiter optimiert werden müssen, gehören die Zellaussaatdichte, die Experimentdauer und/oder die Analysepipeline. IMARIS ist ein leistungsstarkes bildgebendes Analysewerkzeug, das für die Zellmigrationsanalyse verwendet wird und über Funktionen verfügt, die über das hier beschriebene hinausgehen, einschließlich der Klassifizierung aller Objekte innerhalb einer ausgewählten "Oberfläche" in Sätze, die auf verschiedenen Eigenschaften wie Oberfläche, Volumen, Intensität und Entfernung zu anderen erstellten Oberflächen basieren. Es gibt viele Online-Ressourcen, um weitere Analysemethoden zu ermitteln.
Die beiden hier skizzierten Methoden befassen sich nicht nur physiologisch mit dem Anfangszustand der Gewebeeinführung in ein körniges Material, sondern auch mit der anschließenden Zellreaktion, wenn sie vollständig in das Material eingebettet ist. Wie bei allen Migrationsassays sind die vorhandenen Zellen in der Lage, sich parallel zur Bewegung zu vermehren, jedoch stört das Design der beschriebenen Assays die Proliferation nicht und stellt somit sicher, dass die Analyse nicht übermäßig beeinflusst wird. Beide Methoden sind neben der longitudinalen Bildgebung auch mit der Endpunktfärbung kompatibel, bei der die PFA-Fixierung verwendet wird, um Metriken wie Zytoskelett, Kollagenablagerung, Proliferation und mehr zu erkennen. Die Verwendung der skizzierten Methoden bewegt sich in Richtung einer genaueren räumlich-zeitlichen Darstellung der 3D-Zellmigration, die die Zellinfiltration als messbaren Parameter verwendet, im Gegensatz zu früheren Methoden 1,6,14,15,25,26,27.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Die Finanzierung dieser Arbeit wurde teilweise durch den US National Institutes of Health High Priority, Short-Term Project Award (1R56DK126020-01) und eine philanthropische Spende des Kurtin Trust unterstützt. J.T. wurde durch das Graduate Research Fellowship der National Science Foundation finanziert. Figurenschemata, die mit BioRender.com erstellt wurden.
Materialien
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 647 Phalloidin | ThermoFisher | A22287 | |
| Bovine Serum Albumin | VWR International | 332 | |
| CellTracker Green CMFDS Dye, 1 mg | ThermoFisher | C2925 | 20 x 50 ug units, 492/517 nm |
| Centrifuge | ThermoFisher | 75016085 | ST Plus Series |
| Clear 96 well plate | MilliPore Sigma | CLS3997-50EA | |
| Dimethyl Sulfoxide | Fisher Scientific | MT-25950CQC | 250 mL |
| Fibroblast Basal Medium | ATCC | PCS-201-030 | 480 mL, phenol-red-free |
| Fibroblast Growth Kit - Low Serum | ATCC | PCS-201-041 | 7.5 mM L-glut,5 ng/mL rh FGF basic, 5 ug/mL rh Insulin, 1 ug/mL Hydrocortisone, 50 ug/mL Ascorbic acid, 2% FBS |
| FIJI (ImageJ) | NIH | Public access download | |
| Human Dermal Fibroblasts | ATCC | PCS-201-012 | Adult human dermal fibroblasts |
| ImageXpress Micro Confocal | Molecular Devices | Spinning Disc confocal microscope with 4x, 10x magnifications | |
| IMARIS | Oxford Instruments | 3/4D Imaged Visualizaiton and Analysis Software, Proprietary | |
| Incubator | ThermoFisher | Finnpipette F2 Variable volume Pipettes | HeraCell Vios 160i CO2 Incubator, 165L |
| M-20 Microplate Swinging Bucket Rotor | ThermoFisher | 75003624 | |
| Methylcellulose | Fisher Scientific | 9004-67-5 | Lab grade, powder form |
| Microcentrifuge tube | Fisherbrand | 05-408-129 | 1.5 mL microcentrifuge tubes |
| Paraformaldehyde (4%) | Alfa Aesar | AAJ19943K2 | For fixing |
| Petri dish | Corning | 08-757-100A | Bacteriological Petri Dishes with Lid 35 x 10 mm |
| Pipettes | ThermoFisher | 4642080 | Finnpipette F2 Variable volume Pipettes |
| Sterile PBS | Gibco | 10010-023 | |
| Triton-X | Fisher Scientific | 327371000 |
Referenzen
- Jerka, D., et al. Unraveling endothelial cell migration: Insights into fundamental forces, inflammation, biomaterial applications, and tissue regeneration strategies. ACS Appl Bio Mater. 7 (4), 2054-2069 (2024).
- Edmondson, R., Broglie, J. J., Adcock, A. F., Yang, L. Three-dimensional cell culture systems and their applications in drug discovery and cell-based biosensors. Assay Drug Dev Technol. 12 (4), 207-218 (2014).
- Yamada, K. M., Sixt, M. Mechanisms of 3D cell migration. Nat Rev Mol Cell Biol. 20 (12), 738-752 (2019).
- Grinnell, F. Fibroblast biology in three-dimensional collagen matrices. Trends Cell Biol. 13 (5), 264-269 (2003).
- Kloxin, A. M., Kloxin, C. J., Bowman, C. N., Anseth, K. S. Mechanical properties of cellularly responsive hydrogels and their experimental determination. Adv Mater. 22 (31), 3484-3494 (2010).
- Solbu, A. A., et al. Assessing cell migration in hydrogels: An overview of relevant materials and methods. Mater Today Bio. 18, 100537 (2023).
- Madl, C. M., Katz, L. M., Heilshorn, S. C. Tuning bulk hydrogel degradation by simultaneous control of proteolytic cleavage kinetics and hydrogel network architecture. ACS Macro Lett. 7 (11), 1302-1307 (2018).
- Daly, A. C., Riley, L., Segura, T., Burdick, J. A. Hydrogel microparticles for biomedical applications. Nat Rev Mater. 5 (1), 20-43 (2020).
- Qazi, T. H., Burdick, J. A. Granular hydrogels for endogenous tissue repair. Biomater Biosyst. 1, 100008 (2021).
- Tanner, G. I., Schiltz, L., Narra, N., Figueiredo, M. L., Qazi, T. H. Granular hydrogels improve myogenic invasion and repair after volumetric muscle loss. Adv Healthc Mater. 25, e2303576 (2024).
- Sussman, E. M., Halpin, M. C., Muster, J., Moon, R. T., Ratner, B. D. Porous implants modulate healing and induce shifts in local macrophage polarization in the foreign body reaction. Ann Biomed Eng. 42 (7), 1508-1516 (2014).
- Roosa, C. A., et al. Conjugation of IL-33 to microporous annealed particle scaffolds enhances type 2-like immune responses in vitro and in vivo. Adv Healthc Mater. 13 (25), 2400249 (2024).
- Jaberi, A., et al. Engineering microgel packing to tailor the physical and biological properties of gelatin methacryloyl granular hydrogel scaffolds. Adv Healthc Mater. 13 (25), 2402489 (2024).
- Nandi, S., Brown, A. C. Characterizing cell migration within three-dimensional in vitro wound environments. J Vis Exp. 126, e56099 (2017).
- Qazi, T. H., et al. Anisotropic rod-shaped particles influence injectable granular hydrogel properties and cell invasion. Adv Mater. 34 (12), 2109194 (2022).
- Griffin, D. R., Weaver, W. M., Scumpia, P. O., Di Carlo, D., Segura, T. Accelerated wound healing by injectable microporous gel scaffolds assembled from annealed building blocks. Nat Mater. 14 (7), 737-744 (2015).
- Pruett, L. J., Jenkins, C. H., Singh, N. S., Catallo, K. J., Griffin, D. R. Heparin microislands in microporous annealed particle scaffolds for accelerated diabetic wound healing. Adv Funct Mater. 31 (35), 2104337 (2021).
- Roosa, C., et al. Microfluidic synthesis of microgel building blocks for microporous annealed particle scaffold. J Vis Exp. (184), e64119 (2022).
- Pfaff, B. N., et al. Selective and improved photoannealing of microporous annealed particle (MAP) scaffolds. ACS Biomater Sci Eng. 7 (2), 422-427 (2021).
- Riley, L., Schirmer, L., Segura, T. Granular hydrogels: Emergent properties of jammed hydrogel microparticles and their applications in tissue repair and regeneration. Curr Opin Biotechnol. 60, 1-8 (2019).
- Jo, C. H., Roh, Y. H., Kim, J. E., Shin, S., Yoon, K. S. Optimizing platelet-rich plasma gel formation by varying time and gravitational forces during centrifugation. J Oral Implantol. 39 (5), 525-532 (2013).
- Mironov, V., et al. Fabrication of tubular tissue constructs by centrifugal casting of cells suspended in an in situ. crosslinkable hyaluronan-gelatin hydrogel. Biomaterials. 26 (36), 7628-7635 (2005).
- Vu, L. T., Jain, G., Veres, B. D., Rajagopalan, P. Cell migration on planar and three-dimensional matrices: A hydrogel-based perspective. Tissue Eng Part B Rev. 21 (1), 67-74 (2015).
- Cialdai, F., Risaliti, C., Monici, M. Role of fibroblasts in wound healing and tissue remodeling on Earth and in space. Front Bioeng Biotechnol. 10, 958381 (2022).
- Qazi, T. H., Muir, V. G., Burdick, J. A. Methods to characterize granular hydrogel rheological properties, porosity, and cell invasion. ACS Biomater Sci Eng. 8 (4), 1427-1442 (2022).
- Shaik, R., et al. Cardiac matrix-derived granular hydrogel enhances cell function in 3D culture. ACS Appl Mater Interfaces. 16 (43), 58346-58356 (2024).
- Puiggalí-Jou, A., Asadikorayem, M., Maniura-Weber, K., Zenobi-Wong, M. Growth factor-loaded sulfated microislands in granular hydrogels promote hMSCs migration and chondrogenic differentiation. Acta Biomater. 166, 69-84 (2023).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten