Method Article
Quantifying three-dimensional cell migration within and into granulular hydrogel biomaterials(과립 하이드로겔 생체 물질로의 3차원 세포 이동 정량화)
요약
과립 하이드로겔 내부 및 계면으로의 3D 세포 이동에 대한 정량적 평가를 위한 프로토콜이 여기에 제시되어 있습니다.
초록
과립형 하이드로겔 스캐폴드는 재생 의학에서 상당한 잠재력을 가지고 있으며, 세포 전달을 위한 운반체 또는 조직 통합을 위한 인터페이스로 기능합니다. 이 기사에서는 과립 하이드로겔 내부 및 과립 하이드로겔로의 세포 이동을 정량화하기 위한 두 가지 새로운 접근 방식을 소개하고 이러한 스캐폴드의 고유한 응용 분야를 강조합니다. 먼저, 통합 목적을 위해 과립 하이드로겔로의 조직 성장을 시뮬레이션하는 세포 단층 계면 분석이 제시됩니다. 둘째, 하이드로겔 매트릭스 내에서 세포 이동을 추적하도록 설계된 스페로이드 기반 분석에 대해 설명하며, 특히 세포 전달과 관련된 응용 분야에 적합합니다. 두 방법 모두 세포 이동을 정확하고 제어된 방식으로 측정할 수 있으며, 과립형 하이드로겔 스캐폴드를 활용하는 연구자에게 포괄적인 툴킷을 제공합니다. 이러한 방법의 동기는 특정 응용 분야에 맞게 골격 내 세포 이동을 맞춤화하여 제어해야 할 필요성에서 비롯됩니다. 이러한 정량화 기법을 최적화하고 표준화함으로써 연구자들은 과립 하이드로겔 특성을 반복적으로 정제하여 다양한 재생 의학 맥락에서 효과를 보장할 수 있습니다. 이 강력한 정량 도구 세트는 과립 하이드로겔 스캐폴드를 향상시킬 수 있는 새로운 기회를 제공하여 세포 전달 및 조직 통합 응용 분야 모두에서 사용을 촉진합니다.
서문
치료 응용 분야를 위한 생체 재료는 조직 통합을 연구하기 위해 보다 복잡하고 관련성 있는 세포 환경 모델로 점점 더 진화하고 있습니다. 생체 재료 스캐폴드는 세포 성장을 위한 3차원(3D) 구조를 제공하며 원하는 조직을 모방하는 것을 목표로 합니다 1,2. 3차원 세포 배양 모델에는 촉각 또는 화학주성 신호를 통해 세포에 복잡성을 더하는 천연 매트릭스 및 합성 골격이 포함됩니다 3,4. 전통적인 하이드로겔 스캐폴드는 대량으로 가교되어 소분자의 확산을 허용하는 나노다공성 메쉬를 생성하지만(5,6), 복구가 필요한 조직 영역으로의 세포 규모 이동을 위해서는 분해가 필요합니다7. 과립형 하이드로겔(granulular hydrogel)은 생체 적합성(biocompatibility), 불규칙한 모양에 순응하는 능력, 그리고 많은 경우 주입 가능성(injectability)으로 인해 임상적 번역(clinical translation)에 대한 높은 잠재력을 가진 생체 물질의 하위 집합입니다 8,9. 이들의 빌딩 블록 특성은 세포 규모 다공성의 이점을 제공하여 조직 침투 및 혈관 신생을 향상시킬 뿐만 아니라 모듈성을 향상시켜 세포 행동에 대한 이질적인 단서를 추가할 수 있도록 합니다 10,11,12. 3D 골격 내에서 세포 반응과 움직임을 이해하는 것은 조직 통합을 위해 생체 재료를 사용하는 모든 응용 분야에서 생리학적 관련성을 위해 매우 중요합니다.
그러나 3차원에서 조직 성장을 연구하는 것은 정량적 정확성으로 달성하기 어렵다는 것이 입증되었습니다. 3D 환경의 복잡성이 증가함에 따라 세포 거동에 대한 통찰력뿐만 아니라 재료 상태 최적화를 제공할 수 있는 세포 이동의 체외 모델이 필요합니다. 이전에 발표된 3D 과립 골격 세포 이동에 대한 보고서는 국소 파종을 사용하여 세포 거동을 탐색하고, 다공성 구조 및 세포 형태13 및 기타 스페로이드 발아14,15로의 침투를 보고하여 새싹의 성장 길이와 수를 측정했습니다. 국소 파종(Topical seeding) 이동 길이는 중력의 영향을 불균일하게 받을 수 있으며, 현미경 검사의 한계로 인해 결과가 종단적일 수 없습니다. 스페로이드 발아 방법은 최대 투영을 통한 2D 정량화로 제한되어 제어된 침입 메커니즘을 포착할 수 없습니다. 두 방법 모두 xy-평면에서 측정되며, 이는 3D 세포 이동과 스캐폴드 침투를 완전히 재현하는 데 필요한 뉘앙스가 부족합니다.
이 프로토콜은 특히 MAP(Microporous Annealed Particle) 스캐폴드 16,17,18,19를 사용하여 다공성 3D 과립 하이드로겔 스캐폴드로의 체외 침투와 같은 세포 이동을 정량화하기 위한 두 가지 접근 방식을 설명합니다. 다음 방법의 목적은 3차원 분석을 위해 이동 방향을 제어하여 과립 겔의 세포 거동을 연구하는 것입니다. 첫 번째 단층 기반 상승 이동 분석법(MAMA) 접근법은 균일한 세포-물질 상호 작용을 보여주고 세포가 과립 하이드로겔과 인터페이스하는 초기 환경을 나타내고 골격에 침투하기 전에 개별 거동을 분리하는 플랫폼 역할을 하는 단순화된 내인성 세포 통합 모델입니다. PLOSMA(Parallel Layered Outward Spheroid Migration Assay) 방법이라고 하는 두 번째 방법은 3D 세포 스페로이드 이동 분석법으로, 복잡한 골격 환경에 완전히 둘러싸여 있을 때의 세포 이동을 탐색하고 전달 후의 세포 이동과 세포가 과립 겔에 완전히 들어간 후의 움직임을 모델링합니다.
두 방법 모두 3D 이미지 분석으로 정량화할 수 있으며, 세포 이동을 촉진하거나 제한하는 것이 설계 기준 내에 있는 재생 의학 및 조직 공학 응용 분야의 종적 시점을 사용하여 재료-세포 상호 작용을 연구하고 최적화하는 데 적용할 수 있습니다. 또한 이러한 방법은 균일한 다중 웰 분석 준비를 위해 플레이트 원심분리를 활용합니다.
프로토콜
연구에 사용된 시약 및 장비의 세부 사항은 재료 표에 나열되어 있습니다.
1. 과립 하이드로겔 준비
참고: 이 프로토콜 전반에 걸쳐 사용되는 MAP 입자는 45.88 mg/mL PEG-MAL (10 kDa), 0.82 mg/mL RGD, 8.06 mg/mL MethMal19 및 5.62 mg/mL MMP-2 분해성 교차결합제가 있는 3.2 wt% w/v gel입니다. 젤의 기계적 강성은 15-20kPa로 피부 강성17과 일치합니다.
- 과립 하이드로겔 입자를 생성하고 평소와 같이 세포 배양을 준비합니다.
참고: 이 프로토콜은 Microporous Annealed Particle gel의 멸균 제제를 설명하며, 그 생산은 Roosa et al.18에 의해 자세히 설명되어 있습니다. - 70% 이소프로필 알코올을 3회 세척한 후 멸균 1x PBS를 3회 세척하여 시험관 내 사용을 위해 과립 하이드로겔 입자를 준비합니다.
- LAP 분말을 초순수에 용해시키고 용액을 0.22μm 멸균 필터에 통과시켜 매체 용액에 0.2mM 리튬 페닐-2,4,6-트리메틸벤조일포스피네이트(LAP)를 멸균 준비합니다. 이 용액을 실험 수행에 필요한 겔의 양과 1:1 v/v로 추가합니다.
- 0.2mM LAP, 겔 및 배지 용액을 37°C의 튜브 로테이터에서 20rpm으로 최소 30분 동안 배양하여 미립자 전체에 LAP가 확산될 수 있도록 진행합니다.
- 30분이 경과하면 25°C에서 5분 동안 18,000 x g 에서 입자 현탁액을 원심분리합니다. 상층액을 흡인합니다.
참고: MAP 입자의 전반적인 건조도는 화학적 성질, 친수성 및 입자 크기의 차이로 인해 달라질 수 있습니다. 일관성을 위해 설명된 대로 입자 현탁액을 원심분리하고, 용적식 피펫을 사용하여 튜브의 입자를 혼합한 다음 원심분리 단계를 반복하는 것이 가장 좋습니다.
2. 단층 기반 상승 이동 분석법(MAMA) 방법: 세포 배양 및 이미징
참고: 세포는 FITC 채널(488nm)을 사용하여 이미지화되었습니다. 세포에 사용된 염료는 492nm에서 여기(excitation)와 517nm에서 방출(emission a)을 가졌습니다. 10배 배율은 4배 배율보다 디테일이 증가하지만 둘 다 사용할 수 있습니다.
- 제조업체의 프로토콜에 따라 인간 피부 섬유아세포(HDF)를 해동합니다. 원하는 통과가 달성될 때까지 필요에 따라 통과합니다. 일반적으로 일차 세포는 P5를 통해 유전적 구성과 표현형 구성을 유지합니다.
- 플레이트 120,000 cells/cm2 for least n = 6 in 96-well plate or desired plate size using recommended media volume per well for suspension, each addition 전에 부드럽게 흡인합니다. 그림 1A와 같이 세포가 밤새 부착되도록 하면 다음 날 약 80%의 포화도가 발생합니다.
참고: 일반적으로 중앙 컬럼 및 열 웰은 최적의 겔 확산에 가장 적합합니다. 저자는 96웰 플레이트(B-G 행 및 5-8열)에 최대 24개의 웰을 사용합니다. - 위에서 언급한 바와 같이 겔 조건을 준비하고 LAP에서 배양하는 동안 세포의 웰 플레이트에서 흡인기 또는 피펫으로 배지를 제거합니다. 접시 바닥을 방해하지 않도록 주의하세요.
- 그림 1B에 표시된 제조업체 지침에 따라 well에 세포 추적 염료를 추가합니다. 웰에서 세포 추적 염료를 흡입하기 전에 1단계에서 설명한 대로 겔이 완전히 준비되었는지 확인합니다.
- 각 겔 조건 20 μL를 플레이트 바닥을 건드리지 않고 용적식 피펫으로 웰에 추가합니다.
참고: 최상의 분석 조건은 겔을 웰 중앙에 직접 피펫으로 주입할 때 발생합니다. - 25°C에서 플레이트 회전 로터 원심분리기 부착물을 사용하여 100 x g 에서 15초 동안 회전하고 가속 및 감속을 8로 하여 겔을 평평하게 만듭니다. 플레이트를 180° 뒤집고 100 x g 에서 15초 동안 다시 회전하여 그림 1C에서 볼 수 있듯이 웰 바닥 전체에 균일한 젤 분포를 보장합니다.
- 모든 스캐폴드가 형성된 후 세포의 각 웰에 200μL의 배지를 첨가하기 위해 30초 동안 샘플에 집속광(365nm, 34.4mW/cm2)을 적용하여 상단에서 겔을 무균 광교차 결합(그림 1D)합니다. 세포를 37°C에서 30분 동안 배양하여 이미징 전에 과립 골격에 부착할 수 있도록 합니다.
- 그림 1E에 요약된 이동 거동을 캡처하기 위해 컨포칼 현미경을 사용하여 세포를 이미지화합니다. 세포가 80% 이상 합류하는 플레이트 영역에 대한 가장 낮은 초점 지점을 찾아 z 스택의 아래쪽 가장자리로 설정합니다.
- 세포 형광 신호의 가장 높은 지점을 찾아 z 스택의 위쪽 가장자리로 설정합니다. 최상의 분해능을 위해 5μm 이하의 단계 크기를 사용하십시오.
- 세포 단층(cell monolayer) 및 이동하지 않는 세포 높이를 나타내기 위해 최소 3개의 웰(well)을 이미지화합니다. t = 0 시점은 그림 2A의 평면도와 측면 모두에서 표시됩니다. 이미징이 완료된 후 밤새 배양합니다.
참고: 이 세포는 4x 대물렌즈로 이미지화되었으며 노출은 모든 웰에 대해 일관되게 유지되었습니다. - t = 24 h에서, 세포는 그림 2B 의 상단 및 측면 보기에서 볼 수 있듯이 입상 골격을 통해 상승하기 시작합니다. 동일한 파라미터를 사용하여 t = 0 h 동안 수행된 이미징 단계를 반복합니다. 스테이지의 이전 높이를 참조로 사용하거나 아직 마이그레이션되지 않은 셀을 찾아 z 스택의 아래쪽 가장자리로 설정합니다.
- 모든 웰에 대해 단계를 반복하여 분석의 용이성을 위해 각 웰이 별도의 이미지로 저장되었는지 확인합니다.
참고: 타임포인트는 실험적 제약 조건 및 세포 추적 형광 방법에 따라 더 긴 타임포인트에 대해 이미지화될 수 있습니다. - 스캐폴드는 추가 메트릭을 위해 고정되고 염색될 수 있습니다. 원하는 종료 시점에서, 피펫으로 웰의 배지를 흡인하고 폐기합니다. 200μL의 멸균 PBS로 각 웰을 각각 5분씩 2회 부드럽게 세척합니다. 200μL의 4% 파라포름알데히드(PFA)를 20분 동안 첨가한 다음 흡입하고 버립니다. 웰은 즉시 염색하거나 1x PBS에서 최대 일주일 동안 4°C에서 보관할 수 있습니다.
3. MAMA(Monolayer-based Ascending Migration Assay) 방법: 3D 이미지 분석
- 일괄 변환의 경우 IMARIS 이미지 변환 소프트웨어를 엽니다. 현미경 이미지를 변환 소프트웨어로 끌어다 놓고 가져올 소프트웨어 Arena 내의 폴더를 선택합니다. 모두 시작을 누릅니다. 변환된 이미지는 아레나에 .ims 파일로 표시됩니다.
참고: 복셀 크기가 이미지 메타데이터에 포함되지 않은 경우 개별 컨포칼 이미징 사양을 참조하여 값을 찾으십시오. 또는 복셀 크기는 이미징 중에 사용되는 단계 크기로 근사화할 수 있습니다. - 바탕 화면 IMARIS Arena 아이콘을 두 번 클릭하고 Arena에서 이미지를 선택하여 소프트웨어를 엽니다.
- 이미지는 상단의 도구 모음 아이콘 패널에 표시되는 '3D View' 분석 탭에 자동으로 로드됩니다. 메인 도구 모음에서 Image Proc 탭을 클릭합니다.
- 측면 패널의 왼쪽 상단에서 Channel 1의 드롭다운 메뉴를 클릭하고 Background Subtraction(배경 빼기)을 선택합니다. 패널 하단의 확인을 눌러 '3D View'로 돌아갑니다. t=0h 및 t=24h에 대한 대표 이미지는 각각 그림 2A,B에 나와 있습니다.
- 측면 패널 메뉴 바로 위에 있는 작은 도구 모음에서 둥근 파란색 모양이 있는 아이콘인 Add new Surfaces를 클릭하여 'Surfaces 1'이라는 편집 가능한 개체 탭을 만듭니다.
- 생성 파라미터 알고리듬 설정을 위한 인터페이스가 메뉴 패널 아래쪽으로 열릴 것입니다. 모든 복제에 사용할 파라미터를 인터페이스 하단에 있는 파란색 화살표 버튼을 클릭하여 수동으로 생성합니다. 올바른 소스 채널이 선택되었는지 확인하고 'Smooth' 상자를 선택합니다.
- 표면 디테일을 0.7μm로 설정하고 배경 빼기(로컬 대비)를 선택합니다. 'Diameter of the largest Sphere which fits into the Object' 상자에 평균 셀 길이를 입력합니다. 완료되면 하단에 있는 동일한 파란색 화살표를 누릅니다.
참고: 이 값은 도구 모음의 '슬라이스' 탭을 사용하여 추정하고 평균 셀의 너비를 측정할 수 있습니다.
- 표면 디테일을 0.7μm로 설정하고 배경 빼기(로컬 대비)를 선택합니다. 'Diameter of the largest Sphere which fits into the Object' 상자에 평균 셀 길이를 입력합니다. 완료되면 하단에 있는 동일한 파란색 화살표를 누릅니다.
- 임계값 설정의 경우 가장 밝은 셀만 분할되는 강도 히스토그램을 결정합니다. 슬라이서를 사용하여 이미지 스택을 위아래로 이동하여 가능한 한 정확한지 확인합니다.
- 'Split touching objects (Region Growing)'에 대해 Enable 을 선택하고 Seed Points Diameter를 이전에 사용한 것과 동일한 직경으로 설정합니다. 'Intensity Based' 임계값이 선택되었는지 확인하고 파란색 오른쪽 화살표 버튼을 클릭합니다.
- 다음 두 단계인 Filter Seed Points 및 Filter Surfaces는 생성된 표면이 정확한지 확인하기 위해 여러 측정을 통해 조정할 수 있습니다. 그러나 기준선 분석에는 추가 필터링이 필요하지 않습니다. 변경할 필요가 없는 경우 파란색 화살표 버튼을 클릭합니다.
참고: 마지막 단계에서는 원하는 출력에 따라 표면을 추가로 분류할 수 있습니다. 편집이 완료되면 녹색 화살표 버튼을 클릭하여 표면 작성을 마칩니다. - 배치 분석을 위해 생성 매개변수를 저장하려면 지팡이 아이콘 Creation을 클릭합니다. Store Parameters for Batch를 클릭하고 이름을 지정한 다음 Ok를 클릭합니다. t=0 및 t=24h에 대한 대표적인 처리된 이미지들이 각각 도 2C,D에 도시되어 있다.
참고: 선택한 '표면' 내의 모든 개체는 표면 면적, 부피, 강도 및 생성된 다른 표면과의 거리와 같은 이미지 변환 소프트웨어가 제공하는 다양한 속성에 따라 세트로 분류할 수 있습니다. - 표면 속성은 통계 탭을 선택하여 찾을 수 있습니다. 모든 셀 높이를 수집하려면 세부 탭을 클릭하고 연속 드롭다운 메뉴에서 특정 값 및 위치 Z 를 선택합니다. 단일 저장 아이콘을 클릭하여 모든 z-위치와 모든 분류를 .xls 파일로 저장합니다. 모든 이미지에 대해 반복합니다.
- 대표 이미지에서 이동하지 않는 셀의 중앙값 z-위치를 찾고 각 테스트 조건 웰에서 해당 숫자 아래의 모든 z-위치를 뺍니다.
참고: 마이그레이션 값은 비이동 셀 높이보다 높은 각 기술 반복 조건에 대한 중앙값의 평균으로 보고됩니다. 이는 그림 3A에서 볼 수 있는 중간 높이로 보고되거나 그림 3B에서 볼 수 있는 t = 0과 비교하여 원하는 시점에 대한 마이그레이션 높이의 접힌 변화로 보고될 수 있습니다.
4. PLOSMA(Parallel Layered Outward Spheroid Migration Assay) 방법: 3D 스페로이드를 위한 세포 배양 및 매달린 액적 배양
참고: 이 프로토콜은 Nandi et al.14에 의해 저술된 프로토콜에서 채택된 세포 배양 및 행잉 드롭 배양에 대해 설명합니다.
- 제조업체의 프로토콜에 따라 HDF를 해동합니다. 원하는 통과에 도달할 때까지 필요에 따라 통과합니다.
- 무균 세포 배양 후드에서 바닥에 10mL PBS를 추가하고 뚜껑을 뒤집어 외부가 세포 배양 후드 위에 놓이도록 페트리 접시를 준비합니다.
- 적절한 부피의 세포(세포 수에서 결정됨)를 마이크로 원심분리기 튜브에 추가합니다. 배지에 희석된 세포 추적 염료를 추가합니다. 예열된 매체로 총 부피를 1mL로 가져옵니다.
참고: 스페로이드는 스페로이드당 약 8000개의 세포여야 하지만 세포 유형에 따라 변경될 수 있습니다. - 세포 용액을 37°C에서 45분 동안 배양합니다. 제조업체가 권장하는 속도에 따라 세포를 회전시키고 상층액을 흡입합니다.
- 배지에서 1:100 메틸셀룰로오스로 세포를 재현탁합니다.
- 셀/배지 용액의 20μL 방울을 페트리 접시의 뚜껑에 피펫팅합니다.
- 자신 있게 빠르고 조심스럽게 뚜껑을 뒤집어 PBS가 들어 있는 페트리 접시의 아래쪽 절반 위에 놓습니다.
- 방울을 최소 24시간 동안 배양하십시오.
참고: 스페로이드 형성은 명시야 현미경으로 모니터링할 수 있습니다.
5. PLOSMA(Parallel Layered Outward Spheroid Migration Assay) 방법: 세포 스페로이드를 과립 하이드로겔에 파스딩
참고: 다음 프로세스는 그림 4A에 요약되어 있습니다.
- 그림 4A에 요약된 PLOSMA 분석법을 설정하려면 용적치환 피펫을 사용하여 투명한 96웰 플레이트의 웰에 15μL의 겔을 무균 방식으로 첨가합니다.
- 플레이트 회전 로터 원심분리기 부착물을 사용하여 1000 x g 에서 10초 동안 회전하여 젤을 평평하게 만듭니다. 플레이트를 180° 뒤집고 1000 x g 에서 10초 동안 다시 회전하여 웰 바닥에 젤이 균일하게 분포되도록 합니다.
- 균일한 평탄도가 달성되면 비계를 어닐링하기 위해 30초 동안 샘플에 집중된 빛(365nm, 33.4mW/cm2)을 적용하여 상단에서 젤을 무균 방식으로 광 교차 결합합니다.
- 매달려 있는 물방울의 페트리 접시를 무균 조직 배양 후드로 이동하고 뚜껑을 뒤집습니다.
- 20μL 피펫을 사용하여 스페로이드가 피펫 팁에 들어갈 때까지 물방울을 천천히 흡수합니다. 우물 중앙의 비계로 물방울을 배출합니다.
- 모든 웰에 대해 이전 단계를 반복합니다. 각 웰에 스페로이드가 있는지 명시야 또는 형광 현미경으로 확인하여 확인합니다.
- 웰 플레이트를 37°C에서 2시간 동안 배양하여 스페로이드가 골격에 부착될 수 있도록 합니다.
- 각 스페로이드 위에 15μL의 젤을 추가로 피펫팅합니다. 균일한 겔 분포를 보장하기 위해 300 x g 에서 각 방향으로 15초 동안 플레이트를 원심분리합니다.
- 33.4mW/cm2에서 UV(365nm) 광을 사용하여 30초 동안 젤의 최상층을 어닐링합니다. 웰의 총 부피를 200μL로 끌어올리기 위해 각 스캐폴드 위에 피펫 배지를 놓습니다.
알림: 이 시점에서 골격은 매우 건조하므로 스페로이드가 분리되지 않도록 웰 측면 아래로 미디어를 떨어뜨려 추가합니다.
6. PLOSMA(Parallel Layered Outward Spheroid Migration Assay) 방법: 스페로이드의 공초점 이미징
참고: 이미징의 용이성은 이미징 시스템에 따라 다릅니다. 낮은 노출 시간에 웰 내에서 스페로이드를 찾습니다. 세포는 FITC 채널(488nm)을 사용하여 이미지화되었습니다. 세포에 사용된 염료는 492nm에서 여기(excitation)와 517nm에서 방출(emission a)을 가졌습니다. 10배 배율은 4배 배율보다 디테일이 향상됩니다.
- 셀에 여전히 초점이 맞춰져 있는 가장 낮은 스테이지 수준(z 높이)을 찾습니다. 이를 z 스택의 하한으로 설정합니다.
- 셀에 여전히 초점이 맞춰져 있는 가장 높은 단계 수준(z-높이)을 찾습니다. 이를 z 스택의 상한으로 설정합니다.
참고: 최상의 이미징 결과에는 세포 규모 해상도를 유지하기 위해 5μm 이하의 단계 크기가 포함됩니다. 컨포칼 현미경 시스템에 따라 이미징 속도와 해상도 사이에 절충이 있을 수 있습니다. - t = 0 및 24h에 설명된 대로 모든 스페로이드를 이미지화합니다. 48시간 시점은 실험적 제약 조건에 따라 이미지화될 수도 있습니다. t = 0 및 t = 24의 대표적인 최대 투영 이미지가 그림 4B에 나와 있습니다.
7. PLOSMA(Parallel Layered Outward Spheroid Migration Assay) 방법: 3D 이미지 분석
- 3.1단계부터 3.4단계까지 설명된 대로 분석 소프트웨어로 이미지를 가져옵니다. 왼쪽 패널의 오른쪽 상단에서 채널 1의 드롭다운 메뉴를 클릭하고 배경 빼기를 선택합니다. 패널 아래쪽에 있는 확인을 누릅니다.
- 3D 보기로 돌아온 후 디스플레이 조정 팝업 창에서 모든 채널 자동 조정을 누르고 필요에 따라 수정합니다.
- 사이드 메뉴 바로 위에 있는 작은 도구 모음에서 그림 5A에 표시된 세 개의 직교 화살표가 있는 Add new Reference Frame 아이콘을 클릭하여 'Reference Frame 1'이라는 새 탭을 추가합니다.
- 그림 5B에 시각화된 것처럼 세 평면 모두에서 원점을 회전 타원체의 중심으로 이동합니다.
- 세 개의 직교 화살표와 동일한 도구 모음에서 주황색 구가 있는 아이콘을 클릭하고 새 스폿을 추가하여 '스폿 1'이라는 탭을 만듭니다. 파란색 화살표 버튼을 누릅니다.
- Spot Detection(스폿 탐지)에서 Estimated XY Diameter(추정된 XY 직경)를 셀의 추정 직경으로 설정합니다. 파란색 화살표 버튼을 누릅니다.
참고: HDF의 경우 이 수치는 15.0μm입니다. - 임계값을 적용하려면 가장 밝은 부분만 둘러싸도록 강도 히스토그램을 조정합니다. 슬라이서를 사용하여 이미지 스택을 위아래로 이동하여 가능한 한 정확한지 확인합니다. 파란색 다음 화살표를 누르십시오.
- 녹색 Execute 버튼을 눌러 분석을 마칩니다.
- 슬라이서에서 렌더링을 선택 취소하거나 설정 패널의 오른쪽에 있는 노란색 사각형 아이콘을 클릭합니다.
- Statistics 탭을 클릭합니다. 첫 번째 드롭다운 메뉴에서 Specific Values를 선택합니다. 두 번째 드롭다운 메뉴에서 Distance from Origin Reference Frame을 선택합니다. 선택한 표면의 모든 값이 그림 5C와 같이 표시됩니다. 단일 저장 아이콘을 클릭하면 .xls 파일이 다운로드됩니다.
- 이미지와 분석에 대한 변경 사항을 기본 도구 모음에서 Save(저장 ) 아이콘을 눌러 저장합니다. 그림 6A 는 24시간 후에 이미징된 스페로이드의 3D 렌더링을 나타내며, 그림 6B 는 원점 참조 프레임으로부터의 거리에 따라 색상으로 구분된 확산 셀을 표시하는 IMARIS 스폿 기능을 나타냅니다.
- 내보낸 데이터를 t = 0 이미지로 정규화하고 각 스페로이드에 대한 이동 거리와 z-높이의 평균을 계산하여 각 샘플에 대한 단일 값을 얻습니다. 그림 7A,B는 각각 각 출력에 대한 대표 그래프를 나타냅니다.
결과
이 프로토콜은 두 가지 새로운 과립 스캐폴드 마이그레이션 분석에 필요한 단계를 자세히 설명하는 것을 목표로 합니다. MAMA 방법은 조직 계면에서 세포 침투를 평가하는 데 사용할 수 있습니다. 과립 하이드로겔은 벌크 하이드로겔보다 더 복잡한 시스템이므로 본질적으로 마이그레이션을 위한 처리가 더 복잡합니다 9,20. 그림 1에 요약된 단계적 프로세스를 이해하는 것이 중요합니다. 각 단계는 다음 단계를 기반으로 하며 이 프로토콜에서 최적화되었습니다. 120,000 cells/cm2의 밀도에서 HDF를 파종하면 하룻밤 사이에 최소 80%의 합류가 발생하며(그림 1A), 이러한 비형광 세포는 이미징 가능성을 극대화하기 위해 실험 당일에 세포 추적 염료로 가장 잘 태그됩니다(그림 1B). 이 프로토콜은 세포 생존력을 유지하기 위해 HDF 계대성 계승에 사용되는 낮은 원심력과 일치합니다. 단일 원심분리 단계에서 생성되는 각도로 인해 웰의 바닥 표면을 완전히 덮을 수 있도록 겔 이동을 보장하기 위해 플레이트를 180° 뒤집어야 합니다(그림 1C). 어닐링 후 30분 동안 인큐베이터에서 세포가 회복되도록 하면(그림 1D) 세포 생존율을 유지하고 최적의 이동을 얻을 수 있습니다(그림 1E). 96웰 플레이트의 넓은 영역을 4x 대물렌즈로 이미징하고 0-24시간 시점(그림 2A, B)에서 일치시켜 세포 거동을 광범위하게 평가할 수 있습니다. 분석 소프트웨어에서 결과 z-stack 이미지를 처리하면 사용하기 쉬운 인터페이스에서 여러 대규모 데이터 세트에 대한 고급 분석을 제공할 수 있습니다. 이 프로토콜은 그림 2B,C의 대표 이미지로 시각화된 각 시점에서 셀 높이 또는 위치 Z에 대한 데이터 세트를 생성하는 단계를 요약합니다. 처리된 데이터의 분석은 그림 3A에서 볼 수 있으며, 중앙 높이의 평균과 각 시점의 표준 편차를 사용하여 시각화되었으며, t = 0 h에서 이동하지 않는 셀의 폴드 변화 높이가 그림 3B에 나와 있습니다. 이 방법의 기본 데이터는 일반적으로 비정규 분포이므로 중위수는 비교를 위한 더 강력한 측정값이므로 데이터를 요약하는 데 사용됩니다.
마찬가지로, PLOSMA 방법을 사용하여 3D 과립 하이드로겔 스캐폴드 내에서 전달된 세포의 운동성을 평가할 수 있습니다. 그림 4A는 PLOSMA 방법의 단계를 간략하게 설명하며, 웰 중앙에 스페로이드를 파종하는 것이 특히 중요합니다. 스페로이드를 시야의 중앙에 배치하는 것이 좋지만 현미경의 기능에 따라 다릅니다. 도 4B는 FITC 채널(488nm)에서 10배 배율로 촬영한 t = 0 h 및 t = 24 시간 동안 스페로이드 확산의 대표적인 이미지를 보여줍니다. 소프트웨어에서 원점 참조 프레임을 생성하고 각 z-스택에 맞게 조정할 수 있습니다(그림 5A,B). 이 소프트웨어는 해당 원점 참조 프레임에서 방사형 거리를 추적하고 원하는 데이터 세트로 내보낼 수 있습니다(그림 5C). 그림 6A는 t = 24시간 스페로이드의 3D 렌더링을 보여주는 대표적인 이미지이며, 그림 6B는 소프트웨어의 스폿 기능을 보여줍니다. 처리된 데이터의 예가 그림 7에 나와 있습니다. 그림 7A는 정규화된 중심에서 Day 0 거리로 이동한 평균 거리를 나타냅니다. 그림 7B는 PLOSMA 방법이 연구하고자 하는 방향이기 때문에 z-평면에서만 이동한 거리를 분리합니다.
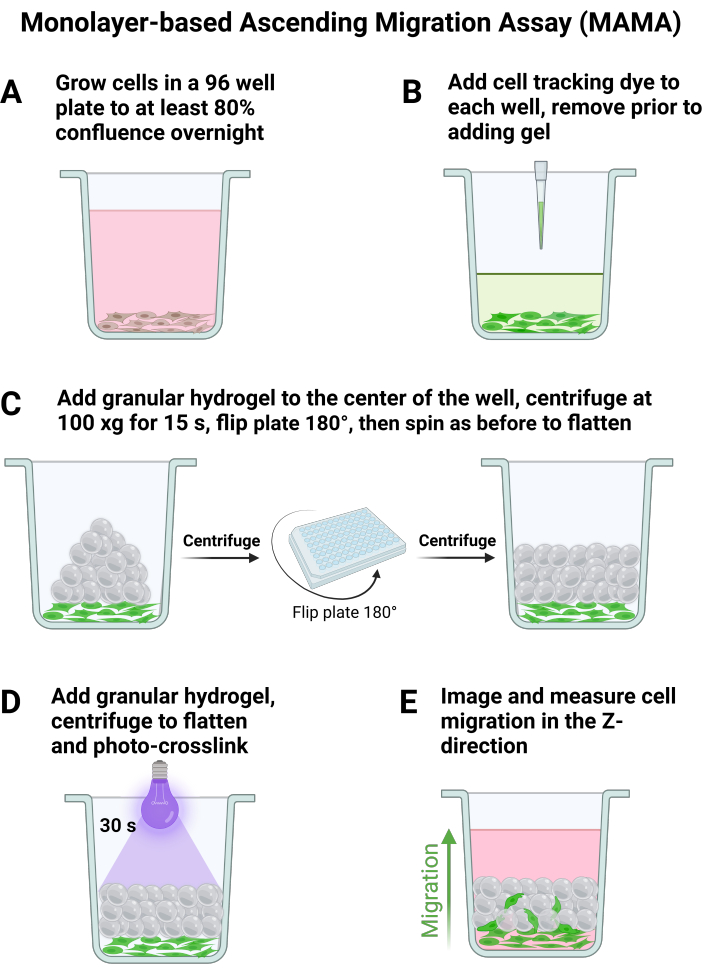
그림 1: 단층 기반 상행 이동 분석 세포 배양 및 이미징. MAMA를 위한 세포 및 겔 처리의 주요 단계에 대한 개략도. (A) 세포는 하룻밤 사이에 합류하도록 성장하고, (B) 세포 추적 염료는 과립 겔을 첨가하기 직전에 첨가됩니다. 스캐폴드는 (C) 플레이트 원심분리를 통해 조립되고 (D) 광 가교로 안정화됩니다. 여러 시점에서의 이미징을 통해 상향 세포 이동을 (E) 시각화할 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
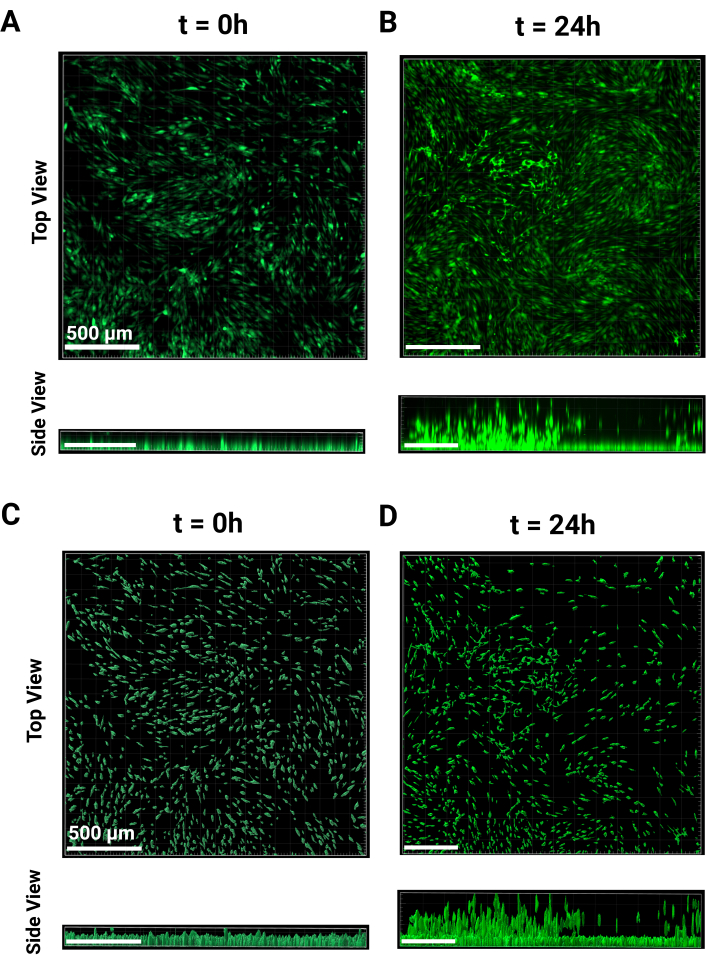
그림 2: MAMA 이미지 처리. 이미지 처리의 대표적인 이미지입니다. FITC 채널에서 (A) t = 0 및 (B) t = 24h에서 4배 배율로 원시 컨포칼 이미지의 상단 및 측면 보기 비교. (B) (C) t = 0 및 (D) t = 24 h에서 처리 된 셀 위치 Z 높이의 상부 및 측면도 비교. 24시간 동안 처리에는 중앙값 비이동 z-높이의 빼기가 포함됩니다. 눈금 막대 = 500μm. 약어: MAMA = Monolayer-based ascending migration assay. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
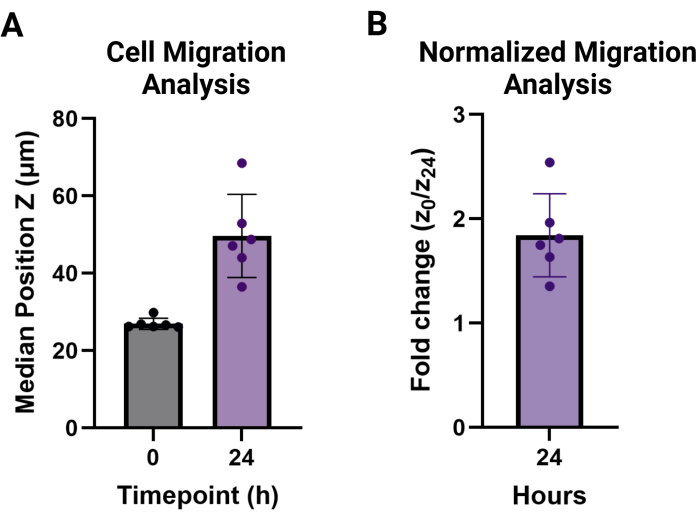
그림 3: MAMA 세포 이동 출력 분석. (A) 시점 t = 0(27.0 μm ± 1.4 μm) 및 t = 24 h(46.6 μm ± 10.8 μm)에서 각 반복(n = 6)에서 세포 높이의 중앙값 위치 Z 및 표준 편차. (B) 24시간에서 세포의 이동을 0시간으로 정규화하고 접힘 변화(1.8 ± 0.4)로 보고했습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
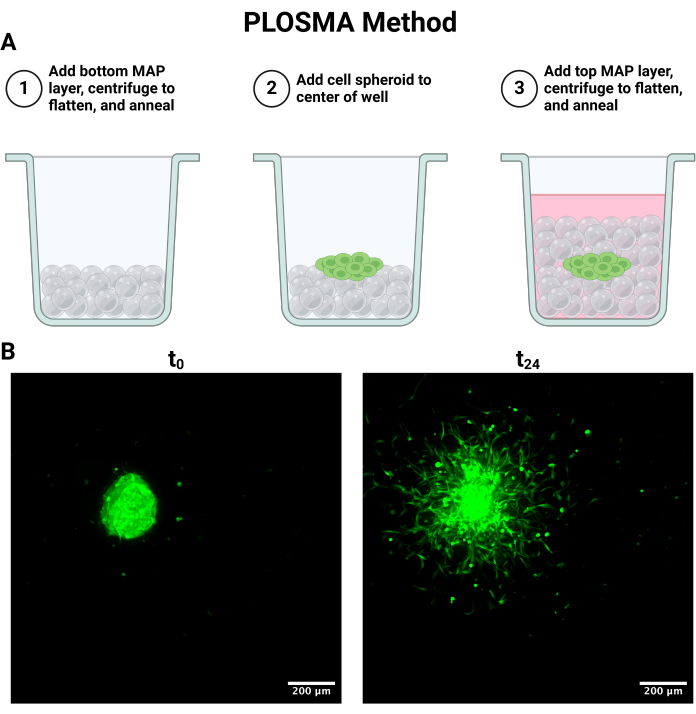
그림 4: PLOSMA(Parallel Layer Outward Spheroid Migration Assay) 세포 배양 및 이미징. (A) 스캐폴드 적층 단계를 설명하는 개략도. (B) 0 및 24 h에서 취한 스페로이드의 최대 강도 투영. 이미지는 10배 배율로 FITC 채널(488nm) 에서 컨포 칼 형광 현미경을 통해 촬영되었습니다. 스케일 바 = 200μm. 약어: PLOSMA = Parallel Layer Outward Spheroid Migration Assay. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
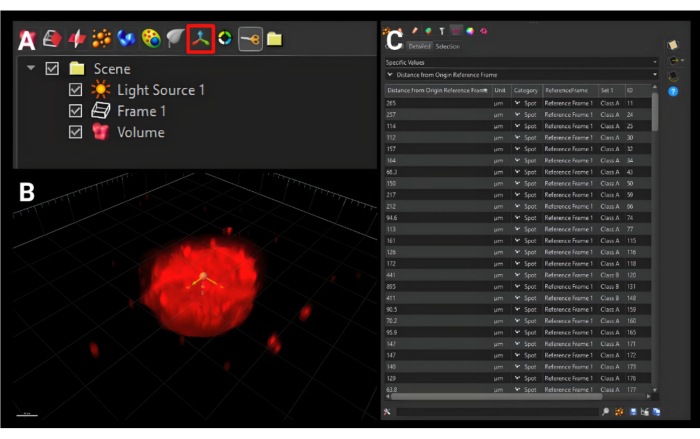
그림 5: 새 원점 참조 프레임 생성. (A) 빨간색 상자로 윤곽선이 표시된 새 원점 참조 프레임 버튼. (B) 새 원점은 3차원 모두에서 회전 타원체의 중심에 있도록 설정됩니다. (C) 표시된 출력 메트릭은 원점 참조 프레임에서 셀 표면까지의 거리이며, 이는 세포가 중심에서 얼마나 멀리 이동했는지를 나타냅니다. 스케일 바 = 120μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
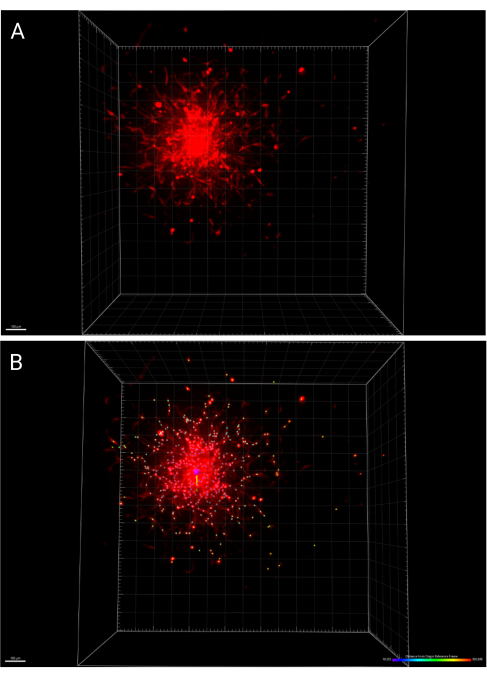
그림 6: 24시간 동안 포함된 스페로이드의 3D 렌더링. (A) 3D 공간에서 처리된 스페로이드. (B) 동일한 스페로이드의 중심은 IMARIS의 원점 참조 프레임 기능을 사용하여 결정되었으며, 세포 확산은 원점으로부터의 거리에 따라 색상으로 구분됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
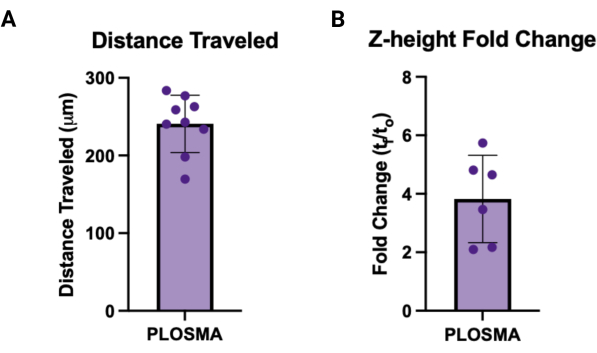
그림 7: PLOSMA의 출력 예. (A) 이동 거리를 μm 단위로 보여주는 PLOSMA 결과의 예. 평균 이동 거리는 240.8μm± 36.87μm였습니다. (B) 스페로이드 발아의 Z-높이 접힘 변화(tf/t0). 평균 폴드 체인지는 3.82 ± 1.495였습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
이 프로토콜은 상처 치유 및 조직 통합을 위해 3D로 세포 이동을 특성화하기 위한 두 가지 체외 모델을 설명합니다. 첫 번째 모델인 단층 기반 이동 분석은 적절하게 부착되고 융합된 세포에 의존합니다. 이 프로토콜은 섬유아세포 유형으로 개발되었으며 1,20,000 cells/cm2의 파종 밀도에서 최적화되었습니다. 이 밀도는 세포가 하룻밤 사이에 웰 플레이트의 바닥을 가로질러 균일하게 최소 80% 포화도까지 성장할 수 있도록 합니다. 이 단계는 최소 24시간 이내에 z 방향으로 마이그레이션을 보장합니다. 겔 층을 추가할 때 포화도가 너무 낮으면 세포가 조직 배양 플라스틱과 겔 내부로 계속 퍼져 나가 균일하지 않고 느린 이동 패턴이 발생할 수 있으며, 이는 최적화 중에 관찰되었습니다. 고르지 않은 이동 높이는 밀도가 낮은 세포 영역에서도 여전히 관찰될 수 있으며, 심지어 80%의 포화도에서도 관찰될 수 있습니다. 음, 복제는 이러한 세포 행동의 소음을 줄일 것입니다. 세포가 과도하게 결합되면 원심분리 기간 동안 세포가 들떠 있고 잠재적으로 세포가 사멸할 수 있습니다. 이러한 변동성은 일관된 수의 세포에서 시딩(seeding)을 통해 해결되며, 적절한 데이터 비교를 위해 일관된 이미지 영역을 캡처합니다. 저자가 아는 한, 플레이트 원심분리는 겔 평탄화에 대해 발표되지 않았지만 원심분리는 세포 통과 및 생체 물질 처리에 일반적으로 사용됩니다21,22. 패시징 속도에 맞게 속도를 조정하면 최적의 세포 처리를 위해 세포 생존력을 유지할 수 있습니다.
이 방법의 주요 과제는 이미징 해상도와 깊이를 최대화하는 동시에 이미징 시간을 최소화하여 최상의 분석을 보장하는 것입니다. 녹색 세포 추적 염료는 5μm 이하의 단계 크기와 1000ms의 노출 시간으로 96웰을 이미징할 수 있을 만큼 충분히 밝습니다. 노출 시간을 줄이면 세포가 배양 상태가 아닌 시간이 줄어들지만 해상도도 감소합니다. 이러한 매개변수는 개별 현미경을 기반으로 최적화해야 하지만, 하나의 연구 내에서 모든 이미지를 동일한 설정으로 캡처하면 변동성을 줄일 수 있습니다.
MAMA 분석에서 중요한 점은 단층 높이 이하의 셀을 제거하여 이동하는 셀만 통계 테스트에 고려되도록 해야 한다는 것입니다. 따라서, 복제 웰의 중앙값은 필터링 후 셀 위치의 비가우스 분포 특성으로 인해 보고됩니다. 그룹 간의 비교는 히스토그램으로 시각화할 수 있으며, 중앙값은 비모수 검정으로 통계적으로 분석할 수 있습니다.
이러한 어려움에도 불구하고 단층 기반 상향 이동 방법은 가장 간단하게도 다공성 스캐폴드의 3D 세포 침투를 위한 재현 가능한 분석법입니다. 세포 이동의 기계론적 효과를 연구하려면 매개변수가 연구 중인 세포 유형에 맞는지 확인하십시오. 여기에는 겔 내 또는 배지 내에 화학주성 또는 촉각성 성분의 첨가가 포함될 수 있습니다. 인간 진피 섬유아세포 완전 배지에는 이동 케모카인이 포함되지만, 보다 특이적인 단서를 사용하는 다른 세포 유형은 그에 따라 분석의 적응이 필요합니다. 이 분석은 여러 유형의 변수를 테스트하는 데 적합합니다. 그러나 이러한 범위는 이 프로토콜에서 다루지 않습니다. MAMA는 생체 내에서 벌크 조직에서 주입된 다공성 하이드로겔로 세포가 이동하는 것과 유사한 생리학적으로 관련된 환경을 제공합니다.
PLOSMA 방법의 경우, 스페로이드를 골격 중앙에 배치하는 것은 성공적인 이미징과 3차원의 의미 있는 세포 이동에 매우 중요합니다. 겔 중앙에 있는 스페로이드의 정확한 파종은 사용자에 따라 다릅니다. 이를 위해 사용자의 주로 사용하지 않는 손으로 배럴에 피펫을 고정하면 센터링에 도움이 되며, 시딩 위치의 효과는 명시야 또는 형광 현미경을 사용하여 확인할 수 있습니다. 중심에서 벗어난 스페로이드는 동일한 스캐폴드 또는 새 스캐폴드에서 새 스페로이드를 사용하여 두 번째 시도로 해결할 수 있습니다. 이러한 이유로 저자는 필요한 것보다 더 많은 스페로이드를 만들고 필요한 것보다 더 많은 MAP 젤을 준비할 것을 권장합니다.
두 번째 층 원심분리 단계는 스페로이드가 (1) 겔에 의해 고르게 덮이고 (2) 전달된 세포를 연구하는 데 중요한 겔 내부로 위아래로 고르게 퍼질 수 있는지 확인합니다. 원심분리는 또한 스페로이드가 중심에서 웰 가장자리로 이동하도록 할 수 있으며, 이 프로토콜은 균일한 분포를 위해 각 층(15μL)에 사용되는 겔의 부피와 원심분리 단계를 최적화하여 이러한 현상을 제한하지만 그 움직임을 완전히 제거하지는 않습니다. 스페로이드 이동을 줄이는 데 필요한 정확한 원심분리 속도와 타이밍은 원심분리기 모델에 따라 조정해야 할 수 있습니다. 그러나, 이 프로토콜에 기술된 사양은 개별 최적화를 위한 벤치마크로 사용될 수 있다. 또 다른 접근법은 젤의 두 번째 층을 추가하기 전에 스페로이드가 스캐폴드에 부착되도록 2시간의 배양 시간을 허용하는 것입니다. 스페로이드 이동은 두 가지 전략이 모두 구현될 때 특히 잘 완화됩니다. 마지막으로, 다단계 원심분리 공정으로 인해 이 방법은 내한성이 낮은 세포주에는 적합하지 않을 수 있습니다.
PLOSMA 방법에서 스페로이드를 도금하는 물류 외에도 이미지 획득 중에는 제한이 있습니다. 스페로이드는 4배 또는 10배 배율을 사용하여 이미징할 수 있지만 최상의 결과를 얻으려면 최소 10배 배율을 사용하고 z 스택의 단계 크기를 2-5μm로 줄이는 것이 좋습니다. 배율은 연구 전반에 걸쳐 일관되어야 합니다. 이미징 시간은 해상도가 높을수록 증가하므로 각 웰 플레이트의 샘플 수(플레이트당 4-8웰)를 제한하여 인큐베이터 외부의 시간을 최소화하십시오. 라이브 이미징 설정은 또한 추적을 개선하고 더 큰 통찰력을 제공할 수 있습니다.
과립 하이드로겔은 고유한 부피, 다공성, 기계적 강도 및 경우에 따라 생체 활성을 포함하는 고유한 토폴로지 및 설계 매개변수를 가지고 있기 때문에 이러한 측면과 관련된 세포 거동을 가능한 한 충실하게 연구해야 합니다. PLOSMA 방법은 전달 후 또는 세포가 과립 겔에 완전히 들어간 후 세포 이동을 모델링하도록 설계되었습니다. 세포는 과립 하이드로겔 기하학에 내재된 기공을 통해 강제로 이동하기 때문에 PLOSMA 방법은 세포 거동에 영향을 미치는 다공성을 효과적으로 분리합니다. 이 분석법의 잠재적인 응용 분야는 입상 골격 내에서의 세포 전달 및 조직 통합, 특히 상처 치유 공간23입니다.
두 프로토콜 모두 조직 복구및 리모델링에서 섬유아세포 이동의 역할로 인해 1차 인간 진피 섬유아세포로 개발되었지만, 4,24, 모든 부착 세포의 이동 거동은 성장 인자의 추가 및 겔의 표면/벌크 구성을 포함하여 다공성 골격의 변경에 대한 반응으로 측정될 수 있습니다. 이러한 변화로 인해 상당한 결과를 얻기 위해 이러한 분석법을 조정해야 할 수 있습니다. 추가 최적화가 필요한 파라미터에는 세포 시딩 밀도, 실험 기간 및/또는 분석 파이프라인이 포함됩니다. IMARIS는 세포 이동 분석에 활용되는 강력한 이미징 분석 도구로, 선택한 '표면' 내의 모든 물체를 표면적, 부피, 강도 및 다른 생성된 표면과의 거리와 같은 다양한 속성을 기반으로 세트로 분류하는 등 여기에 설명된 것 이상의 기능을 갖추고 있습니다. 추가 분석 방법을 결정하기 위한 많은 온라인 리소스가 있습니다.
여기에 요약된 두 가지 방법은 생리학적 방식으로 과립 물질에 대한 조직 도입의 초기 상태뿐만 아니라 물질 내에 완전히 내장되었을 때 후속 세포 반응을 다룹니다. 모든 이동 분석과 마찬가지로 존재하는 세포는 이동과 병행하여 증식할 수 있지만, 설명된 분석의 설계는 증식을 방해하지 않으므로 분석에 과도한 영향을 미치지 않습니다. 두 방법 모두 종단 이미징 외에도 종단 염색과 호환되며, PFA 고정을 사용하여 세포 골격, 콜라겐 침착, 증식 등과 같은 메트릭을 검출합니다. 요약된 방법의 사용은 이전 방법 1,6,14,15,25,26,27과 달리 세포 침투를 측정 가능한 매개변수로 사용하는 3D 세포 이동의 보다 정확한 시공간 표현으로 이동합니다.
공개
저자는 공개할 이해 상충이 없습니다.
감사의 말
이 작업을 위한 자금은 미국 국립보건원(National Institutes of Health) High Priority, Short-Term Project Award(1R56DK126020-01)와 Kurtin Trust의 자선 기부를 통해 부분적으로 지원되었습니다. J.T.는 National Science Foundation Graduate Research Fellowship의 지원을 받았습니다. BioRender.com 로 만든 그림 회로도.
자료
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 647 Phalloidin | ThermoFisher | A22287 | |
| Bovine Serum Albumin | VWR International | 332 | |
| CellTracker Green CMFDS Dye, 1 mg | ThermoFisher | C2925 | 20 x 50 ug units, 492/517 nm |
| Centrifuge | ThermoFisher | 75016085 | ST Plus Series |
| Clear 96 well plate | MilliPore Sigma | CLS3997-50EA | |
| Dimethyl Sulfoxide | Fisher Scientific | MT-25950CQC | 250 mL |
| Fibroblast Basal Medium | ATCC | PCS-201-030 | 480 mL, phenol-red-free |
| Fibroblast Growth Kit - Low Serum | ATCC | PCS-201-041 | 7.5 mM L-glut,5 ng/mL rh FGF basic, 5 ug/mL rh Insulin, 1 ug/mL Hydrocortisone, 50 ug/mL Ascorbic acid, 2% FBS |
| FIJI (ImageJ) | NIH | Public access download | |
| Human Dermal Fibroblasts | ATCC | PCS-201-012 | Adult human dermal fibroblasts |
| ImageXpress Micro Confocal | Molecular Devices | Spinning Disc confocal microscope with 4x, 10x magnifications | |
| IMARIS | Oxford Instruments | 3/4D Imaged Visualizaiton and Analysis Software, Proprietary | |
| Incubator | ThermoFisher | Finnpipette F2 Variable volume Pipettes | HeraCell Vios 160i CO2 Incubator, 165L |
| M-20 Microplate Swinging Bucket Rotor | ThermoFisher | 75003624 | |
| Methylcellulose | Fisher Scientific | 9004-67-5 | Lab grade, powder form |
| Microcentrifuge tube | Fisherbrand | 05-408-129 | 1.5 mL microcentrifuge tubes |
| Paraformaldehyde (4%) | Alfa Aesar | AAJ19943K2 | For fixing |
| Petri dish | Corning | 08-757-100A | Bacteriological Petri Dishes with Lid 35 x 10 mm |
| Pipettes | ThermoFisher | 4642080 | Finnpipette F2 Variable volume Pipettes |
| Sterile PBS | Gibco | 10010-023 | |
| Triton-X | Fisher Scientific | 327371000 |
참고문헌
- Jerka, D., et al. Unraveling endothelial cell migration: Insights into fundamental forces, inflammation, biomaterial applications, and tissue regeneration strategies. ACS Appl Bio Mater. 7 (4), 2054-2069 (2024).
- Edmondson, R., Broglie, J. J., Adcock, A. F., Yang, L. Three-dimensional cell culture systems and their applications in drug discovery and cell-based biosensors. Assay Drug Dev Technol. 12 (4), 207-218 (2014).
- Yamada, K. M., Sixt, M. Mechanisms of 3D cell migration. Nat Rev Mol Cell Biol. 20 (12), 738-752 (2019).
- Grinnell, F. Fibroblast biology in three-dimensional collagen matrices. Trends Cell Biol. 13 (5), 264-269 (2003).
- Kloxin, A. M., Kloxin, C. J., Bowman, C. N., Anseth, K. S. Mechanical properties of cellularly responsive hydrogels and their experimental determination. Adv Mater. 22 (31), 3484-3494 (2010).
- Solbu, A. A., et al. Assessing cell migration in hydrogels: An overview of relevant materials and methods. Mater Today Bio. 18, 100537 (2023).
- Madl, C. M., Katz, L. M., Heilshorn, S. C. Tuning bulk hydrogel degradation by simultaneous control of proteolytic cleavage kinetics and hydrogel network architecture. ACS Macro Lett. 7 (11), 1302-1307 (2018).
- Daly, A. C., Riley, L., Segura, T., Burdick, J. A. Hydrogel microparticles for biomedical applications. Nat Rev Mater. 5 (1), 20-43 (2020).
- Qazi, T. H., Burdick, J. A. Granular hydrogels for endogenous tissue repair. Biomater Biosyst. 1, 100008 (2021).
- Tanner, G. I., Schiltz, L., Narra, N., Figueiredo, M. L., Qazi, T. H. Granular hydrogels improve myogenic invasion and repair after volumetric muscle loss. Adv Healthc Mater. 25, e2303576 (2024).
- Sussman, E. M., Halpin, M. C., Muster, J., Moon, R. T., Ratner, B. D. Porous implants modulate healing and induce shifts in local macrophage polarization in the foreign body reaction. Ann Biomed Eng. 42 (7), 1508-1516 (2014).
- Roosa, C. A., et al. Conjugation of IL-33 to microporous annealed particle scaffolds enhances type 2-like immune responses in vitro and in vivo. Adv Healthc Mater. 13 (25), 2400249 (2024).
- Jaberi, A., et al. Engineering microgel packing to tailor the physical and biological properties of gelatin methacryloyl granular hydrogel scaffolds. Adv Healthc Mater. 13 (25), 2402489 (2024).
- Nandi, S., Brown, A. C. Characterizing cell migration within three-dimensional in vitro wound environments. J Vis Exp. 126, e56099 (2017).
- Qazi, T. H., et al. Anisotropic rod-shaped particles influence injectable granular hydrogel properties and cell invasion. Adv Mater. 34 (12), 2109194 (2022).
- Griffin, D. R., Weaver, W. M., Scumpia, P. O., Di Carlo, D., Segura, T. Accelerated wound healing by injectable microporous gel scaffolds assembled from annealed building blocks. Nat Mater. 14 (7), 737-744 (2015).
- Pruett, L. J., Jenkins, C. H., Singh, N. S., Catallo, K. J., Griffin, D. R. Heparin microislands in microporous annealed particle scaffolds for accelerated diabetic wound healing. Adv Funct Mater. 31 (35), 2104337 (2021).
- Roosa, C., et al. Microfluidic synthesis of microgel building blocks for microporous annealed particle scaffold. J Vis Exp. (184), e64119 (2022).
- Pfaff, B. N., et al. Selective and improved photoannealing of microporous annealed particle (MAP) scaffolds. ACS Biomater Sci Eng. 7 (2), 422-427 (2021).
- Riley, L., Schirmer, L., Segura, T. Granular hydrogels: Emergent properties of jammed hydrogel microparticles and their applications in tissue repair and regeneration. Curr Opin Biotechnol. 60, 1-8 (2019).
- Jo, C. H., Roh, Y. H., Kim, J. E., Shin, S., Yoon, K. S. Optimizing platelet-rich plasma gel formation by varying time and gravitational forces during centrifugation. J Oral Implantol. 39 (5), 525-532 (2013).
- Mironov, V., et al. Fabrication of tubular tissue constructs by centrifugal casting of cells suspended in an in situ. crosslinkable hyaluronan-gelatin hydrogel. Biomaterials. 26 (36), 7628-7635 (2005).
- Vu, L. T., Jain, G., Veres, B. D., Rajagopalan, P. Cell migration on planar and three-dimensional matrices: A hydrogel-based perspective. Tissue Eng Part B Rev. 21 (1), 67-74 (2015).
- Cialdai, F., Risaliti, C., Monici, M. Role of fibroblasts in wound healing and tissue remodeling on Earth and in space. Front Bioeng Biotechnol. 10, 958381 (2022).
- Qazi, T. H., Muir, V. G., Burdick, J. A. Methods to characterize granular hydrogel rheological properties, porosity, and cell invasion. ACS Biomater Sci Eng. 8 (4), 1427-1442 (2022).
- Shaik, R., et al. Cardiac matrix-derived granular hydrogel enhances cell function in 3D culture. ACS Appl Mater Interfaces. 16 (43), 58346-58356 (2024).
- Puiggalí-Jou, A., Asadikorayem, M., Maniura-Weber, K., Zenobi-Wong, M. Growth factor-loaded sulfated microislands in granular hydrogels promote hMSCs migration and chondrogenic differentiation. Acta Biomater. 166, 69-84 (2023).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유