Method Article
Cuantificación de la migración celular tridimensional dentro y hacia biomateriales de hidrogel granular
En este artículo
Resumen
Aquí se presenta un protocolo para la evaluación cuantitativa de la migración celular 3D dentro y hacia la interfaz de hidrogeles granulares.
Resumen
Los andamios de hidrogel granular tienen un potencial significativo en la medicina regenerativa, ya que funcionan como portadores para la entrega de células o como interfaces para la integración de tejidos. Este artículo presenta dos enfoques novedosos para cuantificar la migración celular dentro y dentro de los hidrogeles granulares, destacando las distintas aplicaciones de estos andamios. En primer lugar, se presenta un ensayo de interfaz monocapa celular que simula el crecimiento de tejidos en hidrogeles granulares con fines de integración. En segundo lugar, se describe un ensayo basado en esferoides, diseñado para rastrear el movimiento celular dentro de la matriz de hidrogel, específicamente adecuado para aplicaciones que involucran la administración de células. Ambos métodos permiten mediciones precisas y controladas de la migración celular, proporcionando un conjunto de herramientas completo para los investigadores que utilizan andamios de hidrogel granular. La motivación de estos métodos se deriva de la necesidad de un control personalizado sobre la migración de células dentro del andamio para alinearse con aplicaciones específicas. Al optimizar y estandarizar estas técnicas de cuantificación, los investigadores pueden refinar iterativamente las propiedades del hidrogel granular, asegurando su efectividad en diversos contextos de medicina regenerativa. Este sólido conjunto de herramientas cuantitativas ofrece nuevas oportunidades para mejorar los andamios de hidrogel granular, avanzando en su uso tanto en aplicaciones de administración de células como de integración de tejidos.
Introducción
Los biomateriales para aplicaciones terapéuticas están evolucionando cada vez más hacia modelos más complejos y relevantes de entornos celulares para estudiar la integración de tejidos. Los andamios de biomateriales proporcionan una estructura tridimensional (3D) para el crecimiento celular y tienen como objetivo imitar un tejido deseado 1,2. Los modelos tridimensionales de cultivo celular incluyen matrices naturales y andamios sintéticos que proporcionan a las células una mayor complejidad a través de señales haptotácticas o quimiotácticas 3,4. Los andamios de hidrogel tradicionales se entrecruzan a granel, produciendo una malla nanoporosa que permite la difusión de moléculas pequeñas 5,6, pero requiere degradación para la migración a escala celular a un área de tejido que necesita reparación7. Los hidrogeles granulares son un subconjunto de biomateriales que tienen un alto potencial de traslación clínica debido a su biocompatibilidad, capacidad para adaptarse a formas irregulares y, en muchos casos, su inyectabilidad 8,9. Su naturaleza de bloque de construcción proporciona la ventaja de la porosidad a escala celular para mejorar la infiltración de tejidos y la angiogénesis, así como la modularidad, lo que permite la adición de señales heterogéneas para el comportamiento celular 10,11,12. Comprender la respuesta y el movimiento de las células dentro de un andamio 3D es vital para la relevancia fisiológica en todas las aplicaciones que utilizan biomateriales para la integración de tejidos.
Sin embargo, el estudio del crecimiento tisular en tres dimensiones ha demostrado ser difícil de lograr con precisión cuantitativa. La complejidad ampliada de un entorno 3D requiere modelos in vitro de migración celular que no solo puedan proporcionar información sobre el comportamiento de las células, sino también la optimización de las condiciones de los materiales. Los informes publicados anteriormente sobre la migración de células de andamio granular en 3D han utilizado la siembra tópica para explorar el comportamiento de las células, informando de la infiltración en la estructura porosa y la morfología de la célula13 y otros brotes esferoides.14,15, midiendo la longitud del crecimiento y el número de brotes. Las longitudes de migración de la siembra tópica pueden estar influenciadas de manera desigual por las fuerzas de gravedad y, debido a las limitaciones de la microscopía, los resultados no pueden ser longitudinales. El método de germinación de esferoides se ha limitado a la cuantificación 2D a través de la proyección máxima, que es incapaz de capturar el mecanismo de invasión controlada. Ambos métodos se miden en un plano xy, que carece de los matices necesarios para recapitular completamente el movimiento celular 3D y la infiltración del andamio.
Este protocolo describe dos enfoques para cuantificar la migración celular, como la infiltración in vitro en los andamios porosos de hidrogel granular 3D, específicamente utilizando andamios de partículas recocidas microporosas (MAP) 16,17,18,19. El propósito de los siguientes métodos es estudiar el comportamiento celular en geles granulares mediante el control de la direccionalidad de su migración para el análisis tridimensional. El primer enfoque, el ensayo de migración ascendente basado en una sola capa (MAMA), es un modelo simplificado de integración celular endógena que ilustra las interacciones uniformes entre la célula y el material y sirve como plataforma para representar el entorno inicial en el que las células interactúan con hidrogeles granulares, así como para aislar el comportamiento individual antes de la infiltración del andamio. El segundo, llamado método de ensayo de migración de esferoides hacia afuera en capas paralelas (PLOSMA), es un ensayo de migración de esferoides celulares en 3D, que explora el movimiento de las células cuando están completamente rodeadas por un entorno de andamio complejo y modela el movimiento de las células después de la entrega, así como el movimiento después de que las células han entrado completamente en un gel granular.
Ambos métodos son cuantificables mediante el análisis de imágenes 3D y se pueden aplicar para estudiar y optimizar las interacciones material-célula utilizando puntos de tiempo longitudinales para aplicaciones de medicina regenerativa e ingeniería de tejidos, donde promover o restringir el movimiento celular está dentro de los criterios de diseño. Además, estos métodos aprovechan la centrifugación de placas para la preparación uniforme de ensayos de múltiples pocillos.
Protocolo
Los detalles de los reactivos y el equipo utilizado en el estudio se enumeran en la Tabla de Materiales.
1. Preparación de hidrogel granular
NOTA: Las partículas MAP utilizadas en este protocolo son gel de 3,2 % en peso p/v con 45,88 mg/mL PEG-MAL (10 kDa), 0,82 mg/mL RGD, 8,06 mg/mL MethMal19 y 5,62 mg/mL de reticulante degradable MMP-2. La rigidez mecánica del gel es de 15-20 kPa para que coincida con la rigidez dérmica17.
- Genere partículas de hidrogel granular y prepárese para el cultivo celular con normalidad.
NOTA: Este protocolo describe la preparación estéril de gel de partículas recocidas microporosas, cuya producción se detalla en Roosa et al.18. - Prepare las partículas de hidrogel granular para su uso in vitro esterilizándolas con tres lavados de alcohol isopropílico al 70% seguidos de tres lavados de PBS estéril 1x.
- Prepare un fenil-2,4,6-trimetilbenzoilfosfinato (LAP) de litio estéril de 0,2 mM en una solución de medios disolviendo el polvo de LAP en agua ultrapura y pasando la solución a través de un filtro estéril de 0,22 μm. Añadir esta solución 1:1 v/v con la cantidad de gel necesaria para realizar el experimento.
- Incubar la solución de LAP, gel y medio de 0,2 mM a 37 °C en un rotor de tubo a 20 rpm durante al menos 30 minutos antes de proceder para permitir la difusión de LAP a través de las micropartículas.
- Una vez transcurridos 30 minutos, centrifugar la suspensión de partículas a 18.000 x g durante 5 minutos a 25 °C. Aspirar el sobrenadante.
NOTA: La sequedad general de las partículas MAP puede variar debido a las diferencias en la química, la hidrofilicidad y el tamaño de las partículas. Para mantener la consistencia, se recomienda centrifugar la suspensión de partículas como se describe, mezclar las partículas en el tubo con una pipeta de desplazamiento positivo y luego repetir el paso de centrifugación.
2. Método de ensayo de migración ascendente basado en monocapa (MAMA): cultivo celular e imagen
NOTA: Las células se obtuvieron utilizando el canal FITC (488 nm). El colorante utilizado para las células tenía una excitación a 492 nm y una emisión a 517 nm. El aumento de 10x proporciona un mayor detalle en comparación con el aumento de 4x, pero se puede usar cualquiera de los dos.
- Descongele los fibroblastos dérmicos humanos (HDF) de acuerdo con el protocolo del fabricante. Paso según sea necesario hasta que se logre el paso deseado; generalmente, las células primarias mantienen la composición genética y fenotípica a través de P5.
- Placa de 120.000 células/cm2 para al menos n = 6 en una placa de 96 pocillos o del tamaño de placa deseado utilizando el volumen de medio recomendado por pocillo para la suspensión, aspirando suavemente antes de cada adición. Deje que las células se adhieran durante la noche, como se muestra en la Figura 1A, lo que dará como resultado aproximadamente un 80% de confluencia al día siguiente.
NOTA: Por lo general, los pocillos de la columna central y de la hilera son los mejores para una distribución óptima del gel. Los autores utilizan hasta 24 pocillos para una placa de 96 pocillos (filas B-G y columnas 5-8). - Prepare las condiciones de gel como se indicó anteriormente y, mientras incuba en LAP, retire el medio con un aspirador o una pipeta de la placa de pocillos de las células. Tenga cuidado de no alterar el fondo del plato.
- Agregue el tinte de seguimiento celular a los pocillos de acuerdo con las instrucciones del fabricante, representadas en la Figura 1B. Asegúrese de que el gel esté completamente preparado como se describe en el paso 1 antes de aspirar el tinte de seguimiento celular de los pocillos.
- Agregue 20 μL de cada condición de gel a los pocillos con una pipeta de desplazamiento positivo sin tocar el fondo de la placa.
NOTA: Las mejores condiciones de ensayo ocurren cuando el gel se pipetea directamente al centro del pocillo. - Utilizando un accesorio de centrífuga de rotor giratorio de placas a 25 °C, gire a 100 x g durante 15 s con una aceleración y desaceleración de 8 para aplanar el gel. Voltee la placa 180° y vuelva a girar a 100 x g durante 15 s para asegurar una distribución uniforme del gel en el fondo del pozo, como se ve en la Figura 1C.
- Reticulación fotoaséptica el gel desde la parte superior (Figura 1D) aplicando luz focalizada (365 nm, 34,4 mW/cm2) a la muestra durante 30 s para recocer el andamio, añadiendo 200 μL de medio a cada pocillo de células después de que se hayan formado todos los andamios. Deje que las células se incuben a 37 °C durante 30 minutos para permitir que se adhieran al andamio granular antes de la obtención de imágenes.
- Para capturar el comportamiento de migración resumido en la Figura 1E, tome imágenes de las células utilizando un microscopio confocal. Encuentre el punto de enfoque más bajo para un área de la placa donde las celdas sean al menos 80% confluentes y establézcalo como el borde inferior de la pila z.
- Encuentre el punto más alto de la señal fluorescente de la celda y configúrelo como el borde superior de la pila z. Utilice un tamaño de paso de 5 μm o menor para obtener la mejor resolución.
- Visualice al menos tres pocillos para representar la monocapa de la célula y las alturas de las células no migratorias. El punto de tiempo t = 0 se muestra tanto desde la vista superior como desde la vista lateral en la Figura 2A. Incubar durante la noche después de que se completen las imágenes.
NOTA: Estas células se fotografiaron con un objetivo 4x y la exposición se mantuvo constante para todos los pocillos. - A t = 24 h, las células comenzarán a ascender a través del andamio granular, como se ve en la Figura 2B desde las vistas superior y lateral. Repita los pasos de imagen realizados para t = 0 h utilizando los mismos parámetros. Utilice la altura anterior del escenario como referencia o busque las celdas que aún no se hayan migrado y establézcalas como el borde inferior de la pila z.
- Repita los pasos para todos los pocillos, asegurándose de que cada pocillo se guarde como una imagen separada para facilitar el análisis.
NOTA: Se pueden obtener imágenes de puntos de tiempo más largos dependiendo de las restricciones experimentales y el método de fluorescencia de seguimiento celular. - Los andamios se pueden fijar y teñir para métricas adicionales. En el punto de tiempo final deseado, aspire el medio de los pocillos con una pipeta y deséchelo. Lave suavemente cada pocillo con 200 μL de PBS estéril dos veces durante 5 minutos cada una. Agregue 200 μL de paraformaldehído (PFA) al 4% durante 20 minutos, luego aspire y deséchelo. Los pocillos se pueden teñir inmediatamente o almacenar a 4 °C en 1x PBS hasta por una semana.
3. Método de ensayo de migración ascendente (MAMA) basado en monocapa: análisis de imágenes en 3D
- Para conversiones por lotes, abra el software de conversión de imágenes IMARIS. Arrastre y suelte las imágenes de microscopía en el software de conversión y elija una carpeta dentro del software Arena para importar. Presione Iniciar todo. Las imágenes convertidas aparecerán en la Arena como archivos .ims.
NOTA: Si el tamaño del vóxel no está incluido en los metadatos de la imagen, consulte las especificaciones de imágenes confocales individuales para encontrar el valor. Alternativamente, el tamaño del vóxel se puede aproximar como el tamaño del paso utilizado durante la creación de imágenes. - Abra el software haciendo doble clic en el icono de IMARIS Arena en el escritorio y seleccionando una imagen de la Arena.
- La imagen se carga automáticamente en la pestaña de análisis 'Vista 3D' que se ve en el panel de iconos de la barra de herramientas en la parte superior. Haga clic en la pestaña Image Proc en la barra de herramientas principal.
- En la parte superior izquierda del panel lateral, haz clic en el menú desplegable del canal 1 y selecciona Sustracción de fondo. Presione Ok en la parte inferior del panel para volver a 'Vista 3D'. Las imágenes representativas para t = 0 h y t = 24 h se encuentran en la Figura 2A, B, respectivamente.
- En la pequeña barra de herramientas justo encima del menú del panel lateral, haga clic en el icono con formas azules redondeadas, Agregar nuevas superficies, para crear una pestaña de objetos editables llamada 'Superficies 1'.
- En la parte inferior del panel de menú se abrirá una interfaz para la configuración del algoritmo de los parámetros. Genere manualmente los parámetros que se utilizarán para todas las réplicas haciendo clic en el botón de flecha azul en la parte inferior de la interfaz. Asegúrese de seleccionar el canal de origen correcto y marque la casilla 'Suavizar'.
- Establezca el detalle de la superficie en 0,7 μm y seleccione Resta de fondo (contraste local). Escriba la longitud promedio de la celda en el cuadro 'Diámetro de la esfera más grande que quepa en el objeto'. Presione la misma flecha azul en la parte inferior cuando termine.
NOTA: Este valor se puede estimar utilizando la pestaña 'Corte' en la barra de herramientas y midiendo el ancho de las celdas promedio.
- Establezca el detalle de la superficie en 0,7 μm y seleccione Resta de fondo (contraste local). Escriba la longitud promedio de la celda en el cuadro 'Diámetro de la esfera más grande que quepa en el objeto'. Presione la misma flecha azul en la parte inferior cuando termine.
- Para el umbral, determine el histograma de intensidad en el que solo se segmentan las celdas más brillantes. Con la segmentación, muévase hacia arriba y hacia abajo por la pila de imágenes para asegurarse de que sea lo más precisa posible.
- Seleccione Habilitar para 'Dividir objetos que se tocan (Región creciente)' y establezca el Diámetro de los puntos de semilla en el mismo diámetro que se utilizó anteriormente. Asegúrese de que el umbral "Basado en intensidad" esté seleccionado y haga clic en el botón de flecha azul hacia la derecha.
- Los dos pasos siguientes, Filtrar puntos de semilla y Filtrar superficies, se pueden ajustar mediante varias mediciones para garantizar que las superficies generadas sean precisas; Sin embargo, el análisis de línea base no requiere ningún filtrado adicional. Cuando no sea necesario realizar ningún cambio, haga clic en el botón de flecha azul .
NOTA: El paso final permite una clasificación adicional de la superficie en función de la salida deseada. Una vez realizadas las ediciones, haga clic en el botón de flecha verde para terminar de crear las superficies. - Para guardar los parámetros de creación para el análisis por lotes, haga clic en el icono de la varita Creación. Haga clic en Almacenar parámetros para lote y asígnele un nombre, luego haga clic en Aceptar. Las imágenes procesadas representativas para t = 0 y t = 24 h se muestran en las Figuras 2C, D, respectivamente.
NOTA: Todos los objetos dentro de una 'Superficie' seleccionada se pueden clasificar en conjuntos en función de varias propiedades proporcionadas por el software de conversión de imágenes, como el área de la superficie, el volumen, la intensidad y la distancia de otras superficies creadas. - Las propiedades de la superficie se encuentran seleccionando la pestaña Estadísticas . Para reunir todas las alturas de celda, haga clic en la pestaña Detallado y seleccione Valores específicos y Posición Z en los menús desplegables sucesivos. Haga clic en el icono Guardar para guardar todas las posiciones z y las clasificaciones realizadas en un archivo .xls. Repita el procedimiento para todas las imágenes.
- Encuentre la posición z mediana de las células que no migran de la imagen representativa y reste todas las posiciones z por debajo de ese número de cada pocillo de condición de prueba.
NOTA: Los valores de migración se indican como medias para cada condición de réplica técnica por encima de la altura de celda no migratoria. Pueden informarse como altura mediana, como se observa en la Figura 3A, o como cambio de pliegue en la altura de migración para el punto de tiempo deseado en comparación con t = 0, que se ve en la Figura 3B.
4. Método del ensayo de migración de esferoides hacia afuera en capas paralelas (PLOSMA): cultivo celular y cultivo de gotas colgantes para esferoides 3D
NOTA: Este protocolo describe el cultivo celular y el cultivo en gota colgante adaptado del protocolo escrito por Nandi et al.14.
- Descongele los discos duros de acuerdo con el protocolo del fabricante. Paso según sea necesario hasta que se logre el paso deseado.
- En una campana de cultivo celular aséptico, prepare una placa de Petri agregando 10 ml de PBS en el fondo y volteando la tapa para que el exterior quede encima de la campana de cultivo celular.
- Agregue el volumen apropiado de células (determinado a partir del recuento de células) a un tubo de microcentrífuga. Agregue el tinte de rastreo celular diluido en el medio. Lleve el volumen total a 1 ml con medios calientes.
NOTA: Los esferoides deben ser aproximadamente 8000 celdas por esferoide, pero pueden cambiar según el tipo de celda. - Incubar la solución celular durante 45 min a 37 °C. Gire las celdas de acuerdo con la velocidad recomendada por el fabricante y aspire el sobrenadante.
- Resuspender las células en metilcelulosa 1:100 en los medios.
- Pipetee 20 μL de gotas de la solución de célula/medio en la tapa de la placa de Petri.
- Invierta la tapa con confianza, rapidez y cuidado y colóquela encima de la mitad inferior de la placa de Petri que contiene PBS.
- Incubar las gotitas durante al menos 24 h.
NOTA: La formación de esferoides se puede monitorear mediante microscopía de campo claro.
5. Método de ensayo de migración de esferoides hacia afuera en capas paralelas (PLOSMA): siembra de esferoides celulares en hidrogel granular
NOTA: El siguiente proceso se resume en la Figura 4A.
- Para configurar el método PLOSMA descrito en la Figura 4A, agregue asépticamente 15 μL de gel usando una pipeta de desplazamiento positivo a los pocillos en una placa transparente de 96 pocillos.
- Con un accesorio de centrífuga de rotor giratorio de placas, gire a 1000 x g durante 10 s para aplanar el gel. Voltee la placa 180° y vuelva a girar a 1000 x g durante 10 s para garantizar una distribución uniforme del gel en el fondo del pozo.
- Una vez que se haya logrado una planitud uniforme, se foto-entrecruza asépticamente el gel desde la parte superior aplicando luz focalizada (365 nm, 33,4 mW/cm2) a la muestra durante 30 s para recocer el andamio.
- Mueva asépticamente la placa de Petri de gotas colgantes a la campana de cultivo de tejidos aséptico e invierta la tapa.
- Con una pipeta de 20 μL, absorba lentamente una gota hasta que el esferoide entre en la punta de la pipeta. Expulse la gota sobre el andamio en el centro del pozo.
- Repita los pasos anteriores para todos los pozos. Asegúrese de que cada pocillo tenga un esferoide confirmándolo con microscopía de campo claro o fluorescente.
- Incubar la placa del pocillo a 37 °C durante 2 h para permitir que los esferoides se adhieran al andamio.
- Pipetee 15 μL adicionales de gel encima de cada esferoide. Para asegurarse de que haya una distribución uniforme del gel, centrifugue la placa a 300 x g durante 15 s en cada dirección.
- Recocer la capa superior de gel durante 30 s utilizando luz UV (365 nm) a 33,4 mW/cm2. Pipetea los medios en la parte superior de cada andamio para llevar el volumen total del pocillo a 200 μL.
NOTA: Los andamios estarán muy secos en este punto, así que agregue medios gota abajo por el costado del pozo para evitar que se desprenda el esferoide.
6. Método de ensayo de migración de esferoides hacia afuera en capas paralelas (PLOSMA): imágenes confocales de esferoides
NOTA: La facilidad de obtención de imágenes depende del sistema de imágenes. Ubique el esferoide dentro del pozo a un tiempo de exposición bajo. Las imágenes de las células se utilizaron utilizando el canal FITC (488 nm). El colorante utilizado para las células tenía una excitación a 492 nm y una emisión a 517 nm. El aumento de 10x proporciona un mayor detalle en comparación con el aumento de 4x.
- Encuentre el nivel de etapa más bajo (altura z) en el que las celdas aún están enfocadas. Establézcalo como el límite inferior de la pila z.
- Encuentre el nivel de etapa más alto (altura z) en el que las celdas aún están enfocadas. Establézcalo como el límite superior de la pila z.
NOTA: Los mejores resultados de imagen incluyen un tamaño de paso menor o igual a 5 μm para mantener la resolución a escala celular. Puede haber compensaciones entre la velocidad y la resolución de la imagen dependiendo del sistema de microscopio confocal. - Imagine todos los esferoides como se describe en t = 0 y 24 h. También se puede obtener una imagen de un punto de tiempo de 48 horas dependiendo de las restricciones experimentales. En la Figura 4B se observan imágenes representativas de proyección máxima de t = 0 y t = 24.
7. Método de ensayo de migración de esferoides hacia afuera en capas paralelas (PLOSMA): análisis de imágenes en 3D
- Importe imágenes en el software de análisis como se describe en los pasos 3.1 a 3.4. En la esquina superior derecha del panel izquierdo, haz clic en el menú desplegable del Canal 1 y selecciona Sustracción en segundo plano. Presione Ok en la parte inferior del panel.
- Una vez de vuelta en la vista 3D, presione Ajuste automático de todos los canales en la ventana emergente Ajuste de pantalla y corrija según sea necesario.
- En la barra de herramientas más pequeña justo encima del menú lateral, haga clic en el icono Agregar nuevo marco de referencia que se muestra en la Figura 5A con tres flechas ortogonales para agregar una nueva pestaña llamada 'Marco de referencia 1'.
- Mueva el origen al centro del esferoide en los tres planos, como se visualiza en la Figura 5B.
- En la misma barra de herramientas que las tres flechas ortogonales, haga clic en el icono con esferas naranjas y agregue nuevos Puntos para crear una pestaña llamada 'Puntos 1'. Presione el botón de flecha azul.
- En Detección puntual, establezca el Diámetro XY estimado en el diámetro estimado de las celdas. Presione el botón de flecha azul.
NOTA: En el caso de las HDF, este número es de 15,0 μm. - Para el umbral, ajuste el histograma de intensidad para rodear solo las partes más brillantes. Con la segmentación, muévase hacia arriba y hacia abajo por la pila de imágenes para asegurarse de que sea lo más precisa posible. Pulsa la siguiente flecha azul.
- Presione el botón verde Ejecutar para finalizar el análisis.
- Desmarque Renderizar en segmentación o haga clic en el icono cuadrado amarillo en el lado derecho del panel de configuración.
- Haga clic en la pestaña Estadísticas . En el primer menú desplegable, seleccione Valores específicos. En el segundo menú desplegable, seleccione Distancia desde el marco de referencia de origen. Se mostrarán todos los valores de las superficies seleccionadas, como se ve en la Figura 5C. Haga clic en el icono de guardado único, que descarga un archivo .xls.
- Guarde los cambios realizados en la imagen y los análisis pulsando el icono Guardar en la barra de herramientas principal. La Figura 6A representa una representación en 3D de un esferoide fotografiado a las 24 horas, mientras que la Figura 6B representa la función IMARIS Spots que marca las celdas dispersas, codificadas por colores de acuerdo con la distancia desde el marco de referencia de origen.
- Normalice los datos exportados a las imágenes t = 0 y calcule la media de la distancia celular recorrida y la altura z para cada esferoide para obtener un valor único para cada muestra. Las figuras 7A y B muestran gráficos representativos para cada salida, respectivamente.
Resultados
Este protocolo tiene como objetivo detallar los pasos necesarios para dos nuevos ensayos de migración de andamios granulares. El método MAMA se puede utilizar para evaluar la infiltración celular en una interfaz tisular. Los hidrogeles granulares son un sistema más complejo que los hidrogeles a granel y, por lo tanto, son inherentemente más complejos de procesar para la migración 9,20. Es importante comprender el proceso paso a paso descrito en la Figura 1. Cada paso se basa en el siguiente y se ha optimizado en este protocolo. La siembra de HDF a una densidad de 120.000 células/cm2 dará como resultado al menos un 80% de confluencia durante la noche (Figura 1A), y estas células no fluorescentes se etiquetan mejor con tinte de seguimiento celular el día del experimento para maximizar el potencial de imagen (Figura 1B). Este protocolo coincide con la fuerza centrífuga más baja utilizada en el paso de HDF para mantener la viabilidad de la celda. Debido al ángulo que se produce para un solo paso de centrifugación, es necesario voltear la placa 180° para asegurar que el gel se desplace y cubra completamente la superficie inferior de los pocillos (Figura 1C). Permitir que las células se recuperen en la incubadora durante 30 minutos después del recocido (Figura 1D) mantendrá la viabilidad de la célula y dará como resultado una migración óptima (Figura 1E). Se puede obtener una imagen de un área grande de una placa de 96 pocillos con un objetivo 4x y compararla desde el punto de tiempo 0-24 h (Figura 2A, B) para evaluar ampliamente el comportamiento de la célula. El procesamiento de las imágenes de pila z resultantes en el software de análisis proporciona análisis avanzados para múltiples conjuntos de datos grandes en una interfaz fácil de usar. Este protocolo resume los pasos para crear conjuntos de datos para la altura de la celda, o posición Z, en cada punto de tiempo visualizado con las imágenes representativas de la Figura 2B, C. El análisis de los datos procesados se observa en la Figura 3A, visualizado utilizando la media de las alturas medianas y su desviación estándar de cada punto de tiempo, y la altura de cambio de pliegue de las celdas no migratorias a t = 0 h se muestra en la Figura 3B. Los datos subyacentes de este método suelen tener una distribución no normal, por lo que las medianas son medidas más sólidas para la comparación y, por lo tanto, se utilizan para resumir los datos.
Del mismo modo, el método PLOSMA se puede utilizar para evaluar la motilidad de las células entregadas dentro de un andamio de hidrogel granular 3D. La Figura 4A describe los pasos del método PLOSMA, y es especialmente importante sembrar el esferoide en el centro del pozo. Se recomienda centrar el esferoide en el campo de visión, pero depende de las capacidades del microscopio. La Fig. 4B muestra imágenes representativas de la dispersión del esferoide para t = 0 h y t = 24 h tomadas con un aumento de 10x en el canal FITC (488 nm). En el software, se puede crear un marco de referencia de origen y ajustarlo a cada pila z (Figura 5A, B). El software puede rastrear la distancia radial desde ese marco de referencia de origen y exportarlo como el conjunto de datos deseado (Figura 5C). La Figura 6A muestra una imagen representativa de la representación 3D del esferoide t = 24 h, mientras que la Figura 6B muestra la función Spots del software. En la Figura 7 se muestra un ejemplo de los datos procesados. La Figura 7A representa la distancia promedio recorrida desde el centro normalizada a las distancias del Día 0. La Figura 7B aísla las distancias recorridas solo en el plano z, ya que esa es la dirección que el método PLOSMA pretende estudiar.
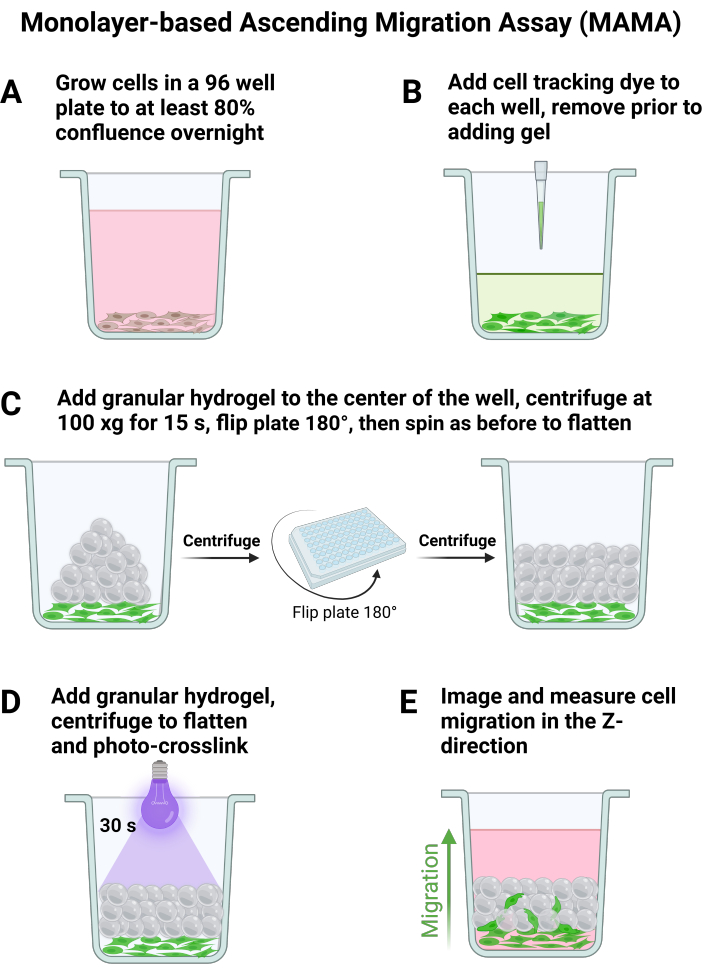
Figura 1: Ensayo de migración ascendente basado en monocapa cultivo e imágenes celulares. Esquema de los principales pasos en el procesamiento de células y geles para MAMA. (A) Las células se cultivan hasta la confluencia durante la noche, y (B) se agrega un tinte de seguimiento celular justo antes de la adición de gel granular. El andamio se ensambla mediante centrifugación de placas (C) y se estabiliza con fotorreticulación (D). La obtención de imágenes en múltiples puntos de tiempo permite (E) la visualización de la migración celular ascendente. Haga clic aquí para ver una versión más grande de esta figura.
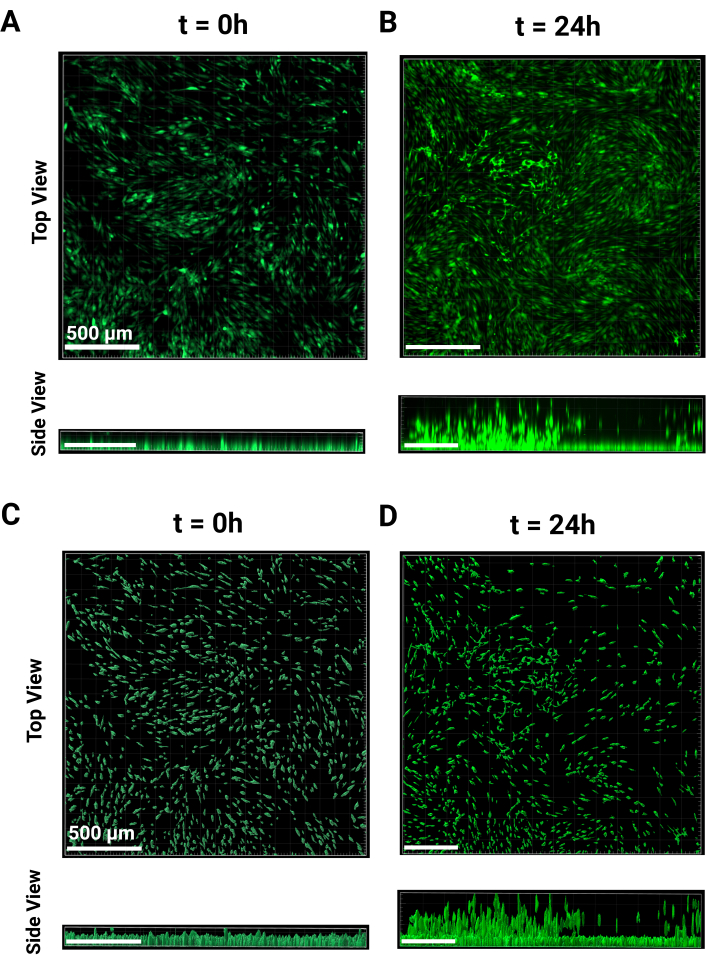
Figura 2: Procesamiento de imágenes MAMA. Imágenes representativas del procesamiento de imágenes. Comparación de la vista superior y lateral de imágenes confocales sin procesar a (A) t = 0 y (B) t = 24 h en el canal FITC con un aumento de 4x. (B) Comparación de la vista superior y lateral de las alturas Z de la posición de la celda procesada a (C) t = 0 y (D) t = 24 h. El procesamiento durante 24 h incluye la sustracción de las alturas z medianas no migratorias. Barras de escala = 500 μm. Abreviaturas: MAMA = Ensayo de Migración Ascendente basado en Monocapa. Haga clic aquí para ver una versión más grande de esta figura.
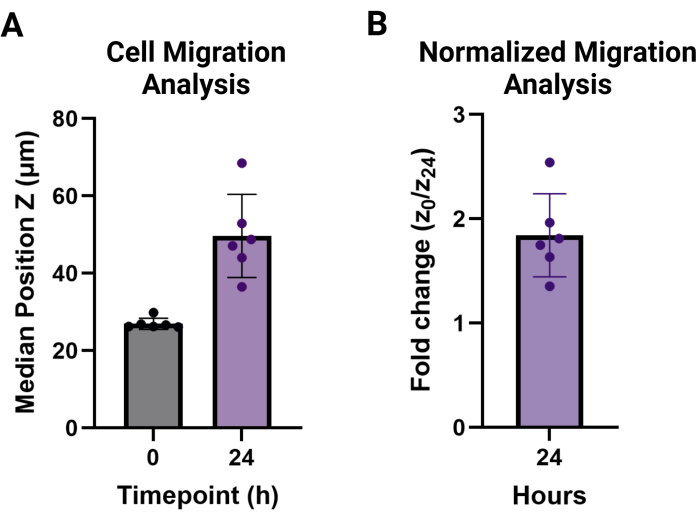
Figura 3: Análisis de la salida de la migración de células MAMA. (A) Posición mediana Z y desviación estándar de las alturas de las celdas en cada réplica (n = 6) en los puntos de tiempo t = 0 (27,0 μm ± 1,4 μm) y t = 24 h (46,6 μm ± 10,8 μm). (B) La migración de las células a las 24 h se normalizó a 0 h y se informó como cambio de pliegue (1,8 ± 0,4). Haga clic aquí para ver una versión más grande de esta figura.
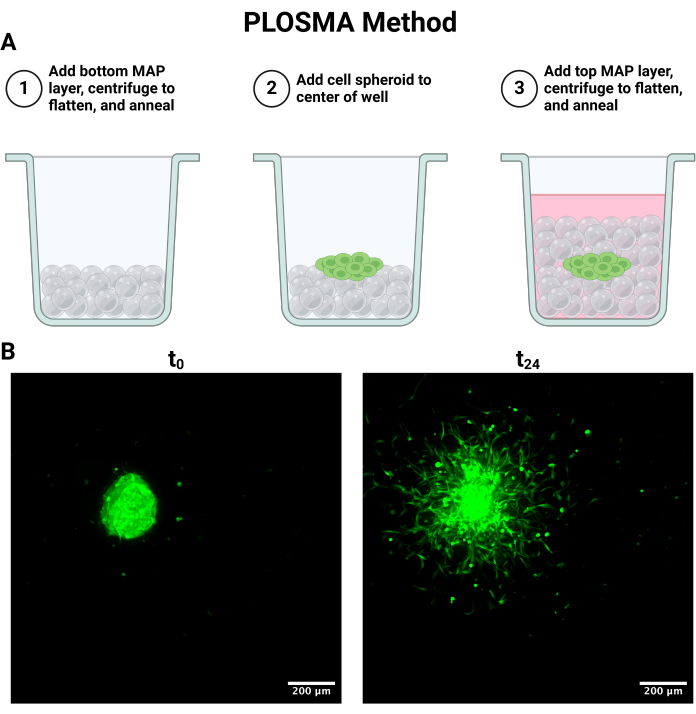
Figura 4: Cultivo e imágenes celulares del ensayo de migración de esferoides hacia afuera de capa paralela (PLOSMA). (A) Esquema que describe los pasos de la estratificación del andamio. (B) Proyecciones de intensidad máxima del esferoide tomadas a las 0 y 24 h. Las imágenes se tomaron mediante microscopía fluorescente confocal en el canal FITC (488 nm) con un aumento de 10x. Barras de escala = 200 μm. Abreviaturas: PLOSMA = Ensayo de Migración de Esferoides hacia el Exterior de Capa Paralela. Haga clic aquí para ver una versión más grande de esta figura.
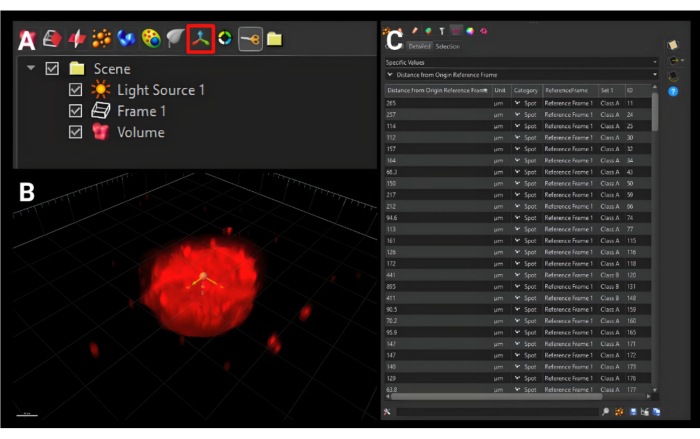
Figura 5: Creación de un nuevo marco de referencia de origen. (A) El botón del nuevo marco de referencia de origen delineado con un cuadro rojo. (B) El nuevo origen está configurado para estar en el centro del esferoide en las tres dimensiones. (C) Las métricas de salida que se muestran son las distancias de las superficies de las celdas desde el marco de referencia de origen, que describe qué tan lejos han migrado las celdas desde el centro. Barra de escala = 120 μm. Haga clic aquí para ver una versión más grande de esta figura.
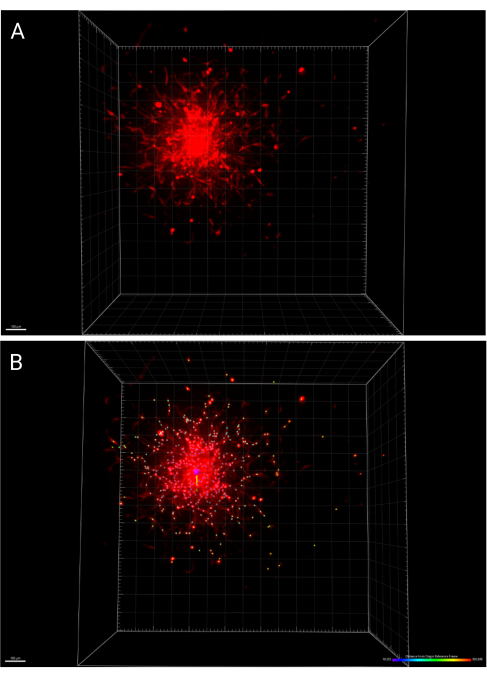
Figura 6: Representaciones 3D del esferoide incrustado a las 24 h. (A) Esferoide procesado en el espacio 3D. (B) El centro del mismo esferoide se determinó utilizando la función de marco de referencia de origen en IMARIS, y la dispersión de celdas está codificada por colores según la distancia desde el origen. Haga clic aquí para ver una versión más grande de esta figura.
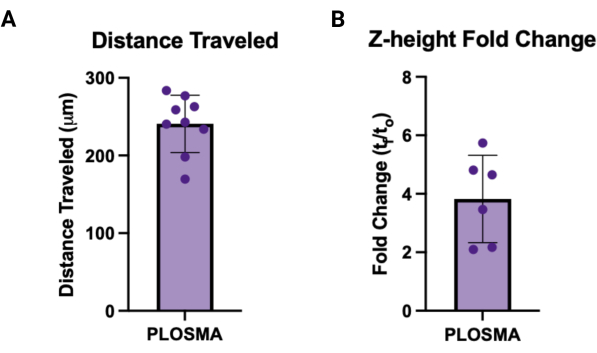
Figura 7: Ejemplos de salidas para PLOSMA. (A) Ejemplos de resultados de PLOSMA que muestran la distancia recorrida en μm. La distancia media recorrida fue de 240,8 μm ± 36,87 μm. (B) Cambio de plegamiento en la altura Z (tf/t0) de la brotación de esferoides. El cambio medio de plegado fue de 3,82 ± 1,495. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Este protocolo describe dos modelos in vitro para caracterizar la migración celular en 3D para la cicatrización de heridas y la integración de tejidos. El primer modelo, el ensayo de migración basado en una sola capa, se basa en células correctamente unidas y confluentes. Este protocolo se desarrolló con un tipo de célula de fibroblastos y se optimizó a una densidad de siembra de 1,20,000 células/cm2. Esta densidad permite que las células crezcan durante la noche hasta al menos un 80% de confluencia uniforme en el fondo de la placa del pocillo. Este paso asegura la migración en la dirección z en al menos 24 h; Si la confluencia es demasiado baja tras la adición de la capa de gel, las células pueden continuar extendiéndose a través del plástico de cultivo de tejidos, así como en el gel, lo que resulta en un patrón de migración lento y no uniforme, que se observó durante la optimización. Todavía se pueden observar alturas de migración desiguales en áreas de células menos densas, incluso con un 80% de confluencia. Las réplicas bien reducirán el ruido de estos comportamientos celulares. Las células demasiado confluentes pueden causar el levantamiento de las células durante el período de centrifugación y, potencialmente, la muerte celular. Esta variabilidad se aborda mediante la siembra en un número constante de celdas y la captura de un área de imagen coherente para permitir comparaciones de datos adecuadas. Hasta donde sabe el autor, la centrifugación en placa no se ha publicado para el aplanamiento en gel, pero la centrifugación se usa comúnmente para el paso de células y el manejo de biomateria21,22. El ajuste de la velocidad para que coincida con las velocidades de paso mantendrá la viabilidad de la célula para un procesamiento óptimo de la célula.
El principal desafío de este método es maximizar la resolución y la profundidad de las imágenes, al tiempo que se minimiza el tiempo de obtención de imágenes para garantizar el mejor análisis. El tinte de seguimiento de células verdes es lo suficientemente brillante como para obtener imágenes de 96 pocillos con un tamaño de paso de 5 μm o menos y hasta 1000 ms de tiempo de exposición. Reducir el tiempo de exposición reduce la cantidad de tiempo que las células no están en condiciones de incubación, pero también reduce la resolución. Estos parámetros deben optimizarse según el microscopio individual, pero la variabilidad se reduce al garantizar que todas las imágenes se capturen con los mismos ajustes dentro de un estudio.
Una nota importante para el análisis de MAMAs es que requiere eliminar las células a la altura de la monocapa o por debajo de ella para garantizar que solo las células migratorias se consideren para las pruebas estadísticas. En consecuencia, las medianas de los pozos replicados se informan debido a la naturaleza de distribución no gaussiana de las posiciones de las celdas después del filtrado. La comparación entre grupos se puede visualizar con un histograma, y las medianas se pueden analizar estadísticamente con una prueba no paramétrica.
A pesar de estos desafíos, el método de migración ascendente basado en una sola capa es, en su forma más simple, un ensayo reproducible para la infiltración celular 3D de andamios porosos. Para estudiar los efectos mecanicistas de la migración celular, asegúrese de que los parámetros se ajusten al tipo de célula que se está estudiando. Esto puede incluir la adición de componentes quimiotácticos o haptotácticos, dentro del gel o en los medios. Los medios completos de fibroblastos dérmicos humanos incluyen quimiocinas migratorias, pero otros tipos de células que utilizan señales más específicas requieren una adaptación del ensayo en consecuencia. Este ensayo se presta para probar múltiples tipos de variables; Sin embargo, el ámbito de aplicación de estos no está cubierto en el presente Protocolo. El MAMA proporciona un entorno fisiológicamente relevante análogo al movimiento celular de tejido a granel a un hidrogel poroso inyectado in vivo.
Para el método PLOSMA, la colocación de los esferoides en el centro del andamio es fundamental para obtener imágenes exitosas y una migración celular significativa en tres dimensiones. La siembra exacta del esferoide en el centro del gel depende del usuario. Con este fin, estabilizar la pipeta en el barril con la mano no dominante del usuario ayuda a centrar, y la eficacia de la posición de siembra se puede confirmar mediante microscopía de campo claro o fluorescente. Un esferoide descentrado se puede remediar mediante un segundo intento con un nuevo esferoide, ya sea en el mismo andamio o en un andamio nuevo. Por esta razón, los autores recomiendan crear más esferoides de los necesarios y preparar más gel MAP de lo necesario.
El paso de centrifugación de la segunda capa garantiza que el esferoide esté (1) cubierto uniformemente por el gel y (2) pueda extenderse uniformemente hacia arriba y hacia abajo en el gel, lo cual es crucial para estudiar las células administradas. La centrifugación también puede hacer que el esferoide se mueva desde el centro hacia los bordes del pocillo, y aunque este protocolo limita este fenómeno al optimizar los pasos de centrifugación y el volumen del gel utilizado para cada capa (15 μL) para una distribución uniforme, no elimina por completo su movimiento. Es posible que sea necesario ajustar la velocidad y el tiempo exactos de centrifugación necesarios para reducir el movimiento del esferoide de acuerdo con el modelo de la centrífuga; Sin embargo, la especificación descrita en este protocolo puede utilizarse como punto de referencia para la optimización individual. Otro enfoque es permitir que los esferoides 2 h de tiempo de incubación se adhieran al andamio antes de agregar la segunda capa de gel. El movimiento de los esferoides se mitiga particularmente bien cuando se implementan ambas estrategias. Finalmente, debido al proceso de centrifugación de varios pasos, este método puede no ser adecuado para líneas celulares menos resistentes.
Aparte de la logística de la siembra de los esferoides en el método PLOSMA, existen limitaciones durante la adquisición de imágenes. El esferoide se puede visualizar con un aumento de 4x o 10x, pero para obtener los mejores resultados, use al menos un aumento de 10x y reduzca el tamaño del paso de las pilas z a 2-5 μm. El aumento debe ser constante a lo largo del estudio. El tiempo de obtención de imágenes aumenta con una resolución más alta, por lo tanto, limite el número de muestras en cada placa de pocillos (4-8 pocillos por placa) para minimizar el tiempo fuera de la incubadora. Una configuración de imágenes en vivo también podría mejorar el seguimiento y proporcionar más información.
Debido a que los hidrogeles granulares tienen una topología y parámetros de diseño únicos que incluyen volumen inherente, porosidad, resistencia mecánica y, en algunos casos, bioactividad, es necesario estudiar el comportamiento de las células en relación con estos aspectos con la mayor fidelidad posible. El método PLOSMA está diseñado para modelar el movimiento de las células después del parto o después de que las células hayan entrado completamente en un gel granular. Debido a que las células se ven obligadas a migrar a través de los poros inherentes a la geometría del hidrogel granular, el método PLOSMA aísla eficazmente la porosidad como una influencia en el comportamiento de la célula. Las aplicaciones potenciales de este ensayo son la administración de células in situ y la integración de tejidos dentro de un andamio granular, particularmente en el espacio de cicatrización de heridas23.
Ambos protocolos se desarrollaron con fibroblastos dérmicos humanos primarios debido al papel de la migración de fibroblastos en la reparación y remodelación de los tejidos 4,24, sin embargo, el comportamiento migratorio de las células adherentes puede medirse en respuesta a la alteración del andamio poroso, incluidas las adiciones de factores de crecimiento y la composición superficial/masiva del gel. Estos cambios pueden requerir la adaptación de estos ensayos para obtener resultados apreciables. Los parámetros que requieren una mayor optimización incluyen la densidad de siembra de celdas, la duración del experimento y/o la canalización de análisis. IMARIS es una poderosa herramienta de análisis de imágenes que se utiliza para el análisis de migración celular y tiene capacidades más allá de lo que se describe aquí, que incluyen la clasificación de todos los objetos dentro de una 'Superficie' seleccionada en conjuntos basados en varias propiedades, como el área de superficie, el volumen, la intensidad y la distancia de otras superficies creadas. Hay muchos recursos en línea para determinar otros métodos de análisis.
Los dos métodos descritos aquí no solo abordan el estado inicial de la introducción del tejido a un material granular de una manera fisiológica, sino también la respuesta celular posterior cuando está completamente incrustado dentro del material. Al igual que con todos los ensayos de migración, las células presentes son capaces de proliferar en paralelo al movimiento, sin embargo, el diseño de los ensayos descritos no interrumpe la proliferación y, por lo tanto, garantiza que no haya un impacto excesivo en el análisis. Ambos métodos son compatibles con la tinción de punto final, además de la obtención de imágenes longitudinales, que utiliza la fijación de PFA para detectar métricas como el citoesqueleto, la deposición de colágeno, la proliferación, etc. El uso de los métodos descritos avanza hacia una representación espacio-temporal más precisa de la migración celular 3D que utiliza la infiltración celular como parámetro medible en contraste con los métodos anteriores 1,6,14,15,25,26,27.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
La financiación de este trabajo fue parcialmente financiada a través del Premio al Proyecto a Corto Plazo de Alta Prioridad de los Institutos Nacionales de Salud de EE. UU. (1R56DK126020-01) y una donación filantrópica de Kurtin Trust. J.T. fue financiado por la Beca de Investigación de Posgrado de la Fundación Nacional de Ciencias. Esquemas de figuras creados con BioRender.com.
Materiales
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 647 Phalloidin | ThermoFisher | A22287 | |
| Bovine Serum Albumin | VWR International | 332 | |
| CellTracker Green CMFDS Dye, 1 mg | ThermoFisher | C2925 | 20 x 50 ug units, 492/517 nm |
| Centrifuge | ThermoFisher | 75016085 | ST Plus Series |
| Clear 96 well plate | MilliPore Sigma | CLS3997-50EA | |
| Dimethyl Sulfoxide | Fisher Scientific | MT-25950CQC | 250 mL |
| Fibroblast Basal Medium | ATCC | PCS-201-030 | 480 mL, phenol-red-free |
| Fibroblast Growth Kit - Low Serum | ATCC | PCS-201-041 | 7.5 mM L-glut,5 ng/mL rh FGF basic, 5 ug/mL rh Insulin, 1 ug/mL Hydrocortisone, 50 ug/mL Ascorbic acid, 2% FBS |
| FIJI (ImageJ) | NIH | Public access download | |
| Human Dermal Fibroblasts | ATCC | PCS-201-012 | Adult human dermal fibroblasts |
| ImageXpress Micro Confocal | Molecular Devices | Spinning Disc confocal microscope with 4x, 10x magnifications | |
| IMARIS | Oxford Instruments | 3/4D Imaged Visualizaiton and Analysis Software, Proprietary | |
| Incubator | ThermoFisher | Finnpipette F2 Variable volume Pipettes | HeraCell Vios 160i CO2 Incubator, 165L |
| M-20 Microplate Swinging Bucket Rotor | ThermoFisher | 75003624 | |
| Methylcellulose | Fisher Scientific | 9004-67-5 | Lab grade, powder form |
| Microcentrifuge tube | Fisherbrand | 05-408-129 | 1.5 mL microcentrifuge tubes |
| Paraformaldehyde (4%) | Alfa Aesar | AAJ19943K2 | For fixing |
| Petri dish | Corning | 08-757-100A | Bacteriological Petri Dishes with Lid 35 x 10 mm |
| Pipettes | ThermoFisher | 4642080 | Finnpipette F2 Variable volume Pipettes |
| Sterile PBS | Gibco | 10010-023 | |
| Triton-X | Fisher Scientific | 327371000 |
Referencias
- Jerka, D., et al. Unraveling endothelial cell migration: Insights into fundamental forces, inflammation, biomaterial applications, and tissue regeneration strategies. ACS Appl Bio Mater. 7 (4), 2054-2069 (2024).
- Edmondson, R., Broglie, J. J., Adcock, A. F., Yang, L. Three-dimensional cell culture systems and their applications in drug discovery and cell-based biosensors. Assay Drug Dev Technol. 12 (4), 207-218 (2014).
- Yamada, K. M., Sixt, M. Mechanisms of 3D cell migration. Nat Rev Mol Cell Biol. 20 (12), 738-752 (2019).
- Grinnell, F. Fibroblast biology in three-dimensional collagen matrices. Trends Cell Biol. 13 (5), 264-269 (2003).
- Kloxin, A. M., Kloxin, C. J., Bowman, C. N., Anseth, K. S. Mechanical properties of cellularly responsive hydrogels and their experimental determination. Adv Mater. 22 (31), 3484-3494 (2010).
- Solbu, A. A., et al. Assessing cell migration in hydrogels: An overview of relevant materials and methods. Mater Today Bio. 18, 100537 (2023).
- Madl, C. M., Katz, L. M., Heilshorn, S. C. Tuning bulk hydrogel degradation by simultaneous control of proteolytic cleavage kinetics and hydrogel network architecture. ACS Macro Lett. 7 (11), 1302-1307 (2018).
- Daly, A. C., Riley, L., Segura, T., Burdick, J. A. Hydrogel microparticles for biomedical applications. Nat Rev Mater. 5 (1), 20-43 (2020).
- Qazi, T. H., Burdick, J. A. Granular hydrogels for endogenous tissue repair. Biomater Biosyst. 1, 100008 (2021).
- Tanner, G. I., Schiltz, L., Narra, N., Figueiredo, M. L., Qazi, T. H. Granular hydrogels improve myogenic invasion and repair after volumetric muscle loss. Adv Healthc Mater. 25, e2303576 (2024).
- Sussman, E. M., Halpin, M. C., Muster, J., Moon, R. T., Ratner, B. D. Porous implants modulate healing and induce shifts in local macrophage polarization in the foreign body reaction. Ann Biomed Eng. 42 (7), 1508-1516 (2014).
- Roosa, C. A., et al. Conjugation of IL-33 to microporous annealed particle scaffolds enhances type 2-like immune responses in vitro and in vivo. Adv Healthc Mater. 13 (25), 2400249 (2024).
- Jaberi, A., et al. Engineering microgel packing to tailor the physical and biological properties of gelatin methacryloyl granular hydrogel scaffolds. Adv Healthc Mater. 13 (25), 2402489 (2024).
- Nandi, S., Brown, A. C. Characterizing cell migration within three-dimensional in vitro wound environments. J Vis Exp. 126, e56099 (2017).
- Qazi, T. H., et al. Anisotropic rod-shaped particles influence injectable granular hydrogel properties and cell invasion. Adv Mater. 34 (12), 2109194 (2022).
- Griffin, D. R., Weaver, W. M., Scumpia, P. O., Di Carlo, D., Segura, T. Accelerated wound healing by injectable microporous gel scaffolds assembled from annealed building blocks. Nat Mater. 14 (7), 737-744 (2015).
- Pruett, L. J., Jenkins, C. H., Singh, N. S., Catallo, K. J., Griffin, D. R. Heparin microislands in microporous annealed particle scaffolds for accelerated diabetic wound healing. Adv Funct Mater. 31 (35), 2104337 (2021).
- Roosa, C., et al. Microfluidic synthesis of microgel building blocks for microporous annealed particle scaffold. J Vis Exp. (184), e64119 (2022).
- Pfaff, B. N., et al. Selective and improved photoannealing of microporous annealed particle (MAP) scaffolds. ACS Biomater Sci Eng. 7 (2), 422-427 (2021).
- Riley, L., Schirmer, L., Segura, T. Granular hydrogels: Emergent properties of jammed hydrogel microparticles and their applications in tissue repair and regeneration. Curr Opin Biotechnol. 60, 1-8 (2019).
- Jo, C. H., Roh, Y. H., Kim, J. E., Shin, S., Yoon, K. S. Optimizing platelet-rich plasma gel formation by varying time and gravitational forces during centrifugation. J Oral Implantol. 39 (5), 525-532 (2013).
- Mironov, V., et al. Fabrication of tubular tissue constructs by centrifugal casting of cells suspended in an in situ. crosslinkable hyaluronan-gelatin hydrogel. Biomaterials. 26 (36), 7628-7635 (2005).
- Vu, L. T., Jain, G., Veres, B. D., Rajagopalan, P. Cell migration on planar and three-dimensional matrices: A hydrogel-based perspective. Tissue Eng Part B Rev. 21 (1), 67-74 (2015).
- Cialdai, F., Risaliti, C., Monici, M. Role of fibroblasts in wound healing and tissue remodeling on Earth and in space. Front Bioeng Biotechnol. 10, 958381 (2022).
- Qazi, T. H., Muir, V. G., Burdick, J. A. Methods to characterize granular hydrogel rheological properties, porosity, and cell invasion. ACS Biomater Sci Eng. 8 (4), 1427-1442 (2022).
- Shaik, R., et al. Cardiac matrix-derived granular hydrogel enhances cell function in 3D culture. ACS Appl Mater Interfaces. 16 (43), 58346-58356 (2024).
- Puiggalí-Jou, A., Asadikorayem, M., Maniura-Weber, K., Zenobi-Wong, M. Growth factor-loaded sulfated microislands in granular hydrogels promote hMSCs migration and chondrogenic differentiation. Acta Biomater. 166, 69-84 (2023).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados