É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Estudo da Checkpoint Dano ao DNA usando
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Xenopus egg extracto é um sistema modelo útil para investigar o checkpoint danos no DNA. Este protocolo é para a preparação de extractos de ovos de Xenopus e os danos do ADN reagentes indutores de ponto de verificação. Estas técnicas são adaptáveis a uma variedade de abordagens que danificam o ADN no estudo da sinalização checkpoint danificam o DNA.
Resumo
Numa base diária, as células são sujeitas a uma variedade de insultos endógenos e ambientais. Para combater esses insultos, as células desenvolveram posto de controle de danos ao DNA sinalização como um mecanismo de vigilância para detectar danos no DNA e diretos respostas celulares a danos no DNA. Há diversos grupos de proteínas chamados sensores, transdutores e efetores envolvidos na sinalização de DNA checkpoint de dano (Figura 1). Neste caminho complexo de sinalização, ATR (ATM e Rad3-relacionados) é uma das quinases importantes que podem responder a danos no ADN e stress replicação. Activated ATR pode fosforilar os seus substratos a jusante, tais como o Chk1 (Ponto de verificação quinase 1). Consequentemente, fosforiladas e activadas Chk1 conduz a muitos efeitos a jusante do ponto de verificação, incluindo danos no DNA paragem do ciclo celular, a activação da transcrição, reparação de danos do ADN, e a apoptose ou senescência (Figura 1). Quando o DNA é danificado, não para ativar o DNA resultados checkpoint danos em unrepdanos ao ar e, posteriormente, instabilidade genômica. O estudo dos danos de ADN do ponto de verificação será elucidar como as células a manter a integridade do genoma e proporcionar um melhor entendimento de como as doenças humanas, tais como o cancro, se desenvolver.
Extratos de ovos de Xenopus laevis estão surgindo como um poderoso modelo de sistema livre de células extrato no DNA pesquisa checkpoint danos. De baixa velocidade de extracto (LSE) foi inicialmente descrita por grupo Masui 1. A adição de cromatina espermática demembranated para os resultados na formação de núcleos de LSE, onde o DNA é replicado em uma forma semiconservativa uma vez por ciclo celular.
A via de sinalização ATR/Chk1-mediated checkpoint é acionado por dano de DNA ou estresse replicação 2. Dois métodos são usados atualmente para induzir o posto danos ao DNA: abordagens de DNA e DNA danos prejudiciais imitando-estruturas 3. Dano do ADN pode ser induzida por irradiação com ultravioleta (UV), γ-irradiação, metil metanesulfonate (MMS), mitomicina C (MMC), 4-nitroquinolina-1-óxido de (4-NQO), ou afidicolina 3, 4. O MMS é um agente alquilante que inibe a replicação do DNA e ativa o ponto de verificação danos ATR/Chk1-mediated DNA 4-7. Irradiação UV também provoca a ATR/Chk1-dependent DNA 8 checkpoint danos. A estrutura de ADN que imita dano-AT70 é um complexo de recozido de dois oligonucleótidos de poli-(dA) 70 e poli-(dT) 70. O sistema AT70 foi desenvolvido no laboratório Bill Dunphy e é amplamente utilizada para induzir a sinalização de checkpoint ATR/Chk1 9-12.
Aqui, nós descrevemos protocolos (1) para preparar extractos isentos de células de ovos (LSE), (2) para o tratamento de Xenopus com cromatina de esperma dois ADN diferentes abordagens danificar (MMS e UV), (3) para preparar o DNA de danos estrutura imita AT70, e (4) para accionar o ponto de verificação ATR/Chk1-mediated danos no DNA LSE com cromatina espermática danificado ou uma estrutura de ADN que imita danos.
Protocolo
1. Preparação LSE
- Rãs do sexo feminino (Xenopus laevis) são injetadas duas vezes para coleta de ovos. A primeira injecção (ferragem) é de 100 U PMSG (gonadotrofina de égua prenhe) por sapo. Rãs deve ser preparada pelo menos dois dias antes de ovo indução assentamento e rãs condicionadas são utilizáveis por até duas semanas. Para rãs primos, injetar por via subcutânea PMSG nos sacos linfáticos dorsal usando uma seringa de 3 ml e 27 agulha G.
- Para induzir a postura de ovos, injetar 500 U hCG (gonadotrofina coriônica humana) por sapo preparado por via subcutânea nos sacos linfáticos dorsal usando uma agulha de 27 G. Incubar rãs injetados em baldes separados contendo 2 litros de solução modificado 1x Marc Ringer (MMR). Permitir rãs 14-20 horas para colocar ovos antes da coleta.
- Retire as rãs dos baldes e despeje a solução MMR até cerca de 100 ml está à esquerda. Obter ovos dos baldes, transferindo os ovos para uma proveta de 250 ml.
- Ovos Dejelly por adição de 100 ml de 2% de cisteína (ajustarpH a 7,8 com KOH 10 M). Agite suavemente os ovos com uma pipeta de vidro invertida Pasteur (0,7 cm de diâmetro), aproximadamente a cada 30 seg. Decantar e substituir com cisteína fresco duas vezes durante a incubação. O processo está completo dejellying em cerca de 5-15 min quando os ovos formar uma camada mais condensada no fundo da proveta.
- Rejeitar a solução cisteína e lavar os ovos três vezes com MMR 0,25 x. Redemoinho os ovos na solução por uma pipeta de vidro. "Bad" ovos são determinadas por inspeção visual simples e são brancos "inchado" na aparência. Os "maus" ovos irá acumular-se no centro do copo, após agitação. Remover "maus" ovos por uma pipeta de Pasteur.
- Lavar os ovos com Lise Egg Buffer (ELB) três vezes. Remover quaisquer adicionais "maus" ovos por uma pipeta de Pasteur. Despeje os ovos em um tubo Falcon 14 ml.
- Gire o tubo de Falcon por 55 segundos a 188 xg (1.100 rpm) com o CL2 IEC centrífuga de mesa clínica com um rotor basculante para compactar os ovos. Remover o excesso de tampãotopo da camada de ovo. Adicione 0,5 ul da Aprotinina estoque Leupeptin / e 0,5 ul de estoque citocalasina B por ml de ovos compactadas.
- Centrifugar ovos em 16.500 xg (10.000 rpm) durante 15 min a 4 ° C utilizando o RC6 Sorvall mais centrifugadora superspeed com HB6 rotor basculante. Após centrifugação, os ovos são fraccionados em três camadas do tubo: extracto de lípidos, e / gema de pigmento a partir de cima para baixo, respectivamente. Perfurar a parede do tubo Falcon na porção inferior da camada de meio de extracto com uma agulha G 21. Remova cuidadosamente a agulha como a agulha de perfuração pode ser obstruída por plástico. Insira uma nova agulha 21 G ligada a uma seringa de 1 ml para o local de punção para recolher o extracto. Lentamente, aspirar o extracto para dentro da seringa, para evitar as bolhas de ar e contaminação com o lípido e / gema de pigmento camadas.
- Colocar-se os extractos num tubo refrigerado microcentrífuga de 1,5 ml. Por cada mililitro de extracto de ovo, adicionar as seguintes soluções de produtos químicos para o emconcentração final dicated (mostrada entre parêntesis): (1) 10 ul de cicloheximida (100 ug / ml), (2) 1 ul de aprotinina / leupeptina (10 | ig / ml de cada), (3) 1 ul de citocalasina B (5 ug / ml ), (4) 1 ul de ditiotreitol (1 mM), e (5) Nocodazole 0,33 ul (3 ug / ml). Inverter o ovo extrair pelo menos 10 vezes, com cuidado. Este é o LSE, a qual deve ser feita e utilizada dentro de 4 hr. A qualidade da LSE é comprometido após 4 h ou congelação-descongelação.
2. Tratamento da cromatina de espermatozóides com abordagens danificar o DNA
- Prepare cromatina espermática normal de acordo com o método descrito previamente 13.
- Prepare MMS-tratado cromatina espermática
- Ressuspender cromatina espermática normal em 0,5 ml de tampão de X.
- Adicionar ~ pi 5,5 de MMS (9,1 M em stock) a 500 ul de cromatina ressuspendidas a uma concentração final de 100 mM.
- Incubar cromatina espermática em tubo de microcentrífuga à temperatura ambiente durante 30 min, com rotação.
- Gire a mtubo icrocentrifuge a 686 xg (2100 rpm) durante 10 min à temperatura ambiente usando a centrífuga IEC CL2 clínico com rotor basculante e adaptadores de tubos.
- Descartar o sobrenadante e ressuspender o sedimento em 0,5 ml de tampão X mais BSA (3%), DTT (0,1 mM) e aprotinina / leupeptina (10 | ig / ml de cada).
- Repetir os passos (4) e (5) duas vezes para lavar a cromatina de esperma.
- Determinar a concentração de cromatina espermática com um hemocitómetro e diluir a 100.000 esperma / ul. Guardar aliquotas de 5 uL a -80 ° C congelador para posterior utilização.
- Prepare UV-tratado cromatina espermática
- Adicionar a quantidade desejada (por exemplo, 10 ul) da cromatina espermática normal sobre a superfície de uma peça de Parafilm.
- Coloque o Parafilm num reticulador de UV. Ajuste o parâmetro de energia desejado, dependendo da sua experiência, e começar a danificar a cromatina de espermatozóides através da luz UV. Por exemplo, demora aproximadamente 21 segundos para atingir 1000 J / m 2.
- Depois de irradiação UVdiação, cromatina espermática UV-tratados devem ser adicionadas a extractos de ovos imediatamente.
3. Preparação de um DNA Damage Estrutura mimetizando-(AT70)
- Dissolver os dois oligonucleótidos sintéticos de poli-(dA) 70 (designada como A70) e poli-(dT) 70 (designado como T70) em água até uma concentração de 2 ug / uL, respectivamente.
- Adicionar 100 ul de A70 e 100 ul de T70 para um tubo de microcentrífuga de 1,5 ml. Ferver solução mista oligo sintético durante 5 min a 95 ° C em um heatblock.
- Leve o bloco de banho seco para fora (com o tubo contendo EM mistura no mesmo), e deixe esfriar até a temperatura ambiente em um banco de laboratório. Este ajustamento de temperatura leva cerca de 45-60 min.
- A mistura arrefecida é AT70, com uma concentração final de 2 ug / uL. Ul da loja 10 da AT70 alíquotas em congelador -20 ° C para posterior utilização.
4. Provocando o Checkpoint danos no DNA em LSE com cromatina esperma danificado ou um dano de DNAImitando-Estrutura
- Induzir checkpoint danos no DNA em LSE com cromatina de esperma danificado
- Adicionar 50 ul de LSE para um tubo de microcentrífuga de 1,5 ml e complementar com 1 ul de mistura de energia e 2 ul de cromatina espermática danificado (concentração final ~ 4000 esperma / ul de reacção).
- Incubar o tubo de reacção à temperatura ambiente durante 90 min e apertar o tubo a cada 10 min.
- Pipetar 1 ul de mistura de reacção sobre uma lâmina de microscópio e suplementado com 1 ul de solução de corante nuclear após 30 min de incubação. Coloque uma lamela sobre a mistura de reacção e verificar se a formação de núcleos de via microscópio de fluorescência. Tipicamente, os núcleos redondos irá formar após 30 min de incubação, indicando que a replicação do ADN tenha iniciado.
- Tomar 10 ul da mistura de reacção em 90 ul de tampão de amostra. As amostras são analisadas através de imunotransf erência utilizando anticorpos anti-P-S344 Chk1 ou anti-anticorpos de Chk1.
- Checkpoint induzir danos no DNA em LSE com aAT70
- Adicionar 50 ul de LSE para um tubo de microcentrífuga de 1,5 ml suplementado com 1 ul de mistura de energia e de 1,6 ul de estoque Tautomycin.
- Adicionar 1,25 ul de pré-fabricados AT70 (2 ug / uL) e água (como controlo negativo) a cada reacção. Incubar as reacções à temperatura ambiente durante 90 min. Flick os tubos de reacção a cada 10 minutos.
- Adicionam-se 10 ul da mistura de reacção em 90 ul de tampão de amostra. As amostras são analisadas por meio de imunoblot utilizando anti-P-S344 Chk1 ou anti-anticorpos de Chk1.
5. Resultados representativos
A cromatina espermática danificadas ou estrutura danificam o DNA imita pode desencadear o checkpoint danos ATR/Chk1-mediated ADN no sistema de extracto de ovo de Xenopus. Figura 2a mostra que o MMS induz fosforilação Chk1 a Ser344 (Chk1 P-S344), o qual é um indicador da ATR activação da cinase. Figura 2B mostra que AT70, como um dano do ADN que imita-structure, também aciona Chk1 fosforilação. Amostras totais Chk1 são usados como controlos de carga em ambos os exemplos.
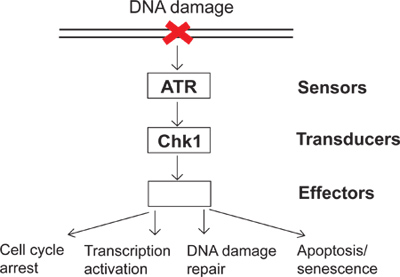
Figura 1. Um diagrama de ponto de verificação da sinalização de danos ao DNA.
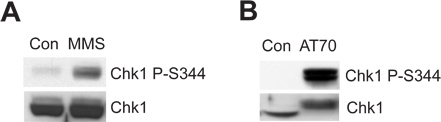
Figura 2. Chk1 fosforilação é induzida por uma ou MMS AT70 tratamentos em extractos de Xenopus ovo. (A) MMS-danificada cromatina espermática (MMS) ou cromatina espermática normal (Con) são incubados em extractos de ovos durante 90 min. Chk1 fosforilação em Ser344 (Chk1 P-S344) e Chk1 total em extractos de ovos são examinadas através de imunotransf erência. (B) ou de água AT70 (Con) são adicionados em extractos de ovos, respectivamente. As amostras são também analisados através de imunotransf erência como em (A).
Access restricted. Please log in or start a trial to view this content.
Discussão
Existem várias vantagens em estudar o posto danos no DNA utilizando extratos de Xenopus ovo. A utilização de extractos de ovo fornece uma grande quantidade de extractos livres de células sincronizadas em interfase do ciclo celular. Os extractos de ovos pode ser feita fácil e barata. É relativamente fácil para danificar o DNA ou cromatina e revelar um defeito no ponto de verificação, após danos no ADN immunodepleting uma proteína alvo a partir de extracto de ovo. Subsequentemente, um defeito da funç?...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Este trabalho é apoiado em parte por fundos fornecidos pela Universidade de Carolina do Norte em Charlotte, fundo Wachovia base para a excelência do corpo docente, e uma bolsa de NIGMS (R15GM101571).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Reagentes | |||
| Anticorpo anti-P-Chk1 S344 | Sinalização celular | 2348L | |
| Anticorpo anti-Chk1 | Santa Cruz | SC7898 | |
| Aprotinina | Biomedicals MP | 0219115880 | |
| Cicloheximida | Sigma | C7698-5G | |
| Citocalasina B | EMD | 250233 | |
| Ditiotreitol (DTT) | VWR | JTF780-2 | |
| hCG | Sigma | CG10-10VL | |
| L-Cisteína | Sigma | C7352-1KG | |
| Leupeptina | VWR | 97063-922 | |
| Metanossulfonato de metilo (MMS) | Sigma | 129.925-5G | |
| Nocodazol | Sigma | M1404-2mg | |
| PMSG | Calbiochem | 367222 | |
| Tampão de amostra | Sigma | S3401 | |
| Tautomycin | Wako Chemicals EUA | 209-12041 | |
| Equipamento | |||
| Balde para a postura de ovos | Produtos Rubbermaid comerciais | 6308 | |
| CL2 centrífuga IEC com rotor basculante | Thermo Scientific | 004260F | |
| HB6 rotor de caçamba móvel | Thermo Scientific | 11860 | |
| Sorvall RC6 mais centrífuga superspeed | Thermo Scientific | 46910 | |
| Reticulador UV | UVP | 95-0174-01 | |
| Soluções | |||
| 1x MMR | 100 mM de NaCl, 2 mM de KCl, 0,5 mM de MgSO4, 2,5 mM de CaCl 2, 5 mM de HEPES, pH ajustado a 7,8 com NaOH 10 M | ||
| Aprotinina / estoque Leupeptin | 10 mg / ml de cada uma em água. Armazenar 20 ul de aliquotas a -80 ° C. | ||
| Tampão X | 0,2 M de sacarose, 80 mM de KCl, 15 mM NaCl, 5 mM de MgCl2, 1 mM de EDTA, 10 mM de HEPES, pH ajustado a 7,5 por HCl | ||
| Estoque cicloheximida | 10 mg / ml em água. Loja de aliquotas de 1 ml a -20 ° C. | ||
| Citocalasina B estoque | 5 mg / ml em DMSO. Armazenar 20 ul de aliquotas a -20 ° C. | ||
| Ditiotreitol estoque (TDT) | 1 M em água. Loja de aliquotas de 1 ml a -20 ° C. | ||
| ELB | Sacarose 0,25 M, 1 mM de DTT, 50 ug / ml de cicloheximida, 2,5 mM MgCl2, 50 mM de KCl, 10 mM de HEPES, pH7.7 | ||
| Nocodazol estoque | 10 mg / ml em DMSO. Armazenar aliquotas de 5 uL a -80 ° C. | ||
| Mistura de energia | 375 mM de fosfato de creatina, ATP 50 mM e 25 mM de MgCl 2. Alíquotas são guardadas a -80 ° C. | ||
| Solução de corante nuclear | 0,4 ug / ml de Hoechst 33258 de glicerol, 25%(V / v), em PBS 1x | ||
| Estoque Tautomycin | 100 uM em DMSO. Armazenar aliquotas de 10 uL a -80 ° C. |
Referências
- Lohka, M. J., Masui, Y. Formation in vitro of sperm pronuclei and mitotic chromosomes induced by amphibian ooplasmic components. Science. 220 (4598), 719-721 (1983).
- Cimprich, K. A., Cortez, D. ATR: an essential regulator of genome integrity. Nat. Rev. Mol. Cell Biol. 9 (8), 616-627 (2008).
- Lupardus, P. J., Van, C., Cimprich, K. A. Analyzing the ATR-mediated checkpoint using Xenopus egg extracts. Methods. 41 (2), 222-231 (2007).
- Lupardus, P. J., Byun, T., Yee, M. C., Hekmat-Nejad, M., Cimprich, K. A. A requirement for replication in activation of the ATR-dependent DNA damage checkpoint. Genes Dev. 16 (18), 2327-2332 (2002).
- Stokes, M. P., Van Hatten, R., Lindsay, H. D., Michael, W. M. DNA replication is required for the checkpoint response to damaged DNA in Xenopus egg extracts. J. Cell Biol. 158 (5), 863-872 (2002).
- Kato, K., Strauss, B. Accumulation of an intermediate in DNA synthesis by HEp.2 cells treated with methyl methanesulfonate. Proc. Natl. Acad. Sci. U.S.A. 71 (5), 1969-1973 (1974).
- Paulovich, A. G., Hartwell, L. H. A checkpoint regulates the rate of progression through S phase in S. cerevisiae in response to DNA damage. Cell. 82 (5), 841-847 (1995).
- Guo, Z., Kumagai, A., Wang, S. X., Dunphy, W. G. Requirement for Atr in phosphorylation of Chk1 and cell cycle regulation in response to DNA replication blocks and UV-damaged DNA in Xenopus egg extracts. Genes Dev. 14 (21), 2745-2756 (2000).
- Jazayeri, A., Balestrini, A., Garner, E., Haber, J. E., Costanzo, V. Mre11-Rad50-Nbs1-dependent processing of DNA breaks generates oligonucleotides that stimulate ATM activity. EMBO. J. 27 (14), 1953-1962 (2008).
- Kumagai, A., Dunphy, W. G. Claspin, a novel protein required for the activation of Chk1 during a DNA replication checkpoint response in Xenopus egg extracts. Mol. Cell. 6 (4), 839-849 (2000).
- Yan, S., Lindsay, H. D., Michael, W. M. Direct requirement for Xmus101 in ATR-mediated phosphorylation of Claspin bound Chk1 during checkpoint signaling. J. Cell Biol. 173 (2), 181-186 (2006).
- Shiotani, B., Zou, L. Single-stranded DNA orchestrates an ATM-to-ATR switch at DNA breaks. Mol. Cell. 33 (5), 547-558 (2009).
- Tutter, A. V., Walter, J. C. Chromosomal DNA replication in a soluble cell-free system derived from Xenopus eggs. Methods Mol. Biol. 322, 121-137 (2006).
- Cross, M. K., Powers, M. Preparation and Fractionation of Xenopus laevis Egg Extracts. J. Vis. Exp. (18), e891(2008).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados