Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
ДНК-привязанная РНК-полимераза для программируемой транскрипции in vitro и молекулярных вычислений
В этой статье
Резюме
Мы описываем разработку новой ДНК-привязанной РНК-полимеразы Т7 для регулирования реакций транскрипции in vitro. Мы обсуждаем этапы синтеза и характеристик белка, проверяем доказательство концепции транскрипционной регуляции и обсуждаем ее приложения в молекулярных вычислениях, диагностике и обработке молекулярной информации.
Аннотация
Нанотехнология ДНК позволяет программируемую самосборку нуклеиновых кислот в предписанные пользователем формы и динамику для различных применений. Эта работа демонстрирует, что концепции из нанотехнологий ДНК могут быть использованы для программирования ферментативной активности фаговой РНК-полимеразы T7 (RNAP) и построения масштабируемых синтетических регуляторных сетей генов. Во-первых, Олигонуклеотид-привязанный T7 RNAP спроектирован путем экспрессии N-терминально помеченного SNAP RNAP и последующей химической связи SNAP-метки с бензилгуанином (BG)-модифицированным олигонуклеотидом. Затем смещение нитей нуклеиновых кислот используется для программирования транскрипции полимеразы по требованию. Кроме того, вспомогательные сборки нуклеиновых кислот могут использоваться в качестве «искусственных факторов транскрипции» для регулирования взаимодействия между запрограммированным ДНК РНКП Т7 с его шаблонами ДНК. Этот регуляторный механизм транскрипции in vitro может реализовывать различные варианты поведения схем, такие как цифровая логика, обратная связь, каскадирование и мультиплексирование. Компонуемость этой генной регуляторной архитектуры облегчает абстракцию, стандартизацию и масштабирование дизайна. Эти функции позволят быстро создавать прототипы генетических устройств in vitro для таких применений, как биозондирование, обнаружение заболеваний и хранение данных.
Введение
ДНК-вычисления используют набор разработанных олигонуклеотидов в качестве среды для вычислений. Эти олигонуклеотиды запрограммированы последовательностями для динамической сборки в соответствии с заданной пользователем логикой и реагируют на конкретные входы нуклеиновых кислот. В экспериментальных исследованиях результат вычислений обычно состоит из набора флуоресцентно меченых олигонуклеотидов, которые могут быть обнаружены с помощью гелевого электрофореза или флуоресцентных пластинчатых считывателей. За последние 30 лет были продемонстрированы все более сложные вычислительные схемы ДНК, такие как различные цифровые логические каскады, сети химических реакций и нейронные сети1,2,3. Чтобы помочь в подготовке этих схем ДНК, математические модели были использованы для прогнозирования функциональности синтетических генных схем4,5,а вычислительные инструменты были разработаны для проектирования ортогональных последовательностей ДНК6,7,8,9,10 . По сравнению с компьютерами на основе кремния, к преимуществам ДНК-компьютеров можно отнести их способность напрямую взаимодействовать с биомолекулами, работать в растворе при отсутствии источника питания, а также их общую компактность и стабильность. С появлением секвенирования следующего поколения стоимость синтеза ДНК-компьютеров снижалась в течение последних двух десятилетий быстрее, чем закон Мура11. В настоящее время начинают появляться приложения таких компьютеров на основе ДНК, такие как для диагностики заболеваний12,13,для питания молекулярной биофизики14и в качестве платформ хранения данных15.
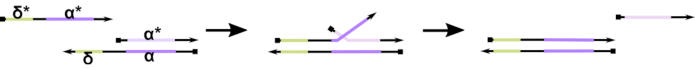
Рисунок 1:Механизм смещения нитей ДНК, опосредованных носком. Toehold, δ, представляет собой свободную, несвязанную последовательность на частичном дуплексе. Когда комплементарный домен (δ*) вводится на второй нити, свободный δ домен служит опорой для гибридизации, позволяя остальной части нити (ɑ*) медленно вытеснять своего конкурента через молниеносную / расстегивающую обратимую реакцию, известную как миграция нитей. По мере увеличения длины δ ΔG для прямой реакции уменьшается, и смещение происходит более легко. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
На сегодняшний день большинство ДНК-компьютеров используют хорошо зарекомендовавший себя мотив в области динамической нанотехнологии ДНК, известный как смещение цепей ДНК(TMDSD, рисунок 1)16. Этот мотив состоит из частично двухцепочечного дуплекса ДНК (dsDNA), отображающего короткие свесы «toehold» (т.е. от 7 до 10 нуклеотидов (nt)). «Входные» нити нуклеиновых кислот могут взаимодействовать с частичными дуплексами через носок. Это приводит к смещению одной из нитей из частичного дуплекса, и эта освобожденная нить может затем служить входом для нисходящих частичных дуплексов. Таким образом, TMDSD обеспечивает каскадирование сигналов и обработку информации. В принципе, ортогональные мотивы TMDSD могут работать независимо в решении, обеспечивая параллельную обработку информации. Существует ряд вариаций реакции TMDSD, таких как опосредованный носком ниточный обмен (TMDSE)17,«непротекающие» пальцы ног с двойными длинными доменами18,несоответствующие последовательности носок19и «ручное» опосредованное смещение нитей20. Эти инновационные принципы проектирования позволяют более точно настроить энергетику и динамику TMDSD для повышения производительности днк-вычислений.
Синтетические генные схемы, такие как транскрипционные генные цепи, также способны вычислять21,22,23. Эти схемы регулируются факторами транскрипции белка, которые активируют или подавляют транскрипцию гена путем связывания со специфическими регуляторными элементами ДНК. По сравнению со схемами на основе ДНК, транскрипционные схемы имеют несколько преимуществ. Во-первых, ферментативная транскрипция имеет гораздо более высокую скорость оборота, чем существующие каталитические схемы ДНК, тем самым генерируя больше копий выходного сигнала на одну копию входа и обеспечивая более эффективные средства усиления сигнала. Кроме того, транскрипционные схемы могут производить различные функциональные молекулы, такие как аптамеры или матричная РНК (мРНК), кодирующая терапевтические белки, в качестве вычислительных выходов, которые могут быть использованы для различных применений. Однако основным ограничением современных транскрипционных схем является отсутствие у них масштабируемости. Это связано с тем, что существует очень ограниченный набор ортогональных факторов транскрипции на основе белка, и разработка de novo новых факторов транскрипции белка остается технически сложной и трудоемкой.
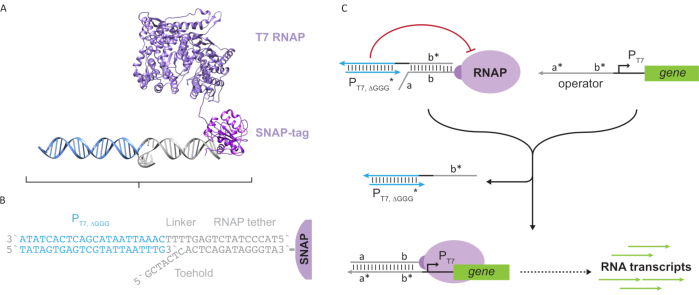
Рисунок 2:Абстракция и механизм полимеразного комплекса «трос» и «клетка». ( А иВ ) Олигонуклеотидный трос ферментативно мечуется на полимеразу Т7 посредством реакции SNAP-метки. Клетка, состоящая из «искусственного» промотора Т7 с навесом троса-комплемента, позволяет ему гибридизоваться с тросом и блокировать транскрипционную активность. (C) Когда оператор (a*b*) присутствует, он связывается с пальцем ноги на олигонуклеотидном тросе(ab)и смещает область b* клетки, позволяя происходить транскрипции. Эта цифра была изменена с Чжоу и Ши27. Сокращения: RNAP = РНК-полимераза. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
В этой статье представлен новый строительный блок для молекулярных вычислений, который сочетает в себе функциональные возможности транскрипционных схем с масштабируемостью схем на основе ДНК. Этот строительный блок представляет собой Ковалентный Т7-РНК, присоединенный одноцепочечным тросом ДНК(рисунок 2А). Чтобы синтезировать этот ПРИВЯЗАННЫЙ к ДНК T7 RNAP, полимеразу сплавляли с N-концевой SNAP-меткой24 и рекомбинантно экспрессировали в кишечной палочке. Затем SNAP-метку реагировали с олигонуклеотидом, функционализированным с субстратом BG. Олигонуклеотидный трос позволяет позиционировать молекулярные гости в непосредственной близости от полимеразы посредством гибридизации ДНК. Одним из таких гостей был конкурентный блокатор транскрипции, называемый «клеткой», который состоит из «искусственного» дуплекса ДНК промотора T7 без гена вниз по течению(рисунок 2B). При связывании с RNAP через его олигонуклеотидный трос клетка останавливает активность полимеразы, превосходя другие шаблоны ДНК для связывания RNAP, приводя RNAP в состояние «OFF»(рисунок 2C).
Чтобы активировать полимеразу до состояния «ON», были разработаны шаблоны ДНК Т7 с одноцепочечными «операторными» доменами выше промотора Т7 гена. Операторный домен (т.е. домен a*b* Рисунок 2C)может быть спроектирован так, чтобы вытеснить клетку из РНКП через TMDSD и позиционировать РНКП проксимально к промотору Т7 гена, тем самым инициируя транскрипцию. Альтернативно, шаблоны ДНК также были разработаны, где операторная последовательность была комплементарна вспомогательным нитям нуклеиновых кислот, которые называются «факторами искусственной транскрипции» (т.е. нитями TFA и TFB на рисунке 3A). Когда обе нити будут введены в реакцию, они будут собираться на операторской площадке, создавая новый псевдосмежный домен a*b*. Затем этот домен может вытеснить клетку через TMDSD для инициирования транскрипции(рисунок 3B). Эти пряди могут поставляться либо экзогенно, либо производиться.
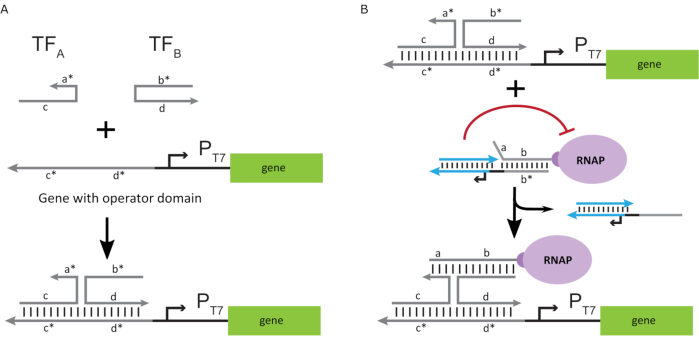
Рисунок 3:Селективное программирование активности полимеразы с помощью трехкомпонентного переключателя активатора. (A)Когда факторы транскрипции (TFA и TFB)присутствуют, они связываются с операторным доменом перед промотором, образуя псевдо-одноцепочечную последовательность(a*b*),способную вытеснять клетку через опосредованное смещение ДНК. (B) Этот домен a*b* может смещать клетку через TMDSD, чтобы инициировать транскрипцию. Эта цифра была изменена с Чжоу и Ши27. Сокращения: TF = транскрипционный фактор; RNAP = РНК-полимераза; TMDSD = смещение нитей ДНК, опосредованное носком. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Использование факторов транскрипции на основе нуклеиновых кислот для транскрипционной регуляции in vitro позволяет масштабируемую реализацию сложных поведений схем, таких как цифровая логика, обратная связь и каскадирование сигналов. Например, можно построить каскады логических ворот, спроектировав последовательности нуклеиновых кислот таким образом, чтобы транскрипты из гена вверх по течению активировали ген нисходящего потока. Одним из приложений, использующих каскадирование и мультиплексирование, которые стали возможными с помощью этой предлагаемой технологии, является разработка более сложных молекулярных вычислительных схем для портативной диагностики и обработки молекулярных данных. Кроме того, интеграция возможностей молекулярных вычислений и синтеза РНК de novo может обеспечить новые приложения. Например, молекулярная схема может быть спроектирована для обнаружения одной или комбинации определяемых пользователем РНК в качестве входных и выходных терапевтических РНК или мРНК, кодирующих функциональные пептиды или белки для медицинских применений в местах оказания медицинской помощи.
Access restricted. Please log in or start a trial to view this content.
протокол
1. Подготовка буфера
ПРИМЕЧАНИЕ: Очистка белка буферным препаратом может происходить в любой день; здесь это было сделано до начала экспериментов.
- Приготовьте буфер лизиса/равновесия, содержащий 50 мМ трис (гидроксиметил)аминометан (Tris), 300 мМ хлорида натрия (NaCl), 5% глицерина и 5 мМ β-меркаптоэтанола (BME), рН 8. Добавьте 1,5 мл 1M Tris, 1,8 мл 5M NaCl, 1,5 мл глицерина, 25,2 мл деионизированной воды (ddH2O) в 50 мл центрифужной трубки и добавьте 10,5 мкл 14,2 M BME непосредственно перед использованием.
ПРИМЕЧАНИЕ: Трис может вызвать острую токсичность; Следовательно, избегайте вдыхания его пыли и избегайте контакта с кожей и глазами. BME токсичен и должен использоваться только в вытяжном шкафу. Важно добавить BME последним, непосредственно перед повторной суспензией и лизисом клеток. Формула буфера лизиса приведена в таблице 1. - Подготовьте промывочный буфер (рН 8), содержащий 50 мМ Tris, 800 мМ NaCl, 5% глицерина, 5 мМ BME и 20 мМ имидазола. Добавьте 1,5 мл 1 M Tris, 4,8 мл 5 M NaCl, 1,5 мл глицерина и 22,2 мл ddH2O в 50 мл центрифужной трубки. Непосредственно перед применением добавляют 7 мкл 14,2 М БМЭ и 200 мкл 2 М имидазола к 20 мл вышеуказанного раствора.
ПРИМЕЧАНИЕ: Для предотвращения острой токсичности из-за имидазола используйте средства индивидуальной защиты. Важно добавлять BME и имидазол последними, непосредственно перед вымыванием белка из колонки. Формула промывочного буфера приведена в таблице 2. - Готовят буфер элюирования (pH8), содержащий 50 мМ Tris, 800 мМ NaCl, 5% глицерина, 5 мМ BME и 200 мМ имидазола. Добавьте 0,5 мл 1 M Tris, 1,6 мл 5 M NaCl, 0,5 мл глицерина и 6,4 мл ddH2O в 15 мл центрифужной трубки. Непосредственно перед применением добавляют 3,5 мкл 14,2 М БМЭ и 1 мл 2 М имидазола к 10 мл вышеуказанного раствора.
ПРИМЕЧАНИЕ: Важно добавить BME и имидазол в последнюю очередь, непосредственно перед элюированием белка из колонки. Формула буфера элюирования приведена в таблице 3. - Подготовьте 2-кратный буфер хранения (для смешивания 1:1 с глицерином), содержащий 100 мМ Tris, 200 мМ NaCl, 40 мМ BME и 2 мМ этилендиаминеттрауксусной кислоты (ЭДТА), 0,2% неионного поверхностно-активного вещества (см. Таблицу материалов). Подготовьте 50 мл буфера хранения, добавив 5 мл 1 M Tris, 2 мл 5 M NaCl, 42,56 мл ddH2O, 200 мкл 0,5 M EDTA, 100 мкл неионного поверхностно-активного вещества в 50 мл центрифужной трубки. Перемешайте до однородности раствора, процедите буфер хранения через шприцевой фильтр 0,2 мкм и добавьте 140,8 мкл BME к вышеуказанному раствору перед использованием.
ПРИМЕЧАНИЕ: Чтобы избежать острой токсичности из-за ЭДТА, избегайте вдыхания его пыли и избегайте контакта с кожей и глазами. Важно добавить BME последним и смешать весь буфер хранения 1:1 с глицерином, непосредственно перед хранением очищенного белка. Формула буфера хранилища приведена в таблице 4.
2. Ночной рост культуры: День 1
- Приготовьте 1000x канамицина, растворив 500 мг канамицина в 10 мл ddH2O.
ПРИМЕЧАНИЕ: Используйте средства индивидуальной защиты для предотвращения острой токсичности из-за канамицина. - Добавьте 20 мкл из 1000x канамицинового бульона в 20 мл лизогенного бульона. Используя стерильный наконечник пипетки, ткните преобразованный бульон глицерина BL21 E. coli, а затем привите культуру, введя наконечник в бульон из среды роста.
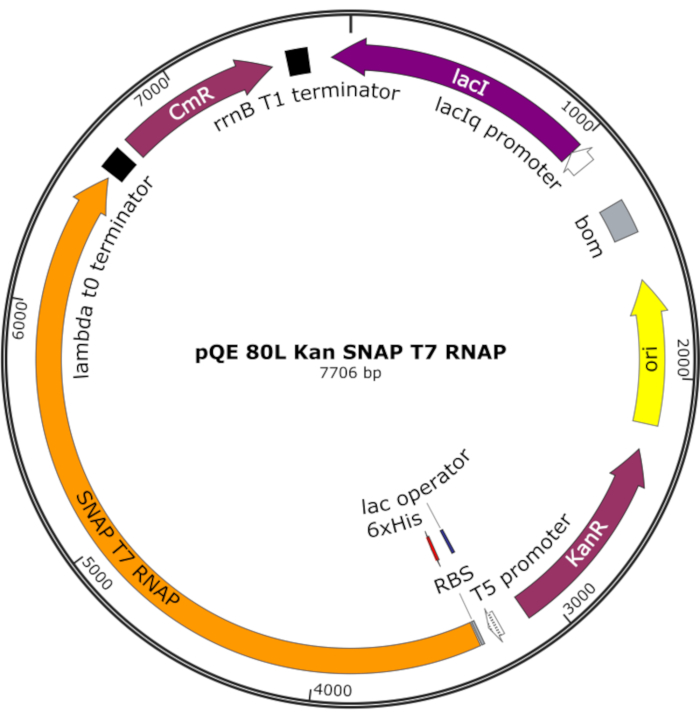
Рисунок 4:Плазмидная карта для SNAP T7 RNAP. Плазмида кодирует T7 RNAP, содержащий N-концевую гистидиновую метку (6x His) и домен SNAP-метки (SNAP T7 RNAP) под лак-репрессором (lacI) на позвоночнике pQE-80L. Другие особенности включают гены устойчивости к канамицину (KanR) и резистентности к хлорамфениколу (CmR). Аббревиатура: RNAP = РНК-полимераза. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
ПРИМЕЧАНИЕ: Плазмида кодирует T7 RNAP, содержащий N-концевую гистидиновую метку и домен SNAP-метки (SNAP T7 RNAP), а также ген устойчивости к канамицину под основой pQE-80L(Рисунок 4)25.
- Опять же, добавьте 20 мкл из 1000x канамицинового бульона в отдельную культуральную колбу, содержащую 20 мл лизогенного бульона, и инкубируйте его в качестве контроля.
- Инкубируют два образца (из стадий 2.2 и 2.3) в течение 12-18 ч при 37 °C, вращая при этом 10 × г.
3. Рост и индукция клеток: День 2
- Инокулируют 400 мл лизогенного бульона, содержащего 400 мкл канамицина, 4 мл ночной культуры роста со стадии 2.4. Инкубируют колбы для культивирования при 37 °C, вращая при этом при 10 × г.
- Как только культура достигнет оптической плотности (OD) при 600 нм ~ 0,5, извлеките 1 мл образца из колбы роста в качестве контроля. Храните контрольный образец при температуре 4 °C.
- Индуцировать клетки изопропиловым β-D-1-тиогалакопиранозидом (IPTG) путем добавления 40 мкл 1M IPTG на 100 мл культуры для достижения конечной концентрации 0,4 мМ IPTG. Инкубируют образец в течение 3 ч при 37 °C, вращая при 10 × г,а затем вращают индуцированную культуру при 8000 × г в течение 10 мин, чтобы гранулировать клетки. Удалите супернатант и храните гранулы при -20 °C до дальнейшего использования.
ПРИМЕЧАНИЕ: Чтобы избежать острой токсичности из-за IPTG, избегайте вдыхания его пыли и избегайте контакта с кожей и глазами. При необходимости можно приостановить эксперимент здесь и продолжить на следующий день.
4. Лизис клеток, очистка белка: День 3
- Повторно суспендируйте хранимую ячейку гранулы с 10 мл лизисного буфера на льду и осторожно закрутите, чтобы обеспечить повторное суспендирование всей гранулы. Затем пипетку 1 мл образца в десять пробирок по 1,5 мл, которые хранят на льду.
- Обрабатывайте ультразвуком каждый образец с амплитудой «1», импульсный в течение 2 с с рабочим циклом 50% в течение 30 с. До и после каждого образца очищайте наконечник ультразвуком 70% этанолом и ddH2O. Держите все образцы на льду во время и после обработки ультразвуком.
ПРИМЕЧАНИЕ: Держите 70% этанола вдали от тепла и открытого огня. - Уравновешивайте никелевую спиновую колонну очистки нитрилотриаценовой кислоты (Ni-NTA) до рабочей температуры 4 °C. Поместите/храните колонну при температуре 4 °C и держите на льду во время использования.
- Центрифугирование десяти образцов по 1 мл при 15 000 × г в течение 20 мин при 4 °C. Осторожно выбрасывайте супернатант, содержащий рекомбинантный RNAP, не нарушая гранулу. При необходимости используйте дополнительный буфер равновесия для регулировки общего объема до ≥ 6 мл.
- Осторожно снимите нижнюю вкладку из колонки спина Ni-NTA, чтобы обеспечить поток через столбец. Поместите колонну в центрифужную трубку и держите ее на льду.
ПРИМЕЧАНИЕ: Используйте центрифужную трубку объемом 50 мл со спиновыми колоннами Ni-NTA объемом 3 мл. - Центрифугируйте колонну при 700 × г и 4 °C в течение 2 мин, чтобы удалить буфер хранения. Уравновесьте столбец, добавив в него 6 мл буфера равновесия. Позвольте буферу полностью войти в слой смолы.
- Снимите буфер равновесия с колонны центрифугированием при 700 × г и 4 °C в течение 2 мин. Перед добавлением подготовленного экстракта клеток в столбец поместите нижнюю пробку на колонку, чтобы избежать потери продукта. Затем добавьте экстракт клеток в колонку и перемешайте на орбитальном шейкерном миксере в течение 30 мин при 4 °C.
- Снимите нижнюю пробку из колонны и поместите колонну в 50-литровую центрифужную трубку с маркировкой flow through. Центрифугируют колонну при 700 × г в течение 2 мин для сбора потока.
- Добавьте 6 мл промывочного буфера в колонку для промывки смолы. Центрифугируют колонну при 700 × г в течение 2 мин для сбора фракции в новую центрифужную трубку с маркировкой промывки 1. Повторите этот шаг еще два раза, в общей сложности 3 отдельные фракции, и соберите фракции в отдельные центрифужные трубки(промывка 2 и промывка 3).
- Добавьте 3 мл буфера элюирования, чтобы элюдировать помеченные His белки из смолы. Центрифугируют колонну при 700 × г в течение 2 мин для сбора фракции в новую центрифужную трубку с маркировкой элюат 1. Повторите этот шаг еще два раза, в общей сложности 3 отдельные фракции, и соберите фракции в отдельные центрифужные трубки(элюат 2 и элюат 3).
- Смешайте элюаты и выполните обессоливание для удаления солей из белкового раствора.
- Пипетка 15 мл 0,05 % мас./v полисорбата 20 над центробежным фильтрующим блоком 100 кДа. Центрифугу при 4000 × г в течение 40 мин и отбросьте сквозной.
- Используйте фильтр с покрытием для концентрирования элюатов 1, 2 и 3 (9 мл общего количества элюата белка + 6 мл буфера хранения) до ~1 500 мкл. Центрифугируйте фильтр при 3 220 × г в течение 20 мин и осторожно промывайте мембрану пипеткой, чтобы предотвратить осаждение.
- Разбавить образец до 15 мл с помощью буфера хранения. Выполните буферный обмен с помощью буфера хранилища 1:1 000, повторив шаг 4.11.2 еще два раза.
- Количественно оценить очищенный белок, измерив поглощение фракции при 280 нм. Очистите спектрофотометр с буфером хранения (2x буфер хранения при 4 °C). Аккуратно перемешайте образец комбинированных элуатов и измерьте его абсорбцию.
ПРИМЕЧАНИЕ: Выполните три отдельных показания при 1-кратном, 10-кратном и 50-кратном разведении образца белка для усреднения и количественной оценки белка. Разбавляйте образцы в буфере хранения. - Отрегулируйте образцы белка до 100 мкМ с помощью 2-кратного буфера хранения. Разбавьте скорректированный образец 1:1 по объему 100% глицерином. Хранить полученный белковый раствор при -80 °C.
5. Анализ белкового продукта на додецилсульфат-полиакриламидный гель электрофорез (SDS-PAGE): День 3
- Запустите гель SDS-PAGE для анализа белка. Смешайте 9 мкл образца с 3 мкл 4x красителя для загрузки белка додецилсульфата лития (LDS). Нагревайте образцы при 95 °C в течение 10 мин.
- Загрузите образцы на 4-12% гель Bis-Tris SDS-PAGE. Загрузите белковую лестницу в скважину 1, затем с образцами (слева направо): проточная, промывка 1, промывка 2, промывка 3, элюирование 1, элюирование 2, элюирование 3 и полное обессоленное элюирование.
ПРИМЕЧАНИЕ: Таблица 5 содержит таблицу загрузки образцов для геля SDS-PAGE. - Запустите загруженные образцы геля в буфере 2-(N-морфолино) этанесульфоновой кислоты (MES) в течение 35 мин при 200 В. Промойте гель в чистом лотке три раза в течение 10 мин каждый, используя 200 мл ddH2O,с мягким перемешиванием, чтобы удалить любой SDS из гелевой матрицы.
ПРИМЕЧАНИЕ: Носите средства индивидуальной защиты, чтобы избежать острой токсичности из-за MES. - Окрасьте гель 20 мл синего coomassie и высиживайте гель на ночь при комнатной температуре с мягким перемешиванием. Обезжиривание геля дважды в течение 1 ч каждый с 200 мл ddH2Oс мягким перемешиванием на орбитальном шейкере.
ПРИМЕЧАНИЕ: Мытье геля в течение более длительного периода времени или частая замена воды повысит чувствительность. Кроме того, помещение сложенной деликатной салфетки в контейнер для поглощения избытка красителя ускорит процесс окрашивания.
6. Функциональная верификация SNAP T7 RNAP через транскрипцию in vitro
ПРИМЕЧАНИЕ: Этот протокол использует шаблон ДНК, который кодирует флуоресцентный аптамер РНК брокколи и позволяет использовать флуоресценцию для мониторинга кинетики транскрипции на считывателе флуоресцентных пластин.
- Настройте три реакции транскрипции in vitro (IVT) для сравнения активности SNAP T7 RNAP с RNAP T7 дикого типа (WT) из коммерческого источника и буферного контроля. Отрегулируйте объем каждой реакции до 20 мкл.
- Готовят реакцию SNAP T7 RNAP IVT путем смешивания 2 мкл 10-кратного транскрипционного буфера, 0,4 мкл смеси рибонуклеозидтрифосфата (rNTP) 25 мМ, 5 мкл шаблона ДНК 500 нМ, 2 мкл 500 нМ SNAP T7 RNAP и 10,6 мкл ddH2O.
- Готовят реакцию WT RNAP IVT путем смешивания 2 мкл 10x транскрипционного буфера, 0,4 мкл смеси rNTP 25 мМ, 5 мкл шаблона ДНК 500 нМ, 2 мкл WT T7 RNAP и 10,6 мкл ddH2O.
- Приготовьте реакцию IVT только с буфером, смешивая 2 мкл 10-кратного транскрипционного буфера, 0,4 мкл смеси rNTP 25 мМ, 5 мкл шаблона ДНК 500 нМ и 12,6 мкл ddH2O.
ПРИМЕЧАНИЕ: Добавьте RNAP последним, сохраняя образцы на льду до его введения. Таблица 6, Таблица 7и Таблица 8 содержат формулы реакции ИВТ.
- Мониторинг кинетики транскрипции на считывателе флуоресцентной пластины в течение 2 ч с интервалом 2 мин при 37 °C с использованием длины волны возбуждения 470 нм и длины волны излучения 512 нм.
7. Получение BG-модифицированных олигонуклеотидов: День 1
- Растворяют олигонуклеотид с 3'-аминной модификацией в ddH2Oдо конечной концентрации 1 мМ. Пометьте этот S1.
- Смешайте 25 мкл 1 М бикарбоната натрия (NaHCO3),284 мкл 100% диметилсульфоксида (ДМСО), 125 мкл S1 (олигонуклеотидный материал) и 66 мкл 50 мМ эфира BG-N-гидроксисукцинимида (NHS) (BG-GLA-NHS), разбавленного ДМСО, отрегулируйте объем до 500 мкл и инкубируйте в течение ночи при комнатной температуре при 100 × г.
ПРИМЕЧАНИЕ: Держите DMSO вдали от тепла и пламени, так как это горючая жидкость. Таблица 9 содержит формулу реакции для конъюгации BG с олигонуклеотидом.
- Смешайте 25 мкл 1 М бикарбоната натрия (NaHCO3),284 мкл 100% диметилсульфоксида (ДМСО), 125 мкл S1 (олигонуклеотидный материал) и 66 мкл 50 мМ эфира BG-N-гидроксисукцинимида (NHS) (BG-GLA-NHS), разбавленного ДМСО, отрегулируйте объем до 500 мкл и инкубируйте в течение ночи при комнатной температуре при 100 × г.
8. Осаждение этанола/ацетона конъюгата BG-олигонуклеотидов: День 2
- Центрифуга - произведение стадии 7.1.1. при 13 000 × г в течение 5 мин. Осторожно перенесите супернатант в свежую трубку и выбросьте любой осажденный BG. Разделите реакцию на две равные аликвоты по 250 мкл, чтобы предотвратить переполнение, и выполните следующие шаги на обеих аликвотах.
- Добавьте 1/10-ю объема 3 М ацетата натрия (25 мкл), а затем 2,5-кратный объем в 100% этаноле (625 мкл). Инкубировать при -80 °C в течение 1 ч.
ПРИМЕЧАНИЕ: Используйте средства индивидуальной защиты при обращении как с ацетатом натрия (может вызвать раздражение глаз, кожи, пищеварительных и дыхательных путей), так и с этанолом (чрезвычайно легковоспламеняющимся, вызывает раздражение при контакте). При необходимости приостановите эксперимент здесь и продолжите на следующий день. - Поместите трубки в центрифугу и отметьте внешний край. Центрифугируйте пробирки по 17 000 × г в течение 30 мин при 4 °C.
ПРИМЕЧАНИЕ: Олигонуклеотидная гранула появится на отмеченном краю трубки. - Не нарушая гранулу, выбросьте супернатант. Долить 750 мкл охлажденного 70% этанола и отжимать при 17 000 × г в течение 10 мин при 4 °C.
- Не нарушая гранулу, выбросьте супернатант. Долить 750 мкл 100% ацетона и вращать при 17 000 × г в течение 10 мин при 4 °C.
ПРИМЕЧАНИЕ: Используйте средства индивидуальной защиты при обращении с ацетоном, так как он чрезвычайно легковоспламеняется и вызывает раздражение при контакте. - С открытой крышкой трубки высушите на воздухе в течение 5 минут, чтобы удалить избыток ацетона путем испарения. Повторно растворяют олигонуклеотид в 250 мкл 1x буфера Tris-EDTA (TE) с получением ~850 мкМ BG-олигонуклеотидного раствора.
- Повторите шаги с 8.2 по 8.6 и повторно растворите в 70 мкл 1x TE буфера. Пометьте этот S2.
9. Очистка BG-олигонуклеотидов с помощью гель-фильтрационной хроматографии
- Приостановите матрицу, энергично перевернув колонны несколько раз; снимите верхнюю крышку и защелкните нижнюю кончик колонки. Поместите колонну в центрифужную трубку объемом 1,5 мл и центрифугируйте трубку при 1000 × г в течение 1 мин при комнатной температуре. Выбросьте элюированный буфер и сборную трубку.
ПРИМЕЧАНИЕ: Важно предотвратить образование вакуума. Немедленно используйте подготовленные столбцы. - Поместите упакованные колонны в чистые центрифужные трубки объемом 1,5 мл. Добавьте 300 мкл 1x TE буфера в центр слоя колонны и центрифугу на 1000 × г в течение 2 мин для обмена буферным раствором. Еще раз отбросьте элюированный буфер и коллекционную трубку.
- Поместите буферно-обменные колонны в чистые центрифужные трубки объемом 1,5 мл. Приложите до 75 мкл образца к центру кровати. Открутите при 1000 × г в течение 4 мин.
ПРИМЕЧАНИЕ: Не беспокойте кровать и не прикасайтесь к боковым сторонам колонны; самая высокая точка гелевой среды должна указывать на внешний ротор. - Соберите элюат из коллекционной трубки, так как он содержит очищенную нуклеиновую кислоту. Для количественной оценки образца измерьте его поглощение при 260 нм; пометьте этот S3.
ПРИМЕЧАНИЕ: Обратите внимание на длину пути, используемую при измерении, и рассчитайте концентрацию, используя закон Бира-Ламберта.
10. Денатурирующий PAGE анализ конъюгата BG-олигонуклеотидов
- Отлить 18% трис-борат-ЭДТА (ТБЭ)-мочевину PAGE гель. Растворить 4,8 г мочевины, 4,5 мл 40% акриламида (19:1) и 1 мл 10x TBE в 2,8 мл ddH2O; добавить 5 мкл тетраметилэтилендиамина (TEMED) и тщательно перемешать. Повторить со 100 мкл 10% персульфата аммония (АФС). Вылейте раствор в пустую гелевую кассету и оставьте полимеризацию в течение 40 мин.
ПРИМЕЧАНИЕ: Используйте соответствующие средства индивидуальной защиты при обращении с мочевиной (вызывает раздражение глаз и кожи), акриламидом (токсичным и канцерогенным) и TEMED (токсичным, легковоспламеняющимся, коррозионным). Таблица 10 содержит реакционную формулу для 18% полиакриламидного геля TBE-UREA. - В микроволновой печи 500 мл буфера TBE (0,5x) в течение 2 мин и 30 с или до ~70 °C и влейте в гель-аппарат. Готовят формамид (денатурирующий) нагрузочный краситель, содержащий 95% формамида + 1 мМ ЭДТА и бромфенол синего цвета. Смешайте загрузочный краситель с каждым образцом и загрузите смесь на полиакриламидный гель.
ПРИМЕЧАНИЕ: Используйте соответствующие средства индивидуальной защиты при обращении с формамидом, поскольку он является канцерогенным. В таблице 11 приведена таблица загрузки образца геля. - Запускайте гель при 270 В в течение 35 мин или до тех пор, пока передняя часть красителя не мигрирует в конец. Поместите гель в гелевую коробку и окрасьте цианиновым красителем для нуклеиновых кислот в течение 15 минут при комнатной температуре перед визуализацией.
ПРИМЕЧАНИЕ: Используйте соответствующие средства индивидуальной защиты при обращении с цианиновым красителем, поскольку он горючий.
11. Конъюгация олигонуклеотида с SNAP T7 RNAP и PAGE анализом
- Подготовьте реагенты для аналитической шкалы связи BG-олигонуклеотида с SNAP T7 RNAP: сделайте 9 разведений одноцепочечной ДНК (ssDNA) олиго с ddH2O для создания соотношения олиго:RNAP в диапазоне от 5:1 до 1:5. Разбавить белковый запас до 50 мкМ.
ПРИМЕЧАНИЕ: Примеры соотношений можно найти в таблице 12; эти соотношения рассчитываются с использованием концентрации RNAP 50 мкМ. - Для каждого разбавления олиго ssDNA вводят 10 мкл реакционной смеси, содержащей 2 мкл буфера SNAP, 4 мкл BG-олигонуклеотида и 4 мкл SNAP T7 RNAP.
ПРИМЕЧАНИЕ: Таблица 13 содержит формулы реакций для реакции маркировки SNAP-меток.- Подготовьте еще два контрольных образца: 1) контроль RNAP путем замены BG-олигонуклеотида на ddH2O; 2) контроль ДНК путем замены SNAP T7 RNAP на ddH2O(для самой низкой концентрации олигонуклеотидов SNAP T7 RNAP). Инкубировать все образцы при комнатной температуре в течение 1 ч и держать на льду до тех пор, пока это не понадобится.
- Настройте одиннадцать реакций по 10 мкл, добавив 2 мкл каждого образца к 4 мкл буфера SNAP и 2 мкл красителя с белковой нагрузкой, и нагрейте при 70 °C в течение 10 мин. Загрузите 2 мкл каждого образца на 4-12% белковый гель Bis-Tris и выполните гель-электрофорез на льду при 200 В в течение 35 мин.
ПРИМЕЧАНИЕ: В таблице 14 приведены реакционные формулы для образцов, нагружающих гель.- Смойте SDS с помощью 3-кратного водообмена на шейкере, каждая стирка длится 10 минут каждая. Окрашивание цианиновым красителем на нуклеиновые кислоты за 15 мин перед визуализацией. Окрасьте гель еще раз, используя 20 мл синего пятна Coomassie в течение 1 ч. Обезжиривание ddH2Oв течение 1 ч (или на ночь) перед визуализацией.
ПРИМЕЧАНИЕ: В геле одна из реакций будет производить наиболее привязанную полимеразу вместе с наименьшим количеством избыточного свободного BG-олигонуклеотида; это оптимальное соотношение.
- Смойте SDS с помощью 3-кратного водообмена на шейкере, каждая стирка длится 10 минут каждая. Окрашивание цианиновым красителем на нуклеиновые кислоты за 15 мин перед визуализацией. Окрасьте гель еще раз, используя 20 мл синего пятна Coomassie в течение 1 ч. Обезжиривание ddH2Oв течение 1 ч (или на ночь) перед визуализацией.
- Готовят реагенты для препаративной шкалы связи BG-олигонуклеотида с SNAP T7 RNAP. Выполните реакцию соединения с оптимальным соотношением, найденным в аналитической шкале.
ПРИМЕЧАНИЕ: Сведите к минимуму воздействие белка на комнатную температуру, поместив белок на лед, когда он не используется.
12. Очистка олигонуклеотидно-привязанного SNAP-T7 с помощью ионообменных колонн
- Следуйте инструкциям производителя по настройке трубки, если она отклоняется от инструкций, перечисленных здесь. Приготовьте буфер очистки с рН выше изоэлектрической точки белка.
ПРИМЕЧАНИЕ: Для примера белка в этом протоколе использовался буфер очистки 10 мМ буфера фосфата натрия (рН 7).- Готовят 1000 мкл буфера элюирования, содержащего конечные концентрации 50 мМ Tris и 0,5 М NaCl. Смешайте 50 мкл 1 M Tris, 100 мкл 5 M NaCl и 850 мкл ddH2O.
ПРИМЕЧАНИЕ: Таблица 15 содержит реакционную формулу для буфера элюирования.
- Готовят 1000 мкл буфера элюирования, содержащего конечные концентрации 50 мМ Tris и 0,5 М NaCl. Смешайте 50 мкл 1 M Tris, 100 мкл 5 M NaCl и 850 мкл ddH2O.
- Поместите колонну в центрифужную трубку объемом 2 мл и промывайте буфером очистки при 2000 × г в течение 15 мин или до тех пор, пока весь буфер не будет элюирован. Удалите элюированный буфер.
- Разбавьте каждый образец буфером очистки при соотношении 3:1 буфер очистки:проба и загрузите образец в колонку 400 мкл за один раз. Вращайтесь при 2000 × г в течение 10 минут или до тех пор, пока весь буфер не будет элюирован. Соберите сквозной поток и пометьте его как проточный.
- Добавьте 400 мкл буфера очистки в центр колонны. Вращайтесь при 2000 × г в течение 15 мин или до тех пор, пока весь буфер не будет элюирован. Соберите сквозной поток и пометьте его как промывку 1. Повторите еще два раза для стирки 2 и стирки 3.
- Добавьте 50 мкл буфера элюирования в центр столбца. Вращайтесь при 2000 × г в течение 5 минут или до тех пор, пока весь буфер не будет элюирован. Соберите сквозной поток и пометьте его как элюат 1. Повторите еще два раза для элюата 2 и элюата 3.
- Пул элюирует 1, 2 и 3 (маркируйте этот общий элюат),оставляя небольшую долю каждого элюата для геля, и измеряйте поглощение при 260 нм (A260) и 280 нм (A280). После измерения добавляют глицерин в соотношении 1:1 и хранят при -20 °C до дальнейшего использования.
- Используйте центробежный фильтрующий блок (0,5 мл; 30 кДа) для буферно-обменного общего элюирования с 2-кратным буфером хранения (~1:100) (маркировка этого продукта). Снова мера A260/280. Добавьте глицерин в соотношении 1:1 и храните при -20 °C до дальнейшего использования.
- Загрузите каждый элюат: проточный, промывочный 1-3, общий элюат и продукт в 4-12% гель Bis-Tris SDS-PAGE вместе с белковой лестницей. Работайте при напряжении 200 В в течение 35 минут или до тех пор, пока передняя часть красителя не мигрирует в конец.
13. Демонстрация контроля по требованию активности привязанной РНК-полимеразы
- Подготовьте 5-кратный буфер отжига, содержащий 25 мМ Tris, 5 мМ ЭДТА и 25 мМ хлорида магния (MgCl2). Смешайте 2,4 мкл каждого шаблона (1 мкМ) с 5 мкл буфера отжига и 14,2 мкл ddH2Oс образованием 25 мкл клетки dsDNA 1 мкМ. Инкубируют этот раствор при 75 °C в течение 2 мин. Аналогично отжиг смысла и антисмысловых нитей промотора и малахитового зеленого аптамера шаблона ДНК. Приготовьте 1мМ раствор малахитового зеленого оксалата.
ПРИМЕЧАНИЕ: Таблица 16 содержит реакционную формулу для 5-кратного буфера отжига, Таблица 17 содержит реакционную формулу для отжига двух шаблонов ssDNA. - Инкубируют привязанный SNAP T7 RNAP с клеткой dsDNA в молярном соотношении 1:5 при комнатной температуре в течение 15 мин до конечной концентрации 500 нМ РНКП. Держитесь на льду до тех пор, пока это не понадобится.
- Разогрейте пластинчатый считыватель до 37 °C. Настройте три реакции IVT 25 мкл на льду
- Наладить реакцию, содержащую клетку SNAP T7RNAP с факторами транскрипции нуклеиновых кислот. Смешайте 2,5 мкл 10x буфера IVT, 1 мкл смеси rNTP 25 мМ, 1 мкл 1 мМ малахитового зеленого, 2,5 мкл смеси RNAP-клетка, 2,5 мкл каждой из 1 мкМ транскрипционного фактора А и В олигонуклеотидных нитей и 3 мкл 1 мМ малахитового зеленого аптамера в 10 мкл ddH2O.
- Наладите реакцию, содержащую клетку SNAP T7RNAP без факторов транскрипции нуклеиновых кислот. Смешайте 2,5 мкл 10x буфера IVT, 1 мкл смеси rNTP 25 мМ, 1 мкл 1 мМ малахитового зеленого, 2,5 мкл смеси RNAP-клетка и 3 мкл 1 мМ шаблона малахитового зеленого аптамера в 15 мкл ddH2O.
- Настройте реакцию, содержащую только буфер. Смешайте 2,5 мкл 10x буфера IVT, 1 мкл смеси rNTP 25 мМ, 1 мкл 1 мМ малахитового зеленого и 3 мкл 1 мМ малахитового зеленого аптамера в 17,5 мкл ddH2O.
ПРИМЕЧАНИЕ: Таблица 18 содержит общий справочник по реакциям транскрипции in vitro.
- Перенесите каждую реакцию на пластину из 384 скважин. Мониторинг транскрипции малахитового зеленого аптамера на флуоресцентном пластинчатом считывателе в течение 2 ч при 37 °C и с возбуждением 610 нм и эмиссией 655 нм. После того, как закончите, держите пластину на льду до тех пор, пока это не понадобится.
- Микроволновая печь 0,5x TBE буфер в течение 2 мин 30 с или до ~70 °C. Запускают РНК-продукты каждой лунки в денатурирующем 12% полиакриламидном геле TBE-мочевины в нагретом 0,5x TBE буфере при 280 В в течение 20 мин или до тех пор, пока передняя часть красителя не достигнет конца. Окрашивайте гель окрашиванием нуклеиновой кислоты цианинового красителя в течение 10 мин на орбитальном шейкере перед визуализацией.
ПРИМЕЧАНИЕ: Таблица 19 содержит реакционную формулу для денатурирующего 12% Геля TBE-Urea PAGE.
Access restricted. Please log in or start a trial to view this content.
Результаты
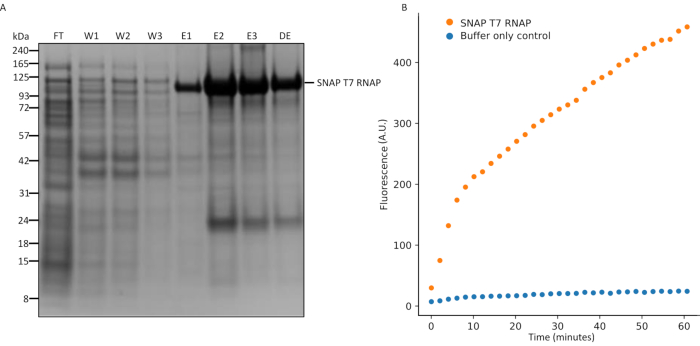
Рисунок 5:SDS-PAGE анализ экспрессии SNAP T7 RNAP и анализа транскрипции in vitro. (A) Анализ очистки белка SNAP T7 RNAP, молекулярная масса SNAP T7 RNAP: 119,4 кДа. FT = протекание из колонны, W1 = фракции элюирования про?...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Это исследование демонстрирует основанный на нанотехнологиях подход к контролю активности РНК-полимеразы Т7 путем ковалентного соединения N-терминально помеченного SNAP рекомбинантного Т7-РНКП с BG-функционализированным олигонуклеотидом, который впоследствии использовался для програ?...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Нет никаких конкурирующих финансовых интересов, которые мог бы декларировать ни один из авторов.
Благодарности
L.Y.T.C признает щедрую поддержку со стороны Фонда исследований «Новые рубежи в исследованиях» (NFRF-E), Гранта Совета по естественным наукам и инженерным исследованиям Канады (NSERC) Discovery Grant и Инициативы «Медицина по дизайну» Университета Торонто, которая получает финансирование от Канадского фонда первого научного совершенства (CFREF).
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| 0.5% polysorbate 20 (TWEEN 20) | BioShop | TWN510.5 | |
| 0.5M ethylenediaminetetraacetic acid (EDTA) | Bio Basic | SD8135 | |
| 10 mM sodium phosphate buffer (pH 7) | Bio Basic | PD0435 | Tablets used to make 10 mM buffer |
| 10% ammonium persulfate (APS) | Sigma Aldrich | A3678-100G | |
| 100 kDa Amicon Ultra-15 Centrifugal Filter Unit | Fisher Scientific | UFC910008 | |
| 100% acetone | Fisher Chemical | A18P4 | |
| 100% ethanol (EtOH) | House Brand | 39752-P016-EAAN | |
| 10x in vitro transcription (IVT) buffer | New England Biolabs | B9012 | |
| 10x Tris-Borate-EDTA (TBE) buffer | Bio Basic | A0026 | |
| 1M Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Sigma Aldrich | I5502-1G | |
| 1M sodium bicarbonate buffer | Sigma Aldrich | S6014-500G | |
| 1M Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 648311-1KG | |
| 1X Tris-EDTA (TE) buffer | ThermoFisher | 12090015 | |
| 2M imidazole | Sigma Aldrich | 56750-100G | |
| 2-mercaptoethanol (BME) | Sigma Aldrich | M3148 | |
| 3M sodium acetate | Bio Basic | SRB1611 | |
| 40% acrylamide (19:1) | Bio Basic | A00062 | |
| 4x LDS protein sample loading buffer | Fisher Scientific | NP0007 | |
| 5M sodium chloride (NaCl) | Bio Basic | DB0483 | |
| 5mM dithiothreitol (DTT) | Sigma Aldrich | 43815-1G | |
| 6x gel loading dye | New England Biolabs | B7024S | |
| agarose B powder | Bio Basic | AB0014 | |
| BG-GLA-NHS | New England Biolabs | S9151S | |
| BL21 competent E. coli | Addgene | C2530H | |
| BLUeye prestained protein ladder | FroggaBio | PM007-0500 | |
| bromophenol blue | Bio Basic | BDB0001 | |
| coomassie blue (SimplyBlue SafeStain) | ThermoFisher | LC6060 | |
| cyanine dye (SYBR Gold nucleic acid gel stain) | Fisher Scientific | S11494 | |
| cyanine dye (SYBR Safe nucleic acid gel stain) | Fisher Scientific | S33102 | |
| dry dimethyl sulfoxide (DMSO) | Fisher Scientific | D12345 | |
| formamide | Sigma Aldrich | F9037-100ML | |
| glycerol | Bio Basic | GB0232 | |
| kanamycin sulfate | BioShop | KAN201.5 | |
| lysogeny broth | Sigma Aldrich | L2542-500ML | |
| malachite green oxalate | Sigma Aldrich | 2437-29-8 | |
| N,N,N'N'-Tetramethylethane-1,2-diamine (TEMED) | Sigma Aldrich | T9281-25ML | |
| NuPAGE MES SDS running buffer (20x) | Fisher Scientific | LSNP0002 | |
| NuPAGE Novex 4-12% Bis-Tris gel 1.0 mm 12-well | Life Technologies | NP0322BOX | |
| oligonucleotide (cage antisense) | IDT | N/A | TATAGTGAGTCGTATTAATTTG |
| oligonucleotide (cage sense) | IDT | N/A | TCAGTCACCTATCTGTTTCAAA TTAATACGACTCACTATA |
| oligonucleotide (malachite green aptamer antisense) | IDT | N/A | GGATCCATTCGTTACCTGGCT CTCGCCAGTCGGGATCCTATA GTGAGTCGTATTACAGTTCCAT TATCGCCGTAGTTGGTGTACT |
| oligonucleotide (malachite green aptamer sense) | IDT | N/A | TAATACGACTCACTATAGGATC CCGACTGGCGAGAGCCAGGT AACGAATGGATCC |
| oligonucleotide (Transcription Factor A) | IDT | N/A | AGTACACCAACTACGAGTGAG |
| oligonucleotide (Transcription Factor B) | IDT | N/A | TCAGTCACCTATCTGGCGATAA TGGAACTG |
| oligonucleotide with 3’ Amine modification (tether) | IDT | N/A | GCTACTCACTCAGATAGGTGAC TGA/3AmMO/ |
| Pierce strong ion exchange spin columns | Fisher Scientific | 90008 | |
| plasmid encoding SNAP T7 RNAP and kanamycin resistance genes | Genscript | N/A | custom gene insert |
| protein purification column (HisPur Ni-NTA spin column) | Fisher Scientific | 88226 | |
| rNTP mix | New England Biolabs | N0466S | |
| Roche mini quick DNA spin column | Sigma Aldrich | 11814419001 | |
| Triton X-100 | Sigma Aldrich | T8787-100ML | |
| Ultra Low Range DNA ladder | Fisher Scientific | 10597012 | |
| urea | BioShop | URE001.1 |
Ссылки
- Cherry, K. M., Qian, L. Scaling up molecular pattern recognition with DNA-based winner-take-all neural networks. Nature. 559 (7714), 370-376 (2018).
- Qian, L., Winfree, E., Bruck, J. Neural network computation with DNA strand displacement cascades. Nature. 475 (7356), 368-372 (2011).
- Chen, Y. -J., et al. Programmable chemical controllers made from DNA. Nature Nanotechnology. 8 (10), 755-762 (2013).
- di Bernardo, D., Marucci, L., Menolascina, F., Siciliano, V. Predicting synthetic gene networks. Synthetic Gene Networks: Methods and Protocols. 813, 57-81 (2012).
- Xiang, Y., Dalchau, N., Wang, B. Scaling up genetic circuit design for cellular computing: advances and prospects. Natural Computing. 17 (4), 833-853 (2018).
- Gould, N., Hendy, O., Papamichail, D. Computational tools and algorithms for designing customized synthetic genes. Frontiers in Bioengineering and Biotechnology. 2, (2014).
- MacDonald, J. T., Siciliano, V. Computational sequence design with R2oDNA Designer. Mammalian Synthetic Promoters. 1651, 249-262 (2017).
- Cervantes-Salido, V. M., Jaime, O., Brizuela, C. A., Martínez-Pérez, I. M. Improving the design of sequences for DNA computing: A multiobjective evolutionary approach. Applied Soft Computing. 13 (12), 4594-4607 (2013).
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. Journal of Computational Chemistry. 32 (1), 170-173 (2011).
- Fornace, M. E., Porubsky, N. J., Pierce, N. A. A unified dynamic programming framework for the analysis of interacting nucleic acid strands: enhanced models, scalability, and speed. ACS Synthetic Biology. 9 (10), 2665-2678 (2020).
- Wetterstrand, K. DNA sequencing costs: Data. Genome.gov. , (2020).
- Lopez, R., Wang, R., Seelig, G. A molecular multi-gene classifier for disease diagnostics. Nature Chemistry. 10 (7), 746-754 (2018).
- Pardee, K., et al. low-cost detection of Zika virus using programmable biomolecular components. Cell. 165 (5), 1255-1266 (2016).
- Yurke, B., Turberfield, A. J., Mills, A. P., Simmel, F. C., Neumann, J. L. A DNA-fuelled molecular machine made of DNA. Nature. 406 (6796), 605-608 (2000).
- Lin, K. N., Volkel, K., Tuck, J. M., Keung, A. J. Dynamic and scalable DNA-based information storage. Nature Communications. 11 (1), 2981(2020).
- Yurke, B., Mills, A. P. Using DNA to power nanostructures. Genetic Programming and Evolvable Machines. 4 (2), 111-122 (2003).
- Zhang, D. Y., Turberfield, A. J., Yurke, B., Winfree, E. Engineering entropy-driven reactions and networks catalyzed by DNA. Science. 318 (5853), 1121-1125 (2007).
- Wang, B., Thachuk, C., Ellington, A. D., Winfree, E., Soloveichik, D. Effective design principles for leakless strand displacement systems. Proceedings of the National Academy of Sciences. 115 (52), 12182-12191 (2018).
- Machinek, R. R. F., Ouldridge, T. E., Haley, N. E. C., Bath, J., Turberfield, A. J. Programmable energy landscapes for kinetic control of DNA strand displacement. Nature Communications. 5 (1), 5324(2014).
- Cabello-Garcia, J., Bae, W., Stan, G. -B. V., Ouldridge, T. E. Handhold-mediated strand displacement: a nucleic acid-based mechanism for generating far-from-equilibrium assemblies through templated reactions. bioRxiv. , (2020).
- Brophy, J. A. N., Voigt, C. A. Principles of genetic circuit design. Nature Methods. 11 (5), 508-520 (2014).
- Khalil, A. S., et al. A synthetic biology framework for programming eukaryotic transcription functions. Cell. 150 (3), 647-658 (2012).
- Swank, Z., Laohakunakorn, N., Maerkl, S. J. Cell-free gene-regulatory network engineering with synthetic transcription factors. Proceedings of the National Academy of Sciences. 116 (13), 5892-5901 (2019).
- Howland, S. W., Tsuji, T., Gnjatic, S., Ritter, G., Old, L. J., Wittrup, K. D. Inducing efficient cross-priming using antigen-coated yeast particles. Journal of immunotherapy. 31 (7), 607(2008).
- Abil, Z., Ellefson, J. W., Gollihar, J. D., Watkins, E., Ellington, A. D. Compartmentalized partnered replication for the directed evolution of genetic parts and circuits. Nature Protocols. 12 (12), 2493-2512 (2017).
- Baugh, C., Grate, D., Wilson, C. 2.8 Å crystal structure of the malachite green aptamer11. Journal of Molecular Biology. Doudna, J. A. 301 (1), 117-128 (2000).
- Chou, L. Y. T., Shih, W. M. In vitro transcriptional regulation via nucleic acid-based transcription factors. ACS Synthetic Biology. 8 (11), 2558-2565 (2019).
- Lykke-Andersen, J., Christiansen, J. The C-terminal carboxy group of T7 RNA polymerase ensures efficient magnesium ion-dependent catalysis. Nucleic Acids Research. 26 (24), 5630-5635 (1998).
- Pu, J., Disare, M., Dickinson, B. C. Evolution of C-terminal modification tolerance in full-length and split T7 RNA Polymerase biosensors. Chembiochem. 20 (12), 1547-1553 (2019).
- Gardner, L. P., Mookhtiar, K. A., Coleman, J. E. Initiation, elongation, and processivity of carboxyl-terminal mutants of T7 RNA polymerase. Biochemistry. 36 (10), 2908-2918 (1997).
- Yin, J., Lin, A. J., Golan, D. E., Walsh, C. T. Site-specific protein labeling by Sfp phosphopantetheinyl transferase. Nature Protocols. 1 (1), 280-285 (2006).
- Warden-Rothman, R., Caturegli, I., Popik, V., Tsourkas, A. Sortase-tag expressed protein ligation: combining protein purification and site-specific bioconjugation into a single step. Analytical Chemistry. 85 (22), 11090-11097 (2013).
- Zhang, W. -B., Sun, F., Tirrell, D. A., Arnold, F. H. Controlling macromolecular topology with genetically encoded SpyTag-SpyCatcher chemistry. Journal of the American Chemical Society. 135 (37), 13988-13997 (2013).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены