È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un modello cellule endoteliali Planar for Imaging immunologiche Synapse Dynamics
In questo articolo
Riepilogo
Adaptive immunity is controlled by dynamic 'immunological synapses' formed between T cells and antigen presenting cells. This protocol describes methods for investigating endothelial cells both as understudied physiologic APCs and as a novel type of 'planar cellular APC model'.
Abstract
Immunità adattativa è regolata da interazioni dinamiche tra cellule T e cellule presentanti l'antigene ('APC') denominati «sinapsi immunologica. All'interno di queste interfacce cellula-cellula intime discrete sub-cellulari cluster di MHC / Ag-TCR, F-actina, adesione e molecole di segnalazione si formano e rimodellano rapidamente. Queste dinamiche si pensa siano fattori determinanti sia per l'efficienza e la qualità delle risposte immunitarie che sviluppano e quindi di protezione contro l'immunità patologica. Attuale comprensione delle sinapsi immunologiche con fisiologica APC è limitata dalla inadeguatezza della risoluzione delle immagini ottenibili. Anche se i modelli substrato artificiale (ad esempio, doppi strati lipidici planari) offrono un'eccellente risoluzione e sono stati strumenti estremamente utili, essi sono intrinsecamente non-fisiologica e semplicistica. Cellule endoteliali vascolari e linfatiche sono emersi come un importante tessuto periferico (o stromale) Vano di 'semi-professionaleAl APC '. Questi APC (che esprimono la maggior parte del macchinario molecolare di professionisti APC) hanno la caratteristica unica di formare superficie cellulare praticamente piana e sono immediatamente transfectable (ad esempio, con i giornalisti proteina fluorescente). Qui un approccio di base per l'attuazione cellule endoteliali come un romanzo e fisiologica 'planare modello APC cellulare' per migliorare l'imaging e l'interrogazione dei processi di segnalazione antigeniche fondamentali sarà descritto.
Introduzione
Linfociti T sono un ramo del sistema immunitario adattativo caratterizzato dalla capacità di riconoscere efficientemente antigene peptide (Ag) vincolato al complesso maggiore di istocompatibilità (MHC) molecole attraverso i recettori delle cellule T (TCR) 1. Linfociti naïve costitutivamente migrano e Scan 'Ag cellule presentanti professionali »(APC, ad esempio, cellule dendritiche) all'interno dei linfonodi, mentre le cellule di memoria / T effettrici devono sorvegliare efficacemente una gamma estremamente ampia di APC e potenziali cellule bersaglio all'interno dei tessuti periferici.
Nel min seguente iscrizione iniziale delle cognate Ag su un APC, linfociti arrestare la loro migrazione e cominciano a formare un professionista intimo un'interfaccia cellula-cellula chiamato 'sinapsi immunologica' (IS). Sostenuta (cioè, 30-60 min) È contatti sono tenuti per amplificare e sostenere segnalazione 2-7. Studi emergenti identificano che all'interno della IS, è la formazione continua e rapida remodeling di discrete segnalazione sub-cellulare micro-cluster (ad esempio, contengono MHC / Ag-TCR, F-actina, adesione e molecole di segnalazione), che determinano la forza e la qualità della conseguente risposta immunitaria 2-7. Tuttavia, i dettagli dinamici e meccanismo di regolazione di questo processo non sono completamente compresi 8,9. Ciò deriva in gran parte da problemi tecnici connessi con topologie irregolari di superfici APC e l'orientamento mal controllata dei piani di interazione cellula-cellula, i problemi che limitano profondamente imaging spazio-temporale richiesto avvicina 8-10 (Figure1A).
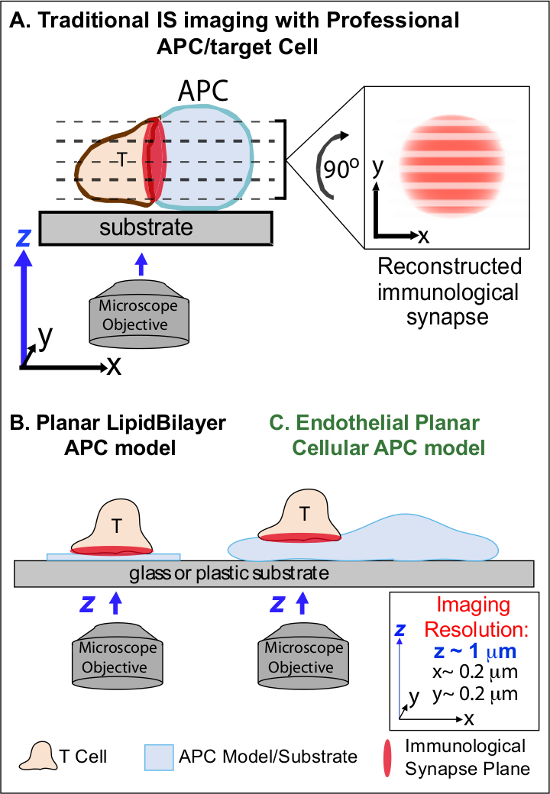
Figura 1. Una fisiologica Planar APC cellulare Modello for Imaging immunologiche Synapse Dynamics. Lo schema mostra l'imaging tradizionale di sinapsi immunologica tra una cellula T e un professio nal APC (A) e cellule T e un modello tradizionale lipidico planare doppio strato APC (B) rispetto a questo romanzo endoteliale modello planare APC (C). APC professionali forniscono sinapsi immunologiche fisiologiche ma offrono interfaccia mal orientata cellula-cellula (cioè, rispetto al piano ottimale di imaging xy; risoluzione ~ 0,2 micron), che compromette notevolmente spaziale (piano di imaging z risoluzione ~ 1 micron) e temporale (cioè, a causa della necessità di eseguire la scansione ripetutamente attraverso tutti i piani di imaging z) Risoluzione di imaging. Modelli a doppio strato hanno una topologia planare che fornisce la risoluzione ottimale delle immagini spazio-temporale, ma sono anche molto semplificate, non fisiologica e rigida. Questo modello delle cellule endoteliali combina la topologia planare di doppi strati lipidici con il substrato fisiologico di un classico APC per fornire la risoluzione ottimale di imaging spaziale e temporale in un ambiente fisiologico.m / files / ftp_upload / 53288 / 53288fig1large.jpg "target =" _ blank "> Clicca qui per vedere una versione più grande di questa figura.
Il lavoro precedente ha parzialmente aggirato questi ostacoli attraverso lo sviluppo di modelli di substrato planare (ad esempio, doppi strati lipidici e superfici rivestiti con anticorpo) che forniscono la risoluzione spazio-temporale ottimale (cioè, mediante il fissaggio alla superficie di attivazione delle cellule T in un unico piano che è parallelo al di imaging ottimale xy piano) 11-15 (Figura 1B). Questi modelli hanno facilitato importanti conoscenze sulle dinamiche subcellulari / molecolari che controllano la segnalazione antigenico nelle cellule T, tra cui la scoperta di actina dinamico / TCR segnalazione micro-cluster 7,11-14. Tuttavia, tali modelli sono intrinsecamente avevano semplificato, così come rigida (precludendo sviluppo / studio delle caratteristiche topologiche 3-dimensionali) (Figura 1B). Pertanto, rimane incerto su come relazionarsi tali risultati al PHYsiologic cellula-cellula sorveglianza immunitaria.
Sebbene ancora poco studiato, vascolari e cellule endoteliali linfatiche stanno emergendo come un grande (vale a dire, maggiore in numero rispetto a tutti APC professionali, da ~ 1.000 volte) compartimento periferico di 'semi-professionale' APC 16-18. Queste cellule esprimono MHC-I-, MHC-II e una moltitudine di molecole co-stimolatore (ad esempio, CD40, LFA3, ICOSL, 4-1BB, OX40L, TL1A, PD-L1, ma non CD80 e CD86) e sono strategicamente posizionato all'interfaccia sangue tessuto in cui servono le funzioni di sentinella specializzate 16-18. Studi precedenti hanno dimostrato che le cellule endoteliali possono effettivamente ri-stimolare effettori / memoria, ma non ingenua, le cellule T 19-25. Così, le cellule endoteliali sono suscettibili di svolgere ruoli unici APC in fase effettrice delle risposte immunitarie adattive all'interno dei tessuti periferici, come ad esempio l'influenza locale all'attivazione delle cellule T, la differenziazione, la memoria e la tolleranza 16,17,26. Criticamente, quando coltivate in vitro, le cellule endoteliali formano superfici cellulari praticamente piane e sono immediatamente transfectable (ad esempio, con i reporter proteina fluorescente). Queste caratteristiche sono ideali per imaging ad alta risoluzione spazio-temporale delle dinamiche topologiche durante le interazioni cellula-cellula 19,27. Così le cellule endoteliali potrebbe servire come 'cellulare planare APC' modello fisiologico distintamente adatto per lo studio dei meccanismi di rimodellamento sottocellulari / molecolare che guidano riconoscimento dell'antigene e regolano le risposte (Figura 1C) 19,20.
Precedentemente stabilito tecniche di imaging complementari (comprese trasfezione di cellule endoteli con accessori proteina fluorescente della membrana plasmatica e citosol) per studiare i dettagli di interazione leucociti-endotelio durante adesione e migrazione transendoteliale 27, hanno mostrato che i leucociti sondare attivamente superficie dell'endotelio dalla dinamica l'inserimento di und retrazione del sub-micron-scala, sporgenze cilindriche ricco di actina (~ 200-1.000 nm di diametro e profondità) definito invadosome-come sporgenze (vale a dire, 'ILPS') 27,28. Questi approcci di imaging sono state ulteriormente espansa insieme alla creazione di protocolli di sfruttare funzione endoteliale APC per sviluppare i primi metodi per un'alta risoluzione spazio-temporale della cellula endoteliale sinapsi immunologica T come riportato 19,20 e ulteriormente descrivere qui. Un risultato centrale derivato da questo romanzo planare cellulare modello APC è che ILPS cellule T funzionano sia nel promuovere il riconoscimento iniziale Ag e nel sostenere la successiva segnalazione. In effetti, gli array di più ILPS (che sono stati stabilizzati e maturati in risposta alla prima flusso di calcio) spettacolo di arricchimento in TCR e molecole suggestivi della segnalazione attiva tale PKC-Q, ZAP-70, phosphotyrosine e HS1. Pertanto, ILPS sembrano rappresentare un equivalente fisiologica tridimensionale al micro TCR segnalazionecluster visto in modelli doppio strato planari. Questo approccio, quindi, sensibilmente rivela / report dinamica molecolare e architettonici (e implicite biomeccanica) non altrimenti rilevabile.
Il metodo qui descritto dovrebbe essere utile per colmare il divario tra APC professionali e modelli substrato artificiale APC, al fine di migliorare la nostra capacità di interrogare meccanismi di base della risposta immunitaria adattativa. Mentre qui l'attenzione è sull'attivazione di CD4 + di tipo Th1 effettore / cella di memoria, questo approccio di base può essere facilmente modificato per studiare una vasta gamma di tipi di cellule T e Ags, come discusso di seguito.
Protocollo
Tutti gli esperimenti descritti in questo protocollo sono condotte con cellule T umane primarie e disponibili in commercio cellule endoteliali umane primarie (per via cutanea o microvascolare polmone EC) .Any protocollo di ricerca che coinvolge soggetti umani deve essere approvato da un comitato istituzionale di revisione e consenso informato scritto deve essere fornita da ogni donatore di sangue. Esperimenti condotti utilizzando questo protocollo è stato approvato dal IRB di Beth Israel Deaconess Medical Center.
1. Preparazione cellule CD4 + Th1 umana Effector / T di memoria
- Applicare laccio emostatico al braccio del donatore, pulire vena con alcool, e inserire l'ago. Disegnare lentamente 15 ml di sangue in vacutainer con EDTA come anticoagulante. Quando il sangue è stato redatto, slegare il laccio emostatico prima di rimuovere l'ago. Applicare immediatamente pressione a ferire con garza sterile quando l'ago è stato rimosso.
- Trasferire il sangue in una provetta da 50 ml. Aggiungere RPMI-1640 a RT ad una diluizione 1: 1 (volume finale 30 ml). Sovrapporre con cautela il diluitad sangue su due provette da 50 ml contenente 15 ml di pre-filtrata linfociti terreno di isolamento come Ficoll-Paque a RT.
- Centrifugare il gradiente a temperatura ambiente per 30 min a 1200 xg in un rotore oscillante secchio. Mentre centrifugazione preparare medio delle cellule T (500 ml di RPMI-1640, 50 ml di FCS, 5 ml di penicillina / streptomicina).
- Alla rimozione del tubo 50 ml dalla centrifuga, osservare quattro strati: un pellet di globuli rossi nel fondo, il Paque, uno strato di cellule che contiene globuli bianchi (inclusi linfociti) e il plasma. Rimuovere accuratamente lo strato di globuli bianchi con una pipetta Pasteur e trasferire in un tubo Falcon da 50 ml.
- Lavare lo strato di globuli bianchi aggiungendo RT RPMI-1460 (fino a 20 ml) e centrifugazione a RT per 5 min a 1200 x g. Risospendere le cellule bianche del sangue in 1 ml di terreno cellule T. Aggiungere 5 ml di sospensione 1 ml di cellule di 250 microlitri medio delle cellule T in una provetta da centrifuga da 1,5 ml e mescolare delicatamente su e giù con una pipetta.
- Aggiungere 25 ml diluiresospensione cellulare d al 25 microlitri 0,4% tripan blu. Aggiungere 10 ml di miscela da ciascun lato di un emocitometro standard.
- Mettere emocitometro su un microscopio a luce bassa potenza. Utilizzando un obiettivo 10X contare il numero di cellule che hanno escluso il colorante blu Trypan e sono presenti nella piazza centrale di entrambi i lati della emocitometro vivente.
- Per calcolare la concentrazione cellulare, moltiplicare la media dei 2 quadrati 100 (fattore di diluizione) e quindi moltiplicare per 10 4 invia il numero di cellule / ml.
- Regolare la concentrazione finale di 0,5 x 10 6 cellule / ml in mezzo di cellule T. Aggiungere una concentrazione finale di 1 mg / ml di superantigeni batteriche stafilococco enterotossina B (SEB) e sindrome da shock tossico tossina 1 (TSST) alle cellule. Cultura per 72 ore (37 ° C e 5% di CO 2) per espandere la popolazione di cellule T CD4 +.
- Pellet cellule T (1.200 XG, 5 min) e risospendere a 0.5 x 10 6 cellule / ml in mezzo di cellule T con ilaggiunta di IL-15 (20 ng / ml). Trasferimento linfociti in un pallone T150. Continuare a espandere / celle divise in pieno medio-IL-15 ogni 24-48 ore, se necessario (in base al colore del supporto, cioè, ogni volta che i media si trasforma da rosa a leggermente giallo) in seguito. Mantenere la popolazione linfocitaria risultante per un massimo di 15 giorni.
NOTA: In base alla progettazione, questo protocollo si attiva e ampliare specificamente sottogruppo di cellule T CD4 + che sono reattivi a SEB e TSST e poi spingerli verso un fenotipo Th1 come effettori / memoria. Altri globuli bianchi riescono a sopravvivere e crescere in queste condizioni, tali che passo 1.10 cellule saranno almeno il 95% CD4 +, CD45RO + T cellule 19, come si può facilmente valutata mediante citometria a flusso. Se lo si desidera, ulteriore purificazione può essere facilmente raggiunto attraverso kit positivi o negativi di selezione basati anticorpo disponibile in commercio / magnetiche stallonatori.
2. Avvio di primaria Cultura cellule endoteliali umane
- Coat un pallone T25 con fibronectina (FN) 20 mg / ml in PBS in condizioni sterili. Lasciare a temperatura ambiente per 30-60 min. Rimuovere FN e aggiungere 5 ml di terreno completo (endoteliale basale medio (EBM-2) media integrato con Endothelial Growth Medium (EGM-2) singlequots). Pre-incubazione a 37 ° C coltura cellulare incubatore per almeno 30 min.
- Scongelare una fiala di surgelati polmone o cutanea microvascolari umano cellule endoteliali (HLMVECs o HDMVECs) in un bagno d'acqua a 37 °, con occasionale agitazione delicata per ~ 2-3 minuti. Trasferire immediatamente cellule a pallone T25 contenente supporti pre-riscaldato. Turbolenza e posto in incubatrice a 37 ° C con delicatezza.
- Cambiare la media dopo ~ 4-6 ore. Continuare a cambiare supporto circa ogni 48 ore (o quando i media diventa leggermente gialla) fino a quando raggiunge il piatto ~ 90-95% di confluenza.
3. Divisione Generale ed espansione delle cellule endoteliali
- Crescere le cellule a ~ 90-95 confluenza. Questa operazione può richiedere 2-5 giorni. Per la divisione,rimuovere i supporti e risciacquare con PBS. Rimuovere PBS e sostituirlo con volume minimo di 1x fresco tripsina (0,5 ml per T25 o 1,5 ml per T75). Agitare delicatamente per coprire tutta la superficie con tripsina. Incubare a 37 ° C per ~ 5 min. Monitorare il distacco delle cellule dalla piastra usando un microscopio a luce bassa potenza.
- Quando maggioranza delle cellule appaiono arrotondati o rimosso, aggiungere 5 volumi (cioè, rispetto al volume tripsina aggiunto) di completa EGM-2 di media preriscaldata e pipettare delicatamente sulla superficie del pallone di staccare tutte le cellule.
- Contare le cellule endoteliali con un emocitometro come descritto in 1,6-1,7. Agglomerare le cellule mediante centrifugazione (5 min, 1.200 XG). Rimuovere il surnatante. Regolare la concentrazione a 0,5 milioni di cellule per ml con l'aggiunta di completa EGM-2 MV supporti pre-riscaldato.
- Trasferire aliquote di cellule ai piatti o matracci FN rivestite appropriate per la manutenzione. Agitare delicatamente e metterli in incubatrice. Modificare i media all'interno 6-12 ore di placcatura. Sho mediaULD essere cambiato circa ogni 48 ore in seguito.
4. endoteliali Trasfezione cellulare
NOTA: primaria cellule endoteliali sono refrattari a trasfezione dalla maggior parte dei metodi chimici e elettroporazione comune. Il metodo basato trasfezione nucleare descritto di seguito permette relativamente alta efficienza di trasfezione (~ 50-70%). Un metodo alternativo efficace è l'uso di infezione da parte di vettori virali appropriati (vedere i commenti a materiali Tabella).
- Preparare T25 o T75 fiaschi (se necessario) di HLMVECs o HDMVECs per una densità finale di 90-95% confluency.Coat con fibronectina (FN) 20ug / ml in PBS in condizioni sterili sia microscopio piastre di coltura come piatti Delta-T (per step 5) o 12 mm vetrini circolari posizionati all'interno di un pozzo di una piastra di coltura cellulare di 24 pozzetti (per il punto 6) con come descritto in precedenza (2.1).
- Aggiungere 1 ml di completo EGM-2 di coltura alla cultura microscopio platesor 0,5 ml per ogni 24 bene ed equilibrare piastreun umidificata 37 ° C / 5% CO 2 incubatore.
- Harvest e contare le cellule endoteliali come nei passi 3.1-3.3.Centrifuge il volume richiesto di celle (0,5 milioni di cellule per campione) a 1200 xg per 5 minuti a temperatura ambiente. Risospendere accuratamente il pellet cellulare in 100 ml di RT soluzione di trasfezione nucleare per campione.
- Combina 100 l di sospensione cellulare con 1-5 mg di DNA. Trasferire cellule / sospensione DNA in provetta certificata; campione deve coprire il fondo della provetta senza bolle d'aria.
NOTA: Costrutti di targeting YFP o DsRed alla membrana cellulare (attraverso gli N-terminali 20 aminoacidi di neuromodulin che contiene un segnale per palmitoylation posttranslational) sono stati utilizzati da soli (solo membrana YFP o membrana-DsRed alone) o co-trasfettate con marcatore volumetrico citoplasmatica (per esempio, la membrana-YFP e DsRed solubile). Molti permutazioni di proteine marker fluorescenti possono essere usati. - Chiudere la provetta con il tappo. Inserire la cuvetta consospensione cellulare / DNA nel portacuvette del elettroporatore e applicare il programma elettroporazione S-005. Prendere la cuvetta dal supporto una volta che il programma è finito.
- Aggiungi ~ 500 ml di terreno di coltura pre-equilibrata alla cuvetta e rimuovere delicatamente la sospensione cellulare dalla provetta utilizzando le pipette di plastica fornite nel kit trasfezione nucleare.
- Per gli esperimenti che utilizzano piastre di coltura microscopio partizionare la sospensione cellulare da una reazione in parti uguali tra due piatti contenenti supporti pre-riscaldato (Piazza di 4.2-4.3). Per gli esperimenti che utilizzano 24 bene / piastra, una partizione reazione equamente tra 3 pozzi.
- Incubare le cellule in un umidificata 37 ° C / 5% di CO 2 incubatore e modificare i media 4-6 ore, e di nuovo a 12-16 ore dopo la trasfezione.
5. Imaging cellulare dal vivo e analisi
- Preparazione Endotelio
- Giorno 0: Co-trasfezione HLMVECs primarie con membrana YFP e DsRed solubile tramite un nucleofeLa tecnologia ction come descritto al punto 4 e la piastra sul live-cell piastre di coltura di imaging.
- Giorno 1: Sostituire media con terreno fresco contenente IFN-γ (100 ng / ml) per indurre l'espressione MHC-II. Il giorno 2. Stimolare cellule trasfettate con l'aggiunta di 20 ng / ml TNF-α ai media esistenti.
- Il giorno 3, incubare l'endotelio con 1 mg / ml ciascuna di superantigeni batteriche enterotossina stafilococcica B (SEB) e sindrome da shock tossico tossina 1 (TSST) a 37 ° C per 30-60 min immediatamente prima esperimenti. Omettere questo passaggio per condizioni di controllo '-AG'.
- Preparazione linfociti
- In parallelo con passo 5.1.3, preparare tampone A (fenolo aneritro HBSS) supplementato con 20 mM Hepes, pH 7,4 e 0,5% v / v di albumina sierica umana pre-riscaldato a 37 ° C. Prelevare un campione di linfociti in coltura e determinare la densità contando con un emocitometro (Step 1,12-1,17).
- Centrifuga 2 milioni di cellule per campione a 1,200 xg per 5 minuti a temperatura ambiente in un tubo da 15 ml. Aspirare i media e pellet di cellule delicatamente risospendere in 2 ml di tampone A tale che nessuna gruppi di cellule rimangono.
- Rimuovere una nuova aliquota di Fura-2 colorante calcio e risospendere in DMSO per fare una concentrazione magazzino di 1 mm.
- Aggiungere 2 ml di Fura-2 stock soluzione per la sospensione cellulare T (2 micron concentrazione finale), tappare la provetta e miscelare immediatamente invertendo il tubo al fine di garantire anche la dispersione di colorante. Incubare a 37 ° C per 30 min.
- Centrifugare come al punto 5.2.2. Aspirare Buffer-A e linfociti delicatamente ma accuratamente risospendere in 20-40 ml di tampone fresco-A.
- Live-cell Setup Imaging e Acquisition
NOTA: una vasta gamma di sistemi può essere impiegato per live-cell imaging di fluorescenza su microscopi ottici verticali e dall'alto in basso. Requisiti di base sono una fonte di luce e di fluorescenza filtri, una camera CCD, motorizzata commutazione filtro e persiane, uno stadio riscaldato (o microscope-mountable camera riscaldata) e software per l'acquisizione automatica delle immagini. Per questo protocollo ad alta apertura numerica, alto ingrandimento (cioè, 40X, 63X) lenti ad immersione olio sono necessari per ottenere la risoluzione spaziale necessaria. Particolare cura deve essere presa nella scelta della fonte di fluorescenza appropriata e lenti per l'imaging a base di calcio-Fura-2 come non tutti sono compatibili con i necessari 340/380 lunghezze d'onda nm di eccitazione. Un approccio alternativo (compatibile con gli insiemi verde e rosso standard filtro fluorescenza) può essere utilizzato con coloranti non ratiomentic calcio-sensibili (ad esempio, Fluo-4, Rhod-3), anche se questi non possono quantificare con precisione il flusso di calcio e solo fornire un parente / lettura quantitativa.- Attivare sistema di microscopia (PC per il funzionamento, microscopio, macchina fotografica del CCD, ruota portafiltri e lampada allo xeno).
- Aprire il software designato.
- Impostare microscopio / software per il multicanale automatizzata time-lapse imaging. Includere acquisizione sequenziale di annunciocontrasto ifferenziale interferenza (DIC), fluorescenza verde di serie, fluoresce rosso di serie e di serie di eccitazione 340 e 380 nm Fura-2 immagini. Impostare l'intervallo di acquisizione per 10-30 secondi e ha una durata complessiva di ~ 20-60 min.
- Impostare i tempi di esposizione per Fura-2 imaging.
- Aggiungere l'olio fresco obiettivo e montare un piatto di microscopio che contiene solo 0,5 ml di tampone-A sull'adattatore fase di riscaldamento e girare subito a equilibrare a 37 ° C (si terrà ~ 2-3 minuti).
- Aggiungere a riposo linfociti Fura2 caricati alla camera piatto microscopio montato con una pipetta 20 ml.
- Attivare l'imaging in campo chiaro. Selezionare il percorso della luce per gli oculari. Utilizzare la manopola di messa a fuoco di massima per portare obiettivo in contatto con la parte inferiore del piatto miscoscope. Utilizzare l'oculare e la manopola fuoco fine di concentrarsi sulle cellule T regolati alla parte inferiore del piatto.
- Utilizzare i controlli XY per selezionare un campo contenente almeno 10 celle. Evitaresovraffollati campi e grumi di cellule come queste creano artefatti di imaging.
- Passare da campo chiaro a fluorescente sorgente luminosa. Passare dalle immagini oculare CCD. Parametri di acquisizione impostati (per esempio, il tempo di esposizione, guadagno rivelatore e binning). Utilizzando il software di acquisire riposo Fura2-340 e immagini Fura2-380 (a partire con il tempo di esposizione identico per ciascuna, di solito nel range di ~ 200-1.000 msec).
- Utilizzare i metodi descritti al punto 5.4.1 per calcolare la Fura2-340 / Fura2-380 per ogni linfocita. Effettuare ripetute iterazioni regolare la Fura2-340 e tempi di esposizione Fura2-380, l'acquisizione di immagini e calcolando i rapporti fino a quando i valori medi sono vicini a 1.
- Impostare i tempi di esposizione per mem-YFP e DsRed.
- Sostituire il piatto microscopio utilizzato in fase 5.3.3.1 con un piatto contenente miscroscope trasfettate, attivato e Sag trattati (o non trattati; controllo) HLMVECs o HDMVECs da colture cellulari incubatore (Steps 4.5.1). Utilizzare una pipetta di trasferimento monouso per rimuovere rapidamente i media, lavare una volta con l'aggiunta di ~ 1 ml di pre-riscaldato Buffer-A. Aspirare e poi aggiungere 0,5 ml di tampone-A.
- Individuare i campi in cui sono presenti le cellule endoteliali transfectant positive luminose fluorescenti e sembrano in buona salute con giunzioni intercellulari ben formati.
- Regolare i parametri di acquisizione (ad esempio, il tempo di esposizione, guadagno rivelatore e binning) per mem-YFP e DsRed. Assicurarsi che medio di intensità di fluorescenza del segnale in ciascun canale cade tra il 25% e il 75% della gamma dinamica del rivelatore.
- Impostare i tempi di esposizione per Fura-2 imaging.
- Condurre esperimento live-cell imaging.
- Utilizzare il software automatizzato per iniziare l'acquisizione di immagini e catturare diversi intervalli di immagini per stabilire basale.
- Durante l'acquisizione applica ~ 5 microlitri di linfociti Fura-2-caricati concentrati (dal punto 5.2) al centro del campo dell'imaging piatto microscopio inserendo la punta di un piccolo volume (P-5o P-20) della pipetta nel supporto vicino al centro dell'obiettivo ed espulsione lentamente.
- Come linfociti stabilirsi nel campo dell'imaging, effettuare regolazioni fini della messa a fuoco per assicurare che la cellula-endotelio interfaccia delle cellule T (sinapsi immunologiche) sono mantenuti nel piano focale. Con il 40 e obiettivo 63x, ~ 10-20 cellule per campo sono ottimali. Se minor numero di cellule si osservano nel settore dell'imaging ripetere passo 5.3.4.2.
- Dopo l'intervallo di osservazione desiderata dell'esperimento è in competizione, continuare imaging e pipettare immediatamente ionomicina direttamente nel piatto miscoscope (usando la tecnica come in 5.3.4.2) ad una concentrazione finale di 2 mM di indurre / segnale segnalazione flusso calcio massima Fura-2 (ie , un mezzo di calibrazione, vedere l'analisi 5.4.).
- In alternativa 5.3.4.4after l'intervallo di osservazione desiderata dell'esperimento è in competizione, aspirare immediatamente il Buffer-A e sostituirlo con 0,5 ml di soluzione fissativa (3,7% di formaldeide in PBS) per 5 minuti aRT, seguita da risciacquo tre volte con PBS. Quindi procedere al punto 6.
- Analisi di Imaging Live-cell
NOTA: Dopo aver salvato i file acquisiti, possono essere analizzati direttamente o esportati per l'analisi da una vasta gamma di applicazioni software di analisi di immagine off-line. ImageJ è un pacchetto particolarmente prezioso, altamente versatile che è liberamente disponibile e compatibile con quasi tutti i pacchetti software di acquisizione. La progettazione degli esperimenti di imaging descritti nei passi 5.1-5.3 produrrà alta dinamica spaziale e temporale di risoluzione di interazione tra linfociti e cellule endoteliali APC in assenza e presenza di cognate SAG. Quasi analisi illimitate sono possibili quando si affrontano i dati di imaging di dinamica morfometrica / segnalazione cellulare. Gli obiettivi specifici descritti in questo particolare protocollo sono da quantificare in modo coordinato la migrazione dei linfociti e di segnalazione (ad esempio, la cinetica e livelli di flusso calcio intracellulare) insieme dinamicoAl cambiamenti nell'architettura sinapsi immunologica con particolare attenzione alla caratteristiche specifiche (ad esempio, ILPS / PODO-stampe). I seguenti sono esempi di serie separati e non standard (ad esempio, sviluppati appositamente per questi esperimenti / domande) analisi.- Misurare linfociti flusso di calcio
- Selezionare le iniziali Fura2-340 (340 nm EX-510 nm EM) immagini e Fura2-380 (380 nm EX-510 nm EM) che sono stati acquisiti prima aggiunta di linfociti. Utilizzare questi come immagini di sfondo e digitalmente li sottrarre tutte le immagini nella serie temporale per ciascun rispettivo canale.
- Per ogni punto di tempo creare un'immagine digitale del rapporto di background-sottratto Fura2-340 e le immagini di sfondo Fura2-380-sottratto (vale a dire, 340 nm EX-510 nm EM-sfondo / 380 nm EX-510 nm EM-sfondo).
- Selezionate lo strumento di disegno software appropriato. Fare clic sullo schermo per disegnare una regione circolare di interest (ROI) intorno ad ogni linfocita. Questi genereranno un valore del rapporto di pixel-media per ogni fotogramma linfociti e di tempo.
- Tracciare il rapporto calcolato in funzione del tempo utilizzando un'applicazione software (per es, programma di fogli). Calcolare flusso di calcio in media per l'esperimento sommando i valori del rapporto di tutti i linfociti per campo e dividendo questo valore per il numero totale di linfociti per ogni punto temporale
- Condurre lympocyte analisi di monitoraggio della migrazione.
- Analizzare cellule migrazione delle cellule T usando un programma adeguato di monitoraggio cella software (ad esempio, ImageJ) utilizzando il canale DIC da ogni video. Utilizzando ImageJ cellulare di monitoraggio Plugin, identificare in ogni fotogramma il baricentro di ogni cella manualmente facendo clic su di essa in ogni fotogramma progressiva.
- Utilizzare i risultanti coordinate XY di serie (ad esempio, tracce di percorsi di migrazione) per calcolare la velocità media (distanza totale migrato / Intervallo totale of per immagini) e la tortuosità (distanza totale migrazione / la distanza lineare end-to-end tra la posizione cella nella prima e ultima immagine).
- Cross-correlare questi parametri con flusso di calcio (5.4.1) dinamica su base cella per cella.
- Valutare la densità Podo-stampa / ILP all'interno IS
- Contare il numero totale di ILP formata in ogni cellula T intervalli è definita (ad esempio, 5 minuti dopo l'aggiunta di cellule T). Questi possono essere identificati discreti micron scala fluorescenti anelli membrana YFP che co-localizzano con occhiaie in citoplasmatica DsRed sul endoteliale APC in un sito di linfociti adesione.
- Cross-correlare le risultanti 'indici ILP' con flusso di calcio (5.4.1) dinamica su base cella per cella.
- Misurare il podo-stampa / vite ILP.
- Per ogni podo-stampa / ILP in un determinato IS calcolare i propri tempi di vita come l'ultimo punto momento in cui un ILP era visibile - il momento in cui tale punto podo-stampa / ILP fiapparso prima. Media ILP vite sia per IS.
- Cross-correlare tale durata con flusso di calcio (5.4.1) dinamica su base cella per cella.
- Valutare la relazione temporale tra la formazione iniziale e ILP flusso di calcio.
- Per ogni linfocita identificare il punto di tempo (telaio) in cui il calcio si alza prima di sopra del basale 19.
- Per ogni linfocita identificare il punto di tempo in cui appare la prima podo-stampa / ILP.
- Per ogni linfocita calcolare un 'offset' sottraendo il punto iniziale di tempo del flusso di calcio da quello della prima podo-stampa / ILP. Calcolare la media dei valori per tutti i linfociti in un campo. I valori positivi indicano la formazione ILP precede flusso di calcio, mentre i numeri negativi indicano il contrario.
NOTA: Questi esperimenti può essere facilmente eseguita sotto fisiologicamente rilevanti flusso laminare shear utilizzando parallelo parete camere di imaging flusso disponibili in commercio come descritto 19.
- Misurare linfociti flusso di calcio
Imaging 6. celle fissi e analisi
- Endpoint fisso cellule T-endoteliale è la formazione e colorazione
- Preparare le cellule T, come descritto al punto 1. Preparare cellule endoteliali (- / + SAG) come al punto 5.1 (trasfezione, passaggio 5.1.1 è opzionale). Prelevare un campione di linfociti in coltura e determinare la densità contando con un emocitometro (sezione 1).
- Trasferire un volume equivalente coltura linfociti ad almeno 3 x 10 5 linfociti / campione ad un tubo da 15 ml e centrifugare a 200 xg per 3 min. Rimuovere il surnatante e risospendere il pellet in linfociti Buffer-A (punto 5.2.1) la concentrazione di 6 x 10 5 linfociti / ml.
- Rimuovere le cellule endoteliali dal termostato coltura cellulare e (procedendo un campione alla volta) Mezzi successivamente aspirato e sostituirlo con 0,5 ml di sospensione di linfociti (3 x 10 5 linfociti / campione) e sostituire a 37 ° C incubator.
- Dopo opportuni tempi di incubazione, osservato in precedenza, media aspirare dai pozzetti e aggiungere sufficiente la soluzione fissativa (3,7% di formaldeide in PBS) per coprire completamente il campione (ad esempio, in 24 pozzetti ~ 300-500 ml). Incubare a temperatura ambiente per 5-10 minuti.
- Risciacquare campione 3 volte con PBS. Stain aggiungendo l'anticorpo primario per 60 minuti a temperatura ambiente (vedi elenco). Se l'anticorpo primario è diretto ad una proteina intracellulare, un passo permeabilizzazione addizionale deve essere eseguita aggiungendo una soluzione di permeabilizzazione sufficiente (0,01% TritonX-100 in PBS) per coprire completamente il campione per 1 min a RT. Risciacquare campione 3 volte con PBS. Aggiungere anticorpi secondari appropriate (in alcuni casi falloidina concomitanza fluorescente) per 60 minuti a RT.
- Montare il vetrino di vetro circolare su uno scivolo di imaging. Endpoint fisso cellule T-endoteliale è la formazione e colorazione. Per iniziare posto di imaging la diapositiva di immagini contenente campioni sul palco microscopio e selezionare l'olio 63Xobbiettivo. Applicare l'olio di immersione fresco.
- Utilizzare la modalità di imaging in campo chiaro e lenti oculari per individuare il piano focale. Passare a epifluorescenza e ispezionare campione tramite le lenti oculari. Selezionare un campo di interesse. Passare alla modalità di scansione laser.
- Utilizzando la modalità fast-scan e lavorare nel più ampio campo possibile regolare singolarmente potenza del laser e guadagno di ciascun canale per ottimizzare il segnale in modo tale che, idealmente, l'intensità del segnale specifico raggiunga almeno ~ 25% e non più del 75% della gamma dinamica dei rivelatori.
- Utilizzando controlli messa a fuoco manuale, scansione rapidamente attraverso l'asse Z e identificare i limiti superiore e inferiore di sezionamento (tipicamente tutte le informazioni devono essere contenuti in uno spessore di 15 micron) ~. Selezionare la sezione di spessore Z nella gamma di ~ 0,2-1,0 micron.
- Infine, zoom / ritagliare il campo di imaging per la regione di interesse e la scansione condotta. Oltre ad avere molto luminoso e fluorescenza specifica sisegnal nel campione, acquisendo alta risoluzione 3D imaging richiede iterazioni di scansione e le regolazioni dei parametri di acquisizione. L'obiettivo è quello di ottenere xy massima risoluzione e asse z, senza apprezzabile fotometabolismo.
- Qualitativamente analizzare la topologia di 3D topologia della IS conducendo ricostruzione digitale in 3D delle sezioni ottiche risultanti attraverso un'applicazione software (per es, Immagine J). Attraverso applicazioni simili quantitativamente valutare distribuzione segnale di fluorescenza (ad esempio, co-localizzazione e intensità di fluorescenza correlativa linea-scan analisi di Pearson e la visualizzazione ortogonale).
Risultati
Un nuovo approccio di imaging utilizzando cellule endoteliali e combinando i vantaggi risoluzione della planare bistrati lipidici modello con la complessità fisiologica e deformabilità professionale APC stato sviluppato (Figura 1). La Figura 2 fornisce esempi di migrazione tipica, flusso di calcio e dinamiche topologici osservato con questo approccio. In assenza di SAG in endotelio, SAG-specifici linfociti CD4 + Th1 rapidame...
Discussione
Nel complesso, questo protocollo descrive i metodi per indagare le cellule endoteliali come i) understudied APC fisiologici e ii) come un nuovo tipo di 'planare cellulare modello APC'. Per quanto riguarda il primo, è diventato sempre più apprezzato che APC non ematopoietiche periferiche (o 'stromali') svolgono, un ruolo non ridondanti critici (ad esempio, rispetto a ematopoietiche APC) nel modellare le risposte immunitarie adattive 16-18. Tra tali 'semi-professionale' APC, v...
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
We thank Dr. Peter T. Sage for his assistance in generating some of the representative images. This work was supported by an NIH R01 grant to C.V.C. (HL104006).
Materiali
| Name | Company | Catalog Number | Comments |
| BD Vacutainer stretch latex free tourniquet | BD Biosciences | 367203 | |
| BD alcohol swabs | BD Biosciences | 326895 | |
| BD Vacutainer Safety-Lok | BD Biosciences | 367861 | K2 EDTA |
| BD Vacutainer Push Button Blood Collection Set | BD Biosciences | 367335 | |
| RPMI-1640 | Sigma-Aldrich | R8758-1L | |
| Ficoll-Paque | Sigma-Aldrich | GE17-1440-02 | Bring to RT before use |
| FCS-Optima | Atlanta Biologics | s12450 | Heat inactivated |
| Penicillin-Streptomycin | Sigma-Aldrich | P4458-100ML | |
| Trypan blue | Sigma-Aldrich | T8154-20ML | |
| staphylococcal enterotoxin B | Toxin Technology | BT202RED | Stock solution 1mg/ml in PBS |
| toxic shock syndrome toxin 1 | Toxin Technology | TT606RED | Stock solution 1mg/ml in PBS |
| human IL-15 | R&D Systems | 247-IL-025 | Stock solution 50ug/ml in PBS |
| PBS | Life Technologies | 10010-049 | |
| Fibronectin | Life Technologies | 33016-015 | Stock solution 1mg/ml in H20 |
| HMVEC-d Ad-Dermal MV Endo Cells | Lonza | CC-2543 | Other Human Microvascular ECs can be used, i.e. HLMVECs |
| EGM-2 MV bullet kit | Lonza | CC-3202 | |
| Trypsin-EDTA | Sigma-Aldrich | T-4174 | Stock solution 10x, dilute in PBS |
| amaxa-HMVEC-L Nucleofector Kit | Lonza | vpb1003 | Required Kit for step 4 |
| IFN-g | Sigma-Aldrich | I3265 | Stock solution 1mg/ml in H20 |
| TNF-alpha 10ug, human | Life Technologies | PHC3015 | Stock solution 1mg/ml in H20 |
| phenol Red-free HBSS | Life Technologies | 14175-103 | |
| Hepes | Fisher Scientific | BP299-100 | |
| Calcium Chloride | Sigma-Aldrich | C1016-100G | Stock solution 1M in H20 |
| Magnesium chloride | Sigma-Aldrich | 208337 | Stock solution 1M in H20 |
| Human Serum albumin | Sigma-Aldrich | A6909-10ml | |
| Immersol 518 F fluorescence free Immersion oil | Fisher Scientific | 12-624-66A | |
| Fura-2 AM 20x50ug | Life Technologies | F1221 | Stock solution 1mM in DMSO |
| pEYFP-Mem (Mem-YFP) | Clontech | 6917-1 | |
| pDsRed-Monomer (Soluble Cytoplasmic DsRed) | Clontech | 632466 | |
| pDsRed-Monomer Membrane (Mem-DsRed) | Clontech | 632512 | |
| pEGFP-Actin | Clontech | 6116-1 | |
| Alexa Fluor 488 Phalloidin | Life Technologies | A12379 | |
| Formaldehyde solution 37% | Fisher Scientific | BP531-500 | Toxic, use fumehood |
| Triton X-100 | Sigma-Aldrich | X100-5ML | |
| Falcon 15mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-70C | |
| Falcon 50mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-49A | |
| Falcon Tissue Culture Treated Flasks T25 | Fisher Scientific | 10-126-9 | |
| Falcon Tissue Culture Treated Flasks T75 | Fisher Scientific | 13-680-65 | |
| Corning Cell Culture Treated T175 | Fisher Scientific | 10-126-61 | |
| Glass coverslips | Fisher Scientific | 12-545-85 | 12 mm diameter |
| Falcon Tissue Culture Plates 24-well | Fisher Scientific | 08-772-1 | |
| Delta-T plates | Bioptechs | 04200415B | |
| Wheaton Disposable Pasteur Pipets | Fisher Scientific | 13-678-8D | |
| 1.5 ml Eppendorf tube | Fisher Scientific | 05-402-25 | |
| ICAM1 mouse anti-human | BD Biosciences | 555509 | |
| HS1 mouse anti-human | BD Biosciences | 610541 | |
| Anti-Human CD11a (LFA-1alpha) Purified | ebioscience | BMS102 | |
| Anti-Human CD3 Alexa Fluor® 488 | ebioscience | 53-0037-41 | |
| Anti-MHC Class II antibody | Abcam | ab55152 | |
| Anti-Talin 1 antibody | Abcam | ab71333 | |
| Anti-PKC theta antibody | Abcam | ab109481 | |
| phosphotyrosine (4G10 Platinum) | Millipore | 50-171-463 | |
| Nucleofector II | Amaxa Biosystems | Required electroporator for step 4 | |
| Zeiss Axiovert | Carl Zeiss MicroImaging | ||
| Zeiss LSM510 | Carl Zeiss MicroImaging | ||
| Zeiss Axiovison Software | Carl Zeiss MicroImaging | ||
| NU-425 (Series 60) Biological Safety Cabinet | NuAIRE | Nu-425-600 | |
| Forma STRCYCLE 37 °C, 5% CO2 Cell culture Incubator | Fisher Scientific | 202370 | |
| Centrifuge 5810 | Eppendorf | EW-02570-02 | |
| Hemocytometer | Sigma-Aldrich | Z359629 | Bright-Line Hemocytometer |
| Isotemp Waterbath model 202 | Fisher Scientific | 15-462-2 |
Riferimenti
- von Andrian, U. H., Mackay, C. R. T-cell function and migration. Two sides of the same coin. N. Engl. J. Med. 343 (14), 1020-1034 (2000).
- Springer, T. A. Adhesion receptors of the immune system. Nature. 346 (6283), 425-434 (1990).
- Shaw, A. S., Dustin, M. L. Making the T cell receptor go the distance: a topological view of T cell activation. Immunity. 6 (4), 361-369 (1997).
- Monks, C. R., Freiberg, B. A., Kupfer, H., Sciaky, N., Kupfer, A. Three-dimensional segregation of supramolecular activation clusters in T cells. Nature. 395 (6697), 82-86 (1998).
- Delon, J., Stoll, S., Germain, R. N. Imaging of T-cell interactions with antigen presenting cells in culture and in intact lymphoid tissue. Immunol Rev. 189, 51-63 (2002).
- Brossard, C., et al. Multifocal structure of the T cell - dendritic cell synapse. Eur J Immunol. 35 (6), 1741-1753 (2005).
- Dustin, M. L. The cellular context of T cell signaling. Immunity. 30 (4), 482-492 (2009).
- Balagopalan, L., Sherman, E., Barr, V. A., Samelson, L. E. Imaging techniques for assaying lymphocyte activation in action. Nat Rev Immunol. 11 (1), 21-33 (2011).
- Cebecauer, M., Spitaler, M., Serge, A., Magee, A. I. Signalling complexes and clusters: functional advantages and methodological hurdles. J Cell Sci. 123, 309-320 (2010).
- Oddos, S., et al. High-speed high-resolution imaging of intercellular immune synapses using optical tweezers. Biophys J. 95 (10), L66-L68 (2008).
- Grakoui, A., et al. The immunological synapse: a molecular machine controlling T cell activation. Science. 285 (5425), 221-227 (1999).
- Bunnell, S. C., et al. T cell receptor ligation induces the formation of dynamically regulated signaling assemblies. J Cell Biol. 158 (7), 1263-1275 (2002).
- Yokosuka, T., et al. Newly generated T cell receptor microclusters initiate and sustain T cell activation by recruitment of Zap70 and SLP-76. Nat Immunol. 6 (12), 1253-1262 (2005).
- Seminario, M. C., Bunnell, S. C. Signal initiation in T-cell receptor microclusters. Immunol Rev. 221, 90-106 (2008).
- Dustin, M. L. Supported bilayers at the vanguard of immune cell activation studies. J Struct Biol. 168 (1), 152-160 (2009).
- Martinelli, R., Carman, C. V., Bradshaw, R. A., Stahl, P. Lymphocyte Endothelilal Interactions. Encyclopedia of Cell Biology. , (2015).
- Choi, J., Enis, D. R., Koh, K. P., Shiao, S. L., Pober, J. S. T lymphocyte-endothelial cell interactions. Annu Rev Immunol. 22, 683-709 (2004).
- Marelli-Berg, F. M., Jarmin, S. J. Antigen presentation by the endothelium: a green light for antigen-specific T cell trafficking?. Immunol Lett. 93 (2-3), 109-113 (2004).
- Sage, P. T., et al. Antigen recognition is facilitated by invadosome-like protrusions formed by memory/effector T cells. J Immunol. 188 (8), 3686-3699 (2012).
- Kumari, S., et al. Actin foci facilitate activation of the phospholipase C-gama in primary T lymphocytes via the WASP pathway . eLife. , (2015).
- Marelli-Berg, F. M., et al. Major histocompatibility complex class II-expressing endothelial cells induce allospecific nonresponsiveness in naive T cells. J Exp Med. 183 (4), 1603-1612 (1996).
- Ma, W., Pober, J. S. Human endothelial cells effectively costimulate cytokine production by, but not differentiation of, naive CD4+ T cells. J Immunol. 161 (5), 2158-2167 (1998).
- Perez, V. L., Henault, L., Lichtman, A. H. Endothelial antigen presentation: stimulation of previously activated but not naive TCR-transgenic mouse T cells. Cell Immunol. 189 (1), 31-40 (1998).
- Epperson, D. E., Pober, J. S. Antigen-presenting function of human endothelial cells. Direct activation of resting CD8 T cells. J Immunol. 153 (12), 5402-5412 (1994).
- Shiao, S. L., et al. Human effector memory CD4+ T cells directly recognize allogeneic endothelial cells in vitro and in vivo. J Immunol. 179 (7), 4397-4404 (2007).
- Marelli-Berg, F. M., Okkenhaug, K., Mirenda, V. A two-signal model for T cell trafficking. Trends Immunol. 28 (6), 267-273 (2007).
- Carman, C. V., et al. Transcellular diapedesis is initiated by invasive podosomes. Immunity. 26 (6), 784-797 (2007).
- Carman, C. V. Mechanisms for transcellular diapedesis: probing and pathfinding by 'invadosome-like protrusions'. J Cell Sci. 122 ((Pt 17)), 3025-3035 (2009).
- Dustin, M. L., Tseng, S. Y., Varma, R., Campi, G. T. T cell-dendritic cell immunological synapses. Curr Opin Immunol. 18 (4), 512-516 (2006).
- Saito, T., Yokosuka, T. Immunological synapse and microclusters: the site for recognition and activation of T cells. Curr Opin Immunol. 18 (3), 305-313 (2006).
- Gomez, T. S., et al. HS1 functions as an essential actin-regulatory adaptor protein at the immune synapse. Immunity. 24 (6), 741-752 (2006).
- Burbach, B. J., Medeiros, R. B., Mueller, K. L., Shimizu, Y. T-cell receptor signaling to integrins. Immunol Rev. 218, 65-81 (2007).
- Vicente-Manzanares, M., Sanchez-Madrid, F. Role of the cytoskeleton during leukocyte responses. Nat Rev Immunol. 4 (2), 110-122 (2004).
- Ma, Z., Janmey, P. A., Finkel, T. H. The receptor deformation model of TCR triggering. Faseb J. 22 (4), 1002-1008 (2008).
- Ma, Z., Sharp, K. A., Janmey, P. A., Finkel, T. H. Surface-anchored monomeric agonist pMHCs alone trigger TCR with high sensitivity. PLoS Biol. 6 (2), e43 (2008).
- Groves, J. T. Bending mechanics and molecular organization in biological membranes. Annu Rev Phys Chem. 58, 697-717 (2007).
- Xu, C., et al. Regulation of T cell receptor activation by dynamic membrane binding of the CD3epsilon cytoplasmic tyrosine-based motif. Cell. 135 (4), 702-713 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon