Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Evaluación In Vitro de la Transformación Oncogénica en Células Epiteliales Mamarias Humanas
En este artículo
Resumen
Este protocolo proporciona herramientas experimentales in vitro para evaluar la transformación de las células mamarias humanas. Se describen los pasos detallados para el seguimiento de la tasa de proliferación celular, la capacidad de crecimiento independiente del anclaje y la distribución de linajes celulares en cultivos 3D con matriz de membrana de sótano.
Resumen
La tumorigenesis es un proceso de varios pasos en el que las células adquieren capacidades que permiten su crecimiento, supervivencia y diseminación en condiciones hostiles. Diferentes pruebas buscan identificar y cuantificar estas señas de identidad de las células cancerosas; sin embargo, a menudo se centran en un solo aspecto de la transformación celular y, de hecho, se requieren múltiples pruebas para su caracterización adecuada. El propósito de este trabajo es proporcionar a los investigadores un conjunto de herramientas para evaluar la transformación celular in vitro desde una perspectiva amplia, lo que permite sacar conclusiones sólidas.
Una activación de señalización proliferativa sostenida es la característica principal de los tejidos tumorales y puede ser fácilmente monitoreada en condiciones in vitro calculando el número de duplicaciones de población logradas con el tiempo. Además, el crecimiento de las células en cultivos 3D permite su interacción con las células circundantes, que se asemeja a lo que ocurre in vivo. Esto permite la evaluación de la agregación celular y, junto con el etiquetado inmunofluorescente de marcadores celulares distintivos, obtener información sobre otra característica relevante de la transformación tumoral: la pérdida de una organización adecuada. Otra característica notable de las células transformadas es su capacidad para crecer sin un apego a otras células y a la matriz extracelular, que se puede evaluar con el ensayo de anclaje.
Se proporcionan procedimientos experimentales detallados para evaluar la tasa de crecimiento celular, realizar el etiquetado inmunofluorescente de marcadores de linaje celular en cultivos 3D y para probar el crecimiento celular independiente del anclaje en el agar blando. Estas metodologías están optimizadas para las células epiteliales primarias de mama (BPEC) debido a su relevancia en el cáncer de mama; sin embargo, los procedimientos se pueden aplicar a otros tipos de celda después de algunos ajustes.
Introducción
Se requieren varios eventos sucesivos para el desarrollo de neoplasias. En 2011, Hanahan y Weinberg describieron 10 capacidades que permiten transformar el crecimiento, la supervivencia y la difusión de las células: las llamadas "Marcas del Cáncer"1. La metodología descrita aquí compila tres herramientas diferentes para evaluar la transformación celular in vitro centrándose en algunas de las características distintivas de las células tumorales. Estas técnicas evalúan la tasa de proliferación celular, el comportamiento de las células cuando se cultivan en 3D y su capacidad para formar colonias con independencia de anclaje.
Los modelos celulares son cruciales para probar la hipótesis in vitro. Se han desarrollado diferentes enfoques para generar modelos experimentales de transformación celular para el estudio del cáncer2,3,4. Dado que el cáncer de mama es el cáncer más común entre las mujeres en todo el mundo y es responsable de aproximadamente el 15% de las muertes por cáncer entre las mujeres5,proporcionar modelos celulares adecuados de células epiteliales mamarias es de suma importancia para la investigación posterior. En este artículo, hemos ilustrado el potencial de tres técnicas para evaluar la transformación celular utilizando un modelo experimental de transformación de células epiteliales primarias de senos (BPEC) descrita inicialmente por Ince y sus colegas en 20076 y posteriormente implementada en nuestro laboratorio7. Este modelo experimental se basa en la alteración secuencial de tres genes dirigidos (SV40 Grandes T y antígenos t pequeños aquí conocidos como Ttag, hTERTy HRAS)al genoma de BPEC no transformados. Además, el método utilizado para la derivación de BPEC favorece el mantenimiento de células epiteliales mamarias con marcadores luminales o mioepteliales, dando como resultado un cultivo celular heterogéneo que conserva algunos de los rasgos fisiológicos de la glándula mamaria.
En la glándula mamaria, las células epiteliales mamarias luminales, que son responsables de la producción de leche, se encuentran cerca del lumen, mientras que las células mioepteliales se eliminan alrededor de las células luminales y se encargan de los movimientos de contracción que conducen la leche al pezón. La pérdida de una organización adecuada entre estos linajes celulares es una característica de la transformación tumoral8 que se puede evaluar in vitro después de la detección inmunofluorescente de marcadores de linaje distintivos en cultivos celulares 3D. Otra característica importante de las células tumorales es su capacidad para crecer sin un apego a otras células y a la matriz extracelular1. Cuando las células sanas se ven obligadas a crecer en suspensión, se activan mecanismos como los anoikis u2012 un tipo de muerte celular inducida en respuesta al desprendimiento de la matriz extracelular de la matriz extracelular9. La evasión de la muerte celular es una de las señas de identidad distintivas del cáncer y, por lo tanto, las células transformadas son capaces de inactivar los anoikis y sobrevivir de una manera independiente del ancla. Esta capacidad se puede evaluar in vitro con el ensayo independiente del anclaje utilizando agar blando. Además, una característica inherente de los tejidos tumorales es su capacidad de señalización proliferativa sostenida, que puede controlarse fácilmente en condiciones in vitro midiendo el aumento del número de células a lo largo del tiempo, no sólo en los ensayos de suspensión, sino también mediante el seguimiento de la tasa de crecimiento de los cultivos monocapa adherentes.
A pesar del mejor modelo para probar el potencial tumorigénico es la inoculación de células tumorales en modelos murinos y la evaluación del desarrollo tumoral in situ, es importante minimizar el número de animales empleados en procedimientos experimentales tanto como sea posible. Por lo tanto, tener pruebas adecuadas para evaluar la transformación in vitro es una prioridad. Aquí, proporcionamos un conjunto de herramientas para evaluar el potencial tumorigénico de las células epiteliales mamarias parcialmente y totalmente transformadas que se pueden implementar fácilmente en la mayoría de los laboratorios que trabajan con modelos de transformación celular.
Protocolo
Las muestras humanas utilizadas en los siguientes experimentos se obtuvieron a partir de mamoplastias de reducción llevadas a cabo en la Clínica Pilar Sant Jordi (Barcelona) bajo el consentimiento de procedimiento estándar. Todos los procedimientos se realizan en un Gabinete de Seguridad Biológica de Clase II a menos que se indique lo contrario.
1. Cultivo in vitro de células epiteliales mamarias humanas y acumulación de la trama de la curva de crecimiento
-
Cultivo in vitro de células epiteliales primarias mamarias (BPEC): transaprovisionamiento celular
NOTA: Para la derivación de BPEC y el cultivo celular siga las instrucciones descritas por Ince et al., 20076.- Preparación media.
- Suplemento WIT medio definido basal con P o T suplementos, proporcionado por el fabricante, dependiendo de si primario o transformado BPECs se cultivan.
- Añadir toxina de cólera al medio WIT suplementado a una concentración final de 100 ng/ml para primario o 25 ng/ml para BPEC transformados.
ADVERTENCIA: La toxina del cólera es mortal si se ingiere. Utilice equipo de protección personal. Evite su liberación al medio ambiente.
- Mantenimiento y passaging de cultivo celular.
NOTA: Para los siguientes pasos, tenga en cuenta que las células están creciendo en un matraz T25. No obstante, los volúmenes se pueden adaptar a otros formatos de cultivo celular manteniendo la proporcionalidad en términos de superficie.- Compruebe la confluencia celular todos los días. Cuando el cultivo es 90% confluente, realice el passaging de celdas.
- Adquirir 1x PBS, 3x tripsina, medio y un tubo cónico de 15 ml que contenga 2 ml de suero bovino fetal (FBS) para cada matraz.
- Retire el medio del matraz y guárdalo en el tubo cónico de 15 ml que contiene FBS.
- Enjuague las células con 1x PBS.
- Separe las células de la superficie añadiendo 1 ml de 3x tripsina. Incubar durante 5 min a 37oC.
- Compruebe si las celdas se han separado. Aplique agitación vigorosa si las células no están completamente desprendidas.
- Inactivar la trippsina añadiendo el medio reservado complementado con FBS.
- Cosecha la suspensión celular y colóquela en el tubo cónico de 15 ml.
- Centrifugar a 500 x g durante 5 min, eliminar el sobrenadante, y resuspender las células peletadas moviendo la parte inferior del tubo con un dedo.
- Añadir 1–2 ml de medios frescos al pellet y medir la concentración celular utilizando un contador celular automático o un hemociclomekil. Estos datos se utilizarán más adelante para calcular las duplicaciones de la población y dibujar la curva de crecimiento. Semilla 12.000 células/cm2 (por ejemplo, 300.000 células para un matraz T25) en matraces de superficie de cultivo celular modificados (ver Tabla de materiales).
NOTA: Diluir la solución de suspensión celular si la concentración es demasiado alta para garantizar una cuantificación adecuada. - Añadir un volumen medio a un volumen final de 5 ml e incubar las células a 37oC y 5% de CO2 atmósfera.
- Reemplace el medio de cultivo celular cada 48 h.
- Preparación media.
-
Cálculo de duplicación de población y visualización de datos
- Utilizando los datos del recuento de celdas obtenidos en el paso 1.1.2.10, aplique la siguiente fórmula para obtener los valores de duplicación de población acumulada (PD):
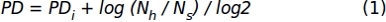
Donde, PDi denota el número de duplicaciones de población alcanzadas por las células hasta la subcultura anterior (se refiere a los datos de la P.I. acumulados en la subcultura anterior), Nh es el número de células cosechadas, y Ns es el número de células de semilla. - Representar datos para un intervalo de tiempo específico mediante un gráfico XY donde se representan el número de días en referencia cultural (ejex)y el PD acumulado(eje y).
- Obtenga la línea de mejor ajuste y la ecuación de ajuste:

NOTA: Un aumento de la pendiente (b) significa un aumento de la tasa de proliferación.
- Utilizando los datos del recuento de celdas obtenidos en el paso 1.1.2.10, aplique la siguiente fórmula para obtener los valores de duplicación de población acumulada (PD):
2. Cultivo tridimensional (3D) en matriz de membrana base y detección de proteínas inmunofluorescentes
- Cultivo 3D en matriz de membrana de sótano
NOTA: Este protocolo ha sido adaptado de Debnath et al., 200310 y está optimizado para 24 placas de pozos (ver Tabla de Materiales).- Preparar el material un día antes del experimento: matriz de membrana del sótano pre-enfriado durante la noche a 4 oC y dejar que las puntas de pipeta, los tubos de microcentrífuga y las placas de pozo se enfríen en el congelador.
NOTA: La matriz debe mantenerse a -20 oC para un almacenamiento a largo plazo. Haga alícuotas para evitar múltiples ciclos de congelación y descongelación. - El día del experimento, coloque material preenfriado sobre hielo.
- Enjuagar los pozos con 1 PBS estériles en frío para reducir la tensión superficial.
- Cubra la parte inferior de cada pozo con 100 ml de matriz de membrana del sótano.
NOTA: Dispensar la matriz lentamente y extenderla por todo el pozo; es crucial evitar la formación de burbujas en la capa inferior para evitar el crecimiento del cultivo celular monocapa. - Colocar la placa en la incubadora, a 37 oC, para dejar que la capa de matriz se solidifique.
NOTA: Por lo general, se tarda unos 20 minutos en solidificarse. - Mientras tanto, tripsina las células como se explicó anteriormente en el paso 1.1. Centrifugar las células a 500 x g durante 5 min y resuspend en medio. Preparar una suspensión de 400.000 células/ml y desagregar suavemente cualquier grupo de células pipeteando.
- Preparar el medio con matriz de membrana de sótano del 8% y mezclar 1:1 (v/v) con suspensión celular para obtener una solución de 200.000 células/ml en matriz del 4%.
NOTA: Calcule la cantidad de medio necesaria para evitar residuos innecesarios de matriz. - Colocar 500 l de suspensión celular en solución de matriz en la parte superior de la capa de matriz ya solidificada para sembrar una cantidad total de 100.000 células en medio con 4% matriz de membrana de sótano.
- Incubar las células a 37oC durante unos minutos y luego, añadir 500 l del medio con matriz de membrana de sótano al 4%. Incubar las células a 37oC en una incubadora con 5% deCO2 durante 14 días. Las células de semillas se agruparán y proliferarán para originar las estructuras similares a los acini.
NOTA: La motilidad celular y la agregación se pueden supervisar mediante lapso de tiempo durante el proceso de formación 3D. Utilice software de análisis de imágenes (por ejemplo, Fiji/ImageJ o Imaris) para evaluar estos eventos. El número y el tamaño del acini dependen del proceso de agregación y de la tasa de proliferación y pueden variar entre los tipos de células. Ajuste la concentración de la matriz de membrana del sótano y las células sembradas para obtener las estructuras 3D deseadas. - Añadir 500 l del medio con matriz de membrana de sótano del 4% 2-3 veces por semana.
NOTA: Evite la perturbación de las capas que llevan la placa suavemente durante la manipulación. - Si se desea, el número y el tamaño de acini se pueden medir durante el período de cultivo. Para ello, tome fotografías aleatorias en diferentes momentos después de la siembra utilizando un contraste de fase o un microscopio invertido DIC. Utilice el software de análisis de imágenes para medir el diámetro de 100 a 200 estructuras 3D.
- Preparar el material un día antes del experimento: matriz de membrana del sótano pre-enfriado durante la noche a 4 oC y dejar que las puntas de pipeta, los tubos de microcentrífuga y las placas de pozo se enfríen en el congelador.
- Immunostaining
NOTA: No se requieren condiciones estériles durante esta parte del protocolo.- Retire el medio de cultivo.
- Rasgue la matriz de membrana del sótano con una punta de pipeta p200 con el extremo cortado. Colocar 50 l de matriz desagregada en la parte superior de un portaobjetos de vidrio y frotarla en un área de 1–2 cm2.
- Deje que la muestra se seque completamente a temperatura ambiente o utilice una placa calefactora a 37 oC para acelerar el proceso. Corrija las muestras con metanol:acetona (1:1, v/v) a -20 oC durante 30 min.
NOTA: Se borrará la señal fluorescente de marcadores anteriores, como la de las proteínas fluorescentes expresadas por las células.
ADVERTENCIA: El metanol es inflamable, tóxico si se inhala, se traga o en caso de que entre en contacto con la piel. Use equipo de protección personal y trabaje dentro de una campana de humos. - Deseche la solución de fijación y elimine el exceso, si existe, reclinando la diapositiva sobre el papel de filtro.
NOTA: El protocolo se puede pausar aquí. Una vez secos, los portaobjetos se pueden almacenar a -20 oC durante varios meses. - Muestras de bloques epitopos con 5% de suero de cabra normal y 0.1% tritón-X-100 en 1x PBS (solución de bloqueo) durante 2 h a temperatura ambiente.
- Mientras tanto, preparar las soluciones de trabajo de anticuerpos diluyendo los anticuerpos primarios o secundarios a la concentración deseada en la solución de bloqueo.
NOTA: La concentración de anticuerpos debe ajustarse con precisión en función del tipo de célula y de la referencia del anticuerpo. Como guía, para identificar células de linajes luminales y mioepitheliales en BPEC, se pueden utilizar anticuerpos primarios anti-Cytokeratin 14 y anti-Claudin-IV (ver Tabla de Materiales). La concentración recomendada de la solución de trabajo es de 1:100 para estos anticuerpos primarios y 1:500 para los anticuerpos secundarios antiratón y anti-conejo (ver Tabla de materiales). - Añadir 30 l de solución de trabajo de anticuerpos primarios y cubrirla con una tira de película de envoltura de laboratorio para evitar la evaporación. Incubar durante la noche a 4oC en una cámara húmeda.
- Lavar tres veces con 1x PBS durante 1 h cada uno.
- Repita el paso 2.2.7 para los anticuerpos secundarios. La incubación debe realizarse en la oscuridad.
- Lavar con 1x PBS durante 2 h.
NOTA: Ajuste la concentración de anticuerpos, el tiempo de incubación y la dureza del lavado para mejorar la relación señal/ruido para muestras específicas. - Retire el PBS restante y, una vez seco, contracargo con DAPI a 0,25 g/ml diluido en medio de montaje antifada. Cubra las diapositivas con un cubreobjetos dejándolo asentar sin aplicar presión. Sellar con esmalte de uñas.
NOTA: Las muestras se pueden almacenar a 4 oC durante varias semanas. Para un almacenamiento a largo plazo, manténgalos a -20 oC. - Analizar la distribución de la señal fluorescente para cada acinus utilizando un microscopio confocal.
NOTA: La configuración del microscopio confocal debe determinarse con precisión en función del equipo utilizado y de los anticuerpos aplicados a la muestra. Como guía, con el equipo y los reactivos detallados en la Tabla de Materiales,utilice un objetivo de 40x y los siguientes ajustes de láser y detector: para el uso DAPI excitación con un láser 405 (3%-5%), detección con detector PMT (800V, Offset: -9) y una banda espectral de 410 nm a 500 nm; para A488 (Claudin-IV) utilizan excitación con un láser 488 (7%-10%), detección con detector de PMT (800V, Desplazamiento: -20) y una banda espectral de 490 nm a 550 nm; y para Cy3 (Cytokeratin 14) utilizan excitación con un láser 555 (2%-10%), detección con un detector PMT (800V, Offset: -35) y un espectral de 560 nm a 600 nm.
3. Ensayo independiente de anclaje, tinción MTT y cuantificación automática de colonias
- Ensayo independiente de anclaje: agar y revestimiento de suspensión celular
NOTA: El protocolo ha sido adaptado de Borowicz et al., 201411 para realizar experimentos en BPECs.- Preparar una solución de agar al 1,2% diluida en agua ultrapura en una botella estéril. Autoclave la solución y mantenla a 42oC durante el experimento. La solución de agar se puede almacenar a 4 oC; cuando sea necesario, caliente la solución de agar hasta que esté de nuevo líquida.
ADVERTENCIA: Use guantes resistentes al calor para evitar quemaduras después del autoclave.
NOTA: A partir de ahora, deben mantenerse las condiciones estériles. - Prepare una solución de agar al 0,6% mezclando un medio precalegado completo 1:1 (v/v) con una solución de agar al 1,2%. Mantener a 42 oC para evitar la solidificación prematura.
NOTA: El medio se puede complementar previamente para obtener una solución completa de 0,6% de agar + medio una vez mezclada. - Cubrir la parte inferior de un pozo de 35 mm con 1,5 ml de 0,6% de agar en solución media y dejar que se solidifique a temperatura ambiente. Asegúrese de que la parte inferior de la placa está completamente cubierta antes de la solidificación del agar, de lo contrario, las células pueden adherirse a la placa y crecer en monocapa.
NOTA: Se pueden utilizar placas de superficie adherentes y no adherentes. - Mientras tanto, tripsinar las células y, una vez centrifugadas y resuspendidas en medio, preparar una solución de 50.000 células/ml y desagregar suavemente cualquier grupo celular pipeteando repetidamente.
- Preparar una suspensión de 0,3% de agar + célula en el medio a una concentración final de 25.000 células/ml.
NOTA: La concentración óptima de células puede diferir entre los tipos de células. Pruebe diferentes concentraciones hasta que se forme colonias individualizadas.- Coloque un filtro de colador de 40 m encima de un tubo estéril de 50 ml y filtre la solución de 50.000 células/ml dejando que caiga en la parte inferior del tubo.
- Retire el filtro del tubo estéril de 50 ml, incline el tubo que contiene la célula hasta un ángulo de 45o y deje caer el mismo volumen de 0,6% de agar + solución mediana vertiente a través de la pared interna del tubo. Esto permitirá que la solución de agar se enfríe lo suficiente para no dañar las células y evitar su solidificación prematura.
- Homogeneizar la mezcla y depositar 1 ml de 0,3% de agar + suspensión celular en el medio (que contiene 25.000 células) en la parte superior de la capa de agar inferior previamente solidificada.
- Visualice las células sembradas utilizando un microscopio invertido para asegurarse de que las células están individualizadas. De lo contrario, el experimento debe repetirse.
- Espere hasta que la capa de agar esté completamente solidificada, luego agregue cuidadosamente 1 ml del medio fresco en la parte superior sin alterar las delicadas capas de agar debajo.
- Incubar las células a 37oC y 5% deCO2 en una incubadora durante 3 semanas.
NOTA: El tiempo necesario para la formación de colonias puede variar entre diferentes tipos de células, pero por lo general 3 semanas son suficientes. - Cambie el medio dos veces por semana. Para ello, incline suavemente la placa hacia usted, aspire el medio en la esquina inferior y agregue 1 ml de medio fresco.
NOTA: Evite tocar las capas de agar, ya que se separan fácilmente de la placa.
- Preparar una solución de agar al 1,2% diluida en agua ultrapura en una botella estéril. Autoclave la solución y mantenla a 42oC durante el experimento. La solución de agar se puede almacenar a 4 oC; cuando sea necesario, caliente la solución de agar hasta que esté de nuevo líquida.
- Tinción MTT
- Preparar la solución en stock de Bromuro de Tetrazolio Azul (MTT) de Thiazolyl a 6 mg/ml en agua ultrapura en una botella estéril y solución de filtro utilizando filtros de 0,2 m. Esta solución MTT se puede almacenar hasta 6 meses a -20 oC.
ADVERTENCIA: La MTT puede causar irritación y se sospecha que causa defectos genéticos. Use gafas de seguridad, guantes y un filtro respiratorio.
NOTA: Evite ciclos repetidos de congelación y descongelación. - Preparar una solución de trabajo de MTT a 1 mg/ml diluyendo la solución de stock con agua ultrapura estéril.
- Una vez concluido el período de formación de la colonia, retire el medio de la placa y añada 1 ml de 1 mg/ml de MTT a cada pozo.
- Incubar durante 24 h en la incubadora. Retire la solución MTT a aspirando suavemente. Las placas se pueden almacenar a 4 oC durante varias semanas.
NOTA: Evite la exposición a la luz para evitar la formación de cristales no específicos.
- Preparar la solución en stock de Bromuro de Tetrazolio Azul (MTT) de Thiazolyl a 6 mg/ml en agua ultrapura en una botella estéril y solución de filtro utilizando filtros de 0,2 m. Esta solución MTT se puede almacenar hasta 6 meses a -20 oC.
- Cuantificación de colonias
- Obtenga imágenes de cada placa con un microscopio invertido. Ajustar el aumento con el fin de adquirir el campo de visión máximo con menos imágenes y todavía ser capaz de detectar pequeñas colonias (generalmente 4x o 10x objetivos).
NOTA: Asegúrese de que las imágenes presenten un fondo homogéneo. No se requiere contraste de fase ni contraste de interferencia diferencial, ya que no se cuantificarán las colonias no manchadas. - Cargue imágenes en el software ImageJ/Fiji12 para contar el número de colonias y el área de cada colonia MTT positiva.
NOTA: Se proporciona un script para la cuantificación automática como archivo suplementario. Para ejecutar el código, péguelo en el editor de macros (Plugins | Nueva | Macro) y siga las instrucciones.- Obtenga una máscara binaria mediante el umbral de la imagen original(imagen | Ajustar | Umbral) para obtener colonias bien delimitadas (Figura 1).
NOTA: Las imágenes de 8 o 16 bits suelen ser necesarias para realizar este paso. Se recomienda el método "Umbral mínimo". - Ejecute Extended Particle Analyzer desde el plugin Biovoxxel13 (Plugins | | BioVoxxel Analizador de partículas ampliado) para identificar colonias positivas MTT (Figura 2). Condiciones de guía iniciales: Tamaño (m2) a 250–Infinity; Solidez: 0,75–1,00.
- Obtenga una máscara binaria mediante el umbral de la imagen original(imagen | Ajustar | Umbral) para obtener colonias bien delimitadas (Figura 1).
- Estimar el diámetro medio (D) de cada valor de área de colonia (A) de acuerdo con la fórmula:

- Filtrar los resultados excluyendo las colonias proliferativas bajas (por ejemplo, de bajo diámetro).
- Elija un número mínimo de divisiones por semana (m) que se considerarán (por ejemplo, 1).
- Estimar el radio de una colonia (R) con n celdas de acuerdo con la siguiente fórmula:

Donde, r es el radio promedio de las células individuales en suspensión, n es el número de células que forman una colonia que sufrió m divisiones cada semana durante las semanas en el cultivo. En crecimiento exponencial: n a 2(m*w). La eficiencia del embalaje es la de embalaje. Tenga en cuenta que, en el movimiento aleatorio, la eficiencia del embalaje es de 0,64 euros y la fracción de embalaje más densa posible para esferas idénticas es 0,7414. - Deseche todas las colonias que presenten un diámetro inferior a 2R, ya que sus células no han alcanzado el número mínimo de divisiones consideradas en el paso 3.3.4.1.
- Obtenga imágenes de cada placa con un microscopio invertido. Ajustar el aumento con el fin de adquirir el campo de visión máximo con menos imágenes y todavía ser capaz de detectar pequeñas colonias (generalmente 4x o 10x objetivos).
Resultados
Se eligió un modelo experimental de transformación celular con la introducción de tres elementos genéticos en BPEC para generar resultados representativos de la transformación oncogénica6,7 (Figura 3). Los BCE no transformados (N) se derivaron del tejido mamario libre de enfermedades descrito por Ince y sus colegas6 y se cultivaron siguiendo el protocolo indicado aquí. Después...
Discusión
Los protocolos experimentales descritos en este documento proporcionan herramientas útiles para evaluar la transformación oncogénica de las células cultivadas in vitro. Cada técnica evalúa aspectos específicos del proceso de transformación y, por lo tanto, se debe prestar especial atención al extraer conclusiones de un solo análisis. La acumulación de curvas de crecimiento es un enfoque que exige información ya disponible al cultivar células para otros fines. Esto hace que esta técnica sea más barata y fá...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
El laboratorio AG está financiado por el Consejo Español de Seguridad Nuclear. T.A. y A.G. son miembros de un grupo de investigación reconocido por la Generalitat de Catalunya (2017-SGR-503). MT tiene un contrato financiado por la Fundación Científica Asociación Española Contra el Cáncer. El contrato de G.F. es financiado por una subvención de cellex Foundation.
Materiales
| Name | Company | Catalog Number | Comments |
| 1 ml Serological Pipettes | Labclinics | PLC91001 | |
| 1.5 ml Eppendorfs | Thermo Fisher Scientific | 3451 | Dark eppendorfs are preferred for MTT long-term storage |
| 10 μl Pipette tips w/o filter | Biologix | 20-0010 | |
| 100 ml glass bottle | With cap, autoclavable | ||
| 1000 μl Pipette tips w/ filter | Labclinics | LAB1000ULFNL | |
| 1000 μl Pipette tips w/o filter | Biologix | 20-1000 | |
| 15 ml Conical tubes | VWR | 525-0400 | |
| 2 ml Serological Pipettes | Labclinics | PLC91002 | |
| 200 μl Pipette tips w/ filter | Labclinics | FTR200-96 | |
| 5 ml Serological Pipettes | Labclinics | PLC91005 | |
| 50 ml Conical Tubes | VWR | 525-0304 | |
| Acetone | PanReac AppliChem | 211007 | Used for 3D structure fixation prior to immunofluorescent labelling |
| Agar | Sigma-Aldrich | A1296 | Used for anchorage assay |
| Anti-Claudin 4 antibody | Abcam | 15104, RRID:AB_301650 | Working dilution 1:100, host: rabbit |
| Anti-Cytokeratin 14 [RCK107] antibody | Abcam | 9220, RRID:AB_307087 | Working dilution 1:100, host: mouse |
| Anti-mouse Cyanine Cy3 antibody | Jackson ImmunoResearch Inc. | 115-165-146, RRID:AB_2338690 | Working dilution 1:500, host: goat |
| Anti-rabbit Alexa Fluor 488 antibody | Thermo Fisher Scientific | A-11034, RRID:AB_2576217 | Working dilution 1:500, host: goat |
| Autoclave | |||
| BioVoxxel Toolbox | RRID:SCR_015825 | ||
| Cell culture 24-well Plate | Labclinics | PLC30024 | Used for 3D cultures in Matrigel. Flat Bottom |
| Cell culture 6-well Plate | Labclinics | PLC30006 | Used for anchorage assay |
| Cell incubator (37 ºC and 5 % CO2) | |||
| Cell Strainers | Fisherbrand | 11587522 | Mesh size: 40 μm |
| CellSense software | Olympus | Used to image acquisition | |
| Centrifuge | |||
| Cholera Toxin from Vibrio cholerae | Sigma-Aldrich | C8052 | Used to supplement cell culture medium |
| Class II Biological Safety Cabinet | Herasafe | HAEREUS HS12 | |
| Confocal inverted Microscope | Leica | TCS SP5 | |
| Cover glasses | Witeg Labortechnik GmbH | 4600122 | 22 X 22 mm, thickness 0.13 - 0.17 mm |
| DAPI | 2-(4-amidinophenyl)-1H -indole-6-carboxamidine | ||
| Fetal Bovine Serum | Biowest | S1810 | Used to inactivate trypsine action |
| Fiji software (ImageJ) | National Institutes of Health | RRID:SCR_002285 | Free download, no license needed |
| Glass Pasteur Pipettes | |||
| Glass slides | Fisherbrand | 11844782 | |
| Goat Serum | Biowest | S2000 | Used for immunofluorescence of 3D structures |
| Heat-Resistant Gloves | Used for agar manipulation after autoclave | ||
| Heater bath (37 ºC) | Used to temper solutions prior to cell subculture | ||
| Heater bath (42 ºC) | Used to keep agar warm | ||
| Heating plate | Used for Matrigel dehydration | ||
| Humid chamber | Used for the incubation of antibodies during immunofluorescence | ||
| Ice | Used during Matrigel manipulation | ||
| Ice-box | |||
| Inverted Optic Microscope | Olympus | IX71 | |
| Matrigel Matrix | Becton Dickinson | 354234 | Store at -20 ºC and keep cold when in use. Referred to as basement membrane matrix |
| Methanol | PanReac AppliChem | 131091 | Used for 3D structure fixation prior to immunofluorescent labelling |
| Micropipette | p1000, p200 and p10 | ||
| Microsoft Office Excel | Microsoft | RRID:SCR_016137 | Used to calculate population doubling and to obtain growth rate equation |
| MilliQ water | Referred to as ultrapure water | ||
| Nail Polish | Used to seal samples after mounting | ||
| Parafilm M | Bemis | PM-999 | Used to cover antibody solution during incubation |
| PBS pH 7.4 (w/o calcium & magnesium) | Gibco | 10010-056 | Sterile. Used for cell subculture |
| PBS tablets | Sigma-Aldrich | P4417 | Dilute in milliQ water. No sterility required. Used for immunofluorescence |
| Pipette Aid | |||
| Primaria T25 flasks | Corning | 353808 | Used for BPEC culture |
| Scepter Automated Cell Counter | Millipore | PHCC20060 | Alternatively, use an haemocytometer |
| Scissors | Used to cut pipette tips and parafilm | ||
| Sterile filters 0.22 μm | Millipore | SLGP033RS | Used to filter MTT solution |
| Thiazolyl Blue Tetrazolium Bromide (MTT) | Sigma-Aldrich | M2128 | Store at -20 ºC |
| Triton X-100 | Sigma-Aldrich | T8787 | Used for immunofluorescence of 3D structures |
| Trypsin-EDTA 10X | Biowest | X0930 | Dilute in PBS to obtain 3X solution |
| Vectashield Antifade Mounting Medium | Vector Laboratories | H-1000 | |
| WIT-P-NC Culture Medium | Stemgent | 00-0051 | Used for primary BPEC culture |
| WIT-T Culture Medium | Stemgent | 00-0047 | Used for transformed BPEC culture |
Referencias
- Hanahan, D., Weinberg, R. A. Hallmarks of cancer: the next generation. Cell. 144 (5), 646-674 (2011).
- Stampfer, M. R., Yaswen, P. Culture models of human mammary epithelial cell transformation. Journal of Mammary Gland Biology and Neoplasia. 5 (4), 365-378 (2000).
- Schinzel, A. C., Hahn, W. C. Oncogenic transformation and experimental models of human cancer. Frontiers in Bioscience : A Journal and Virtual Library. 13 (13), 71 (2008).
- Balani, S., Nguyen, L. V., Eaves, C. J. Modeling the process of human tumorigenesis. Nature Communications. 8 (1), 15422 (2017).
- Bray, F., et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA: A Cancer Journal for Clinicians. 68 (6), 394-424 (2018).
- Ince, T. A., et al. Transformation of different human breast epithelial cell types leads to distinct tumor phenotypes. Cancer Cell. 12 (2), 160-170 (2007).
- Repullés, J., et al. Radiation-induced malignant transformation of preneoplastic and normal breast primary epithelial cells. Molecular Cancer Research. , 1-13 (2019).
- Weigelt, B., Bissell, M. J. Unraveling the microenvironmental influences on the normal mammary gland and breast cancer. Seminars in Cancer Biology. 18 (5), 311-321 (2008).
- Paoli, P., Giannoni, E., Chiarugi, P. Anoikis molecular pathways and its role in cancer progression. Biochimica et Biophysica Acta. 1833 (12), 3481-3498 (2013).
- Debnath, J., Muthuswamy, S. K., Brugge, J. S. Morphogenesis and oncogenesis of MCF-10A mammary epithelial acini grown in three-dimensional basement membrane cultures. Methods. 30 (3), 256-268 (2003).
- Borowicz, S., et al. The soft agar colony formation assay. Journal of Visualized Experiments. (92), (2014).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Brocher, J. The BioVoxxel Image Processing and Analysis Toolbox. European BioImage Analysis Symposium. 8 (2), 67112 (2015).
- Torquato, S., Truskett, T. M., Debenedetti, P. G. Is random close packing of spheres well defined. Physical Review Letters. 84 (10), 2064-2067 (2000).
- LaBarge, M. A., Garbe, J. C., Stampfer, M. R. Processing of human reduction mammoplasty and mastectomy tissues for cell culture. Journal of Visualized Experiments. (71), (2013).
- Zubeldia-Plazaola, A., et al. Glucocorticoids promote transition of ductal carcinoma in situ to invasive ductal carcinoma by inducing myoepithelial cell apoptosis. Breast Cancer Research. 20 (1), 65 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados