Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Évaluation in vitro de la transformation oncogène des cellules épithéliales mammaires humaines
Dans cet article
Résumé
Ce protocole fournit des outils expérimentaux in vitro pour évaluer la transformation des cellules mammaires humaines. Des étapes détaillées pour le taux de prolifération cellulaire de suivi, la capacité de croissance ancre-indépendante, et la distribution des lignées cellulaires dans les cultures 3D avec la matrice de membrane de sous-sol sont décrites.
Résumé
Tumorigenesis est un processus en plusieurs étapes dans lequel les cellules acquièrent des capacités qui permettent leur croissance, survie et diffusion dans des conditions hostiles. Différents tests visent à identifier et quantifier ces caractéristiques des cellules cancéreuses; cependant, ils se concentrent souvent sur un seul aspect de la transformation cellulaire et, en fait, plusieurs tests sont nécessaires pour leur caractérisation appropriée. Le but de ces travaux est de fournir aux chercheurs un ensemble d’outils pour évaluer la transformation cellulaire in vitro d’un point de vue large, permettant ainsi de tirer des conclusions solides.
Une activation proliférative soutenue de signalisation est la principale caractéristique des tissus tumoraux et peut être facilement surveillée dans des conditions in vitro en calculant le nombre de doublons de population réalisés au fil du temps. En outre, la croissance des cellules dans les cultures 3D permet leur interaction avec les cellules environnantes, ressemblant à ce qui se passe in vivo. Ceci permet l’évaluation de l’agrégation cellulaire et, avec l’étiquetage immunofluorescent des marqueurs cellulaires distinctifs, d’obtenir l’information sur une autre caractéristique pertinente de la transformation tumorale : la perte de l’organisation appropriée. Une autre caractéristique remarquable des cellules transformées est leur capacité à croître sans attachement à d’autres cellules et à la matrice extracellulaire, qui peut être évaluée avec l’analyse d’ancrage.
Des procédures expérimentales détaillées pour évaluer le taux de croissance cellulaire, pour effectuer l’étiquetage immunofluorescent des marqueurs de lignée cellulaire dans les cultures 3D, et pour tester la croissance cellulaire indépendante de l’ancrage chez les agars mous sont fournies. Ces méthodologies sont optimisées pour les cellules épithéliales primaires du sein (BPEC) en raison de sa pertinence dans le cancer du sein; cependant, les procédures peuvent être appliquées à d’autres types de cellules après quelques ajustements.
Introduction
Plusieurs événements successifs sont nécessaires pour le développement du néoplasme. En 2011, Hanahan et Weinberg ont décrit 10 capacités qui permettent la croissance, la survie et la diffusion des cellules transformées : les « caractéristiques du cancer» 1. La méthodologie décrite ici compile trois outils différents pour évaluer la transformation cellulaire in vitro en se concentrant sur certaines des caractéristiques distinctives des cellules tumorales. Ces techniques évaluent le taux de prolifération cellulaire, le comportement des cellules lorsqu’elles sont cultivés en 3D et leur capacité à former des colonies avec l’indépendance de l’ancrage.
Les modèles cellulaires sont essentiels pour tester l’hypothèse in vitro. Différentes approches ont été développées pour générer des modèles expérimentaux de transformation cellulaire pour l’étude du cancer2,3,4. Étant donné que le cancer du sein est le cancer le plus fréquent chez les femmes dans le monde et est responsable d’environ 15% des décès par cancer chezles femmes 5, fournir des modèles cellulaires appropriés de cellules épithéliales mammaires est de la plus haute importance pour une enquête plus approfondie. Dans cet article, nous avons illustré le potentiel de trois techniques pour évaluer la transformation cellulaire à l’aide d’un modèle expérimental de transformation des cellules épithéliales primaires du sein (BPECs) initialement décrite par Ince et ses collègues en 20076 et mise en œuvre ultérieurement dans notrelaboratoire 7. Ce modèle expérimental est basé sur l’altération séquentielle de trois gènes ciblés (SV40 Gros T et petits antigènes t ci-dessous appelés Ttag, hTERTet HRAS)au génome des BPECs non transformés. En outre, la méthode utilisée pour la dérivation bpecs favorise le maintien des cellules épithéliales mammaires avec des marqueurs luminaux ou myoepithelial, résultant en une culture cellulaire hétérogène qui conserve certains des traits physiologiques de la glande mammaire.
Dans la glande mammaire, les cellules épithéliales mammaires luminaires, qui sont responsables de la production laitière, sont situées près du lumen, tandis que les cellules myoepitheliales sont éliminées autour des cellules luminaires et prennent soin des mouvements de contraction menant le lait au mamelon. La perte de l’organisation appropriée entre ces lignées cellulaires est une caractéristique de la transformation tumorale8 qui peut être évaluée in vitro après détection immunofluorescente des marqueurs distinctifs de lignée dans les cultures cellulaires 3D. Une autre caractéristique majeure des cellules tumorales est leur capacité à croître sans attachement à d’autres cellules et à la matrice extracellulaire1. Lorsque les cellules saines sont forcées de croître en suspension, des mécanismes tels que anoikis \u2012 un type de mort cellulaire induite en réponse au détachement de la matrice extracellulaire \u2012 sont activés9. L’évasion de la mort cellulaire est l’une des caractéristiques distinctives du cancer et, par conséquent, les cellules transformées sont capables d’inactiver les anoikis et de survivre d’une manière indépendante de l’ancre. Cette capacité peut être évaluée in vitro avec l’analyse indépendante de l’ancrage à l’aide d’une agar molle. En outre, une caractéristique inhérente des tissus tumoraux est leur capacité de signalisation proliférative soutenue, qui peut être facilement surveillée dans des conditions in vitro en mesurant l’augmentation du nombre de cellules le long du temps, non seulement dans les tests de suspension, mais aussi en surveillant le taux de croissance des cultures adhérentes monocouches.
En dépit du meilleur modèle pour tester le potentiel tumorigenic est l’inoculation des cellules tumorales dans les modèles murins et l’évaluation du développement de tumeur in situ, il est important de réduire au minimum le nombre d’animaux employés dans les procédures expérimentales autant que possible. Par conséquent, avoir des tests appropriés pour évaluer la transformation in vitro est une priorité absolue. Ici, nous fournissons un ensemble d’outils pour évaluer le potentiel tumoriogène des cellules épithéliales du sein partiellement et entièrement transformées qui peuvent être facilement mises en œuvre dans la plupart des laboratoires qui travaillent avec des modèles de transformation cellulaire.
Protocole
Des échantillons humains utilisés dans les expériences suivantes ont été obtenus à partir de mammoplasties de réduction réalisées à Clínica Pilar Sant Jordi (Barcelone) sous le consentement standard de la procédure. Toutes les procédures sont effectuées dans un cabinet de sécurité biologique de classe II, sauf indication contraire.
1. Culture in vitro des cellules épithéliales mammaires humaines et accumulation de parcelles courbes de croissance
-
Culture in vitro des cellules épithéliales primaires du sein (BPECs) : passage cellulaire
REMARQUE : Pour la dérivation et la culture cellulaire du BPEC, suivez les instructions décrites par Ince et coll., 20076.- Préparation moyenne.
- Supplément WIT milieu basal défini avec des suppléments de P ou T, fournis par le fabricant, selon que les BPECs primaires ou transformés sont cultivés.
- Ajouter la toxine du choléra au milieu wit complété à une concentration finale de 100 ng/mL pour les BPECs primaires ou de 25 ng/mL pour les BPECs transformés.
ATTENTION : La toxine du choléra est mortelle si elle est avalée. Utilisez de l’équipement de protection individuelle. Évitez sa libération dans l’environnement.
- Entretien et passage de la culture cellulaire.
REMARQUE : Pour les étapes suivantes, gardez à l’esprit que les cellules poussent dans un flacon T25. Néanmoins, les volumes peuvent être adaptés à d’autres formats de culture cellulaire en conservant la proportionnalité en termes de surface.- Vérifiez la confluence cellulaire tous les jours. Lorsque la culture est 90% confluente, effectuer le passage cellulaire.
- Acquérir 1x PBS, trypsine 3x, moyenne et un tube conique de 15 mL contenant 2 mL de sérum bovin fœtal (FBS) pour chaque flacon.
- Retirez le milieu du flacon et gardez-le dans le tube conique de 15 mL contenant du FBS.
- Rincer les cellules avec 1x PBS.
- Détachez les cellules de la surface en ajoutant 1 mL de trypsine 3x. Incuber pendant 5 min à 37 °C.
- Vérifiez si les cellules ont été détachées. Appliquer des secousses vigoureuses si les cellules ne sont pas complètement détachées.
- Inactivez la trypsine en ajoutant le milieu réservé complété par FBS.
- Récoltez la suspension cellulaire et placez-la dans le tube conique de 15 mL.
- Centrifugeuse à 500 x g pendant 5 min, éliminer le supernatant, et resuspendre les cellules granulées en feuilletant le fond du tube avec un doigt.
- Ajouter 1 à 2 mL de médias frais à la pastille et mesurer la concentration cellulaire à l’aide d’un compteur cellulaire automatique ou d’un hémocytomètre. Ces données seront ensuite utilisées pour calculer le doublement de la population et tracer la courbe de croissance. Graine 12 000 cellules/cm2 (p. ex., 300 000 cellules pour un flacon T25) dans des flacons de surface de culture cellulaire modifiée (voir tableau des matériaux).
REMARQUE : Diluer la solution de suspension cellulaire si la concentration est trop élevée pour assurer une quantification adéquate. - Ajouter du milieu à un volume final de 5 mL et incuber les cellules à 37 °C et 5 % d’atmosphère de CO2.
- Remplacer le milieu de culture cellulaire toutes les 48 h.
- Préparation moyenne.
-
Calcul du doublement de la population et visualisation des données
- À l’aide des données du nombre de cellules obtenues au cours de l’étape 1.1.2.10, appliquez la formule suivante pour obtenir les valeurs de doublement de la population accumulée :
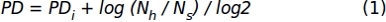
Lorsque, PDi désigne le nombre de doublements de population atteints par les cellules jusqu’à la sous-culture précédente (il se réfère à la MP accumulée sur la sous-culture précédente), Nhest le nombre de cellules récoltées, et Nsest le nombre de cellules ensemencées. - Représenter les données pour un intervalle de temps spécifique à l’aide d’un graphique XY où le nombre de jours en culture(x-axe)et la accumulée(y-axe)sont représentés.
- Obtenez la ligne la mieux adaptée et l’équation d’ajustement :

REMARQUE : Une pente accrue b) signifie un taux de prolifération accru.
- À l’aide des données du nombre de cellules obtenues au cours de l’étape 1.1.2.10, appliquez la formule suivante pour obtenir les valeurs de doublement de la population accumulée :
2. Culture tridimensionnelle (3D) dans la matrice membranaire du sous-sol et la détection des protéines immunofluorescentes
- Culture 3D dans la matrice membranaire du sous-sol
REMARQUE : Ce protocole a été adapté de Debnath et coll., 200310 et est optimisé pour 24 plaques de puits (voir Tableau des matériaux).- Préparez le matériau un jour avant l’expérience : pré-refroidissement de la matrice membranaire du sous-sol pendant la nuit à 4 °C et laissez refroidir les pointes de pipette, les tubes de microcentrifugeuse et les plaques de puits dans le congélateur.
REMARQUE : La matrice doit être maintenue à -20 °C pour le stockage à long terme. Faire des aliquots pour éviter plusieurs cycles de gel-dégel. - Le jour de l’expérience, placez du matériel pré-refroidi sur la glace.
- Rincer les puits à l’aide d’un 1x PBS stérile à froid afin de réduire la tension de surface.
- Couvrir le fond de chaque puits de 100 μL de matrice membranaire du sous-sol.
REMARQUE : Distribuez lentement la matrice et étalez-la dans tout le puits; il est crucial d’éviter la formation de bulles dans la couche inférieure pour éviter la croissance de la culture cellulaire monocouche. - Placez la plaque dans l’incubateur, à 37 °C, pour laisser la couche matricielle se solidifier.
REMARQUE : Il faut habituellement environ 20 min pour se solidifier. - Pendant ce temps, trypsinize cellules comme expliqué précédemment dans l’étape 1.1. Centrifugeuse les cellules à 500 x g pendant 5 min et resuspendre à moyen. Préparer une suspension de 400 000 cellules/mL et désaggréger délicatement toute touffe cellulaire par pipetting.
- Préparer le milieu avec une matrice membranaire de sous-sol de 8 % et mélanger 1:1 (v/v) avec une suspension cellulaire pour obtenir une solution de 200 000 cellules/mL dans une matrice de 4 %.
REMARQUE : Calculez la quantité de milieu nécessaire pour éviter les déchets inutiles de matrice. - Placez 500 μL de suspension cellulaire dans la solution matricielle au-dessus de la couche matricielle déjà solidifiée pour ensemencer une quantité totale de 100 000 cellules en moyenne avec une matrice membranaire de sous-sol de 4 %.
- Incuber les cellules à 37 °C pendant quelques minutes, puis ajouter 500 μL du milieu avec une matrice membranaire de sous-sol de 4 %. Incuber les cellules à 37 °C dans un incubateur avec 5 % de CO2 pendant 14 jours. Les cellules ensemencées se regroupent et prolifèrent pour être à l’origine des structures acini-like.
REMARQUE : La motilité et l’agrégation cellulaires peuvent être surveillées par time-lapse pendant le processus de formation 3D. Utilisez un logiciel d’analyse d’images (p. ex., Fidji/ImageJ ou Imaris) pour évaluer ces événements. Le nombre et la taille de l’acini dépendent du processus d’agrégation et du taux de prolifération et peuvent varier d’un type de cellule à l’autre. Ajuster la concentration de la matrice membranaire du sous-sol et les cellules ensemencées pour obtenir les structures 3D souhaitées. - Ajouter 500 μL du milieu avec une matrice membranaire de sous-sol de 4 % 2 à 3 fois par semaine.
REMARQUE : Évitez les perturbations des couches portant doucement la plaque pendant la manipulation. - Si vous le souhaitez, le nombre et la taille de l’acini peuvent être mesurés pendant la période de culture. Pour ce faire, prenez des photos aléatoires à différents moments après l’ensemencement à l’aide d’un contraste de phase ou d’un microscope inversé DIC. Utilisez un logiciel d’analyse d’images pour mesurer le diamètre des structures 3D de 100 à 200.
- Préparez le matériau un jour avant l’expérience : pré-refroidissement de la matrice membranaire du sous-sol pendant la nuit à 4 °C et laissez refroidir les pointes de pipette, les tubes de microcentrifugeuse et les plaques de puits dans le congélateur.
- Immunomarquage
REMARQUE : Les conditions stériles ne sont pas requises pendant cette partie du protocole.- Supprimer le milieu de culture.
- Déchirer la matrice membranaire du sous-sol à l’aide d’une pointe de pipette p200 avec l’extrémité coupée. Placez ~50 μL de matrice désagrégée sur une lame de verre et enduitez-la dans une zone de 1-2 cm2.
- Laissez sécher complètement l’échantillon à température ambiante ou utilisez une plaque chauffante à 37 °C pour accélérer le processus. Fixer les échantillons avec du méthanol: acétone (1:1, v/v) à -20 °C pendant 30 min.
REMARQUE : Le signal fluorescent des marqueurs précédents, comme celui des protéines fluorescentes exprimées par les cellules, sera effacé.
MISE EN GARDE : Le méthanol est inflammable, toxique s’il est inhalé, avalé ou au cas où il entre en contact avec la peau. Portez de l’équipement de protection individuelle et travaillez à l’intérieur d’un capot à fumée. - Jetez la solution de fixation et retirez l’excédent, le cas échéant, en inclinant la glissière sur du papier filtre.
REMARQUE : Le protocole peut être mis en pause ici. Une fois sèches, les glissières peuvent être stockées à -20 °C pendant plusieurs mois. - Les échantillons de blocs épitopes avec 5% de sérum de chèvre normal et 0,1% triton-X-100 en 1x PBS (solution de blocage) pendant 2 h à température ambiante.
- Pendant ce temps, préparer des anticorps solutions de travail en diluant les anticorps primaires ou secondaires à la concentration désirée dans la solution de blocage.
REMARQUE : La concentration d’anticorps doit être ajustée avec précision en fonction du type de cellule et de la référence des anticorps. À titre de guide, pour identifier les cellules des lignées luminales et myoepitheliales dans BPEC, l’anti cytokératine primaire 14 et les anticorps anti-Claudin-IV (voir tableau des matériaux)peuvent être utilisés. La concentration recommandée de solution de travail est de 1:100 pour ces anticorps primaires et de 1:500 pour les anticorps secondaires anti-souris et anti-lapin (voir tableau des matériaux). - Ajouter 30 μL d’anticorps primaires solution de travail et le couvrir avec une bande de film d’emballage de laboratoire pour éviter l’évaporation. Incuber toute la nuit à 4 °C dans une chambre humide.
- Laver trois fois avec 1x PBS pendant 1 h chacun.
- Répétez l’étape 2.2.7 pour les anticorps secondaires. L’incubation doit être effectuée dans l’obscurité.
- Laver avec 1x PBS pendant 2 h.
REMARQUE : Ajustez la concentration des anticorps, le temps d’incubation et la dureté du lavage afin d’améliorer le rapport signal/bruit pour des échantillons spécifiques. - Retirer le PBS restant et, une fois sec, contre-détenir avec dapi à 0,25 μg/mL dilué dans un milieu de montage antifade. Couvrir les diapositives d’un coverslip en le laissant s’installer sans appliquer de pression. Sceller avec du vernis à ongles.
REMARQUE : Les échantillons peuvent être conservés à 4 °C pendant plusieurs semaines. Pour un stockage à long terme, gardez-les à -20 °C. - Analyser la distribution du signal fluorescent pour chaque acinus à l’aide d’un microscope confocal.
REMARQUE : La configuration du microscope confocal doit être déterminée avec précision en fonction de l’équipement utilisé et des anticorps appliqués à l’échantillon. À titre de guide, avec l’équipement et les réadageurs détaillés dans le tableau des matériaux, utilisez un objectif 40x et les paramètres laser et détecteur suivants: pour l’excitation utilisation DAPI avec un laser 405 (3%-5%), détection avec un détecteur PMT (800V, Offset: -9) et une bande spectrale de 410 nm à 500 nm; pour l’A488 (Claudin-IV) utiliser excitation avec un laser 488 (7%-10%), détection avec un détecteur PMT (800V, Offset: -20) et une bande spectrale de 490 nm à 550 nm; et pour Cy3 (Cytokeratin 14) utiliser excitation avec un laser 555 (2%-10%), détection avec un détecteur PMT (800V, Offset: -35) et une bande spectrale de 560 nm à 600 nm.
3. Analyse indépendante d’Anchorage, coloration de MTT et quantification automatique de colonie
- Analyse indépendante de l’ancrage : placage d’agar et de suspension cellulaire
REMARQUE : Le protocole a été adapté de Borowicz et coll., 201411 pour effectuer des expériences dans les BPECs.- Préparer une solution d’agar de 1,2 % diluée dans de l’eau ultrapure dans une bouteille stérile. Autoclavez la solution et maintenez-la à 42 °C pendant l’expérience. La solution d’agar peut être stockée à 4 °C; au besoin, chauffer la solution d’agar jusqu’à ce qu’elle soit à nouveau liquide.
ATTENTION : Utilisez des gants résistants à la chaleur pour éviter les brûlures après l’autoclave.
REMARQUE : Désormais, les conditions stériles doivent être maintenues. - Préparez une solution d’agar de 0,6 % en mélangeant 1:1 (v/v) milieu préchauffé complet avec une solution d’agar de 1,2 %. Maintenir à 42 °C pour éviter une solidification prématurée.
REMARQUE: Moyen peut être préalablement double complété pour obtenir un entièrement complété 0,6% agar + solution moyenne une fois mélangé. - Couvrez le fond d’un puits de 35 mm avec 1,5 mL d’agar de 0,6 % en solution moyenne et laissez-le se solidifier à température ambiante. Assurez-vous que le fond de la plaque est complètement couvert avant la solidification de l’agar, sinon, les cellules peuvent adhérer à la plaque et se développer en monomeuse.
REMARQUE : Des plaques de surface adhérentes et non adhérentes peuvent être utilisées. - Pendant ce temps, trypsiniser les cellules et, une fois centrifugées et résuspendées en milieu, préparer une solution de 50 000 cellules/ml et désaggréger délicatement n’importe quelle touffe cellulaire par pipetting à plusieurs reprises.
- Préparer une suspension de 0,3% d’agar + cellule dans le milieu à une concentration finale de 25.000 cellules/mL.
REMARQUE : La concentration optimale de cellules peut différer d’un type de cellule à l’autre. Essayez différentes concentrations jusqu’à formation de colonies individualisées.- Placez un filtre de passoire de 40 μm sur un tube stérile de 50 mL et filtrez la solution de 50 000 cellules/mL en la laissant tomber au fond du tube.
- Retirez le filtre du tube stérile de 50 mL, inclinez le tube contenant des cellules à un angle de 45° et laissez tomber le même volume de 0,6% d’agar + solution moyenne le déversant à travers la paroi interne du tube. Cela permettra à la solution d’agar de refroidir juste assez pour ne pas endommager les cellules et éviter sa solidification prématurée.
- Homogénéiser le mélange et déposer 1 mL de 0,3% d’agar + suspension cellulaire dans le milieu (contenant 25.000 cellules) sur le dessus de la couche d’agar inférieur précédemment solidifié.
- Visualisez les cellules ensemencées à l’aide d’un microscope inversé pour s’assurer que les cellules sont individualisées. Sinon, l’expérience doit être répétée.
- Attendez que la couche d’agar soit complètement solidifiée, puis ajoutez soigneusement 1 mL du milieu frais sur le dessus sans déranger les couches délicates d’agar en dessous.
- Incuber les cellules à 37 °C et 5 % de CO2 dans un incubateur pendant 3 semaines.
REMARQUE : Le temps requis pour la formation des colonies peut varier d’un type de cellule à l’autre, mais habituellement 3 semaines sont suffisantes. - Changez moyen deux fois par semaine. Pour ce faire, inclinez doucement la plaque vers vous, aspirez le milieu dans le coin inférieur et ajoutez 1 mL de milieu frais.
REMARQUE : Évitez de toucher les couches d’agar car elles se détachent facilement de la plaque.
- Préparer une solution d’agar de 1,2 % diluée dans de l’eau ultrapure dans une bouteille stérile. Autoclavez la solution et maintenez-la à 42 °C pendant l’expérience. La solution d’agar peut être stockée à 4 °C; au besoin, chauffer la solution d’agar jusqu’à ce qu’elle soit à nouveau liquide.
- Coloration MTT
- Préparer la solution de bouillon thiazolyl bleu tetrazolium bromure (MTT) à 6 mg/mL dans de l’eau ultrapure dans une bouteille stérile et une solution de filtre à l’aide de filtres de 0,2 μm. Cette solution MTT peut être stockée jusqu’à 6 mois à -20 °C.
ATTENTION: MTT peut causer une irritation et est soupçonné de causer des défauts génétiques. Utilisez des lunettes de sécurité, des gants et un filtre respiratoire.
REMARQUE : Évitez les cycles répétés de gel-dégel. - Préparer une solution de travail de MTT à 1 mg/mL en diluant la solution de stock avec de l’eau ultrapure stérile.
- Une fois la période de formation de la colonie terminée, retirer le milieu de la plaque et ajouter 1 mL de 1 mg/mL de MTT à chaque puits.
- Incuber pendant 24 h dans l’incubateur. Retirez la solution MTT en l’aspirant doucement. Les plaques peuvent être conservées à 4 °C pendant plusieurs semaines.
REMARQUE : Évitez l’exposition à la lumière pour prévenir la formation de cristaux non spécifiques.
- Préparer la solution de bouillon thiazolyl bleu tetrazolium bromure (MTT) à 6 mg/mL dans de l’eau ultrapure dans une bouteille stérile et une solution de filtre à l’aide de filtres de 0,2 μm. Cette solution MTT peut être stockée jusqu’à 6 mois à -20 °C.
- Quantification des colonies
- Obtenez des images de chaque plaque à l’aide d’un microscope inversé. Ajustez le grossissement afin d’acquérir le champ de vision maximum avec moins d’images tout en étant capable de détecter de petites colonies (généralement des objectifs 4x ou 10x).
REMARQUE : Assurez-vous que les images présentent un arrière-plan homogène. Ni le contraste de phase ni le contraste différentiel n’est nécessaire puisque les colonies non tachées ne seront pas quantifiées. - Téléchargez des images sur le logiciel ImageJ/Fiji12 pour compter le nombre de colonies et la superficie de chaque colonie positive au MTT.
REMARQUE : Un script pour la quantification automatique est fourni sous forme de fichier supplémentaire. Pour exécuter le code, coller à l’éditeur macro(Plugins | Nouveau | Macro) et suivez les instructions.- Obtenez un masque binaire en seuilant l’imaged’origine (Image | Ajuster | Seuil) pour obtenir des colonies bien délimitées (figure 1).
REMARQUE : Des images 8 ou 16 bits sont généralement nécessaires pour effectuer cette étape. La méthode du « seuil minimum » est recommandée. - Exécutez l’analyseur de particules étendus à partir du plugin Biovoxxel13 (Plugins | BioVoxxel | Analyseur de particules étendu) pour identifier les colonies positives du MTT( figure 2). Conditions directrices initiales : Taille (μm2) = 250-Infinity; Solidité = 0,75-1,00.
- Obtenez un masque binaire en seuilant l’imaged’origine (Image | Ajuster | Seuil) pour obtenir des colonies bien délimitées (figure 1).
- Estimer le diamètre moyen (D) de chaque valeur de la zone de la colonie (A) selon la formule :

- Filtrer les résultats en excluant les colonies proliférantes faibles (p. ex., faible diamètre).
- Choisissez un nombre minimum de divisions par semaine(m)à considérer (p. ex., 1).
- Estimer le rayon d’une colonie (R) avec des cellules n selon la formule suivante:

Où, r est le rayon moyen des cellules individuelles en suspension, n est le nombre de cellules formant une colonie qui a souffert de divisions m chaque semaine pendant les semaines w dans la culture. En croissance exponentielle : n = 2(m*w). c’est l’efficacité de l’emballage. Notez que, dans le mouvement aléatoire, l’efficacité de l’emballage est ~0.64 et la fraction d’emballage la plus dense possible pour les sphères identiques est de 0.7414. - Jetez toutes les colonies qui présentent un diamètre inférieur à 2R car leurs cellules n’ont pas atteint le nombre minimum de divisions considérées à l’étape 3.3.4.1.
- Obtenez des images de chaque plaque à l’aide d’un microscope inversé. Ajustez le grossissement afin d’acquérir le champ de vision maximum avec moins d’images tout en étant capable de détecter de petites colonies (généralement des objectifs 4x ou 10x).
Résultats
Un modèle expérimental de transformation cellulaire avec l’introduction de trois éléments génétiques dans les BPEC a été choisi pour générer des résultats représentatifs de la transformationoncogène 6,7 (Figure 3). Les BPECs non transformés (N) ont été dérivés du tissu mammaire sans maladie tel que décrit par Ince et sescollègues 6 et cultivés suivant le protoco...
Discussion
Les protocoles expérimentaux décrits dans cet article fournissent des outils utiles pour évaluer la transformation oncogène des cellules de culture in vitro. Chaque technique évalue des aspects spécifiques du processus de transformation et, par conséquent, une attention particulière doit être accordée lors de l’élaboration de conclusions à partir d’une seule analyse. L’accumulation des courbes de croissance est une approche qui exige des informations déjà disponibles lors de la culture des cellules à...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Le laboratoire AG est financé par le Conseil espagnol de sûreté nucléaire. T.A. et A.G. font partie d’un groupe de recherche reconnu par la Generalitat de Catalunya (2017-SGR-503). MT détient un contrat financé par la Fondation scientifique Asociación Española Contra el Cáncer [AECC-INVES19022TERR]. Le contrat G.F. est financé par une subvention de la Fondation Cellex.
matériels
| Name | Company | Catalog Number | Comments |
| 1 ml Serological Pipettes | Labclinics | PLC91001 | |
| 1.5 ml Eppendorfs | Thermo Fisher Scientific | 3451 | Dark eppendorfs are preferred for MTT long-term storage |
| 10 μl Pipette tips w/o filter | Biologix | 20-0010 | |
| 100 ml glass bottle | With cap, autoclavable | ||
| 1000 μl Pipette tips w/ filter | Labclinics | LAB1000ULFNL | |
| 1000 μl Pipette tips w/o filter | Biologix | 20-1000 | |
| 15 ml Conical tubes | VWR | 525-0400 | |
| 2 ml Serological Pipettes | Labclinics | PLC91002 | |
| 200 μl Pipette tips w/ filter | Labclinics | FTR200-96 | |
| 5 ml Serological Pipettes | Labclinics | PLC91005 | |
| 50 ml Conical Tubes | VWR | 525-0304 | |
| Acetone | PanReac AppliChem | 211007 | Used for 3D structure fixation prior to immunofluorescent labelling |
| Agar | Sigma-Aldrich | A1296 | Used for anchorage assay |
| Anti-Claudin 4 antibody | Abcam | 15104, RRID:AB_301650 | Working dilution 1:100, host: rabbit |
| Anti-Cytokeratin 14 [RCK107] antibody | Abcam | 9220, RRID:AB_307087 | Working dilution 1:100, host: mouse |
| Anti-mouse Cyanine Cy3 antibody | Jackson ImmunoResearch Inc. | 115-165-146, RRID:AB_2338690 | Working dilution 1:500, host: goat |
| Anti-rabbit Alexa Fluor 488 antibody | Thermo Fisher Scientific | A-11034, RRID:AB_2576217 | Working dilution 1:500, host: goat |
| Autoclave | |||
| BioVoxxel Toolbox | RRID:SCR_015825 | ||
| Cell culture 24-well Plate | Labclinics | PLC30024 | Used for 3D cultures in Matrigel. Flat Bottom |
| Cell culture 6-well Plate | Labclinics | PLC30006 | Used for anchorage assay |
| Cell incubator (37 ºC and 5 % CO2) | |||
| Cell Strainers | Fisherbrand | 11587522 | Mesh size: 40 μm |
| CellSense software | Olympus | Used to image acquisition | |
| Centrifuge | |||
| Cholera Toxin from Vibrio cholerae | Sigma-Aldrich | C8052 | Used to supplement cell culture medium |
| Class II Biological Safety Cabinet | Herasafe | HAEREUS HS12 | |
| Confocal inverted Microscope | Leica | TCS SP5 | |
| Cover glasses | Witeg Labortechnik GmbH | 4600122 | 22 X 22 mm, thickness 0.13 - 0.17 mm |
| DAPI | 2-(4-amidinophenyl)-1H -indole-6-carboxamidine | ||
| Fetal Bovine Serum | Biowest | S1810 | Used to inactivate trypsine action |
| Fiji software (ImageJ) | National Institutes of Health | RRID:SCR_002285 | Free download, no license needed |
| Glass Pasteur Pipettes | |||
| Glass slides | Fisherbrand | 11844782 | |
| Goat Serum | Biowest | S2000 | Used for immunofluorescence of 3D structures |
| Heat-Resistant Gloves | Used for agar manipulation after autoclave | ||
| Heater bath (37 ºC) | Used to temper solutions prior to cell subculture | ||
| Heater bath (42 ºC) | Used to keep agar warm | ||
| Heating plate | Used for Matrigel dehydration | ||
| Humid chamber | Used for the incubation of antibodies during immunofluorescence | ||
| Ice | Used during Matrigel manipulation | ||
| Ice-box | |||
| Inverted Optic Microscope | Olympus | IX71 | |
| Matrigel Matrix | Becton Dickinson | 354234 | Store at -20 ºC and keep cold when in use. Referred to as basement membrane matrix |
| Methanol | PanReac AppliChem | 131091 | Used for 3D structure fixation prior to immunofluorescent labelling |
| Micropipette | p1000, p200 and p10 | ||
| Microsoft Office Excel | Microsoft | RRID:SCR_016137 | Used to calculate population doubling and to obtain growth rate equation |
| MilliQ water | Referred to as ultrapure water | ||
| Nail Polish | Used to seal samples after mounting | ||
| Parafilm M | Bemis | PM-999 | Used to cover antibody solution during incubation |
| PBS pH 7.4 (w/o calcium & magnesium) | Gibco | 10010-056 | Sterile. Used for cell subculture |
| PBS tablets | Sigma-Aldrich | P4417 | Dilute in milliQ water. No sterility required. Used for immunofluorescence |
| Pipette Aid | |||
| Primaria T25 flasks | Corning | 353808 | Used for BPEC culture |
| Scepter Automated Cell Counter | Millipore | PHCC20060 | Alternatively, use an haemocytometer |
| Scissors | Used to cut pipette tips and parafilm | ||
| Sterile filters 0.22 μm | Millipore | SLGP033RS | Used to filter MTT solution |
| Thiazolyl Blue Tetrazolium Bromide (MTT) | Sigma-Aldrich | M2128 | Store at -20 ºC |
| Triton X-100 | Sigma-Aldrich | T8787 | Used for immunofluorescence of 3D structures |
| Trypsin-EDTA 10X | Biowest | X0930 | Dilute in PBS to obtain 3X solution |
| Vectashield Antifade Mounting Medium | Vector Laboratories | H-1000 | |
| WIT-P-NC Culture Medium | Stemgent | 00-0051 | Used for primary BPEC culture |
| WIT-T Culture Medium | Stemgent | 00-0047 | Used for transformed BPEC culture |
Références
- Hanahan, D., Weinberg, R. A. Hallmarks of cancer: the next generation. Cell. 144 (5), 646-674 (2011).
- Stampfer, M. R., Yaswen, P. Culture models of human mammary epithelial cell transformation. Journal of Mammary Gland Biology and Neoplasia. 5 (4), 365-378 (2000).
- Schinzel, A. C., Hahn, W. C. Oncogenic transformation and experimental models of human cancer. Frontiers in Bioscience : A Journal and Virtual Library. 13 (13), 71 (2008).
- Balani, S., Nguyen, L. V., Eaves, C. J. Modeling the process of human tumorigenesis. Nature Communications. 8 (1), 15422 (2017).
- Bray, F., et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA: A Cancer Journal for Clinicians. 68 (6), 394-424 (2018).
- Ince, T. A., et al. Transformation of different human breast epithelial cell types leads to distinct tumor phenotypes. Cancer Cell. 12 (2), 160-170 (2007).
- Repullés, J., et al. Radiation-induced malignant transformation of preneoplastic and normal breast primary epithelial cells. Molecular Cancer Research. , 1-13 (2019).
- Weigelt, B., Bissell, M. J. Unraveling the microenvironmental influences on the normal mammary gland and breast cancer. Seminars in Cancer Biology. 18 (5), 311-321 (2008).
- Paoli, P., Giannoni, E., Chiarugi, P. Anoikis molecular pathways and its role in cancer progression. Biochimica et Biophysica Acta. 1833 (12), 3481-3498 (2013).
- Debnath, J., Muthuswamy, S. K., Brugge, J. S. Morphogenesis and oncogenesis of MCF-10A mammary epithelial acini grown in three-dimensional basement membrane cultures. Methods. 30 (3), 256-268 (2003).
- Borowicz, S., et al. The soft agar colony formation assay. Journal of Visualized Experiments. (92), (2014).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Brocher, J. The BioVoxxel Image Processing and Analysis Toolbox. European BioImage Analysis Symposium. 8 (2), 67112 (2015).
- Torquato, S., Truskett, T. M., Debenedetti, P. G. Is random close packing of spheres well defined. Physical Review Letters. 84 (10), 2064-2067 (2000).
- LaBarge, M. A., Garbe, J. C., Stampfer, M. R. Processing of human reduction mammoplasty and mastectomy tissues for cell culture. Journal of Visualized Experiments. (71), (2013).
- Zubeldia-Plazaola, A., et al. Glucocorticoids promote transition of ductal carcinoma in situ to invasive ductal carcinoma by inducing myoepithelial cell apoptosis. Breast Cancer Research. 20 (1), 65 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon