Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
In Vitro-Bewertung der onkogenen Transformation in menschlichen Mammaepithelzellen
In diesem Artikel
Zusammenfassung
Dieses Protokoll bietet experimentelle In-vitro-Werkzeuge zur Bewertung der Transformation menschlicher Brustzellen. Detaillierte Schritte zur Nachverfolgung der Zellproliferationsrate, der verankerungsunabhängigen Wachstumskapazität und der Verteilung von Zelllinien in 3D-Kulturen mit Kellermembranmatrix werden beschrieben.
Zusammenfassung
Tumorigenese ist ein mehrstufiger Prozess, bei dem Zellen Fähigkeiten erwerben, die ihr Wachstum, überleben und verbreiten unter feindlichen Bedingungen ermöglichen. Verschiedene Tests zielen darauf ab, diese Merkmale von Krebszellen zu identifizieren und zu quantifizieren; Sie konzentrieren sich jedoch häufig auf einen einzigen Aspekt der zellulären Transformation, und tatsächlich sind mehrere Tests für ihre korrekte Charakterisierung erforderlich. Ziel dieser Arbeit ist es, Forschern eine Reihe von Instrumenten zur Verfügung zu stellen, um die zelluläre Transformation in vitro aus einer breiten Perspektive zu bewerten und so fundierte Schlussfolgerungen zu ziehen.
Eine nachhaltige proliferative Signalaktivierung ist das Hauptmerkmal von Tumorgeweben und kann leicht unter In-vitro-Bedingungen überwacht werden, indem die Anzahl der im Laufe der Zeit erreichten Bevölkerungsverdoppelungen berechnet wird. Außerdem ermöglicht das Wachstum von Zellen in 3D-Kulturen ihre Interaktion mit umgebenden Zellen, ähnlich dem, was in vivo vorkommt. Dies ermöglicht die Bewertung der zellulären Aggregation und zusammen mit der immunfluoreszenzenten Kennzeichnung von ausgeprägten zellulären Markern, Um Informationen über ein weiteres relevantes Merkmal der Tumortransformation zu erhalten: den Verlust der richtigen Organisation. Ein weiteres bemerkenswertes Merkmal transformierter Zellen ist ihre Fähigkeit, ohne Bindung an andere Zellen und an die extrazelluläre Matrix zu wachsen, die mit dem Anker-Assay ausgewertet werden kann.
Detaillierte experimentelle Verfahren zur Bewertung der Zellwachstumsrate, zur immunfluoreszenzenten Kennzeichnung von Zelllinienmarkern in 3D-Kulturen und zur Erprobung des verankerungsunabhängigen Zellwachstums in weichem Agar werden bereitgestellt. Diese Methoden sind für primäre Epithelzellen (BPEC) der Brust aufgrund ihrer Relevanz bei Brustkrebs optimiert; Nach einigen Anpassungen können jedoch Prozeduren auf andere Zelltypen angewendet werden.
Einleitung
Für die Entwicklung von Neoplasma sind mehrere aufeinanderfolgende Ereignisse erforderlich. Im Jahr 2011 beschrieben Hanahan und Weinberg 10 Fähigkeiten, die das Wachstum, überleben und die Verbreitung transformierter Zellen ermöglichen: die sogenannten "Hallmarks of Cancer"1. Die hier beschriebene Methodik stellt drei verschiedene Werkzeuge zur Bewertung der in vitro zellulären Transformation zusammen, indem sie sich auf einige der Besonderheiten der Tumorzellen konzentriert. Diese Techniken bewerten die Zellproliferationsrate, das Verhalten von Zellen, wenn sie in 3D kultiviert werden, und ihre Fähigkeit, Kolonien mit Ankerunabhängigkeit zu bilden.
Zellmodelle sind entscheidend, um Hypothesen in vitro zu testen. Verschiedene Ansätze wurden entwickelt, um experimentelle Modelle der zellulären Transformation für die Untersuchung von Krebs zu generieren2,3,4. Da Brustkrebs weltweit die häufigste Krebsart bei Frauen ist und für etwa 15 % der Krebstoten bei Frauen5verantwortlich ist, ist die Bereitstellung geeigneter Zellmodelle von Brustepithelzellen von größter Bedeutung für die weitere Untersuchung. In diesem Artikel haben wir das Potenzial von drei Techniken zur Bewertung der zellulären Transformation anhand eines experimentellen Modells der Transformation von Breast Primary Epithelial Cells (BPECs) veranschaulicht, die ursprünglich von Ince und Kollegen im Jahr 20076 beschrieben und später in unserem Labor implementiert wurden7. Dieses experimentelle Modell basiert auf der sequenziellen Veränderung von drei gezielten Genen (SV40 Large T und small t Antigene hierin, die als Ttag, hTERTund HRASbezeichnet werden) in das Genom von nicht transformierten BPECs. Darüber hinaus begünstigt die Methode, die für die Ableitung von BPECs verwendet wird, die Aufrechterhaltung von Brustepithelzellen mit luminalen oder myoepithelialen Markern, was zu einer heterogenen Zellkultur führt, die einige der physiologischen Merkmale der Brustdrüse beibehält.
In der Brustdrüse befinden sich luminale Brustepithelzellen, die für die Milchproduktion verantwortlich sind, in der Nähe des Lumens, während Myoepithelzellen um Leuchtkörperzellen angeordnet sind und sich um Kontraktionsbewegungen kümmern, die die Milch zur Brustwarze führen. Der Verlust der richtigen Organisation zwischen diesen Zelllinien ist ein Merkmal der tumoralen Transformation8, das in vitro nach immunfluoreszierendem Nachweis von markanten Linienmarkern in 3D-Zellkulturen beurteilt werden kann. Ein weiteres wesentliches Merkmal von Tumorzellen ist ihre Fähigkeit, ohne Bindung an andere Zellen und an die extrazelluläre Matrix1zu wachsen. Wenn gesunde Zellen gezwungen werden, in Suspension zu wachsen, werden Mechanismen wie anoikis - u2012 eine Art von Zelltod induziert als Reaktion auf die Ablösung von der extrazellulären Matrix aktiviert. Die Umgehung des Zelltodes ist eines der kennzeichens Merkmale von Krebs und somit sind transformierte Zellen in der Lage, Anoikis zu inaktivieren und ankerunabhängig zu überleben. Diese Kapazität kann in vitro mit dem anchorage-unabhängigen Assay mit weichem Agar ausgewertet werden. Darüber hinaus ist ein inhärentes Merkmal von Tumorgeweben ihre anhaltende proliferative Signalfähigkeit, die leicht unter In-vitro-Bedingungen überwacht werden kann, indem der Anstieg der Zellzahl entlang der Zeit gemessen wird, nicht nur in Suspensionstests, sondern auch durch die Überwachung der Wachstumsrate von monolayer-anhaftenden Kulturen.
Trotz des besten Modells, um tumorigenic Potenzial zu testen ist die Impfung von Tumorzellen in murinen Modellen und Bewertung der Tumorentwicklung in situ, ist es wichtig, die Anzahl der Tiere in experimentellen Verfahren so weit wie möglich verwendet zu minimieren. Daher hat die Bewertung der Transformation in vitro geeignete Tests oberste Priorität. Hier stellen wir eine Reihe von Werkzeugen zur Verfügung, um das tumorgene Potenzial von teilweise und vollständig transformierten Brustepithelzellen zu bewerten, die leicht in den meisten Laboratorien implementiert werden können, die mit zellulären Transformationsmodellen arbeiten.
Protokoll
Menschliche Proben, die in den folgenden Experimenten verwendet wurden, wurden aus Reduktionsmammoplastiken gewonnen, die in der Clénica Pilar Sant Jordi (Barcelona) im Rahmen der Standardprozedur-Genehmigung durchgeführt wurden. Sofern nicht anders angegeben, werden alle Verfahren in einem biologischen Sicherheitskabinett der Klasse II durchgeführt.
1. In-vitro-Kultur der menschlichen Brustepithelzellen und Wachstumskurven-Plot-Aufbau
-
In-vitro-Kultur der primären Epithelzellen der Brust (BPECs): Zellpassaging
HINWEIS: Für BPEC-Ableitung und Zellkultur folgen Sie den Anweisungen von Ince et al., 20076.- Mittlere Vorbereitung.
- Ergänzung WIT basal definiertes Medium mit P oder T Ergänzungen, vom Hersteller zur Verfügung gestellt, je nachdem, ob primäre oder transformierte BPECs kultiviert sind.
- Fügen Sie dem ergänzten WIT-Medium Choleratoxin zu einer Endkonzentration von 100 ng/ml für primäre oder 25 ng/ml für transformierte BPECs hinzu.
VORSICHT: Cholera-Toxin ist tödlich, wenn es geschluckt wird. Verwenden Sie persönliche Schutzausrüstung. Vermeiden Sie die Freigabe in die Umgebung.
- Zellkulturpflege und Passaging.
HINWEIS: Bei den folgenden Schritten sollten Sie bedenken, dass Zellen in einem T25-Kolben wachsen. Dennoch können Volumen an andere Zellkulturformate angepasst werden, wobei die Proportionalität in Bezug auf die Oberfläche erhalten bleibt.- Überprüfen Sie die Zellkonfluency jeden Tag. Wenn die Kultur zu 90 % konfluent ist, führen Sie die Zelldurchlaufung durch.
- Erwerben Sie 1x PBS, 3x Trypsin, Medium und ein 15 ml kegelförmiges Rohr mit 2 ml Fetal Bovine Serum (FBS) für jeden Kolben.
- Entfernen Sie das Medium aus dem Kolben und bewahren Sie es in dem 15 ml konischen Rohr auf, das FBS enthält.
- Spülen Sie Zellen mit 1x PBS.
- Trennen Sie Zellen von der Oberfläche, indem Sie 1 ml 3x Trypsin hinzufügen. 5 min bei 37 °C inkubieren.
- Überprüfen Sie, ob Zellen getrennt wurden. Tragen Sie kräftiges Schütteln auf, wenn die Zellen nicht vollständig getrennt sind.
- Inaktivieren Sie Trypsin, indem Sie das reservierte Medium hinzufügen, das mit FBS ergänzt wird.
- Die Zellsuspension ernten und in das 15 ml konische Rohr legen.
- Zentrifuge bei 500 x g für 5 min, beseitigen Sie den Überstand, und setzen Sie die pelletierten Zellen durch Streichen der Unterseite des Rohres mit einem Finger.
- Fügen Sie dem Pellet 1–2 ml frische Medien hinzu und messen Sie die Zellkonzentration mit einem automatischen Zellzähler oder einem Hämozytometer. Diese Daten werden später verwendet, um die Verdoppelungen der Bevölkerung zu berechnen und die Wachstumskurve zu zeichnen. Samen 12.000 Zellen/cm2 (z. B. 300.000 Zellen für einen T25-Kolben) in modifizierten Zellkultur-Oberflächenkolben (siehe Materialtabelle).
HINWEIS: Verdünnen Sie die Zellsuspensionslösung, wenn die Konzentration zu hoch ist, um eine ordnungsgemäße Quantifizierung zu gewährleisten. - Fügen Sie mittlere zu einem Endvolumen von 5 ml hinzu und inkubieren Sie die Zellen bei 37 °C und 5%CO2-Atmosphäre.
- Ersetzen Sie das Zellkulturmedium alle 48 h.
- Mittlere Vorbereitung.
-
Berechnung der Bevölkerungsverdoppelung und Datenvisualisierung
- Verwenden Sie die Daten der Zellenzahl, die in Schritt 1.1.2.10 ermittelt wurde, wenden Sie die folgende Formel an, um die PPD-Werte (ACCUMULATED Population Double) zu erhalten:
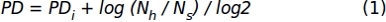
Wobei PDi die Anzahl der von den Zellen erreichten Bevölkerungsverdoppelungen bis zur vorherigen Subkultur angibt (es bezieht sich auf die PD, die in der vorherigen Subkultur angesammelt wurde), ist Nh die Anzahl der geernteten Zellen und Ns die Anzahl der gesäten Zellen. - Stellen Sie Daten für ein bestimmtes Zeitintervall mithilfe eines XY-Diagramms dar, in dem die Anzahl der Tage in der Kultur (x-Achse)und die akkumulierte PD (y-achse) dargestellt werden.
- Holen Sie sich die am besten geeignete Linie und die passende Gleichung:

HINWEIS: Eine erhöhte Steigung (b) bedeutet eine erhöhte Proliferationsrate.
- Verwenden Sie die Daten der Zellenzahl, die in Schritt 1.1.2.10 ermittelt wurde, wenden Sie die folgende Formel an, um die PPD-Werte (ACCUMULATED Population Double) zu erhalten:
2. Dreidimensionale (3D) Kultur in der Kellermembranmatrix und immunfluoreszierender Proteindetektion
- 3D-Kultur in Derkellermembranmatrix
HINWEIS: Dieses Protokoll wurde von Debnath et al., 200310 angepasst und ist für 24 Wellplatten optimiert (siehe Tabelle der Materialien).- Bereiten Sie das Material einen Tag vor dem Experiment vor: Kellermembranmatrix über Nacht bei 4 °C vorkühlen und Pipettenspitzen, Mikrozentrifugenrohre und Brunnenplatten im Gefrierschrank abkühlen lassen.
HINWEIS: Die Matrix muss zur Langzeitlagerung bei -20 °C gehalten werden. Machen Sie Aliquots, um mehrere Frost-Tau-Zyklen zu vermeiden. - Am Tag des Experiments vorgekühltes Material auf Eis legen.
- Spülen Sie Brunnen mit kaltsterilen 1x PBS, um Oberflächenspannung zu reduzieren.
- Bedecken Sie den Boden jedes Brunnens mit 100 l Kellermembranmatrix.
ANMERKUNG: Geben Sie die Matrix langsam und verteilen Sie sie im gesamten Brunnen; es ist wichtig, Blasenbildung in der unteren Schicht zu vermeiden, um monolayer ZellkulturWachstum zu verhindern. - Legen Sie die Platte bei 37 °C in den Inkubator, um die Matrixschicht verfestigen zu lassen.
HINWEIS: Es dauert in der Regel etwa 20 min zu erstarren. - In der Zwischenzeit, Trypsinize Zellen, wie zuvor in Schritt 1.1 erklärt. Zentrifugieren Sie die Zellen bei 500 x g für 5 min und resuspendieren Sie im Medium. Bereiten Sie eine 400.000 Zellen/ml Suspension vor und disaggregieren Sie jeden Zellklumpen durch Pipettieren.
- Bereiten Sie das Medium mit 8% Kellermembranmatrix vor und mischen Sie 1:1 (v/v) mit zellulärer Suspension, um eine 200.000 Zellen/ml-Lösung in 4% Matrix zu erhalten.
HINWEIS: Berechnen Sie die Menge an Medium benötigt, um Matrix unnötige Abfälle zu vermeiden. - Legen Sie 500 l Zellsuspension in Matrixlösung auf die bereits erstarrte Matrixschicht, um eine Gesamtmenge von 100.000 Zellen in Medium mit 4% Kellermembranmatrix auszusäen.
- Inkubieren Sie Zellen bei 37 °C für ein paar Minuten und fügen Sie dann 500 l des Mediums mit 4% Kellermembranmatrix hinzu. Inkubieren Sie die Zellen bei 37 °C in einem Inkubator mit 5%CO2 für 14 Tage. Gesäte Zellen gruppieren sich und vermehren sich, um die acini-ähnlichenStrukturen zu entstehen.
HINWEIS: Die Zellmotilität und Aggregation kann durch Zeitraffer während des 3D-Bildungsprozesses überwacht werden. Verwenden Sie Bildanalysesoftware (z. B. Fidschi/ImageJ oder Imaris), um diese Ereignisse auszuwerten. Anzahl und Größe der Acini hängen vom Aggregationsprozess und der Proliferationsrate ab und können je nach Zelltyp variieren. Passen Sie die Konzentration der Kellermembranmatrix und der gesäten Zellen an, um gewünschte 3D-Strukturen zu erhalten. - Fügen Sie 2-3 Mal pro Woche 500 l des Mediums mit 4% Kellermembranmatrix hinzu.
HINWEIS: Vermeiden Sie Störungen der Schichten, die die Platte während der Manipulation sanft tragen. - Falls gewünscht, können Anzahl und Größe der Acini während der Kulturperiode gemessen werden. Nehmen Sie dazu zu verschiedenen Zeiten nach der Aussaat mit einem Phasenkontrast oder einem invertierten DIC-Mikroskop zufällige Bilder auf. Verwenden Sie Bildanalysesoftware, um den Durchmesser von 100–200 3D-Strukturen zu messen.
- Bereiten Sie das Material einen Tag vor dem Experiment vor: Kellermembranmatrix über Nacht bei 4 °C vorkühlen und Pipettenspitzen, Mikrozentrifugenrohre und Brunnenplatten im Gefrierschrank abkühlen lassen.
- Immunostaining
HINWEIS: Sterile Bedingungen sind während dieses Teils des Protokolls nicht erforderlich.- Entfernen Sie das Kulturmedium.
- Zerreißen Sie die Kellermembranmatrix mit einer p200 Pipettenspitze mit abgeschnittenem Ende. Legen Sie die disaggregierte Matrix in höhe von 50 l auf eine Glasrutsche und schmieren Sie sie in einem Bereich von 1–2cm2.
- Lassen Sie die Probe bei Raumtemperatur vollständig trocknen oder verwenden Sie eine Heizplatte bei 37 °C, um den Prozess zu beschleunigen. Proben mit Methanol fixieren:Aceton (1:1, v/v) bei -20 °C für 30 min.
HINWEIS: Das fluoreszierende Signal früherer Marker, wie das von den Zellen exprimierte fluoreszierende Proteine, wird ausgegart.
VORSICHT: Methanol ist entzündlich, giftig beim Einatmen, Verschlucken oder bei Hautkontakt. Tragen Sie persönliche Schutzausrüstung und arbeiten Sie in einer Dunstabzugshaube. - Verwerfen Sie die Fixierungslösung, und entfernen Sie ggf. den Überschuss, indem Sie die Folie auf Filterpapier legen.
HINWEIS: Das Protokoll kann hier angehalten werden. Nach dem Trocknen können die Dias mehrere Monate bei -20 °C gelagert werden. - Blockproben Epitope mit 5% normalem Ziegenserum und 0,1% Triton-X-100 in 1x PBS (Blockierlösung) für 2 h bei Raumtemperatur.
- In der Zwischenzeit bereiten Antikörper Arbeitslösungen durch Verdünnung primäre oder sekundäre Antikörper in der gewünschten Konzentration in Blockierlösung.
HINWEIS: Die Antikörperkonzentration muss je nach Zelltyp und Antikörperreferenz genau angepasst werden. Als Leitfaden können Zellen aus luminalen und myoepithelialen Linien in BPEC, primäre Anti-Cytokeratin 14 und Anti-Claudin-IV-Antikörper (siehe Materialtabelle) verwendet werden. Die empfohlene Konzentration der Arbeitslösung beträgt 1:100 für diese primären Antikörper und 1:500 für Anti-Maus- und Anti-Rabbit-Sekundärantikörper (siehe Tabelle der Materialien). - Fügen Sie 30 l primäre Antikörper Arbeitslösung hinzu und bedecken Sie sie mit einem Streifen Laborverpackungsfolie, um Verdunstung zu vermeiden. Über Nacht bei 4 °C in einer feuchten Kammer inkubieren.
- Waschen Sie dreimal mit 1x PBS für je 1 h.
- Wiederholen Sie Schritt 2.2.7 für sekundäre Antikörper. Die Inkubation sollte in der Dunkelheit durchgeführt werden.
- Mit 1x PBS für 2 h waschen.
HINWEIS: Passen Sie die Antikörperkonzentration, inkubationszeit und Waschhärte an, um das Signal-Rausch-Verhältnis für bestimmte Proben zu verbessern. - Entfernen Sie die verbleibende PBS und, einmal trocken, Gegenfleck mit DAPI bei 0,25 g/ml in Antifade-Montagemedium verdünnt. Abdeckung strotzt mit einem Deckelschlupf, indem Sie es ohne Druck absetzen lassen. Siegel mit Nagellack.
HINWEIS: Proben können mehrere Wochen bei 4 °C gelagert werden. Für eine langfristige Lagerung bei -20 °C aufbewahren. - Analysieren Sie die fluoreszierende Signalverteilung für jeden Acinus mit einem konfokalen Mikroskop.
HINWEIS: Die Konfiguration des konfokalen Mikroskops muss in Abhängigkeit von der verwendeten Ausrüstung und den Antikörpern, die auf die Probe aufgebracht werden, genau bestimmt werden. Als Leitfaden verwenden Sie mit den in der Tabelle der Materialienaufgeführten Geräten und Reagenzien ein 40-faches Objektiv und die folgenden Laser- und Detektoreinstellungen: für den DAPI-Einsatz anregung mit einem 405-Laser (3%–5%), detektion mit einem PMT-Detektor (800V, Offset: -9) und ein Spektralband von 410 nm bis 500 nm; für A488 (Claudin-IV) Anregung mit einem 488 Laser (7%–10%), Detektion mit einem PMT-Detektor (800V, Offset: -20) und ein Spektralband von 490 nm bis 550 nm; und für Cy3 (Cytokeratin 14) Verwenden Sie Anregung mit einem 555 Laser (2%–10%), Detektion mit einem PMT-Detektor (800V, Offset: -35) und einem Spektralband von 560 nm bis 600 nm.
3. Anchorage-unabhängiger Assay, MTT-Färbung und automatische Koloniequantifizierung
- Anchorage-unabhängiger Assay: Agar und zelluläre Suspensionsbeschichtung
HINWEIS: Das Protokoll wurde von Borowicz et al., 201411 angepasst, um Experimente mit BPECs durchzuführen.- Bereiten Sie eine 1,2% Agar-Lösung in Reinstwasser in einer sterilen Flasche verdünnt. Autoklaven Sie die Lösung und halten Sie sie während des Experiments bei 42 °C. Die Agarlösung kann bei 4 °C gelagert werden; Bei Bedarf die Agarlösung erhitzen, bis sie wieder flüssig ist.
VORSICHT: Verwenden Sie hitzebeständige Handschuhe, um eine Verbrennung nach dem Autoklaven zu vermeiden.
HINWEIS: Von nun an müssen sterile Bedingungen aufrechterhalten werden. - Bereiten Sie eine 0,6% Agar-Lösung vor, indem Sie 1:1 (v/v) komplettes vorgewärmtes Medium mit 1,2% Agar-Lösung mischen. Bei 42 °C aufbewahren, um eine vorzeitige Erstarrung zu vermeiden.
HINWEIS: Medium kann zuvor doppelt ergänzt werden, um eine vollständig ergänzte 0,6% Agar + mittlere Lösung zu erhalten, sobald sie gemischt ist. - Bedecken Sie den Boden eines 35 mm Brunnens mit 1,5 ml 0,6% Agar in mittlerer Lösung und lassen Sie ihn bei Raumtemperatur erstarren. Stellen Sie sicher, dass der Boden der Platte vollständig vor der Agar-Erstarrung bedeckt ist, sonst können Zellen an der Platte haften und in Monolayer wachsen.
HINWEIS: Es können haftende und nicht haftende Oberflächenplatten verwendet werden. - In der Zwischenzeit versuchen, Zellen zu trypsinisieren und, einmal zentrifugiert und in Medium resuspendiert, bereiten eine 50.000 Zellen / ml Lösung und leicht disaggregieren jede Zellklumpen durch Pipetten wiederholt.
- Bereiten Sie eine 0,3% Agar + Zellsuspension im Medium bei einer Endkonzentration von 25.000 Zellen/ml vor.
HINWEIS: Die optimale Zellkonzentration kann sich je nach Zelltyp unterscheiden. Probieren Sie verschiedene Konzentrationen aus, bis individualisierte Kolonien gebildet werden.- Legen Sie einen 40-mm-Siebfilter auf ein 50 ml steriles Rohr und filtern Sie die 50.000-Zellen/ml-Lösung, die sie in den Boden des Rohres fallen lässt.
- Entfernen Sie den Filter aus dem 50 ml sterilen Rohr, neigen Sie das zellhaltige Rohr in einen 45°-Winkel und lassen Sie das gleiche Volumen von 0,6% Agar + Medium Lösung gießen es durch die Innenwand des Rohres. Dadurch kann die Agar-Lösung gerade genug abkühlen, um die Zellen nicht zu beschädigen und ihre vorzeitige Erstarrung zu vermeiden.
- Homogenisieren Sie das Gemisch und legen Sie 1 ml 0,3% Agar + Zellsuspension im Medium (mit 25.000 Zellen) auf der zuvor erstarrten unteren Agarschicht ab.
- Visualisieren Sie gesäte Zellen mit einem invertierten Mikroskop, um sicherzustellen, dass die Zellen individualisiert werden. Andernfalls sollte das Experiment wiederholt werden.
- Warten Sie, bis die Agarschicht vollständig verfestigt ist, und fügen Sie dann vorsichtig 1 ml des frischen Mediums oben hinzu, ohne die darunter liegenden empfindlichen Agarschichten zu stören.
- Inkubieren Sie die Zellen 3 Wochen lang bei 37 °C und 5%CO2 in einem Inkubator.
HINWEIS: Die zeitabhängige Zeit für die Koloniebildung kann zwischen verschiedenen Zelltypen variieren, aber in der Regel sind 3 Wochen ausreichend. - Wechseln Sie das Medium zweimal pro Woche. Um dies zu tun, neigen Sie die Platte vorsichtig zu Ihnen, aspirieren Medium in der unteren Ecke, und fügen Sie 1 ml frisches Medium.
HINWEIS: Vermeiden Sie es, die Agarschichten zu berühren, da sie sich leicht von der Platte lösen.
- Bereiten Sie eine 1,2% Agar-Lösung in Reinstwasser in einer sterilen Flasche verdünnt. Autoklaven Sie die Lösung und halten Sie sie während des Experiments bei 42 °C. Die Agarlösung kann bei 4 °C gelagert werden; Bei Bedarf die Agarlösung erhitzen, bis sie wieder flüssig ist.
- MTT Färbung
- Bereiten Sie Thiazolyl Blue Tetrazolium Bromid (MTT) Stofflösung bei 6 mg/ml in Reinstwasser in einer sterilen Flasche und Filterlösung mit 0,2 m Filtern vor. Diese MTT-Lösung kann bis zu 6 Monate bei -20 °C gelagert werden.
VORSICHT: MTT kann Reizungen verursachen und steht im Verdacht, genetische Defekte zu verursachen. Verwenden Sie Eine Schutzbrille, Handschuhe und einen Atemfilter.
HINWEIS: Vermeiden Sie wiederholte Frost-Tau-Zyklen. - Bereiten Sie eine Arbeitslösung von MTT bei 1 mg/ml vor, indem Sie die Stammlösung mit sterilem Reinstwasser verdünnen.
- Sobald die Koloniebildungsphase abgeschlossen ist, entfernen Sie das Medium von der Platte und fügen Sie 1 ml von 1 mg/ml MTT zu jedem Brunnen hinzu.
- 24 h im Inkubator inkubieren. Entfernen Sie die MTT-Lösung, indem Sie sie vorsichtig anheben. Die Platten können mehrere Wochen bei 4 °C gelagert werden.
HINWEIS: Vermeiden Sie Lichteinwirkung, um unspezifische Kristallbildung zu verhindern.
- Bereiten Sie Thiazolyl Blue Tetrazolium Bromid (MTT) Stofflösung bei 6 mg/ml in Reinstwasser in einer sterilen Flasche und Filterlösung mit 0,2 m Filtern vor. Diese MTT-Lösung kann bis zu 6 Monate bei -20 °C gelagert werden.
- Koloniequantifizierung
- Erhalten Sie Bilder jeder Platte mit einem invertierten Mikroskop. Passen Sie die Vergrößerung an, um das maximale Sichtfeld mit weniger Bildern zu erfassen und dennoch kleine Kolonien zu erkennen (in der Regel 4x oder 10x Objektive).
HINWEIS: Stellen Sie sicher, dass Die Bilder einen homogenen Hintergrund haben. Weder Phasenkontrast noch Differentialinterferenzkontrast sind erforderlich, da nicht gefärbte Kolonien nicht quantifiziert werden. - Laden Sie Bilder auf ImageJ/Fiji Software12 hoch, um die Anzahl der Kolonien und das Gebiet jeder MTT-positiven Kolonie zu zählen.
HINWEIS: Ein Skript für die automatische Quantifizierung wird als Zusatzdateibereitgestellt. Um den Code auszuführen, fügen Sie ihn in den Makro-Editor(Plugins | Neue | Makro) und folgen Sie den Anweisungen.- Erhalten Sie eine binäre Maske durch Schwellenwerte für das Originalbild(Bild | Anpassen | Schwellenwert), um gut abgegrenzte Kolonien zu erhalten (Abbildung 1).
HINWEIS: Für diesen Schritt sind in der Regel 8- oder 16-Bit-Bilder erforderlich. Die Methode "Mindestschwellenwert" wird empfohlen. - Führen Sie den Extended Particle Analyzer von Biovoxxel plugin13 (Plugins | BioVoxxel | Erweiterte Partikelanalysator) zur Identifizierung von MTT-positiven Kolonien (Abbildung 2). Anfangsbedingungen: Größe (m2) = 250–Unendlichkeit; Festigkeit = 0,75–1,00.
- Erhalten Sie eine binäre Maske durch Schwellenwerte für das Originalbild(Bild | Anpassen | Schwellenwert), um gut abgegrenzte Kolonien zu erhalten (Abbildung 1).
- Schätzen Sie den durchschnittlichen Durchmesser (D) von jedem Kolonieflächenwert (A) nach der Formel:

- Filterergebnisse durch Ausschluss von kolonien niedrigen Proliferativen (z. B. niedrigem Durchmesser).
- Wählen Sie eine Mindestanzahl von Divisionen pro Woche (m), die berücksichtigt werden sollen (z. B. 1).
- Schätzen Sie den Radius einer Kolonie (R) mit n Zellen nach der folgenden Formel:

Wobei r der durchschnittliche Radius einzelner Zellen in Suspension ist, n ist die Anzahl der Zellen, die eine Kolonie bilden, die jede Woche während w Wochen in kultur unter m Teilungen litt. Bei exponentiellem Wachstum: n = 2(m*w). • ist die Verpackungseffizienz. Beachten Sie, dass bei zufälliger Bewegung die Verpackungseffizienz 0,64 USD beträgt und die dichtestmögliche Verpackungsfraktion für identische Kugeln 0,7414beträgt. - Entsorgen Sie alle Kolonien, die einen Durchmesser von weniger als 2R aufweisen, da ihre Zellen nicht die Mindestanzahl der in Schritt 3.3.4.1 berücksichtigten Teilungen erreicht haben.
- Erhalten Sie Bilder jeder Platte mit einem invertierten Mikroskop. Passen Sie die Vergrößerung an, um das maximale Sichtfeld mit weniger Bildern zu erfassen und dennoch kleine Kolonien zu erkennen (in der Regel 4x oder 10x Objektive).
Ergebnisse
Ein experimentelles Modell der zellulären Transformation mit der Einführung von drei genetischen Elementen in BPECs wurde gewählt, um repräsentative Ergebnisse der onkogenen Transformation zu generieren6,7 ( Abbildung3). Nicht transformierte BPECs (N) wurden aus krankheitsfreiem Brustgewebe abgeleitet, wie von Ince und Kollegen6 beschrieben und gemäß dem hier angegebenen Protoko...
Diskussion
Die in diesem Papier beschriebenen experimentellen Protokolle bieten nützliche Instrumente zur Bewertung der onkogenen Transformation von In-vitro-kulturierten Zellen. Jede Technik bewertet bestimmte Aspekte des Transformationsprozesses, und daher muss bei der Schlussfolgerungausarbeit aus einer einzigen Analyse besondere Aufmerksamkeit geschenkt werden. Wachstumskurven-Aufbau ist ein Ansatz, der Informationen erfordert, die bereits verfügbar sind, wenn Zellen für andere Zwecke kultiviert werden. Das macht diese Techn...
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Das Labor der AG wird vom spanischen Rat für nukleare Sicherheit finanziert. T.A. und A.G. sind Mitglieder einer von der Generalitat de Catalunya (2017-SGR-503) anerkannten Forschungsgruppe. MT hat einen Vertrag, der von der Wissenschaftlichen Stiftung Asociacion Espaéola Contra el Céncer [AECC-INVES19022TERR] finanziert wird. Der G.F.-Vertrag wird durch einen Zuschuss der Cellex Foundation finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| 1 ml Serological Pipettes | Labclinics | PLC91001 | |
| 1.5 ml Eppendorfs | Thermo Fisher Scientific | 3451 | Dark eppendorfs are preferred for MTT long-term storage |
| 10 μl Pipette tips w/o filter | Biologix | 20-0010 | |
| 100 ml glass bottle | With cap, autoclavable | ||
| 1000 μl Pipette tips w/ filter | Labclinics | LAB1000ULFNL | |
| 1000 μl Pipette tips w/o filter | Biologix | 20-1000 | |
| 15 ml Conical tubes | VWR | 525-0400 | |
| 2 ml Serological Pipettes | Labclinics | PLC91002 | |
| 200 μl Pipette tips w/ filter | Labclinics | FTR200-96 | |
| 5 ml Serological Pipettes | Labclinics | PLC91005 | |
| 50 ml Conical Tubes | VWR | 525-0304 | |
| Acetone | PanReac AppliChem | 211007 | Used for 3D structure fixation prior to immunofluorescent labelling |
| Agar | Sigma-Aldrich | A1296 | Used for anchorage assay |
| Anti-Claudin 4 antibody | Abcam | 15104, RRID:AB_301650 | Working dilution 1:100, host: rabbit |
| Anti-Cytokeratin 14 [RCK107] antibody | Abcam | 9220, RRID:AB_307087 | Working dilution 1:100, host: mouse |
| Anti-mouse Cyanine Cy3 antibody | Jackson ImmunoResearch Inc. | 115-165-146, RRID:AB_2338690 | Working dilution 1:500, host: goat |
| Anti-rabbit Alexa Fluor 488 antibody | Thermo Fisher Scientific | A-11034, RRID:AB_2576217 | Working dilution 1:500, host: goat |
| Autoclave | |||
| BioVoxxel Toolbox | RRID:SCR_015825 | ||
| Cell culture 24-well Plate | Labclinics | PLC30024 | Used for 3D cultures in Matrigel. Flat Bottom |
| Cell culture 6-well Plate | Labclinics | PLC30006 | Used for anchorage assay |
| Cell incubator (37 ºC and 5 % CO2) | |||
| Cell Strainers | Fisherbrand | 11587522 | Mesh size: 40 μm |
| CellSense software | Olympus | Used to image acquisition | |
| Centrifuge | |||
| Cholera Toxin from Vibrio cholerae | Sigma-Aldrich | C8052 | Used to supplement cell culture medium |
| Class II Biological Safety Cabinet | Herasafe | HAEREUS HS12 | |
| Confocal inverted Microscope | Leica | TCS SP5 | |
| Cover glasses | Witeg Labortechnik GmbH | 4600122 | 22 X 22 mm, thickness 0.13 - 0.17 mm |
| DAPI | 2-(4-amidinophenyl)-1H -indole-6-carboxamidine | ||
| Fetal Bovine Serum | Biowest | S1810 | Used to inactivate trypsine action |
| Fiji software (ImageJ) | National Institutes of Health | RRID:SCR_002285 | Free download, no license needed |
| Glass Pasteur Pipettes | |||
| Glass slides | Fisherbrand | 11844782 | |
| Goat Serum | Biowest | S2000 | Used for immunofluorescence of 3D structures |
| Heat-Resistant Gloves | Used for agar manipulation after autoclave | ||
| Heater bath (37 ºC) | Used to temper solutions prior to cell subculture | ||
| Heater bath (42 ºC) | Used to keep agar warm | ||
| Heating plate | Used for Matrigel dehydration | ||
| Humid chamber | Used for the incubation of antibodies during immunofluorescence | ||
| Ice | Used during Matrigel manipulation | ||
| Ice-box | |||
| Inverted Optic Microscope | Olympus | IX71 | |
| Matrigel Matrix | Becton Dickinson | 354234 | Store at -20 ºC and keep cold when in use. Referred to as basement membrane matrix |
| Methanol | PanReac AppliChem | 131091 | Used for 3D structure fixation prior to immunofluorescent labelling |
| Micropipette | p1000, p200 and p10 | ||
| Microsoft Office Excel | Microsoft | RRID:SCR_016137 | Used to calculate population doubling and to obtain growth rate equation |
| MilliQ water | Referred to as ultrapure water | ||
| Nail Polish | Used to seal samples after mounting | ||
| Parafilm M | Bemis | PM-999 | Used to cover antibody solution during incubation |
| PBS pH 7.4 (w/o calcium & magnesium) | Gibco | 10010-056 | Sterile. Used for cell subculture |
| PBS tablets | Sigma-Aldrich | P4417 | Dilute in milliQ water. No sterility required. Used for immunofluorescence |
| Pipette Aid | |||
| Primaria T25 flasks | Corning | 353808 | Used for BPEC culture |
| Scepter Automated Cell Counter | Millipore | PHCC20060 | Alternatively, use an haemocytometer |
| Scissors | Used to cut pipette tips and parafilm | ||
| Sterile filters 0.22 μm | Millipore | SLGP033RS | Used to filter MTT solution |
| Thiazolyl Blue Tetrazolium Bromide (MTT) | Sigma-Aldrich | M2128 | Store at -20 ºC |
| Triton X-100 | Sigma-Aldrich | T8787 | Used for immunofluorescence of 3D structures |
| Trypsin-EDTA 10X | Biowest | X0930 | Dilute in PBS to obtain 3X solution |
| Vectashield Antifade Mounting Medium | Vector Laboratories | H-1000 | |
| WIT-P-NC Culture Medium | Stemgent | 00-0051 | Used for primary BPEC culture |
| WIT-T Culture Medium | Stemgent | 00-0047 | Used for transformed BPEC culture |
Referenzen
- Hanahan, D., Weinberg, R. A. Hallmarks of cancer: the next generation. Cell. 144 (5), 646-674 (2011).
- Stampfer, M. R., Yaswen, P. Culture models of human mammary epithelial cell transformation. Journal of Mammary Gland Biology and Neoplasia. 5 (4), 365-378 (2000).
- Schinzel, A. C., Hahn, W. C. Oncogenic transformation and experimental models of human cancer. Frontiers in Bioscience : A Journal and Virtual Library. 13 (13), 71 (2008).
- Balani, S., Nguyen, L. V., Eaves, C. J. Modeling the process of human tumorigenesis. Nature Communications. 8 (1), 15422 (2017).
- Bray, F., et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA: A Cancer Journal for Clinicians. 68 (6), 394-424 (2018).
- Ince, T. A., et al. Transformation of different human breast epithelial cell types leads to distinct tumor phenotypes. Cancer Cell. 12 (2), 160-170 (2007).
- Repullés, J., et al. Radiation-induced malignant transformation of preneoplastic and normal breast primary epithelial cells. Molecular Cancer Research. , 1-13 (2019).
- Weigelt, B., Bissell, M. J. Unraveling the microenvironmental influences on the normal mammary gland and breast cancer. Seminars in Cancer Biology. 18 (5), 311-321 (2008).
- Paoli, P., Giannoni, E., Chiarugi, P. Anoikis molecular pathways and its role in cancer progression. Biochimica et Biophysica Acta. 1833 (12), 3481-3498 (2013).
- Debnath, J., Muthuswamy, S. K., Brugge, J. S. Morphogenesis and oncogenesis of MCF-10A mammary epithelial acini grown in three-dimensional basement membrane cultures. Methods. 30 (3), 256-268 (2003).
- Borowicz, S., et al. The soft agar colony formation assay. Journal of Visualized Experiments. (92), (2014).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Brocher, J. The BioVoxxel Image Processing and Analysis Toolbox. European BioImage Analysis Symposium. 8 (2), 67112 (2015).
- Torquato, S., Truskett, T. M., Debenedetti, P. G. Is random close packing of spheres well defined. Physical Review Letters. 84 (10), 2064-2067 (2000).
- LaBarge, M. A., Garbe, J. C., Stampfer, M. R. Processing of human reduction mammoplasty and mastectomy tissues for cell culture. Journal of Visualized Experiments. (71), (2013).
- Zubeldia-Plazaola, A., et al. Glucocorticoids promote transition of ductal carcinoma in situ to invasive ductal carcinoma by inducing myoepithelial cell apoptosis. Breast Cancer Research. 20 (1), 65 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten