É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Avaliação In Vitro da Transformação Oncogênica em Células Epiteliais Mamárias Humanas
Neste Artigo
Resumo
Este protocolo fornece ferramentas in vitro experimentais para avaliar a transformação das células mamárias humanas. São descritas etapas detalhadas para a taxa de proliferação celular de acompanhamento, capacidade de crescimento independente de ancoragem e distribuição de linhagens celulares em culturas 3D com matriz de membrana de porão.
Resumo
Tumorigênese é um processo de várias etapas no qual as células adquirem capacidades que permitem seu crescimento, sobrevivência e disseminação em condições hostis. Diferentes testes buscam identificar e quantificar essas marcas de células cancerosas; no entanto, muitas vezes eles se concentram em um único aspecto da transformação celular e, de fato, múltiplos testes são necessários para sua caracterização adequada. O objetivo deste trabalho é fornecer aos pesquisadores um conjunto de ferramentas para avaliar a transformação celular in vitro de uma perspectiva ampla, possibilitando assim tirar conclusões sonoras.
Uma ativação de sinalização proliferativa sustentada é a principal característica dos tecidos tumorais e pode ser facilmente monitorada em condições in vitro, calculando o número de duplicações populacionais alcançadas ao longo do tempo. Além disso, o crescimento de células em culturas 3D permite sua interação com células circundantes, assemelhando-se ao que ocorre in vivo. Isso permite a avaliação da agregação celular e, juntamente com a rotulagem imunofluorescente de marcadores celulares distintos, obter informações sobre outra característica relevante da transformação tumoral: a perda da organização adequada. Outra característica notável das células transformadas é sua capacidade de crescer sem apego a outras células e à matriz extracelular, que pode ser avaliada com o ensaio de ancoragem.
Procedimentos experimentais detalhados para avaliar a taxa de crescimento celular, para realizar rotulagem imunofluorescente de marcadores de linhagem celular em culturas 3D e para testar o crescimento celular independente de ancoragem em ágar macio são fornecidos. Essas metodologias são otimizadas para células epiteliais primárias de mama (BPEC) devido à sua relevância no câncer de mama; no entanto, os procedimentos podem ser aplicados a outros tipos de células após alguns ajustes.
Introdução
Múltiplos eventos sucessivos são necessários para o desenvolvimento da neoplasia. Em 2011, Hanahan e Weinberg descreveram 10 capacidades que permitem o crescimento, sobrevivência e disseminação das células transformadas: as chamadas "Marcas do Câncer"1. A metodologia descrita aqui compila três diferentes ferramentas para avaliar a transformação celular in vitro, focando em algumas das características distintas das células tumorais. Essas técnicas avaliam a taxa de proliferação celular, o comportamento das células quando cultivadas em 3D e sua capacidade de formar colônias com independência de ancoragem.
Modelos celulares são cruciais para testar hipóteses in vitro. Diferentes abordagens foram desenvolvidas para gerar modelos experimentais de transformação celular para o estudo do câncer2,3,4. Como o câncer de mama é o câncer mais comum entre as mulheres em todo o mundo e é responsável por aproximadamente 15% das mortes por câncer entre mulheres5, fornecer modelos celulares adequados de células epiteliais mamárias é de extrema importância para uma investigação mais aprofundada. Neste artigo, ilustramos o potencial de três técnicas para avaliar a transformação celular utilizando um modelo experimental de transformação de Células Epiteliais Primárias de Mama (BPECs) inicialmente descritas pelo Ince e colegas em 20076 e posteriormente implementadas em nosso laboratório7. Este modelo experimental baseia-se na alteração sequencial de três genes-alvo (SV40 Large T e pequenos antígenos t aqui referidos como Ttag, hTERT, e HRAS) ao genoma de BPECs não transformados. Além disso, o método utilizado para a derivação de BPECs favorece a manutenção de células epiteliais mamárias com marcadores luminosos ou mioepitheliais, resultando em uma cultura celular heterogênea que retém alguns dos traços fisiológicos da glândula mamária.
Na glândula mamária, as células epiteliais amamárias luminais, responsáveis pela produção do leite, estão localizadas perto do lúmen, enquanto as células mioepiteliais são dispostas ao redor de células luminais e cuidam dos movimentos de contração que levam o leite ao mamilo. A perda de organização adequada entre essas linhagens celulares é uma característica da transformação tumoral8 que pode ser avaliada in vitro após a detecção imunofluorescente de marcadores de linhagem distintivos em culturas celulares 3D. Outra característica importante das células tumorais é sua capacidade de crescer sem apego a outras células e à matriz extracelular1. Quando as células saudáveis são forçadas a crescer em suspensão, mecanismos como anoikis \u2012 um tipo de morte celular induzida em resposta ao descolamento da matriz extracelular \u2012 são ativados9. A evasão da morte celular é uma das marcas distintas do câncer e, portanto, as células transformadas são capazes de inativar anoikis e sobreviver de forma independente da âncora. Esta capacidade pode ser avaliada in vitro com o ensaio independente de ancoragem usando ágar macio. Além disso, uma característica inerente dos tecidos tumorais é sua capacidade de sinalização proliferativa sustentada, que pode ser facilmente monitorada em condições in vitro medindo o aumento do número celular ao longo do tempo, não apenas em ensaios de suspensão, mas também monitorando a taxa de crescimento das culturas aderentes de monocamadas.
Apesar do melhor modelo para testar o potencial tumorigênico é a inoculação de células tumorais em modelos murinos e a avaliação do desenvolvimento do tumor in situ, é importante minimizar o número de animais empregados em procedimentos experimentais o máximo possível. Portanto, ter testes adequados para avaliar a transformação in vitro é uma prioridade máxima. Aqui, fornecemos um conjunto de ferramentas para avaliar o potencial tumorigênico de células epiteliais mamárias parcial e totalmente transformadas que podem ser facilmente implementadas na maioria dos laboratórios que trabalham com modelos de transformação celular.
Protocolo
Amostras humanas utilizadas nos seguintes experimentos foram obtidas a partir de mamografias de redução realizadas na Clínica Pilar Sant Jordi (Barcelona) sob o consentimento do procedimento padrão. Todos os procedimentos são realizados em um Gabinete de Segurança Biológica classe II, a menos que seja declarado em contrário.
1. Cultura in vitro de células epiteliais mamárias humanas e acúmulo de parcelas de curva de crescimento
-
Cultura in vitro de células epiteliais primárias mamárias (BPECs): passagem celular
NOTA: Para a derivação bpec e cultura celular siga instruções descritas por Ince et al., 20076.- Preparação média.
- Suplemento de peso basal definido com suplementos P ou T, fornecidos pelo fabricante, dependendo se os BPECs primários ou transformados são cultivados.
- Adicione a toxina da cólera ao meio wit suplementado a uma concentração final de 100 ng/mL para BPECs primários ou 25 ng/mL para BPECs transformados.
ATENÇÃO: A toxina da cólera é fatal se engolida. Use equipamento de proteção individual. Evite sua liberação para o meio ambiente.
- Manutenção de cultura celular e passagem.
NOTA: Para as seguintes etapas tenha em mente que as células estão crescendo em um frasco T25. No entanto, os volumes podem ser adaptados a outros formatos de cultura celular mantendo a proporcionalidade em termos de área de superfície.- Verifique a confluência celular todos os dias. Quando a cultura é 90% confluente, realize a passagem celular.
- Adquira 1x PBS, trippsina 3x, médio e um tubo cônico de 15 mL contendo 2 mL de Soro Bovino Fetal (FBS) para cada frasco.
- Retire o meio do frasco e mantenha-o no tubo cônico de 15 mL contendo FBS.
- Enxágüe células com 1x PBS.
- Desprender células da superfície adicionando 1 mL de trippsina 3x. Incubar por 5 min a 37 °C.
- Verifique se as células foram separadas. Aplique um tremor vigoroso se as células não estiverem completamente separadas.
- Inativar trippsina adicionando o meio reservado complementado com FBS.
- Colher a suspensão celular e colocá-la no tubo cônico de 15 mL.
- Centrífuga a 500 x g por 5 min, elimine o supernasce, e resuspenha as células pelotas, apertando a parte inferior do tubo com um dedo.
- Adicione 1-2 mL de mídia fresca à pelota e meça a concentração celular usando um contador automático de células ou um hemócito. Esses dados serão posteriormente utilizados para calcular as duplicações populacionais e desenhar a curva de crescimento. Semente 12.000 células/cm2 (por exemplo, 300.000 células para um frasco T25) em frascos de superfície de cultura celular modificada (ver Tabela de Materiais).
NOTA: Diluir a solução de suspensão celular se a concentração for muito alta para garantir a quantificação adequada. - Adicione o médio a um volume final de 5 mL e incubar as células a 37 °C e 5% de co2 atmosfera.
- Substitua o meio de cultura celular a cada 48 horas.
- Preparação média.
-
Cálculo de duplicação populacional e visualização de dados
- Utilizando dados da contagem celular obtida na etapa 1.1.2.10, aplique a seguinte fórmula para obter os valores de duplicação populacional acumulada (DP):
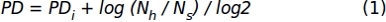
Onde, PDi denota o número de duplicações populacionais alcançadas pelas células até a subcultura anterior (refere-se ao DP acumulado na subcultura anterior), Nh é o número de células colhidas, e Ns é o número de células semeadas. - Represente os dados para um intervalo específico de tempo usando um gráfico XY onde o número de dias na cultura (x-eixo) e o PD acumulado(y-eixo) são representados.
- Obtenha a linha mais adequada e a equação de montagem:

NOTA: Uma inclinação aumentada (b) significa uma taxa de proliferação aumentada.
- Utilizando dados da contagem celular obtida na etapa 1.1.2.10, aplique a seguinte fórmula para obter os valores de duplicação populacional acumulada (DP):
2. Cultura tridimensional (3D) na matriz da membrana do porão e detecção de proteínas imunofluorescentes
- Cultura 3D na matriz de membrana do porão
NOTA: Este protocolo foi adaptado de Debnath et al., 200310 e é otimizado para 24 placas de poço (ver Tabela de Materiais).- Prepare o material um dia antes do experimento: matriz de membrana de porão pré-frio durante a noite a 4 °C e deixe pontas de pipeta, tubos de microcentrifusagem e placas de bem esfriar no congelador.
NOTA: A matriz deve ser mantida a -20 °C para armazenamento a longo prazo. Faça alíquotas para evitar vários ciclos de congelamento. - No dia do experimento, coloque material pré-resfriado no gelo.
- Enxágüe poços com 1x PBS estéril a frio, a fim de reduzir a tensão superficial.
- Cubra a parte inferior de cada poço com 100 μL de matriz de membrana de porão.
NOTA: Dispense a matriz lentamente e espalhe-a por todo o poço; é crucial evitar a formação de bolhas na camada inferior para evitar o crescimento da cultura celular monocamada. - Coloque a placa na incubadora, a 37 °C, para deixar a camada de matriz solidificar.
NOTA: Geralmente leva cerca de 20 minutos para solidificar. - Enquanto isso, células de trypsinize como explicado anteriormente na etapa 1.1. Centrifugar as células a 500 x g por 5 min e resuspend em médio. Prepare uma suspensão de 400.000 células/mL e desagrega suavemente qualquer aglomerado de células por pipetação.
- Prepare o meio com matriz de membrana de 8% do porão e misture 1:1 (v/v) com suspensão celular para obter uma solução de 200.000 células/mL em matriz de 4%.
NOTA: Calcule a quantidade de meio necessário para evitar resíduos desnecessários da matriz. - Coloque 500 μL de suspensão celular na solução matricial em cima da já solidificada camada matricial para semear uma quantidade total de 100.000 células em média com matriz de membrana de 4% porão.
- Incubar células a 37 °C por alguns minutos e, em seguida, adicionar 500 μL do meio com 4% de matriz de membrana de porão. Incubar as células a 37 °C em uma incubadora com 5% de CO2 durante 14 dias. As células semeadas se agruparão e proliferarão para originar as estruturas semelhantes à acini.
NOTA: A motilidade celular e a agregação podem ser monitoradas por lapso de tempo durante o processo de formação 3D. Use software de análise de imagem (por exemplo, Fiji/ImageJ ou Imaris) para avaliar esses eventos. O número e o tamanho do acini dependem do processo de agregação e da taxa de proliferação e podem variar entre os tipos celulares. Ajuste a concentração da matriz da membrana do porão e as células semeadas para obter estruturas 3D desejadas. - Adicione 500 μL do meio com matriz de membrana de 4% de porão 2-3 vezes por semana.
NOTA: Evite a perturbação das camadas que carregam a placa suavemente durante a manipulação. - Se desejar, o número e o tamanho do acini podem ser medidos durante o período cultural. Para isso, tire fotos aleatórias em diferentes momentos após a semeadura usando um contraste de fase ou microscópio invertido DIC. Use software de análise de imagens para medir o diâmetro de estruturas 3D 100-200.
- Prepare o material um dia antes do experimento: matriz de membrana de porão pré-frio durante a noite a 4 °C e deixe pontas de pipeta, tubos de microcentrifusagem e placas de bem esfriar no congelador.
- Imunostaining
NOTA: As condições estéreis não são necessárias durante esta parte do protocolo.- Remova o meio de cultura.
- Rasgue a matriz de membrana do porão usando uma ponta de pipeta p200 com a extremidade cortada. Coloque ~50 μL de matriz desagregada em cima de um escorregador de vidro e manche-o em uma área de 1-2 cm2.
- Deixe a amostra secar completamente à temperatura ambiente ou use uma placa de aquecimento a 37 °C para acelerar o processo. Fixar amostras com metanol:acetona (1:1, v/v) a -20 °C por 30 min.
NOTA: O sinal fluorescente de marcadores anteriores, como o das proteínas fluorescentes expressas pelas células, será apagado.
ATENÇÃO: O metanol é inflamável, tóxico se inalado, engolido ou no caso de entrar em contato com a pele. Use equipamentos de proteção individual e trabalhe dentro de um capô de fumaça. - Descarte a solução de fixação e remova o excesso, se houver, reclinando o slide no papel do filtro.
NOTA: O protocolo pode ser pausado aqui. Uma vez secos, os slides podem ser armazenados a -20 °C por vários meses. - Bloqueie amostras de epítopos com soro de cabra 5% normal e triton-X-100 em 1x PBS (solução de bloqueio) por 2h à temperatura ambiente.
- Enquanto isso, prepare soluções de trabalho de anticorpos diluindo anticorpos primários ou secundários na concentração desejada na solução de bloqueio.
NOTA: A concentração de anticorpos deve ser ajustada com precisão dependendo do tipo celular e da referência do anticorpo. Como guia, para identificar células de linhagens luminais e mioepiteliais em BPEC, podem ser utilizados anticorpos anti-Citokeratina 14 primários e anticorpos anti-Claudin-IV (ver Tabela de Materiais). A concentração recomendada de solução de trabalho é de 1:100 para estes anticorpos primários e 1:500 para anticorpos secundários anti-rato e anti-coelho (ver Tabela de Materiais). - Adicione 30 μL de solução de trabalho de anticorpos primários e cubra-o com uma tira de filme de embalagem de laboratório para evitar a evaporação. Incubar durante a noite a 4 °C em uma câmara úmida.
- Lave três vezes com 1x PBS por 1h cada.
- Repita o passo 2.2.7 para anticorpos secundários. A incubação deve ser realizada na escuridão.
- Lave com 1x PBS por 2 h.
NOTA: Ajuste a concentração de anticorpos, o tempo de incubação e a dureza da lavagem para melhorar a relação sinal/ruído para amostras específicas. - Remova o PBS restante e, uma vez seco, contraten com DAPI a 0,25 μg/mL diluído em meio de montagem antifade. Cubra slides com uma mancha de cobertura, deixando-a se acomodar sem aplicar pressão. Sele com esmalte.
NOTA: As amostras podem ser armazenadas a 4 °C durante várias semanas. Para armazenamento a longo prazo, mantenha-os a -20 °C. - Analise a distribuição de sinal fluorescente para cada acinus usando um microscópio confocal.
NOTA: A configuração do microscópio confocal deve ser determinada com precisão, dependendo do equipamento utilizado e dos anticorpos aplicados à amostra. Como guia, com os equipamentos e reagentes detalhados na Tabela de Materiais,utilize um objetivo de 40x e as seguintes configurações de laser e detector: para uso de DAPI excitação com laser de 405 (3%-5%), detecção com detector pmt (800V, Deslocamento: -9) e uma banda espectral de 410 nm a 500 nm; para A488 (Claudin-IV) usam excitação com um laser 488 (7%-10%), detecção com detector pmt (800V, Offset: -20) e uma banda espectral de 490 nm a 550 nm; e para Cy3 (Cytokeratin 14) usam excitação com um laser de 555 nm (2%-10%), detecção com um detector PMT (800V, Offset: -35) e uma banda espectral de 560 nm a 600 nm.
3. Ensaio independente de ancoragem, coloração MTT e quantificação automática da colônia
- Ensaio independente de ancoragem: ágar e suspensão celular emplacando
NOTA: O protocolo foi adaptado de Borowicz et al., 201411 para realizar experimentos em BPECs.- Prepare uma solução de 1,2% de ágar diluída em água ultrauso em uma garrafa estéril. Autoclave a solução e mantenha-a a 42 °C durante o experimento. A solução de ágar pode ser armazenada a 4 °C; quando necessário, aqueça a solução de ágar até que seja líquida novamente.
ATENÇÃO: Use luvas resistentes ao calor para evitar queimaduras após a autoclave.
NOTA: A partir de agora, as condições estéreis devem ser mantidas. - Prepare uma solução de 0,6% de ágar misturando 1:1 (v/v) meio pré-aquecido completo com solução de 1,2% de ágar. Mantenha-se a 42 °C para evitar a solidificação prematura.
NOTA: O médio pode ser previamente suplementado para obter uma solução de ágar + médio totalmente suplementada de 0,6% uma vez misturada. - Cubra a parte inferior de um poço de 35 mm com 1,5 mL de 0,6% de ágar em solução média e deixe-o solidificar à temperatura ambiente. Certifique-se de que a parte inferior da placa está completamente coberta antes da solidificação do ágar, caso contrário, as células podem aderir à placa e crescer em monocamada.
NOTA: Podem ser utilizadas placas de superfície aderentes e não aderentes. - Enquanto isso, células de trypsinize e, uma vez centrifadas e resuspendidas em médio, preparem uma solução de 50.000 células/ml e desagreguem suavemente qualquer aglomerado de células por pipetar repetidamente.
- Prepare uma suspensão de 0,3% de ágar + célula no meio em uma concentração final de 25.000 células/mL.
NOTA: A concentração celular ideal pode diferir entre os tipos de células. Tente diferentes concentrações até que colônias individualizadas sejam formadas.- Coloque um filtro de coador de 40 μm em cima de um tubo estéril de 50 mL e filtre a solução de 50.000 células/mL permitindo que ele caia na parte inferior do tubo.
- Remova o filtro do tubo estéril de 50 mL, incline o tubo contendo células para um ângulo de 45° e solte o mesmo volume de 0,6% de solução ágar + média derramando-o através da parede interna do tubo. Isso permitirá que a solução de ágar esfrie o suficiente para não danificar as células e evitar sua solidificação prematura.
- Homogeneize a mistura e deposite 1 mL de 0,3% de suspensão de ágar + célula no meio (contendo 25.000 células) em cima da camada de ágar inferior previamente solidificada.
- Visualize células semeadas usando um microscópio invertido para garantir que as células sejam individualizadas. Caso contrário, o experimento deve ser repetido.
- Aguarde até que a camada de ágar esteja completamente solidificada, em seguida, adicione cuidadosamente 1 mL do meio fresco em cima sem perturbar as delicadas camadas de ágar abaixo.
- Incubar as células a 37 °C e 5% de CO2 em uma incubadora por 3 semanas.
NOTA: O tempo necessário para a formação de colônias pode variar entre diferentes tipos de células, mas geralmente 3 semanas são suficientes. - Troque meio duas vezes por semana. Para isso, incline suavemente a placa em sua direção, aspire meio no canto inferior e adicione 1 mL de meio fresco.
NOTA: Evite tocar nas camadas de ágar à medida que elas se desprendem facilmente da placa.
- Prepare uma solução de 1,2% de ágar diluída em água ultrauso em uma garrafa estéril. Autoclave a solução e mantenha-a a 42 °C durante o experimento. A solução de ágar pode ser armazenada a 4 °C; quando necessário, aqueça a solução de ágar até que seja líquida novamente.
- Coloração MTT
- Prepare a solução de estoque Thiazolyl Blue Tetrazolium Bromide (MTT) a 6 mg/mL em água ultrauso em uma garrafa estéril e solução de filtro usando filtros de 0,2 μm. Esta solução MTT pode ser armazenada por até 6 meses a -20 °C.
ATENÇÃO: A MTT pode causar irritação e é suspeita de causar defeitos genéticos. Use óculos de segurança, luvas e um filtro respiratório.
NOTA: Evite ciclos repetidos de congelamento. - Prepare uma solução de trabalho de MTT a 1 mg/mL diluindo a solução de estoque com água ultrapura estéril.
- Uma vez concluído o período de formação da colônia, remova o meio da placa e adicione 1 mL de 1 mg/mL MTT a cada poço.
- Incubar por 24 h na incubadora. Remova a solução MTT aspirando-a suavemente. As placas podem ser armazenadas a 4 °C por várias semanas.
NOTA: Evite a exposição à luz para evitar a formação de cristais não específicos.
- Prepare a solução de estoque Thiazolyl Blue Tetrazolium Bromide (MTT) a 6 mg/mL em água ultrauso em uma garrafa estéril e solução de filtro usando filtros de 0,2 μm. Esta solução MTT pode ser armazenada por até 6 meses a -20 °C.
- Quantificação da colônia
- Obtenha imagens de cada placa usando um microscópio invertido. Ajuste a ampliação para adquirir o campo máximo de visão com menos imagens e ainda ser capaz de detectar pequenas colônias (geralmente objetivos 4x ou 10x).
NOTA: Certifique-se de que as imagens apresentem um fundo homogêneo. Nem o contraste de fase nem o contraste de interferência diferencial são necessários, uma vez que colônias não manchadas não serão quantificadas. - Envie imagens para o software ImageJ/Fiji12 para contar o número de colônias e a área de cada colônia MTT positivo.
NOTA: Um script para quantificação automática é fornecido como um arquivo suplementar. Para executar o código, cole-o ao editor de macro(Plugins | Nova | Macro) e siga as instruções.- Obtenha uma máscara binária através do limiar da imagem original(Imagem | Ajuste | Limiar) para obter colônias bem delimitadas(Figura 1).
NOTA: Imagens de 8 ou 16 bits geralmente são necessárias para executar esta etapa. Recomenda-se o método "Limiar mínimo". - Execute o Analisador de Partículas Estendidas a partir do plugin Biovoxxel13 (Plugins | | BioVoxxel Analisador de partículas estendidas) para identificar colônias positivas MTT(Figura 2). Condições orientais iniciais: Tamanho (μm2) = 250-Infinito; Solidez = 0,75-1,00.
- Obtenha uma máscara binária através do limiar da imagem original(Imagem | Ajuste | Limiar) para obter colônias bem delimitadas(Figura 1).
- Estime o diâmetro médio (D) de cada valor da área da colônia (A) de acordo com a fórmula:

- Filtrar resultados excluindo colônias de baixa proliferação (por exemplo, baixo diâmetro).
- Escolha um número mínimo de divisões por semana (m) a serem consideradas (por exemplo, 1).
- Estime o raio de uma colônia(R)com células n de acordo com a seguinte fórmula:

Onde, r é o raio médio de células individuais em suspensão, n é o número de células formando uma colônia que sofreu divisões m a cada semana durante w semanas na cultura. Em crescimento exponencial: n = 2(m*w). ρ é a eficiência da embalagem. Note-se que, em movimento aleatório, a eficiência da embalagem é de ~0,64 e a fração de embalagem mais densa possível para esferas idênticas é de 0,7414. - Descarte todas as colônias que apresentem diâmetro inferior ao 2R, pois suas células não atingiram o número mínimo de divisões consideradas na etapa 3.3.4.1.
- Obtenha imagens de cada placa usando um microscópio invertido. Ajuste a ampliação para adquirir o campo máximo de visão com menos imagens e ainda ser capaz de detectar pequenas colônias (geralmente objetivos 4x ou 10x).
Resultados
Um modelo experimental de transformação celular com a introdução de três elementos genéticos em BPECs foi escolhido para gerar resultados representativos de transformação oncogênica6,7 (Figura 3). Os BPECs não transformados (N) foram derivados do tecido mamário livre de doenças, conforme descrito pelo Ince e pelos colegas6 e cultivados seguindo o protocolo aqui indicado. A...
Discussão
Os protocolos experimentais descritos neste artigo fornecem ferramentas úteis para avaliar a transformação oncogênica de células cultivadas in vitro. Cada técnica avalia aspectos específicos do processo de transformação e, portanto, deve-se prestar atenção especial ao tirar conclusões de uma única análise. O acúmulo de curvas de crescimento é uma abordagem que exige informações já disponíveis ao criar células para outros fins. Isso torna essa técnica mais barata e fácil de aplicar em comparação c...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
O laboratório ag é financiado pelo Conselho Espanhol de Segurança Nuclear. T.A. e A.G. são membros de um grupo de pesquisa reconhecido pela Generalitat de Catalunya (2017-SGR-503). A MT detém um contrato financiado pela Fundação Científica Asociación Española Contra el Cáncer [AECC-INVES19022TERR]. O contrato da G.F. é financiado por uma bolsa da Fundação Cellex.
Materiais
| Name | Company | Catalog Number | Comments |
| 1 ml Serological Pipettes | Labclinics | PLC91001 | |
| 1.5 ml Eppendorfs | Thermo Fisher Scientific | 3451 | Dark eppendorfs are preferred for MTT long-term storage |
| 10 μl Pipette tips w/o filter | Biologix | 20-0010 | |
| 100 ml glass bottle | With cap, autoclavable | ||
| 1000 μl Pipette tips w/ filter | Labclinics | LAB1000ULFNL | |
| 1000 μl Pipette tips w/o filter | Biologix | 20-1000 | |
| 15 ml Conical tubes | VWR | 525-0400 | |
| 2 ml Serological Pipettes | Labclinics | PLC91002 | |
| 200 μl Pipette tips w/ filter | Labclinics | FTR200-96 | |
| 5 ml Serological Pipettes | Labclinics | PLC91005 | |
| 50 ml Conical Tubes | VWR | 525-0304 | |
| Acetone | PanReac AppliChem | 211007 | Used for 3D structure fixation prior to immunofluorescent labelling |
| Agar | Sigma-Aldrich | A1296 | Used for anchorage assay |
| Anti-Claudin 4 antibody | Abcam | 15104, RRID:AB_301650 | Working dilution 1:100, host: rabbit |
| Anti-Cytokeratin 14 [RCK107] antibody | Abcam | 9220, RRID:AB_307087 | Working dilution 1:100, host: mouse |
| Anti-mouse Cyanine Cy3 antibody | Jackson ImmunoResearch Inc. | 115-165-146, RRID:AB_2338690 | Working dilution 1:500, host: goat |
| Anti-rabbit Alexa Fluor 488 antibody | Thermo Fisher Scientific | A-11034, RRID:AB_2576217 | Working dilution 1:500, host: goat |
| Autoclave | |||
| BioVoxxel Toolbox | RRID:SCR_015825 | ||
| Cell culture 24-well Plate | Labclinics | PLC30024 | Used for 3D cultures in Matrigel. Flat Bottom |
| Cell culture 6-well Plate | Labclinics | PLC30006 | Used for anchorage assay |
| Cell incubator (37 ºC and 5 % CO2) | |||
| Cell Strainers | Fisherbrand | 11587522 | Mesh size: 40 μm |
| CellSense software | Olympus | Used to image acquisition | |
| Centrifuge | |||
| Cholera Toxin from Vibrio cholerae | Sigma-Aldrich | C8052 | Used to supplement cell culture medium |
| Class II Biological Safety Cabinet | Herasafe | HAEREUS HS12 | |
| Confocal inverted Microscope | Leica | TCS SP5 | |
| Cover glasses | Witeg Labortechnik GmbH | 4600122 | 22 X 22 mm, thickness 0.13 - 0.17 mm |
| DAPI | 2-(4-amidinophenyl)-1H -indole-6-carboxamidine | ||
| Fetal Bovine Serum | Biowest | S1810 | Used to inactivate trypsine action |
| Fiji software (ImageJ) | National Institutes of Health | RRID:SCR_002285 | Free download, no license needed |
| Glass Pasteur Pipettes | |||
| Glass slides | Fisherbrand | 11844782 | |
| Goat Serum | Biowest | S2000 | Used for immunofluorescence of 3D structures |
| Heat-Resistant Gloves | Used for agar manipulation after autoclave | ||
| Heater bath (37 ºC) | Used to temper solutions prior to cell subculture | ||
| Heater bath (42 ºC) | Used to keep agar warm | ||
| Heating plate | Used for Matrigel dehydration | ||
| Humid chamber | Used for the incubation of antibodies during immunofluorescence | ||
| Ice | Used during Matrigel manipulation | ||
| Ice-box | |||
| Inverted Optic Microscope | Olympus | IX71 | |
| Matrigel Matrix | Becton Dickinson | 354234 | Store at -20 ºC and keep cold when in use. Referred to as basement membrane matrix |
| Methanol | PanReac AppliChem | 131091 | Used for 3D structure fixation prior to immunofluorescent labelling |
| Micropipette | p1000, p200 and p10 | ||
| Microsoft Office Excel | Microsoft | RRID:SCR_016137 | Used to calculate population doubling and to obtain growth rate equation |
| MilliQ water | Referred to as ultrapure water | ||
| Nail Polish | Used to seal samples after mounting | ||
| Parafilm M | Bemis | PM-999 | Used to cover antibody solution during incubation |
| PBS pH 7.4 (w/o calcium & magnesium) | Gibco | 10010-056 | Sterile. Used for cell subculture |
| PBS tablets | Sigma-Aldrich | P4417 | Dilute in milliQ water. No sterility required. Used for immunofluorescence |
| Pipette Aid | |||
| Primaria T25 flasks | Corning | 353808 | Used for BPEC culture |
| Scepter Automated Cell Counter | Millipore | PHCC20060 | Alternatively, use an haemocytometer |
| Scissors | Used to cut pipette tips and parafilm | ||
| Sterile filters 0.22 μm | Millipore | SLGP033RS | Used to filter MTT solution |
| Thiazolyl Blue Tetrazolium Bromide (MTT) | Sigma-Aldrich | M2128 | Store at -20 ºC |
| Triton X-100 | Sigma-Aldrich | T8787 | Used for immunofluorescence of 3D structures |
| Trypsin-EDTA 10X | Biowest | X0930 | Dilute in PBS to obtain 3X solution |
| Vectashield Antifade Mounting Medium | Vector Laboratories | H-1000 | |
| WIT-P-NC Culture Medium | Stemgent | 00-0051 | Used for primary BPEC culture |
| WIT-T Culture Medium | Stemgent | 00-0047 | Used for transformed BPEC culture |
Referências
- Hanahan, D., Weinberg, R. A. Hallmarks of cancer: the next generation. Cell. 144 (5), 646-674 (2011).
- Stampfer, M. R., Yaswen, P. Culture models of human mammary epithelial cell transformation. Journal of Mammary Gland Biology and Neoplasia. 5 (4), 365-378 (2000).
- Schinzel, A. C., Hahn, W. C. Oncogenic transformation and experimental models of human cancer. Frontiers in Bioscience : A Journal and Virtual Library. 13 (13), 71 (2008).
- Balani, S., Nguyen, L. V., Eaves, C. J. Modeling the process of human tumorigenesis. Nature Communications. 8 (1), 15422 (2017).
- Bray, F., et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA: A Cancer Journal for Clinicians. 68 (6), 394-424 (2018).
- Ince, T. A., et al. Transformation of different human breast epithelial cell types leads to distinct tumor phenotypes. Cancer Cell. 12 (2), 160-170 (2007).
- Repullés, J., et al. Radiation-induced malignant transformation of preneoplastic and normal breast primary epithelial cells. Molecular Cancer Research. , 1-13 (2019).
- Weigelt, B., Bissell, M. J. Unraveling the microenvironmental influences on the normal mammary gland and breast cancer. Seminars in Cancer Biology. 18 (5), 311-321 (2008).
- Paoli, P., Giannoni, E., Chiarugi, P. Anoikis molecular pathways and its role in cancer progression. Biochimica et Biophysica Acta. 1833 (12), 3481-3498 (2013).
- Debnath, J., Muthuswamy, S. K., Brugge, J. S. Morphogenesis and oncogenesis of MCF-10A mammary epithelial acini grown in three-dimensional basement membrane cultures. Methods. 30 (3), 256-268 (2003).
- Borowicz, S., et al. The soft agar colony formation assay. Journal of Visualized Experiments. (92), (2014).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Brocher, J. The BioVoxxel Image Processing and Analysis Toolbox. European BioImage Analysis Symposium. 8 (2), 67112 (2015).
- Torquato, S., Truskett, T. M., Debenedetti, P. G. Is random close packing of spheres well defined. Physical Review Letters. 84 (10), 2064-2067 (2000).
- LaBarge, M. A., Garbe, J. C., Stampfer, M. R. Processing of human reduction mammoplasty and mastectomy tissues for cell culture. Journal of Visualized Experiments. (71), (2013).
- Zubeldia-Plazaola, A., et al. Glucocorticoids promote transition of ductal carcinoma in situ to invasive ductal carcinoma by inducing myoepithelial cell apoptosis. Breast Cancer Research. 20 (1), 65 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados