Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Identifizierung der Bindungsproteine kleiner Liganden mit der differentiellen radialen Kapillarwirkung des Ligandenassays (DRaCALA)
In diesem Artikel
Zusammenfassung
Der Differential Radial Capillary Action of Ligand Assay (DRaCALA) kann verwendet werden, um kleine ligandenbindende Proteine eines Organismus unter Verwendung einer ORFeome-Bibliothek zu identifizieren.
Zusammenfassung
In den letzten zehn Jahren gab es enorme Fortschritte beim Verständnis kleiner Signalmoleküle in der bakteriellen Physiologie. Insbesondere die Zielproteine mehrerer nukleotid-abgeleiteter Sekundärbotenstoffe (NSMs) wurden systematisch identifiziert und in Modellorganismen untersucht. Diese Erfolge sind hauptsächlich auf die Entwicklung mehrerer neuer Techniken zurückzuführen, darunter die Capture-Compound-Technik und die differentielle radiale Kapillarwirkung des Ligandenassays (DRaCALA), mit denen Zielproteine dieser kleinen Moleküle systematisch identifiziert wurden. Dieser Artikel beschreibt die Verwendung der NSMs, Guanosinpenta- und Tetraphosphate (p)ppGpp, als Beispiel und Videodemonstration der DRaCALA-Technik. Unter Verwendung von DRaCALA wurden 9 von 20 bekannten und 12 neuen Zielproteinen von (p)ppGpp im Modellorganismus Escherichia coli K-12 identifiziert, was die Leistungsfähigkeit dieses Assays demonstriert. Prinzipiell könnte DRaCALA zur Untersuchung kleiner Liganden verwendet werden, die durch radioaktive Isotope oder Fluoreszenzfarbstoffe markiert werden können. Die kritischen Schritte, Vor- und Nachteile von DRaCALA werden hier für die weitere Anwendung dieser Technik diskutiert.
Einleitung
Bakterien verwenden mehrere kleine Signalmoleküle, um sich an sich ständig ändernde Umgebungen anzupassen1,2. Zum Beispiel vermitteln die Autoinduzierer, N-Acylhomoserinlactone und ihre modifizierten Oligopeptide, die interzelluläre Kommunikation zwischen Bakterien, um das Populationsverhalten zu koordinieren, ein Phänomen, das als Quorum Sensing bekanntist 2. Eine weitere Gruppe kleiner Signalmoleküle sind die NSMs, darunter das umfassend untersuchte zyklische Adenosinmonophosphat (cAMP), cyclisches Di-AMP, cyclisches Di-Guanosinmonophosphat (cyclisches Di-GMP) und Guanosin-Penta- und Tetraphosphate (p)ppGpp1. Bakterien produzieren diese NSMs als Reaktion auf eine Vielzahl von verschiedenen Stressbedingungen. Einmal produziert, binden diese Moleküle an ihre Zielproteine und regulieren mehrere verschiedene physiologische und metabolische Wege, um den angetroffenen Belastungen standzuhalten und das Überleben der Bakterien zu verbessern. Daher ist die Identifizierung der Zielproteine eine unumgängliche Voraussetzung, um die molekularen Funktionen dieser kleinen Moleküle zu entschlüsseln.
In den letzten zehn Jahren gab es einen Wissensboom über diese kleinen Signalmoleküle, hauptsächlich aufgrund mehrerer technischer Innovationen, die die Zielproteine dieser kleinen Moleküle enthüllten. Dazu gehören die Capture-Compound-Technik3,4,5und die differentielle radiale Kapillarwirkung des Ligandenassays (DRaCALA)6, die in diesem Artikel diskutiert werden soll.
DRaCALA wurde 2011 von Vincent Lee und Mitarbeitern6erfunden und nutzt die Fähigkeit einer Nitrocellulosemembran, freie und proteingebundene Liganden differentiell zu sequestriert. Moleküle wie Proteine können nicht auf einer Nitrocellulosemembran diffundieren, während kleine Liganden wie die NSMs dazu in der Lage sind. Durch Mischen des NSM (z. B., ppGpp) mit dem zu testenden Protein und Aufträgen auf der Membran sind zwei Szenarien zu erwarten (Abbildung 1): Wenn (p)ppGpp an das Protein bindet, wird das radioaktiv markierte (p)ppGpp vom Protein in der Mitte des Flecks zurückgehalten und diffundiert nicht nach außen, wodurch ein intensiver kleiner Punkt (d.h. B. starkes radioaktives Signal) unter einem Phosphorimager. Wenn (p)ppGpp jedoch nicht an das Protein bindet, diffundiert es frei nach außen, um einen großen Fleck mit gleichmäßigem radioaktivem Hintergrundsignal zu erzeugen.
Darüber hinaus kann DRaCALA die Wechselwirkung zwischen einem kleinen Molekül und einem ungereinigten Protein in einem ganzen Zelllysat nachweisen, wenn das Protein in ausreichender Menge vorhanden ist. Diese Einfachheit ermöglicht die Verwendung von DRaCALA bei der schnellen Identifizierung von Proteinzielen mithilfe einer ORFeome-Expressionsbibliothek. Tatsächlich wurden Zielproteine von cAMP7, cyclischem di-AMP8, cyclischem di-GMP9,10und (p)ppGpp11,12,13 systematisch unter Verwendung von DRaCALA identifiziert. Dieser Videoartikel verwendet (p)ppGpp als Beispiel, um die kritischen Schritte und Überlegungen bei der Durchführung eines erfolgreichen DRaCALA-Screenings zu demonstrieren und zu beschreiben. Es wird dringend empfohlen, eine ausführlichere Beschreibung von DRaCALA14 in Kombination mit diesem Artikel zu lesen, bevor Sie DRaCALA durchführen.
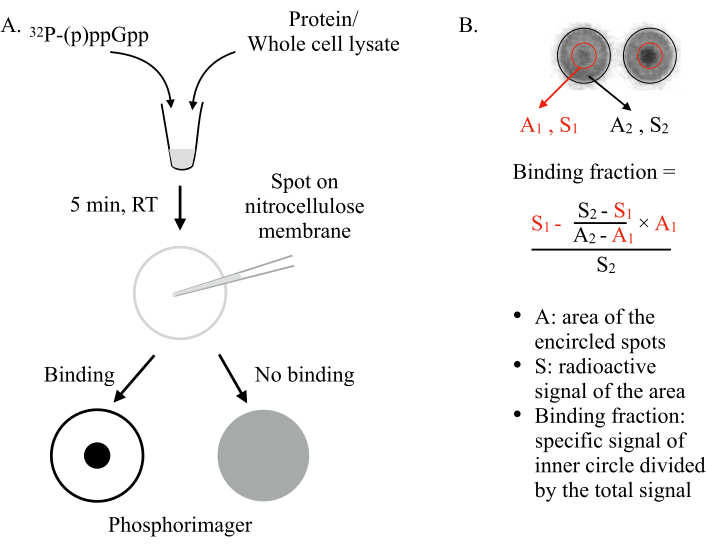
Abbildung 1: Das Prinzip von DRaCALA. (A) Schematische Darstellung des DRaCALA-Assays. Weitere Informationen finden Sie im Text. (B) Quantifizierung und Berechnung des Bindungsanteils. Weitere Informationen finden Sie im Text. Kurz gesagt, die DRaCALA-Flecken werden analysiert, indem zwei Kreise gezeichnet werden, die den gesamten Fleck und den inneren dunklen Punkt(dhdas zurückgehaltene (p) ppGpp aufgrund der Bindung des getesteten Proteins) umschreiben. Das spezifische Bindungssignal ist das radioaktive Signal des inneren Kreises (S1) nach Subtraktierung des unspezifischen Hintergrundsignals (berechnet durch A1 × ((S2 -S1)/(A2-A1))). Der Bindungsanteil ist das spezifische Bindungssignal dividiert durch das gesamte radioaktive Signal (S2). Abkürzungen: DRaCALA = Differential Radial Capillary Action of Ligand Assay; (p)ppGpp = Guanosinpenta- und Tetraphosphate; RT = Raumtemperatur. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
1. Herstellung von Ganzzelllysaten
- Impfen Sie die E. coli K-12 ASKA ORFeome Collection Stämme15 in 1,5 ml Lysogeny Brühe (LB) mit 25 μg/ml Chloramphenicol in 96-Well-Tiefbrunnenplatten. Über Nacht (O/N) für 18 h bei 30 °C mit Schütteln bei 160 U/min wachsen. Am nächsten Tag isopropyl β-d-1-thiogalactopyranosid (IPTG) (letzte 0,5 mM) in die O/N-Kulturen geben, um die Proteinexpression bei 30 °C für 6 h zu induzieren.
- Pelletzellen bei 500 x g für 10 min. Die Pellets bis zum Gebrauch bei -80 °C einfrieren. Um die Zellen zu lysieren, fügen Sie 150 μL Lysepuffer L1 (40 mM Tris pH 7,5, 100 mM NaCl, 10 mMMgCl2,ergänzt mit 2 mM Phenylmethylsulfonylfluorid (PMSF), 40 μg/ml DNase 1 und 0,5 mg/ml Lysozym) hinzu, um das Pellet wieder zu resuspendieren.
- Die Zellen 30 min lang bei -80 °C einfrieren und dann 20 min bei 37 °C auftauen. Wiederholen Sie diesen Zyklus dreimal, um die Zellen zu lysieren. Lagern Sie die Lysate vor Gebrauch bei -80 °C.
2. Reinigung von Relseq und GppA
HINWEIS: Die rekombinanten Proteine Relseq aus Streptococcus equisimilis und GppA aus E. coli K-12 werden verwendet, um die radioaktiv markierten ppGpp bzw. ppGpp zu synthetisieren.
- Wachsen und sammeln Sie Zellen, die jedes Protein überexprimieren.
- Züchte den E. coli BL21 DE3-Stamm bis zu einer exponentiellen Phase (optische Dichte (OD) ~ 0,3-0,4) in LB-Brühe und spinne 1 ml Kultur bei 6000 x g für 5 min herunter. Dekantieren Sie den Überstand und resuspendieren Sie die Zellen mit 100 μL eiskalter TSB-Brühe (LB-Brühe ergänzt mit 0,1 g/ml PEG3350, 0,05 ml/ml Dimethylsulfoxid, 20 mMMgCl2).
- Mischen Sie die Plasmide mit dem histidin-markierten relseq und gppA,jeweils 100 ng, mit den oben genannten Zellsuspensionen in TSB und inkubieren Sie 30 Minuten lang auf Eis. Hitzeschock die Zellen bei 42 °C für 40 s. Legen Sie die Mischung für 2 min auf Eis und fügen Sie 1 ml LB-Brühe bei Raumtemperatur hinzu, damit sich die Zellen für 1 h bei 37 ° C mit Rühren bei 160 U / min erholen können.
- Platten die zurückgewonnenen Zellen auf LB-Agarplatten, ergänzt mit den entsprechenden Antibiotika (Rel seq:100 μg/ml Ampicillin; GppA: 30 μg/ml Kanamycin). Am nächsten Tag die Kolonien in LB-Brühe impfen, um O/N-Vorkulturen beider Stämme bei 37 °C zu starten.
- Nach 18 h impfen Sie 500 ml LB-Medium mit 10 ml der O/N-Kulturen und den entsprechenden Antibiotika. Züchten Sie die Kulturen durch Schütteln bei 160 U /min bei 37 °C. Wenn der OD600nm 0,5-0,7 erreicht, induzieren Sie die Proteinexpression, indem Sie 0,5 mM IPTG hinzufügen und 3 h bei 30 °C mit Schütteln bei 160 U / min wachsen.
- Sammeln Sie die Zellen, indem Sie bei 6084 x g für 10 min bei 4 °C drehen. Das Pellet in 20 ml eiskaltem 1x phosphatgepufferter Kochsalzlösung (PBS) wieder aufsuchen und bei 1912 x g für 20 min bei 4 °C erneut zentrifugieren. Dekantieren Sie den Überstand und frieren Sie die Pellets vor Gebrauch bei -20 °C ein.
- Nickel-Nitrilotretsäure (Ni-NTA) Affinitätsreinigung
HINWEIS: Stellen Sie ab diesem Zeitpunkt sicher, dass die Proben kalt sind.- Fügen Sie 40 mL eiskalten Lysepuffer L2 (50 mM Tris pH 7,5, 150 mM NaCl, 5% Glycerin, 10 mM Imidazol, 10 mM β-Mercaptoethanol mit Proteasehemmern (EDTA-freie Tablette; siehe Materialtabelle)hinzu, um das Pellet wiederzuberuhen. Lyse der Zellen durch Beschallung (60% Amplitude, 2 s ON/ 4 s OFF für 8 min ON). Das Lysat durch Drehen bei 23.426 x g für 40 min bei 4 °C reinigen und mit dem Überstand zur Reinigung fortfahren.
- Bereiten Sie während der obigen Zentrifugation das Ni-NTA-Harz vor.
- 500 μL homogenisiertes Ni-NTA-Harz in eine stehende Polypropylen-Chromatographiesäule geben und 15 minuten ruhen lassen und die Speicherlösung durchgehen. Waschen Sie das Harz zweimal mit 15 ml Reinstwasser und waschen Sie dann die Säule mit 15 ml des Lysepuffers L2.
- Laden Sie den geklärten Überstand des Zelllysats aus Schritt 1 auf die Säule und lassen Sie ihn durchfließen. Waschen Sie die Säule mit 30 mL Waschpuffer (50 mM Tris pH 7,5, 150 mM NaCl, 5% Glycerin, 20 mM Imidazol).
- Eluieren Sie die Proteine mit 400 μL des Elutionspuffers (50 mM Tris pH 7,5, 150 mM NaCl, 5% Glycerin, 500 mM Imidazol) dreimal. Wiederholen Sie dann die Elution mit weiteren 300 μL des Elutionspuffers. Kombinieren Sie die eluierten Proteine zu einem Endvolumen von 700 μL.
- Gelfiltration
- Gelfiltrationspuffer vorbereiten (50 mM Tris, pH 7,5; 200 mM NaCl; 5% Glycerin). Waschen Sie die Größenausschlusssäule mit einem Säulenvolumen (25 ml) des Gelfiltrationspuffers.
- Laden Sie die oben genannten 700 μL-Probe mit einer 500-μL-Schleife, laufen Sie mit 0,5 ml / min und sammeln Sie 2-3 Fraktionen mit jeweils 0,5 ml Volumen, die die jeweiligen Proteine enthalten.
- Kombinieren und konzentrieren Sie die Fraktionen, die jedes der Proteine enthalten, mit einer Spinsäule und messen Sie die Proteinkonzentration mit dem Bradford-Assay.
3. Synthese von 32P-markierten pppGpp und ppGpp
- Eine kleine Relseq-Reaktion wird in einem Schraubverschlussrohr montiert (siehe Tabelle 1).
HINWEIS: Arbeiten Sie mit radioaktiven Reagenzien nur an einem lizenzierten Ort und mit persönlicher Schutzausrüstung.
| Volumen (μL) | ||
| Kleiner Maßstab | Großer Maßstab | |
| Reinstwasser | ||
| 10x Relseq Puffer* | 2 | 50 |
| ATP (8 mM finale) | ||
| Relseq (4 μM Finale) | ||
| 32 P-α-Guanosintriphosphat (GTP) (letzte 120 nM) (VORSICHT) | 0.2 | 5 |
| gesamt | 20 | 500 |
Tabelle 1: Assemblierungsinformationen für die klein- und großräumigen Synthesereaktionen von 32P-markierten pppGpp. *10x Relseq-Puffer enthält 250 mM Tris-HCl, pH 8,6; 1M NaCl; 80 mMMgCl2. Abkürzung: pppGpp = Guanosinpentaphosphat.
- Das Röhrchen bei 37 °C in einem Thermomixer für 1 h, dann bei 95 °C für 5 min inkubieren und für 5 min auf Eis legen. Drehen Sie das ausgefällte Protein bei 15.700 x g für 5 min herunter und übertragen Sie den Überstand (synthetisiert 32P-pppGpp) in ein neues Schraubverschlussrohr.
- Um 32P-ppGpp aus 32P-pppGpp zu synthetisieren, übertragen Sie die Hälfte des 32p-pppGpp-Produkts in ein neues Schraubverschlussrohr und fügen Sie 1 μM GppA hinzu. Inkubieren Sie das Röhrchen bei 37 °C für 10 min, bei 95 °C für 5 min und legen Sie es dann für 5 min auf Eis.
- Drehen Sie das ausgefällte Protein bei 15.700 x g für 5 min herunter und übertragen Sie den Überstand (synthetisiert 32P-ppGpp) in ein neues Schraubverschlussrohr.
- Analysieren Sie die 32P-pppGpp und 32P-ppGpp, indem Sie 1 μL der Proben auf einer Dünnschichtchromatographieplatte (TLC) (Polyethylenimin-modifizierte Cellulose-TLC-Platten) mit dem 1,5 M KH2PO4,pH 3,4, als mobile Phase laufen lassen.
HINWEIS: Verwenden Sie dasα-32P-markierte Guanosin-5'-triphosphat(32P-α-GTP) als Kontrolle. - Trocknen Sie die TLC-Platte vollständig, legen Sie sie zwischen eine transparente Kunststoffmappe und setzen Sie sie 5 Minuten lang einem Phosphorspeicher aus. Visualisieren und quantifizieren Sie die Signale mit einem Phosphorimager.
HINWEIS: Wenn die Verhältnisse von 32P-pppGpp und 32P-ppGpp höher als 85% sind, könnte eine großräumige Reaktion (500 μL, ausreichend für das Screening von 20 96-Well-Platten) unter Verwendung von Tabelle 1zusammengesetzt und synthetisiert werden.
4. DRaCALA-Screening der Zielproteine von (p)ppGpp
- Auftauen und 20 μL ganzzellige Lysate auf eine 96-Well-V-Boden-Mikrotiterplatte übertragen. Fügen Sie 2,5 U/Well Endonuklease aus Serratia marcescenshinzu und inkubieren Sie bei 37 °C für 15 minuten, um die Lysatviskosität zu reduzieren. Die Lysate für 20 min auf Eis legen.
- Mischen Sie die 32P-pppGpp und 32P-ppGpp in einem Verhältnis von 1: 1 und fügen Sie 1x Lysepuffer L1 hinzu, um die Endkonzentration von (p) ppGpp gleich 4 nM zu machen.
HINWEIS: Angesichts der chemischen Ähnlichkeit zwischen pppGpp und ppGpp vereinfacht eine Mischung aus beiden Chemikalien den Screening-Prozess. - Verwenden Sie eine Mehrkanalpipette und gefilterte Pipettenspitzen, um 10 μL der (p)ppGpp-Mischung mit dem Zelllysat hinzuzufügen und zu mischen. Bei Raumtemperatur (RT) für 5 min inkubieren.
- Waschen Sie das 96 x Pin-Werkzeug, indem Sie 30 s lang eine 0,01%ige Lösung eines nichtionischen Reinigungsmittels einlegen und 30 s auf einem Seidenpapier trocknen. Wiederholen Sie das Waschen des Stiftwerkzeugs 3x.
- Legen Sie das Stiftwerkzeug in die obigen 96-Well-Probenplatten und warten Sie 30 s. Heben Sie das Stiftwerkzeug gerade nach oben und legen Sie es für 30 s gerade nach unten auf eine Nitrocellulosemembran.
HINWEIS: Wenn ein Punkt fehlt, spoten Sie 2 μL der entsprechenden Proben mit einer Pipette und gefilterten Spitzen. Es ist ratsam, einen doppelten Punkt der gleichen Probe wie unten angegeben zu machen. - Trocknen Sie die Membran für 5 min bei RT. Legen Sie die Membran zwischen eine transparente Kunststoffmappe und setzen Sie sie für 5 min einem Speicherphosphorsieb aus. Visualisieren Sie mit einem Phosphorimager.
5. Quantifizierung und Identifizierung potenzieller Zielproteine
- Verwenden Sie die Analysesoftware, die dem Phosphorimager zugeordnet ist, um die GEL-Datei der visualisierten Platten zu öffnen. Verwenden Sie die Array-Analysefunktion, um die 96 Punkte zu definieren, indem Sie ein Raster mit 12 Spalten x 8 Zeilen einrichten.
- Definieren Sie große Kreise, um den äußeren Rand der gesamten Flecken zu umschreiben (siehe Abbildung 1B). Exportieren Sie volumn+Background und Area der definierten großen Kreise und speichern Sie sie in einer Tabelle.
HINWEIS: Positionieren Sie bei Bedarf jeden einzelnen Kreis neu, um sich perfekt mit den Flecken zu überlappen, und ändern Sie die Größe jedes einzelnen Kreises, um ihn etwas größer als den tatsächlichen Punkt zu machen. - Vergrößern Sie die definierten Kreise, um die kleinen inneren Punkte zu umschreiben. Exportieren Sie volumn+Backgroundund Area der definierten kleinen Kreise und speichern Sie sie in einer Tabelle.
- Berechnen Sie die Bindungsbrüche in der Tabelle mithilfe der Gleichung in Abbildung 1B, und zeichnen Sie die Daten. Identifizieren Sie die potenziellen Bindungsproteine in den Vertiefungen, die im Vergleich zu den meisten anderen Vertiefungen hohe Bindungsfraktionen aufweisen.

Abbildung 2:Gesamtworkflow des DRaCALA-Screening-Prozesses. Die Proteinproduktion aus einer Escherichia coli ASKA-Sammlung wird induziert und die Zellen werden lysiert. Inzwischen werden die rekombinanten Proteine Relseq-Hisund GppA-His gereinigt und zur Synthese von 32P-markierten pppGpp und ppGpp aus 32P-α-GTP verwendet. Die radioaktiv markierten (p)ppGpp-Moleküle werden dann mit den Lysaten gemischt, und ein 96-Pin-Tool wird verwendet, um die Mischungen auf einer Nitrocellulosemembran zu erkennen, um sie anschließend einem Phosphorspeicherbildschirm, der Bildgebung und quantifizierung der radioaktiven Signale zu erweisen. Abkürzungen: DRaCALA = Differential Radial Capillary Action of Ligand Assay; (p)ppGpp = Guanosinpenta- und Tetraphosphate; RT = Raumtemperatur; IPTG = Isopropyl β-d-1-thiogalactopyranosid; GTP = Guanosin-5'-triphosphat; SDS-PAGE = Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese ; TLC = Dünnschichtchromatographie. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Das Befolgen des oben beschriebenen Protokolls führt in der Regel zu zwei Arten von Ergebnissen (Abbildung 3).
Abbildung 3A zeigt eine Platte mit relativ geringen Hintergrundbindungssignalen (Bindungsfraktionen < 0,025) aus der Mehrzahl der Vertiefungen. Das positive Bindungssignal der Bohrung H3 ergibt einen Bindungsanteil von ~0,35, der viel höher ist als der, der für die anderen Vertiefungen beobachtet wurde. Selbst ohne Quantif...
Diskussion
Einer der kritischen Schritte bei der Durchführung eines DRaCALA-Screenings besteht darin, gute Lysate ganzer Zellen zu erhalten. Zunächst sollten die getesteten Proteine in großen Mengen und in löslichen Formen hergestellt werden. Zweitens sollte die Lyse der Zellen vollständig sein und die Viskosität des Lysats muss minimal sein. Die Einbeziehung von Lysozym und die Verwendung von drei Gefrier-Tau-Zyklen reichen oft aus, um Zellen vollständig zu lysieren. Die freigesetzte chromosomale DNA macht das Lysat jedoch ...
Offenlegungen
Die Autoren haben keinen Interessenkonflikt offenzulegen.
Danksagungen
Die Arbeit wird durch einen NNF-Projektzuschuss (NNF19OC0058331) für YEZ und das Forschungs- und Innovationsprogramm Horizon 2020 der Europäischen Union im Rahmen der Marie Skłodowska-Curie-Finanzhilfevereinbarung (Nr. 801199) an MLS unterstützt.

Materialien
| Name | Company | Catalog Number | Comments |
| 32P-α-GTP | Perkinelmer | BLU006X250UC | |
| 96 x pin tool | V&P Scientific | VP 404 | 96 Bolt Replicator, on 9 mm centers, 4.2 mm Bolt Diameter, 24 mm long |
| 96-well V-bottom microtiter plate | Sterilin | MIC9004 | Sterilin Microplate V Well 611V96 |
| Agar | OXOID - Thermo Fisher | LP0011 | Agar no. 1 |
| ASKA collection strain | NBRP, SHIGEN, JAPAN | Ref: DNA Research, Volume 12, Issue 5, 2005, Pages 291–299. https://doi.org/10.1093/dnares/dsi012 | |
| Benzonase | SIGMA | E1014-25KU | genetically engineered endonuclease from Serratia marcescens |
| Bradford Protein Assay Dye | Bio-Rad | 5000006 | Reagent Concentrate |
| DMSO | SIGMA | D8418 | ≥99.9% |
| DNase 1 | SIGMA | DN25-1G | |
| gel filtration10x300 column | GE Healthcare | 28990944 | contains 20% ethanol as preservative |
| Glycerol | PanReac AppliChem | 122329.1214 | Glycerol 87% for analysis |
| Hypercassette | Amersham | RPN 11647 | 20 x 40 cm |
| Imidazole | SIGMA | 56750 | puriss. p.a., ≥ 99.5% (GC) |
| IP Storage Phosphor Screen | FUJIFILM | 28956474 | BAS-MS 2040 20x 40 cm |
| Isopropyl β-d-1-thiogalactopyranoside (IPTG) | SIGMA | I6758 | Isopropyl β-D-thiogalactoside |
| Lysogeny Broth (LB) | Invitrogen - Thermo Fisher | 12795027 | Miller's LB Broth Base |
| Lysozyme | SIGMA | L4949 | from chicken egg white; BioUltra, lyophilized powder, ≥98% |
| MgCl2 (Magnesium chloride) | SIGMA | 208337 | |
| MilliQ water | ultrapure water | ||
| multichannel pipette | Thermo Scientific | 4661110 | F1 - Clip Tip; 1-10 ul, 8 x channels |
| NaCl | VWR Chemicals | 27810 | AnalaR NORMAPUR, ACS, Reag. Ph. Eur. |
| Ni-NTA Agarose | Qiagen | 30230 | |
| Nitrocellulose Blotting Membrane | Amersham Protran | 10600003 | Premium 0.45 um 300 mm x 4 m |
| PBS | OXOID - Thermo Fisher | BR0014G | Phosphate buffered saline (Dulbecco A), Tablets |
| PEG3350 (Polyethylene glycol 3350) | SIGMA | 202444 | |
| phenylmethylsulfonyl fluoride (PMSF) | SIGMA | 93482 | Phenylmethanesulfonyl fluoride solution - 0.1 M in ethanol (T) |
| Phosphor-imager | GE Healthcare | 28955809 | Typhoon FLA-7000 Phosphor-imager |
| Pipette Tips, filtered | Thermo Scientific | 94410040 | ClipTip 12.5 μl nonsterile |
| Poly-Prep Chromatography column | Bio-Rad | 7311550 | polypropylene chromatography column |
| Protease inhibitor Mini | Pierce | A32955 | Tablets, EDTA-free |
| screw cap tube | Thermo Scientific | 3488 | Microcentifuge Tubes, 2.0 ml with screw cap, nonsterile |
| SLS 96-deep Well plates | Greiner | 780285 | MASTERBLOCK, 2 ML, PP, V-Bottom, Natural |
| spin column | Millipore | UFC500396 | Amicon Ultra -0.5 ml Centrifugal Filters |
| Thermomixer | Eppendorf | 5382000015 | Thermomixer C |
| TLC plate (PEI-cellulose F TLC plates) | Merck Millipore | 105579 | DC PEI-cellulose F (20 x 20 cm) |
| Tris | SIGMA | BP152 | Tris Base for Molecular Biology |
| Tween 20 | SIGMA | P1379 | viscous non-ionic detergent |
| β-mercaptoethanol | SIGMA | M3148 | 99% (GC/titration) |
Referenzen
- Kalia, D., et al. Nucleotide, c-di-GMP, c-di-AMP, cGMP, cAMP, (p)ppGpp signaling in bacteria and implications in pathogenesis. Chemical Society Reviews. 42 (1), 305-341 (2013).
- Camilli, A., Bassler, B. L. Bacterial small-molecule signaling pathways. Science. 311 (5764), 1113-1116 (2006).
- Luo, Y., et al. The cAMP capture compound mass spectrometry as a novel tool for targeting cAMP-binding proteins: from protein kinase A to potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channels. Molecular & Cellular Proteomics. 8 (12), 2843-2856 (2009).
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. Journal of Proteomics. 75 (15), 4874-4878 (2012).
- Laventie, B. J., et al. Capture compound mass spectrometry--a powerful tool to identify novel c-di-GMP effector proteins. Journal of Visual Experiments. (97), e51404 (2015).
- Roelofs, K. G., Wang, J., Sintim, H. O., Lee, V. T. Differential radial capillary action of ligand assay for high-throughput detection of protein-metabolite interactions. Proceedings of the National Academy of Sciences of the United States of America. 108 (37), 15528-15533 (2011).
- Zhang, Y., et al. Evolutionary adaptation of the essential tRNA methyltransferase TrmD to the signaling molecule 3 ',5 '-cAMP in bacteria. Journal of Biological Chemistry. 292 (1), 313-327 (2017).
- Corrigan, R. M., et al. Systematic identification of conserved bacterial c-di-AMP receptor proteins. Proceedings of the National Academy of Sciences of the United States of America. 110 (22), 9084-9089 (2013).
- Roelofs, K. G., et al. Systematic identification of cyclic-di-GMP binding proteins in Vibrio cholerae reveals a novel class of cyclic-di-GMP-binding ATPases associated with type II secretion systems. PLoS Pathogen. 11 (10), 1005232 (2015).
- Fang, X., et al. GIL, a new c-di-GMP-binding protein domain involved in regulation of cellulose synthesis in enterobacteria. Molecular Microbiology. 93 (3), 439-452 (2014).
- Corrigan, R. M., Bellows, L. E., Wood, A., Grundling, A. ppGpp negatively impacts ribosome assembly affecting growth and antimicrobial tolerance in Gram-positive bacteria. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1710-1719 (2016).
- Zhang, Y., Zbornikova, E., Rejman, D., Gerdes, K. Novel (p)ppGpp binding and metabolizing proteins of Escherichia coli. Mbio. 9 (2), 02188 (2018).
- Yang, J., et al. The nucleotide pGpp acts as a third alarmone in Bacillus, with functions distinct from those of (p) ppGpp. Nature Communications. 11 (1), 5388 (2020).
- Orr, M. W., Lee, V. T. Differential radial capillary action of ligand assay (DRaCALA) for high-throughput detection of protein-metabolite interactions in bacteria. Methods in Molecular Biology. 1535, 25-41 (2017).
- Kitagawa, M., et al. Complete set of ORF clones of Escherichia coli ASKA library (A complete Set of E. coli K-12 ORF archive): Unique resources for biological research. DNA Research. 12 (5), 291-299 (2005).
- Hochstadt-Ozer, J., Cashel, M. The regulation of purine utilization in bacteria. V. Inhibition of purine phosphoribosyltransferase activities and purine uptake in isolated membrane vesicles by guanosine tetraphosphate. Journal of Biological Chemistry. 247 (21), 7067-7072 (1972).
- Zhang, Y. E., et al. p)ppGpp regulates a bacterial nucleosidase by an allosteric two-domain switch. Molecular Cell. 74 (6), 1239-1249 (2019).
- Wang, B., et al. Affinity-based capture and identification of protein effectors of the growth regulator ppGpp. Nature Chemical Biology. 15 (2), 141-150 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten