Method Article
Nanopore sıralamayı kullanarak tüm kan mRNA sıralaması
Bu Makalede
Özet
Nanopore sıralamasını uzak konumlarda ve kaynak kötü ayarlarında maliyet-etkili sıralamaya izin veren bir yeni teknoloji. Burada, mRNAs 'ın bu şartlar ile uyumlu olan tüm kandan sıralanması için bir protokol sunuyoruz.
Özet
Uzak konumlarda ve kaynak yetersiz ayarlarında sıralama benzersiz zorluklar sunar. Nanopore sıralama başarıyla bu koşullarda kullanılabilir, ve Batı Afrika 'ya son Ebola virüs salgını sırasında dağıtılan, bu olasılığı vurgulayarak. Pratik avantajları (düşük maliyetli, ekipman taşıma ve kullanım kolaylığı) ek olarak, bu teknoloji aynı zamanda ikinci nesil sıralama yaklaşımlar, özellikle çok uzun okuma uzunluğu, doğrudan dizi yeteneği üzerinde temel avantajları sağlar RNA ve gerçek zamanlı veri kullanılabilirliği. Ham okuma doğruluğu, bu teknolojinin ana sınırlamasının temsil ettiği diğer sıralama platformlarından daha düşüktür; Ancak, bu kısmen oluşturulan yüksek okuma derinliği tarafından hafifletilebilir. Burada, ebolaviruses için hücresel reseptör olan Niemann-Pick C1 için mRNAs kodlamasının sıralanması için alan uyumlu bir protokol sunuyoruz. Bu protokol, hayvan kanı örneklerinden RNA 'nın çıkarılması, hedef zenginleştirme, barkodlama, Kütüphane hazırlama ve sıralamanın kendisi için RT-PCR tarafından izlenen ve diğer DNA veya RNA hedefleri ile kullanılmak üzere kolayca adapte edilebilir.
Giriş
Sıralama, biyolojik ve Biyomedikal araştırmalarda güçlü ve önemli bir araçtır. Genomlar, genetik varyasyonlar ve RNA ifade profillerinin analizine izin verir ve böylece insan ve hayvan hastalıklarının1,2' deki araştırmasında önemli bir rol oynamaktadır. DNA sıralaması için kullanılabilen en eski yöntemlerden biri olan Sanger sıralaması, hala rutin olarak bu güne kadar kullanılır ve moleküler biyolojinin köşe taşı olmuştur. Son 50 yıl içinde, bu teknoloji, 1.000 NT 'den fazla okuma-uzunlukları ve% 99,9991kadar yüksek bir doğruluk elde etmek için geliştirilmiştir. Ancak, Sanger sıralama da sınırlamalar vardır. Daha büyük bir numune kümesi veya bu yöntemle tüm genomlar Analizi sıralama zaman alıcı ve pahalı1,3. 454 pyrosequencing ve ıllına teknolojisi gibi ikinci nesil (yeni nesil) DNA sıralama yöntemleri, son on yıl içinde sıralamanın gerektirdiği maliyet ve iş yükünü önemli ölçüde azaltmamıza ve büyük bir artış sağladı biyolojik sıralı bilgi miktarı kullanılabilir4. Yine de, bu ikinci nesil teknolojileri kullanarak bireysel sıralı çalışır pahalıdır, ve alan koşullarında sıralama zordur, gerekli ekipman hantal ve kırılgan olduğu gibi (Sanger sıralama cihazları benzer), ve genellikle Özel eğitimli personel tarafından kalibre edilmiş ve servis edilir. Ayrıca, ikinci nesil teknolojilerin çoğu için okuma-uzunlukları oldukça sınırlıdır, genellikle bu verilerin aşağı akım Biyoinformatik Analizi zorlu yapar.
Cep boyutunda nanopore sıralama cihazları kullanarak üçüncü nesil sıralama (bkz. malzeme tablosu) bu kurulan sıralama platformlarına alternatif olarak hizmet verebilir. Bu cihazlarda tek telli bir DNA veya RNA molekül bir nanopore ile aynı anda bir sensör ile ölçülen iyonik akım ile geçer (Şekil 1). Strand nanopore geçer gibi, herhangi bir zamanda gözenek mevcut nükleotidler tarafından akım modülasyon tespit edilir, ve hesaplamalı geri nükleotid sırası5tercüme. Bu operasyonel prensip nedeniyle, nanopore sıralamaları çok uzun okuma üretimi sağlar (yakın 1 x 106 nükleotidler6) ve gerçek zamanlı olarak sıralama veri analizi. Barkodlama, bir örnekteki nükleik asitlere tanımlanmış nükleotid dizileri takarak, tek bir sıralamanın çalıştırılmasıyla birden fazla numunenin analizine olanak sağlayan, böylece örnek verimi artırarak ve örnek maliyetleri düşürerek mümkündür. Yüksek taşınabilirlik ve kullanım kolaylığı nedeniyle, nanopore sıralama cihazları Batı Afrika 'da son Ebola virüs hastalığı salgını sırasında alanında başarıyla kullanılmıştır, uzak bölgelere hızlı dağıtım için uygunluğu vurgulayarak7 , 8' den itibaren.
Burada, Niemann-Pick C1 (NPC1) proteini için mRNA kodlama sıralaması için ayrıntılı bir alan uyumlu protokol tarif, ebolaviruses gibi filovirüsler için zorunlu giriş reseptörü olan, ve türler duyarlılık sınırlamak için gösterilmiştir Bu virüsler9,10. Protokol, tüm RNA 'nın kan örneklerinden çıkarılması, NPC1 mRNA 'nın RT-PCR ile spesifik amplifikasyonu, numune barkodları, Kütüphane hazırlığı ve nanopore sıralama cihazı ile sıralamayı kapsar. Veri analizi, alan sınırlamaları nedeniyle tartışılamaz, ancak bazı temel yönergeler temsili sonuçlarında sağlanır; Ancak, ilgili okuyucu daha ayrıntılı bilgi için diğer12,13,14 tarafından yayınlarının yanı sıra, kullanılan iş akışının daha ayrıntılı bir açıklaması için bir önceki yayın11 adlandırılır Bu iş akışında kullanılan analiz araçlarıyla ilgili olarak.
Protokol
Örnekleri Njala Üniversitesi Kurumsal Inceleme Kurulu (NUıRB) protokol No sonra toplandı. IRB00008861/FWA00018924.
1. kan örneklerinden RNA ekstraksiyonu
- Türlerden tüm kan 3 mL toplamak 6 mL DNA/RNA stabilize reaktif reaksiyonu ile önceden doldurulan bir kan toplama tüpü içine analiz edilecek ( malzeme tablosunabakın) ve 5 kez tersine çevirme ile karıştırın. Kan örneğini 4 °C ' de bir aya kadar saklayın.
- Kan toplama tüpünün içeriğini 50 ml toplama tüpüne aktarın, 120 μL proteinaz K ekleyin ve 5 s için vortekslenir ile karıştırın. numuneyi oda sıcaklığında 30 dakika boyunca kulyattır.
- 5 s için karışımı ve girdap için izopropanol 9 ml ekleyin.
- Bir RNA arıtma döndürme sütununa bir rezervuar yerleştirin ( malzeme tablosunabakın) ve montajı bir vakum manifold üzerine yerleştirin (bkz. malzeme tablosu). Örnek karışımı rezervuar içine ekleyin. Tüm sıvı sütundan geçtikten kadar bir vakum uygulayın.
- Alternatif olarak, herhangi bir vakum manifold varsa, kan 700 μL bölümlerinde sütun üzerinden 30 s için tekrarlanan santrifüjleme ile 12.000 x g de santrifüjleme adımları arasında atılan akış ile geçirilebilir. Ancak, bu yaklaşık 26 santrifüjleme adımları gerektirir.
- RNA arıtma döndürme sütununu bir toplama tüpüne yerleştirin ve 400 μL DNA/RNA hazırlık tamponunu ekleyin (bkz. malzeme tablosu). 30 s için 12.000 x g 'de santrifüjün ve akışını atın.
- Eklemek 400 μL DNA/RNA yıkama tamponu (bkz. malzeme tablosu) sütuna, santrifüjden 12.000 x g için 30 s ve atın akış-through.
- 5 μL DNase ı (1 U/μL) karışımı (bkz. malzeme tablosu) 75 μL DNA sindirim tamponu (bkz. malzeme tablosu) ve karışımı sütuna ekleyin. Oda sıcaklığında 15 dakika boyunca inküye yapın.
- Sütun için 400 μL DNA/RNA hazırlık tamponunu ekleyin ve 30 s için 12.000 x g 'de Santrifüjü atın. akışını atın.
- 700 μL DNA/RNA yıkama tamponunu kullanarak sütunu yıkayın ve 30 s için 12.000 x g 'de Santrifüjü atın. akışını atın.
- 400 μL DNA/RNA yıkama tamponu ile adım 1,9 yineleyin ve 2 dakika boyunca 12.000 x g 'de santrifüjün tüm yıkama tamponunu çıkarıp sütunu kurutun. Koleksiyon tüpünden sütun kaldırırken, sütun alt sütunu toplama tüpünde arabellek ile ıslak emin olun.
-
Sütunu yeni bir 1,5 mL mikrosantrifüjü tüpüne yerleştirin ve 70 μL çekirdeksiz su ekleyin. Oda sıcaklığında 1 dakika boyunca inküye ve 12.000 x g'de 30 s santrifüj yapın. RNA at-80 °C ' ye daha fazla kullanım (veya hemen kullanımı) kadar saklayın.
- Isteğe bağlı: RNA ölçmek için, bir kısım almak ve bir UV spektrofotometre kullanarak konsantrasyonu belirlemek (bkz. malzeme tablosu).
2. cDNA içine NPC1 mRNA Ters transkripsiyon
- Bir 0,2 mL reaksiyon tüpünde, 8 μL şablon RNA (1 pg-2,5 μg RNA) ve 1 μL her biri 10X DNase tampon ve DNase enziminin (bkz. malzeme tablosu) ekleyin. 37 °C ' de 2 dak. Daha sonra reaksiyonu kısaca santrifüjün ve buzun üzerine yerleştirin.
- 4 μL ters transkriptaz ana karışımı ekleyin (bkz. malzeme tablosu) ve 6 μL reaksiyonsuz su, reaksiyon tüpüne ve hafifçe karıştırın. Reaksiyonu 10 dk 25 °C ' de (astar tavlama için), 50 °C ' de 10 dak (RNA 'nın ters transkripsiyonu için) ile tersten takip et. Enzimi etkisiz hale getirmek için 85 °C ' de 5 dakika boyunca inküye yapın.
- CDNA 'yı yeni 1,5 mL mikrosantrifüjü tüpüne aktarın ve daha fazla kullanım (veya hemen kullanın) kadar-80 °C ' ye saklayın.
3. NPC1 açık okuma çerçevesinin amplifikasyonu
-
İlk amplifikasyon adımı
- Bir touchdown PCR kurmak15,16 ile NPC1 cDNA yükseltmek için primer Set 1 (bkz. Tablo 1), sıcak başlangıç yüksek doğruluk DNA polimeraz kullanarak ( malzeme tablosunabakın) 50 μL reaksiyonunda uygun reaksiyon tamponu ile 1 μL şablonu ile birim. Mümkünse, buzun üzerindeki reaksiyonu veya 4 °C ' lik serin blokta ayarlayın.
- 98 °C ' de 30 s 'ye kadar ilk denatürasyon adımıyla, 10 s için 98 °C ' de denaturasyon ile 10 döngü, 65 °C ' de 20 s için astar tavlama, döngüsü başına 0,5 °C ' ye kadar sıcaklık düşürücü ve 72 °C ' de 1 dakika süreyle uzama ile reaksiyonu Tersiyere atın. Daha sonra, 98 °C ' de 10 s, 60 °C ' de 20 s, 72 °C ' de 1 dak, sonra da 72 °C ' de 5 dakika son uzama adımıyla ek 20 döngü çalıştırın.
-
Manyetik boncuklar kullanarak PCR arıtma
- 50 μL PCR ürününün 1,5 mL DNA-düşük bağlayıcı reaksiyon tüpüne aktarımı (bkz. malzeme tablosu). Resuspend manyetik boncuk (bkz. malzeme tablosu) tarafından iyice vortekslenir ve eklemek 50 μL boncuk PCR reaksiyon. İyi karıştırın. Numuneyi dönen bir mikserde (bkz. malzeme tablosu) Oda sıcaklığında 5 dakika (15 RPM 'de) ile kulyın.
- Kısaca numuneyi döndürün ve 1,5 mL mikrosantrifüjün tüpünü manyetik bir rafa yerleştirin (bkz. malzeme tablosu) manyetik boncuklar Pelet. Bir sonraki adıma devam etmeden önce süpernatant tamamen açıklığa kavuşturulana kadar bekleyin.
- Boncuk Pelet rahatsız etmeden süpernatant aspirate ve atın.
- Pipet 200 μL% 70 etanol reaksiyon tüpü içine ve 30 s için inküye. Pelet rahatsız etmeden etanol Aspirate ve atın. Toplam iki yıkanıyor için tekrarlayın. Hiçbir etanol kaldığını emin olun. Daha büyük bir pipet (örn. 1000 μL) ile ilk Aspire etmek ve daha sonra kalan etanol damlacıkları daha küçük bir pipet (örn. 10 μL) ile çıkarmak gerekebilir.
- Oda sıcaklığında 1 dakika boyunca hava-kuru Pelet.
- Manyetik rafa reaksiyon tüpü çıkarın, 30 μL çekirdeksiz su içinde Pelet pelletini ve oda sıcaklığında 2 dakika boyunca inküye.
- Reaksiyon tüpünü manyetik rafa geri yerleştirin ve boncuklar tamamen peletlenmiş olana kadar bekleyin.
- Pelet rahatsız etmeden süpernatant çıkarın ve yeni bir 1,5 ml reaksiyon tüpü aktarmak.
-
Yuvalanmış PCR tarafından barkod adaptörlerinin eklenmesi
- 5x reaksiyon tampon ve primer Set 2 (bkz. Tablo 1) ile sıcak başlangıç yüksek sadakat DNA polimeraz Ile 50 μL TOUCHDOWN PCR ayarlayın. Bu setteki astarlar, 3,2 adımda oluşturulan PCR ürünlerinin bağlanmasına izin vermek için hedef serisine özgü bir bölgeden oluşur (astar 1 dizilerinin içinde), sonraki barkodlama PCR reaksiyonunda hedef olarak kullanılan bir adaptör dizisi (CF. Bölüm 4). Mümkünse, buzun üzerindeki reaksiyonu veya 4 °C ' lik serin blokta ayarlayın. 3,2 bölümünde hazırlanmış 1 μL temizleşmiş PCR ürününü şablon olarak kullanın.
- 98 °C ' de 30 s 'nin ilk denatürasyon adımını kullanarak, 10 s için 98 °C ' de denaturasyon ile 10 döngü takiben, 65 0,5 °C ' de 20 s için astar tavlama ve 72 °C ' de 1 dakika boyunca uzama. Daha sonra, 98 °C ' de 10 s, 71 °C ' de 20 s, 72 °C ' de 1 dak, sonra da 72 °C ' de 5 dk. son uzama adımının reaksiyonu için daha fazla 30 döngü.
- 3,2 bölümünde açıklandığı gibi manyetik boncuklar kullanarak PCR ürününü temizleyin.
4. NPC1 Amplicons barcoding
- Bölüm 3 ' te oluşturulan her PCR ürünü için, 0,2 ml reaksiyon tüpünde bir barkodlama PCR reaksiyonu kullanarak 50 μL Taq DNA polimeraz 2x Master Mix (bkz. malzeme tablosu), bir PCR barkodu setinden barkod astarlardan biri 2 μL (bkz. malzeme tablosu ve Tablo 2), ve 1 μL TEMIZLEŞMIŞ PCR ürün adım 3.3.2 şablon olarak. 100 μL son hacmi elde etmek için 47 μL çekirdeksiz su kirası ekleyin.
- 95 °C ' de ilk denatürasyon olarak 3 dakika boyunca bir termocycte reaksiyonu inkübasyon yapın. Daha sonra, 95 °C ' de 15 s, 62 °C ' de 15 s ve 65 °C ' de 1,5 dak için 15 döngü çalıştırın. Son uzama olarak, 65 °C ' de 5 dakika boyunca reaksiyonu inkübasyon yapın.
- 3,2 altında açıklandığı şekilde PCR ürününü arındırın, ancak 30 μL ' lık çekirdeksiz su içinde 100 μL manyetik boncuk ve elute kullanın.
- Mümkünse, numuneyi bir UV spektrofotometresi kullanarak ölçmek.
5. Kütüphane hazırlığı
- 45 μL 'Lik bir hacimde 1 μg DNA toplamı için her örnekteki barkodlu DNA 'Yı eşit miktarda birleştirin (gerekirse, 0,2 mL reaksiyon tüpüne çekirdeksiz su ekleyin). UV spektrofotometresi yoksa, her numunenin eşit hacimleri kullanın. DA-tailing için, 7 μL End Prep reaksiyon tamponu (bkz. malzeme tablosu), 3 μL End-Prep enzim karışımı (bkz. malzeme tablosu) ve 5 μL çekirdeksiz su. Hafifçe tüp Flicking karıştırın.
- Reaksiyonu 20 °C ' de 5 dakika, ardından 65 °C ' de 5 dak.
- Reaksiyon ürününü Bölüm 3,2 ' de açıklandığı gibi arındırın, ancak 25 μL 'ye kadar çekirdeksiz su ile 60 μL manyetik boncuk ve elüt kullanın.
- Opsiyonel: UV spektrofotometresi kullanılarak numunenin konsantrasyonunu ölçmek Için 1 μL alın. Toplam tutar 700 ng 'den fazla olmalıdır.
- Yeni bir 1,5 mL DNA-düşük bağlayıcı reaksiyon tüpünde 2,5 μL 1D2 adaptör ile (bkz. malzeme tablosu) ve 25 μL künt/ta ligaz ana karışımı (bkz. malzemetablo) ile adım 5,3 ' den arındırılmış DNA 22,5 μL birleştirin, hafifçe Flicking ile karıştırın ve kısaca spin Aşağı. Oda sıcaklığında 10 dakika boyunca inküye yapın.
- Reaksiyon ürününü Bölüm 3,2 ' de açıklandığı gibi arındırın, ancak 20 μL manyetik boncuk kullanın, DNA 'ya 10 dakika bağlamanın inkübasyon süresini artırın, her biri 1 mL etanol ile iki yıkama adımı gerçekleştirin ve 46 μL 'de çekirdeksiz su ile elute yapın.
- Reaksiyonunun 45 μL 'yi 5 μL barkod adaptör karışımı ile adım 5,6 ile birleştirin (bkz. malzeme tablosu) ve bir DNA-düşük bağlayıcı reaksiyon tüpünde k/ta ligaz ana karışımı 50 μL. Hafifçe Flicking ile karıştırın ve oda sıcaklığında 10 dakika boyunca inküye.
- Reaksiyon ürününü Bölüm 3,2 ' de açıklandığı gibi arındırın, ancak 40 μL mıknatıslı boncuk kullanın, iki yıkama adımını, 140 μL ABB tampon ile yapın ( malzeme tablosunabakın) etanol yerine, bir manyetik raf üzerinde Flicking ve pelet tarafından boncuk pelletini. 15 μL 'de elüsyon tamponu (bkz. malzeme tablosu). DNA 'nın ilk bağlamasının boncuklar için inkübasyon sürelerini ve elüsyon adımını 10 dakikaya kadar artırın. elde edilen ürünü buzda veya 4 °C ' ye kadar kullanıma kadar saklayın.
6. akış hücresi kalite kontrolü
- Kullanmadan önce akış hücresinde kalite denetimi gerçekleştirin. Bu amaçla, sıralı aygıtı ana bilgisayara bağlayın ve yazılımı açın.
- Sıralı cihaza bir akış hücresi ekleyin ( malzeme tablosunabakın) ve seçici kutusundan akış hücresi tipini seçin ve kullanılabilir'i tıklatarak onaylayın.
- Ekranın altındaki akış hücresini denetle 'yi tıklatın ve doğru akış hücresi türünü seçin.
- Kalite denetimi başlatmak için Testi Başlat 'ı tıklatın. Akış hücresinin kullanılabilir olması için en az 800 aktif Nanopores gereklidir.
7. akış hücresi yükleniyor ve sıralı çalıştırma başlatılıyor
- Astar bağlantı noktası kapağını saat yönünde kaydırarak açın. 200 μL 'ye bir P1000 pipet ayarlayın ve ucu astar bağlantı noktasına takın. Pipeti 230 μL 'ye ayarlayın, ucu astar bağlantı noktasında tutarken, 20-30 μL tampon kadar çizmek ve hava kabarcıklarını çıkarın.
- Yeni bir 1,5 mL DNA-düşük bağlayıcı reaksiyon tüpü, 576 μL RBF tampon (bkz. malzeme tablosu) Ile 624 μL çekirdeksiz su ile birleştirerek astar karışımını hazırlar.
- Hazırlanan astar karışımının μL 'sini dikkatle pipet 800, astar girişine yerleştirin ve 5 dakika bekleyin. örnek bağlantı noktası kapağını kaldırın ve hazırlanan astar karışımının ek 200 μL 'sini astar girişine pipet atın.
- Pipet 35 μL RBF tampon yeni, temiz 1,5 mL DNA düşük bağlayıcı reaksiyon tüpü içine. Pipetleme ile LLB boncuk (bkz . malzeme tablosu) iyice karıştırın ve 25,5 μL boncuk RBF tampon ekleyin. 5,8 adım 2,5 μL çekirdeksiz su ve 12 μL DNA Kütüphanesi ekleyin ve pipetleme ile karıştırın.
- Örnek bağlantı noktası aracılığıyla akış hücresine yavaş dropwise moda numune karışımı 75 μL ekleyin.
- Örnek bağlantı noktası kapağını değiştirin, astar bağlantı noktasını kapatın ve sıralama cihazının kapağını kapatın.
- Yazılım içinde, akış hücresinin hala kullanılabilir olduğunu onaylayın, yeni bir deneme açın ve kullanılan kiti seçerek çalıştırma parametrelerini ayarlayın. Canlı temel arama 'yı seçin. Denemeyi Başlat 'ı tıklatarak sıralama çalışmasını başlatın. Yeterli deneysel veri toplanıncaya kadar sıralamayı Çalıştır devam edin.
Sonuçlar
Sunulan Protokolü test etmek için temsili bir denemede, RNA 'ya beş hayvan türünün 10 farklı kan örneğinden (örn. türler başına 2 kişi (keçi, koyun, domuz, köpek, sığır)) (Tablo 3) elde ettik. RNA verimleri ve kalite aşağıdaki ekstraksiyon özellikle örnek işleme ve depolama farklılıkları nedeniyle, yaygın olarak değişebilir. Temsilci deneyimizde 43 NG ile 543 NG başına μL (Tablo 3) arasındaki RNA konsantrasyonlarını gözlemledik. Ayrıca, RT-PCR amplifikasyon sonra, NPC-1 PCR-ürünleri jel Analizi çeşitli sonuçlar gösterdi (Şekil 2), örnekler için belirgin zayıf BANTLARı ile BC01 ve BC02 (her ikisi de keçi). Bu farklılıklar, örnek kalitedeki farklılıklar nedeniyle büyük olasılıkla, farklı türlerin NPC1 geni ile astar bağlamada farklılıklar yüzünden PCR etkinliğinin farklılıklarına rağmen hariç tutulamaz. Ancak, bu verim ve/veya amplifikasyon verimliliğinde farklılıklar Genel sıralama sonucunu belirgin bir şekilde etkilemez. Ayrıca, ek bir non-spesifik PCR ürün örnek BC10 (sığır) oluştu. Bu okur, veri analizinin bir parçası olarak bir başvuru sırasına elde edilen okuma eşleme sırasında atılır gibi Sanger sıralamasının aksine, bu tür spesifik olmayan ürünler nanopore sıralamanın sonuçlarını olumsuz etkilemez.
Her sıralamanın çalıştırılmadan önce, kullanılacak akış hücresinin kalite kontrolü, 800 toplam gözenekleri minimum gereksinimi ile şiddetle tavsiye edilir. Temsilcilik deneyimizde, bu kalite kontrolü 1.102 döndürülüyor. Veriler gerçek zamanlı olarak sağlanır ve hemen analiz edilebilir olduğundan, bir sıralı çalışma uzunluğu bireysel uygulama için ayarlanabilir (yani, istenen analiz için yeterli sıralı veri üretilinceye kadar). Bizim deneyler, sıralama çalışır genellikle bir gecede gerçekleştirilir, ve bizim temsilci deney durumunda yaklaşık 1.400.000 okuma sırasında böyle bir 14 saat çalıştırmak.
Gerçekleştirilecek veri çözümleme türüne bağlı olarak, yalnızca bir alt kümesini elde edilen okuma işlemek için tavsiye edilebilir. Temsilci denememiz durumunda, 10.000 bir alt kümesi daha fazla analiz için seçildi. Bu amaçla, sıralama çalıştırmak sırasında oluşturulan fastq dosyaları daha da bir Ubuntu 18,04 LTS ortamında işlenmiş ve nanopore sıralı veri demultiplexing için optimize parametreleri ile Flexbar v 3.0.3 kullanarak demultiplexed (barkod-kuyruk-Uzunluk 300 , barkod-hata oranı 0,2, barkod-Gap-ceza-1)12. Demultiplexing sonra, harita ve fikir birliği oluşturma okuma daha sonra farklı araçlar bir dizi kullanılarak yapılabilir, ancak nanopore sıralamanın Biyoinformatik yönü ayrıntılı bir tartışma Bu makalenin kapsamı dışındadır. Ancak, temsilcilik sonuçlarımız durumunda, Geneious 10.2.3 kullanılarak bir referans sırasına eşleme okuma yapıldı. 10.000 incelendiğinde, 5.457, 1.750 ve 2.000 nükleotidler arasında bir uzunluk gösterdi ve bu da iş akışımızın bir parçası olarak amplifikatörleşmiş PCR parçaları için beklenen boyutlara uyan (1.769 NT, Şekil 3). Okuma uzunluğu dağılımı ek bir zirve 250 arasında görüldü 500 nükleotidler, hangi spesifik olmayan PCR ürünlere atfedilebilir. Okunma demultiplexing 10 barkodlardan birine okuma% 87,6 atamasına izin/numuneler (Şekil 4) analiz. Her barkod için demultiplexed okuma oranı barkod 1 için% 3,4, barkodlu 10 için% 16,9 arasında değişmektedir; Ancak, genel olarak çok sayıda nedeniyle bu hala bu düşük bolluk barkod veri kümeleri için bile yüksek okuma derinliği ile arama anlamlı uzlaşma izin okur. Nitekim, NPC1 bir referans dizisi için sıralanmış okuma haritalama% 31,7 arasında sonuçlandı (Barkod 2) ve 100% (barkod 7 ve 8) referans haritalama okur, daha fazla okuma derinliği vererek 90 her örnek için herhangi bir pozisyonda okur. Bu daha sonra emin uzlaşma baz-ihmal edilebilir bir hata oranı ile arama izin vermek için yeterli daha fazladır.
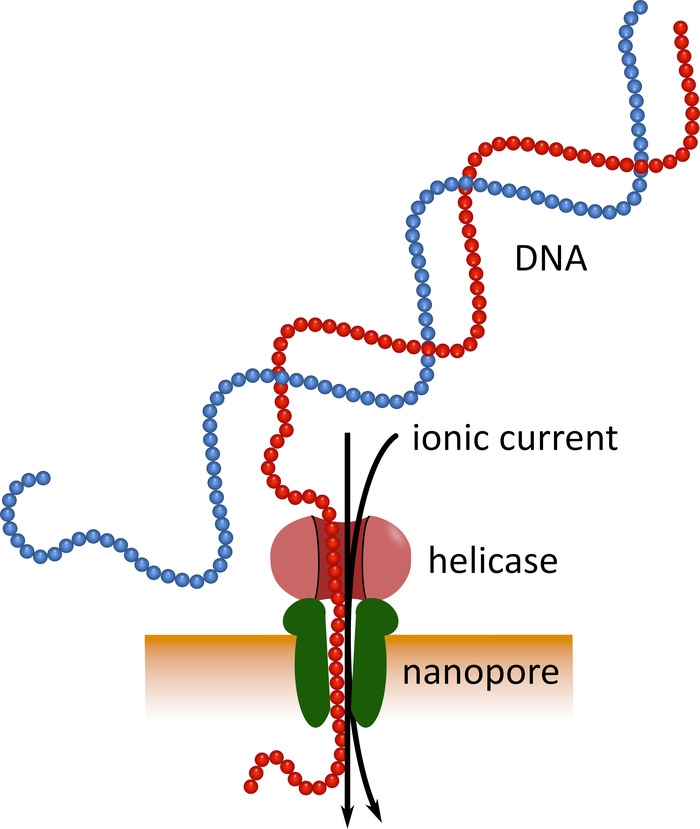
Şekil 1: nanopore teknolojisini kullanarak DNA sıralamanın şematik temsili. Tek telli bir DNA molekülü, elektrik dirençli bir membrana gömülü bir nanopore ile geçiş hızını düzenleyen bir helikazı ile geçer. Bir iyonik akım aynı anda gözenek geçer ve sürekli ölçülür. Gözenek içinde bulunan nükleotidler tarafından kaynaklanan akım modülasyonları algılanır ve DNA iplikteki nükleotid serisine Hesaplamalı olarak geri çevrilir. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
Şekil 2: mRNA 'Dan Niemann-Pick C1 'ın PCR ürünlerinin amplifikasyonu. mRNA keçi (BC01 ve 02), koyun (BC03 ve 04), domuz (BC05 ve 06), köpek (BC07 ve 08), ve sığır (BC09 ve 10) izole edildi. Yuvalanmış PCR ürünler 1x Tae tampon bir 0,8% agaroz jel ayrıldı (50x Tae tampon itibaren hazırlanmış: 242,28 g Tris tabanı, 57,1 ml buzul asetik asit, 100 ml 0,5 M EDTA, DH2O için 1 L, pH ayarlanmış 8,0) için 45 dk 100 V ve ile lekelenmiş Sybr güvenli. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
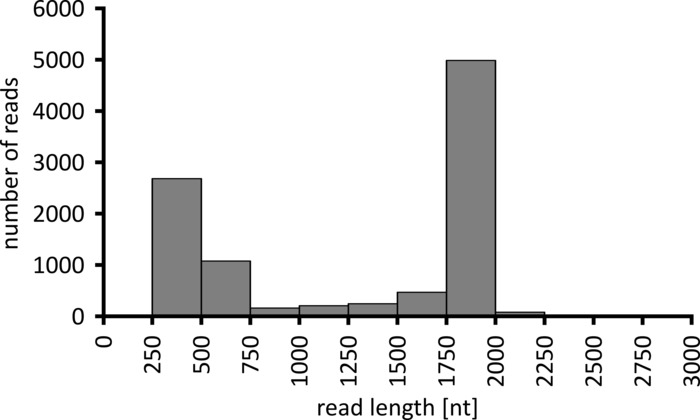
Şekil 3:10.000 okuma-uzunluk dağılımı temsili deneyden okur. Verilen okuma uzunluğu aralığına sahip alınan okuma sayısı belirtilir. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
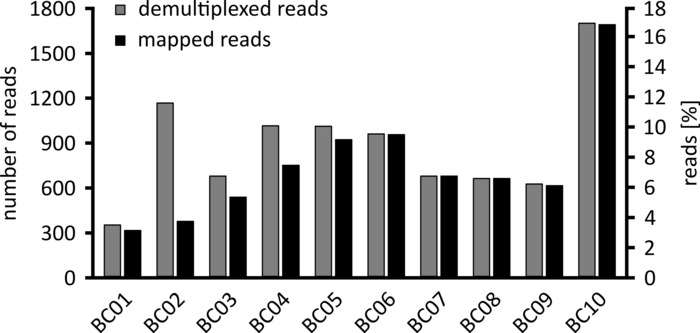
Şekil 4: demültiplexing sonra okuma dağılımı. Her barkod için demultiplexed (gri) ve eşleştirilen okur (siyah) sayısı ve yüzdesi gösterilir. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
Tablo 1: kullanılan astar setleri genel bakış. Hedef dizilerin ilk amplifikasyonu primer Set 1 ile yapılmıştır. Primer Set 2 daha sonra iç içe amplifikasyon ve adaptör ilavesi için kullanılmıştır. Adaptörler kırmızı renkte belirtilir. Bu dosyayı indirmek Için lütfen buraya tıklayın.
Tablo 2: barkod dizilerine genel bakış. Her sıralı numuneyi tanımlamak için bireysel barkodlar kullanılmıştır. Bu dosyayı indirmek Için lütfen buraya tıklayın.
Tablo 3: temsili denemede sıralanen kan örneklerinden ALıNAN RNA konsantrasyonları. Her beş türün iki kişinin RNA konsantrasyonları gösterilir ve 260/280 nm ve 260/230 nm 'de optik yoğunluklarının oranları gösterilir. Bu dosyayı indirmek Için lütfen buraya tıklayın.
Tartışmalar
Son yirmi yıl içinde, biyolojik numunelerin sıralanması, çok çeşitli konu alanlarındaki çalışmaların giderek önemli bir yönü haline gelmiştir. Enzimatik manipülasyon ve görüntü tabanlı veri edinme1 yinelemeli DÖNGÜLERI kullanarak DNA özelliklerinin yoğun bir dizi sıralamasına dayalı ikinci nesil sıralama sistemlerinin geliştirilmesi önemli ölçüde artış verimi ile karşılaştırıldığında geleneksel Sanger sıralama tekniği ve çoklu numunelerin yanı sıra belirli bir örnekteki çeşitli nükleik asit türlerinin çözümlerine paralel4' te izin verir. Ancak, sık kullanılan ikinci nesil sistemlerin çoğu için, sadece kısa okuma üretilir ve tüm platformlar hassas, hantal ve pahalı ekipman3,4güveniyor.
İkinci nesil sıralama platformlarının aksine, bu protokolde kullanılan sıralama aygıtı nanopore teknolojisine dayanır. Burada tek telli nüklik asit molekül bir nanopore üzerinden geçer, aynı zamanda aynı nanopore üzerinden akan bir iyonik akım modülasyon sonuçlanan, ve hangi ölçülebilir ve geri-nüklik asit molekül dizisini anlamak için tercüme. Bu üçüncü nesil sıralama yaklaşımı diğer yaklaşımlar üzerinde bir dizi avantaj sunar. Bu teknolojinin benzersiz çalışma prensibi ile doğrudan ilgili ana avantajları son derece uzun okuma uzunluğu üretilen (8,8 x 105 nükleotidler kadar okuma uzunlukları6BILDIRILMIŞTIR), sadece DNA sırası yeteneği ama Ayrıca RNA doğrudan, son zamanlarda tam bir grip virüs genom17için gösterildi, ve yeteneği gerçek zamanlı olarak onlar oluşturulan gibi veri analiz etmek, hangi dakika içinde patojenler hızlı metagenomics tespiti sağlar18. Ek pratik avantajları nanopore sıralı cihazın son derece küçük boyutu, herhangi bir laboratuar veya alan misyonları uzak yerlere19,20ve diğer sıralamaya kıyasla düşük fiyat kullanımına izin verir Platform. Çalışan maliyetleri açısından, şu anda yeni bir akış hücresi her sıralama çalışması için gereklidir, hangi maliyetleri sonuçları yaklaşık $1.100 akış hücresi ve kitaplık hazırlama reaktifler için çalışma başına. Bu maliyetler, bazı durumlarda akış hücresini yıkayarak ve yeniden kullanarak veya barkodlama ve tek bir çalışma içinde birden çok numune sıralamasıyla azaltılabilir. Ayrıca, akış hücresi bir roman türü şu anda bir akış hücresi adaptörü ("flongle" olarak adlandırılır) kullanımı gerektirecektir laboratuvarları, az sayıda tarafından beta-test ediliyor ve önemli ölçüde akış hücresi fiyatını azaltmak ve böylece maliyetleri çalışıyor.
Nanopore sıralamanın büyük eksiklik doğruluğu kalır, 83 aralığında tek okuma hassasiyeti ile 86%6bildirildi,21,22, ve yanlışlıkların çoğu ekleme/silme nedeniyle neden olan ( ındels)5,21. Ancak, yüksek okuma derinliği bu yanlışlıkları telafi edebilir ve yeni bir çalışma > 10 ' daki okuma derinliğinin genel doğruluğu > 99.8%21' e yükselebileceğini kuramsal hususları temel alarak önerdi. Yine de, özellikle analiz bir fikir birliği sırası düzeyinde yerine tek bir molekül düzeyinde gerçekleştirilecek ise, doğruluk daha da iyileştirmeler gerekli olacaktır. 1D 2 teknolojisinin, aynı nanopore tarafından sıralanan tek bir DNA molekülün her iki ipine de yol açmasına neden olan 1 adet ve barkod adaptörünün (CF. Bölüm 5,5) ilavesi temelinde bulunan bu protokolde açıklandığı şekilde kullanımı, okuma Her iki DNA ipinden gelen bilgi sıra belirlenmesi için kullanılabilir beri doğruluğu. Ayrıca, diğer sıralı teknolojilerin daha yüksek doğruluk ile nanopore sıralamanın (özellikle uzun okuma uzunluğu) avantajlarını birleştirmek için takip edilebilir bir geçici çözüm stratejisi, bir iskele olarak nanopore sıralama bilgileri kullanmaktır sonra diğer platformlardan sıralama verileri kullanılarak parlatılır6.
Burada sunulan protokolün başarısı için en kritik faktör, örnek kalite ve özellikle çıkarılan RNA 'nın miktarı ve kalitesidir. RNA 'nın uygun depolama ve istemi ekstraksiyonu, yeterli RNA verimi elde etmeye yardımcı olur. Uygun kan toplama tüpleri kullanımı bir aya kadar kan numunelerinin depolanması sağlar, ancak kan pıhtılaşması bir sorun olabilir, özellikle örnekleri yüksek sıcaklıklarda depolanmakta olan, alan koşullarında durumda olabilir hangi. İkinci kritik adım, hedef sıraların amplifikasyonu ve özellikle alan koşullarında PCR reaksiyonları genellikle standart Laboratuar koşulları altında daha az iyi performans7. Bu amaçla, dikkatli astar tasarımı ve optimizasyonu güçlü amplifikasyon elde etmek için en önemlisi. Ayrıca, bu protokolde kullanılan iç içe PCR yaklaşımlar ve touchdown PCR, hem özgüllüğü ve hedef gen amplifikasyon duyarlılığı artırabilir4,7. Nitekim, Liberya ve Gine 'de bu teknoloji ile iç içe protokollere sahip olan deneyimlerimiz, laboratuvar örneklerinden hedeflerin amplifikasyonu ve laboratuvar koşulları altında izin verilen astar setleri için bile alan örnekleriyle alan koşulları altında gerekli tek bir yuvarlak PCR (7 ve yayımlanmamış sonuçlar).
Bu daha kritik adımların aksine, kitaplık hazırlığı ve sıralamanın kendisi oldukça sağlam prosedürlerden ibaret. Ancak, alan koşullarında belirli ekipman parçalarının kullanılabilirliği gibi pratik sorunlar sorunlu olabilir. Örneğin, barkodlu numunelerin kütüphaneye hazırlanması öncesinde DNA konsantrasyonlarını belirlemek için bir UV spektrofotometresi gereklidir. Ancak, böyle bir cihaz alan koşulları altında kullanılabilir olmamalıdır, her örnek eşit hacimli sadece büyük tarafından hafifletilmiş sonra örnek giriş malzemesi farklılıkları ile, Kütüphane hazırlama için gerekli 45 μL yapmak için kombine edilebilir okuma sayısı. Benzer şekilde, sıralı çalıştırma için internet bağlantısı gereksinimi, temel arama artık çevrimiçi olarak gerçekleştirilebilecek ancak yerel olarak yapılabilir olsa da bir sorun olabilir; Ancak, gerekirse bu zorunluluk üretici tarafından belirli koşullarda kaldırılabilir.
Özetle, sunulan protokol, uzak konumlarda da dahil olmak üzere geleneksel sıralı donanıma erişimi olmayan konumlarda nispeten düşük maliyetli sıralamaya izin verir. Kolayca herhangi bir hedef RNA veya DNA adapte edilebilir, böylece araştırmacılar çok sayıda biyolojik sorulara cevap sağlar.
Açıklamalar
TH 2014 gelen Oxford nanopore Technologies (ONT) MinION erken erişim programına katıldı 2015, ve bir önceki çalışma için MinION cihazlar ve akış hücreleri aldı 7 Ulusal Sağlık Enstitüleri tarafından GERÇEKLEŞTIRILEN, ABD, ücretsiz veya düşük maliyetler . O, Londra Calling 2015 toplantı Londra 'da bu çalışmanın bir parçası sunmak için ONT tarafından davet edildi INGILTERE, ve ONT ulaşım ve konaklama için ödenir. Bu yazıda sunulan çalışmalar için, ONT 'tan hiçbir yararı (örneğin, düşük maliyetli donanım veya reaktifler, seyahat geri iadeler vb.) elde edilmiştir. , KF ve RS ifşa hiçbir şey yok.
Teşekkürler
Yazarlar, el yazması kritik okuma için Allison Groseth teşekkür ederiz. Bu çalışma, Federal tarım ve gıda (BLE) ofisi aracılığıyla Almanya Federal Cumhuriyeti Parlamentosu kararı temelinde Alman gıda ve Tarım Bakanlığı (BMEL) tarafından mali olarak destekleniyordu.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 1D2 adapter, barcode adapter mix, ABB buffer, elution buffer, RBF buffer, LBB beads | Oxford Nanopore Technologies | SQK-LSK308 | 1D² Sequencing Kit |
| Blood collection tube with DNA/RNA stabilizing reagent | Zymo Research | R1150 | DNA/RNA Shield - Blood Collection Tube |
| Blunt/TA ligase master mix | New England Biolabs | M0367S | Blunt/TA Ligase Master Mix |
| DNA-low binding reaction tube | Eppendorf | 30108051 | DNA LoBind Tube |
| DNase buffer and DNase | ThermoFisher Scientific | 11766050 | SuperScript™ IV VILO™ Master Mix with ezDNase™ Enzyme |
| Flow cell | Oxford Nanopore Technologies | FLO-MIN105.24 | flow cell R9.4 |
| Hot start high fidelity DNA polymerase | New England Biolabs | M0493L | Q5 Hot Start High-Fidelity DNA Polymerase (500 U) |
| Magnetic beads | Beckman Coulter | A63881 | Agencourt AMPure XP beads |
| Magnetic rack | ThermoFisher Scientific | 12321D | DynaMag-2 Magnet |
| Nanopore sequencing device | Oxford Nanopore Technologies | - | MinION Mk 1B |
| PCR barcoding kit | Oxford Nanopore Technologies | EXP-PBC001 | PCR Barcoding Kit I (R9) |
| Reverse transcriptase master mix | ThermoFisher Scientific | 11766050 | SuperScript™ IV VILO™ Master Mix with ezDNase™ Enzyme |
| RNA purification spin column, DNA/RNA prep buffer, DNA/RNA wash buffer, DNase I, DNA digestion buffer | Zymo Research | R1151 | Quick-DNA/RNA Blood Tube Kit |
| Rotating mixer | ThermoFisher Scientific | 15920D | HulaMixer Sample Mixer |
| Taq DNA polymerase | New England Biolabs | M0287S | LongAmp Taq 2x Master Mix |
| Ultra II End-prep kit | New England Biolabs | E7546S | NEBNext Ultra II End-Repair/dA-tailing Modul |
| UV spectrophotometer | Implen | - | NanoPhotometer |
| Vacuum manifold | Zymo Research | S7000 | EZ-Vac Vacuum Manifold |
Referanslar
- Shendure, J., Ji, H. Next-generation DNA sequencing. Nature Biotechnology. 26 (10), 1135-1145 (2008).
- Shendure, J., Lieberman Aiden, E. The expanding scope of DNA sequencing. Nature Biotechnology. 30 (11), 1084-1094 (2012).
- Liu, L., et al. Comparison of next-generation sequencing systems. Journal of Biomedicine and Biotechnology. 2012, 251364 (2012).
- Levy, S. E., Myers, R. M. Advancements in Next-Generation Sequencing. Annual Review of Genomics and Human Genetics. 17, 95-115 (2016).
- Lu, H., Giordano, F., Ning, Z. Oxford Nanopore MinION Sequencing and Genome Assembly. Genomics, Proteomics & Bioinformatics. 14 (5), 265-279 (2016).
- Jain, M., et al. Nanopore sequencing and assembly of a human genome with ultra-long reads. Nature Biotechnology. 36 (4), 338-345 (2018).
- Hoenen, T., et al. Nanopore Sequencing as a Rapidly Deployable Ebola Outbreak Tool. Emerging Infectious Diseases. 22 (2), 331-334 (2016).
- Quick, J., et al. Real-time, portable genome sequencing for Ebola surveillance. Nature. 530 (7589), 228-232 (2016).
- Carette, J. E., et al. Ebola virus entry requires the cholesterol transporter Niemann-Pick C1. Nature. 477 (7364), 340-343 (2011).
- Ndungo, E., et al. A Single Residue in Ebola Virus Receptor NPC1 Influences Cellular Host Range in Reptiles. mSphere. 1 (2), (2016).
- Martin, S., et al. A genome-wide siRNA screen identifies a druggable host pathway essential for the Ebola virus life cycle. Genome Medicine. 10 (1), 58 (2018).
- Roehr, J. T., Dieterich, C., Reinert, K. Flexbar 3.0 - SIMD and multicore parallelization. Bioinformatics. 33 (18), 2941-2942 (2017).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Kielbasa, S. M., Wan, R., Sato, K., Horton, P., Frith, M. C. Adaptive seeds tame genomic sequence comparison. Genome Research. 21 (3), 487-493 (2011).
- Don, R. H., Cox, P. T., Wainwright, B. J., Baker, K., Mattick, J. S. Touchdown' PCR to circumvent spurious priming during gene amplification. Nucleic Acids Research. 19 (14), 4008 (1991).
- Korbie, D. J., Mattick, J. S. Touchdown PCR for increased specificity and sensitivity in PCR amplification. Nature Protocols. 3 (9), 1452-1456 (2008).
- Keller, M. W., et al. Direct RNA Sequencing of the Coding Complete Influenza A Virus Genome. Scientific Reports. 8 (1), 14408 (2018).
- Greninger, A. L., et al. Rapid metagenomic identification of viral pathogens in clinical samples by real-time nanopore sequencing analysis. Genome Medicine. 7, 99 (2015).
- Castro-Wallace, S. L., et al. Nanopore DNA Sequencing and Genome Assembly on the International Space Station. Scientific Reports. 7 (1), 18022 (2017).
- Goordial, J., et al. In Situ Field Sequencing and Life Detection in Remote (79 degrees 26'N) Canadian High Arctic Permafrost Ice Wedge Microbial Communities. Frontiers in Microbiology. 8, 2594 (2017).
- Runtuwene, L. R., et al. Nanopore sequencing of drug-resistance-associated genes in malaria parasites, Plasmodium falciparum. Scientific Reports. 8 (1), 8286 (2018).
- Rang, F. J., Kloosterman, W. P., de Ridder, J. From squiggle to basepair: computational approaches for improving nanopore sequencing read accuracy. Genome Biology. 19 (1), 90 (2018).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır