Method Article
Секвенирование мРНК из цельной крови с использованием секвенирования Наноруды
В этой статье
Резюме
Секвенирование наноруды является новой технологией, которая позволяет экономически эффективной последовательности в отдаленных местах и ресурсов бедных условиях. Здесь мы представляем протокол для секвенирования mRNAs из цельной крови, совместимый с такими условиями.
Аннотация
Секвенирование в удаленных местоположениях и в условиях нехватки ресурсов создает уникальные проблемы. Секвенирование наноруды может быть успешно использовано в таких условиях и было развернуто в Западной Африке во время недавней эпидемии вируса Эбола, подчеркивая эту возможность. В дополнение к практическим преимуществам (низкая стоимость, простота транспортировки и использования оборудования), эта технология также обеспечивает фундаментальные преимущества по сравнению со вторым поколением подходов секвенирования, особенно очень долго читать длина, способность непосредственно последовательности РНК и доступность данных в режиме реального времени. Сырая точность чтения ниже, чем при других платформах секвенирования, что представляет собой основное ограничение этой технологии; Однако, это может быть частично смягчено высокой глубиной чтения, генерируемой. Здесь мы представляем протокол, совместимый с полем для секвенирования mRNAs, кодирующего Ниманн-пика С1, который является сотовым рецептором эболавивирусов. Этот протокол включает в себя извлечение РНК из образцов крови животных, а затем РТ-СР для обогащения цели, штриховое кодирование, подготовка библиотеки, и последовательность запуска сам, и может быть легко адаптирована для использования с другими целями ДНК или РНК.
Введение
Секвенирование является мощным и важным инструментом в биологических и биомедицинских исследованиях. Он позволяет анализировать геномы, генетические вариации и профили РНК, и, таким образом, играет важную роль в расследовании заболеваний человека и животных, так1,2. Секвенирование Сэнгера, один из старейших методов секвенирования ДНК, до сих пор обычно используется по сей день и является угловым камнем молекулярной биологии. За последние 50 лет, эта технология была улучшена для достижения чтения длины более 1 000 NT и точность выше, чем 99,999%1. Однако секвенирование Сэнгера также имеет свои ограничения. Секвенирование большего набора образцов или анализ целых геномов с этим методом является трудоемким и дорогостоящим1,3. Методы секвенирования ДНК второго поколения, такие как 454 Пиросеквенирование и технология «Озарина», позволили нам значительно сократить затраты и рабочую нагрузку, необходимые для секвенирования в течение последнего десятилетия, и привели к огромному увеличению количество биологической последовательности информации, доступной4. Тем не менее, Индивидуальная секвенирование выполняется с использованием технологий второго поколения, и секвенирование в полевых условиях является сложным, так как необходимое оборудование громоздко и хрупко (аналогично устройствам секвенирования Сэнгера) и часто приходится быть откалиброван и обслуживается специально подготовленным персоналом. Кроме того, для многих из второго поколения технологии чтения длины довольно ограничены, что часто делает ниже по течению биоинформатики анализа этих данных сложной.
Секвенирование третьего поколения с использованием карманных устройств секвенирования наноруды (см. таблицу материалов) может послужить альтернативой этим установленным секвенированию платформ. В этих устройствах молекула одноцепочечной ДНК или РНК проходит через наноруду одновременно с ионного тока, который затем измеряется датчиком (Рисунок 1). По мере того как прядь пересекает наноруду, обнаруживается модуляция тока нуклеотидами, присутствния в поры в любой момент времени, и вычислительно назад-переведена в нуклеотидный последовательность5. Из-за этого оперативного принципа, секвенирование наноруды позволяет как поколения очень долго читает (близко к 1 х 106 нуклеотидов6) и анализ последовательности данных в режиме реального времени. Баркодирование возможно путем присоединения определенных последовательностей нуклеотидов к нуклеиновых кислот в образце, что позволяет анализировать несколько образцов в одном запуске последовательности, таким образом увеличивая пропускную способность выборки и понижая затраты на образец. Из-за высокой переносимости и простоты использования устройства для секвенирования наноруд успешно используются в полевых условиях в ходе недавней эпидемии болезни, вызванной вирусом Эбола, в Западной Африке, что подчеркивает их пригодность для быстрого развертывания в отдаленных регионах7 , 8.
Здесь мы описываем подробный протокол, совместимый с полем для секвенирования мРНК, кодирующего белок Ниманн-пика С1 (NPC1), который является облигатными рецептором для филовирусов, таких как эболавивирусы, и, как было доказано, ограничивает восприимчивость видов к Эти вирусы9,10. Протокол включает в себя извлечение всей РНК из образцов крови, специфическое усиление NPC1 мРНК путем RT-СР, баркокодирование образцов, подготовку библиотеки и секвенирование с устройством секвенирования наноруды. Анализ данных не может обсуждаться из-за ограниченности пространства, хотя в репрезентативных результатах представлены некоторые базовые указания; Тем не менее, заинтересованном читателю упоминается Предыдущая публикация11 для более подробного описания рабочего процесса, который мы использовали, а также публикаций других12,13,14 для детальной информации в отношении инструментов анализа, используемых в этом рабочем процессе.
протокол
Образцы были собраны после того, как протокол Совета по институциональному обзору Ньябы (НЮСИБ) нет. IRB00008861/FWA00018924.
1. РНК извлечение из образцов крови
- Соберите 3 мл цельной крови из видов, которые будут проанализированы на трубке сбора крови, предварительно заполненной 6 мл ДНК/РНК стабилизирующего реагента (см. таблицу материалов) и смешайте инвертирование 5 раз. Храните образец крови на уровне 4 °C в течение одного месяца.
- Перенесите содержимое трубки для сбора крови на коллекционные трубки 50 mL, добавьте 120 мкл протеиназы K и смешайте с помощью вихрей для 5 с. инкубировать образец в течение 30 минут при комнатной температуре.
- Добавить 9 мл изопропанола в смесь и вихрь на 5 с.
- Поместите резервуар на колонку вращения РНК (см. таблицу материалов) и поместите сборку на вакуумный коллектор (см. таблицу материалов). Добавьте образец смеси в резервуар. Нанесите вакуум до тех пор, пока вся жидкость не пропроходит через колонну.
- В качестве альтернативы, если не имеется вакуумный коллектор, кровь может передаваться через столбец в 700 мкл частей путем повторного центрифугирования для 30 s при 12 000 x g с потоком-через отбрасываются между шагами центрифугирования. Однако для этого потребуется примерно 26 этапов центрифугирования.
- Поместите колонну РНК очищения в коллекционные трубки и добавьте буфер ДНК/РНК подготовки 400 мкл (см. таблицу материалов). Центрифуга на 12 000 x g для 30 s и отбросить поток-через.
- Добавить 400 мкл ДНК/РНК мыть буфер (см. таблицу материалов) в колонке, центрифуга на 12 000 x g для 30 s и отбросить поток через.
- Смешайте 5 мкл дна I (1 U/мкл) (см. таблицу материалов) с 75 мкл в буфере пищеварения ДНК (см. таблицу материалов) и добавьте смесь на колонку. Инкубировать в течение 15 минут при комнатной температуре.
- Добавьте 400 мкл в буфер подготовки ДНК/РНК к колонне и центрифуге при 12 000 x g для 30 с. Выбросьте поток через.
- Вымойте колонку с 700 мкл ДНК/РНК мыть буфер и центрифуги на 12 000 x g для 30 с. отбросить поток через.
- Повторите шаг 1,9 с 400 мкл ДНК/РНК мыть буфер и центрифуги на 12 000 x g в течение 2 минут, чтобы удалить все остаточный буфер мытья и высушить колонку. При удалении столбца из коллекционных труб убедитесь в том, чтобы не намочить нижнюю часть столбца с буфером в трубной коллекции.
-
Поместите колонну в новую пробирку микроцентрифуги 1,5 mL и добавьте 70 мл воды, свободной от нуклеазы. Инкубировать в течение 1 мин при комнатной температуре и центрифуге на 30 с в 12 000 x g. Хранить РНК при температуре-80 ° c до дальнейшего использования (или использовать немедленно).
- Необязательно: Для количественной оценки РНК, принимать Алиготе и определить концентрацию с помощью УФ-спектрофотометра (см. таблицу материалов).
2. Обратная транскрипция NPC1 мРНК в кДНК
- В реакционной трубке 0,2 mL добавьте 8 мкл шаблона РНК (1 ПГ до 2,5 мкг РНК) и 1 мкл каждого из 10-х буферного буфера и дндазы фермента (см. таблицу материалов). Инкубировать при температуре 37 ° c в течение 2 мин. Затем, центробежная реакция кратко и положите ее на лед.
- Добавьте 4 мкКл мастер-микса обратной транскриптазы (см. таблицу материалов) и 6 мкл Нуклеаза-свободной воды к реакционной трубке и аккуратно перемешайте. Инкубировать реакцию в термоциклера на 10 мин при температуре 25 ° c (для грунтовки отжига), а затем 10 мин при температуре 50 ° c (для обратной транскрипции РНК). Для инактивировать фермент, инкубации в течение 5 минут при температуре 85 ° c.
- Передача cDNA к новой 1,5 мл трубки микроцентрифуги и хранить при-80 ° c до дальнейшего использования (или использовать немедленно).
3. усиление рамок открытого чтения NPC1
-
Начальный шаг усиления
- Настройка приземления КЦР15,16 , чтобы усилить NPC1 cDNA с грунтовки набор 1 (см. таблицу 1), с помощью горячего начала высокой точности ДНК-полимеразы (см. таблицу материалов) с соответствующим буфером реакции в 50 мкл реакции объемом с 1 мкл шаблоном. Если возможно, установите реакцию на льду или в прохладном блоке 4 °C.
- Инкубировать реакцию в Термоциклер с начальным шагом денатурации 30-х при температуре 98 ° c, а затем 10 циклов с денатурации при 98 ° c для 10 s, грунтовка отжига для 20 s при 65 ° c, понижая температуру на 0,5 ° c за цикл, и удлинение в течение 1 мин при температуре 72 ° c. Затем, запустите дополнительные 20 циклов с 10 s при температуре 98 ° c, 20 s при 60 ° c, и 1 мин при 72 ° c, после чего заключительный шаг удлинения 5 мин при 72 ° c.
-
Очистка ЦР с использованием магнитных бусин
- Передача 50 мкл препарата ЦР в 1,5 mL ДНК-низкая связывания реакции трубки (см. таблицу материалов). Ресуспензируем магнитные бусы (см. таблицу материалов) тщательно потряхивая и добавить 50 мкл шарики к реакции ЦР. Хорошо перемешайте. Инкубировать образец на вращающемся смесителе (см. таблицу материалов) в течение 5 минут при комнатной температуре (при 15 оборотов в минуту).
- Кратко закрутить вниз образец и поместить пробку микроцентрифуги 1,5 mL на магнитной стойке (см. таблицу материалов) для того чтобы гранулы магнитные шарики. Подождите, пока супернатант полностью не проясняется, прежде чем продолжить следующий шаг.
- Аспирин супернатант, не нарушая шарик гранул и выбросить.
- Пипетка 200 мкл 70% этанола в реакционную трубку и инкубировать в течение 30 с. Аспоте этанола, не нарушая гранул и выбросить. Повторите в общей сложности две мойки. Убедитесь, что этанол не остается. Это может быть необходимо сначала аспирин с большим пипетки (например, 1000 мкл), а затем, чтобы удалить оставшиеся капли этанола с меньшим пипетки (например, 10 мкл).
- Воздушно-сухой гранулы на 1 мин при комнатной температуре.
- Снимите реакцию трубки от магнитной стойки, ресуспензируем гранулы в 30 мкл Нуклеаза-свободной воды и инкубировать в течение 2 мин при комнатной температуре.
- Поместите реакционную трубку обратно на магнитную стойку и подождите, пока бисер полностью не окупить.
- Снимите супернатант, не нарушая гранулят, и перенесите его на новый 1,5 mL-реакционную трубку.
-
Добавление адаптеров штрих-кодов на вложенный ЦР
- Настройка 50 мкл приземления СР с горячим началом высокой четкости ДНК полимеразы с 5-кратным буфером реакции и грунтом Set 2 (см. таблицу 1). Праймеры в этом наборе состоят из определенной области, имеющей определенную последовательность, чтобы обеспечить связывание продуктов ЦР, генерируемых в шаге 3,2 (внутри набора грунтовки 1), а также последовательность адаптеров, которая используется в качестве мишени при последующей реакции баркодировки (СР. раздел 4). Если возможно, установите реакцию на льду или в прохладном блоке 4 °C. Использование 1 мкл очищенного продукта ЦР, подготовленного в разделе 3,2 в качестве шаблона.
- Инкубировать реакцию смесь в Термоциклер с помощью начального шага денатурации 30-х при температуре 98 ° c, а затем 10 циклов с денатурации при 98 ° c для 10 s, грунтовка отжига для 20 s при 65 ° c, понижая температуру на 0,5 ° c за цикл , и удлинение на 1 мин при температуре 72 ° c. Впоследствии, инкубировать реакцию на еще 30 циклов для 10 s при 98 ° c, 20 s при 71 ° c, и 1 мин при 72 ° c, после чего окончательный шаг удлинения 5 мин при 72 ° c.
- Очистите продукт ЦР, используя магнитные бусы, как описано в разделе 3,2.
4. баркодирование NPC1 ам,
- Для каждого продукта КЦР, образующихся в разделе 3, создать штриховое кодирование реакцию ЦР в 0,2 ml реакционной трубки с использованием 50 мкл из Taq ДНК-полимеразы 2x Мастер микс (см. таблицу материалов), 2 мкл одного из праймеров штрих-кода от комплекта баркодировки ЦР (см. таблицу материалов и Таблица 2) и 1 мкл очищенного продукта ЦР от 3.3.2 шага в качестве шаблона. Добавьте 47 мкл свободной воды нуклеазы, чтобы получить окончательный объем 100 мкл.
- Инкубировать реакцию в термоциклера при температуре 95 ° c в течение 3 мин в качестве начальной денатурации. Впоследствии, запустить 15 циклов для 15 s при 95 ° c, 15 с при 62 ° c, и 1,5 мин при 65 ° c. В качестве конечного удлинения, инкубировать реакцию при температуре 65 ° c в течение 5 мин.
- Очистка продукта ЦР, как описано в 3,2, но использовать 100 мкл магнитных бусин и элюировать в 30 мкл Нуклеаза свободной воды.
- Если возможно, количественно измерить образец с помощью УФ-спектрофотометра.
5. Подготовка к библиотеке
- Объединить равное количество баркодированной ДНК из каждого образца в общей сложности 1 мкг ДНК в объеме 45 мкл (при необходимости, добавить Нуклеаза свободной воды) в 0,2 mL реакционной трубки. Если УФ-спектрофотометр не доступен, используйте равные объемы каждого образца. Для dA-хвостохранилища, добавить 7 мкл End-приготовительный буфер реакции (см. таблицу материалов), 3 мкл End-приготовительная смесь фермента (см. таблицу материалов), и 5 мкл Нуклеаза свободной воды. Смешайте осторожно, стряхивая трубку.
- Инкубировать реакцию в течение 5 минут при температуре 20 °C, а затем 5 минут при температуре 65 ° c в термоциклера.
- Очищаем реакцию продукта, как описано в разделе 3,2, но использовать 60 мкл магнитных бусин и элюировать в 25 мкл Нуклеаза свободной воды.
- Необязательно: Принимайте 1 мкл для количественной оценки концентрации образца с помощью УФ-спектрофотометра. Общая сумма должна составлять более 700 нг.
- Комбинат 22,5 мкл очищенной ДНК из шага 5,3 с 2,5 мкл 1d2 адаптер (см. таблицу материалов) и 25 мкл из тупой/TA лигазы Мастер микс (см. таблицу материалов) в новой 1,5 мл ДНК-низкая связывания реакции трубки, перемешать осторожно, щелкая, и кратко спина Вниз. Инкубировать в течение 10 минут при комнатной температуре.
- Очистить реакцию продукта, как описано в разделе 3,2, но использовать 20 мкл магнитных бусин, увеличить время инкубации для связывания ДНК до 10 мин, выполните два шага мыть с 1 мл этанола каждый, и elуты в 46 мкл Нуклеаза свободной воды.
- Комбинат 45 мкл из реакции продукта от шага 5,6 с 5 мкл штрих-кода адаптер микс (см. таблицу материалов) и 50 мкл тупой/TA лигазы мастер смеси в ДНК-низкая связывания реакции трубки. Смешайте осторожно, щелкая и инкубировать в течение 10 минут при комнатной температуре.
- Очистить реакцию продукта, как описано в разделе 3,2, но использовать 40 мкл магнитных бусин, делать два шага мыть с 140 мкл из АББ буфер (см. таблицу материалов) вместо этанола, ресуспензируем бусины, щелкая и гранулы на магнитной стойке. Элют в 15 Мкl в буфере (см. таблицу материалов). Увеличьте время инкубации для начального связывания дна к бусинкам так же как для шага делавания до 10 минут. Храните полученный продукт на льду или при температуре 4 °C до использования.
6. Проверка качества ячейки потока
- Выполните проверку качества на ячейку потока перед использованием. С этой целью Подключите устройство секвенирования к компьютеру-хозяину и откройте программное обеспечение.
- Вставьте ячейку потока (см. таблицу материалов) в устройство секвенирования и выберите тип потока ячейки из селекционером и подтвердите, нажав кнопку доступны.
- Нажмите кнопку потока проверки в нижней части экрана и выберите правильный тип ячейки потока.
- Щелкните Начало теста , чтобы начать проверку качества. Для использования ячейки потока требуется как минимум 800 активных наноруд.
7. Загрузка ячейки потока и запуск запуска секвенирования
- Откройте покрытие порта грунтовки, сдвинув его в направлении по часовой стрелке. Установите пипетку P1000 на 200 мкл и вставьте наконечник в порт грунтовки. Отрегулируйте пипетку до 230 мкл, сохраняя кончик в порту грунтовки, чтобы составить буфер 20-30 мкл и удалить пузырьки воздуха.
- В новом 1,5 mL ДНК-низкая связывающая реакция трубки подготовить смеси грунтовки путем объединения 576 мкл из буфера RBF (см. таблицу материалов) с 624 мкл Нуклеаза свободной воды.
- Тщательно Пипетка 800 мкл из подготовленного грунтовки смешать в порту грунтовка и подождите 5 мин. Поднимите крышку образца порта, и пипетки дополнительные 200 мкл из подготовленных грунтовки смесь в грунтовки порта.
- Пипетка 35 МКН в буфер RBF в новый, чистый 1,5 мл ДНК с низким уровнем связывания реакции трубки. Тщательно смешайте шарики LLB (см. таблицу материалов) по пипетки и добавить 25,5 мкл из бисера в БУФЕР RBF. Добавить 2,5 мкл Нуклеаза-свободной воды и 12 мкл библиотеки ДНК из шага 5,8 и смешать по пипетки.
- Добавить 75 мкл образца смеси в медленной выдрок в ячейку потока через образец порта.
- Замените образец портовой крышки, закройте порт грунтовки и закройте крышку устройства секвенирования.
- В рамках программы подтвердите, что ячейка по-прежнему доступна, откройте новый эксперимент и установите параметры запуска, выбрав используемый набор. Выберите живого базового вызова. Запустите секвенирование, нажав начать эксперимент. Продолжайте секвенирование до тех пор, пока не будут собраны достаточные экспериментальные данные.
Результаты
В ходе репрезентативного эксперимента по проверке представленного протокола мы извлекли РНК из 10 различных образцов крови из пяти видов животных (т.е. 2 особи на одного вида (коза, овца, свинья, собака, крупный рогатый скот)) (Таблица 3). Урожайность РНК и качество после извлечения могут сильно варьироваться, в частности из-за различий в обработке образцов и хранении. В нашем представительном эксперименте мы наблюдали концентрацию РНК между 43 нг и 543 нг на мкл (Таблица 3). Кроме того, после усиления по RT-СР, гель анализ ВСНП-1 ЦР-продуктов показали различные исходы (рис. 2), с заметно слабее полос для образцов BC01 и BC02 (оба козла). Эти различия, скорее всего, вызваны различиями в качестве образцов, хотя различия в эффективности СЦР из-за различий в грунтовки до гена NPC1 различных видов не могут быть исключены. Однако эти различия в эффективности доходности и/или усиления не заметно повлияли на общий исход секвенирования. Кроме того, в образце BC10 (крупный рогатый скот) было совершено дополнительное неспецифическое средство для ЦР. В отличие от секвенирования Сэнгера, такие неспецифическая продукция не оказывает негативного влияния на результаты секвенирования наноруды, так как они отбрасываются при отображении полученных считений в эталонных последовательности в рамках анализа данных.
До каждого запуска последовательности рекомендуется качественная проверка используемой ячейки потока, при минимальном требовании в 800 общих пор. В нашем представительном эксперименте эта проверка качества вернула 1 102 поры, доступные для секвенирования. Поскольку данные предоставляются в режиме реального времени и могут быть проанализированы немедленно, длина последовательности выполнения может быть скорректирована для индивидуального приложения (т.е. до тех пор, пока не будет произведена достаточная последовательность данных для требуемого анализа). В наших экспериментах, последовательность запусков, как правило, выполняются в одночасье, и в случае нашего представителя эксперимента мы получили около 1 400 000 читает в течение такого 14 h перспективе.
В зависимости от типа анализа данных, который будет выполняться, может быть целесообразно обработать только подмножество полученных считывает. В случае нашего репрезентативного эксперимента для дальнейшего анализа была выбрана подгруппа из 10 000 считывает. С этой целью, FASTQ файлы, созданные во время секвенирования запуска были дополнительно обработаны в Ubuntu 18,04 LTS окружающей среды, и демультилеплет с помощью сгибает v 3.0.3 с параметрами оптимизированы для демультилирования данных последовательности наноруды (штрих-хвост длины 300 , штрих-код-частота ошибок 0,2, штрих-код-штраф-1)12. После демультилирования чтение карт и формирование консенсуса может быть сделано с использованием ряда различных инструментов, но подробное обсуждение аспекта биоинформатики в последовательности наноруд выходит за рамки этой рукописи. Однако, в случае с нашими репрезентативными результатами, считка сопоставления с ссылкой последовательность была выполнена с использованием Geneious 10.2.3. Из 10 000 считывает проанализированы, 5 457 показал длину между 1 750 и 2 000 нуклеотиды, который соответствует ожидаемым размерам для фрагментов ЦР усиливается как часть нашего рабочего процесса (1 769 NT, рис. 3). Дополнительный пик в распределении длины читает наблюдалось между 250 к 500 нуклеотиды, которые могут быть отнесены к неспецифическое продуктов ЦР. Demultiplexing считывает позволил назначение 87,6% из читает до один из 10 штрих-кодов/образцов проанализированных (Диаграмма 4). Доля демултиплезированных считывает для каждого штрих-кода варьировалась от 3,4% для штрих-кодов от 1 до 16,9% для штрих-кодов 10; Однако, из-за общего большого количества читает это все еще позволенные содержательный консенсус вызывая с высокой глубиной прочитаны даже для этих наборов данных по штрихкодов более низкого изобилия. Действительно, отображение отсортированных считывает в эталонных последовательности NPC1 в результате между 31,7% (штрих-код 2) и 100% (штрих-код 7 и 8) считывает отображение на ссылку, давая читать глубину более 90 читает в любом положении для каждого образца. Это то более чем достаточно, чтобы позволить уверенно консенсус базового вызова с незначительным частота ошибок.
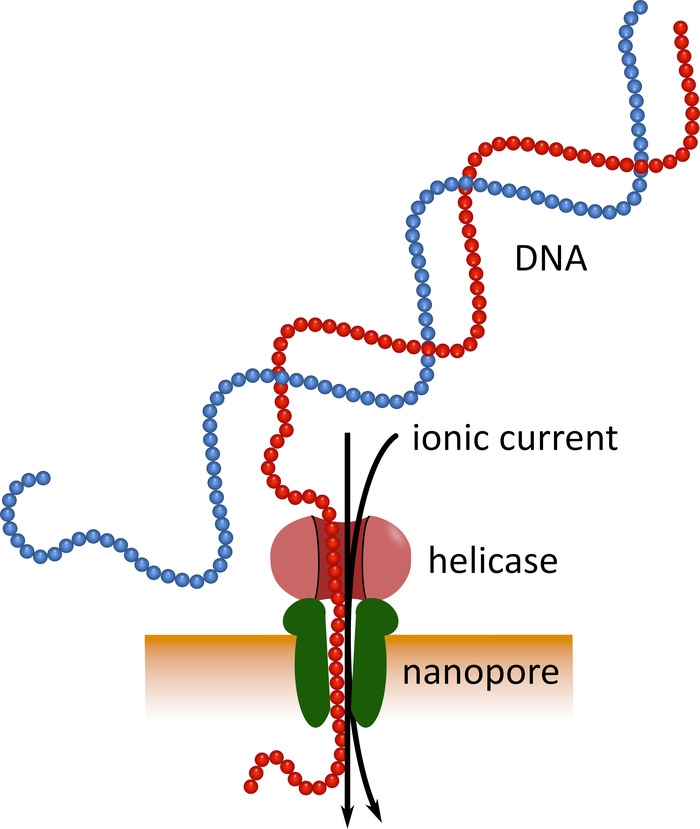
Рисунок 1: Схематическое представление секвенирования ДНК с использованием технологии наноруды. Одноцепочечной молекулы ДНК проходит через наноруды встроенных в электрически устойчивой мембраны, с гелиаза регулирующих скорость перехода. Ионный ток одновременно проходит через поры и непрерывно измеряется. Модуляции тока, вызванные нуклеотидами, присутствними в поре, выявляются и возвращаются назад-переведены в нуклеотидный последовательность нити ДНК. Пожалуйста, нажмите здесь, чтобы посмотреть увеличенном варианте этой фигуры.
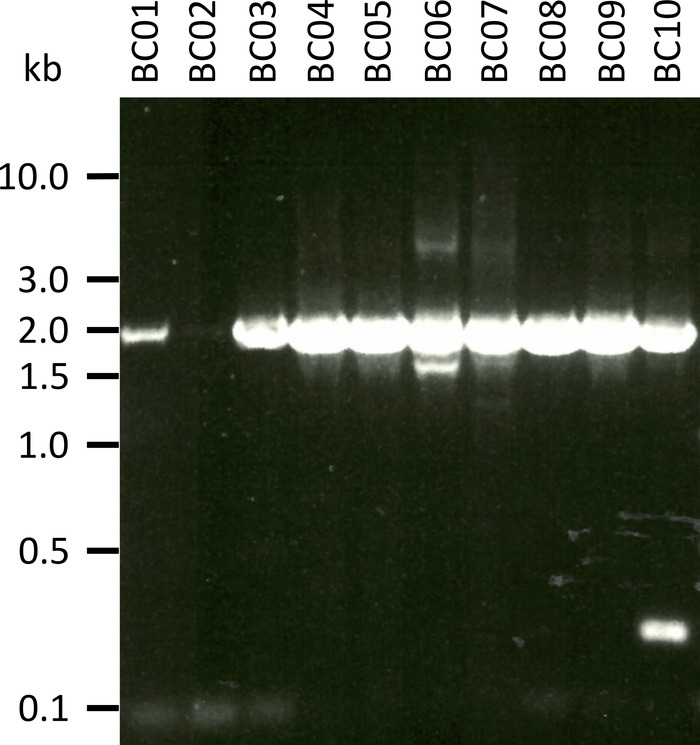
Рисунок 2: усиление продуктов ЦР Ниманн-пика С1 из мРНК. мРНК была изолирована от козы (BC01 и 02), овец (BC03 и 04), свиней (BC05 и 06), собак (BC07 и 08), и крупного рогатого скота (BC09 и 10). Вложенные продукты ЦР были разделены в 0,8% агарозы гель в 1x ТЭ буфера (подготовленный из 50x ТЭ буфер: 242,28 g из тарис базы, 57,1 мл ледниковой ацетотической кислоты, 100 мл 0,5 м ЭДТА, dH2O к 1 L, pH с поправкой на 8,0) для 45 мин при 100 V и окрашенных Sybr Сейф. Пожалуйста, нажмите здесь, чтобы посмотреть увеличенном варианте этой фигуры.
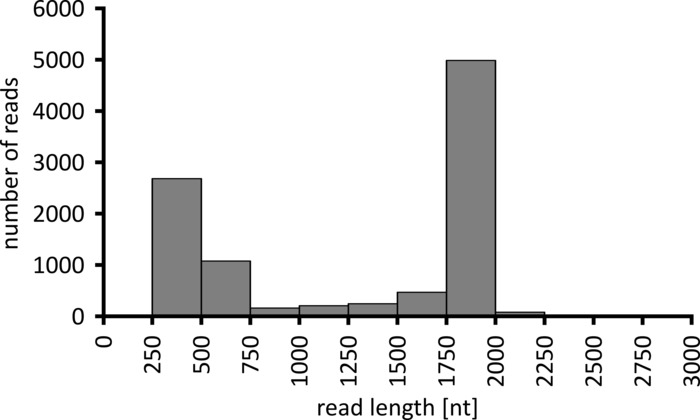
Рисунок 3: распространение 10 000 считывает из репрезентативного эксперимента. Указывается количество считений, полученных с учетом интервала считывания длины. Пожалуйста, нажмите здесь, чтобы посмотреть увеличенном варианте этой фигуры.
Рисунок 4: распределение считывает после демультиплетений. Показаны количество и процент демултиплезированных (серый) и отображается (черный) для каждого штрих-кода. Пожалуйста, нажмите здесь, чтобы посмотреть увеличенном варианте этой фигуры.
Таблица 1: обзор используемых наборов праймера. Начальное усиление целевых последовательностей было выполнено с набором грунтовки 1. Грунт комплект 2 затем использовался для вложенного усиления и дополнения адаптера. Адаптеры указаны красным цветом. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Таблица 2: обзор последовательности штрих-кодов. Для идентификации каждого секвенированного образца использовались индивидуальные штрих-коды. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Таблица 3: концентрации РНК, полученные после извлечения из образцов крови, последовательности в репрезентативной эксперимента. Показаны концентрации РНК двух особей от каждого из пяти видов, и указаны соотношения оптических плотностей на 260/280 Нм и 260/230 Нм. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
За последние два десятилетия секвенирование биологических образцов стало все более важным аспектом исследований в широком диапазоне предметно-значимых областей. Разработка систем секвенирования второго поколения на основе последовательности плотных массивов ДНК с использованием итеративных циклов ферментативной манипуляции и сбора данных на основе изображений1 резко увеличила пропускную способность по сравнению с традиционного метода секвенирования Сэнгера, и позволяет анализировать несколько образцов, а также различные виды нуклеиновых кислот в данном образце параллельно4. Однако для большинства широко используемых систем второго поколения производится только короткое считывает, и все платформы полагаются на чувствительное, громоздкое и дорогостоящее оборудование3,4.
В отличие от платформ секвенирования второго поколения, используемое в данном протоколе устройство секвенирования основано на технологии наноруды. Здесь молекула одноцепочечной нуклеиновой кислоты проходит через наноруду, что приводит к модуляции ионного течения, которое также течет через те же наноруды, и которые могут быть измерены и обратно-перевод вывести последовательность молекулы нуклеиновой кислоты. Такой подход к секвенированию третьего поколения придает ряд преимуществ по сравнению с другими подходами. Основные преимущества, которые непосредственно связаны с уникальным рабочим принципом этой технологии являются чрезвычайно долго читать длина производства (читать длины до 8,8 х 105 нуклеотиды были зарегистрированы6), способность последовательности не только ДНК, но также РНК напрямую, которая недавно была продемонстрирована для полного генома вируса гриппа17, и способность анализировать данные в режиме реального времени, как они создаются, что позволяет быстрое метагеномики обнаружения патогенов в течение минут18. Дополнительными практическими преимуществами являются крайне небольшие размеры устройства секвенирования наноруд, позволяющие использовать его в любой лаборатории или на полевых миссиях в отдаленные места19,20, и низкую цену по сравнению с другими секвенированием Платформ. Что касается затрат на запуск, то в настоящее время для каждого запуска последовательности требуется новая ячейка потока, которая приводит к затратам в размере около $1 100 за пробег для реагентов по подготовке потока и библиотек. Эти затраты могут быть сокращены в некоторых случаях путем промывки и повторного использования ячейки потока, или путем штрихкодирования и секвенирования нескольких образцов в одном запуске. Кроме того, новый тип потока ячейки в настоящее время бета-тестирования небольшого числа лабораторий, которые потребуют использования адаптера ячейки потока (так называемый "flongle"), и должны значительно уменьшить расход ячейки цена и, таким образом, расходы.
Основные недостатки последовательности наноруды остается его точностью, с одним читать точности в диапазоне от 83 до 86% сообщается6,21,22, и большинство из неточностей, вызванных вставки/удалений ( indels)5,21. Тем не менее, высокая глубина чтения может компенсировать эти неточности, и недавнее исследование предложил на основе теоретических соображений, что чтение глубина > 10 может увеличить общую точность, чтобы > 99.8%21. Тем не менее потребуются дальнейшие улучшения в точности, особенно если анализ будет выполняться на одном уровне молекул, а не на уровне консенсусной последовательности. Использование технологии 1D2 , описанной в этом протоколе, основанный на добавлении адаптеров 1d2 и штрих-кодов (см. раздел 5,5), которые приводят к тому, что обе нити одной молекулы ДНК секвенированы одним и тем же наноштуф, увеличивает прочитанное точность, поскольку информация из обеих нитей ДНК может быть использована для определения последовательности. Кроме того, обходной стратегии, которые могут быть продолжены для того, чтобы объединить преимущества секвенирования наноруды (особенно долго читать длина) с более высокой точностью других технологий секвенирования является использование наноруды секвенирования информации как эшафот, который является затем полируется с помощью секвенирования данных с других платформ6.
Наиболее важным фактором успеха протокола, представленного здесь, является качество выборки и, в частности, количество и качество извлеченной РНК. Надлежащее хранение и быстрое извлечение РНК помогают в достижении адекватного выхода РНК. Использование надлежащих труб для сбора крови позволяет хранить пробы крови в течение одного месяца, но свертывание крови может быть проблемой, особенно когда образцы хранятся при повышенных температурах, которые могут быть в случае полевых условиях. Второй критический шаг является усиление целевых последовательностей, и особенно в полевых условиях, реакции ЦР часто выполняют менее хорошо, чем в стандартных лабораторных условиях7. С этой целью тщательное проектирование грунтовки и оптимизация имеет первостепенное значение для достижения надежного усиления. Кроме того, вложенные ЦР-подходы и СР-посадки, используемые в этом протоколе, могут увеличить как специфичность, так и чувствительность целевого амплификации генов4,7. В самом деле, по нашему опыту в Либерии и Гвинее с этой технологией вложенные протоколы требовались в полевых условиях с образцами полей даже для наборов грунтовки, которые позволяли усиления целевых показателей из лабораторных образцов и в лабораторных условиях с один раунд ЦР (7 и неопубликованные результаты).
В отличие от этих более критических шагов, подготовка библиотек и последовательность выполнения сами по себе являются довольно надежными процедурами. Однако в полевых условиях практические вопросы, такие, как наличие определенных единиц оборудования, могут быть проблематичными. Например, УФ-спектрофотометр необходим для определения концентраций ДНК до подготовки библиотеки образцов баркодированных. Однако, если такое устройство не будет доступно в полевых условиях, равное количество каждого образца может быть просто объединено, чтобы составить 45 мкл, необходимый для подготовки библиотеки, с различиями в образце входных материалах, которые обычно смягчаются большим количество считывает. Аналогичным образом, необходимость подключения к Интернету для выполнения секвенирования может быть проблемой, даже если базовый вызов больше не должен выполняться онлайн, но может быть выполнен локально; Однако, эта необходимость может быть удалена при определенных обстоятельствах производителем, если требуется.
Таким образом, представленный Протокол допускает относительно низкую стоимость секвенирования в местах, где отсутствует доступ к традиционному оборудованию секвенирования, в том числе в удаленных местах. Она может быть легко адаптирована к любой целевой РНК или ДНК, тем самым позволяя исследователям ответить на многочисленные биологические вопросы.
Раскрытие информации
TH участвовал в программе «Оксфорд Наномор-технологии» (ОНТ) по раннему доступу с 2014 до 2015, и получил устройства Миньона и ячейки потока для предыдущего исследования 7 , проведенного национальными институтами здоровья, США, бесплатно или по сниженным расходам . Он был приглашен ОНТ представить часть этой работы на Лондонской вызова 2015 совещание в Лондоне, Великобритания, и ОНТ оплачивается для транспортировки и проживания. За работу, представленную в этой рукописи, не было получено никаких пособий (например, аппаратных средств или реагентов по сниженным затратам, возмещению путевых расходов и т.д.) из ОНТ. AM, КФ и РС нечего раскрывать.
Благодарности
Авторы благодарят Элисон Гросет за критическое чтение рукописи. Эта работа была финансово поддержана федеральным министерством продовольствия и сельского хозяйства Германии (BMEL) на основе решения парламента Федеративной Республики Германия через Федеральное ведомство по сельскому хозяйству и продовольствию (BLE).
Материалы
| Name | Company | Catalog Number | Comments |
| 1D2 adapter, barcode adapter mix, ABB buffer, elution buffer, RBF buffer, LBB beads | Oxford Nanopore Technologies | SQK-LSK308 | 1D² Sequencing Kit |
| Blood collection tube with DNA/RNA stabilizing reagent | Zymo Research | R1150 | DNA/RNA Shield - Blood Collection Tube |
| Blunt/TA ligase master mix | New England Biolabs | M0367S | Blunt/TA Ligase Master Mix |
| DNA-low binding reaction tube | Eppendorf | 30108051 | DNA LoBind Tube |
| DNase buffer and DNase | ThermoFisher Scientific | 11766050 | SuperScript™ IV VILO™ Master Mix with ezDNase™ Enzyme |
| Flow cell | Oxford Nanopore Technologies | FLO-MIN105.24 | flow cell R9.4 |
| Hot start high fidelity DNA polymerase | New England Biolabs | M0493L | Q5 Hot Start High-Fidelity DNA Polymerase (500 U) |
| Magnetic beads | Beckman Coulter | A63881 | Agencourt AMPure XP beads |
| Magnetic rack | ThermoFisher Scientific | 12321D | DynaMag-2 Magnet |
| Nanopore sequencing device | Oxford Nanopore Technologies | - | MinION Mk 1B |
| PCR barcoding kit | Oxford Nanopore Technologies | EXP-PBC001 | PCR Barcoding Kit I (R9) |
| Reverse transcriptase master mix | ThermoFisher Scientific | 11766050 | SuperScript™ IV VILO™ Master Mix with ezDNase™ Enzyme |
| RNA purification spin column, DNA/RNA prep buffer, DNA/RNA wash buffer, DNase I, DNA digestion buffer | Zymo Research | R1151 | Quick-DNA/RNA Blood Tube Kit |
| Rotating mixer | ThermoFisher Scientific | 15920D | HulaMixer Sample Mixer |
| Taq DNA polymerase | New England Biolabs | M0287S | LongAmp Taq 2x Master Mix |
| Ultra II End-prep kit | New England Biolabs | E7546S | NEBNext Ultra II End-Repair/dA-tailing Modul |
| UV spectrophotometer | Implen | - | NanoPhotometer |
| Vacuum manifold | Zymo Research | S7000 | EZ-Vac Vacuum Manifold |
Ссылки
- Shendure, J., Ji, H. Next-generation DNA sequencing. Nature Biotechnology. 26 (10), 1135-1145 (2008).
- Shendure, J., Lieberman Aiden, E. The expanding scope of DNA sequencing. Nature Biotechnology. 30 (11), 1084-1094 (2012).
- Liu, L., et al. Comparison of next-generation sequencing systems. Journal of Biomedicine and Biotechnology. 2012, 251364 (2012).
- Levy, S. E., Myers, R. M. Advancements in Next-Generation Sequencing. Annual Review of Genomics and Human Genetics. 17, 95-115 (2016).
- Lu, H., Giordano, F., Ning, Z. Oxford Nanopore MinION Sequencing and Genome Assembly. Genomics, Proteomics & Bioinformatics. 14 (5), 265-279 (2016).
- Jain, M., et al. Nanopore sequencing and assembly of a human genome with ultra-long reads. Nature Biotechnology. 36 (4), 338-345 (2018).
- Hoenen, T., et al. Nanopore Sequencing as a Rapidly Deployable Ebola Outbreak Tool. Emerging Infectious Diseases. 22 (2), 331-334 (2016).
- Quick, J., et al. Real-time, portable genome sequencing for Ebola surveillance. Nature. 530 (7589), 228-232 (2016).
- Carette, J. E., et al. Ebola virus entry requires the cholesterol transporter Niemann-Pick C1. Nature. 477 (7364), 340-343 (2011).
- Ndungo, E., et al. A Single Residue in Ebola Virus Receptor NPC1 Influences Cellular Host Range in Reptiles. mSphere. 1 (2), (2016).
- Martin, S., et al. A genome-wide siRNA screen identifies a druggable host pathway essential for the Ebola virus life cycle. Genome Medicine. 10 (1), 58 (2018).
- Roehr, J. T., Dieterich, C., Reinert, K. Flexbar 3.0 - SIMD and multicore parallelization. Bioinformatics. 33 (18), 2941-2942 (2017).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Kielbasa, S. M., Wan, R., Sato, K., Horton, P., Frith, M. C. Adaptive seeds tame genomic sequence comparison. Genome Research. 21 (3), 487-493 (2011).
- Don, R. H., Cox, P. T., Wainwright, B. J., Baker, K., Mattick, J. S. Touchdown' PCR to circumvent spurious priming during gene amplification. Nucleic Acids Research. 19 (14), 4008 (1991).
- Korbie, D. J., Mattick, J. S. Touchdown PCR for increased specificity and sensitivity in PCR amplification. Nature Protocols. 3 (9), 1452-1456 (2008).
- Keller, M. W., et al. Direct RNA Sequencing of the Coding Complete Influenza A Virus Genome. Scientific Reports. 8 (1), 14408 (2018).
- Greninger, A. L., et al. Rapid metagenomic identification of viral pathogens in clinical samples by real-time nanopore sequencing analysis. Genome Medicine. 7, 99 (2015).
- Castro-Wallace, S. L., et al. Nanopore DNA Sequencing and Genome Assembly on the International Space Station. Scientific Reports. 7 (1), 18022 (2017).
- Goordial, J., et al. In Situ Field Sequencing and Life Detection in Remote (79 degrees 26'N) Canadian High Arctic Permafrost Ice Wedge Microbial Communities. Frontiers in Microbiology. 8, 2594 (2017).
- Runtuwene, L. R., et al. Nanopore sequencing of drug-resistance-associated genes in malaria parasites, Plasmodium falciparum. Scientific Reports. 8 (1), 8286 (2018).
- Rang, F. J., Kloosterman, W. P., de Ridder, J. From squiggle to basepair: computational approaches for improving nanopore sequencing read accuracy. Genome Biology. 19 (1), 90 (2018).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены