A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
طريقة لقياس التمثيل الغذائي في التصنيف القطعان المجتمعات خلية مجمع عن طريق مستقر النظائر للبحث عن المفقودين
In This Article
Summary
This article describes a method for studying cellular metabolism in complex communities of multiple cell types, using a combination of stable isotope tracing, cell sorting to isolate specific cell types, and mass spectrometry.
Abstract
Mammalian cell types exhibit specialized metabolism, and there is ample evidence that various co-existing cell types engage in metabolic cooperation. Moreover, even cultures of a single cell type may contain cells in distinct metabolic states, such as resting or cycling cells. Methods for measuring metabolic activities of such subpopulations are valuable tools for understanding cellular metabolism. Complex cell populations are most commonly separated using a cell sorter, and subpopulations isolated by this method can be analyzed by metabolomics methods. However, a problem with this approach is that the cell sorting procedure subjects cells to stresses that may distort their metabolism.
To overcome these issues, we reasoned that the mass isotopomer distributions (MIDs) of metabolites from cells cultured with stable isotope-labeled nutrients are likely to be more stable than absolute metabolite concentrations, because MIDs are formed over longer time scales and should be less affected by short-term exposure to cell sorting conditions. Here, we describe a method based on this principle, combining cell sorting with liquid chromatography-high resolution mass spectrometry (LC-HRMS). The procedure involves analyzing three types of samples: (1) metabolite extracts obtained directly from the complex population; (2) extracts of "mock sorted" cells passed through the cell sorter instrument without gating any specific population; and (3) extracts of the actual sorted populations. The mock sorted cells are compared against direct extraction to verify that MIDs are indeed not altered by the cell sorting procedure itself, prior to analyzing the actual sorted populations. We show example results from HeLa cells sorted according to cell cycle phase, revealing changes in nucleotide metabolism.
Introduction
الكائنات العليا تحتوي على مجتمعات معقدة من أنواع الخلايا المتميزة التي تتعاون لتحقيق المزيد من الوظائف المعقدة. على سبيل المثال، وأورام تحتوي ليس فقط الخلايا السرطانية، ولكن أيضا الخلايا الليفية، الخلايا التي تشكل الأوعية الدموية، والخلايا في كثير من الأحيان المناعة تتسرب 1؛ يحتوي الدم على خليط معقد من عشرات الأنواع الفرعية الخلايا المناعية 2. ويمكن أن تتألف حتى خطوط خلايا مستنبتة من القطعان متعددة، مثل اللمعية وفرعية القاعدية من خلايا سرطان الثدي 3. وعلاوة على ذلك، أنواع الخلايا المتميزة التي تتعايش يمكن أن تظهر الأيض "التعاون". على سبيل المثال، في الدماغ، ويعتقد أن الخلايا النجمية لتحويل الجلوكوز إلى اللاكتات، وهو بعد ذلك "تغذية" إلى الخلايا العصبية التي تؤكسد هذه الركيزة 4؛ اللمفاويات التائية هي في بعض السياقات التي تعتمد على الخلايا الجذعية المجاورة كمصدر للالسيستين 5؛ والخلايا السرطانية قد تتعاون مع أسوالليفية ciated في الأورام 6. لفهم سلوك الأيض مثل هذه الأنظمة، لا بد من فصل وقياس الأنشطة الأيضية لمختلف أنواع الخلايا الحالية.
إلى حد بعيد الأسلوب الأكثر استخداما على نطاق واسع لفصل أنواع الخلايا غير مضان تنشيط الفرز الخلية. هذه الطريقة تنطبق على نطاق واسع، شريطة أن يكون نوع من الخلايا أو دولة من الفائدة يمكن أن يكون "المسمى" باستخدام الأجسام المضادة الفلورسنت والتعبير عن البروتينات الفلورية هندسيا، أو الأصباغ الأخرى. خيار واحد هو أن خلايا أنواع منفصلة في البداية من خلال فارز الخلية، وإعادة ثقافة أنواع الخلايا الفردية التي تم الحصول عليها، ثم قم بإجراء دراسات التمثيل الغذائي لهذه الثقافات 7. ومع ذلك، وهذا أمر ممكن فقط إذا كان نوع من الخلايا أو النمط الظاهري مستقرة في ظروف ثقافة، ولا يمكن التقاط السلوك عابرة مثل دول دورة الخلية، ولا التعاون الأيض في المشترك الثقافات. لمثل هذه الحالات، يجب أن تقاس عملية التمثيل الغذائي مباشرة على ذلكخلايا rted. هذا هو التحدي منذ الخلية فرز إجراء تخضع خلايا للضغوط التي قد تشوه عملية الأيض لديهم 8، ونحن ندرك فقط عدد قليل من الدراسات اتخاذ هذا النهج 9 و 10. على وجه الخصوص، وجدنا أن نواتج رئيسية مثل الأحماض الأمينية قد تسرب من خلايا أبقى في الخلية فرز العازلة، بحيث قياسات وفرة الأيض المطلقة لم تعد موثوقة 11 (على الرغم من أن المقارنة النسبية بين كسور فرزها قد يكون لا يزال قيما).
للتحايل على هذه القضايا، ونحن تسمية الخلايا مع نظائر مستقرة قبل الفرز، والتركيز على أجهزة الإنترنت النقالة في الأيض الخلوية، بدلا من وفرة الأيض. منذ تتشكل أجهزة الإنترنت النقالة على مدى فترات زمنية أطول، ينبغي أن تكون أقل تأثرا التعرض على المدى القصير لشروط الفرز. نحن قياس أجهزة الإنترنت النقالة باستخدام كامل المسح الضوئي عالية الدقة الطيفي، وهي حساسة بما يكفي لتوفير داتا على مئات من المركبات بدءا من حوالي 500،000 خلايا فرزها، والتي تتطلب حوالي 30-60 دقيقة من الخلايا وقت الفرز. مقارنة بين "وهمية فرز" السيطرة (خلايا مرت الصك خلية فارز دون النابضة أية مجموعة من السكان معين) واستخراج المستقلب مباشرة من يرصد الطبق الثقافة للتأكد من أن أجهزة الإنترنت النقالة المرصودة هي تمثيلية من تلك الموجودة في الثقافة الأصلية. اعتمادا على اختيار استشفاف النظائر المستقرة، ومختلف المسارات الأيضية يمكن دراستها مع هذا الأسلوب.
Protocol
1. المستقلب استخراج
- استخراج من الطبق
- خلايا الثقافة في لوحة 6 جيدا في يثلث في النظائر وصفت وسائل الإعلام ثقافة مستقرة + ملاحق مدال (المصل أو المكملات النمو الأخرى) حتى تصبح الخلايا 75٪ متموجة.
ملاحظة: هنا ثقافة خلايا هيلا لمدة 48 ساعة في RPMI تحتوي على 40٪ U- 13 C-الجلوكوز و 70٪ U- 13 C، 15 N2-الجلوتامين و 5٪ مدال FBS (مصل بقري جنيني). يستخدم مدال FBS للتخلص من نواتج الأيض الوزن الجزيئي الصغيرة التي قد تلوث وسائل الإعلام المسمى. ينصح زراعة الخلايا في ملحق مدال قبل التجربة الحقيقية لضمان الخلايا تنمو عادة في المتوسط. ومدال ملاحق في 0.15 حل M كلوريد الصوديوم بين عشية وضحاها باستخدام جلد الثعبان غسيل الكلى الأنابيب. - في يوم سائل الإعلام والثقافة استخراج فضلات، وشطف الآبار مرتين مع HBSS 500 ميكرولتر البارد ثم تخلص منه.
ملاحظة: هنا استخدام HBSS التي تحتوي على 40٪ U-13 C-الجلوكوز لأنه هو أيضا في وسائل الإعلام والثقافة. - إضافة 600 ميكرولتر 100٪ الميثانول قبل تبريده على الثلج الجاف.
- نقل طبق لتجف الجليد وإزالة المواد خلية مع مكشطة الخلية.
- بعناية ماصة مقتطفات الخلية إلى أنبوب microcentrifuge ومخزن في -80 درجة مئوية حتى تحليل الطيف الكتلي.
- خلايا الثقافة في لوحة 6 جيدا في يثلث في النظائر وصفت وسائل الإعلام ثقافة مستقرة + ملاحق مدال (المصل أو المكملات النمو الأخرى) حتى تصبح الخلايا 75٪ متموجة.
- استخراج الخلايا مرتبة وهمية
- خلايا الثقافة في صحن 100 ملم في يثلث في النظائر وصفت وسائل الإعلام ثقافة + ملاحق مدال مستقرة.
ملاحظة: هنا ثقافة خلايا هيلا لمدة 48 ساعة في صحن 100 ملم لعدد كبير من الخلايا (~ 4 × 10 6) هناك حاجة إلى الحصول على 500،000 خلايا فرزها. وقد يتطلب هذا العدد من هيلا خلايا استخراج للحصول على قياس جيد الأيض. وكان وسائل الإعلام ثقافة استخدام RPMI تحتوي على 40٪ U- 13 C-الجلوكوز و 70٪ U- 13 C، 15 N2-الجلوتامين و 5٪ مدال FBS. - في يوم من الاستخراج، وسائل الإعلام ثقافة نبذ، وشطف الآبار ثإيث 1.5 مل دافئ HBSS ثم تخلص منه.
- فصل الخلايا عن طريق إضافة 1.5 مل التربسين / EDTA لمدة 4 دقائق عند 37 درجة مئوية. نفذ الخطوات التالية في 4 درجات مئوية أو في الجليد.
- تعطيل التربسين وذلك بإضافة 3 مل الجليد HBSS الباردة (هانك المتوازن محلول الملح) + ملحق مدال.
- جمع الخلايا في أنبوب 15 مل وأجهزة الطرد المركزي في 750 x ج لمدة 3 دقائق.
- Resuspend وبيليه في HBSS + مدال ملحق + 1 ملم EDTA بتركيز 1-2 × 10 6 خلية / مل، تمر عبر 40 ميكرومتر مصافى الخلية للحصول على خلايا واحدة، ونقل إلى أنبوب 5 مل.
- خلايا من خلال فرز النابضة خلية فارز فقط لsinglets، ووأجهزة الطرد المركزي فرز الخلايا في 750 x ج لمدة 3 دقائق في 4 درجات مئوية.
ملاحظة: هنا نوع خلايا هيلا بمعدل 1000 أحداث / ثانية، مع الضغط أداة 27 رطل، وباستخدام فوهة 100 ميكرومتر. خلايا هيلا كبيرة ولذلك يجب فرز بمعدل الحدث بطيئة وفوهة كبيرة للحصول على بيليه كما سليمة قدر الإمكان. إبقاء الخلايا في كتل الباردة throughouر الفرز لتقليل عملية التمثيل الغذائي. وصفت وصفا شاملا لإجراء الفرز سابقا 12. - تجاهل طاف و resuspend بيليه في 50 ميكرولتر الجليد درهم البارد 2 O للحصول على بيليه متجانسة قبل إضافة الميثانول. إضافة الميثانول البارد مباشرة إلى بيليه تشكل بيليه الصلبة والتي من الصعب ل resuspend.
- استخراج الأيض وذلك بإضافة 540 ميكرولتر الميثانول يوضع في الثلج الجاف والحفاظ على مقتطفات في -80 درجة مئوية حتى اللوني السائل - عالية الدقة الطيف الكتلي (LC-HRMS) التحليل.
- خلايا الثقافة في صحن 100 ملم في يثلث في النظائر وصفت وسائل الإعلام ثقافة + ملاحق مدال مستقرة.
- استخراج دورة الخلية فرز الخلايا
- خلايا الثقافة في صحن 100 ملم في يثلث في وسائل الإعلام ثقافة + ملاحق مدال.
ملاحظة: هنا ثقافة خلايا هيلا تحتوي على Geminin Fucci الأخضر (mAG1-hGem) التحقيق 13 الذي يسمح للفرز من G1 (السلبي) وSG2M الخلايا (إيجابية). الخلايا يمكن تربيتها في وسائل الإعلام النظائر تتبع مستقرة لليالي طويلة كما هو مطلوب. هنا لدينا مثقف لهم لمدة 46 ساعة في وسائل الإعلام الخالي من الملصقات، ثم قمنا تحولت إلى RPMI تحتوي على 40٪ U- 13 C-الجلوكوز و 70٪ U- 13 C، 15 N2-الجلوتامين 2 ساعة قبل بدء الفرز. وقد تم ذلك من أجل أن تكون قادرة على دراسة مراحل دورة الخلية مما يتطلب وضع العلامات نبضة قصيرة. - في يوم من الاستخراج، وسائل الإعلام ثقافة نبذ، وشطف الآبار مع 1.5 مل HBSS الدافئ ثم تخلص منه.
- فصل الخلايا عن طريق إضافة 1.5 مل التربسين / EDTA لمدة 4 دقائق عند 37 درجة مئوية. نفذ الخطوات التالية في 4 درجات مئوية أو في الجليد.
- تعطيل التربسين وذلك بإضافة 3 مل الجليد الباردة HBSS + ملحق مدال.
- جمع الخلايا في أنبوب 15 مل وأجهزة الطرد المركزي في 750 x ج لمدة 3 دقائق.
- Resuspend وبيليه في HBSS + مدال ملحق + 1 ملم EDTA بتركيز 1-2 × 10 6 خلية / مل، تمر عبر 40 ميكرومتر مصافى الخلية للحصول على خلايا واحدة، ونقل إلى أنبوب 5 مل.
- خلايا الفرز من خلال خلية فارز زating من الحطام والحلل، ثم النابضة للعلامة الخلية من الفائدة، وأجهزة الطرد المركزي فرز الخلايا في 750 x ج لمدة 3 دقائق في 4 درجات مئوية.
ملاحظة: هنا نوع خلايا هيلا بمعدل 1000 أحداث / ثانية، مع الضغط أداة 27 رطل، وباستخدام فوهة 100 ميكرومتر. إبقاء الخلايا في كتل الباردة طوال الفرز لخفض معدل الأيض. - تجاهل طاف و resuspend بيليه في 50 ميكرولتر الجليد درهم البارد 2 O للحصول على بيليه متجانسة قبل إضافة الميثانول. إضافة الميثانول البارد مباشرة إلى بيليه تشكل بيليه الصلبة والتي من الصعب ل resuspend.
- استخراج الأيض وذلك بإضافة 540 ميكرولتر الميثانول يوضع في الثلج الجاف والحفاظ على مقتطفات في -80 درجة مئوية حتى التحليل LC-إدارة الموارد البشرية.
- خلايا الثقافة في صحن 100 ملم في يثلث في وسائل الإعلام ثقافة + ملاحق مدال.
تحليل 2. الطيف الكتلي
ملاحظة: نحن هنا وصف بروتوكول لتحليل مقتطفات خلية على نظام LC-إدارة الموارد البشرية. أي طرق الايض لتحليل مقتطفات خلية يمكن استخدامها. مسح كاملقد يكون من المفيد تحليل للكشف عن مجموعة واسعة من المركبات.
- معايرة أداة باستخدام مزيج معايرة الطيف إشارة الشامل.
- مقتطفات الخلية ذوبان الجليد على الجليد لمدة 30 دقيقة ودوامة لمدة 15 ثانية.
- نقل 100 ميكرولتر من استخراج الخلايا إلى تصفية تدور وأجهزة الطرد المركزي لمدة 10 دقيقة في 13000 x ج في 4 درجات مئوية.
- ضخ 12.5 ميكرولتر من الترشيح على النظام LC-إدارة الموارد البشرية.
- الأيض منفصلة باستخدام Zwitterionic التفاعل ماء اللوني السائل (زيوريخ-HILIC) عمود (150 ملم × 4.6 ملم، 5 ميكرون حجم الجسيمات) مزودة العمود حارس زيوريخ-HILIC (20 مم × 2.1 مم) باستخدام شطف التدرج من 0.1٪ الفورمات الحمض الموجود في الماء (مذيب) والأسيتونتريل (المذيبات B). بدء شطف التدرج في 20٪ من المذيبات ألف وزيادة تصل إلى 80٪ في 17 دقيقة. الحفاظ على هذه النسبة خلال 4 دقائق مع تدفق 400 ميكرولتر دقيقة -1 و درجة حرارة العمود وصينية عينة في 23 درجة مئوية و 4 درجات مئوية، على التوالي.
- استخدام طnstrument بالإضافة إلى الفصل الكروماتوغرافي للكشف عن الأيض، وelectrospray ساخنة (H-ESI الثاني) في كل من وسائط الإيجابية والسلبية كمصدر التأين، وكامل طريقة اكتساب المسح في كتلة حل السلطة من 70000 كاملة العرض نصف الحد الأقصى (FWHM) ( م / ض 200).
- استخدام النيتروجين (نقاء> 99.995٪) للغاز غمد والغاز مساعد بمعدل تدفق 45 و 10 او (وحدات التعسفي) وضبط درجة الحرارة المرذاذ في 350 ° C والجهد electrospray في 4 كيلو فولت في وضع إيجابي و-3.5 كيلو فولت في وضع سلبي.
تحليل 3. البيانات
- اختيار عدد من المركبات التي تتوفر المعايير والتي تظهر قمم نوعية جيدة في العينات. قمم نوعية جيدة لها إشارة إلى ارتفاع نسبة الضوضاء. ومن المهم أن تحقق الذروة الجودة والتأكد من عدم إدراج النظائر كاذبة. قمم النظائر التي تختلف في الشكل و / أو الوقت الاحتفاظ هي على الأرجح زائفة.
- للحصول على عينات المسمى النظير حساب isotopomer الشامل (MI) كسور بتقسيم منطقة ذروة كل MI مع مجموع المناطق الذروة من كل سوء.
- حساب المسمى كسور الكربون / النيتروجين، والإثراء من 13 C و 15 N، على التوالي، كما
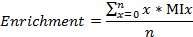
حيث n هو عدد ذرات الكربون (أو نيتروجين، على التوالي) في الأيض، ومزج هو جزء MI من س.
ملاحظة: يمكن تنفيذ جميع العمليات الحسابية باستخدام لغات البرمجة.
النتائج
وكمثال على ذلك، ونحن هنا وصف تجربة التحقيق في عملية التمثيل الغذائي للخلايا هيلا مرتبة وفقا لمرحلة دورة الخلية. لتسمية مجموعة واسعة من المركبات المركزية في كل من الكربون ونيتروجين، ونحن مثقف الخلايا لمدة 48 ساعة باستخدام U- 13 C-الجلوكوز وU- 13
Discussion
ويستند طريقتنا على مبدأ أن أجهزة الإنترنت النقالة في الأيض الخلوية تعكس "التاريخ" من الأنشطة الأيضية للخلية. هذا يجعل من الممكن للتحقيق في الأنشطة الأيضية في حيوانية من الخلايا، لأنها وقعت في المجتمع المعقدة من الخلايا، قبل الخلية إجراء الفرز. في المقابل، مناط?...
Disclosures
الكتاب ليس لديهم ما يكشف.
Acknowledgements
The authors would like to thank Dr. Anas Kamleh for valuable help with optimizing mass spectrometry methods, and Annika von Vollenhoven for assistance with cell sorting. This research was supported by the Swedish Foundation for Strategic Research (grant no. FFL12-0220) and the Strategic Programme in Cancer Research (IR, RN); the Swedish Heart-Lung Foundation (CEW, HG); and Mary Kay Foundation (JW, MJ).
Materials
| Name | Company | Catalog Number | Comments |
| HBSS | Sigma | H6648 | |
| INFLUX (inFlux v7 Sorter) | BD Biosciences | ||
| U-13C-Glucose | Cambridge isotopes | 40762-22-9 / GLC-018 | |
| U-13C,15N2-Glutamine | Cambridge isotopes | CNLM-1275-H-0.1 | |
| Methanol (JT Baker), HPLC grade | VWR | BAKR8402.2500 | |
| Ultrafree - MC - VV centrifugal Filters. Durapore PVDF 0.1 µm | Millipore | UFC30VV00 | |
| Ultimate 3,000 UHPLC | Thermo Fisher scientific | ||
| Q-Exactive Orbitrap Mass spectrometer | Thermo Fisher scientific | ||
| Merk-Sequant ZIC HILIC column (150 mm x 4.6 mm, 5 µm) | Merck KGaA | 1.50444.0001 | |
| Merk-Sequant ZIC HILIC guard column (20 mm x 2.1 mm) | Merck KGaA | ||
| Acetonitrile Optima LC-MS, amber glass | Fisher Scientific | A955-212 | |
| Milli-Q water | Millipore | Produced with a Milli-Q Gradient system | |
| Myrsyra 99.5% Optima (Formic acid) | Fisher Scientific | 11423423 | |
| X100 Screw Vial 1.5 ml, 8-425 32x11.6 mm, amber, 100 units | Thermo Fisher scientific | 10560053 | |
| X100 Lock Skruv Vitt PTFE Packing 8-425 (Screw caps) | Thermo Fisher scientific | 12458636 | |
| ProteoMass LTQ/FT-Hybrid ESI Pos. Mode Cal Mix | Sigma-Aldrich | MSCAL5 | Calibration kit |
| SNAKESKIN 10K MWCO | Thermo Fisher scientific | 88245 | |
| Mathematica v.10 | Wolfram Research |
References
- Gregersen, P. K. Cell type-specific eQTLs in the human immune system. Nat. Genet. 44 (5), 478-480 (2012).
- Heppner, G. H. Tumor heterogeneity. Cancer Res. 44 (6), 2259-2265 (1984).
- Prat, A., et al. Characterization of cell lines derived from breast cancers and normal mammary tissues for the study of the intrinsic molecular subtypes. Breast Cancer Res. Treat. 142 (2), 237-255 (2013).
- Magistretti, P. J., Allaman, I. A Cellular Perspective on Brain Energy Metabolism and Functional Imaging. Neuron. 86 (4), 883-901 (2015).
- Angelini, G., et al. Antigen-presenting dendritic cells provide the reducing extracellular microenvironment required for T lymphocyte activation. Proc. Natl. Acad. Sci. U. S. A. 99, 1491-1496 (2002).
- Koukourakis, M. I., Giatromanolaki, A., Harris, A. L., Sivridis, E. Comparison of metabolic pathways between cancer cells and stromal cells in colorectal carcinomas: A metabolic survival role for tumor-associated stroma. Cancer Res. 66 (2), 632-637 (2006).
- Hollenbaugh, J. A., Munger, J., Kim, B. Metabolite profiles of human immunodeficiency virus infected CD4+ T cells and macrophages using LC-MS/MS analysis. Virology. 415 (2), 153-159 (2011).
- Richardson, G. M., Lannigan, J., Macara, I. G. Does FACS perturb gene expression. Cytom. Part A. 87 (2), 166-175 (2015).
- Moussaieff, A., et al. High-resolution metabolic mapping of cell types in plant roots. Proc. Natl. Acad. Sci. U. S. A. 110 (13), E1232-E1241 (2013).
- Johnson, C., et al. A metabolic signature of colon cancer initiating cells. Cancer Metab. 2, 32 (2014).
- Roci, I., et al. Metabolite Profiling and Stable Isotope Tracing in Sorted Subpopulations of Mammalian Cells. Anal. Chem. 88 (5), 2707-2713 (2016).
- Shapiro, H. . Practical flow cytometry. , (2003).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Noack, S., Wiechert, W. Quantitative metabolomics: A phantom. Trends Biotechnol. 32 (5), 238-244 (2014).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved